Method Article
Nomogramme à risque compétitif pour prédire la survie spécifique au cancer chez plusieurs patients atteints de cancer colorectal primitif après une intervention chirurgicale
Dans cet article
Résumé
L’étude a révélé que le sexe masculin, le mauvais grade de la tumeur et le stade avancé de métastase du ganglion tumoral étaient associés à une survie spécifique au cancer (CSS) plus faible chez plusieurs patients atteints de cancer colorectal primitif (MPCC) après la chirurgie. Nous avons développé un nomogramme pour prédire le CSS des patients MPCC et contribuer à la prise de décision de traitement clinique.
Résumé
La survie spécifique au cancer (CSS) chez plusieurs patients atteints de cancer colorectal primitif (MPCC) est influencée de manière compétitive par les décès dus à d’autres causes. Cette étude visait à étudier le CSS et les facteurs de risque associés en comparant l’analyse des risques chez les patients MPCC. Les données de cette étude proviennent de la base de données SEER. En utilisant l’analyse univariée et multivariée du modèle de risque concurrentiel pour affaiblir l’impact des événements compétitifs, il explore les facteurs de risque du CSS et développe un modèle de nomogramme. Ensuite, les performances du modèle sont vérifiées par la courbe ROC, la courbe d’étalonnage et le DCA. L’étude porte sur un total de 8931 patients, dont 6255 dans la cohorte d’entraînement et 2676 dans la cohorte de validation. Des analyses univariées et multivariées ont montré que le sexe, le stade de métastase du ganglion tumoral (TNM) et le grade de la tumeur sont des facteurs de risque indépendants de survie spécifique au cancer chez les patients atteints de MPCC. Sur la base des facteurs de risque, nous avons développé un modèle de diagramme pour prédire le CSS. La courbe ROC, la courbe d’étalonnage et le DCA donnent également de bons résultats. En conclusion, un nomogramme est mis au point qui sert d’outil précieux pour prédire le CSS chez les patients MPCC, fournissant aux cliniciens des informations cruciales pour une planification personnalisée du traitement.
Introduction
Le cancer colorectal, l’une des tumeurs du système digestif les plus courantes dans le monde, a connu une augmentation continue de son incidence au cours des dernières décennies. La persistance des taux élevés d’incidence et de mortalité a attiré beaucoup d’attention. Selon les dernières statistiques, le cancer colorectal se classe au troisième rang des tumeurs malignes courantes dans le monde, son taux de mortalité se classant au deuxième rang mondial1. Le cancer colorectal primitif multiple (MPCC) est un sous-type particulier de cancer colorectal qui a suscité une attention croissante de la part des chercheurs ces dernières années. Il est défini comme le diagnostic de deux ou plusieurs lésions indépendantes du cancer colorectal chez le même patient, avec une distance de plus de 5 cm entre les bords des lésions. Dans le MPCC, lorsque plusieurs tumeurs sont diagnostiquées simultanément ou dans un délai de moins de 6 mois, il est défini comme un cancer colorectal synchrone (SCRC), tandis que si l’intervalle entre les diagnostics est supérieur à 6 mois, il est défini comme un cancer colorectal métachrone (MCRC)2,3,4,5.
La proportion de MPCC dans tous les cancers colorectaux est relativement faible, et les rapports sur son incidence varient selon les régions et les études. Des études récentes ont révélé que le CPPM représente entre 2 % et 10 % des cancers colorectaux 2,3,4,5. Comparé au cancer colorectal solitaire, le MPCC a un pronostic plus sombre 3,6. Actuellement, l’évaluation clinique du pronostic du MPCC repose principalement sur le système de stadification des métastases tumorales ganglionnaires (TNM) de l’American Joint Committee of Cancer, qui utilise le stade le plus avancé des lésions multiples comme stade réel du MPCC. Cependant, il ne suffit pas de baser les prédictions de pronostic uniquement sur la stadification TNM. Il n’existe toujours pas d’outil efficace pour prédire le pronostic post-opératoire du MPCC. À l’ère actuelle de la médecine de précision, les modèles de prédiction clinique permettant de quantifier le risque sont largement utilisés dans la prise de décision clinique et l’évaluation du pronostic du patient 7,8. Ces dernières années, les nomogrammes basés sur des facteurs de risque pronostiques indépendants ont été largement acceptés pour prédire le pronostic tumoral9. Les nomogrammes peuvent visualiser des modèles statistiques complexes, ce qui les rend plus faciles à appliquer dans des contextes cliniques. Dans l’évaluation du pronostic tumoral, la survie globale (SG) et la survie spécifique au cancer (CSS) sont des indicateurs de résultats couramment utilisés9. La SG fait référence au temps écoulé entre la confirmation d’un cancer chez un patient et le décès quelle qu’en soit la cause. Le CSS fait référence au temps écoulé entre le diagnostic d’une tumeur et le décès causé spécifiquement par le cancer, offrant un reflet plus précis du risque de décès dû au cancer. Lorsque la CSS est utilisée comme indicateur de résultat, les décès causés par d’autres facteurs peuvent affecter la probabilité de mortalité spécifique au cancer, introduisant une relation de risque concurrente entre les deux événements10,11. Par conséquent, les études de survie spécifique à la tumeur devraient utiliser des modèles de risque concurrents pour éliminer l’impact des événements concurrents. Des études antérieures ont construit des modèles pour prédire le pronostic du MPCC, mais ceux-ci se sont limités au cancer colorectal synchrone et n’ont pas utilisé de modèles de risque concurrents pour tenir compte de l’impact des événements concurrents sur les CSS 12,13,14.
Dans cette étude, nous avons exploré les facteurs de risque indépendants concurrents affectant la survie spécifique au cancer pour le MPCC après une chirurgie. La raison d’être de l’utilisation d’un modèle de risques concurrents découle de sa capacité à tenir compte de la possibilité que les patients meurent de causes autres que le cancer, ce qui est essentiel pour obtenir des estimations de survie non biaisées15,16. Les techniques traditionnelles d’analyse de survie, telles que le modèle de Cox, peuvent surestimer les probabilités de survie en présence d’événements concurrents, ce qui rend l’utilisation de modèles de risque concurrents plus appropriée dans ces scénarios17.
Sur la base des facteurs de risque indépendants identifiés, nous avons construit un nomogramme pour prédire la probabilité de survie et validé ses performances. Les nomogrammes ont gagné en popularité dans les milieux cliniques parce qu’ils fournissent une représentation graphique conviviale de modèles statistiques complexes, permettant aux cliniciens de calculer facilement des scores de risque individualisés18,19. Contrairement à d’autres outils prédictifs, les nomogrammes intègrent plusieurs facteurs de risque et fournissent des estimations de pronostic plus précises et personnalisées. Cette approche a été de plus en plus validée dans divers cancers, démontrant des performances supérieures à celles des systèmes de stadification traditionnels 20,21,22. Notre outil vise à aider les cliniciens à faire des estimations de pronostic personnalisées et plus précises pendant le diagnostic et le traitement, améliorant ainsi la prise de décision dans la prise en charge du MPCC.
Protocole
Cette étude comprend deux étapes. Tout d’abord, les données cliniques et de survie du MPCC ont été obtenues à partir de la base de données SEER. Ensuite, le logiciel R (version 4.3.3) a été utilisé pour analyser et construire un modèle de risque concurrent. Le schéma de déroulement de l’étude est présenté à la figure 1. Cette étude n’a pas besoin d’une approbation éthique et d’un consentement pour y participer. Les données utilisées dans cette étude ont été obtenues à partir de bases de données.
1. Acquisition de données
- Téléchargez le SEER. Logiciel Stat 8.4.3 du site Web de la base de données SEER (http://seer.cancer.gov/about/overview.html). Inscrivez-vous et connectez-vous à SEER. Stat 8.4.3 pour obtenir des données pertinentes sur les patients.
- Après s’être connecté à SEER. Stat 8.4.3, cliquez sur Case Listing Session > Data et sélectionnez la base de données Incidence SEER Research Data, 17 Registries, Nov 2022 Sub (2000-2020).
- Cliquez sur Sélection > Modifier et choisissez {Race, Sexe, Année Dx. Année du diagnostic} = '2004', '2005', '2006', '2007', '2008', '2009', '2010', '2011', '2012', '2013', '2014', '2015' ET {Site et morphologie. Recodage du site ICD-O-3/OMS 2008} = 'Côlon et rectum' ET {Site et morphologie. Confirmation diagnostique} = 'Histologie positive' ET {Plusieurs champs primaires. Numéro de séquence} ! = 'Un seul primaire'.
- Cliquez sur OK et enregistrez la sélection. Cliquez sur Tableau, et dans l’interface des variables disponibles, sélectionnez Recodage de l’âge avec âges simples et 85+, Sexe, Recodage du site ICD-O-3/WHO 2008, Taille de la tumeur CS, Recodage du grade (jusqu’en 2017), AJCC T dérivé, 6e édition (2004-2015), AJCC N, 6e édition (2004-2015), AJCC M dérivé, 6e édition (2004-2015), Recodage des radiations, Recodage de la chimiothérapie (oui, non/unk), Classification des décès spécifiques à la cause SEER, Classification SEER des autres causes de décès, mois de survie, ID du patient, RX Summ-Surg Prim Site (1998+), et cliquez sur Colonne.
- Cliquez sur Sortie, nommez les données, puis cliquez sur Exécuter pour afficher et enregistrer les données.
- La base de données SEER ne fournit pas directement d’informations sur la présence d’un patient MPCC. Après avoir terminé le téléchargement des données comme décrit ci-dessus, utilisez l’ID du patient pour filtrer les patients atteints de MPCC, c’est-à-dire ceux diagnostiqués avec deux occurrences ou plus. Après avoir identifié les patients MPCC, calculez l’intervalle entre les diagnostics de tumeur en fonction de leur temps de survie. En utilisant 6 mois comme seuil, classez les patients en SMPCC et MMPCC.
- En fonction de leur âge au moment du diagnostic initial, classez l’âge des patients comme = 65 ans et> 65 ans. Pour le grade tumoral, le grade I correspond à une bonne différenciation, le grade II correspond à une différenciation modérée, le grade III correspond à une faible différenciation et le grade IV correspond à l’indifférenciation. Classez l’emplacement de la tumeur en fonction de la distribution de plusieurs tumeurs comme côlon droit, colorectum gauche et colorectum entier.
- Il existe des différences évidentes dans le développement embryonnaire et les caractéristiques biologiques entre le colorectum proximal et distal, divisez l’emplacement de la tumeur en fonction de la flexion splénique23. Le côlon droit est défini comme proximal à la flexion splénique, y compris le cæcum, le côlon ascendant, la flexion hépatique et le côlon transverse, tandis que le colorectum gauche comprend la flexion splénique, le côlon descendant, le côlon sigmoïde, la jonction rectosigmoïde et le rectum. Si les tumeurs d’un patient sont entièrement situées dans le côlon droit, définissez l’emplacement de la tumeur comme étant le côlon droit ; Si toutes les tumeurs sont situées dans le colorectum gauche, définissez l’emplacement comme colorectum gauche ; S’il y a des tumeurs dans le côlon droit et le colorectum gauche, définissez l’emplacement comme étant le colorectum entier.
- Déterminez la taille de la tumeur en sélectionnant le plus grand diamètre de tumeur parmi plusieurs tumeurs chez le même patient. Sur la base du diamètre de la tumeur, catégorisez la taille comme = 5 cm ou >5 cm. Au total, 8 931 patients ont été inclus dans l’étude. Pour créer une cohorte d’entraînement et une cohorte de validation, divisez au hasard tous les cas dans un rapport de 7:3.
2. Construction du modèle
- Téléchargez les logiciels RStudio (2023.12.1+402) et R (4.3.3). Ouvrez RStudio pour exécuter le logiciel R. Cliquez sur Nouveau fichier et sélectionnez Script R pour créer une nouvelle interface de programmation R. Entrez le code approprié dans l’éditeur de code et cliquez sur Exécuter pour exécuter le code.
REMARQUE : Le langage R et ses fonctions offrent une grande variété de paramètres. L’ajout ou la modification de ces paramètres peut améliorer l’analyse et la visualisation des données. - Utilisez le code suivant pour effectuer une analyse univariée et tracer la courbe CIF.
Bibliothèque(tidycmprsk)
Bibliothèque(gtsummary)
Bibliothèque(ggsurvfit)
Bibliothèque(ggprism)
données <-read.csv('data.csv')"
data$status<-factor(data$status, levels=c(0,1,2),labels=c(« 0 »,"CSS »,"OCS »))
CIF <- tidycmprsk ::cuminc(Surv(temps, statut) ~ Sexe, données = données)
ggcuminc(CIF,outcome= c(« CSS », « OCS »),size=1.5)
où data.csv sont des données obtenues à partir de la base de données SEER. - Après avoir exécuté le code ci-dessus, cliquez sur Exporter, puis sur Enregistrer en tant qu’image, et enfin sur Enregistrer pour enregistrer l’image. La méthode d’enregistrement des images suivantes sera la même que dans cette étape. Remplacez le sexe dans le code ci-dessus un par un par d’autres facteurs pour effectuer une analyse univariée de tous les facteurs.
- Utilisez le code suivant pour effectuer une analyse et une visualisation BSR et multivariées.
Bibliothèque (Leaps)
bibliothèque(risqueRégression)
Bibliothèque(prodlim)
Bibliothèque(Parcelle forestière)
leaps<-regsubsets(status==0~Sexe+Taille+Grade+T+N+M,données = données)
plot(leaps,scale="adjr2 »)
multi <- FGR(Hist(temps,statut)~Sexe + T + N + M +Grade+ Taille,cause=1,données=données)
Résumé(Multi)
Multi <- read.csv(« multi.csv »,en-tête = T)
forestplot(labeltext=as.matrix(Multi [,1:4]), mean= Multi $HR_mean, ower= Multi $HR_1, upper= Multi $HR_2)
où les données de multi.csv proviennent des résultats du code précédent. - Suivez l’étape 2.3. pour enregistrer l’image.
- Utilisez le code suivant pour tracer le nomogramme, la courbe ROC, la courbe d’étalonnage et la courbe DCA.
library(QHScrnomo)
Bibliothèque (RMS)
bibliothèque(risqueRégression)
Bibliothèque(prodlim)
Données <- read.csv (« data.csv »)
d <- datadist(data)
options(datadist = « d »)
e <-cph(Surv(temps,statut==1)~Sexe + T + N + M +Grade,données = données,
x=T, y=T, surv=T,time.inc=60)
nomo <- crr.fit(e,failcode=1,cencode = 0)
nomogram.crr(fit =nomo, lp = F, xfrac = 0.5, fun.at =seq(from=0, to=1, by= 0.1) , failtime =c(12,36,60),funlabel = c(« Incidence cumulative CSS 1 an », « Incidence cumulative CSS 3 ans »,"Incidence cumulative CSS 5 ans »))
set.seed(123)
data$pro <- tenf.crr(m3,temps = 60)
groupci(x=data$pro, ftime = data$time, fstatus = data$status, failcode = 1, cencode = 0, ci = TRUE)
f <- CSC(Hist(temps,statut)~Sexe + T + N + M +Note,données = données)
x <- Score(list(model1=f), Hist(time,status)~1, data=data, cause=1, times=c(12,36,60), se.fit=1L, plots="roc », metrics="auc »)
g <- as.data.frame(x$AUC$score)
h <- g[g$times %in % c(12,36,60),]
col = c(« darkcyan »,"tomate »,"violet »)
plotROC(x, xlab="1-Spécificité », ylab="Sensibilité »,col=col[1], cex=1.5, legend=" », auc.in.legend = F, times = 12)
plotROC(x,col=col[2],legend = '', cex=1.5,times =36,auc.in.legend = F,add=T)
plotROC(x,col=col[3], times =60, add=T, cex=1.5, legend = '',auc.in.legend = F)
jambe <- paste0(c(« 1 an : « ,"3 ans : « ,"5 ans : « ),substr(a$AUC,1,5)) - Suivez l’étape 2.3. pour enregistrer l’image.
- Utilisez le package ggscidca pour tracer la courbe DCA.
Bibliothèque(ggscidca)
df_surv<-read.csv(« data.csv »,en-tête = T)
cox_model <- coxph(Surv(temps, statut==1)~Sexe + T + N + M +Grade, données = df_surv)
cox_model1<-newcrr(cox_model)
scidca(cox_model1, newdata= df_surv,threshold.line = T,threshold.text = T)
où data.csv sont des données obtenues à partir de la base de données SEER. - Suivez l’étape 2.3. pour enregistrer l’image.
Résultats
Caractéristiques du patient
Au total, 8 931 patients ont été inclus dans l’étude. En termes de répartition par sexe, il y avait une proportion plus élevée de patients masculins (56 %) que de patientes (44 %). En ce qui concerne la distribution de l’emplacement des tumeurs, la majorité des tumeurs étaient réparties dans le colorectum total, tandis que le moins de tumeurs étaient situées dans le côlon droit (21 %). En termes de grade tumoral, le grade le plus courant était le grade II, avec 6 251 patients, représentant 70 % du total ; il était suivi par le grade III, avec 2 026 patients (23 %). Les grades I et IV comptaient respectivement 355 et 299 patients, soit 4 % et 3,3 % du total. En termes d’invasion tumorale, les stades T3 et T4 étaient les plus courants. Plus de la moitié des patients n’avaient pas de métastases ganglionnaires, tandis que 17 % des patients ont présenté des métastases à distance. La radiothérapie a été reçue par 1 446 patients (16 %) et la chimiothérapie a été administrée à 3 843 patients (43 %). Le SCRC était plus fréquent, avec 5 327 patients représentant 60 % du total ; Il y avait 3 604 patients atteints d’un cancer colorectal métachrone, soit 40 %. Les patients ont été divisés au hasard en ensembles d’entraînement et de validation dans un rapport de 7:3. Il n’y avait pas de différences statistiquement significatives dans les données de référence entre les cohortes de formation et de validation, comme le montre le tableau 1.
Analyse univariée
Après avoir contrôlé l’influence des événements compétitifs, les résultats de l’analyse univariée ont montré que le sexe, le grade et la taille de la tumeur, le stade TNM, la radiothérapie, la chimiothérapie, le statut synchrone ou métachrone et la localisation de la tumeur étaient les facteurs pronostiques affectant le CSS chez les patients MPCC. Seul l’âge n’est pas un facteur pronostique du CSS chez les patients atteints de MPCC. Nous notons qu’il existe une intersection significative de la CIF dans la radiothérapie, la chimiothérapie, la localisation et le statut synchrone ou métachrone, ce qui indique que les effets pronostiques à court et à long terme de la radiothérapie sur les patients MPCC étaient différents, tout comme la chimiothérapie, la localisation et le statut synchrone ou métachrone. La courbe de risque cumulatif de chaque sous-groupe est illustrée à la figure 2.
Analyse multivariée
Les facteurs pronostiques obtenus par l’analyse univariable ont été incorporés dans la régression des meilleurs sous-ensembles (BSR) et l’analyse multivariée du modèle de risque concurrentiel. Parmi eux, la radiothérapie, la chimiothérapie, la localisation et le statut synchrone ou métachrone sont exclus en raison de leur double effet sur le pronostic. Les résultats de la BSR et de l’analyse multivariée ont tous deux montré que le sexe, le stade TNM et le grade de la tumeur étaient des facteurs de risque indépendants de CSS chez les patients MPCC. (Graphique 2)
Construction et vérification de Nomogram
Sur la base des facteurs de risque indépendants obtenus par l’analyse multivariée, nous construisons un nomogramme linéaire pour prédire le CSS et vérifier les performances du modèle de prédiction (Figure 3). Ensuite, nous utilisons la courbe ROC, la courbe d’étalonnage et le DCA pour évaluer le modèle. La courbe ROC a montré que les AUC de 1 an, 3 ans et 5 ans de la cohorte de formation étaient de 0,762, 0,742 et 0,734, et que les AUC de 1 an, 3 ans et 5 ans de la cohorte de vérification étaient de 0,801, 0,740 et 0,743. Dans la cohorte d’entraînement et la cohorte de vérification, la courbe de calage a montré une forte concordance entre la probabilité projetée et les données réelles (figure 4). Afin de vérifier les performances du modèle en application clinique, nous avons utilisé le DCA pour évaluer la valeur clinique du modèle. Les résultats montrent que le modèle montre un bon bénéfice net (figure 5).
Figure 1 : Schéma de déroulement de l’étude. Cette étude se déroule en deux étapes : tout d’abord, les données ont été obtenues à l’aide du SEER. Les statistiques, puis l’analyse et la visualisation des données ont été effectuées à l’aide de R. Veuillez cliquer ici pour voir une version agrandie de cette figure.
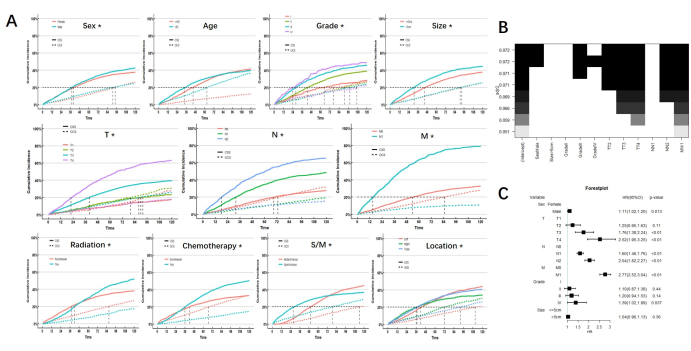
Figure 2 : Résultats de l’analyse. (A) CIF des sous-groupes. * indique p <0,05. (B) Régression des meilleurs sous-ensembles. Dans la meilleure qualité de l’ajustement, le sexe, le grade et le stade TNM ont été pris en compte pour l’inclusion (indiqués par des blocs noirs en haut). (C) L’analyse multivariée a également montré que le sexe, le grade et le stade TNM sont des facteurs de risque indépendants. Veuillez cliquer ici pour voir une version agrandie de cette figure.
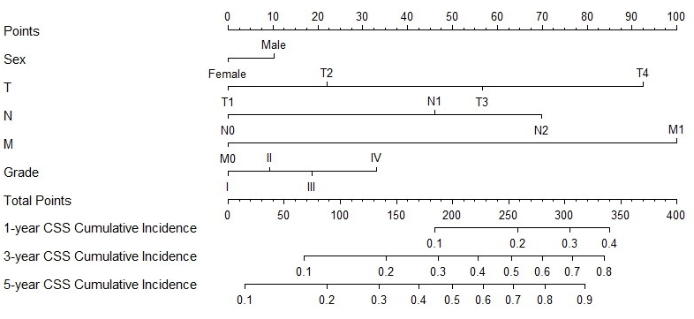
Figure 3 : Nomogramme pour le CSS chez les patients MPCC. Le score total du patient peut être calculé en additionnant les scores correspondant à chaque facteur. Sur la base du score total, la probabilité de décès spécifique au cancer à 1, 3 et 5 ans peut être prédite Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
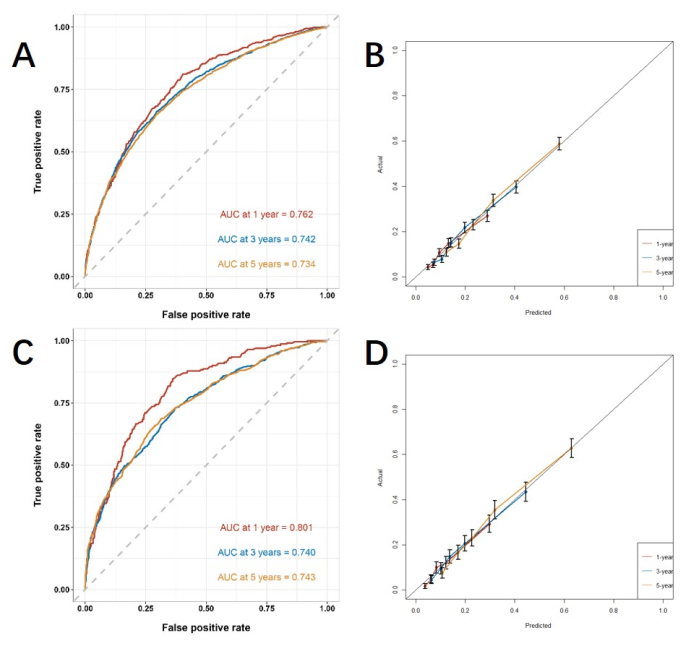
Figure 4 : Courbe ROC et courbe d’étalonnage. (A, B) Les courbes de la cohorte d’entraînement (n=6255) et (C, D) de la cohorte de validation (n=2676). Plus la valeur AUC est proche de 1, meilleures sont les performances de classification du modèle. Les barres d’erreur indiquent l’intervalle de confiance à 95 % pour la probabilité que l’événement réel se produise. Veuillez cliquer ici pour voir une version agrandie de cette figure.
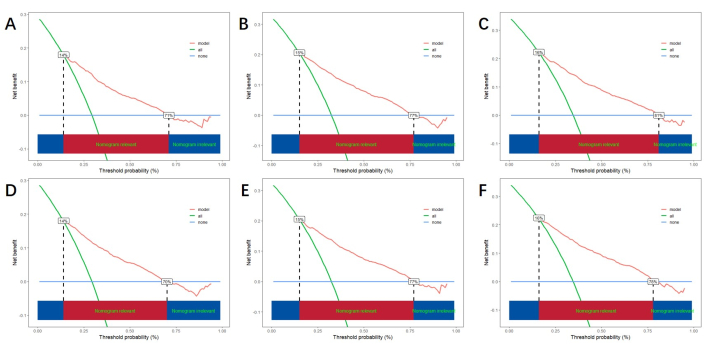
Figure 5 : ACD à 1 an, 3 ans et 5 ans. (A, B, C) ACD dans la cohorte de formation et (D, E, F) cohorte de validation. La ligne verte représente l’avantage net de tous les points positifs, la ligne bleue représente l’avantage net de tous les points positifs et la ligne rouge indique l’avantage net du modèle. La zone rouge ci-dessous représente l’avantage du modèle supérieur à celui de tous les avantages positifs et aucun positif, indiquant la fourchette réelle des avantages du modèle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Caractéristiques clinicopathologiques et initiales des patients. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
En tant que tumeur courante du système digestif, les facteurs influençant le pronostic du cancer colorectal solitaire ont été étudiés et confirmés dans des recherches antérieures24. Cependant, il y a eu peu de recherches sur les facteurs pronostiques pour les patients atteints de MPCC. Cette étude a porté sur 8 931 patients atteints de MPCC qui ont subi une intervention chirurgicale à partir de la base de données SEER entre 2004 et 2015. Nous avons utilisé un modèle de risque concurrent pour étudier les facteurs de risque affectant le CSS et avons construit un modèle prédictif. Dans cette étude, 39,1 % des patients décédés sont décédés de causes autres que le cancer, ce qui souligne la nécessité d’utiliser un modèle de risque concurrent pour analyser les facteurs de risque de CSS.
Des études antérieures ont montré que l’âge avancé est un facteur de risque affectant la SG des patients MPCC12. Cependant, dans cette étude, nous avons constaté que l’âge n’était pas un facteur de risque de CSS. Les patients âgés présentent souvent des problèmes de santé de base moins bons et plus de comorbidités, ce qui peut entraîner une SG plus courte par rapport aux patients plus jeunes. Les personnes âgées sont également plus susceptibles de mourir d’autres causes, telles que des événements cardiovasculaires et des infections graves, ce qui les rend plus sensibles à ces facteurs que les patients plus jeunes. Dans cette étude, nous nous sommes concentrés sur la relation concurrente entre les décès dus à d’autres causes et les décès dus au cancer lui-même. En utilisant un modèle de risque concurrent et en excluant l’interférence des décès causés par d’autres raisons, nous avons constaté que dans ces conditions, l’âge avancé n’était plus un facteur de risque pour le CSS du MPCC. Cela indique que les patients MPCC de différents groupes d’âge peuvent être confrontés à des charges tumorales similaires. Dans l’ensemble, les patients jeunes et âgés atteints de cette maladie ont un SCS similaire, ce qui fournit des conseils précieux pour l’élaboration de stratégies de traitement personnalisées.
Cette étude a également révélé que l’incidence et le pronostic du MPCC présentent des tendances liées au sexe, avec une proportion plus élevée et un CSS plus faible chez les patients masculins. Ceci est cohérent avec la situation observée dans le cancer colorectal solitaire. Des études antérieures ont montré que le cancer colorectal solitaire est plus fréquent chez les hommes et que les patients masculins ont un pronostic plus sombre que les patients féminins. Cela peut être dû à l’influence de l’œstrogène sur l’apparition et la progression du cancer colorectal25. De plus, d’autres recherches ont suggéré que le microbiote intestinal et les métabolites intestinaux chez les patients masculins pourraient être l’une des raisons des différences entre les sexes observées chez les patients atteints de cancer colorectal26. Une étude menée en Allemagne a révélé que le diabète de type 2 a un impact plus important sur le cancer colorectal chez les femmesde 27 ans. De plus, la vitamine D a un effet protecteur contre la néoplasie colorectale chez les femmes, mais aucun résultat similaire n’a été trouvé chez les hommes28.
Une analyse univariée concurrente a révélé que les patients MPCC avec des tumeurs de plus grande taille avaient un CSS plus faible. Dans des études antérieures, la taille de la tumeur a souvent été considérée comme un indicateur de l’agressivité tumorale 29,30,31. Cependant, les résultats d’analyses multivariées concurrentes n’ont pas soutenu la taille de la tumeur en tant que facteur de risque indépendant. Cela suggère que dans les organes creux comme le colorectum, la taille de la tumeur peut avoir une capacité limitée à refléter l’agressivité de la tumeur. Ce phénomène peut provenir de divers facteurs, notamment la complexité de la biologie tumorale et les différents modèles de croissance tumorale et de propagation dans différents organes.
Un mauvais grade tumoral indique des capacités invasives et migratoires plus fortes des cellules tumorales, ce qui s’aligne sur les conclusions de cette étude : un grade tumoral moins bon suggère un CSS plus faible pour les patients MPCC. Le stade TNM est la méthode clinique la plus couramment utilisée pour guider le traitement des patients et prédire le pronostic32,33. Cette étude a révélé que plus l’infiltration tumorale est profonde, plus le nombre de ganglions lymphatiques métastatiques et la présence de métastases d’organes sont importants, plus la survie spécifique au cancer du patient est mauvaise. Ceci est tout à fait conforme au consensus clinique.
Dans le cancer colorectal, le pronostic varie en fonction de la localisation de la tumeur. De nombreuses études ont montré que le côlon droit a un pronostic plus sombre par rapport au colorectum gauche 34,35,36,37. Cependant, certaines recherches suggèrent que dans le cancer colorectal résécable, le côté de l’emplacement de la tumeur n’a pas d’impact sur le pronostic à long terme38. Dans cette étude, le MPCC situé dans le côlon droit avait un CSS plus mauvais à court terme, mais un meilleur pronostic à long terme. De même, les patients MPCC qui ont reçu une radiothérapie et une chimiothérapie avaient un meilleur CSS à court terme, mais un CSS à long terme moins bon. Le SCRC avait un CSS à court terme moins bon que le MCRC, mais un meilleur CSS à long terme. Les raisons de ce double effet sur le pronostic ne sont pas encore claires.
Cette étude présente certaines limites. En raison des contraintes inhérentes à la base de données SEER, nous n’avons pas pu obtenir ou analyser certains indicateurs pronostiques connus, tels que les niveaux d’antigène carcinoembryonnaire (ACE) et l’état des microsatellites. De plus, nous n’avons pas pu exclure les patients atteints d’une maladie inflammatoire de l’intestin, d’un cancer colorectal héréditaire sans polypose et d’une polypose adénomateuse familiale qui présentent des risques plus élevés de MPCC39. De plus, l’immunothérapie est devenue un aspect de plus en plus important du traitement du cancer colorectal, mais nous n’avons pas été en mesure d’obtenir des données à ce sujet. La nature rétrospective de notre analyse et le fait de s’appuyer sur un seul ensemble de données peuvent introduire des biais inhérents.
L’étude basée sur la base de données SEER a révélé que le sexe masculin, le mauvais grade de la tumeur et le stade TNM avancé étaient associés à un CSS plus faible chez les patients MPCC après la chirurgie. Il est important de porter une attention particulière aux patients présentant ces facteurs de risque lors du diagnostic et du traitement. De plus, cette étude a développé un nomogramme pour prédire le CSS des patients MPCC, ce qui peut prédire avec précision le pronostic et contribuer à la prise de décision clinique en matière de traitement.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Aucun.
matériels
| Name | Company | Catalog Number | Comments |
| forestplot package | Comprehensive R Archive Network (CRAN) | forest plot 3.1.3 | A forest plot that allows for multiple confidence intervals per row, custom fonts for each text element, custom confidence intervals, text mixed with expressions, and more. |
| ggprism package | Comprehensive R Archive Network (CRAN) | ggprism 1.0.5 | The ggprism package provides various themes, palettes, and other useful functions to customise ggplots and give them the ‘GraphPad Prism’ look. |
| ggscidca package | Comprehensive R Archive Network (CRAN) | ggscidca package | The 'ggscidca' package adds coloured bars of discriminant relevance to the traditional decision curve. Improved practicality and aesthetics. |
| ggsurvfit package | Comprehensive R Archive Network (CRAN) | ggsurvfit 1.1.0 | The ggsurvfit package eases the creation of time-to-event (aka survival) summary figures with ggplot2. The concise and modular code creates images that are ready for publication or sharing. |
| gtsummary package | Comprehensive R Archive Network (CRAN) | gtsummary 2.0.0 | The gtsummary package provides an elegant and flexible way to create publication-ready analytical and summary tables using the R programming language. The {gtsummary} package summarizes data sets, regression models, and more, using sensible defaults with highly customizable capabilities. |
| QHScrnomo package | Comprehensive R Archive Network (CRAN) | QHScrnomo 3.0.1 | The goal of QHScrnomo is to provide functionality to construct nomograms in the context of time-to-event (survival) analysis in the presence of competing risks. It also contains functions to build, validate, and summarize these models. |
| R Software | R Core Team | R 4.3.3 | Free software environment for statistical computing and graphics |
| riskRegression package | Comprehensive R Archive Network (CRAN) | riskRegression 1.3.7 | Risk Regression Models and Prediction Scores for Survival Analysis with Competing Risks |
| rms package | Comprehensive R Archive Network (CRAN) | rms 6.8-1 | rms does regression modeling, testing, estimation, validation, graphics, prediction, and typesetting by storing enhanced model design attributes in the fit. |
| RStudio | RStudio, Public Benefit Corporation(PBC) | Rstudio 2023.12.1+402 | Integrated Development Environment (IDE) used for running R scripts, data analysis, and model development. Provides a user-friendly interface for R programming with advanced features like script editing, debugging, and version control. |
| SEERstat | National Cancer Institute (NCI) | SEERstat 8.4.3 | Software for statistical analysis of SEER and other cancer-related databases |
| tidycmprsk package | Comprehensive R Archive Network (CRAN) | tidycmprsk 1.0.0 | The tidycmprsk package provides an intuitive interface for working with the competing risk endpoints |
Références
- Bray, F., et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 74 (3), 229-263 (2024).
- Derwinger, K., Gustavsson, B. A study of aspects on gender and prognosis in synchronous colorectal cancer. Clin Med Insights Oncol. 5, 259-264 (2011).
- Chin, C. C., Kuo, Y. H., Chiang, J. M. Synchronous colorectal carcinoma: predisposing factors and characteristics. Colorectal Dis. 21 (4), 432-440 (2019).
- Nikoloudis, N., et al. Synchronous colorectal cancer. Tech Coloproctol. 8 (Suppl 1), s177-s179 (2004).
- Huang, C. S., et al. Synchronous and metachronous colorectal cancers: Distinct disease entities or different disease courses. Hepatogastroenterology. 62 (140), 838-842 (2015).
- He, W., et al. Prognosis of synchronous colorectal carcinoma compared to solitary colorectal carcinoma: a matched pair analysis. Eur J Gastroenterol Hepatol. 31 (12), 1489-1495 (2019).
- Ranstam, J., Cook, J. A., Collins, G. S. Clinical prediction models. Br J Surg. 103 (13), 1886 (2016).
- Smith, T., et al. Comparison of prognostic models to predict the occurrence of colorectal cancer in asymptomatic individuals: a systematic literature review and external validation in the EPIC and UK Biobank prospective cohort studies. Gut. 68 (4), 672-683 (2019).
- Iasonos, A., Schrag, D., Raj, G. V., Panageas, K. S. How to build and interpret a nomogram for cancer prognosis. J Clin Oncol. 26 (8), 1364-1370 (2008).
- Conroy, T., et al. Five-year outcomes of FOLFIRINOX vs Gemcitabine as adjuvant therapy for pancreatic cancer: A randomized clinical trial. JAMA Oncol. 8 (11), 1571-1578 (2022).
- Zhong, M., et al. Impact of lung metastasis versus metastasis of bone, brain, or liver on overall survival and thyroid cancer-specific survival of thyroid cancer patients: A population-based study. Cancers. 14 (13), 3133 (2022).
- Xu, Y., Wang, X., Huang, Y., Ye, D., Chi, P. A LASSO-based survival prediction model for patients with synchronous colorectal carcinomas based on SEER. Transl Cancer Res. 11 (8), 2795-2809 (2022).
- Zhang, X., Zhao, L., Hu, Y., Deng, K., Ren, W. A novel risk prediction nomogram for early death in patients with resected synchronous multiple primary colorectal cancer based on the SEER database. Int J Colorectal Dis. 38 (1), 130 (2023).
- Zhang, X., et al. Developing prognostic nomograms to predict overall survival and cancer-specific survival in synchronous multiple primary colorectal cancer based on the SEER database. J Cancer Res Clin Oncol. 149 (15), 14057-14070 (2023).
- Austin, P. C., Lee, D. S., Fine, J. P. Introduction to the analysis of survival data in the presence of competing risks. Circulation. 133 (6), 601-609 (2016).
- Lau, B., Cole, S. R., Gange, S. J. Competing risk regression models for epidemiologic data. Am J Epidemiol. 170 (2), 244-256 (2009).
- Nolan, E. K., Chen, H. Y. A comparison of the Cox model to the Fine-Gray model for survival analyses of re-fracture rates. Arch Osteoporos. 15 (1), 86 (2020).
- Park, S. Y. Nomogram: An analogue tool to deliver digital knowledge. J Thorac Cardiovasc Surg. 155 (4), 1793 (2018).
- Balachandran, V. P., Gonen, M., Smith, J. J., DeMatteo, R. P. Nomograms in oncology: more than meets the eye. Lancet Oncol. 16 (4), e173-e180 (2015).
- Liang, W., et al. Development and validation of a nomogram for predicting survival in patients with resected non-small-cell lung cancer. J Clin Oncol. 33 (8), 861-869 (2015).
- Chen, D., et al. Predicting postoperative peritoneal metastasis in gastric cancer with serosal invasion using a collagen nomogram. Nat Commun. 12 (1), 179 (2021).
- Niu, X., et al. A prognostic nomogram for patients with newly diagnosed adult thalamic glioma in a surgical cohort. Neuro Oncol. 23 (2), 337-338 (2021).
- Luo, S., et al. Comparison of left- and right-sided colorectal cancer to explore prognostic signatures related to pyroptosis. Heliyon. 10 (7), e28091 (2024).
- Keum, N., Giovannucci, E. Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies. Nat Rev Gastroenterol Hepatol. 16 (12), 713-732 (2019).
- Foster, P. A. Oestrogen and colorectal cancer: mechanisms and controversies. Int J Colorectal Dis. 28 (6), 737-749 (2013).
- Wang, L., et al. Male-biased gut microbiome and metabolites aggravate colorectal cancer development. Adv Sci. 10 (25), e2206238 (2023).
- Krämer, H. U., et al. Type 2 diabetes mellitus and gender-specific risk for colorectal neoplasia. Eur J Epidemiol. 27 (5), 341-347 (2012).
- Aigner, E., et al. Gender- and site-specific differences of colorectal neoplasia relate to vitamin D. Aliment Pharmacol Ther. 40 (11-12), 1341-1348 (2014).
- Dai, W., et al. Does tumor size have its prognostic role in colorectal cancer? Re-evaluating its value in colorectal adenocarcinoma with different macroscopic growth pattern. Int J Surg. 45, 105-112 (2017).
- Rössler, O., et al. Tumor size, tumor location, and antitumor inflammatory response are associated with lymph node size in colorectal cancer patients. Mod Pathol. 30 (6), 897-904 (2017).
- Zhang, Q., et al. Prognostic impact of tumor size on patients with metastatic colorectal cancer: a large SEER-based retrospective cohort study. Updates Surg. 75 (5), 1135-1147 (2023).
- Greene, F. L. TNM: our language of cancer. CA Cancer J Clin. 54 (3), 129-130 (2004).
- Ueno, H., et al. Optimal colorectal cancer staging criteria in TNM classification. J Clin Oncol. 30 (13), 1519-1526 (2012).
- Yahagi, M., Okabayashi, K., Hasegawa, H., Tsuruta, M., Kitagawa, Y. The worse prognosis of right-sided compared with left-sided colon cancers: a systematic review and meta-analysis. J Gastrointest Surg. 20 (3), 648-655 (2016).
- Holch, J. W., Ricard, I., Stintzing, S., Modest, D. P., Heinemann, V. The relevance of primary tumour location in patients with metastatic colorectal cancer: A meta-analysis of first-line clinical trials. Eur J Cancer. 70, 87-98 (2017).
- Petrelli, F., et al. Prognostic survival associated with left-sided vs right-sided colon cancer: A systematic review and meta-analysis. JAMA Oncol. 3 (2), 211-219 (2017).
- Zheng, C., Jiang, F., Lin, H., Li, S. Clinical characteristics and prognosis of different primary tumor location in colorectal cancer: a population-based cohort study. Clin Transl Oncol. 21 (11), 1524-1531 (2019).
- Karim, S., Brennan, K., Nanji, S., Berry, S. R., Booth, C. M. Association between prognosis and tumor laterality in early-stage colon cancer. JAMA Oncol. 3 (10), 1386-1392 (2017).
- Lindberg, L. J., et al. Risk of multiple colorectal cancer development depends on age and subgroup in individuals with hereditary predisposition. Fam Cancer. 18 (2), 183-191 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon