Method Article
수술 후 여러 원발성 대장암 환자의 암 특이적 생존을 예측하기 위한 경쟁 위험 노모그램
요약
이 연구는 남성의 성별, 낮은 종양 등급 및 진행된 종양 노드 전이 단계가 수술 후 다발성 원발성 결장직장암(MPCC) 환자의 암 특이적 생존율 저하와 관련이 있음을 발견했습니다. MPCC 환자의 CSS를 예측하고 임상적 치료 의사결정에 기여하기 위해 노모그램을 개발했습니다.
초록
다발성 원발성 대장암(MPCC) 환자의 암 특이적 생존율(CSS)은 다른 원인으로 인한 사망에 의해 경쟁적으로 영향을 받습니다. 본 연구는 MPCC 환자를 대상으로 경쟁적인 위험분석을 통해 CSS 및 관련 위험인자를 조사하는 것을 목적으로 하였다. 이 연구의 데이터는 SEER 데이터베이스에서 가져온 것입니다. 경쟁 리스크 모델의 일변수 및 다변수 분석을 사용하여 경쟁 이벤트의 영향을 약화시키고 CSS의 위험 요인을 탐색하고 노모그램 모델을 개발합니다. 그런 다음 ROC 곡선, 보정 곡선 및 DCA로 모델의 성능을 검증합니다. 이 연구는 총 8931명의 환자를 대상으로 하며, 이 중 6255명은 훈련 코호트에, 2676명은 검증 코호트에 속합니다. 단변수 및 다변수 분석은 성별, 종양 노드 전이(TNM) 단계 및 종양 등급이 MPCC 환자의 암 특이적 생존에 대한 독립적인 위험 요소임을 보여주었습니다. 위험 요인을 기반으로 CSS를 예측할 수 있는 다이어그램 모델을 개발했습니다. ROC 곡선, 검량선 및 DCA도 좋은 결과를 보여줍니다. 결론적으로, MPCC 환자의 CSS를 예측하는 데 유용한 도구 역할을 하는 노모그램이 개발되어 임상의에게 개인화된 치료 계획을 위한 중요한 통찰력을 제공합니다.
서문
대장암은 전 세계적으로 가장 흔한 소화기 계통 종양 중 하나로, 지난 수십 년 동안 발병률이 지속적으로 증가하고 있습니다. 지속적으로 높은 발병률과 사망률이 주목을 받고 있습니다. 최신 통계에 따르면 대장암은 전 세계적으로 흔한 악성 종양 중 3위를 차지하고 있으며, 사망률은 전 세계적으로 2위를 차지하고 있습니다1. 다발성 원발성 결장직장암(MPCC)은 최근 몇 년 동안 연구자들의 관심을 받고 있는 대장암의 특별한 하위 유형입니다. 이는 동일한 환자에서 두 개 이상의 독립적인 결장직장암 병변을 진단하는 것으로 정의되며, 병변 가장자리 사이의 거리가 5cm 이상인 것으로 정의됩니다. MPCC에서는 여러 종양이 동시에 진단되거나 6개월 이내에 진단되면 동기성 결장직장암(SCRC)으로 정의하고, 진단 간격이 6개월 이상이면 메타크로미스 결장직장암(MCRC)으로 정의합니다2,3,4,5.
모든 결장직장암에서 MPCC의 비율은 상대적으로 낮으며, 발병률에 대한 보고는 지역과 연구에 따라 다릅니다. 최근 연구에 따르면 MPCC는 대장암의 2%-10%를 차지한다고 보고되었습니다 2,3,4,5. 단독성 결장직장암에 비해 MPCC는 예후가 더 나쁘다 3,6. 현재 MPCC 예후의 임상적 평가는 주로 미국암공동위원회(American Joint Committee of Cancer)의 종양림프절 전이(TNM) 병기 결정 시스템에 의존하고 있으며, 이 시스템은 다발성 병변의 가장 진행된 단계를 MPCC의 실제 단계로 사용합니다. 그러나 TNM 병기에만 근거하여 예후 예측을 하는 것은 충분하지 않습니다. MPCC의 수술 후 예후를 예측할 수 있는 효과적인 도구는 아직 부족합니다. 현재 정밀 의학 시대에 위험을 정량화하기 위한 임상 예측 모델은 임상 의사 결정 및 환자 예후 평가에 널리 사용됩니다 7,8. 최근 몇 년 동안 독립적인 예후 위험 인자에 기초한 노모그램(nomogram)이 종양 예후를 예측하는 데 널리 받아들여지고 있다9. 노모그램은 복잡한 통계 모델을 시각화할 수 있어 임상 환경에서 더 쉽게 적용할 수 있습니다. 종양 예후를 평가할 때 전체 생존(full survival, OS)과 암 특이적 생존(cancer-specific survival, CSS)이 일반적으로 사용되는 결과 지표이다9. OS는 환자의 암 확진부터 모든 원인으로 인한 사망까지의 시간을 나타냅니다. CSS는 종양 진단부터 암으로 인한 사망까지의 시간을 말하며, 암으로 인한 사망 위험을 보다 정확하게 반영합니다. CSS를 결과 지표로 사용하면 다른 요인으로 인한 사망이 암 특이적 사망 확률에 영향을 미칠 수 있으며, 이로 인해 두 사건 사이에 경쟁적인 위험 관계가 형성될 수 있습니다10,11. 따라서 종양 특이적 생존에 대한 연구는 경쟁 사건의 영향을 제거하기 위해 경쟁 위험 모델을 사용해야 합니다. 이전 연구에서는 MPCC의 예후를 예측하기 위한 모델을 구축했지만, 이는 동기성 결장직장암에 국한되어 있었고 CSS 12,13,14에 대한 경쟁 사건의 영향을 설명하기 위해 경쟁 위험 모델을 활용하지 않았습니다.
이 연구에서는 MPCC의 수술 후 암 특이적 생존에 영향을 미치는 경쟁적인 독립 위험 요인을 조사했습니다. 경쟁 위험 모델을 사용하는 이유는 환자가 암 이외의 원인으로 사망할 수 있는 가능성을 설명할 수 있는 능력에서 비롯되며, 이는 편향되지 않은 생존 추정치를 얻는 데 매우 중요합니다15,16. 콕스(Cox) 모델과 같은 전통적인 생존 분석 기법은 경쟁 사건이 존재할 때 생존 확률을 과대 추정할 수 있으며, 이로 인해 이러한 시나리오에서 경쟁 위험 모형을 사용하는 것이 더 적절할 수 있다17.
식별된 독립적인 위험 인자를 기반으로 생존 확률을 예측하기 위한 노모그램을 구축하고 그 성능을 검증했습니다. 노모그램은 복잡한 통계 모델을 사용자 친화적인 그래픽 표현으로 제공하여 임상의가 개별화된 위험 점수를 쉽게 계산할 수 있도록 하기 때문에 임상 환경에서 인기를 얻고 있습니다18,19. 다른 예측 도구와 달리 노모그램은 여러 위험 요인을 통합하고 보다 정확하고 개인화된 예후 추정치를 제공합니다. 이 접근법은 다양한 암에서 점점 더 검증되고 있으며, 기존 병기 결정 시스템보다 우수한 성능을 입증하고 있습니다 20,21,22. 당사의 도구는 임상의가 진단 및 치료 중에 개인화되고 보다 정확한 예후 추정치를 만들 수 있도록 지원하여 MPCC 관리에 대한 의사 결정을 향상시키는 것을 목표로 합니다.
프로토콜
이 연구는 두 단계로 구성됩니다. 먼저, MPCC의 임상 및 생존 데이터는 SEER 데이터베이스에서 수집되었습니다. 그런 다음 R 소프트웨어(버전 4.3.3)를 사용하여 경쟁 위험 모델을 분석하고 구성했습니다. 연구의 워크플로우 다이어그램은 그림 1에 나와 있습니다. 본 연구는 참여에 대한 윤리적 승인 및 동의를 필요로 하지 않습니다. 이 연구에 사용된 데이터는 데이터베이스에서 얻은 것입니다.
1. 데이터 수집
- SEER를 다운로드합니다. SEER 데이터베이스 웹 사이트 (http://seer.cancer.gov/about/overview.html)의 Stat 8.4.3 소프트웨어. SEER에 등록하고 로그인합니다. 통계 8.4.3을 사용하여 관련 환자 데이터를 얻습니다.
- SEER에 로그인한 후. 통계 8.4.3에서 사례 목록 세션 > 데이터를 클릭하고 Incidence SEER Research Data, 17 Registries, Nov 2022 Sub (2000-2020) 데이터베이스를 선택합니다.
- >편집을 클릭하고 {인종, 성별, 연도 dx. 진단 연도} = '2004', '2005', '2006', '2007', '2008', '2009', '2010', '2011', '2012', '2013', '2014', '2015' 및 {사이트 및 형태. 사이트 코드 변경 ICD-O-3/WHO 2008} = 'Colon and Rectum' AND {Site and Morphology. Diagnostic Confirmation} = 'Positive histology' AND {Multiple Primary Fields. 시퀀스 번호}! = '1차 1회만'.
- 확인을 클릭하고 선택 항목을 저장합니다. 테이블을 클릭하고 사용 가능한 변수 인터페이스에서 단일 연령 및 85+로 연령 코드 변경, 성별, 사이트 코드 변경 ICD-O-3/WHO 2008, CS 종양 크기, 등급 코드(2017년까지), 파생된 AJCC T, 6판(2004-2015년), 파생된 AJCC N, 6판(2004-2015년), 파생된 AJCC M, 6판(2004-2015년), 방사선 재코드, 화학요법 코드(예, 아니오/unk), SEER 원인별 사망 분류, SEER의 기타 사망 원인 분류, 생존 개월, 환자 ID, RX Summ-Surg Prim Site(1998+)를 선택하고 열을 클릭합니다.
- Output( 출력)을 클릭하고, 데이터 이름을 지정하고, Execute(실행 )를 클릭하여 데이터를 출력하고 저장합니다.
- SEER 데이터베이스는 환자가 MPCC를 가지고 있는지 여부에 대한 정보를 직접 제공하지 않습니다. 위에서 설명한 대로 데이터 다운로드를 완료한 후 환자 ID를 사용하여 MPCC가 있는 환자, 즉 두 번 이상 발생한 환자를 필터링합니다. MPCC 환자를 식별한 후 생존 시간을 기준으로 종양 진단 사이의 간격을 계산합니다. 6개월을 기준점으로 환자를 SMPCC와 MMPCC로 분류합니다.
- 최초 진단 당시의 연령을 기준으로 환자의 연령을 = 65세 및> 65세로 분류합니다. 종양 등급의 경우 등급 I은 양호한 분화, 등급 II는 중간 정도의 분화, 등급 III은 낮은 분화, 등급 IV는 미분화에 해당합니다. 여러 종양의 분포를 기반으로 종양 위치를 오른쪽 결장, 왼쪽 결장직장 및 전체 결장직장으로 분류합니다.
- 근위부와 원위부 결장직장 사이에는 배아 발달과 생물학적 특성에 명백한 차이가 있으며, 비장 굴곡에 따라 종양 위치를 나눕니다23. 오른쪽 결장은 맹장, 상행 결장, 간 굴곡 및 횡단 결장을 포함하는 비장 굴곡에 근접한 것으로 정의되며, 왼쪽 결장에는 비장 굴곡, 하행 결장, S상 결장, 직장 접합 및 직장이 포함됩니다. 환자의 종양이 전적으로 오른쪽 결장에 위치한 경우 종양 위치를 오른쪽 결장으로 정의합니다. 모든 종양이 좌측 결장직장에 위치한 경우 위치를 좌측 결장직장으로 정의합니다. 오른쪽 결장과 왼쪽 결장직장 모두에 종양이 있는 경우 위치를 전체 결장직장으로 정의합니다.
- 동일한 환자의 여러 종양 중에서 가장 큰 종양 직경을 선택하여 종양 크기를 결정합니다. 종양 직경에 따라 크기를 = 5cm 또는 >5cm로 분류합니다. 총 8,931명의 환자가 연구에 참여했습니다. 학습 코호트와 검증 코호트를 만들려면 모든 사례를 7:3 비율로 무작위로 나눕니다.
2. 모델 구성
- RStudio(2023.12.1+402) 및 R 소프트웨어(4.3.3)를 다운로드합니다. RStudio를 열어 R 소프트웨어를 실행합니다. 새 파일을 클릭하고 R 스크립트를 선택하여 새 R 프로그래밍 인터페이스를 만듭니다. 코드 편집기에 관련 코드를 입력하고 실행을 클릭하여 코드를 실행합니다.
참고: R 언어와 그 함수는 다양한 매개변수를 제공합니다. 이러한 매개 변수를 추가하거나 수정하면 데이터 분석 및 시각화를 향상시킬 수 있습니다. - 다음 코드를 사용하여 일변량 분석을 수행하고 CIF 곡선을 플로팅합니다.
도서관(Tidycmprsk)
라이브러리(GTLuffect)
라이브러리(GGSURVFIT)
라이브러리(GGPRISM)
데이터 <-read.csv('data.csv')"
데이터 $ 상태 <-factor (데이터 $ 상태, 수준 = c (0,1,2), 레이블 = c ( "0", "CSS", "OCS"))
CIF <- tidycmprsk::cuminc(Surv(시간, 상태) ~ 성별, 데이터 = 데이터)
ggcuminc(CIF,결과= c("CSS", "OCS"),크기=1.5)
여기서 data.csv는 SEER 데이터베이스에서 가져온 데이터입니다. - 위의 코드를 실행한 후 내보내기를 클릭한 다음 이미지로 저장을 클릭하고 마지막으로 저장을 클릭하여 이미지를 저장합니다. 후속 이미지를 저장하는 방법은 이 단계와 동일합니다. 위 코드의 Sex를 다른 요인으로 하나씩 대체하여 모든 요인에 대한 일변량 분석을 수행합니다.
- 다음 코드를 사용하여 BSR 및 다변량 분석 및 시각화를 수행합니다.
라이브러리(LEAPS)
라이브러리(riskRegression)
라이브러리(prodlim)
라이브러리 (Forestplot)
leaps<-regsubsets(상태==0~성별+크기+등급+T+N+M,데이터 = 데이터)
플롯(leaps,scale="adjr2")
다중 <- FGR(Hist(시간, 상태)~성별 + T + N + M +등급 + 크기, 원인=1, 데이터=데이터)
요약(다중)
다중 <- read.csv("multi.csv",헤더 = T)
forestplot (labeltext = as.matrix (Multi [, 1 : 4]), mean = Multi $HR_mean, ower = Multi $HR_1, upper = Multi $HR_2)
여기서 multi.csv의 데이터는 이전 코드의 결과에서 가져옵니다. - 2.3단계를 따릅니다. 을 클릭하여 이미지를 저장합니다.
- 다음 코드를 사용하여 노모그램, ROC 곡선, 보정 곡선 및 DCA 곡선을 플로팅합니다.
라이브러리(QHScrnomo)
라이브러리(RMS)
라이브러리(riskRegression)
라이브러리(prodlim)
데이터 <- read.csv("data.csv")
d <- datadist(데이터)
옵션(datadist = "d")
e <-cph(Surv(시간,상태==1)~섹스 + T + N + M +등급,데이터 = 데이터,
x=T, y=T, surv=T,time.inc=60)
nomo <- crr.fit(e,failcode=1,cencode = 0)
nomogram.crr(fit =nomo, lp = F, xfrac = 0.5, fun.at =seq(from=0, to=1, by= 0.1) , failtime =c(12,36,60),funlabel = c("1년 CSS 누적 발생률", "3년 CSS 누적 발생률","5년 CSS 누적 발생률"))
세트.시드(123)
data$pro <- tenf.crr(m3,time = 60)
groupci(x=data$pro, ftime = data$time, fstatus = data$status, failcode = 1, cencode = 0, ci = TRUE)
f <- CSC(Hist(시간,상태)~성별 + T + N + M +등급,데이터 = 데이터)
x <- 점수(목록(모델1=f), 역사(시간, 상태)~1, 데이터=데이터, 원인=1, 시간=c(12,36,60), se.fit=1L, plots="roc", metrics="auc")
g <- as.data.frame(x$AUC$score)
h <- g[g$times %in% C(12,36,60),]
col = c("다크시안","토마토","자주색")
plotROC(x, xlab="1-특이도", ylab="민감도", col=col[1], cex=1.5, 범례="", auc.in.legend = F, times = 12)
plotROC(x,col=col[2],범례 = '', cex=1.5,times =36,auc.in.legend = F,더하기=T)
plotROC(x,col=col[3], 횟수 =60, 더하기=T, cex=1.5, 범례 = '',auc.in.legend = F)
다리 <- paste0(c("1년: ","3년: ","5년: "),substr(a$AUC,1,5)) - 2.3단계를 따릅니다. 을 클릭하여 이미지를 저장합니다.
- ggscidca 패키지를 사용하여 DCA 곡선을 플로팅합니다.
라이브러리(GGSCIDCA)
df_surv<-read.csv("data.csv",헤더 = T)
cox_model <- coxph(Surv(시간,상태==1)~성별 + T + N+ M +등급, 데이터 = df_surv)
cox_model1<-newcrr(cox_model)
scidca (cox_model1, newdata = df_surv, threshold.line = T, threshold.text = T)
여기서 data.csv는 SEER 데이터베이스에서 가져온 데이터입니다. - 2.3단계를 따릅니다. 을 클릭하여 이미지를 저장합니다.
결과
환자 특성
총 8,931명의 환자가 연구에 참여했습니다. 성별 분포 측면에서는 남성 환자(56%)가 여성 환자(44%)보다 높았다. 종양 위치 분포와 관련하여, 종양의 대부분은 전체 결장직장에 분포되어 있는 반면, 가장 적은 종양은 우측 결장에 분포되어 있었다(21%). 종양 등급별로 보면 가장 흔한 등급은 II등급으로 6,251명으로 전체의 70%를 차지했다. 그 뒤를 이어 3등급이 2,026명(23%)으로 뒤를 이었다. I등급과 IV등급은 각각 355명과 299명으로 전체의 4%와 3.3%를 차지했다. 종양 침습의 경우, T3 및 T4 단계가 가장 흔했습니다. 환자의 절반 이상은 림프절 전이가 없었으며, 17%는 원격 전이를 경험했다. 방사선 요법은 1,446명(16%), 화학요법은 3,843명(43%)이 시행했다. SCRC는 5,327명의 환자가 전체의 60%를 차지하여 더 흔했습니다. 메타인성 대장암 환자는 3,604명으로 40%를 차지했다. 환자들은 7:3 비율로 훈련 세트와 검증 세트로 무작위로 나뉘었습니다. 표 1에서 볼 수 있듯이 훈련 코호트와 검증 코호트 간의 기준선 데이터에는 통계적으로 유의미한 차이가 없었습니다.
단변량 해석
경쟁 사건의 영향을 통제한 후 일변량 분석 결과에서 성별, 종양 등급 및 크기, TNM 병기, 방사선, 화학 요법, 동기 또는 메타동기 상태, 종양 위치가 MPCC 환자의 CSS에 영향을 미치는 예후 인자인 것으로 나타났습니다. MPCC 환자에서 CSS의 예후 요인은 나이만이 아닙니다. 방사선, 화학요법, 위치, 동기 또는 준동기 상태에서 CIF의 유의미한 교차점이 있다는 점에 주목하며, 이는 MPCC 환자에 대한 방사선의 단기 및 장기 예후 효과가 화학요법, 위치, 동기 또는 준동기 상태와 마찬가지로 다르다는 것을 나타냅니다. 각 부분군의 누적 위험 곡선은 그림 2에 나와 있습니다.
다변수 분석
단변수 분석으로 얻은 예후 요인은 경쟁 위험 모델의 최량 부분 집합 회귀(BSR) 및 다변수 분석에 통합되었습니다. 그 중 방사선, 화학요법, 위치, 동기 또는 준동기 상태는 예후에 대한 이중 효과 때문에 제외됩니다. BSR과 다변량 분석의 결과는 모두 MPCC 환자에서 성별, TNM 병기 및 종양 등급이 CSS에 대한 독립적인 위험 요소임을 보여주었습니다. (그림 2)
Nomogram의 구축 및 검증
다변수 분석으로 얻은 독립적인 위험 요인을 기반으로 라인 노모그램을 구축하여 CSS를 예측하고 예측 모델의 성능을 검증합니다(그림 3). 그런 다음 ROC 곡선, 보정 곡선 및 DCA를 사용하여 모델을 평가합니다. ROC 곡선을 보면 훈련 코호트의 1년, 3년, 5년의 AUC는 0.762, 0.742, 0.734였고, 검증 코호트의 1년, 3년, 5년의 AUC는 0.801, 0.740, 0.743이었다. 훈련 코호트와 검증 코호트에서 보정 곡선은 예상 확률과 실제 데이터 간에 높은 일치도를 보여주었습니다(그림 4). 임상 적용에서 모델의 성능을 검증하기 위해 DCA를 사용하여 모델의 임상적 가치를 평가했습니다. 결과는 모델이 양호한 순이익을 보인다는 것을 보여줍니다(그림 5).
그림 1: 연구의 워크플로우 다이어그램. 본 연구는 두 단계로 구성되는데, 첫째, SEER를 사용하여 데이터를 얻었다. Stat, 그리고 데이터 분석 및 시각화는 R을 사용하여 수행되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
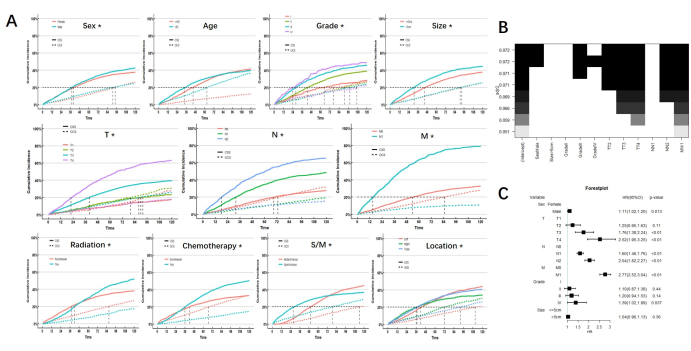
그림 2: 분석 결과. (A) 부분군의 CIF. *는 p <0.05를 나타냅니다. (B) 최량 부분 집합 회귀. 최상의 적합도에서 성별, 등급 및 TNM 단계가 포함 대상으로 고려되었습니다(상단에 검은색 블록으로 표시). (C) 다변량 분석은 또한 성별, 등급 및 TNM 단계가 독립적인 위험 요인임을 보여주었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
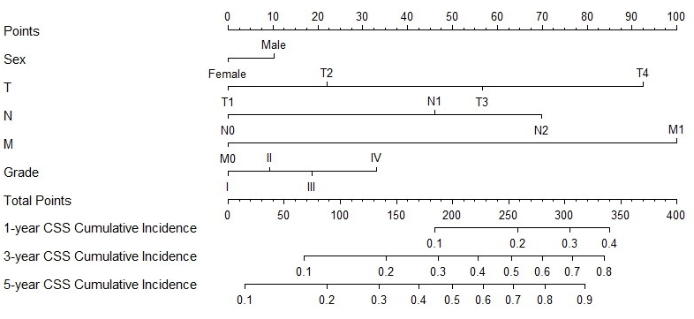
그림 3: MPCC 환자의 CSS에 대한 노모그램. 환자의 총점은 각 요인에 해당하는 점수를 더하여 계산할 수 있습니다. 총점을 기준으로 1년, 3년, 5년 시점에 암으로 인한 사망 확률을 예측할 수 있습니다. 이 수치의 더 큰 버전을 보려면 여기를 클릭하십시오.
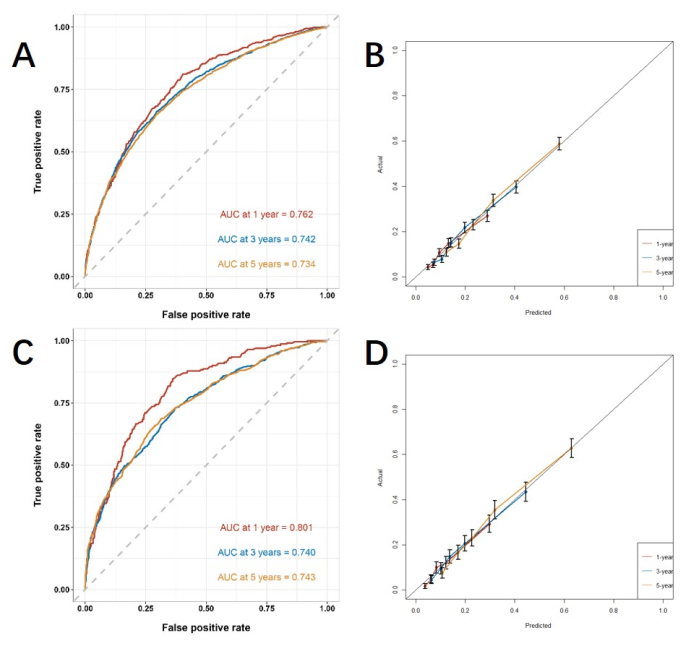
그림 4: ROC 곡선 및 보정 곡선. (A, B) 훈련 코호트(n=6255) 및 (C, D) 검증 코호트(n=2676)의 곡선. AUC 값이 1에 가까울수록 모델의 분류 성능이 향상됩니다. 오차 막대는 실제 사건이 발생할 확률에 대한 95% 신뢰 구간을 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
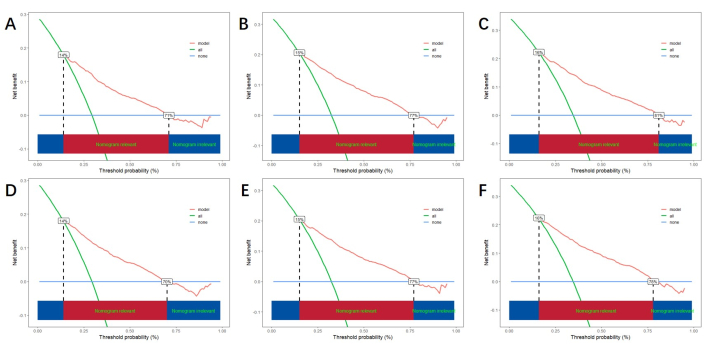
그림 5: 1년, 3년, 5년 DCA. (A, B, C) 교육 코호트 및 (D, E, F) 검증 코호트의 DCA. 녹색 선은 모든 긍정의 순 이익을 나타내고, 파란색 선은 긍정 없음의 순 이익을 나타내고, 빨간색 선은 모델의 순 이익을 나타냅니다. 아래의 빨간색 영역은 모든 긍정 및 긍정 없음 이상의 이점을 초과하는 모델의 이점을 나타내며 모델의 실제 이점 범위를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: 환자의 임상병리학적 및 기준선 특성. 이 표를 다운로드하려면 여기를 클릭하십시오.
토론
흔한 소화기 계통 종양으로서 고립성 결장직장암의 예후에 영향을 미치는 요인이 이전 연구에서 연구되고 확인되었다24. 그러나 MPCC 환자의 예후 인자에 대한 연구는 제한적입니다. 이 연구에는 2004년에서 2015년 사이에 SEER 데이터베이스에서 수술을 받은 8,931명의 MPCC 환자가 포함되었습니다. CSS에 영향을 미치는 위험 요인을 조사하기 위해 경쟁 위험 모델을 사용하고 예측 모델을 구축했습니다. 본 연구에서 사망한 환자의 39.1%가 암 이외의 원인으로 사망한 것으로 나타나 CSS의 위험인자를 분석하기 위해 경쟁적인 위험모형을 사용할 필요성을 강조하였다.
이전 연구에서는 고령이 MPCC 환자의 OS에 영향을 미치는 위험 요인이라는 사실이 밝혀졌다12. 그러나 본 연구에서는 나이가 CSS의 위험 요인이 아니라는 것을 확인했다. 고령 환자는 종종 기본 건강 상태가 좋지 않고 동반 질환이 더 많아 젊은 환자에 비해 OS가 짧아질 수 있습니다. 또한 노인은 심혈관 질환 및 심각한 감염과 같은 다른 원인으로 사망할 가능성이 더 높기 때문에 젊은 환자보다 이러한 요인에 더 취약합니다. 본 연구에서는 다른 원인으로 인한 사망과 암 자체로 인한 사망 간의 경쟁 관계에 초점을 맞췄다. 경쟁 위험 모델을 사용하고 다른 이유로 인한 사망의 간섭을 제외함으로써 이러한 조건에서 고령은 더 이상 MPCC의 CSS에 대한 위험 요소가 아님을 발견했습니다. 이는 서로 다른 연령대의 MPCC 환자가 유사한 종양 부담에 직면할 수 있음을 나타냅니다. 전반적으로 이 질환을 앓고 있는 젊은 환자와 노인 환자는 유사한 CSS를 가지고 있으며, 이는 개인화된 치료 전략을 개발하기 위한 귀중한 지침을 제공합니다.
이 연구는 또한 MPCC의 발병률과 예후가 성별과 관련된 경향을 보이며, 남성 환자의 비율이 더 높고 CSS가 더 낮다는 것을 발견했습니다. 이것은 고립성 결장직장암에서 관찰된 상황과 일치합니다. 이전 연구에서는 고립성 결장직장암이 남성에게 더 흔하고 남성 환자가 여성 환자보다 예후가 더 나쁘다는 것을 보여주었습니다. 이는 에스트로겐이 대장암의 발병과 진행에 미치는 영향 때문일 수 있다25. 또한, 다른 연구에서는 남성 환자의 장내 미생물총(microbiota)과 장내 대사 산물이 대장암 환자에서 관찰되는 성별 차이의 원인 중 하나일 수 있다고 제안했다26. 독일에서 실시된 한 연구는 제2형 당뇨병이 여성27세의 대장암에 더 큰 영향을 미친다는 것을 발견했다. 또한 비타민 D는 여성의 결장직장 신생물에 대한 보호 효과가 있지만 남성에서는 유사한 결과가 발견되지 않았습니다28.
경쟁 일변량 분석에서는 종양 크기가 큰 MPCC 환자가 CSS가 더 낮은 것으로 나타났습니다. 이전 연구에서는 종양 크기가 종양 공격성의 지표로 간주되는 경우가 많았다 29,30,31. 그러나 경쟁적인 다변량 분석의 결과는 종양 크기를 독립적인 위험 인자로 뒷받침하지 않았다. 이는 결장직장과 같은 속이 빈 기관에서 종양 크기가 종양의 공격성을 반영하는 능력이 제한적일 수 있음을 시사합니다. 이러한 현상은 종양 생물학의 복잡성, 종양 성장 및 장기에 걸친 다양한 퍼짐 패턴 등 다양한 요인에서 비롯될 수 있습니다.
낮은 종양 등급은 종양 세포의 더 강한 침습 및 이동 능력을 의미하며, 이는 이 연구의 결론과 일치합니다: 더 나쁜 종양 등급은 MPCC 환자의 더 나쁜 CSS를 암시합니다. TNM 병기는 환자의 치료를 안내하고 예후를 예측하기 위해 가장 일반적으로 사용되는 임상적 방법이다32,33. 이 연구는 종양 침투가 깊을수록 전이성 림프절의 수가 많고 장기 전이가 존재할수록 환자의 암 특이적 생존율이 더 나빠진다는 것을 발견했습니다. 이것은 임상적 합의와 완전히 일치합니다.
대장암의 경우 종양의 위치에 따라 예후가 달라집니다. 많은 연구에서 오른쪽 결장이 좌측 결장직장에 비해 예후가 더 나쁘다는 사실이 밝혀졌다 34,35,36,37. 그러나 일부 연구에서는 절제 가능한 대장암의 경우 종양 위치가 장기 예후에 영향을 미치지 않는다고 제안한다38. 이 연구에서 우측 결장에 위치한 MPCC는 단기적으로는 CSS가 더 나빴지만 장기적으로는 예후가 더 좋았습니다. 마찬가지로, 방사선 및 화학요법을 받은 MPCC 환자는 단기적 CSS가 더 좋았지만 장기 CSS는 더 나빴습니다. SCRC는 MCRC에 비해 단기 CSS가 더 나빴지만 장기 CSS는 더 좋았습니다. 이러한 이중적 효과가 예후에 미치는 이유는 아직 불분명하다.
이 연구에는 몇 가지 제한 사항이 있습니다. SEER 데이터베이스의 내재적 제약으로 인해 발암배아 항원(CEA) 수준 및 현미부수체 상태와 같은 일부 알려진 예후 지표를 얻거나 분석할 수 없었습니다. 또한, MPCC 위험이 높은 염증성 장 질환, 유전성 비용종증 대장암 및 가족성 선종성 용종증 환자를 배제할 수 없었다39. 또한, 면역요법은 대장암 치료에서 점점 더 중요한 측면이 되고 있지만, 이에 대한 데이터를 얻을 수 없었습니다. 분석의 소급적 특성과 단일 데이터 세트에 대한 의존은 내재적 편향을 유발할 수 있습니다.
SEER 데이터베이스를 기반으로 한 연구는 남성의 성별, 낮은 종양 등급 및 진행된 TNM 병기가 수술 후 MPCC 환자의 CSS 저하와 관련이 있음을 발견했습니다. 진단과 치료 과정에서 이러한 위험 요인을 가진 환자에게 세심한 주의를 기울이는 것이 중요합니다. 또한, 본 연구는 MPCC 환자의 CSS를 예측할 수 있는 노모그램을 개발하여 예후를 정확하게 예측하고 임상적 치료 의사결정에 기여할 수 있도록 하였다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
없음.
자료
| Name | Company | Catalog Number | Comments |
| forestplot package | Comprehensive R Archive Network (CRAN) | forest plot 3.1.3 | A forest plot that allows for multiple confidence intervals per row, custom fonts for each text element, custom confidence intervals, text mixed with expressions, and more. |
| ggprism package | Comprehensive R Archive Network (CRAN) | ggprism 1.0.5 | The ggprism package provides various themes, palettes, and other useful functions to customise ggplots and give them the ‘GraphPad Prism’ look. |
| ggscidca package | Comprehensive R Archive Network (CRAN) | ggscidca package | The 'ggscidca' package adds coloured bars of discriminant relevance to the traditional decision curve. Improved practicality and aesthetics. |
| ggsurvfit package | Comprehensive R Archive Network (CRAN) | ggsurvfit 1.1.0 | The ggsurvfit package eases the creation of time-to-event (aka survival) summary figures with ggplot2. The concise and modular code creates images that are ready for publication or sharing. |
| gtsummary package | Comprehensive R Archive Network (CRAN) | gtsummary 2.0.0 | The gtsummary package provides an elegant and flexible way to create publication-ready analytical and summary tables using the R programming language. The {gtsummary} package summarizes data sets, regression models, and more, using sensible defaults with highly customizable capabilities. |
| QHScrnomo package | Comprehensive R Archive Network (CRAN) | QHScrnomo 3.0.1 | The goal of QHScrnomo is to provide functionality to construct nomograms in the context of time-to-event (survival) analysis in the presence of competing risks. It also contains functions to build, validate, and summarize these models. |
| R Software | R Core Team | R 4.3.3 | Free software environment for statistical computing and graphics |
| riskRegression package | Comprehensive R Archive Network (CRAN) | riskRegression 1.3.7 | Risk Regression Models and Prediction Scores for Survival Analysis with Competing Risks |
| rms package | Comprehensive R Archive Network (CRAN) | rms 6.8-1 | rms does regression modeling, testing, estimation, validation, graphics, prediction, and typesetting by storing enhanced model design attributes in the fit. |
| RStudio | RStudio, Public Benefit Corporation(PBC) | Rstudio 2023.12.1+402 | Integrated Development Environment (IDE) used for running R scripts, data analysis, and model development. Provides a user-friendly interface for R programming with advanced features like script editing, debugging, and version control. |
| SEERstat | National Cancer Institute (NCI) | SEERstat 8.4.3 | Software for statistical analysis of SEER and other cancer-related databases |
| tidycmprsk package | Comprehensive R Archive Network (CRAN) | tidycmprsk 1.0.0 | The tidycmprsk package provides an intuitive interface for working with the competing risk endpoints |
참고문헌
- Bray, F., et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 74 (3), 229-263 (2024).
- Derwinger, K., Gustavsson, B. A study of aspects on gender and prognosis in synchronous colorectal cancer. Clin Med Insights Oncol. 5, 259-264 (2011).
- Chin, C. C., Kuo, Y. H., Chiang, J. M. Synchronous colorectal carcinoma: predisposing factors and characteristics. Colorectal Dis. 21 (4), 432-440 (2019).
- Nikoloudis, N., et al. Synchronous colorectal cancer. Tech Coloproctol. 8 (Suppl 1), s177-s179 (2004).
- Huang, C. S., et al. Synchronous and metachronous colorectal cancers: Distinct disease entities or different disease courses. Hepatogastroenterology. 62 (140), 838-842 (2015).
- He, W., et al. Prognosis of synchronous colorectal carcinoma compared to solitary colorectal carcinoma: a matched pair analysis. Eur J Gastroenterol Hepatol. 31 (12), 1489-1495 (2019).
- Ranstam, J., Cook, J. A., Collins, G. S. Clinical prediction models. Br J Surg. 103 (13), 1886(2016).
- Smith, T., et al. Comparison of prognostic models to predict the occurrence of colorectal cancer in asymptomatic individuals: a systematic literature review and external validation in the EPIC and UK Biobank prospective cohort studies. Gut. 68 (4), 672-683 (2019).
- Iasonos, A., Schrag, D., Raj, G. V., Panageas, K. S. How to build and interpret a nomogram for cancer prognosis. J Clin Oncol. 26 (8), 1364-1370 (2008).
- Conroy, T., et al. Five-year outcomes of FOLFIRINOX vs Gemcitabine as adjuvant therapy for pancreatic cancer: A randomized clinical trial. JAMA Oncol. 8 (11), 1571-1578 (2022).
- Zhong, M., et al. Impact of lung metastasis versus metastasis of bone, brain, or liver on overall survival and thyroid cancer-specific survival of thyroid cancer patients: A population-based study. Cancers. 14 (13), 3133(2022).
- Xu, Y., Wang, X., Huang, Y., Ye, D., Chi, P. A LASSO-based survival prediction model for patients with synchronous colorectal carcinomas based on SEER. Transl Cancer Res. 11 (8), 2795-2809 (2022).
- Zhang, X., Zhao, L., Hu, Y., Deng, K., Ren, W. A novel risk prediction nomogram for early death in patients with resected synchronous multiple primary colorectal cancer based on the SEER database. Int J Colorectal Dis. 38 (1), 130(2023).
- Zhang, X., et al. Developing prognostic nomograms to predict overall survival and cancer-specific survival in synchronous multiple primary colorectal cancer based on the SEER database. J Cancer Res Clin Oncol. 149 (15), 14057-14070 (2023).
- Austin, P. C., Lee, D. S., Fine, J. P. Introduction to the analysis of survival data in the presence of competing risks. Circulation. 133 (6), 601-609 (2016).
- Lau, B., Cole, S. R., Gange, S. J. Competing risk regression models for epidemiologic data. Am J Epidemiol. 170 (2), 244-256 (2009).
- Nolan, E. K., Chen, H. Y. A comparison of the Cox model to the Fine-Gray model for survival analyses of re-fracture rates. Arch Osteoporos. 15 (1), 86(2020).
- Park, S. Y. Nomogram: An analogue tool to deliver digital knowledge. J Thorac Cardiovasc Surg. 155 (4), 1793(2018).
- Balachandran, V. P., Gonen, M., Smith, J. J., DeMatteo, R. P. Nomograms in oncology: more than meets the eye. Lancet Oncol. 16 (4), e173-e180 (2015).
- Liang, W., et al. Development and validation of a nomogram for predicting survival in patients with resected non-small-cell lung cancer. J Clin Oncol. 33 (8), 861-869 (2015).
- Chen, D., et al. Predicting postoperative peritoneal metastasis in gastric cancer with serosal invasion using a collagen nomogram. Nat Commun. 12 (1), 179(2021).
- Niu, X., et al. A prognostic nomogram for patients with newly diagnosed adult thalamic glioma in a surgical cohort. Neuro Oncol. 23 (2), 337-338 (2021).
- Luo, S., et al. Comparison of left- and right-sided colorectal cancer to explore prognostic signatures related to pyroptosis. Heliyon. 10 (7), e28091(2024).
- Keum, N., Giovannucci, E. Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies. Nat Rev Gastroenterol Hepatol. 16 (12), 713-732 (2019).
- Foster, P. A. Oestrogen and colorectal cancer: mechanisms and controversies. Int J Colorectal Dis. 28 (6), 737-749 (2013).
- Wang, L., et al. Male-biased gut microbiome and metabolites aggravate colorectal cancer development. Adv Sci. 10 (25), e2206238(2023).
- Krämer, H. U., et al. Type 2 diabetes mellitus and gender-specific risk for colorectal neoplasia. Eur J Epidemiol. 27 (5), 341-347 (2012).
- Aigner, E., et al. Gender- and site-specific differences of colorectal neoplasia relate to vitamin D. Aliment Pharmacol Ther. 40 (11-12), 1341-1348 (2014).
- Dai, W., et al. Does tumor size have its prognostic role in colorectal cancer? Re-evaluating its value in colorectal adenocarcinoma with different macroscopic growth pattern. Int J Surg. 45, 105-112 (2017).
- Rössler, O., et al. Tumor size, tumor location, and antitumor inflammatory response are associated with lymph node size in colorectal cancer patients. Mod Pathol. 30 (6), 897-904 (2017).
- Zhang, Q., et al. Prognostic impact of tumor size on patients with metastatic colorectal cancer: a large SEER-based retrospective cohort study. Updates Surg. 75 (5), 1135-1147 (2023).
- Greene, F. L. TNM: our language of cancer. CA Cancer J Clin. 54 (3), 129-130 (2004).
- Ueno, H., et al. Optimal colorectal cancer staging criteria in TNM classification. J Clin Oncol. 30 (13), 1519-1526 (2012).
- Yahagi, M., Okabayashi, K., Hasegawa, H., Tsuruta, M., Kitagawa, Y. The worse prognosis of right-sided compared with left-sided colon cancers: a systematic review and meta-analysis. J Gastrointest Surg. 20 (3), 648-655 (2016).
- Holch, J. W., Ricard, I., Stintzing, S., Modest, D. P., Heinemann, V. The relevance of primary tumour location in patients with metastatic colorectal cancer: A meta-analysis of first-line clinical trials. Eur J Cancer. 70, 87-98 (2017).
- Petrelli, F., et al. Prognostic survival associated with left-sided vs right-sided colon cancer: A systematic review and meta-analysis. JAMA Oncol. 3 (2), 211-219 (2017).
- Zheng, C., Jiang, F., Lin, H., Li, S. Clinical characteristics and prognosis of different primary tumor location in colorectal cancer: a population-based cohort study. Clin Transl Oncol. 21 (11), 1524-1531 (2019).
- Karim, S., Brennan, K., Nanji, S., Berry, S. R., Booth, C. M. Association between prognosis and tumor laterality in early-stage colon cancer. JAMA Oncol. 3 (10), 1386-1392 (2017).
- Lindberg, L. J., et al. Risk of multiple colorectal cancer development depends on age and subgroup in individuals with hereditary predisposition. Fam Cancer. 18 (2), 183-191 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유