Method Article
体液からの細胞外小胞の特性評価
要約
ここでは、ヒト被験者の涙や唾液などの生体液から採取された細胞外小胞(EV)の特性評価法を示します。この方法で使用されるスキャナーは、サンプルの1μLからEVの表現型、サイズ、および総粒子数を検出することができます。
要約
細胞外小胞(EV)は、細胞から生成され、ある細胞から別の細胞に生体分子を輸送することにより細胞間コミュニケーションに関与する構造です。EVは、体内を短距離および長距離を移動することが分かっており、組織特異的です。EVは組織だけでなく、涙、唾液、脳脊髄液、血液など、実質的にすべての体液にも含まれています。EVは涙や唾液から非侵襲的に採取できるとはいえ、一度に採取できる量は少量で、タンパク質を解析するのに十分なEVの入手に課題が生じることがあります。この論文で取り上げるスキャナーは、この問題の解決策を提供するナノ粒子分析装置であり、わずか1μLの生体液からEVの表現型、サイズ、および総粒子数を特徴付けて研究することができます。このプロトコルは、患者から抽出するのが難しい少量のサンプルからEVの知識を広げます。これにより、患者の快適性が向上し、さまざまな疾患や障害に対する新たな治療標的を特定できる可能性があります。
概要
細胞は、細胞間コミュニケーションにおいて重要な役割を果たす細胞外小胞(EV)の放出など、さまざまなシグナル伝達メカニズムを通じて隣接する細胞と通信します。EVは、DNA、RNA、タンパク質などの遺伝子貨物を1つの細胞から別の細胞に渡すことで、細胞間コミュニケーションに関与しています1,2,3,4,5。現在、EVにはエクソソーム、マイクロベシクル、アポトーシス体の3つのカテゴリーがあり、その大きさが特徴です。エキソソームは最も小さく、直径は30〜150 nm 6,7,8で、エンドソーム膜系9,10,11から形成されます(図1)。微小胞は、100〜1000 nmの範囲12,13,14で、原形質膜11,12,13から芽を出します(図1)。アポトーシス体はEVの中で最も大きく、1000〜5000 nm 12,14,15の範囲であり、原形質膜12,16,17からも芽を出します(図1)。サイズとは別に、EVは、密度、CD63、CD81、CD9などの分子マーカー、さらには生合成メカニズム18などの他の生物物理学的特性に基づいて分類できます。EVは、隣接する細胞間の短い距離と、体全体の長い距離を移動できる19,20,21,22,23。EVは、血液24、25、26、脳脊髄液27、28、29、涙30、31、32、唾液33、34、35などの体液に含まれています。
今日まで、超遠心分離は、サンプル36,37,38からEVを分離するための最もよく知られた方法の1つです。この方法では、複数回の遠心分離と超遠心分離が必要ですが、これは速度を上げてEVを細胞や細胞の破片から分離することで実行できます。この方法は、ペレットセルを使用して低速で開始し、その後に中速で大きな小胞を除去し、最後に超遠心分離ステップでEVをペレット化する18。超遠心分離は単離のための最良の方法と考えられているが、EVの形態を変えるという点でまだいくつかの制限が存在する39,40,41。EVの単離に使用される別の手法はフローサイトメトリーであり、これは複数の時点とエンドポイントの評価、およびハイスループットの単一EV分析の利点を強調しています。しかしながら、フローサイトメトリーの限界には、細孔の詰まりおよび弱いシグナルが含まれるが、これらに限定されない。また、グラジエント遠心分離法では、密度の異なる材料をEVと一緒に遠心分離し、超遠心法に比べてEVの分離性を高めることができます。この手法により分離は改善されますが、労働集約的で時間がかかり、サンプルの大幅な損失につながる可能性があります。さらに、降水とろ過を使用してEVを分離することもできます。これらの手法はどちらもシンプルで高速ですが、どちらもサンプルの汚染につながる可能性があります。EVを分離する手法はいくつかありますが、各手法にはそれぞれ長所と短所があり、以下にリストされています(表1)42,43,44,45,46,47,48,49,50,51,52,53,54、 55、56、57、58、59、60、61、62、63、64、65、66、67。
EVが単離されたら、分子アッセイを実行してEVを特性評価できます。ウェスタンブロットは、表面およびカーゴタンパク質の発現68,69,70を探すための一般的なアッセイであり、ポリメラーゼ連鎖反応(PCR)は、EVのmiRNA発現71,72,73に使用されます74,75,76。これらのアッセイは確立されており、興味深い結果を生み出すことができます。これらの方法の限界は、読み取り値77を得るためにEVからの大量のタンパク質またはRNAを必要とすることであり、これはそもそも少量またはEV濃度のサンプルにとって問題となる。
この論文で取り上げたナノ粒子分析装置は、表1および表2に記載されている多くの制限を克服することを可能にします:78,79,80,81,82,83,84,85,86,87,88,89,90 .この方法では、絶縁技術を利用する必要がないため、EVの歩留まりの低下を克服するのに役立ちます。また、この方法では、わずか1 μLのサンプル量から、表面タンパク質とカーゴタンパク質、総EV数、およびEVサイズを分析できます。これは、図2に示すように、テトラスパニン抗体(CD63、CD81、およびCD9)を備えた抗体マイクロアレイを使用して溶液中のEVを同定する会社が提供するテトラスパニンチップを使用して行われます。蛍光抗体は、EVの存在を確認するだけでなく、汚染粒子が結果を歪めるのを防ぎます。
この手法の全体的な目標は、少量のサンプルからEVを分析するだけでなく、EVを分析するためのより時間のかからない方法を提供することです。このナノ粒子分析装置を使用すると、ユーザーはわずか1μLのサンプルからサイズ、総粒子数、および表面タンパク質を分析できるため、涙液や唾液などの生体液に最適です。
プロトコル
記載されているすべての研究は、ヘルシンキ宣言に準拠しています。研究に含まれる前に、各被験者から書面による同意が得られました。オーフス大学病院(1-10-72-77-14)およびディーン・マギー研究所(1576837-2)からの治験審査委員会(IRB)の承認は、連邦および機関のガイドラインに従って受け取られました。処理前に、すべての涙液および唾液サンプルを匿名化しました。すべての研究は、North Texas Regional Institutional Review Board(#2020-030)によって審査され、承認されました。以下のプロトコルは、上記のようにすべてのガイドラインに準拠し、承認されています。
1. 1日目:サンプル調製とインキュベーション
- 体液を採取しますampすなわち、被験者からの涙と唾液、または冷凍庫からの解凍サンプル。
- 涙:ガラスキャピラリーチューブを使用して、外側半月板から受動的に涙を収集します。
- 角膜に触れないように注意しながら、ガラス毛細管を下まぶたに置きます。
- 10分以内に10μLの涙液を採取します。
- 裂け目はすぐに使用するか、-20 °Cで保管してください。
- 唾液:1.5 mLの微量遠心チューブで受動のよだれから唾液を採取します
- ドナーが口の中に唾液を溜めるのを許してください。
- ドナーに漏斗でチューブによだれを垂らすように頼みます。
- すぐに唾液を使用するか、-20 °Cで保存してください。
- 涙:ガラスキャピラリーチューブを使用して、外側半月板から受動的に涙を収集します。
- テトラスパニンチップをチップワッシャープレートに15分間置いてから、サンプルを室温に戻すことができます。チップをウェルに入れるときは、数字が上を向いて底面にあり、チップがウェルの側壁に触れていないことを確認してください。
- ID番号とチップ番号をノートブックに記録して、チップを追跡します。
- キットに付属のユニバーサルシリアルバス(USB)を接続し、関連付けられたキット番号のフォルダーを抽出して、選択した指定領域に保存します。
- 溶液B99μLを1.5mLチューブに入れてピペットで披露します。
- 前のステップで調製した1.5mLチューブに1μLの生体液を加えます。
- チューブをスピンダウンします。
- ステップ1.6の溶液70μLを取り、テトラスパニンチップにピペットで移します。
- フィルムをチップ洗浄プレートに置き、サンプルが蒸発するのを防ぎます。
- 振動のないベンチで室温(RT)で16時間インキュベートします。
2. 2日目:チップ洗浄
- チップ洗浄機の電源を入れます。
- ステップ1.9のフィルムをプレートから取り外します。
- プレートをチップワッシャーに入れ、所定の位置にロックされていることを確認して、蓋を閉めます。
注意: プレートは、正しく所定の位置にロックされている場合、簡単には動きません。 - チップワッシャーの CW-TETRA v0 オプションを押します。
- 開始行と洗浄する行数を選択します。
- チップ洗浄機の 続行 を押します。
- チップを洗浄している間に、以下のように抗体を含むブロッキング溶液をチューブ内に調製します。
- チップあたり300μLのブロッキング溶液を調製します。
- チップあたり0.6 μLまたは各抗体を添加します(ブロッキング溶液に添加する前にボルテックス抗体とスピンダウン抗体を調製します)。
- ブロッキング溶液+抗体カクテルをボルテックスします。
注:ブロッキング溶液+抗体カクテルを光で覆い、抗体の安定性を確保します。これは使用日に行う必要があります。
- チップワッシャーが鳴ったら、チップの上にブロッキング溶液+抗体カクテル250μLを加えます。
- 蓋を閉じて 、続行を押します。
注:チップワッシャーは、1時間のインキュベーションと、さらに数回の洗浄とすすぎを行います。 - チップワッシャーが鳴ったら、チップ洗浄プレートを取り外し、チップを上に移動しますamp。
- プレートをチップワッシャーに戻し、所定の位置にロックされていることを確認します。蓋を閉じて 、続行を押します。
- チップワッシャーがプログラムを完了したら、プレートを取り外し、チップをペーパータオルの上に置きます。チップをライトから覆います。
3. 2日目:チップのスキャン
- スキャナーの電源を入れ、スキャンソフトウェアを開きます。
注意: スキャナーは、エラーがあるかどうか、またはスキャンを続行できるかどうかを示すために、セルフチェックを受けます。 - [ フォルダの保存 ]を選択し、ファイルを保存する場所を選択します。
- [ChipFileフォルダ]を選択し、手順1.4で保存したフォルダを選択します。
- ドロップダウンオプションを選択して各チップを選択し、チャックの チップ位置 に配置します。スキャンする必要のあるすべてのチップについて、この手順を繰り返します。
- [ チップ位置] の下にある 3 つの四角をクリックして、蛍光をオンにします。正方形はそれぞれ黄色、青、赤に変わります。
- チャックの蓋を押し下げて引っ張るようにして、チャックの蓋を取り外します。
- チップをチャックに置き、しっかりと固定されていることを確認します。
- ペグを穴に置き、押し下げて蓋をスライドさせて所定の位置にロックして、チャックの蓋を元に戻します。
- チャックをステージに置き、所定の位置にロックされていることを確認します。
- [スキャンチップ]を選択します。
- [OK] を選択します。
- ステージがマシンに完全に移動したら、ドアを閉じます。
注:1つのチップをスキャンするには約12分かかります。 - プログラムがチップのスキャンが完了したら、ドアを開けてステージチェックを取り出し、スキャンが成功したかどうかを確認します。
- スキャンが成功した場合は、チップを捨てて次のチップのセットをチャックに置くか、空のチャックをステージに戻します。次に、ソフトウェアを終了し、完了したらマシンの電源を切ります。
- スキャンが成功しなかった場合は、エラー・コードを調べてエラーを修正します。
4. データ処理
- 分析ソフトウェアを開きます。
- [ プレスキャンデータ ]を選択し、2日目のステップ3.2からスキャンしたフォルダーを選択します。
- [次へ] を選択します。
- [サンプル名] 列のチップにサンプルの名前のラベルを付けるには、セルをクリックしてサンプル ID を入力します。
- 次に、[ チャネル名 ]列でテトラスパニンにラベルを付けます。赤 = CD63、緑 = CD81、青 = CD9。
- [次へ] を選択します。
- [Select Spot] グループから [High CV] を選択してドロップダウン ボックスを検査し、1 つまたは 2 つのスポットを無効にして各チップの High CV をオフにします。
- 次に、[ High Count ] を選択し、高カウントの警告をオフにしてみます。
注:一部のサンプルはカウントが高すぎる場合がありますが、それでも分析することは可能です。 - ドロップダウンメニューから [All Spots ]を選択し、[ Spot Montage]を選択して、各チップに変形や傷がないか視覚的に確認します。
- [次へ] を選択します。
- CD63チャネルで、最小値を300に設定して、 Avg % Included行の赤血球を取り除きます。黄色の細胞は問題ないので、CD81チャネルに進みます。最小値を 300 に設定しても赤いセルが黄色または白に変わらない場合は、手順 4.14 に進んでトラブルシューティングを行います。
- CD81チャネルで、最小値を300に設定して、 Avg % Included行の赤血球を取り除きます。黄色の細胞は正常で、CD9チャネルに進みます。最小値を 300 に設定しても赤いセルが黄色または白に変わらない場合は、手順 4.14 に進んでトラブルシューティングを行います。
- CD9チャネルで、最小値を400に設定して、 Avg % Included行の赤血球を取り除きます。黄色のセルは問題ないので、手順4.15に進みます。最小値を 400 に設定しても赤いセルが黄色または白に変わらない場合は、手順 4.14 に進んでトラブルシューティングを行います。
- トラブルシューティング
- 最小値を 100 単位増やします。
- 手順4.14.1が機能しなかった場合は、[作業する チップの選択 ]セクションでチップを選択し、チップを選択します。 [Particle Count ] を選択し、パーティクル数が多いスポットを無効にします。 [強度/サイズ] を選択し、[ すべてのチップ] を選択して、 問題が解決するかどうかを確認します。
- 手順4.14.2が機能しない場合は、[作業する チップの選択 ]セクションでチップを選択し、チップを選択します。 [シングルチップカットオフ ]を選択し、最小値を 100 単位増やします。次に 、[すべてのチップ ]を選択して、赤いセルが黄色になっているかどうかを確認します。これでうまくいかない場合は、[ Avg % Included] セルが黄色または白になるまでこの手順を繰り返します。
- [次へ] を選択します。
- IM チャネルの選択を解除します。
- 右下隅のヒートマップ値をすべての粒子が含まれるように設定し、[ レポートにプロットを追加]を選択します。
- ドロップダウンボックスの最大数を調整し、手動で値を入力します。
- 次に、上部中央の 「Capture Probes 」の下にあるドロップダウンメニューを選択し、「 CD63」を選択します。
- [ レポートにプロットを追加] を選択して、合計粒子数をレポートに追加します。
- [Analysis Mode] のドロップダウン メニューを選択し、[Colocalization] を選択し、[Add Plot to Report] を選択します。
- 次に、[ サイズ] を選択し、[ ビューをプロットに変更] を選択し、[ レポートにプロットを追加] を選択します。
- 最後に、[ 画像 ] を選択し、[ レポートにプロットを追加] を選択します。
- CD81 と CD9 について手順 4.18 〜 4.22 を繰り返し、そのたびに IM チャネルの選択を解除します。
- すべてのアイテムがレポートに追加されたら、[ エクスポート レポート] を選択し、正しいファイルに保存されていることを確認し、[ フォルダー] を選択します。
注:Excelファイルと画像が生成され、さらに分析に使用できるようになりました。
結果
チップ上のスポットを分析するときは、CD63、CD81、およびCD9チャネルで蛍光標識されたEVを観察してEVの最適な濃度を目指し、図3Aに示すように制御チャネルであるMIgGチャネル30,91を黒のままにする必要があります。CD81は体液のために減少します。CD63、CD81、およびCD9チャネルに蛍光がない場合、EVは存在しません(図3B)。チップが飽和しすぎないように注意してください(図3C)。これにより、分析ソフトウェアがEVのサイズ、総粒子数、および表現型の精度を計算することが困難になります。最適な濃度のスポットから、分析ソフトウェアはEVのサイズ(50-200 nm)、総粒子数、およびテトラスパニン発現を測定でき(図4)、さらに分析するために個々のスプレッドシートにエクスポートされます。テトラスパニン分析には、EV上の表面タンパク質の共局在が含まれます。共局在は "/" で表されます (例: "CD63/CD81")。これは、CD63 と CD81 が組み合わされるという意味ではありません。これは、CD63とCD81の両方がEVの表面に配置されていることを意味します。
これらの結果は、健康なサンプルと病気のサンプルとの間の貴重な洞察を提供します。健康なサンプルと病気のサンプルのどちらがより多くのEVを生成するか、より大きなEVまたはより小さいEVを生成するか、およびEVの表現型を判断できるようになります。これらの特性のいずれかまたはすべてが、疾患の生合成および/または進行に関与している可能性があります。これらの結果により、テトラスパニンレベルが存在しないか増加しているかを確認でき、細胞プロセスとEVの形成に関する洞察を得ることができます。
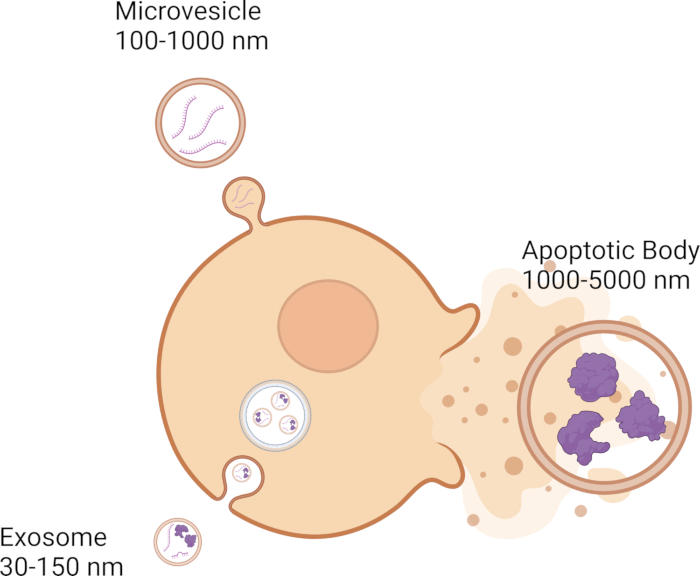
図1:さまざまなEVのタイプとサイズ。 この図の拡大版を表示するには、ここをクリックしてください。
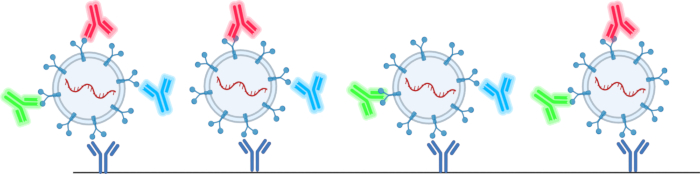
図2:テトラスパニンチップがEVを捕捉して検出する方法の概略図。この図の拡大版を表示するには、ここをクリックしてください。
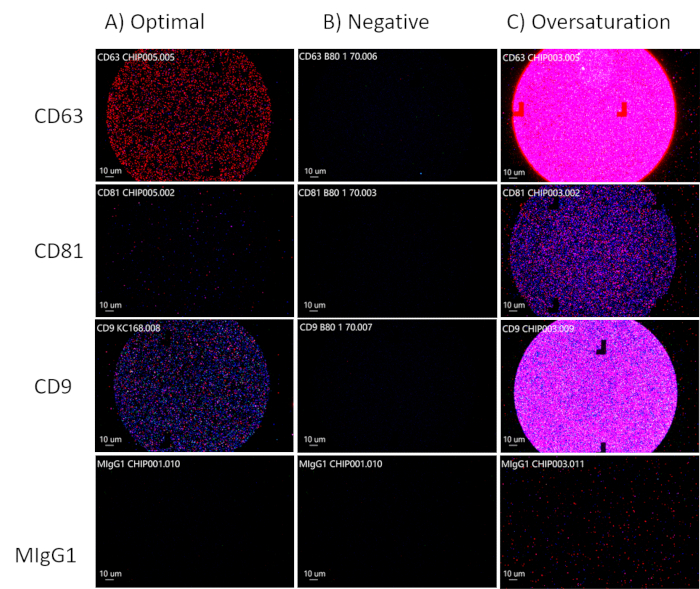
図3:EV濃度の最適化。 (A) EVの最適な濃度。(B)陰性結果:EVは存在しません。(C)EVの過飽和状態。 この図の拡大版を表示するには、ここをクリックしてください。
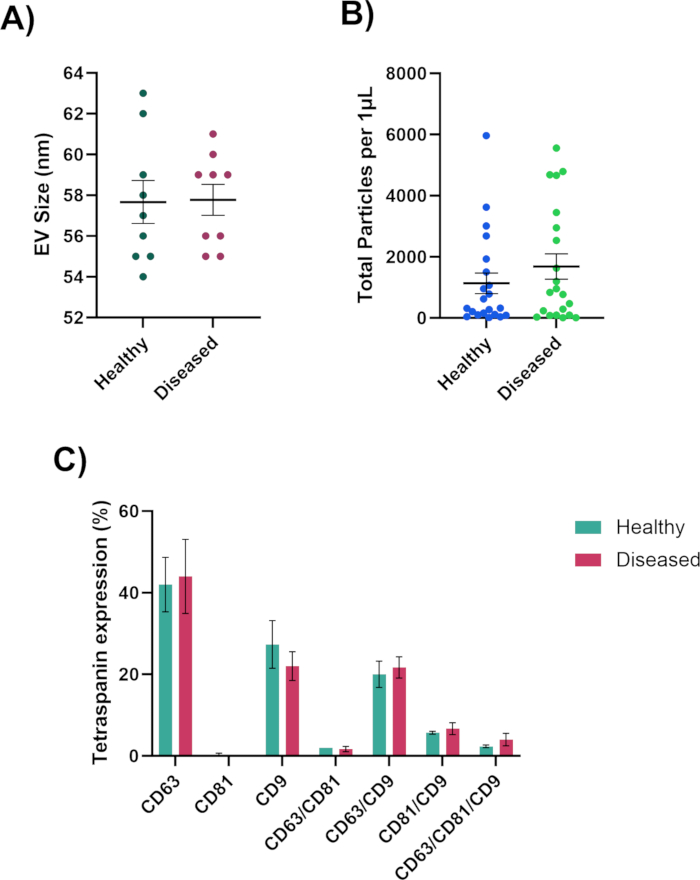
図4:ナノ粒子分析装置で生成されたデータ(A)ナノメートルで測定されたEV直径サイズ。(B)EVの総粒子数。(C)EV共局在表現型。この図の拡大版を表示するには、ここをクリックしてください。
| 技術 | 利点 | 制限 |
| 超遠心分離機 | 高純度42,43 | EVの形態の変化42,44 |
| 均質性42,45 | 大容量が必要42,44,46 | |
| 機能性42,43 | 高価な42,43 | |
| フローサイトメトリー | 複数の時点におけるサンプルの評価47 | 弱い信号48,49 |
| 複数のエンドポイント47,50 | 毛穴の詰まり51 | |
| ハイスループットのシングルEV解析52,53 | ||
| 密度勾配遠心分離 | 高純度のサンプルを作製54 | 労働集約型55,56 |
| サンプルの種類55,57 | 時間のかかる55,58 | |
| 大幅な歩留まり損失54 | ||
| 降水 | シンプル59,60 | 汚染36,44,61 |
| 高速61,62 | 非特定63 | |
| ハイイールド44,59,62 | ||
| 中性pH36 | ||
| 濾過 | シンプル64 | トラップエクソソーム65 |
| ファスト64 | 大きな小胞を損傷する36 | |
| 安価な64 | フィルターケーキ66,67 | |
| エキソソームの詰まり65 |
表1:EVアイソレーション技術の利点と制限。 EVを分離するさまざまな方法(その利点と制限を含む)のリスト。
| 技術 | 利点 | 制限 |
| 動的光散乱 | サイズ1 nmから6 μMの範囲の粒子を測定します78 | 大きなサイズ範囲の複雑なエキソソームサンプルの測定には適していません79 |
| 下限は10nmで、単分散システムに適しています79 | 汚染されたタンパク質とエキソソームを区別できない79 | |
| 透過型電子顕微鏡(TEM) | エクソソームの形態を観察する79,80 | 複雑なサンプル調製79 |
| 内部構造81の観察 | 蛍光シグナルが誇張されているため、サイズと形状に基づいてエクソソームを区別できない82 | |
| ナノ粒子追跡分析 | 10 nmから2 μMの範囲でのエキソソームの濃度、サイズ、およびサイズ分布を測定する78 | EVをタンパク質凝集体や他の汚染物質と区別できない83 |
| 迅速なサンプル調製と測定78,84 | 高価なNTA機器85 | |
| サンプルはネイティブフォーム84で回収可能 | 振動に敏感85 | |
| ウェスタンブロット(WB) | マーカータンパク質を定性的・定量的に解析できる79,86 | 複雑で時間がかかる79 |
| 細胞培養培地からのエクソソームの分析79 | EV分離株は、リポタンパク質および他の汚染物質を含んでいてもよい86 | |
| フローサイトメトリー | より高い感度と高解像度のイメージング | 400 nmの検出限界で時間と手間がかかります79,88 |
| 染色されたエクソソームと封じ込めを区別する87 | ||
| 必要な低サンプル濃度79 | 光信号は精度と解像度88を妨げます | |
| エグゾビュー | エクソソーム上のテトラスパニン(CD9、CD63、およびCD81)を測定する89 | より大きなEVサイズを測定しません75 |
| EVカーゴタンパク質を測定90 |
表2:EV特性評価手法の利点と制限。
ディスカッション
このプロトコルの最も重要なステップは、EVの最適な濃度が達成されていることを確認することです。読み取り値を取得するには十分なEVが存在する必要がありますが、チップを過飽和にするEVが多すぎないようにする必要があります。最適なEV濃度を決定する最良の方法は、1 μLのサンプルで最適化分析を行い、濃度を調整する必要があるかどうかを確認することです。もう1つの重要なステップは、サンプル中に細胞破片が豊富にあるかどうかを確認することですが、これは分析ソフトウェアでチップ上の大きなチャンクを見ることで判断できます。サンプルに細胞破片がある場合は、サンプルの単純な遠心分離またはろ過でこの問題を解決できるはずです。
追加の重要なステップは、チップがウェルの壁に触れないようにし、チップをランプに配置するときにチップの中央領域との接触を避けることです。スキャナーはチップの中央にある正方形から読み取るため、EVの混乱を防ぐために、鉗子でその領域に触れないようにすることが不可欠です。
この方法には、装置で検出するためのEVの濃度が不十分であるなど、いくつかの制限があります。濃度は、サンプルを乾燥させるか、濃縮チューブを使用することで改善できます。別の制限は、分析ソフトウェアが50〜200nmの範囲のEVのみを測定し、サイズ測定75から一部の微小胞とすべてのアポトーシス体を除外することです。
分離には多くの方法があります(表1 42,43,44,45,46,47,48,49,50,51,52, 53,54,55,56,57,58,59,60, 61,62,63,64,65,66,67)およびEVの分析(表2 78,79,80,81,82,83,84,85、 86,87,88,89,90)、そして現在のEV単離のゴールドスタンダードは、ウェスタンブロット68,69,70およびPCR 71,72,73,74などのアッセイのために細胞をペレット化するための超遠心分離36,37,38であり、75,76.このプロトコルは大きなサンプル42,44,46には適していますが、体液の入手は困難であり、多くの場合、一度に少量のサンプルしか収集できないため、超遠心分離には理想的ではありません。一方、このナノ粒子分析装置を使用すると、わずか1μLのサンプルでEVのサイズ、総粒子数、表現型などの貴重なデータを生成することができるため、生体液に最適です。抽出が困難な少量のサンプルからEVの知識を広げることができるため、患者の快適性を高め、さまざまな疾患や疾患の潜在的な治療標的を見つけることができる可能性があります。
開示事項
著者には、開示すべき競合する金銭的利益またはその他の利益相反はありません。
謝辞
NIHの資金提供に感謝します(EY031316およびEY034714)。また、ラボスペースを提供してくださったUNTHSCとNTERIにも感謝いたします。
資料
| Name | Company | Catalog Number | Comments |
| ChipWasher 100 | NanoView | EV-CW100 | Incubates, washes, rinses and dries the tetraspanin chips. This current model is no longer available. Price at time of purchase: $9,995.00 |
| ExoView Analyzer software | NanoView | N/A | Analyzes the chip informations and produces excel files for further analysis. No longer available. |
| ExoView R100 | NanoView | EV-R100 | Used to scan the tetraspanin chips at 3 wavelengths. This current model is no longer available. Price at time of purchase: $110,000.00 |
| ExoView Scanner software | NanoView | N/A | Scans the chips at 3 different wavelengths. No longer available. |
| Human Tetraspanin Kits | Unchained Labs | EV-TETRA-C | Includes 8 tetraspanin chips, Incubation Solution, Blocking Solution, CD63 antibody, CD81 antibody, CD9 antibody, Solution A, Solution B, USB, and plate cover. |
参考文献
- Abels, E. R., Breakefield, X. O. Introduction to extracellular vesicles: Biogenesis, RNA cargo selection, content, release, and uptake. Cell Mol Neurobiol. 36 (3), 301-312 (2016).
- Radler, J., Gupta, D., Zickler, A., Andaloussi, S. E. Exploiting the biogenesis of extracellular vesicles for bioengineering and therapeutic cargo loading. Mol Ther. 31 (5), 1231-1250 (2023).
- Gupta, D., Zickler, A. M., El Andaloussi, S. Dosing extracellular vesicles. Adv Drug Deliv Rev. 178, 113961 (2021).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63 (2018).
- Zhang, H., et al. Exosome-induced regulation in inflammatory bowel disease. Front Immunol. 10, 1464 (2019).
- Barzin, M., et al. Application of plant-derived exosome-like nanoparticles in drug delivery. Pharm Dev Technol. 28 (5), 383-402 (2023).
- Zhou, C., et al. Stem cell-derived exosomes: emerging therapeutic opportunities for wound healing. Stem Cell Res Ther. 14 (1), 107 (2023).
- Kalluri, R., LeBleu, V. S. The biology, function, and biomedical applications of exosomes. Science. 367 (6478), aau6977 (2020).
- Mondal, J., et al. Hybrid exosomes, exosome-like nanovesicles and engineered exosomes for therapeutic applications. J Control Release. 353, 1127-1149 (2023).
- van Niel, G., D'Angelo, G., Raposo, G. Shedding light on the cell biology of extracellular vesicles. Nat Rev Mol Cell Biol. 19 (4), 213-228 (2018).
- Paul, N., Sultana, Z., Fisher, J. J., Maiti, K., Smith, R. Extracellular vesicles- crucial players in human pregnancy. Placenta. 140, 30-38 (2023).
- Zaldivia, M. T. K., McFadyen, J. D., Lim, B., Wang, X., Peter, K. Platelet-derived microvesicles in cardiovascular diseases. Front Cardiovasc Med. 4, 74 (2017).
- Tamasi, V., Nemeth, K., Csala, M. Role of extracellular vesicles in liver diseases. Life (Basel). 13 (5), 1117 (2023).
- Fu, Y., et al. Emerging understanding of apoptosis in mediating mesenchymal stem cell therapy. Cell Death Dis. 12 (6), 596 (2021).
- Santavanond, J. P., Rutter, S. F., Atkin-Smith, G. K., Poon, I. K. H. Apoptotic bodies: Mechanism of formation, isolation and functional relevance. Subcell Biochem. 97, 61-88 (2021).
- Yu, L., et al. Apoptotic bodies: bioactive treasure left behind by the dying cells with robust diagnostic and therapeutic application potentials. J Nanobiotechnology. 21 (1), 218 (2023).
- Momen-Heravi, F., Getting, S. J., Moschos, S. A. Extracellular vesicles and their nucleic acids for biomarker discovery. Pharmacol Ther. 192, 170-187 (2018).
- Liu, Y. J., Wang, C. A review of the regulatory mechanisms of extracellular vesicles-mediated intercellular communication. Cell Commun Signal. 21 (1), 77 (2023).
- Grange, C., Bussolati, B. Extracellular vesicles in kidney disease. Nat Rev Nephrol. 18 (8), 499-513 (2022).
- Cocucci, E., Meldolesi, J. Ectosomes and exosomes: shedding the confusion between extracellular vesicles. Trends Cell Biol. 25 (6), 364-372 (2015).
- Iannotta, D. A. A., Kijas, A. W., Rowan, A. E., Wolfram, J. Entry and exit of extracellular vesicles to and from the blood circulation. Nat Nanotechnol. 19 (1), 13-20 (2024).
- Saint-Pol, J., Gosselet, F., Duban-Deweer, S., Pottiez, G., Karamanos, Y. Targeting and crossing the blood-brain barrier with extracellular vesicles. Cells. 9 (4), 851 (2020).
- Alberro, A., Iparraguirre, L., Fernandes, A., Otaegui, D. Extracellular vesicles in blood: Sources, effects, and applications. Int J Mol Sci. 22 (15), 8163 (2021).
- Nieuwland, R., Siljander, P. R. A beginner's guide to study extracellular vesicles in human blood plasma and serum. J Extracell Vesicles. 13 (1), e12400 (2024).
- Thangaraju, K., Neerukonda, S. N., Katneni, U., Buehler, P. W. Extracellular vesicles from red blood cells and their evolving roles in health, coagulopathy and therapy. Int J Mol Sci. 22 (1), 153 (2020).
- Li, C., et al. Cerebrospinal fluid-derived extracellular vesicles after spinal cord injury promote vascular regeneration via PI3K/AKT signaling pathway. J Orthop Translat. 39, 124-134 (2023).
- Deng, Y., et al. Phosphoproteome analysis of cerebrospinal fluid extracellular vesicles in primary central nervous system lymphoma. Analyst. 148 (15), 3594-3602 (2023).
- Hirschberg, Y., et al. Proteomic comparison between non-purified cerebrospinal fluid and cerebrospinal fluid-derived extracellular vesicles from patients with Alzheimer's, Parkinson's and Lewy body dementia. J Extracell Vesicles. 12 (12), e12383 (2023).
- Hefley, B. S., et al. Revealing the presence of tear extracellular vesicles in Keratoconus. Exp Eye Res. 224, 109242 (2022).
- Cross, T., et al. RNA profiles of tear fluid extracellular vesicles in patients with dry eye-related symptoms. Int J Mol Sci. 24 (20), 15390 (2023).
- Ma, H., et al. Metabolic signatures of tear extracellular vesicles caused by herpes simplex keratitis. Ocul Surf. 31, 21-30 (2024).
- Han, P., Bartold, P. M., Ivanovski, S. The emerging role of small extracellular vesicles in saliva and gingival crevicular fluid as diagnostics for periodontitis. J Periodontal Res. 57 (1), 219-231 (2022).
- Han, P., Li, X., Wei, W., Ivanovski, S. Saliva diagnosis using small extracellular vesicles and salivaomics. Methods Mol Biol. 2588, 25-39 (2023).
- Reseco, L., et al. Characterization of extracellular vesicles from human saliva: Effects of age and isolation techniques. Cells. 13 (1), 95 (2024).
- Tiwari, S., Kumar, V., Randhawa, S., Verma, S. K. Preparation and characterization of extracellular vesicles. Am J Reprod Immunol. 85 (2), e13367 (2021).
- Tomiyama, E., Fujita, K., Nonomura, N. Urinary extracellular vesicles: Ultracentrifugation method. Methods Mol Biol. 2292, 173-181 (2021).
- Xu, K., Jin, Y., Li, Y., Huang, Y., Zhao, R. Recent progress of exosome isolation and peptide recognition-guided strategies for exosome research. Front Chem. 10, 844124 (2022).
- Brennan, K., et al. A comparison of methods for the isolation and separation of extracellular vesicles from protein and lipid particles in human serum. Sci Rep. 10 (1), 1039 (2020).
- Gao, J., et al. Recent developments in isolating methods for exosomes. Front Bioeng Biotechnol. 10, 1100892 (2022).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Ansari, F. J., et al. Comparison of the efficiency of ultrafiltration, precipitation, and ultracentrifugation methods for exosome isolation. Biochem Biophys Rep. 38, 101668 (2024).
- Patel, G. K., et al. Comparative analysis of exosome isolation methods using culture supernatant for optimum yield, purity and downstream applications. Sci Rep. 9 (1), 5335 (2019).
- Stam, J., Bartel, S., Bischoff, R., Wolters, J. C. Isolation of extracellular vesicles with combined enrichment methods. J Chromatogr B Analyt Technol Biomed Life Sci. 1169, 122604 (2021).
- Le Roy, A., et al. AUC and small-angle scattering for membrane proteins. Methods Enzymol. 562, 257-286 (2015).
- Chhoy, P., Brown, C. W., Amante, J. J., Mercurio, A. M. Protocol for the separation of extracellular vesicles by ultracentrifugation from in vitro cell culture models. STAR Protoc. 2 (1), 100303 (2021).
- McDorman, K. S., Chan, C., Rojko, J., Satterwhite, C. M., Morrison, J. P. . Chapter 7 - Special Techniques in Toxicologic Pathology. Haschek and Rousseaux's Handbook of Toxicologic Pathology (Third Edition). , (2013).
- Woud, W. W., et al. An imaging flow cytometry-based methodology for the analysis of single extracellular vesicles in unprocessed human plasma. Commun Biol. 5 (1), 633 (2022).
- Gul, B., Syed, F., Khan, S., Iqbal, A., Ahmad, I. Characterization of extracellular vesicles by flow cytometry: Challenges and promises. Micron. 161, 103341 (2022).
- Black, C. B., Duensing, T. D., Trinkle, L. S., Dunlay, R. T. Cell-based screening using high-throughput flow cytometry. Assay Drug Dev Technol. 9 (1), 13-20 (2011).
- Safford, H. R., Bischel, H. N. Flow cytometry applications in water treatment, distribution, and reuse: A review. Water Res. 151, 110-133 (2019).
- Kobayashi, H., et al. Precise analysis of single small extracellular vesicles using flow cytometry. Sci Rep. 14 (1), 7465 (2024).
- Welsh, J. A., et al. A compendium of single extracellular vesicle flow cytometry. J Extracell Vesicles. 12 (2), e12299 (2023).
- Carnino, J. M., Lee, H. Extracellular vesicles in respiratory disease. Adv Clin Chem. 108, 105-127 (2022).
- Sun, Y., Sethu, P. Low-stress microfluidic density-gradient centrifugation for blood cell sorting. Biomed Microdevices. 20 (3), 77 (2018).
- Strachan, B. C., Xia, H., Voros, E., Gifford, S. C., Shevkoplyas, S. S. Improved expansion of T cells in culture when isolated with an equipment-free, high-throughput, flow-through microfluidic module versus traditional density gradient centrifugation. Cytotherapy. 21 (2), 234-245 (2019).
- Malvezzi, H., Sharma, R., Agarwal, A., Abuzenadah, A. M., Abu-Elmagd, M. Sperm quality after density gradient centrifugation with three commercially available media: a controlled trial. Reprod Biol Endocrinol. 12, 121 (2014).
- Sun, Y., Sethu, P. Microfluidic adaptation of density-gradient centrifugation for isolation of particles and cells. Bioengineering (Basel). 4 (3), 67 (2017).
- Karttunen, J., et al. Precipitation-based extracellular vesicle isolation from rat plasma co-precipitate vesicle-free microRNAs. J Extracell Vesicles. 8 (1), 1555410 (2019).
- De Sousa, K. P., et al. Isolation and characterization of extracellular vesicles and future directions in diagnosis and therapy. Wiley Interdiscip Rev Nanomed Nanobiotechnol. 15 (1), e1835 (2023).
- Sidhom, K., Obi, P. O., Saleem, A. A review of exosomal isolation methods: Is size exclusion chromatography the best option. Int J Mol Sci. 21 (18), 6466 (2020).
- Coughlan, C., et al. Exosome isolation by ultracentrifugation and precipitation and techniques for downstream analyses. Curr Protoc Cell Biol. 88 (1), e110 (2020).
- Konoshenko, M. Y., et al. Isolation of extracellular vesicles from biological fluids via the aggregation-precipitation approach for downstream miRNAs detection. Diagnostics (Basel). 11 (3), 384 (2021).
- Drozdz, A., et al. Low-vacuum filtration as an alternative extracellular vesicle concentration method: A comparison with ultracentrifugation and differential centrifugation. Pharmaceutics. 12 (9), 872 (2020).
- Akbar, A., Malekian, F., Baghban, N., Kodam, S. P., Ullah, M. Methodologies to isolate and purify clinical grade extracellular vesicles for medical applications. Cells. 11 (2), 186 (2022).
- Chen, J., et al. Review on strategies and technologies for exosome isolation and purification. Front Bioeng Biotechnol. 9, 811971 (2021).
- Helling, A., et al. Investigation of microbial cell deformability by filter cake compressibility using ultrafiltration membranes. Colloids Surf B Biointerfaces. 185, 110626 (2020).
- Bagci, C., et al. Overview of extracellular vesicle characterization techniques and introduction to combined reflectance and fluorescence confocal microscopy to distinguish extracellular vesicle subpopulations. Neurophotonics. 9 (2), 021903 (2022).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750 (2018).
- van Maanen, J. C., et al. A combined western and bead-based multiplex platform to characterize extracellular vesicles. Tissue Eng Part C Methods. 29 (11), 493-504 (2023).
- Xu, D., et al. MicroRNAs in extracellular vesicles: Sorting mechanisms, diagnostic value, isolation, and detection technology. Front Bioeng Biotechnol. 10, 948959 (2022).
- Lee, H., He, X., Le, T., Carnino, J. M., Jin, Y. Single-step RT-qPCR for detection of extracellular vesicle microRNAs in vivo: a time- and cost-effective method. Am J Physiol Lung Cell Mol Physiol. 318 (4), L742-L749 (2020).
- Kim, J. A., et al. Small RNA sequencing of circulating small extracellular vesicles microRNAs in patients with amyotrophic lateral sclerosis. Sci Rep. 13 (1), 5528 (2023).
- Fan, Y., et al. Differential proteomics argues against a general role for CD9, CD81 or CD63 in the sorting of proteins into extracellular vesicles. J Extracell Vesicles. 12 (8), e12352 (2023).
- Silva, A. M., et al. Quantification of protein cargo loading into engineered extracellular vesicles at single-vesicle and single-molecule resolution. J Extracell Vesicles. 10 (10), e12130 (2021).
- Salunkhe, S., Basak Dheeraj, M., Chitkara, D., Mittal, A. Surface functionalization of exosomes for target-specific delivery and in vivo imaging, tracking: Strategies and significance. J Control Release. 326, 599-614 (2020).
- Silva, A. K. A., et al. Development of extracellular vesicle-based medicinal products: A position paper of the group "Extracellular Vesicle translatiOn to clinicaL perspectiVEs - EVOLVE France". Adv Drug Deliv Rev. 179, 114001 (2021).
- Gurunathan, S., Kang, M. H., Jeyaraj, M., Qasim, M., Kim, J. H. Review of the isolation, characterization, biological function, and multifarious therapeutic approaches of exosomes. Cells. 8 (4), 307 (2019).
- Zhang, Y., et al. Exosome: A review of its classification, isolation techniques, storage, diagnostic and targeted therapy applications. Int J Nanomedicine. 15, 6917-6934 (2020).
- Colombo, M., Raposo, G., Thery, C. Biogenesis, secretion, and intercellular interactions of exosomes and other extracellular vesicles. Annu Rev Cell Dev Biol. 30, 255-289 (2014).
- Pisitkun, T., Shen, R. F., Knepper, M. A. Identification and proteomic profiling of exosomes in human urine. Proc Natl Acad Sci U S A. 101 (36), 13368-13373 (2004).
- Zaborowski, M. P., Balaj, L., Breakefield, X. O., Lai, C. P. Extracellular vesicles: Composition, biological relevance, and methods of study. Bioscience. 65 (8), 783-797 (2015).
- Kowkabany, G., Bao, Y. Nanoparticle tracking analysis: An effective tool to characterize extracellular vesicles. Molecules. 29 (19), 4672 (2024).
- Li, H. Metastatic characteristics of SY86B human gastric carcinoma in athymic nude mice. Zhonghua Zhong Liu Za Zhi. 10 (6), 421-423 (1988).
- Zhao, Z., Wijerathne, H., Godwin, A. K., Soper, S. A. Isolation and analysis methods of extracellular vesicles (EVs). Extracell Vesicles Circ Nucl Acids. 2, 80-103 (2021).
- Gandham, S., et al. Technologies and standardization in research on extracellular vesicles. Trends Biotechnol. 38 (10), 1066-1098 (2020).
- van der Vlist, E. J., Nolte-'t Hoen, E. N., Stoorvogel, W., Arkesteijn, G. J., Wauben, M. H. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nat Protoc. 7 (7), 1311-1326 (2012).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. J Extracell Vesicles. 4, 25530 (2015).
- Breitwieser, K., et al. Detailed characterization of small extracellular vesicles from different cell types based on tetraspanin composition by ExoView R100 platform. Int J Mol Sci. 23 (15), 8544 (2022).
- An, H. J., Cho, H. K., Song, D. H., Kee, C. Quantitative analysis of exosomes in the aqueous humor of Korean patients with pseudoexfoliation glaucoma. Sci Rep. 12 (1), 12875 (2022).
- Hefley, B. S., McKay, T. B., Hutcheon, A. E. K., Ciolino, J. B., Karamichos, D. Corneal epithelial-stromal constructs to study differences associated with diabetes mellitus. Exp Eye Res. 248, 110100 (2024).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved