Method Article
Characterizing Extracellular Vesicles from Biological Fluids(생물학적 유체로부터 세포외 소포체 특성화)
요약
여기에서는 인간 피험자의 눈물 및 타액과 같은 생물학적 액체에서 수집된 세포외 소포체(EV)의 특성화 방법을 보여줍니다. 이 방법에 사용된 스캐너는 샘플의 1μL에서 EV의 표현형, 크기 및 총 입자 수를 감지할 수 있습니다.
초록
세포외 소포체(EV)는 세포에서 생성되며 한 세포에서 다른 세포로 생체 분자를 운반하여 세포 간 통신에 참여하는 구조입니다. EV는 신체에서 단거리와 원거리를 이동하는 것으로 나타났으며 조직에 따라 다릅니다. EV는 조직뿐만 아니라 눈물, 타액, 뇌척수액, 혈액 등과 같은 거의 모든 체액에서도 발견될 수 있습니다. EV는 눈물과 타액에서 비침습적으로 수집할 수 있지만 한 번에 소량만 수집할 수 있어 단백질을 분석하기에 충분한 EV를 얻는 데 문제가 발생할 수 있습니다. 이 논문에서 논의된 스캐너는 이 문제에 대한 해결책을 제공하는 나노 입자 분석기로, 이를 통해 1μL의 생물학적 유체에서 EV의 표현형, 크기 및 총 입자 수를 특성화하고 연구할 수 있습니다. 이 프로토콜은 환자로부터 추출하기 어려운 소량의 샘플에서 EV에 대한 지식을 확장할 것입니다. 이를 통해 환자의 편안함을 높이고 다양한 질병 및 장애에 대한 새로운 치료 표적을 식별할 수 있습니다.
서문
세포는 세포 간 통신에서 중요한 역할을 하는 세포외 소포체(EV)의 방출을 포함한 다양한 신호 전달 메커니즘을 통해 이웃 세포와 통신합니다. EV는 DNA, RNA 및 단백질과 같은 유전 화물을 한 세포에서 다른 세포로 전달하여 세포 간 통신에 참여합니다 1,2,3,4,5. 현재 EV에는 엑소좀(exosomes), 미세소포체(microvesicles), 자가사멸체(apoptotic body)의 세 가지 범주가 있으며, 이들은 그 크기에 따라 달라집니다. 엑소좀은 직경이 30-150nm인 가장 작은 엑소좀(6,7,8)이며, 엔도솜 막계(endosomal membrane system) 9,10,11로 형성됩니다(그림 1). 미세소포는 100-1000 nm 12,13,14 범위이고 원형질막에서 싹이 11,12,13 떨어져 나온다는 점에서 엑소좀보다 큽니다(그림 1). 자가사멸체(Apoptotic body)는 EV 중 가장 크고 범위는 1000-5000nm 12,14,15이며 원형질막 12,16,17에서도 싹을 틔웁니다(그림 1). 크기 외에도 EV는 밀도, CD63, CD81, CD9와 같은 분자 마커 및 생물 발생 메커니즘18을 포함하는 다른 생물물리학적 특성을 기반으로 분류할 수 있습니다. EV는 인접한 세포 사이의 짧은 거리와 몸 전체의 장거리를 이동할 수 있습니다 19,20,21,22,23. EV는 혈액24,25,26, 뇌척수액 27,28,29, 눈물 30,31,32, 타액33,34,35와 같은 생물학적 액체에서 찾을 수 있습니다.
현재까지 초원심분리는 샘플36,37,38에서 EV를 분리하는 가장 잘 알려진 방법 중 하나입니다. 이 방법은 여러 차례의 원심분리와 초원심분리가 필요하며, 이는 EV를 세포와 세포 파편으로부터 분리하는 속도를 높여 수행할 수 있습니다. 이 방법은 펠릿 셀을 사용한 저속으로 시작하여 더 큰 소포를 제거하기 위해 중간 속도로 시작할 수 있으며, 마지막으로 EV18을 펠릿화하기 위한 초원심분리 단계로 시작할 수 있습니다. 초원심분리는 절연을 위한 최상의 방법으로 간주되지만 EV 39,40,41의 형태를 변경한다는 점에서 몇 가지 제한 사항이 여전히 존재합니다. EV를 분리하는 데 사용되는 또 다른 기법은 유세포 분석으로, 다중 시점 및 종말점 평가와 고처리량 단일 EV 분석의 이점을 강조합니다. 그러나 유세포 분석의 한계에는 공극의 막힘 및 약한 신호가 포함되지만 이에 국한되지 않습니다. 사용되는 또 다른 접근 방식은 기울기 원심분리(gradient centrifugation)로, 밀도가 다른 재료를 사용하여 EV로 원심분리하고 초원심분리에 비해 EV를 더 잘 분리할 수 있습니다. 이 기법은 분리를 개선하지만 노동 집약적이고 시간이 많이 소요되며 상당한 시료 손실로 이어질 수 있습니다. 또한 강수 및 여과를 사용하여 EV를 격리할 수도 있습니다. 이 두 가지 기법 모두 간단하고 빠르지만 둘 다 시료 오염으로 이어질 수 있습니다. EV를 분리하는 몇 가지 기술이 있지만 각 기술에는 다음과 같은 장점과 한계가 있습니다(표 1) 42,43,44,45,46,47,48,49,50,51,52,53,54, 55, 56,57,58,59,60,61,62,63,64,65,66,67.
EV가 분리되면 분자 분석을 수행하여 EV를 특성화할 수 있습니다. 웨스턴 블롯은 표면 및 화물 단백질 발현 68,69,70을 찾기 위한 일반적인 분석법이며, 중합효소 연쇄 반응(PCR)은 EV에 대한 miRNA 발현 71,72,73 74,75,76에 사용됩니다. 이러한 분석은 확립되어 있으며 흥미로운 결과를 생성할 수 있습니다. 이러한 방법의 한계는 판독값77을 얻기 위해 EV에서 많은 양의 단백질 또는 RNA가 필요하다는 것인데, 이는 우선 부피 또는 EV 농도가 작은 샘플의 경우 문제가 됩니다.
이 논문에서 논의된 나노 입자 분석기를 통해 사용자는 표 1 및 표 2 78,79,80,81,82,83,84,85,86,87,88,89,90에 명시된 많은 제한 사항을 극복할 수 있습니다 . 이 방법은 EV의 감소된 수율을 극복하는 데 도움이 되는 절연 기술의 활용을 필요로 하지 않습니다. 또한 이 방법을 통해 사용자는 1μL의 작은 시료 부피에서 표면 및 화물 단백질, 총 EV 수 및 EV 크기를 분석할 수 있습니다. 이는 테트라스파닌 항체, CD63, CD81 및 CD9가 포함된 항체 마이크로어레이를 사용하는 회사에서 제공하는 테트라스파닌 칩을 사용하여 수행되며, 그림 2와 같이 솔루션에서 EV를 식별합니다. 형광 항체는 EV의 존재를 확인할 뿐만 아니라 오염 입자가 결과를 왜곡하는 것을 방지합니다.
이 기술의 전반적인 목표는 소량의 샘플에서 EV를 분석할 뿐만 아니라 EV를 분석하는 데 시간이 덜 걸리는 방법을 제공하는 것입니다. 이 나노 입자 분석기를 사용하면 1μL의 샘플에서 크기, 총 입자 수 및 표면 단백질을 분석할 수 있으며, 이는 눈물 및 타액과 같은 생물학적 유체에 이상적입니다.
프로토콜
설명된 모든 연구는 헬싱키 선언을 준수했습니다. 연구에 포함되기 전에 각 피험자로부터 서면 동의를 받았습니다. 연방 및 기관 지침에 따라 Aarhus University Hospital(1-10-72-77-14) 및 Dean McGee Institute(1576837-2)의 IRB(Institutional Review Board) 승인을 받았습니다. 처리하기 전에 모든 눈물 및 타액 샘플은 비식별화되었습니다. 모든 연구는 North Texas Regional Institutional Review Board(#2020-030)에서 검토 및 승인되었습니다. 다음 프로토콜은 모든 지침을 준수하며 위에서 언급한 바와 같이 승인되었습니다.
1. 1일차: 시료 준비 및 배양
- 생물학적 유체 샘플(예: 피험자의 눈물과 타액)을 수집하거나 냉동실에서 샘플을 해동합니다.
- 눈물: 유리 모세관 튜브를 사용하여 외측 반월판에서 수동적으로 눈물을 수집합니다.
- 유리 모세혈관을 아래 눈꺼풀에 놓고 각막을 만지지 않도록 주의합니다.
- 10분 이내에 10μL의 눈물을 수집합니다.
- 눈물은 즉시 사용하거나 -20 °C에서 보관하십시오.
- 타액: 1.5mL 마이크로 원심분리기 튜브에서 수동 침에서 타액을 수집합니다.
- 기증자가 입에 침을 고일 수 있도록 합니다.
- 기증자에게 깔때기로 튜브에 침을 흘리도록 요청하십시오.
- 즉시 타액을 사용하거나 -20 °C에서 보관하십시오.
- 눈물: 유리 모세관 튜브를 사용하여 외측 반월판에서 수동적으로 눈물을 수집합니다.
- 테트라스파닌 칩을 칩 와셔 플레이트에 15분 동안 넣은 후 s를 추가합니다.amp실온에 도달하도록 합니다. 칩을 우물에 넣는 동안 숫자가 바닥면이 위를 향하도록 하고 칩이 우물의 측벽에 닿지 않는지 확인하십시오.
- 칩을 추적하기 위해 노트북에 ID 번호와 칩 번호를 기록하십시오.
- 키트와 함께 제공되는 USB(범용 직렬 버스)를 연결하고 관련 키트 번호가 있는 폴더를 추출하여 선택한 지정된 영역에 저장합니다.
- 1.5mL 튜브에 99μL의 용액 B를 피펫으로 넣습니다.
- 이전 단계에서 준비한 1.5mL 튜브에 1μL의 생물학적 유체를 추가합니다.
- 튜브를 회전시킵니다.
- 1.6단계에서 용액 70μL를 취하고 테트라스파닌 칩에 피펫팅합니다.
- 샘플이 증발하는 것을 방지하기 위해 칩 세척판에 필름을 놓습니다.
- 진동이 없는 벤치에서 실온(RT)에서 16시간 동안 배양합니다.
2. 2일차: 칩 세척
- 칩 세탁기를 켭니다.
- 플레이트에서 1.9단계의 필름을 제거합니다.
- 접시를 칩 와셔에 넣고 제자리에 고정되었는지 확인하고 뚜껑을 닫습니다.
알림: 플레이트는 제자리에 올바르게 고정되면 쉽게 움직이지 않습니다. - 칩 와셔에서 CW-TETRA v0 옵션을 누릅니다.
- 시작 행과 세척할 행 수를 선택합니다.
- 칩 세탁기에서 계속 을 누릅니다.
- 칩을 세척하는 동안 다음과 같이 튜브에 항체가 포함된 차단 용액을 준비합니다.
- 칩당 300μL의 블로킹 용액을 준비합니다.
- 0.6 μL 또는 칩당 각 항체 추가(차단 용액에 추가하기 전에 와류 및 스핀 다운 항체)
- Vortex 차단 솔루션 + 항체 칵테일.
알림: 항체 안정성을 보장하기 위해 차단액 + 항체 칵테일을 빛으로부터 덮습니다. 이것은 사용 당일에 이루어져야 합니다.
- 칩 와셔가 짹삑 소리를 내면 칩 위에 250μL의 차단 용액 + 항체 칵테일을 추가합니다.
- 덮개를 닫고 계속을 누릅니다.
알림: 칩 와셔는 이제 몇 번의 추가 세척 및 헹굼과 함께 1-h 배양을 수행합니다. - 칩 와셔가 짹짹 소리를 내면 칩 세척판을 제거하고 칩을 램프 위로 이동합니다.
- 플레이트를 칩 와셔에 다시 넣고 제자리에 고정되었는지 확인합니다. 덮개를 닫고 계속을 누릅니다.
- 칩 와셔가 프로그램을 완료하면 플레이트를 제거하고 칩을 종이 타월 위에 놓습니다. 빛에서 칩을 덮으십시오.
3. 2일차: 칩 스캐닝
- 스캐너를 켜고 스캔 소프트웨어를 엽니다.
참고: 스캐너는 오류가 있는지 또는 스캔을 계속할 수 있는지 여부를 나타내기 위해 자체 검사를 거칩니다. - 폴더 저장을 선택하고 파일을 저장할 위치를 선택합니다.
- ChipFile 폴더를 선택하고 1.4단계에서 저장한 폴더를 선택합니다.
- 드롭다운 옵션을 선택하여 각 칩을 선택하고 척의 칩 위치에서 칩을 찾습니다. 스캔해야 하는 모든 칩에 대해 이 단계를 반복합니다.
- Chip Position 아래의 세 사각형을 클릭하여 형광을 켭니다. 사각형은 각각 노란색, 파란색, 빨간색으로 바뀝니다.
- 뚜껑을 아래로 눌러 척 뚜껑을 제거하고 당깁니다.
- 칩을 척에 놓고 단단히 고정되었는지 확인합니다.
- 구멍에 못을 넣어 척 덮개를 다시 끼우고 아래로 누른 다음 덮개를 밀어 제자리에 고정합니다.
- 척을 무대에 놓고 제자리에 고정되었는지 확인합니다.
- Scan Chips(칩 스캔)를 선택합니다.
- 확인을 선택합니다.
- 스테이지가 기계 안으로 완전히 이동되면 문을 닫습니다.
참고: 단일 칩을 스캔하는 데 약 12분이 걸립니다. - 프로그램이 칩 스캔을 완료하면 도어를 열어 스테이지를 꺼내고 스캔이 성공했는지 확인합니다.
- 스캔에 성공한 경우 칩을 버리고 다음 칩 세트를 척에 놓거나 빈 척을 스테이지로 되돌립니다. 그런 다음 소프트웨어를 종료하고 완료되면 컴퓨터를 끕니다.
- 스캔에 성공하지 못한 경우 오류 코드를 확인하고 오류를 수정하십시오.
4. 데이터 처리
- 분석 소프트웨어를 엽니다.
- Prescan Data(데이터 사전 검사)를 선택하고 Day 2 step 3.2에서 스캔한 폴더를 선택합니다.
- 다음을 선택합니다.
- 셀을 클릭하고 샘플 ID를 입력하여 Sample Name 열의 칩에 샘플 이름을 지정합니다.
- 다음으로, 채널 이름 열에서 테트라스패닌에 레이블을 지정합니다. 빨간색 = CD63, 녹색 = CD81, 파란색 = CD9.
- 다음을 선택합니다.
- Select Spot(스폿 선택) 그룹에서 High CV(높은 CV)를 선택하여 드롭다운 상자를 검사하고 하나 또는 두 개의 스폿을 비활성화하여 각 칩에 대한 높은 CV를 끕니다.
- 그런 다음 High Count 를 선택하고 High-Count 경고를 해제해 봅니다.
참고: 일부 샘플의 개수가 너무 많을 수 있지만 여전히 분석할 수 있습니다. - 드롭다운 메뉴에서 All Spots(모든 스폿 )를 선택하고 Spot Montage(스폿 몽타주)를 선택한 다음 각 칩에 존재할 수 있는 변형이나 긁힌 자국이 있는지 육안으로 확인합니다.
- 다음을 선택합니다.
- CD63 채널에서 최소값을 300으로 설정하여 Avg % Included 행에서 빨간색 셀을 제거합니다. 노란색 세포는 괜찮으며 CD81 채널로 진행합니다. 최소값을 300으로 설정해도 빨간색 셀이 노란색 또는 흰색으로 변경되지 않으면 문제 해결을 위해 4.14단계로 진행합니다.
- CD81 채널에서 최소값을 300으로 설정하여 Avg % Included 행의 빨간색 셀을 제거합니다. 노란색 세포는 괜찮으며 CD9 채널로 진행합니다. 최소값을 300으로 설정해도 빨간색 셀이 노란색 또는 흰색으로 변경되지 않으면 문제 해결을 위해 4.14단계로 진행합니다.
- CD9 채널에서 최소값을 400으로 설정하여 Avg % Included 행에서 빨간색 셀을 제거합니다. 노란색 셀은 괜찮으며 4.15단계로 진행합니다. 최소값을 400으로 설정해도 빨간색 셀이 노란색 또는 흰색으로 변경되지 않으면 문제 해결을 위해 4.14단계로 진행합니다.
- 문제 해결
- 최소값을 100 단위 늘립니다.
- 4.14.1단계가 작동하지 않으면 작업할 칩 선택 섹션에서 칩을 선택하고 칩을 선택합니다. Particle Count(파티클 수 )를 선택하고 파티클 수가 많은 지점을 비활성화합니다. Intensity/Size(강도/크기)를 선택하고 All Chips(모든 칩) 를 선택한 다음 문제가 해결되는지 확인합니다.
- 4.14.2단계가 작동하지 않으면 작업할 칩 선택 섹션에서 칩을 선택하고 칩을 선택합니다. Single-Chip Cutoffs를 선택하고 최소값을 100 단위 늘립니다. 그런 다음 모든 칩을 선택하여 빨간색 셀이 이제 노란색인지 확인합니다. 그래도 문제가 해결되지 않으면 Avg % Included 셀이 노란색 또는 흰색이 될 때까지 이 단계를 반복합니다.
- 다음을 선택합니다.
- IM 채널을 선택 취소합니다.
- 모든 파티클을 포함하도록 오른쪽 하단 모서리의 히트맵 값을 설정하고 Add Plot to Report를 선택합니다.
- 드롭다운 상자에서 최대 수를 조정하고 값을 수동으로 입력합니다.
- 그런 다음 Capture Probes(프로브 캡처) 아래 상단 중앙에 있는 드롭다운 메뉴를 선택하고 CD63을 선택합니다.
- Add Plot to Report를 선택하여 총 파티클 수를 보고서에 추가합니다.
- 분석 모드에서 드롭다운 메뉴를 선택하고, Colocalization(공동 국소화)을 선택하고, Add Plot to Report(보고서에 플롯 추가)를 선택합니다.
- 다음으로, 크기를 선택하고, 뷰를 플롯으로 변경을 선택하고, 보고서에 플롯 추가를 선택합니다.
- 마지막으로 이미지를 선택하고 보고서에 플롯 추가를 선택합니다.
- CD81 및 CD9에 대해 4.18-4.22단계를 반복하여 매번 IM 채널을 선택 취소해야 합니다.
- 모든 항목이 보고서에 추가되면 보고서 내보내기를 선택하고, 올바른 파일에 저장되었는지 확인하고, 폴더를 선택합니다.
참고: Excel 파일 및 이미지가 생성되었으며 이제 추가 분석에 사용할 수 있습니다.
결과
칩의 스폿을 분석할 때 CD63, CD81 및 CD9 채널에서 형광 표지된 EV를 관찰하여 최적의 EV 농도를 목표로 하고 MIgG 채널30,91은 그림 3A에서 볼 수 있는 제어 채널이므로 검은색으로 유지되어야 합니다. CD81은 생물학적 유체에 대해 감소될 것입니다. CD63, CD81 및 CD9 채널에 형광이 없으면 EV가 없는 것입니다(그림 3B). 칩이 과포화되지 않도록 주의하십시오(그림 3C). 이로 인해 분석 소프트웨어가 EV의 크기, 총 입자 수 및 표현형의 정확도를 계산하기 어렵게 됩니다. 최적의 농도 지점에서 분석 소프트웨어는 EV의 크기(50-200nm), 총 입자 수 및 테트라스파닌 발현을 측정할 수 있으며(그림 4) 추가 분석을 위해 개별 스프레드시트로 내보낼 수 있습니다. 테트라스파닌 분석에는 EV의 표면 단백질의 공동 국소화가 포함됩니다. 공동 국소화는 "/"로 표시됩니다(예: "CD63/CD81"). 이것은 CD63과 CD81이 결합된다는 것을 의미하지 않습니다. 이는 CD63과 CD81이 모두 EV 표면에 위치한다는 것을 의미합니다.
이러한 결과는 건강한 샘플과 병든 샘플 간에 귀중한 통찰력을 제공합니다. 건강한 샘플 또는 질병에 걸린 샘플이 더 많은 EV, 더 크거나 작은 EV, EV의 표현형을 생성하는지 여부를 결정할 수 있습니다. 이러한 특성의 일부 또는 전부는 질병의 생물 발생 및/또는 진행에 중요한 역할을 할 수 있습니다. 이러한 결과를 통해 테트라스파닌 수치가 없거나 증가하는지 확인할 수 있으며, 이는 세포 과정과 EV의 형성에 대한 통찰력을 제공할 수 있습니다.
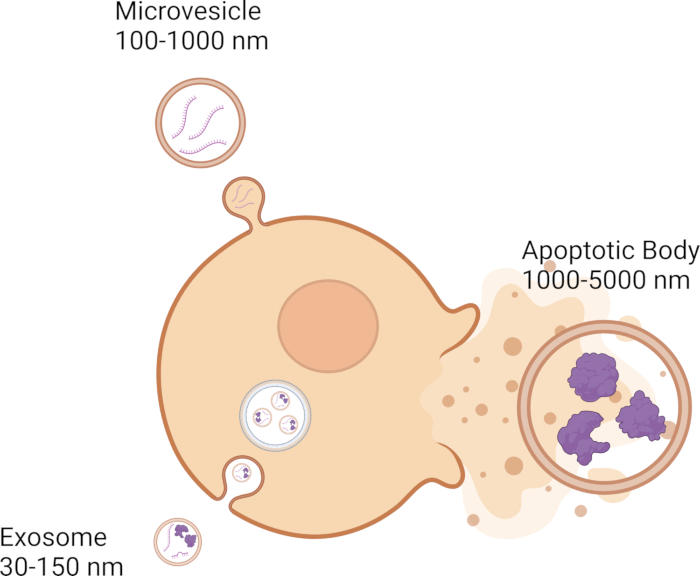
그림 1: 다양한 EV의 유형과 크기. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
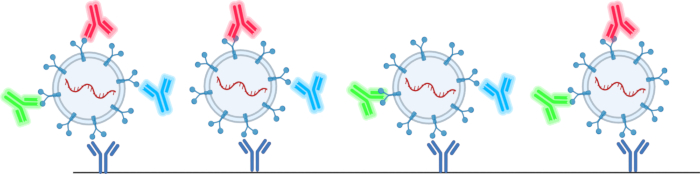
그림 2: 테트라스파닌 칩이 EV를 캡처하고 감지하는 방법의 개략도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
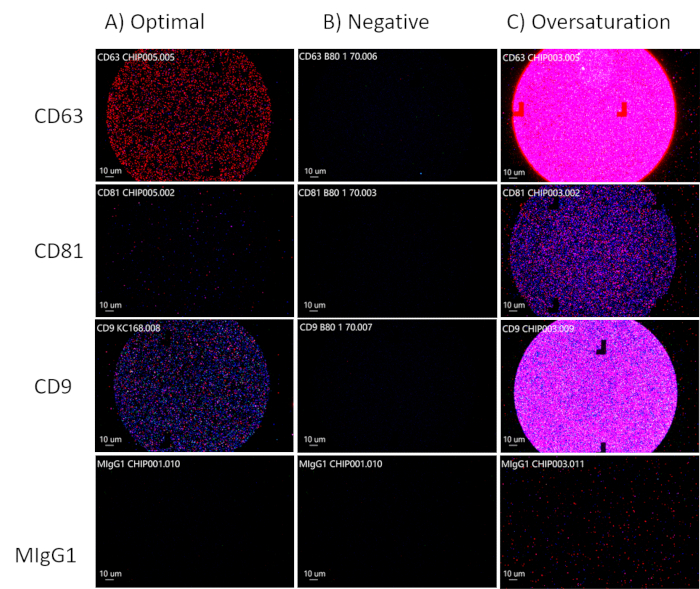
그림 3: EV 농도 최적화. (A) EV의 최적 농도. (B) 음성 결과: EV가 없습니다. (C) EV의 과포화. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
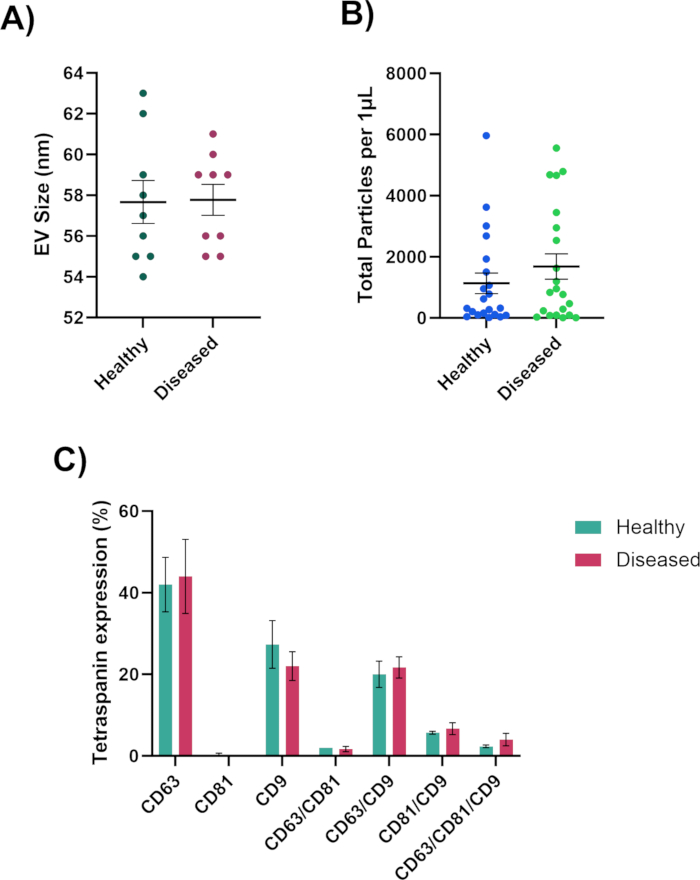
그림 4: 나노 입자 분석기로 생성된 데이터. (A) 나노미터 단위로 측정한 EV 직경 크기. (B) EV의 총 입자 수. (C) EV 공동 국소화 표현형. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 기술 | 장점 | 제한 |
| 초원심분리기 | 고순도42,43 | EV 형태소 변경42,44 |
| 균질성42,45 | 대용량필요 42,44,46 | |
| 기능42,43 | 비싼42,43 | |
| 유세포 분석 | 여러 시점에서 표본 평가47 | 약한 신호48,49 |
| 다중 엔드포인트47,50 | 모공 막힘51 | |
| 고처리량 단일 EV 분석52,53 | ||
| Density Gradient Centrifugation | 고순도 샘플 생산54 | 노동 집약적55,56 |
| 다양한 샘플55,57 | 시간이 많이 소요됨55,58 | |
| 상당한 수율 손실54 | ||
| 강수량 | 심플59,60 | 오염36,44,61 |
| 빠른61,62 | 비특이적63 | |
| 높은 수율44,59,62 | ||
| 중성 pH36 | ||
| 여과 | 심플64 | 트랩 엑소좀:65 |
| 패스트64 | 큰 수포 손상36 | |
| 저렴한64 | 필터 케이크66,67 | |
| 엑소좀 막힘65 |
표 1: EV 절연 기술의 장점과 한계. EV의 장점과 제한 사항을 포함하여 EV를 격리하는 다양한 방법 목록입니다.
| 기술 | 장점 | 제한 |
| 동적 광 산란 | 1nm에서 6μM 크기의 입자 측정78 | 큰 크기 범위의 복잡한 엑소좀 샘플측정에는 적합하지 않음 79 |
| 하한은 10nm이며 단분산 시스템에 적합합니다79 | 오염된 단백질과 엑소좀을 구별할 수 없음79 | |
| 투과 전자 현미경 (TEM) | 엑소좀의 형태 관찰79,80 | 복잡한 시료 전처리79 |
| 내부 구조관찰 81 | 과장된 형광 신호로 인해 크기와 모양에 따라 엑소좀을 구별할 수 없음82 | |
| 나노 입자 추적 분석 | 10 nm에서 2 μM 범위에서 엑소좀의 농도, 크기 및 크기 분포 측정78 | EV를 단백질 응집체 및 기타 오염 물질과 구별할 수 없음83 |
| 빠른 시료 준비 및 측정78,84 | 고가의 NTA 악기85 | |
| 샘플은 기본 형태84로 회수할 수 있습니다. | 진동에 민감함85 | |
| 웨스턴 블롯(WB) | 마커 단백질을 정성적 및 정량적으로 분석할 수 있음79,86 | 복잡하고 시간이 많이 걸린다79 |
| 세포 배양 배지의 엑소좀 분석79 | EV 분리물에는 지단백질 및 기타 오염 물질이 포함될 수 있습니다86 | |
| 유세포 분석 | 더 높은 감도와 고해상도 이미징 | 400nm의 검출 한계로 시간이 많이 걸리고 힘들다79,88 |
| 염색된 엑소좀과 격납 장치 구별87 | ||
| 낮은 시료 농도필요 79 | 광 신호는 정확도와 해상도를 방해합니다88 | |
| 엑소뷰 | 엑소좀에서 테트라스파닌(CD9, CD63, CD81) 측정89 | 더 큰 EV 크기를 측정하지 않음75 |
| EV 화물 단백질측정 90 |
표 2: EV 특성화 기술의 장점과 한계.
토론
이 프로토콜에서 가장 중요한 단계는 EV의 최적 농도가 달성되도록 하는 것입니다. 판독값을 얻으려면 충분한 EV가 있어야 하지만 칩을 과포화시킬 EV가 너무 많아서는 안 됩니다. 최적의 EV 농도를 결정하는 가장 좋은 방법은 1μL의 샘플로 최적화 실행을 수행하고 농도를 조정해야 하는지 확인하는 것입니다. 또 다른 중요한 단계는 샘플에 세포 파편이 풍부한지 확인하는 것인데, 이는 분석 소프트웨어에서 칩의 큰 덩어리를 확인하여 확인할 수 있습니다. 샘플에 세포 파편이 있는 경우 샘플의 간단한 원심분리 또는 필터링으로 이 문제를 해결할 수 있습니다.
추가적으로 중요한 단계는 칩이 우물의 벽에 닿지 않도록 하고 램프에 칩을 배치할 때 칩의 중앙 영역과의 접촉을 피하는 것입니다. 스캐너는 칩 중앙의 사각형에서 읽으므로 EV를 방해하지 않도록 집게로 해당 영역을 만지지 않는 것이 중요합니다.
이 방법에는 기기에서 감지하기 위한 EV의 농도가 충분하지 않은 것과 같은 몇 가지 제한 사항이 있습니다. 농도는 샘플을 건조하거나 농축기 튜브를 사용하여 개선할 수 있습니다. 또 다른 제한 사항은 분석 소프트웨어가 크기 측정75에서 일부 미세 소포와 모든 세포 사멸체를 제외하고 50-200nm 범위의 EV만 측정한다는 것입니다.
분리하는 방법에는 여러 가지가 있습니다(표 1 42,43,44,45,46,47,48,49,50,51,52, 53,54,55,56,57,58,59,60, 61,62,63,64,65,66,67) EV를 분석하고(표 2 78,79,80,81,82,83,84,85, 86,87,88,89,90), EV 분리를 위한 현재 황금 표준은 초원심분리 36,37,38로 웨스턴 블롯 68,69,70 및 PCR 71,72,73,74와 같은 분석을 위해 세포를 펠렛화합니다.75,76. 이 프로토콜은 큰 샘플에 잘 작동하지만 42,44,46 생물학적 유체를 얻는 것은 어려울 수 있으며 종종 한 번에 소량의 샘플만 수집할 수 있어 초원심분리에 적합하지 않습니다. 대조적으로, 이 나노 입자 분석기를 사용하면 사용자가 1μL의 샘플에서 EV의 크기, 총 입자 수 및 표현형과 같은 귀중한 데이터를 생성할 수 있으므로 생물학적 유체에 이상적입니다. 추출하기 어려운 소량의 샘플에서 EV에 대한 지식을 확장할 수 있으며, 이를 통해 환자의 편안함을 높이고 다양한 질병 및 장애에 대한 잠재적인 치료 대상을 찾을 수 있습니다.
공개
저자는 공개할 경쟁 재정적 이해관계나 기타 이해 상충이 없습니다.
감사의 말
자금 지원(EY031316 및 EY034714)에 대해 NIH에 감사드립니다. 또한 실험실 공간을 제공해 주신 UNTHSC와 NTERI에도 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| ChipWasher 100 | NanoView | EV-CW100 | Incubates, washes, rinses and dries the tetraspanin chips. This current model is no longer available. Price at time of purchase: $9,995.00 |
| ExoView Analyzer software | NanoView | N/A | Analyzes the chip informations and produces excel files for further analysis. No longer available. |
| ExoView R100 | NanoView | EV-R100 | Used to scan the tetraspanin chips at 3 wavelengths. This current model is no longer available. Price at time of purchase: $110,000.00 |
| ExoView Scanner software | NanoView | N/A | Scans the chips at 3 different wavelengths. No longer available. |
| Human Tetraspanin Kits | Unchained Labs | EV-TETRA-C | Includes 8 tetraspanin chips, Incubation Solution, Blocking Solution, CD63 antibody, CD81 antibody, CD9 antibody, Solution A, Solution B, USB, and plate cover. |
참고문헌
- Abels, E. R., Breakefield, X. O. Introduction to extracellular vesicles: Biogenesis, RNA cargo selection, content, release, and uptake. Cell Mol Neurobiol. 36 (3), 301-312 (2016).
- Radler, J., Gupta, D., Zickler, A., Andaloussi, S. E. Exploiting the biogenesis of extracellular vesicles for bioengineering and therapeutic cargo loading. Mol Ther. 31 (5), 1231-1250 (2023).
- Gupta, D., Zickler, A. M., El Andaloussi, S. Dosing extracellular vesicles. Adv Drug Deliv Rev. 178, 113961 (2021).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63 (2018).
- Zhang, H., et al. Exosome-induced regulation in inflammatory bowel disease. Front Immunol. 10, 1464 (2019).
- Barzin, M., et al. Application of plant-derived exosome-like nanoparticles in drug delivery. Pharm Dev Technol. 28 (5), 383-402 (2023).
- Zhou, C., et al. Stem cell-derived exosomes: emerging therapeutic opportunities for wound healing. Stem Cell Res Ther. 14 (1), 107 (2023).
- Kalluri, R., LeBleu, V. S. The biology, function, and biomedical applications of exosomes. Science. 367 (6478), aau6977 (2020).
- Mondal, J., et al. Hybrid exosomes, exosome-like nanovesicles and engineered exosomes for therapeutic applications. J Control Release. 353, 1127-1149 (2023).
- van Niel, G., D'Angelo, G., Raposo, G. Shedding light on the cell biology of extracellular vesicles. Nat Rev Mol Cell Biol. 19 (4), 213-228 (2018).
- Paul, N., Sultana, Z., Fisher, J. J., Maiti, K., Smith, R. Extracellular vesicles- crucial players in human pregnancy. Placenta. 140, 30-38 (2023).
- Zaldivia, M. T. K., McFadyen, J. D., Lim, B., Wang, X., Peter, K. Platelet-derived microvesicles in cardiovascular diseases. Front Cardiovasc Med. 4, 74 (2017).
- Tamasi, V., Nemeth, K., Csala, M. Role of extracellular vesicles in liver diseases. Life (Basel). 13 (5), 1117 (2023).
- Fu, Y., et al. Emerging understanding of apoptosis in mediating mesenchymal stem cell therapy. Cell Death Dis. 12 (6), 596 (2021).
- Santavanond, J. P., Rutter, S. F., Atkin-Smith, G. K., Poon, I. K. H. Apoptotic bodies: Mechanism of formation, isolation and functional relevance. Subcell Biochem. 97, 61-88 (2021).
- Yu, L., et al. Apoptotic bodies: bioactive treasure left behind by the dying cells with robust diagnostic and therapeutic application potentials. J Nanobiotechnology. 21 (1), 218 (2023).
- Momen-Heravi, F., Getting, S. J., Moschos, S. A. Extracellular vesicles and their nucleic acids for biomarker discovery. Pharmacol Ther. 192, 170-187 (2018).
- Liu, Y. J., Wang, C. A review of the regulatory mechanisms of extracellular vesicles-mediated intercellular communication. Cell Commun Signal. 21 (1), 77 (2023).
- Grange, C., Bussolati, B. Extracellular vesicles in kidney disease. Nat Rev Nephrol. 18 (8), 499-513 (2022).
- Cocucci, E., Meldolesi, J. Ectosomes and exosomes: shedding the confusion between extracellular vesicles. Trends Cell Biol. 25 (6), 364-372 (2015).
- Iannotta, D. A. A., Kijas, A. W., Rowan, A. E., Wolfram, J. Entry and exit of extracellular vesicles to and from the blood circulation. Nat Nanotechnol. 19 (1), 13-20 (2024).
- Saint-Pol, J., Gosselet, F., Duban-Deweer, S., Pottiez, G., Karamanos, Y. Targeting and crossing the blood-brain barrier with extracellular vesicles. Cells. 9 (4), 851 (2020).
- Alberro, A., Iparraguirre, L., Fernandes, A., Otaegui, D. Extracellular vesicles in blood: Sources, effects, and applications. Int J Mol Sci. 22 (15), 8163 (2021).
- Nieuwland, R., Siljander, P. R. A beginner's guide to study extracellular vesicles in human blood plasma and serum. J Extracell Vesicles. 13 (1), e12400 (2024).
- Thangaraju, K., Neerukonda, S. N., Katneni, U., Buehler, P. W. Extracellular vesicles from red blood cells and their evolving roles in health, coagulopathy and therapy. Int J Mol Sci. 22 (1), 153 (2020).
- Li, C., et al. Cerebrospinal fluid-derived extracellular vesicles after spinal cord injury promote vascular regeneration via PI3K/AKT signaling pathway. J Orthop Translat. 39, 124-134 (2023).
- Deng, Y., et al. Phosphoproteome analysis of cerebrospinal fluid extracellular vesicles in primary central nervous system lymphoma. Analyst. 148 (15), 3594-3602 (2023).
- Hirschberg, Y., et al. Proteomic comparison between non-purified cerebrospinal fluid and cerebrospinal fluid-derived extracellular vesicles from patients with Alzheimer's, Parkinson's and Lewy body dementia. J Extracell Vesicles. 12 (12), e12383 (2023).
- Hefley, B. S., et al. Revealing the presence of tear extracellular vesicles in Keratoconus. Exp Eye Res. 224, 109242 (2022).
- Cross, T., et al. RNA profiles of tear fluid extracellular vesicles in patients with dry eye-related symptoms. Int J Mol Sci. 24 (20), 15390 (2023).
- Ma, H., et al. Metabolic signatures of tear extracellular vesicles caused by herpes simplex keratitis. Ocul Surf. 31, 21-30 (2024).
- Han, P., Bartold, P. M., Ivanovski, S. The emerging role of small extracellular vesicles in saliva and gingival crevicular fluid as diagnostics for periodontitis. J Periodontal Res. 57 (1), 219-231 (2022).
- Han, P., Li, X., Wei, W., Ivanovski, S. Saliva diagnosis using small extracellular vesicles and salivaomics. Methods Mol Biol. 2588, 25-39 (2023).
- Reseco, L., et al. Characterization of extracellular vesicles from human saliva: Effects of age and isolation techniques. Cells. 13 (1), 95 (2024).
- Tiwari, S., Kumar, V., Randhawa, S., Verma, S. K. Preparation and characterization of extracellular vesicles. Am J Reprod Immunol. 85 (2), e13367 (2021).
- Tomiyama, E., Fujita, K., Nonomura, N. Urinary extracellular vesicles: Ultracentrifugation method. Methods Mol Biol. 2292, 173-181 (2021).
- Xu, K., Jin, Y., Li, Y., Huang, Y., Zhao, R. Recent progress of exosome isolation and peptide recognition-guided strategies for exosome research. Front Chem. 10, 844124 (2022).
- Brennan, K., et al. A comparison of methods for the isolation and separation of extracellular vesicles from protein and lipid particles in human serum. Sci Rep. 10 (1), 1039 (2020).
- Gao, J., et al. Recent developments in isolating methods for exosomes. Front Bioeng Biotechnol. 10, 1100892 (2022).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Ansari, F. J., et al. Comparison of the efficiency of ultrafiltration, precipitation, and ultracentrifugation methods for exosome isolation. Biochem Biophys Rep. 38, 101668 (2024).
- Patel, G. K., et al. Comparative analysis of exosome isolation methods using culture supernatant for optimum yield, purity and downstream applications. Sci Rep. 9 (1), 5335 (2019).
- Stam, J., Bartel, S., Bischoff, R., Wolters, J. C. Isolation of extracellular vesicles with combined enrichment methods. J Chromatogr B Analyt Technol Biomed Life Sci. 1169, 122604 (2021).
- Le Roy, A., et al. AUC and small-angle scattering for membrane proteins. Methods Enzymol. 562, 257-286 (2015).
- Chhoy, P., Brown, C. W., Amante, J. J., Mercurio, A. M. Protocol for the separation of extracellular vesicles by ultracentrifugation from in vitro cell culture models. STAR Protoc. 2 (1), 100303 (2021).
- McDorman, K. S., Chan, C., Rojko, J., Satterwhite, C. M., Morrison, J. P. . Chapter 7 - Special Techniques in Toxicologic Pathology. Haschek and Rousseaux's Handbook of Toxicologic Pathology (Third Edition). , (2013).
- Woud, W. W., et al. An imaging flow cytometry-based methodology for the analysis of single extracellular vesicles in unprocessed human plasma. Commun Biol. 5 (1), 633 (2022).
- Gul, B., Syed, F., Khan, S., Iqbal, A., Ahmad, I. Characterization of extracellular vesicles by flow cytometry: Challenges and promises. Micron. 161, 103341 (2022).
- Black, C. B., Duensing, T. D., Trinkle, L. S., Dunlay, R. T. Cell-based screening using high-throughput flow cytometry. Assay Drug Dev Technol. 9 (1), 13-20 (2011).
- Safford, H. R., Bischel, H. N. Flow cytometry applications in water treatment, distribution, and reuse: A review. Water Res. 151, 110-133 (2019).
- Kobayashi, H., et al. Precise analysis of single small extracellular vesicles using flow cytometry. Sci Rep. 14 (1), 7465 (2024).
- Welsh, J. A., et al. A compendium of single extracellular vesicle flow cytometry. J Extracell Vesicles. 12 (2), e12299 (2023).
- Carnino, J. M., Lee, H. Extracellular vesicles in respiratory disease. Adv Clin Chem. 108, 105-127 (2022).
- Sun, Y., Sethu, P. Low-stress microfluidic density-gradient centrifugation for blood cell sorting. Biomed Microdevices. 20 (3), 77 (2018).
- Strachan, B. C., Xia, H., Voros, E., Gifford, S. C., Shevkoplyas, S. S. Improved expansion of T cells in culture when isolated with an equipment-free, high-throughput, flow-through microfluidic module versus traditional density gradient centrifugation. Cytotherapy. 21 (2), 234-245 (2019).
- Malvezzi, H., Sharma, R., Agarwal, A., Abuzenadah, A. M., Abu-Elmagd, M. Sperm quality after density gradient centrifugation with three commercially available media: a controlled trial. Reprod Biol Endocrinol. 12, 121 (2014).
- Sun, Y., Sethu, P. Microfluidic adaptation of density-gradient centrifugation for isolation of particles and cells. Bioengineering (Basel). 4 (3), 67 (2017).
- Karttunen, J., et al. Precipitation-based extracellular vesicle isolation from rat plasma co-precipitate vesicle-free microRNAs. J Extracell Vesicles. 8 (1), 1555410 (2019).
- De Sousa, K. P., et al. Isolation and characterization of extracellular vesicles and future directions in diagnosis and therapy. Wiley Interdiscip Rev Nanomed Nanobiotechnol. 15 (1), e1835 (2023).
- Sidhom, K., Obi, P. O., Saleem, A. A review of exosomal isolation methods: Is size exclusion chromatography the best option. Int J Mol Sci. 21 (18), 6466 (2020).
- Coughlan, C., et al. Exosome isolation by ultracentrifugation and precipitation and techniques for downstream analyses. Curr Protoc Cell Biol. 88 (1), e110 (2020).
- Konoshenko, M. Y., et al. Isolation of extracellular vesicles from biological fluids via the aggregation-precipitation approach for downstream miRNAs detection. Diagnostics (Basel). 11 (3), 384 (2021).
- Drozdz, A., et al. Low-vacuum filtration as an alternative extracellular vesicle concentration method: A comparison with ultracentrifugation and differential centrifugation. Pharmaceutics. 12 (9), 872 (2020).
- Akbar, A., Malekian, F., Baghban, N., Kodam, S. P., Ullah, M. Methodologies to isolate and purify clinical grade extracellular vesicles for medical applications. Cells. 11 (2), 186 (2022).
- Chen, J., et al. Review on strategies and technologies for exosome isolation and purification. Front Bioeng Biotechnol. 9, 811971 (2021).
- Helling, A., et al. Investigation of microbial cell deformability by filter cake compressibility using ultrafiltration membranes. Colloids Surf B Biointerfaces. 185, 110626 (2020).
- Bagci, C., et al. Overview of extracellular vesicle characterization techniques and introduction to combined reflectance and fluorescence confocal microscopy to distinguish extracellular vesicle subpopulations. Neurophotonics. 9 (2), 021903 (2022).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750 (2018).
- van Maanen, J. C., et al. A combined western and bead-based multiplex platform to characterize extracellular vesicles. Tissue Eng Part C Methods. 29 (11), 493-504 (2023).
- Xu, D., et al. MicroRNAs in extracellular vesicles: Sorting mechanisms, diagnostic value, isolation, and detection technology. Front Bioeng Biotechnol. 10, 948959 (2022).
- Lee, H., He, X., Le, T., Carnino, J. M., Jin, Y. Single-step RT-qPCR for detection of extracellular vesicle microRNAs in vivo: a time- and cost-effective method. Am J Physiol Lung Cell Mol Physiol. 318 (4), L742-L749 (2020).
- Kim, J. A., et al. Small RNA sequencing of circulating small extracellular vesicles microRNAs in patients with amyotrophic lateral sclerosis. Sci Rep. 13 (1), 5528 (2023).
- Fan, Y., et al. Differential proteomics argues against a general role for CD9, CD81 or CD63 in the sorting of proteins into extracellular vesicles. J Extracell Vesicles. 12 (8), e12352 (2023).
- Silva, A. M., et al. Quantification of protein cargo loading into engineered extracellular vesicles at single-vesicle and single-molecule resolution. J Extracell Vesicles. 10 (10), e12130 (2021).
- Salunkhe, S., Basak Dheeraj, M., Chitkara, D., Mittal, A. Surface functionalization of exosomes for target-specific delivery and in vivo imaging, tracking: Strategies and significance. J Control Release. 326, 599-614 (2020).
- Silva, A. K. A., et al. Development of extracellular vesicle-based medicinal products: A position paper of the group "Extracellular Vesicle translatiOn to clinicaL perspectiVEs - EVOLVE France". Adv Drug Deliv Rev. 179, 114001 (2021).
- Gurunathan, S., Kang, M. H., Jeyaraj, M., Qasim, M., Kim, J. H. Review of the isolation, characterization, biological function, and multifarious therapeutic approaches of exosomes. Cells. 8 (4), 307 (2019).
- Zhang, Y., et al. Exosome: A review of its classification, isolation techniques, storage, diagnostic and targeted therapy applications. Int J Nanomedicine. 15, 6917-6934 (2020).
- Colombo, M., Raposo, G., Thery, C. Biogenesis, secretion, and intercellular interactions of exosomes and other extracellular vesicles. Annu Rev Cell Dev Biol. 30, 255-289 (2014).
- Pisitkun, T., Shen, R. F., Knepper, M. A. Identification and proteomic profiling of exosomes in human urine. Proc Natl Acad Sci U S A. 101 (36), 13368-13373 (2004).
- Zaborowski, M. P., Balaj, L., Breakefield, X. O., Lai, C. P. Extracellular vesicles: Composition, biological relevance, and methods of study. Bioscience. 65 (8), 783-797 (2015).
- Kowkabany, G., Bao, Y. Nanoparticle tracking analysis: An effective tool to characterize extracellular vesicles. Molecules. 29 (19), 4672 (2024).
- Li, H. Metastatic characteristics of SY86B human gastric carcinoma in athymic nude mice. Zhonghua Zhong Liu Za Zhi. 10 (6), 421-423 (1988).
- Zhao, Z., Wijerathne, H., Godwin, A. K., Soper, S. A. Isolation and analysis methods of extracellular vesicles (EVs). Extracell Vesicles Circ Nucl Acids. 2, 80-103 (2021).
- Gandham, S., et al. Technologies and standardization in research on extracellular vesicles. Trends Biotechnol. 38 (10), 1066-1098 (2020).
- van der Vlist, E. J., Nolte-'t Hoen, E. N., Stoorvogel, W., Arkesteijn, G. J., Wauben, M. H. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nat Protoc. 7 (7), 1311-1326 (2012).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. J Extracell Vesicles. 4, 25530 (2015).
- Breitwieser, K., et al. Detailed characterization of small extracellular vesicles from different cell types based on tetraspanin composition by ExoView R100 platform. Int J Mol Sci. 23 (15), 8544 (2022).
- An, H. J., Cho, H. K., Song, D. H., Kee, C. Quantitative analysis of exosomes in the aqueous humor of Korean patients with pseudoexfoliation glaucoma. Sci Rep. 12 (1), 12875 (2022).
- Hefley, B. S., McKay, T. B., Hutcheon, A. E. K., Ciolino, J. B., Karamichos, D. Corneal epithelial-stromal constructs to study differences associated with diabetes mellitus. Exp Eye Res. 248, 110100 (2024).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유