Method Article
Biyolojik Sıvılardan Hücre Dışı Veziküllerin Karakterizasyonu
Bu Makalede
Özet
Burada, insan deneklerin gözyaşı ve tükürük gibi biyolojik sıvılarından toplanan hücre dışı veziküller (EV'ler) için karakterizasyon yöntemini gösteriyoruz. Bu yöntemde kullanılan tarayıcı, numunenin 1 μL'sinden EV'lerin fenotipini, boyutunu ve toplam partikül sayısını tespit etme yeteneğine sahiptir.
Özet
Hücre dışı veziküller (EV'ler), hücrelerden üretilen ve biyomolekülleri bir hücreden diğerine taşıyarak hücreler arası iletişime katılan yapılardır. EV'lerin vücutta kısa ve uzak mesafeler kat ettiği ve dokuya özgü olduğu gösterilmiştir. EV'ler sadece dokularda bulunmaz, aynı zamanda gözyaşı, tükürük, beyin omurilik sıvısı, kan vb. gibi hemen hemen tüm vücut sıvılarında da bulunabilirler. EV'ler gözyaşı ve tükürükten non-invaziv olarak toplanabilse de, bir seferde sadece küçük hacimler toplanabilir, bu da proteinleri analiz etmek için yeterli EV'nin elde edilmesinde sorunlara neden olabilir. Bu yazıda tartışılan tarayıcı, bu soruna bir çözüm sunan ve EV'lerin fenotipini, boyutunu ve toplam parçacık sayısını 1 μL kadar küçük bir biyolojik sıvıdan karakterize etmemize ve incelememize olanak tanıyan bir nanopartikül analizörüdür. Bu protokol, hastalardan çıkarılması zor olan küçük hacimli numunelerden EV'lerin bilgisini genişletecektir. Bu, hasta konforunu artırabilir ve potansiyel olarak bir dizi hastalık ve bozukluk için yeni terapötik hedefler belirleyebilir.
Giriş
Hücreler, hücreler arası iletişimde önemli bir rol oynayan hücre dışı veziküllerin (EV'ler) salınması da dahil olmak üzere çeşitli sinyal mekanizmaları aracılığıyla komşu hücrelerle iletişim kurar. EV'ler, DNA, RNA ve proteinler gibi genetik kargoları bir hücreden diğerinegeçirerek hücreler arası iletişime katılır 1,2,3,4,5. Şu anda, üç EV kategorisi vardır: büyüklükleri ile karakterize edilen eksozomlar, mikro-parçacıklar ve apoptotik cisimler. Eksozomlar en küçüğüdür, çapı 30-150 nm 6,7,8'dir ve endozomal membran sisteminden oluşur 9,10,11 (Şekil 1). Mikroveziküller, 100-1000 nm 12,13,14 arasında değiştikleri ve plazma zarından 11,12,13 tomurcuklandıkları için eksozomlardan daha büyüktür (Şekil 1). Apoptotik cisimler EV'lerin en büyüğüdür ve 1000-5000 nm 12,14,15 arasında değişir ve ayrıca plazma zarından 12,16,17 tomurcuklanırlar (Şekil 1). Boyutun yanı sıra, EV'ler yoğunluk, CD63, CD81, CD9 gibi moleküler belirteçler ve ayrıca biyogenez mekanizması18 gibi diğer biyofiziksel özelliklere göre sınıflandırılabilir. EV'ler, bitişik hücreler arasında kısa mesafeler ve vücut boyunca uzun mesafeler kat edebilir 19,20,21,22,23. EV'ler kan 24,25,26, beyin omurilik sıvısı 27,28,29, gözyaşı30,31,32 ve tükürük 33,34,35 gibi biyolojik sıvılarda bulunabilir.
Bugüne kadar, ultrasantrifüjleme, EV'leri 36,37,38 örneklerinden izole etmek için en iyi bilinen yöntemlerden biridir. Bu yöntem, EV'leri hücrelerden ve hücre kalıntılarından izole etmek için hızı artırarak gerçekleştirilebilen çok sayıda santrifüjleme ve ultrasantrifüjleme turu gerektirir. Bu yöntem, daha büyük vezikülleri ortadan kaldırmak için orta hız ve son olarak EV'leri peletlemek için bir ultrasantrifüj adımı olan pelet hücreleri ile düşük hızda başlatılabilir18. Ultrasantrifüjleme, izolasyon için en iyi yöntem olarak kabul edilse de, EV'lerin 39,40,41 morfolojisini değiştirmesi bakımından hala bazı sınırlamalar mevcuttur. EV'leri izole etmek için kullanılan başka bir teknik, çoklu zaman noktası ve uç nokta değerlendirmesinin ve yüksek verimli tek EV analizinin avantajlarını vurgulayan akış sitometrisidir. Bununla birlikte, akış sitometrisinin sınırlamaları, gözeneklerin tıkanmasını ve zayıf sinyalleri içerir ancak bunlarla sınırlı değildir. Kullanılan diğer bir yaklaşım, EV'lerle santrifüjlenecek farklı yoğunluklara sahip malzemeleri kullanan ve EV'lerin ultrasantrifüjlemeye kıyasla daha iyi ayrılmasını sağlayan gradyan santrifüjlemedir. Bu teknik ayırmayı iyileştirse de, emek yoğundur, zaman alıcıdır ve önemli ölçüde numune kaybına yol açabilir. Ek olarak, EV'leri izole etmek için yağış ve filtreleme de kullanılabilir. Bu tekniklerin her ikisi de basit ve hızlıdır, ancak her ikisi de numune kontaminasyonuna yol açabilir. EV'leri izole etmek için çeşitli teknikler olsa da, her tekniğin aşağıda listelenen avantajları ve sınırlamaları vardır (Tablo 1) 42,43,44,45,46,47,48,49,50,51,52,53,54, 55, 56,57,58,59,60,61,62,63,64,65,66,67.
EV'ler izole edildikten sonra, EV'leri karakterize etmek için moleküler analizler yapılabilir. Western blotları, yüzey ve kargo protein ekspresyonu 68,69,70'i aramak için yaygın bir testtir ve polimeraz zincir reaksiyonu (PCR), EV'ler71,72,73 için miRNA ekspresyonu 74,75,76 için kullanılır. Bu tahliller kurulmuştur ve ilgi çekici sonuçlar üretebilir. Bu yöntemlerin bir sınırlaması, başlangıçta küçük bir hacme veya EV konsantrasyonuna sahip numuneler için bir sorun olan77 okumasını elde etmek için EV'lerden büyük miktarda protein veya RNA'ya ihtiyaç duymalarıdır.
Bu yazıda tartışılan nanopartikül analizörü, kullanıcının Tablo 1 ve Tablo 2'de belirtilen birçok sınırlamanın üstesinden gelmesine olanak tanır: 78,79,80,81,82,83,84,85,86,87,88,89,90 . Bu yöntem, EV'lerin azalan veriminin üstesinden gelmeye yardımcı olacak izolasyon tekniklerinin kullanılmasını gerektirmez. Bu yöntem aynı zamanda kullanıcının yüzey ve kargo proteinlerini, toplam EV sayısını ve EV boyutunu 1 μL kadar küçük bir numune hacminden analiz etmesine olanak tanır. Bu, Şekil 2'de gösterildiği gibi bir çözeltideki EV'leri tanımlamak için tetraspanin antikorları, CD63, CD81 ve CD9 içeren bir antikor mikrodizisi kullanan şirket tarafından sağlanan tetraspanin çipleri kullanılarak yapılır. Floresan antikorlar, EV'lerin varlığını doğrular ve kirletici parçacıkların sonuçları çarpıtmasını önler.
Bu tekniğin genel amacı, EV'leri analiz etmek için daha az zaman alan bir yöntem sağlamanın yanı sıra EV'leri küçük bir numune hacminden analiz etmektir. Bu nanopartikül analizörünü kullanmak, kullanıcıların gözyaşı ve tükürük gibi biyolojik sıvılar için ideal olan 1 μL kadar küçük bir numuneden boyutu, toplam partikül sayısını ve yüzey proteinlerini analiz etmesine olanak tanır.
Protokol
Açıklanan tüm çalışmalar Helsinki Bildirgesi'ne bağlı kalmıştır. Çalışmaya dahil edilmeden önce her denekten yazılı onam alınmıştır. Federal ve kurumsal yönergelere göre Aarhus Üniversite Hastanesi (1-10-72-77-14) ve Dean McGee Enstitüsü'nden (1576837-2) Kurumsal İnceleme Kurulu (IRB) onayı alındı. İşlemden önce, tüm gözyaşı ve tükürük örnekleri kimliksizleştirildi. Tüm çalışmalar Kuzey Teksas Bölgesel Kurumsal İnceleme Kurulu (#2020-030) tarafından gözden geçirildi ve onaylandı. Aşağıdaki protokol tüm yönergelere uygundur ve yukarıda belirtildiği gibi onaylanmıştır.
1. Gün 1: Numune hazırlama ve inkübasyon
- Biyolojik sıvı örnek(ler)ini, yani bir denek(ler)den gözyaşı ve tükürük toplayın veya örnek(ler)i dondurucudan çözün.
- Gözyaşı: Bir cam kılcal tüp kullanarak lateral menisküsten pasif olarak gözyaşı toplayın.
- Cam kılcal boruyu korneaya dokunmamaya dikkat ederek alt göz kapağına yerleştirin.
- 10 dakika içinde 10 μL gözyaşı hacmi toplayın.
- Gözyaşlarını hemen kullanın veya -20 °C'de saklayın.
- Tükürük: 1,5 mL'lik bir mikrosantrifüj tüpünde pasif salyadan tükürük toplayın
- Bağışçının ağzında tükürük biriktirmesine izin verin.
- Bağışçıdan bir huni ile tüpe salya akıtmasını isteyin.
- Tükürüğü hemen kullanın veya -20 °C'de saklayın.
- Gözyaşı: Bir cam kılcal tüp kullanarak lateral menisküsten pasif olarak gözyaşı toplayın.
- Tetraspanin cips(ler)ini, oda sıcaklığına ulaşmalarını sağlamak için numune eklemeden önce 15 dakika talaş yıkama plakasına yerleştirin. Talaşları kuyuya yerleştirirken, numaraların alt tarafta yukarı baktığından ve talaş(lar)ın kuyu(lar)ın yan duvarlarına temas etmediğinden emin olun.
- Çipleri takip etmek için kimlik numarasını ve çip numarasını dizüstü bilgisayara kaydedin.
- Kit ile birlikte gelen evrensel seri veri yolunu (USB) takın ve ilgili kit numarasıyla birlikte klasörü ayıklayın ve seçtiğiniz belirlenmiş bir alana kaydedin.
- 1.5 mL'lik bir tüpe 99 μL Çözelti B'yi pipetleyin.
- Bir önceki adımda hazırlanan 1,5 mL'lik tüpe 1 μL biyolojik sıvı ekleyin.
- Tüpleri aşağı doğru döndürün.
- Adım 1.6'dan 70 μL çözelti alın ve tetraspanin çipine pipetleyin.
- Numunenin buharlaşmasını önlemek için filmi talaş yıkama plakasına yerleştirin.
- Titreşimsiz bir tezgahta oda sıcaklığında (RT) 16 saat inkübe edin.
2. Gün 2: Talaş yıkama
- Talaş yıkama makinesini açın.
- Adım 1.9'daki filmi plakadan çıkarın.
- Plakayı talaş yıkayıcıya yerleştirin, yerine oturduğundan emin olun ve kapağı kapatın.
NOT: Yerine doğru şekilde kilitlenirse plaka kolayca hareket etmeyecektir. - Talaş yıkayıcıda CW-TETRA v0 seçeneğine basın.
- Başlangıç satırını ve yıkanacak satır sayısını seçin.
- Talaş yıkama makinesinde Continue (Devam ) düğmesine basın.
- Çip(ler) yıkanırken, antikorları içeren blokaj solüsyonunu bir tüpte aşağıdaki gibi hazırlayın:
- Çip başına 300 μL Engelleme çözeltisi hazırlayın.
- Çip başına 0.6 μL veya her bir antikor ekleyin (bloke edici çözeltiye eklemeden önce girdap ve geri döndürme antikorları)
- Bloke edici çözelti + antikor kokteylini girdaplayın.
NOT: Antikor stabilitesini sağlamak için bloke edici solüsyon + antikor kokteylini ışıktan koruyun. Bu, kullanım gününde yapılmalıdır.
- Talaş yıkayıcı cıvıldadığında, talaş(lar)ın üzerine 250 μL bloke edici solüsyon + antikor kokteyli ekleyin.
- Kapağı kapatın ve Devam'a basın.
NOT: Talaş yıkayıcı şimdi birkaç ek yıkama ve durulama turu ile birlikte 1 saatlik bir inkübasyon yapacaktır. - Talaş yıkayıcı cıvıl cıvıl öttüş yaptığında, talaş yıkama plakasını çıkarın ve talaşları rampadan yukarı doğru hareket ettirin.
- Plakayı, yerine kilitlendiğinden emin olarak talaş yıkayıcıya geri yerleştirin. Kapağı kapatın ve Devam'a basın.
- Talaş yıkayıcı programı tamamladığında, plakayı çıkarın ve talaşları bir kağıt havlunun üzerine yerleştirin. Çip(ler)i ışıktan kapatın.
3. Gün 2: Çip(ler)in taranması
- Tarayıcıyı açın ve tarama yazılımını açın.
NOT: Tarayıcı, herhangi bir hata olup olmadığını veya taramaya devam edilip edilemeyeceğini belirtmek için kendi kendini kontrol edecektir. - Klasörü Kaydet'i seçin ve dosyaların kaydedileceği konumu seçin.
- ChipFile Klasörü'nü seçin ve adım 1.4'te kaydedilen klasörü seçin.
- Her bir çipi seçmek için açılır seçeneği seçin ve ayna üzerindeki Çip Konumuna yerleştirin. Taranması gereken tüm çipler için bu adımı tekrarlayın.
- Çip Konumu'nun altındaki üç kareye tıklayarak floresansı açın. Kareler sırasıyla sarı, mavi ve kırmızıya dönecektir.
- Kapağı aşağı bastırarak mandren kapağını çıkarın ve çekin.
- Talaş(lar)ı sıkıca sabitlendiklerinden emin olarak aynanın üzerine yerleştirin.
- Mandalları deliklere yerleştirerek ayna kapağını tekrar takın, aşağı bastırın ve yerine kilitlemek için kapağı kaydırın.
- Mandreni sahneye yerleştirin ve yerine kilitlendiğinden emin olun.
- Scan Chips (Çipleri Tara) öğesini seçin.
- Tamam'ı seçin.
- Sahne tamamen makineye taşındıktan sonra kapıyı kapatın.
NOT: Tek bir çipi taramak yaklaşık 12 dakika sürer. - Program çipleri taramayı bitirdikten sonra, taramaların başarılı olup olmadığını görmek için aşama kontrolünü çıkarmak için kapıyı açın.
- Taramalar başarılı olduysa, çipleri atın ve bir sonraki fiş setini aynaya yerleştirin veya boş aynayı sahneye geri koyun. Ardından, yazılımdan çıkın ve işiniz bittiğinde makineyi kapatın.
- Taramalar başarılı olmadıysa, hata koduna bakın ve hatayı düzeltin.
4. Veri işleme
- Analiz yazılımını açın.
- Verileri Tara'yı seçin ve 2. gün adım 3.2'den itibaren taramanın bulunduğu klasörü seçin.
- İleri'yi seçin.
- Örnek Adı sütunundaki çipleri, hücreye tıklayıp örnek kimliğini yazarak örneğin adıyla etiketleyin.
- Ardından, Kanal Adı sütununda tetraspaninleri etiketleyin. Kırmızı = CD63, Yeşil = CD81 ve Mavi = CD9.
- İleri'yi seçin.
- Açılır kutuyu incelemek için Select Spot (Spot Seç) grubundan High CV'yi (Yüksek CV) seçin ve bir veya iki noktayı devre dışı bırakarak her çip için yüksek CV'leri kapatın.
- Ardından, Yüksek Sayım'ı seçin ve yüksek sayım uyarılarını kapatmayı deneyin.
NOT: Bazı numunelerin sayısı çok yüksek olabilir, ancak yine de analiz edilebilirler. - Açılır menüden Tüm Noktalar'ı seçin, Nokta Montajı'nı seçin ve her bir çipte mevcut olabilecek herhangi bir deformasyon veya çizik olup olmadığını görsel olarak kontrol edin.
- İleri'yi seçin.
- CD63 kanalında, Ort % Dahil satırındaki kırmızı hücrelerden kurtulmak için minimum değeri 300 olarak ayarlayın; sarı hücreler iyidir ve CD81 kanalına ilerleyin. Minimum değerin 300 olarak ayarlanması kırmızı hücreyi sarı veya beyaz olarak değiştirmezse, sorun giderme için adım 4.14'e geçin.
- CD81 kanalında, Ort % Dahil satırındaki kırmızı hücrelerden kurtulmak için minimum değeri 300 olarak ayarlayın; sarı hücreler iyidir ve CD9 kanalına ilerleyin. Minimum değerin 300 olarak ayarlanması kırmızı hücreyi sarı veya beyaz olarak değiştirmezse, sorun giderme için adım 4.14'e geçin.
- CD9 kanalında, Ort % Dahil satırındaki kırmızı hücrelerden kurtulmak için minimum değeri 400 olarak ayarlayın; Sarı hücreler iyidir ve Adım 4.15'e geçin. Minimum değerin 400 olarak ayarlanması kırmızı hücreyi sarı veya beyaz olarak değiştirmezse, sorun giderme için adım 4.14'e geçin.
- Sorun giderme
- Minimum değeri 100 birim artırın.
- Adım 4.14.1 işe yaramadıysa, Üzerinde çalışılacak çipleri seç bölümünde çipi seçin ve çipi seçin. Parçacık Sayımı'nı seçin ve yüksek parçacık sayımına sahip noktaları devre dışı bırakın. Yoğunluk/Boyut'u seçin, Tüm Çipler'i seçin ve bunun sorunu çözüp çözmediğine bakın.
- Adım 4.14.2 işe yaramadıysa, Üzerinde çalışılacak çipleri seç bölümünde çipi seçin ve çipi seçin. Tek Çipli Kesintiler'i seçin ve minimumu 100 birim artırın. Ardından, kırmızı hücrenin şimdi sarı olup olmadığını görmek için Tüm Çipler'i seçin. Bu işe yaramazsa, Ort % Dahil Edilen hücre artık sarı veya beyaz olana kadar bu adımı yineleyin.
- İleri'yi seçin.
- Anlık ileti kanalının seçimini kaldırın.
- Sağ alt köşedeki ısı haritası değerini tüm parçacıkları içerecek şekilde ayarlayın ve Rapora Çizim Ekle'yi seçin.
- Açılır kutuda maksimum sayıyı ayarlayın ve manuel olarak bir değer girin.
- Ardından, Yakalama Probları altındaki üst ortadaki açılır menüyü seçin ve CD63'ü seçin.
- Rapora toplam parçacık sayısını eklemek için Rapora Çizim Ekle'yi seçin.
- Analiz Modu'nun altındaki açılır menüyü seçin, Yerelleştirme'yi seçin ve Rapora Çizim Ekle'yi seçin.
- Ardından Boyut'u seçin, Görünümü Çizim olarak değiştir'i seçin ve Rapora Çizim Ekle'yi seçin.
- Son olarak, Görüntüler'i seçin ve Rapora Çizim Ekle'yi seçin.
- CD81 ve CD9 için 4.18–4.22 arasındaki adımları tekrarlayın ve her seferinde IM kanalının seçimini kaldırdığınızdan emin olun.
- Tüm öğeler rapora eklendikten sonra Raporu Dışa Aktar'ı seçin, doğru dosyaya kaydedildiğinden emin olun ve Klasör'ü seçin.
NOT: Excel dosyaları ve görüntüleri oluşturulmuştur ve artık daha fazla analiz için kullanılabilir.
Sonuçlar
Çipler üzerindeki noktaları analiz ederken, CD63, CD81 ve CD9 kanallarında floresan etiketli EV'leri gözlemleyerek optimum bir EV konsantrasyonu hedefleyin ve MIgG kanalı 30,91 siyah kalmalıdır, çünkü bu, Şekil 3A'da görüldüğü gibi kontrol kanalıdır. Biyolojik sıvılar için CD81 azaltılacaktır. CD63, CD81 ve CD9 kanallarında floresan yoksa, EV'lerde yoktur (Şekil 3B). Çipi aşırı doyurmamaya dikkat edin (Şekil 3C). Bu, analiz yazılımının EV'lerin boyutunun, toplam parçacık sayısının ve fenotiplerinin doğruluğunu hesaplamasını zorlaştıracaktır. Optimal konsantrasyon noktalarından, analiz yazılımı EV'lerin boyutunu (50-200 nm), toplam partikül sayısını ve tetraspanin ekspresyonunu ölçebilecek (Şekil 4) ve daha fazla analiz için ayrı elektronik tablolara aktarılacaktır. Tetraspanin analizi, EV'ler üzerindeki yüzey proteinlerinin kolokalizasyonunu içerecektir. Yerelleştirme "/" ile temsil edilir, örneğin, "CD63/CD81". Bu, CD63 ve CD81'in birleştirildiği anlamına gelmez; bu, hem CD63 hem de CD81'in EV'nin yüzeyinde bulunduğu anlamına gelir.
Bu sonuçlar, sağlıklı ve hastalıklı örnekler arasında değerli bilgiler sağlayacaktır. Sağlıklı örneklerin veya hastalıklı örneklerin daha fazla EV, daha büyük veya daha küçük EV'ler üretip üretmediğini ve EV'lerin fenotipini belirleyebileceğiz. Bu özelliklerin herhangi biri veya tümü hastalığın biyogenezinde ve/veya ilerlemesinde rol oynayabilir. Bu sonuçlarla, hücre süreçleri ve EV'lerin oluşumu hakkında fikir verebilecek tetraspanin seviyelerinde bir eksiklik veya artış olup olmadığını görebileceğiz.
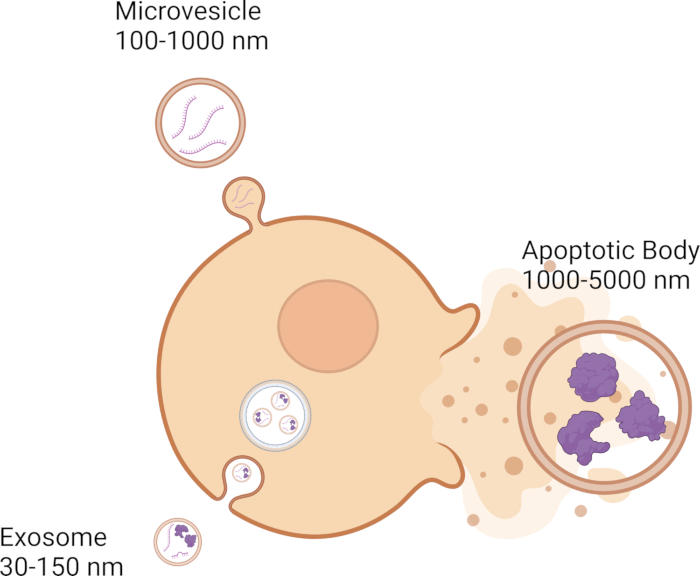
Şekil 1: Farklı EV'lerin türleri ve boyutları. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
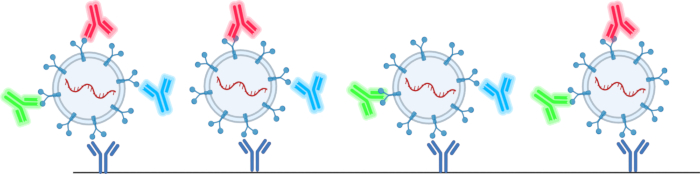
Şekil 2: Tetraspanin çiplerinin EV'leri nasıl yakaladığını ve tespit ettiğini gösteren şema. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
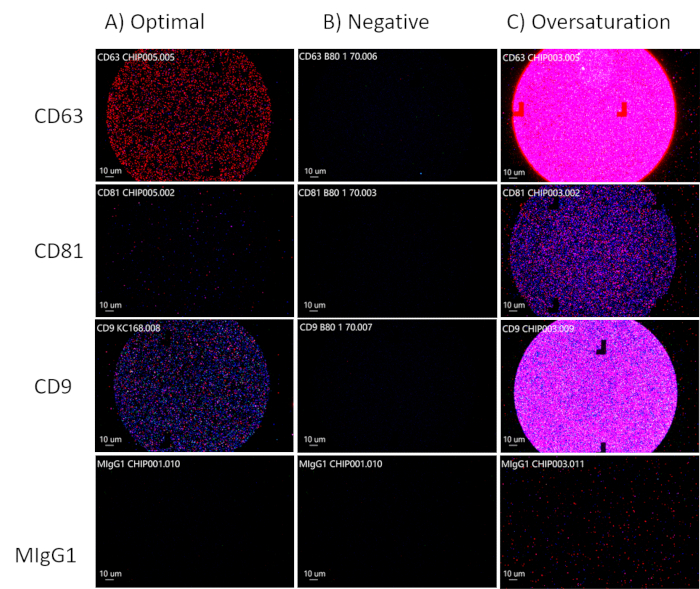
Şekil 3: EV konsantrasyonlarının optimize edilmesi. (A) EV'lerin optimal konsantrasyonu. (B) Olumsuz sonuç: EV yok. (C) EV'lerin aşırı doygunluğu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
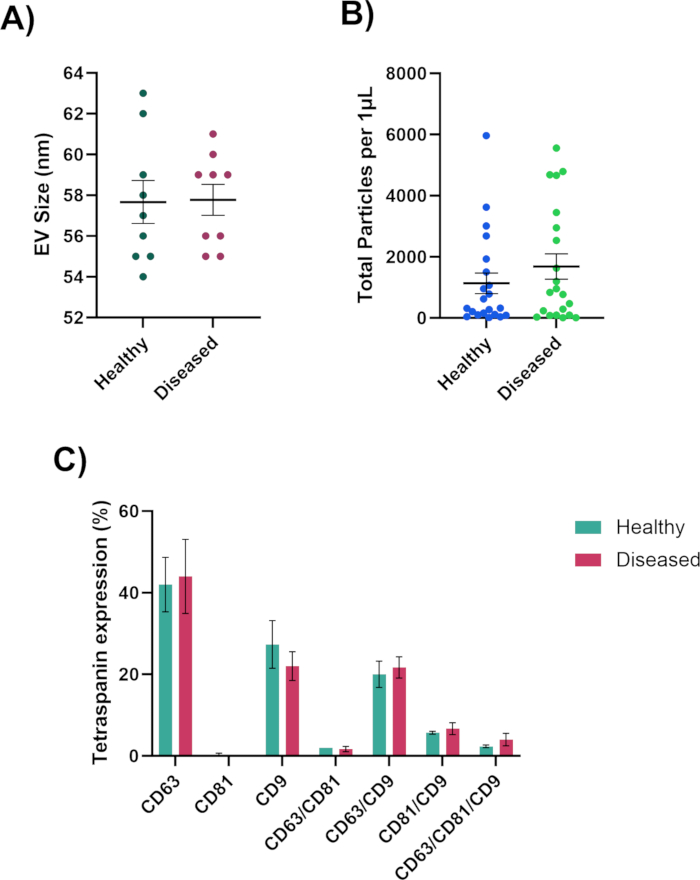
Şekil 4: Nanopartikül analizörü tarafından üretilen veriler. (A) Nanometre cinsinden ölçülen EV çap boyutu. (B) EV'lerin toplam parçacık sayısı. (C) EV kolokalize fenotipi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Teknik | Avantaj -ları | Sınırlama |
| Ultrasantrifüj | Yüksek Saflık42,43 | EV morfolojisini değiştirir42,44 |
| Homojenlik42,45 | Büyük hacim gerektirir42,44,46 | |
| İşlevsellik42,43 | Pahalı42,43 | |
| Akış Sitometrisi | Numuneleri birden fazla zaman noktasında değerlendirin47 | Zayıf sinyal48,49 |
| Çoklu uç noktalar47,50 | Tıkalı gözenekler51 | |
| Yüksek verimli tek EV analizi52,53 | ||
| Yoğunluk Gradyan Santrifüjleme | Son derece saf numuneler üretin54 | Emek yoğun55,56 |
| Numune çeşitliliği55,57 | Zaman alıcı55,58 | |
| Önemli verim kaybı54 | ||
| Yağış | Basit59,60 | Kirlenme36,44,61 |
| Hızlı61,62 | Spesifik Olmayan63 | |
| Yüksek Verim44,59,62 | ||
| Nötr pH36 | ||
| Filtrasyon | Basit64 | Tuzak eksozomları65 |
| Hızlı64 | Büyük veziküllere zarar verir36 | |
| Ucuz64 | Filtre kek66,67 | |
| Eksozom tıkanması65 |
Tablo 1: EV izolasyon tekniklerinin Avantajları ve Sınırlamaları. Avantajları ve sınırlamaları da dahil olmak üzere EV'leri izole etmenin çeşitli yollarının bir listesi.
| Teknik | Avantaj -ları | Sınırlama |
| Dinamik ışık saçılımı | Boyutları 1 nm ila 6 μM arasında değişen partikülleri ölçün78 | Büyük boyut aralığına sahip karmaşık eksozom numunelerinin ölçümü için uygun değildir79 |
| Alt sınır 10 nm'dir, monodispers sistemler için uygundur79 | Kontamine proteinleri eksozomlardan ayırt edememe79 | |
| Transmisyon elektron mikroskobu (TEM) | Eksozomların morfolojisini gözlemleyin79,80 | Karmaşık numune hazırlamaişlemleri 79 |
| İç yapıyı gözlemleyin81 | Abartılı floresan sinyalleri nedeniyle eksozomları boyut ve şekle göre ayırt edememe82 | |
| Nanopartikül İzleme analizi | 10 nm ila 2 μM aralığındaki eksozomların konsantrasyonunu, boyutunu ve boyut dağılımını ölçün78 | EV'leri protein agregatlarından ve diğer kirleticilerden ayırt edemez83 |
| Hızlı numune hazırlama ve ölçme78,84 | Pahalı NTA cihazı85 | |
| Örnekler yerel formda geri kazanılabilir84 | Titreşimlere karşı hassas85 | |
| Batı Lekesi (WB'ler) | Marker proteinleri kalitatif ve kantitatif olarak analiz edebilir79,86 | Karmaşık ve zaman alıcı79 |
| Hücre kültürü ortamından eksozomların analizi79 | EV izolatları lipoproteinler ve diğer kirleticiler içerebilir86 | |
| Akış sitometrisi | Daha yüksek hassasiyet ve yüksek çözünürlüklü görüntüleme | 400 nm79,88 algılama limiti ile zaman alıcı ve zahmetli |
| Lekeli eksozomları muhafazalardan ayırt edin87 | ||
| Gerekli düşük numune konsantrasyonu79 | Optik sinyaller doğruluğu ve çözünürlüğü engelliyor88 | |
| Dış Görünüm | Eksozomlar89 üzerindeki Tetraspaninleri (CD9, CD63 ve CD81) ölçün | Daha büyük EV boyutlarını ölçmez75 |
| EV kargo proteinleriniölçer 90 |
Tablo 2: EV karakterizasyon tekniklerinin avantajları ve sınırlamaları.
Tartışmalar
Bu protokoldeki en kritik adım, optimal bir EV konsantrasyonunun elde edildiğinden emin olmaktır. Bir okuma elde etmek için yeterli EV'nin mevcut olması gerekir, ancak çipi aşırı doyuracak çok fazla EV olmamalıdır. Optimum EV konsantrasyonunu belirlemenin en iyi yolu, 1 μL numune ile bir optimizasyon çalışması yapmak ve konsantrasyonun ayarlanması gerekip gerekmediğini görmektir. Bir diğer kritik adım, numunede bol miktarda hücre kalıntısı olup olmadığını görmektir ve bu, analiz yazılımındaki çipler üzerindeki büyük parçalar görüntülenerek belirlenebilir. Numunede hücre kalıntısı varsa, numunenin basit bir santrifüjlenmesi veya filtrelenmesi bu sorunu çözmelidir.
Ek ve çok önemli bir adım, talaş(lar)ın kuyu duvarlarına temas etmemesini sağlamak ve talaş(lar)ı rampaya yerleştirirken talaş(lar)ın merkez alanıyla temastan kaçınmaktır. Tarayıcı, çipin ortasındaki kareden okuyacaktır, bu nedenle EV'leri bozmamak için o alana forseps ile dokunmaktan kaçınmak önemlidir.
Bu yöntemin, cihaz tarafından tespit için yetersiz EV konsantrasyonuna sahip olması gibi bazı sınırlamaları vardır. Konsantrasyon, numunenin kurutulması veya bir yoğunlaştırıcı tüp kullanılmasıyla iyileştirilebilir. Diğer bir sınırlama, analiz yazılımının, bazı mikro-parçacıkları ve tüm apoptotik cisimleri boyut ölçümlerinden 50 hariç tutarak, yalnızca200-75 nm aralığındaki EV'leri ölçmesidir.
İzole etmenin birçok yolu vardır (Tablo 1 42,43,44,45,46,47,48,49,50,51,52, 53,54,55,56,57,58,59,60, 61,62,63,64,65,66,67) ve EV'leri analiz etmek (Tablo 2 78,79,80,81,82,83,84,85, 86,87,88,89,90) ve EV izolasyonu için mevcut altın standart, Western Blots 36,37,38 ve PCR68,69,70 gibi tahliller için hücreleri peletlemek için 71,72,73,74,75,76. Bu protokol büyük numuneler42,44,46 için iyi çalışsa da, biyolojik sıvıların elde edilmesi zor olabilir ve çoğu zaman bir seferde yalnızca küçük miktarda numune toplanabilir, bu da ultrasantrifüjleme için ideal değildir. Buna karşılık, bu nanopartikül analizörünün kullanılması, kullanıcının EV'lerin boyutu, toplam partikül sayısı ve fenotipi gibi değerli verileri 1 μL kadar küçük bir numunede üretmesine olanak tanır ve bu da onu biyolojik sıvılar için ideal hale getirir. EV'lerin bilgisini, çıkarılması zor olan küçük hacimli örneklerden genişletebileceğiz, bu da hasta konforunu artırabilir ve muhtemelen çeşitli hastalıklar ve bozukluklar için potansiyel terapötik hedefler bulabilir.
Açıklamalar
Yazarların ifşa etmesi gereken rekabet eden finansal çıkarları veya diğer çıkar çatışmaları yoktur.
Teşekkürler
Finansman için NIH'ye teşekkür ederiz (EY031316 ve EY034714). Laboratuvar alanı için UNTHSC ve NTERI'ye de teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| ChipWasher 100 | NanoView | EV-CW100 | Incubates, washes, rinses and dries the tetraspanin chips. This current model is no longer available. Price at time of purchase: $9,995.00 |
| ExoView Analyzer software | NanoView | N/A | Analyzes the chip informations and produces excel files for further analysis. No longer available. |
| ExoView R100 | NanoView | EV-R100 | Used to scan the tetraspanin chips at 3 wavelengths. This current model is no longer available. Price at time of purchase: $110,000.00 |
| ExoView Scanner software | NanoView | N/A | Scans the chips at 3 different wavelengths. No longer available. |
| Human Tetraspanin Kits | Unchained Labs | EV-TETRA-C | Includes 8 tetraspanin chips, Incubation Solution, Blocking Solution, CD63 antibody, CD81 antibody, CD9 antibody, Solution A, Solution B, USB, and plate cover. |
Referanslar
- Abels, E. R., Breakefield, X. O. Introduction to extracellular vesicles: Biogenesis, RNA cargo selection, content, release, and uptake. Cell Mol Neurobiol. 36 (3), 301-312 (2016).
- Radler, J., Gupta, D., Zickler, A., Andaloussi, S. E. Exploiting the biogenesis of extracellular vesicles for bioengineering and therapeutic cargo loading. Mol Ther. 31 (5), 1231-1250 (2023).
- Gupta, D., Zickler, A. M., El Andaloussi, S. Dosing extracellular vesicles. Adv Drug Deliv Rev. 178, 113961 (2021).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63 (2018).
- Zhang, H., et al. Exosome-induced regulation in inflammatory bowel disease. Front Immunol. 10, 1464 (2019).
- Barzin, M., et al. Application of plant-derived exosome-like nanoparticles in drug delivery. Pharm Dev Technol. 28 (5), 383-402 (2023).
- Zhou, C., et al. Stem cell-derived exosomes: emerging therapeutic opportunities for wound healing. Stem Cell Res Ther. 14 (1), 107 (2023).
- Kalluri, R., LeBleu, V. S. The biology, function, and biomedical applications of exosomes. Science. 367 (6478), aau6977 (2020).
- Mondal, J., et al. Hybrid exosomes, exosome-like nanovesicles and engineered exosomes for therapeutic applications. J Control Release. 353, 1127-1149 (2023).
- van Niel, G., D'Angelo, G., Raposo, G. Shedding light on the cell biology of extracellular vesicles. Nat Rev Mol Cell Biol. 19 (4), 213-228 (2018).
- Paul, N., Sultana, Z., Fisher, J. J., Maiti, K., Smith, R. Extracellular vesicles- crucial players in human pregnancy. Placenta. 140, 30-38 (2023).
- Zaldivia, M. T. K., McFadyen, J. D., Lim, B., Wang, X., Peter, K. Platelet-derived microvesicles in cardiovascular diseases. Front Cardiovasc Med. 4, 74 (2017).
- Tamasi, V., Nemeth, K., Csala, M. Role of extracellular vesicles in liver diseases. Life (Basel). 13 (5), 1117 (2023).
- Fu, Y., et al. Emerging understanding of apoptosis in mediating mesenchymal stem cell therapy. Cell Death Dis. 12 (6), 596 (2021).
- Santavanond, J. P., Rutter, S. F., Atkin-Smith, G. K., Poon, I. K. H. Apoptotic bodies: Mechanism of formation, isolation and functional relevance. Subcell Biochem. 97, 61-88 (2021).
- Yu, L., et al. Apoptotic bodies: bioactive treasure left behind by the dying cells with robust diagnostic and therapeutic application potentials. J Nanobiotechnology. 21 (1), 218 (2023).
- Momen-Heravi, F., Getting, S. J., Moschos, S. A. Extracellular vesicles and their nucleic acids for biomarker discovery. Pharmacol Ther. 192, 170-187 (2018).
- Liu, Y. J., Wang, C. A review of the regulatory mechanisms of extracellular vesicles-mediated intercellular communication. Cell Commun Signal. 21 (1), 77 (2023).
- Grange, C., Bussolati, B. Extracellular vesicles in kidney disease. Nat Rev Nephrol. 18 (8), 499-513 (2022).
- Cocucci, E., Meldolesi, J. Ectosomes and exosomes: shedding the confusion between extracellular vesicles. Trends Cell Biol. 25 (6), 364-372 (2015).
- Iannotta, D. A. A., Kijas, A. W., Rowan, A. E., Wolfram, J. Entry and exit of extracellular vesicles to and from the blood circulation. Nat Nanotechnol. 19 (1), 13-20 (2024).
- Saint-Pol, J., Gosselet, F., Duban-Deweer, S., Pottiez, G., Karamanos, Y. Targeting and crossing the blood-brain barrier with extracellular vesicles. Cells. 9 (4), 851 (2020).
- Alberro, A., Iparraguirre, L., Fernandes, A., Otaegui, D. Extracellular vesicles in blood: Sources, effects, and applications. Int J Mol Sci. 22 (15), 8163 (2021).
- Nieuwland, R., Siljander, P. R. A beginner's guide to study extracellular vesicles in human blood plasma and serum. J Extracell Vesicles. 13 (1), e12400 (2024).
- Thangaraju, K., Neerukonda, S. N., Katneni, U., Buehler, P. W. Extracellular vesicles from red blood cells and their evolving roles in health, coagulopathy and therapy. Int J Mol Sci. 22 (1), 153 (2020).
- Li, C., et al. Cerebrospinal fluid-derived extracellular vesicles after spinal cord injury promote vascular regeneration via PI3K/AKT signaling pathway. J Orthop Translat. 39, 124-134 (2023).
- Deng, Y., et al. Phosphoproteome analysis of cerebrospinal fluid extracellular vesicles in primary central nervous system lymphoma. Analyst. 148 (15), 3594-3602 (2023).
- Hirschberg, Y., et al. Proteomic comparison between non-purified cerebrospinal fluid and cerebrospinal fluid-derived extracellular vesicles from patients with Alzheimer's, Parkinson's and Lewy body dementia. J Extracell Vesicles. 12 (12), e12383 (2023).
- Hefley, B. S., et al. Revealing the presence of tear extracellular vesicles in Keratoconus. Exp Eye Res. 224, 109242 (2022).
- Cross, T., et al. RNA profiles of tear fluid extracellular vesicles in patients with dry eye-related symptoms. Int J Mol Sci. 24 (20), 15390 (2023).
- Ma, H., et al. Metabolic signatures of tear extracellular vesicles caused by herpes simplex keratitis. Ocul Surf. 31, 21-30 (2024).
- Han, P., Bartold, P. M., Ivanovski, S. The emerging role of small extracellular vesicles in saliva and gingival crevicular fluid as diagnostics for periodontitis. J Periodontal Res. 57 (1), 219-231 (2022).
- Han, P., Li, X., Wei, W., Ivanovski, S. Saliva diagnosis using small extracellular vesicles and salivaomics. Methods Mol Biol. 2588, 25-39 (2023).
- Reseco, L., et al. Characterization of extracellular vesicles from human saliva: Effects of age and isolation techniques. Cells. 13 (1), 95 (2024).
- Tiwari, S., Kumar, V., Randhawa, S., Verma, S. K. Preparation and characterization of extracellular vesicles. Am J Reprod Immunol. 85 (2), e13367 (2021).
- Tomiyama, E., Fujita, K., Nonomura, N. Urinary extracellular vesicles: Ultracentrifugation method. Methods Mol Biol. 2292, 173-181 (2021).
- Xu, K., Jin, Y., Li, Y., Huang, Y., Zhao, R. Recent progress of exosome isolation and peptide recognition-guided strategies for exosome research. Front Chem. 10, 844124 (2022).
- Brennan, K., et al. A comparison of methods for the isolation and separation of extracellular vesicles from protein and lipid particles in human serum. Sci Rep. 10 (1), 1039 (2020).
- Gao, J., et al. Recent developments in isolating methods for exosomes. Front Bioeng Biotechnol. 10, 1100892 (2022).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Ansari, F. J., et al. Comparison of the efficiency of ultrafiltration, precipitation, and ultracentrifugation methods for exosome isolation. Biochem Biophys Rep. 38, 101668 (2024).
- Patel, G. K., et al. Comparative analysis of exosome isolation methods using culture supernatant for optimum yield, purity and downstream applications. Sci Rep. 9 (1), 5335 (2019).
- Stam, J., Bartel, S., Bischoff, R., Wolters, J. C. Isolation of extracellular vesicles with combined enrichment methods. J Chromatogr B Analyt Technol Biomed Life Sci. 1169, 122604 (2021).
- Le Roy, A., et al. AUC and small-angle scattering for membrane proteins. Methods Enzymol. 562, 257-286 (2015).
- Chhoy, P., Brown, C. W., Amante, J. J., Mercurio, A. M. Protocol for the separation of extracellular vesicles by ultracentrifugation from in vitro cell culture models. STAR Protoc. 2 (1), 100303 (2021).
- McDorman, K. S., Chan, C., Rojko, J., Satterwhite, C. M., Morrison, J. P. . Chapter 7 - Special Techniques in Toxicologic Pathology. Haschek and Rousseaux's Handbook of Toxicologic Pathology (Third Edition). , (2013).
- Woud, W. W., et al. An imaging flow cytometry-based methodology for the analysis of single extracellular vesicles in unprocessed human plasma. Commun Biol. 5 (1), 633 (2022).
- Gul, B., Syed, F., Khan, S., Iqbal, A., Ahmad, I. Characterization of extracellular vesicles by flow cytometry: Challenges and promises. Micron. 161, 103341 (2022).
- Black, C. B., Duensing, T. D., Trinkle, L. S., Dunlay, R. T. Cell-based screening using high-throughput flow cytometry. Assay Drug Dev Technol. 9 (1), 13-20 (2011).
- Safford, H. R., Bischel, H. N. Flow cytometry applications in water treatment, distribution, and reuse: A review. Water Res. 151, 110-133 (2019).
- Kobayashi, H., et al. Precise analysis of single small extracellular vesicles using flow cytometry. Sci Rep. 14 (1), 7465 (2024).
- Welsh, J. A., et al. A compendium of single extracellular vesicle flow cytometry. J Extracell Vesicles. 12 (2), e12299 (2023).
- Carnino, J. M., Lee, H. Extracellular vesicles in respiratory disease. Adv Clin Chem. 108, 105-127 (2022).
- Sun, Y., Sethu, P. Low-stress microfluidic density-gradient centrifugation for blood cell sorting. Biomed Microdevices. 20 (3), 77 (2018).
- Strachan, B. C., Xia, H., Voros, E., Gifford, S. C., Shevkoplyas, S. S. Improved expansion of T cells in culture when isolated with an equipment-free, high-throughput, flow-through microfluidic module versus traditional density gradient centrifugation. Cytotherapy. 21 (2), 234-245 (2019).
- Malvezzi, H., Sharma, R., Agarwal, A., Abuzenadah, A. M., Abu-Elmagd, M. Sperm quality after density gradient centrifugation with three commercially available media: a controlled trial. Reprod Biol Endocrinol. 12, 121 (2014).
- Sun, Y., Sethu, P. Microfluidic adaptation of density-gradient centrifugation for isolation of particles and cells. Bioengineering (Basel). 4 (3), 67 (2017).
- Karttunen, J., et al. Precipitation-based extracellular vesicle isolation from rat plasma co-precipitate vesicle-free microRNAs. J Extracell Vesicles. 8 (1), 1555410 (2019).
- De Sousa, K. P., et al. Isolation and characterization of extracellular vesicles and future directions in diagnosis and therapy. Wiley Interdiscip Rev Nanomed Nanobiotechnol. 15 (1), e1835 (2023).
- Sidhom, K., Obi, P. O., Saleem, A. A review of exosomal isolation methods: Is size exclusion chromatography the best option. Int J Mol Sci. 21 (18), 6466 (2020).
- Coughlan, C., et al. Exosome isolation by ultracentrifugation and precipitation and techniques for downstream analyses. Curr Protoc Cell Biol. 88 (1), e110 (2020).
- Konoshenko, M. Y., et al. Isolation of extracellular vesicles from biological fluids via the aggregation-precipitation approach for downstream miRNAs detection. Diagnostics (Basel). 11 (3), 384 (2021).
- Drozdz, A., et al. Low-vacuum filtration as an alternative extracellular vesicle concentration method: A comparison with ultracentrifugation and differential centrifugation. Pharmaceutics. 12 (9), 872 (2020).
- Akbar, A., Malekian, F., Baghban, N., Kodam, S. P., Ullah, M. Methodologies to isolate and purify clinical grade extracellular vesicles for medical applications. Cells. 11 (2), 186 (2022).
- Chen, J., et al. Review on strategies and technologies for exosome isolation and purification. Front Bioeng Biotechnol. 9, 811971 (2021).
- Helling, A., et al. Investigation of microbial cell deformability by filter cake compressibility using ultrafiltration membranes. Colloids Surf B Biointerfaces. 185, 110626 (2020).
- Bagci, C., et al. Overview of extracellular vesicle characterization techniques and introduction to combined reflectance and fluorescence confocal microscopy to distinguish extracellular vesicle subpopulations. Neurophotonics. 9 (2), 021903 (2022).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750 (2018).
- van Maanen, J. C., et al. A combined western and bead-based multiplex platform to characterize extracellular vesicles. Tissue Eng Part C Methods. 29 (11), 493-504 (2023).
- Xu, D., et al. MicroRNAs in extracellular vesicles: Sorting mechanisms, diagnostic value, isolation, and detection technology. Front Bioeng Biotechnol. 10, 948959 (2022).
- Lee, H., He, X., Le, T., Carnino, J. M., Jin, Y. Single-step RT-qPCR for detection of extracellular vesicle microRNAs in vivo: a time- and cost-effective method. Am J Physiol Lung Cell Mol Physiol. 318 (4), L742-L749 (2020).
- Kim, J. A., et al. Small RNA sequencing of circulating small extracellular vesicles microRNAs in patients with amyotrophic lateral sclerosis. Sci Rep. 13 (1), 5528 (2023).
- Fan, Y., et al. Differential proteomics argues against a general role for CD9, CD81 or CD63 in the sorting of proteins into extracellular vesicles. J Extracell Vesicles. 12 (8), e12352 (2023).
- Silva, A. M., et al. Quantification of protein cargo loading into engineered extracellular vesicles at single-vesicle and single-molecule resolution. J Extracell Vesicles. 10 (10), e12130 (2021).
- Salunkhe, S., Basak Dheeraj, M., Chitkara, D., Mittal, A. Surface functionalization of exosomes for target-specific delivery and in vivo imaging, tracking: Strategies and significance. J Control Release. 326, 599-614 (2020).
- Silva, A. K. A., et al. Development of extracellular vesicle-based medicinal products: A position paper of the group "Extracellular Vesicle translatiOn to clinicaL perspectiVEs - EVOLVE France". Adv Drug Deliv Rev. 179, 114001 (2021).
- Gurunathan, S., Kang, M. H., Jeyaraj, M., Qasim, M., Kim, J. H. Review of the isolation, characterization, biological function, and multifarious therapeutic approaches of exosomes. Cells. 8 (4), 307 (2019).
- Zhang, Y., et al. Exosome: A review of its classification, isolation techniques, storage, diagnostic and targeted therapy applications. Int J Nanomedicine. 15, 6917-6934 (2020).
- Colombo, M., Raposo, G., Thery, C. Biogenesis, secretion, and intercellular interactions of exosomes and other extracellular vesicles. Annu Rev Cell Dev Biol. 30, 255-289 (2014).
- Pisitkun, T., Shen, R. F., Knepper, M. A. Identification and proteomic profiling of exosomes in human urine. Proc Natl Acad Sci U S A. 101 (36), 13368-13373 (2004).
- Zaborowski, M. P., Balaj, L., Breakefield, X. O., Lai, C. P. Extracellular vesicles: Composition, biological relevance, and methods of study. Bioscience. 65 (8), 783-797 (2015).
- Kowkabany, G., Bao, Y. Nanoparticle tracking analysis: An effective tool to characterize extracellular vesicles. Molecules. 29 (19), 4672 (2024).
- Li, H. Metastatic characteristics of SY86B human gastric carcinoma in athymic nude mice. Zhonghua Zhong Liu Za Zhi. 10 (6), 421-423 (1988).
- Zhao, Z., Wijerathne, H., Godwin, A. K., Soper, S. A. Isolation and analysis methods of extracellular vesicles (EVs). Extracell Vesicles Circ Nucl Acids. 2, 80-103 (2021).
- Gandham, S., et al. Technologies and standardization in research on extracellular vesicles. Trends Biotechnol. 38 (10), 1066-1098 (2020).
- van der Vlist, E. J., Nolte-'t Hoen, E. N., Stoorvogel, W., Arkesteijn, G. J., Wauben, M. H. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nat Protoc. 7 (7), 1311-1326 (2012).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. J Extracell Vesicles. 4, 25530 (2015).
- Breitwieser, K., et al. Detailed characterization of small extracellular vesicles from different cell types based on tetraspanin composition by ExoView R100 platform. Int J Mol Sci. 23 (15), 8544 (2022).
- An, H. J., Cho, H. K., Song, D. H., Kee, C. Quantitative analysis of exosomes in the aqueous humor of Korean patients with pseudoexfoliation glaucoma. Sci Rep. 12 (1), 12875 (2022).
- Hefley, B. S., McKay, T. B., Hutcheon, A. E. K., Ciolino, J. B., Karamichos, D. Corneal epithelial-stromal constructs to study differences associated with diabetes mellitus. Exp Eye Res. 248, 110100 (2024).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır