Method Article
Caratterizzazione delle vescicole extracellulari da fluidi biologici
In questo articolo
Riepilogo
Qui, dimostriamo il metodo di caratterizzazione delle vescicole extracellulari (EV) raccolte da fluidi biologici, come lacrime e saliva, di soggetti umani. Lo scanner utilizzato in questo metodo è in grado di rilevare il fenotipo, le dimensioni e il conteggio totale delle particelle delle vescicole extracellulari a partire da 1 μL del campione.
Abstract
Le vescicole extracellulari (EV) sono strutture prodotte dalle cellule e partecipano alla comunicazione intercellulare trasportando biomolecole da una cellula all'altra. È stato dimostrato che le vescicole extracellulari viaggiano su brevi e lunghe distanze nel corpo e sono tessuto-specifiche. Le vescicole extracellulari non si trovano solo nei tessuti, ma possono anche essere trovate praticamente in tutti i fluidi corporei, come lacrime, saliva, liquido cerebrospinale, sangue, ecc. Anche se le vescicole extracellulari possono essere raccolte in modo non invasivo da lacrime e saliva, è possibile raccogliere solo piccoli volumi alla volta, il che può causare problemi nell'ottenere abbastanza vescicole extracellulari per analizzare le proteine. Lo scanner discusso in questo articolo è un analizzatore di nanoparticelle che fornisce una soluzione a questo problema, permettendoci di caratterizzare e studiare il fenotipo, le dimensioni e il numero totale di particelle delle vescicole extracellulari da un minimo di 1 μL di fluido biologico. Questo protocollo amplierà la conoscenza delle vescicole extracellulari da piccoli volumi di campioni difficili da estrarre dai pazienti. Ciò potrebbe migliorare il comfort del paziente e potenzialmente identificare nuovi bersagli terapeutici per una serie di malattie e disturbi.
Introduzione
Le cellule comunicano con le cellule vicine attraverso vari meccanismi di segnalazione, tra cui il rilascio di vescicole extracellulari (EV), che svolgono un ruolo chiave nella comunicazione intercellulare. Le vescicole extracellulari partecipano alla comunicazione intercellulare passando il carico genetico, come DNA, RNA e proteine, da una cellula all'altra 1,2,3,4,5. Attualmente, ci sono tre categorie di vescicole extracellulari: esosomi, microvescicole e corpi apoptotici, che si caratterizzano per le loro dimensioni. Gli esosomi sono i più piccoli, con un diametro di 30-150 nm 6,7,8, e sono formati dal sistema di membrana endosomiale 9,10,11 (Figura 1). Le microvescicole sono più grandi degli esosomi in quanto vanno da 100-1000 nm 12,13,14 e gemma fuori dalla membrana plasmatica 11,12,13 (Figura 1). I corpi apoptotici sono i più grandi delle vescicole extracellulari e vanno da 1000 a 5000 nm 12,14,15, e germogliano anche dalla membrana plasmatica 12,16,17 (Figura 1). Oltre alle dimensioni, le vescicole extracellulari possono essere classificate in base ad altre caratteristiche biofisiche, che includono densità, marcatori molecolari come CD63, CD81, CD9 e anche il meccanismo di biogenesi18. Le vescicole extracellulari possono percorrere brevi distanze tra cellule adiacenti e lunghe distanze in tutto il corpo 19,20,21,22,23. Le vescicole extracellulari possono essere trovate in fluidi biologici come il sangue 24,25,26, il liquido cerebrospinale 27,28,29, le lacrime 30,31,32 e la saliva 33,34,35, solo per citarne alcuni.
Ad oggi, l'ultracentrifugazione è uno dei metodi più noti per isolare le vescicole extracellulari dai campioni 36,37,38. Questo metodo richiede più cicli di centrifugazioni e ultracentrifugazione che possono essere eseguite aumentando la velocità di isolamento delle vescicole extracellulari dalle cellule e dai detriti cellulari. Questo metodo può essere avviato con la bassa velocità con celle a pellet che sarà seguita da una velocità media per eliminare le vescicole più grandi e, infine, una fase di ultracentrifugazione per pellettare EVs18. Sebbene l'ultracentrifugazione sia considerata il metodo migliore per l'isolamento, esistono ancora alcune limitazioni in quanto modifica la morfologia delle vescicole extracellulari 39,40,41. Un'altra tecnica utilizzata per isolare le vescicole extracellulari è la citometria a flusso, che evidenzia i vantaggi della valutazione di più punti temporali e finali e dell'analisi di singole vescicole extracellulari ad alto rendimento. Tuttavia, i limiti della citometria a flusso includono, a titolo esemplificativo ma non esaustivo, l'ostruzione dei pori e i segnali deboli. Un altro approccio utilizzato è la centrifugazione in gradiente, che utilizza materiali con densità diverse da centrifugare con le vescicole extracellulari e consente una migliore separazione delle vescicole extracellulari rispetto all'ultracentrifugazione. Sebbene questa tecnica migliori la separazione, è laboriosa, richiede molto tempo e può portare a una significativa perdita di campione. Inoltre, la precipitazione e la filtrazione possono essere utilizzate anche per isolare le vescicole extracellulari. Entrambe queste tecniche sono semplici e veloci, ma entrambe possono portare alla contaminazione del campione. Sebbene esistano diverse tecniche per isolare le vescicole extracellulari, ciascuna tecnica ha i suoi vantaggi e limiti, che sono elencati di seguito (Tabella 1) 42,43,44,45,46,47,48,49,50,51,52,53,54, 55, 56,57,58,59,60,61,62,63,64,65,66,67.
Una volta che le vescicole extracellulari sono state isolate, possono essere eseguiti saggi molecolari per caratterizzarle. I western blot sono un test comune per cercare l'espressione proteica di superficie e cargo 68,69,70 e la reazione a catena della polimerasi (PCR) è utilizzata per l'espressione di miRNA 71,72,73 per le EV 74,75,76. Questi saggi sono consolidati e possono generare risultati intriganti. Un limite di questi metodi è che richiedono una grande quantità di proteine o RNA dalle vescicole extracellulari per ottenere una lettura77, che è un problema per i campioni che hanno un piccolo volume o concentrazione di vescicole extracellulari, per cominciare.
L'analizzatore di nanoparticelle discusso in questo documento consente all'utente di superare molte delle limitazioni indicate nella Tabella 1 e nella Tabella 2 78,79,80,81,82,83,84,85,86,87,88,89,90 . Questo metodo non richiede l'utilizzo di tecniche di isolamento, che aiuteranno a superare la ridotta resa delle vescicole extracellulari. Questo metodo consente inoltre all'utente di analizzare le proteine di superficie e di carico, la conta totale delle vescicole extracellulari e le dimensioni delle vescicole extracellulari a partire da un volume di campione di appena 1 μl. Ciò viene fatto utilizzando chip di tetraspanina forniti dall'azienda che utilizza un microarray di anticorpi con anticorpi tetraspanina, CD63, CD81 e CD9, per identificare le vescicole extracellulari in una soluzione, come mostrato nella Figura 2. Gli anticorpi fluorescenti confermano la presenza di vescicole extracellulari e impediscono alle particelle contaminanti di distorcere i risultati.
L'obiettivo generale di questa tecnica è quello di fornire un metodo meno dispendioso in termini di tempo per analizzare le vescicole extracellulari e analizzare le vescicole extracellulari da un piccolo volume di campione. L'utilizzo di questo analizzatore di nanoparticelle consente agli utenti di analizzare le dimensioni, il numero totale di particelle e le proteine di superficie da un minimo di 1 μL di campione, ideale per fluidi biologici come lacrime e saliva.
Protocollo
Tutti gli studi descritti hanno aderito alla Dichiarazione di Helsinki. Il consenso scritto è stato ottenuto da ciascun soggetto prima di essere incluso nello studio. L'approvazione dell'Institutional Review Board (IRB) da parte dell'Aarhus University Hospital (1-10-72-77-14) e del Dean McGee Institute (1576837-2) è stata ricevuta secondo le linee guida federali e istituzionali. Prima dell'elaborazione, tutti i campioni di lacrime e saliva sono stati resi anonimi. Tutti gli studi sono stati esaminati e approvati dal North Texas Regional Institutional Review Board (#2020-030). Il seguente protocollo aderisce a tutte le linee guida ed è stato approvato, come menzionato sopra.
1. Giorno 1: Preparazione del campione e incubazione
- Raccogliere campioni di fluido biologico, ad esempio lacrime e saliva da un soggetto o scongelare i campioni dal congelatore.
- Lacrime: Raccogliere passivamente le lacrime dal menisco laterale utilizzando un tubo capillare di vetro.
- Posizionare il tubo capillare di vetro sulla palpebra inferiore, facendo attenzione a non toccare la cornea.
- Raccogliere un volume di 10 μL di lacrime entro 10 minuti.
- Utilizzare immediatamente le lacrime o conservarle a -20 °C.
- Saliva: Raccogliere la saliva dalla bava passiva in una provetta da microcentrifuga da 1,5 ml
- Consenti al donatore di accumulare saliva in bocca.
- Chiedi al donatore di sbavare nel tubo con un imbuto.
- Utilizzare immediatamente la saliva o conservare a -20 °C.
- Lacrime: Raccogliere passivamente le lacrime dal menisco laterale utilizzando un tubo capillare di vetro.
- Posizionare i chip di tetraspanina nella piastra di lavaggio dei trucioli per 15 minuti prima di aggiungere i campioni per consentire loro di raggiungere la temperatura ambiente. Mentre metti le fiches nel pozzo, assicurati che i numeri siano sul lato inferiore rivolto verso l'alto e che le fiches non tocchino le pareti laterali dei pozzetti.
- Registrare il numero ID e il numero di chip nel notebook per tenere traccia dei chip.
- Collegare il bus seriale universale (USB) fornito con il kit, quindi estrarre e salvare la cartella con il numero di kit associato in un'area designata a scelta.
- Pipettare 99 μl di soluzione B in una provetta da 1,5 mL.
- Aggiungere 1 μL di fluido biologico nella provetta da 1,5 mL preparata nel passaggio precedente.
- Centrifugare i tubi.
- Prelevare 70 μl della soluzione dal passaggio 1.6 e pipettare il chip di tetraspanina.
- Posizionare la pellicola sulla piastra di lavaggio dei trucioli per evitare che il campione evapori.
- Incubare per 16 ore a temperatura ambiente (RT) su un banco privo di vibrazioni.
2. Giorno 2: Lavaggio dei trucioli
- Accendi la lavatrice per trucioli.
- Rimuovere la pellicola del passaggio 1.9 dalla piastra.
- Posizionare la piastra nella rondella per trucioli, assicurandosi che si blocchi in posizione, e chiudere il coperchio.
NOTA: La piastra non si muoverà facilmente se bloccata correttamente in posizione. - Premere l'opzione CW-TETRA v0 sulla rondella trucioli.
- Selezionare la riga di partenza e il numero di file da lavare.
- Premere Continua sulla lavatrice per trucioli.
- Durante il lavaggio dei chip, preparare la soluzione bloccante contenente gli anticorpi in una provetta come segue:
- Preparare 300 μl di soluzione bloccante per chip.
- Aggiungere 0,6 μl o ciascun anticorpo per chip (anticorpi vortex e spin down prima di aggiungerli alla soluzione bloccante)
- Vortex la soluzione bloccante + cocktail di anticorpi.
NOTA: Coprire la soluzione bloccante + il cocktail di anticorpi dalla luce per garantire la stabilità degli anticorpi. Questo deve essere effettuato il giorno dell'uso.
- Quando il lavatrucioli emette un segnale acustico, aggiungere 250 μl di soluzione bloccante + cocktail di anticorpi sopra i chip.
- Chiudere il coperchio e premere Continua.
NOTA: La lavatrice per trucioli eseguirà ora un'incubazione di 1–h insieme a diversi cicli aggiuntivi di lavaggio e risciacquo. - Quando la rondella dei trucioli emette un segnale acustico, rimuovere la piastra di lavaggio dei trucioli e spostare i trucioli su per la rampa.
- Riposizionare la piastra nella rondella per trucioli, assicurandosi che si blocchi in posizione. Chiudere il coperchio e premere Continua.
- Quando la lavatrice per trucioli ha completato il programma, rimuovere la piastra e posizionare i trucioli su un tovagliolo di carta. Coprire le patatine dalla luce.
3. Giorno 2: Scansione del/i chip/i
- Accendi lo scanner e apri il software di scansione.
NOTA: Lo scanner verrà sottoposto a controlli automatici per indicare se sono presenti errori o se la scansione può essere continuata. - Seleziona Salva cartella e scegli la posizione in cui salvare i file.
- Seleziona Cartella ChipFile e scegli la cartella salvata nel passaggio 1.4.
- Selezionare l'opzione a discesa per scegliere ciascun truciolo e posizionarlo in Posizione truciolo sul mandrino. Ripetere questo passaggio per tutti i chip che devono essere scansionati.
- Attiva la fluorescenza facendo clic sui tre quadrati sotto Posizione chip. I quadrati diventeranno rispettivamente gialli, blu e rossi.
- Rimuovere il coperchio del mandrino premendo il coperchio verso il basso e tirarlo.
- Posizionare i trucioli sul mandrino, assicurandosi che siano ben fissati.
- Riposizionare il coperchio del mandrino posizionando i pioli nei fori, premere verso il basso e far scorrere il coperchio per bloccarlo in posizione.
- Posizionare il mandrino sul tavolino, assicurandosi che si blocchi in posizione.
- Seleziona Scansiona chip.
- Selezionare OK.
- Una volta che il tavolino si è completamente spostato nella macchina, chiudere lo sportello.
NOTA: La scansione di un singolo chip richiede circa 12 minuti. - Una volta terminata la scansione dei chip, aprire lo sportello per espellere il controllo del tavolino per vedere se le scansioni sono andate a buon fine.
- Se le scansioni hanno avuto esito positivo, scartare i chip e posizionare il set successivo di chip sul mandrino, oppure riportare il mandrino vuoto sul tavolino. Quindi, uscire dal software e spegnere la macchina al termine.
- Se le scansioni non sono andate a buon fine, esaminare il codice di errore e correggere l'errore.
4. Trattamento dei dati
- Aprire il software di analisi.
- Selezionare Prescansione dati e scegliere la cartella che contiene la scansione dal passaggio 3.2 del giorno 2.
- Seleziona Avanti.
- Etichettare i chip nella colonna Nome campione con il nome del campione facendo clic sulla cella e digitando l'ID del campione.
- Quindi, etichettare le tetraspanine nella colonna Nome canale . Rosso = CD63, Verde = CD81 e Blu = CD9.
- Seleziona Avanti.
- Selezionare CV alto dal gruppo Seleziona spot per esaminare la casella a discesa e disattivare i CV alti per ciascun chip disabilitando uno o due spot.
- Quindi, seleziona Conteggio alto e prova a disattivare gli avvisi di conteggio elevato.
NOTA: Alcuni campioni potrebbero avere un conteggio troppo alto, ma possono comunque essere analizzati. - Selezionare Tutti i punti dal menu a discesa, selezionare Montaggio spot e controllare visivamente ogni scheggiatura per eventuali deformazioni o graffi che potrebbero essere presenti.
- Seleziona Avanti.
- Nel canale CD63, imposta il minimo su 300 per eliminare i globuli rossi nella riga Media % inclusa; le cellule gialle vanno bene e procedono verso il canale CD81. Se l'impostazione del valore minimo su 300 non modifica la cella rossa in gialla o bianca, procedere al passaggio 4.14 per la risoluzione dei problemi.
- Nel canale CD81, impostare il valore minimo su 300 per eliminare i globuli rossi nella riga Media % inclusi; le cellule gialle vanno bene e procedono verso il canale CD9. Se l'impostazione del valore minimo su 300 non modifica la cella rossa in gialla o bianca, procedere al passaggio 4.14 per la risoluzione dei problemi.
- Nel canale CD9, imposta il minimo su 400 per eliminare i globuli rossi nella riga Media % inclusa; Le celle gialle vanno bene e procedi al passaggio 4.15. Se l'impostazione del valore minimo su 400 non modifica la cella rossa in gialla o bianca, procedere al passaggio 4.14 per la risoluzione dei problemi.
- Risoluzione dei problemi
- Aumenta il valore minimo di 100 unità.
- Se il passaggio 4.14.1 non ha funzionato, seleziona la nella sezione Scegli fiches su cui lavorare e scegli la. Seleziona Conteggio particelle e disabilita i punti con un numero elevato di particelle. Seleziona Intensità/Dimensione, seleziona Tutte le fiches e verifica se questo risolve il problema.
- Se il passaggio 4.14.2 non ha funzionato, selezionare la nella sezione Scegli fiches su cui lavorare e scegli la. Seleziona i tagli a chip singolo e aumenta il minimo di 100 unità. Quindi seleziona Tutte le fiches per vedere se la cella rossa è ora gialla. Se il problema persiste, ripetere questo passaggio fino a quando la cella % media inclusa non diventa gialla o bianca.
- Seleziona Avanti.
- Deselezionare il canale IM.
- Imposta il valore della mappa di calore nell'angolo in basso a destra per includere tutte le particelle e seleziona Aggiungi grafico al rapporto.
- Regola il numero massimo nella casella a discesa e inserisci manualmente un valore.
- Quindi, selezionare il menu a discesa in alto al centro sotto Sonde di acquisizione e selezionare CD63.
- Selezionare Aggiungi grafico al rapporto per aggiungere il conteggio totale delle particelle al rapporto.
- Selezionare il menu a discesa in Modalità analisi, scegliere Colocalizzazione e Aggiungi grafico al report.
- Quindi, seleziona Dimensione, seleziona Cambia visualizzazione in Grafico e seleziona Aggiungi grafico al rapporto.
- Infine, seleziona Immagini e seleziona Aggiungi grafico al rapporto.
- Ripetere i passaggi da 4.18 a 4.22 per CD81 e CD9, assicurandosi di deselezionare ogni volta il canale IM.
- Una volta aggiunti tutti gli elementi al report, selezionare Esporta report, assicurarsi che venga salvato nel file corretto e selezionare Cartella.
NOTA: I file Excel e le immagini sono stati generati e ora possono essere utilizzati per ulteriori analisi.
Risultati
Quando si analizzano le macchie sui chip, puntare a una concentrazione ottimale di vescicole extracellulari osservando le vescicole extracellulari marcate in fluorescenza nei canali CD63, CD81 e CD9, e il canale MIgG30,91 dovrebbe rimanere nero, poiché questo è il canale di controllo come mostrato nella Figura 3A. Il CD81 sarà ridotto per i fluidi biologici. Se non c'è fluorescenza nei canali CD63, CD81 e CD9, c'è un'assenza di vescicole extracellulari (Figura 3B). Fare attenzione a non saturare eccessivamente il chip (Figura 3C). Ciò renderà difficile per il software di analisi calcolare l'accuratezza delle dimensioni, del conteggio totale delle particelle e dei fenotipi delle vescicole extracellulari. Dai punti della concentrazione ottimale, il software di analisi sarà in grado di misurare le dimensioni (50-200 nm), il conteggio totale delle particelle e l'espressione della tetraspanina delle vescicole extracellulari (Figura 4) e sarà esportato in singoli fogli di calcolo per ulteriori analisi. L'analisi delle tetraspanine includerà la colocalizzazione delle proteine di superficie sulle vescicole extracellulari. La colocalizzazione sarà rappresentata con una "/", ad esempio "CD63/CD81". Ciò non significa che CD63 e CD81 siano combinati; significa che sia il CD63 che il CD81 si trovano sulla superficie dell'EV.
Questi risultati forniranno preziose informazioni tra campioni sani e malati. Saremo in grado di determinare se i campioni sani o i campioni malati producono più vescicole extracellulari, vescicole extracellulari più grandi o più piccole e il fenotipo delle vescicole extracellulari. Alcune o tutte queste caratteristiche potrebbero svolgere un ruolo nella biogenesi e/o nella progressione della malattia. Con questi risultati, saremo in grado di vedere se c'è un'assenza o un aumento dei livelli di tetraspanina, che possono fornire informazioni sui processi cellulari e sulla formazione di vescicole extracellulari.
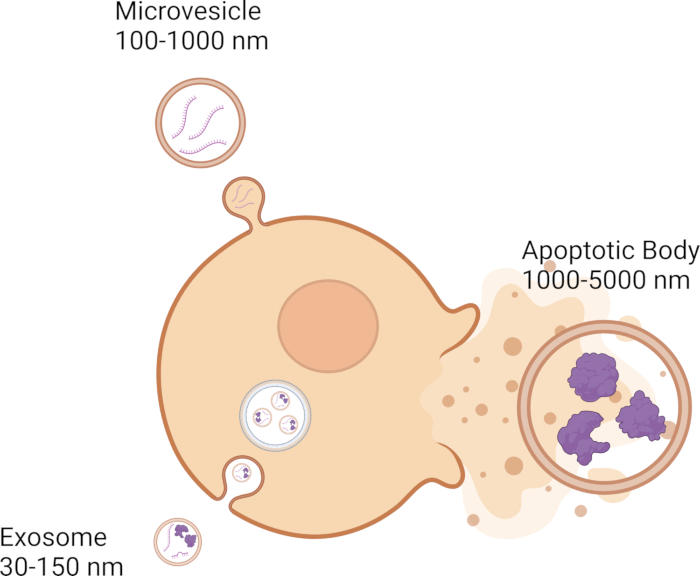
Figura 1: I tipi e le dimensioni dei diversi veicoli elettrici. Fare clic qui per visualizzare una versione più grande di questa figura.
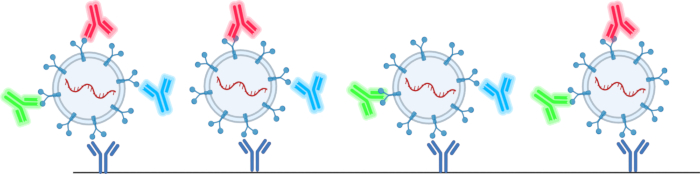
Figura 2: Schema di come i chip di tetraspanina catturano e rilevano le vescicole extracellulari. Fare clic qui per visualizzare una versione più grande di questa figura.
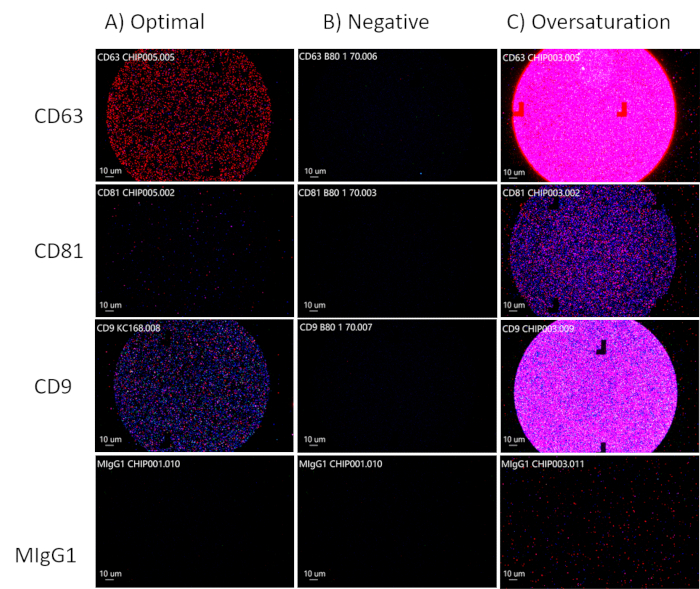
Figura 3: Ottimizzazione delle concentrazioni di EV. (A) Concentrazione ottimale di vescicole extracellulari. (B) Risultato negativo: non sono presenti EV. (C) Sovrasaturazione dei veicoli elettrici. Clicca qui per visualizzare una versione più grande di questa figura.
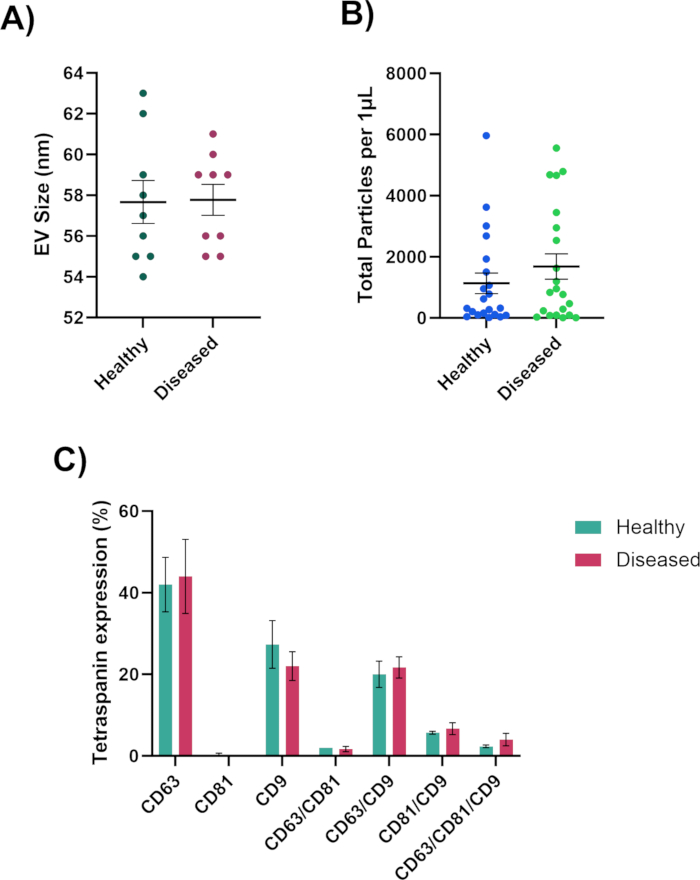
Figura 4: Dati generati dall'analizzatore di nanoparticelle. (A) Dimensione del diametro EV misurata in nanometri. (B) Conteggio totale delle particelle delle vescicole extracellulari. (C) Fenotipo colocalizzato EV. Clicca qui per visualizzare una versione più grande di questa figura.
| Tecnica | Vantaggi | Limitazioni |
| Ultracentrifuga | Elevata purezza42,43 | Modifiche Morfologia EV42,44 |
| Omogeneità42,45 | Richiede un grande volume42,44,46 | |
| Funzionalità42,43 | Costoso42,43 | |
| Citometria a flusso | Valutare i campioni in più punti temporali47 | Segnale debole48,49 |
| Endpoint multipli47,50 | Pori ostruiti51 | |
| Analisi EV singola ad alto rendimento52,53 | ||
| Centrifugazione in gradiente di densità | Produzione di campioni altamente puri54 | Manodopera intensiva55,56 |
| Varietà di campioni55,57 | Richiede tempo55,58 | |
| Perdita di resa significativa54 | ||
| Precipitazione | Semplice59,60 | Contaminazione36,44,61 |
| Veloce61,62 | Non specifico63 | |
| Alta resa44,59,62 | ||
| pH neutro36 | ||
| Filtrazione | Semplice64 | Esosomi trappola65 |
| Veloce64 | Danneggia le grandi vescicole36 | |
| Economico64 | Torta filtrante66,67 | |
| Intasamento degli esosomi65 |
Tabella 1: Vantaggi e limiti delle tecniche di isolamento delle vescicole extracellulari. Un elenco dei vari modi per isolare i veicoli elettrici, compresi i loro vantaggi e limiti.
| Tecnica | Vantaggi | Limitazioni |
| Diffusione dinamica della luce | Misura di particelle di dimensioni comprese tra 1 nm e 6 μM78 | Non adatto per la misurazione di campioni di esosomi complessi con ampio intervallo di dimensioni79 |
| Il limite inferiore è di 10 nm adatto per sistemi monodispersi79 | Non è in grado di distinguere le proteine contaminate dagli esosomi79 | |
| Microscopia elettronica a trasmissione (TEM) | Osservare la morfologia degli esosomi79,80 | Preparazioni complicate dei campioni79 |
| Osservare la struttura interna81 | Non è in grado di distinguere gli esosomi in base alle dimensioni e alla forma a causa di segnali di fluorescenza esagerati82 | |
| Analisi di tracciamento delle nanoparticelle | Misurare la concentrazione, le dimensioni e la distribuzione dimensionale degli esosomi nell'intervallo da 10 nm a 2 μM78 | Non è possibile differenziare le vescicole extracellulari dagli aggregati proteici e da altri contaminanti83 |
| Preparazione e misurazione rapide del campione78,84 | Costoso strumento NTA85 | |
| I campioni possono essere recuperati in forma nativa84 | Sensibile alle vibrazioni85 | |
| Western Blot (WB) | può analizzare qualitativamente e quantitativamente le proteine marcatrici79,86 | Complicato e dispendioso in termini di tempo79 |
| Analisi degli esosomi da terreni di coltura cellulare79 | Gli isolati di EV possono contenere lipoproteine e altri contaminanti86 | |
| Citometria a flusso | Maggiore sensibilità e imaging ad alta risoluzione per | Lungo e laborioso con limite di rilevamento di 400 nm79,88 |
| distinguere gli esosomi colorati dai contenimenti87 | ||
| Bassa concentrazione del campione richiesta79 | I segnali ottici ostacolano la precisione e la risoluzione88 | |
| Exoview | Misurazione delle tetraspanine (CD9, CD63 e CD81) sugli esosomi89 | Non misura veicoli elettrici di dimensioni maggiori75 |
| Misura EV cargo proteine90 |
Tabella 2: Vantaggi e limiti delle tecniche di caratterizzazione delle vescicole extracellulari.
Discussione
Il passaggio più critico di questo protocollo consiste nell'assicurarsi che venga raggiunta una concentrazione ottimale di vescicole extracellulari. Ci devono essere abbastanza veicoli elettrici presenti per ottenere una lettura, ma non troppi veicoli elettrici che satureranno eccessivamente il chip. Il modo migliore per determinare la concentrazione ottimale di EV è eseguire un'analisi con 1 μL di campione e vedere se la concentrazione deve essere regolata. Un altro passaggio critico è vedere se c'è un'abbondanza di detriti cellulari nel campione, che può essere determinata visualizzando grandi pezzi sui chip nel software di analisi. Se il campione contiene detriti cellulari, una semplice centrifugazione o filtraggio del campione dovrebbe risolvere questo problema.
Un ulteriore passaggio cruciale consiste nell'assicurarsi che i trucioli non tocchino le pareti del pozzetto ed evitare il contatto con l'area centrale dei trucioli quando li si posiziona sulla rampa. Lo scanner leggerà dal quadrato al centro del chip, quindi è essenziale evitare di toccare quell'area con la pinza per evitare di interrompere le EV.
Questo metodo presenta alcune limitazioni, come ad esempio una concentrazione insufficiente di vescicole extracellulari per il rilevamento da parte dello strumento. La concentrazione potrebbe essere migliorata essiccando il campione o utilizzando una provetta concentratrice. Un'altra limitazione è che il software di analisi misura solo le vescicole extracellulari nell'intervallo 50-200 nm, escludendo alcune microvescicole e tutti i corpi apoptotici dalle misure di dimensione75.
Ci sono molti modi per isolare (Tabella 1 42,43,44,45,46,47,48,49,50,51,52, 53,54,55,56,57,58,59,60, 61,62,63,64,65,66,67) e analizzare le EV (Tabella 2 78,79,80,81,82,83,84,85, 86,87,88,89,90), e l'attuale gold standard per l'isolamento delle vescicole extracellulari è l'ultracentrifugazione 36,37,38 per pellettare le cellule per saggi come Western Blots 68,69,70 e PCR 71,72,73,74,75,76. Sebbene questo protocollo funzioni bene per campioni di grandi dimensioni 42,44,46, ottenere fluidi biologici può essere difficile e, spesso, è possibile raccogliere solo un piccolo volume di campioni alla volta, il che non è l'ideale per l'ultracentrifugazione. Al contrario, l'utilizzo di questo analizzatore di nanoparticelle consente all'utente di generare dati preziosi, come le dimensioni, il conteggio totale delle particelle e il fenotipo delle vescicole extracellulari, in appena 1 μL di campione, rendendolo ideale per i fluidi biologici. Saremo in grado di ampliare la conoscenza delle vescicole extracellulari a partire da piccoli volumi di campioni difficili da estrarre, che possono aumentare il comfort del paziente e possibilmente trovare potenziali bersagli terapeutici per varie malattie e disturbi.
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti o altri conflitti di interesse da divulgare.
Riconoscimenti
Ringraziamo gli NIH per il finanziamento (EY031316 e EY034714). Vorremmo anche ringraziare UNTHSC e NTERI per lo spazio del laboratorio.
Materiali
| Name | Company | Catalog Number | Comments |
| ChipWasher 100 | NanoView | EV-CW100 | Incubates, washes, rinses and dries the tetraspanin chips. This current model is no longer available. Price at time of purchase: $9,995.00 |
| ExoView Analyzer software | NanoView | N/A | Analyzes the chip informations and produces excel files for further analysis. No longer available. |
| ExoView R100 | NanoView | EV-R100 | Used to scan the tetraspanin chips at 3 wavelengths. This current model is no longer available. Price at time of purchase: $110,000.00 |
| ExoView Scanner software | NanoView | N/A | Scans the chips at 3 different wavelengths. No longer available. |
| Human Tetraspanin Kits | Unchained Labs | EV-TETRA-C | Includes 8 tetraspanin chips, Incubation Solution, Blocking Solution, CD63 antibody, CD81 antibody, CD9 antibody, Solution A, Solution B, USB, and plate cover. |
Riferimenti
- Abels, E. R., Breakefield, X. O. Introduction to extracellular vesicles: Biogenesis, RNA cargo selection, content, release, and uptake. Cell Mol Neurobiol. 36 (3), 301-312 (2016).
- Radler, J., Gupta, D., Zickler, A., Andaloussi, S. E. Exploiting the biogenesis of extracellular vesicles for bioengineering and therapeutic cargo loading. Mol Ther. 31 (5), 1231-1250 (2023).
- Gupta, D., Zickler, A. M., El Andaloussi, S. Dosing extracellular vesicles. Adv Drug Deliv Rev. 178, 113961 (2021).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63 (2018).
- Zhang, H., et al. Exosome-induced regulation in inflammatory bowel disease. Front Immunol. 10, 1464 (2019).
- Barzin, M., et al. Application of plant-derived exosome-like nanoparticles in drug delivery. Pharm Dev Technol. 28 (5), 383-402 (2023).
- Zhou, C., et al. Stem cell-derived exosomes: emerging therapeutic opportunities for wound healing. Stem Cell Res Ther. 14 (1), 107 (2023).
- Kalluri, R., LeBleu, V. S. The biology, function, and biomedical applications of exosomes. Science. 367 (6478), aau6977 (2020).
- Mondal, J., et al. Hybrid exosomes, exosome-like nanovesicles and engineered exosomes for therapeutic applications. J Control Release. 353, 1127-1149 (2023).
- van Niel, G., D'Angelo, G., Raposo, G. Shedding light on the cell biology of extracellular vesicles. Nat Rev Mol Cell Biol. 19 (4), 213-228 (2018).
- Paul, N., Sultana, Z., Fisher, J. J., Maiti, K., Smith, R. Extracellular vesicles- crucial players in human pregnancy. Placenta. 140, 30-38 (2023).
- Zaldivia, M. T. K., McFadyen, J. D., Lim, B., Wang, X., Peter, K. Platelet-derived microvesicles in cardiovascular diseases. Front Cardiovasc Med. 4, 74 (2017).
- Tamasi, V., Nemeth, K., Csala, M. Role of extracellular vesicles in liver diseases. Life (Basel). 13 (5), 1117 (2023).
- Fu, Y., et al. Emerging understanding of apoptosis in mediating mesenchymal stem cell therapy. Cell Death Dis. 12 (6), 596 (2021).
- Santavanond, J. P., Rutter, S. F., Atkin-Smith, G. K., Poon, I. K. H. Apoptotic bodies: Mechanism of formation, isolation and functional relevance. Subcell Biochem. 97, 61-88 (2021).
- Yu, L., et al. Apoptotic bodies: bioactive treasure left behind by the dying cells with robust diagnostic and therapeutic application potentials. J Nanobiotechnology. 21 (1), 218 (2023).
- Momen-Heravi, F., Getting, S. J., Moschos, S. A. Extracellular vesicles and their nucleic acids for biomarker discovery. Pharmacol Ther. 192, 170-187 (2018).
- Liu, Y. J., Wang, C. A review of the regulatory mechanisms of extracellular vesicles-mediated intercellular communication. Cell Commun Signal. 21 (1), 77 (2023).
- Grange, C., Bussolati, B. Extracellular vesicles in kidney disease. Nat Rev Nephrol. 18 (8), 499-513 (2022).
- Cocucci, E., Meldolesi, J. Ectosomes and exosomes: shedding the confusion between extracellular vesicles. Trends Cell Biol. 25 (6), 364-372 (2015).
- Iannotta, D. A. A., Kijas, A. W., Rowan, A. E., Wolfram, J. Entry and exit of extracellular vesicles to and from the blood circulation. Nat Nanotechnol. 19 (1), 13-20 (2024).
- Saint-Pol, J., Gosselet, F., Duban-Deweer, S., Pottiez, G., Karamanos, Y. Targeting and crossing the blood-brain barrier with extracellular vesicles. Cells. 9 (4), 851 (2020).
- Alberro, A., Iparraguirre, L., Fernandes, A., Otaegui, D. Extracellular vesicles in blood: Sources, effects, and applications. Int J Mol Sci. 22 (15), 8163 (2021).
- Nieuwland, R., Siljander, P. R. A beginner's guide to study extracellular vesicles in human blood plasma and serum. J Extracell Vesicles. 13 (1), e12400 (2024).
- Thangaraju, K., Neerukonda, S. N., Katneni, U., Buehler, P. W. Extracellular vesicles from red blood cells and their evolving roles in health, coagulopathy and therapy. Int J Mol Sci. 22 (1), 153 (2020).
- Li, C., et al. Cerebrospinal fluid-derived extracellular vesicles after spinal cord injury promote vascular regeneration via PI3K/AKT signaling pathway. J Orthop Translat. 39, 124-134 (2023).
- Deng, Y., et al. Phosphoproteome analysis of cerebrospinal fluid extracellular vesicles in primary central nervous system lymphoma. Analyst. 148 (15), 3594-3602 (2023).
- Hirschberg, Y., et al. Proteomic comparison between non-purified cerebrospinal fluid and cerebrospinal fluid-derived extracellular vesicles from patients with Alzheimer's, Parkinson's and Lewy body dementia. J Extracell Vesicles. 12 (12), e12383 (2023).
- Hefley, B. S., et al. Revealing the presence of tear extracellular vesicles in Keratoconus. Exp Eye Res. 224, 109242 (2022).
- Cross, T., et al. RNA profiles of tear fluid extracellular vesicles in patients with dry eye-related symptoms. Int J Mol Sci. 24 (20), 15390 (2023).
- Ma, H., et al. Metabolic signatures of tear extracellular vesicles caused by herpes simplex keratitis. Ocul Surf. 31, 21-30 (2024).
- Han, P., Bartold, P. M., Ivanovski, S. The emerging role of small extracellular vesicles in saliva and gingival crevicular fluid as diagnostics for periodontitis. J Periodontal Res. 57 (1), 219-231 (2022).
- Han, P., Li, X., Wei, W., Ivanovski, S. Saliva diagnosis using small extracellular vesicles and salivaomics. Methods Mol Biol. 2588, 25-39 (2023).
- Reseco, L., et al. Characterization of extracellular vesicles from human saliva: Effects of age and isolation techniques. Cells. 13 (1), 95 (2024).
- Tiwari, S., Kumar, V., Randhawa, S., Verma, S. K. Preparation and characterization of extracellular vesicles. Am J Reprod Immunol. 85 (2), e13367 (2021).
- Tomiyama, E., Fujita, K., Nonomura, N. Urinary extracellular vesicles: Ultracentrifugation method. Methods Mol Biol. 2292, 173-181 (2021).
- Xu, K., Jin, Y., Li, Y., Huang, Y., Zhao, R. Recent progress of exosome isolation and peptide recognition-guided strategies for exosome research. Front Chem. 10, 844124 (2022).
- Brennan, K., et al. A comparison of methods for the isolation and separation of extracellular vesicles from protein and lipid particles in human serum. Sci Rep. 10 (1), 1039 (2020).
- Gao, J., et al. Recent developments in isolating methods for exosomes. Front Bioeng Biotechnol. 10, 1100892 (2022).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Ansari, F. J., et al. Comparison of the efficiency of ultrafiltration, precipitation, and ultracentrifugation methods for exosome isolation. Biochem Biophys Rep. 38, 101668 (2024).
- Patel, G. K., et al. Comparative analysis of exosome isolation methods using culture supernatant for optimum yield, purity and downstream applications. Sci Rep. 9 (1), 5335 (2019).
- Stam, J., Bartel, S., Bischoff, R., Wolters, J. C. Isolation of extracellular vesicles with combined enrichment methods. J Chromatogr B Analyt Technol Biomed Life Sci. 1169, 122604 (2021).
- Le Roy, A., et al. AUC and small-angle scattering for membrane proteins. Methods Enzymol. 562, 257-286 (2015).
- Chhoy, P., Brown, C. W., Amante, J. J., Mercurio, A. M. Protocol for the separation of extracellular vesicles by ultracentrifugation from in vitro cell culture models. STAR Protoc. 2 (1), 100303 (2021).
- McDorman, K. S., Chan, C., Rojko, J., Satterwhite, C. M., Morrison, J. P. . Chapter 7 - Special Techniques in Toxicologic Pathology. Haschek and Rousseaux's Handbook of Toxicologic Pathology (Third Edition). , (2013).
- Woud, W. W., et al. An imaging flow cytometry-based methodology for the analysis of single extracellular vesicles in unprocessed human plasma. Commun Biol. 5 (1), 633 (2022).
- Gul, B., Syed, F., Khan, S., Iqbal, A., Ahmad, I. Characterization of extracellular vesicles by flow cytometry: Challenges and promises. Micron. 161, 103341 (2022).
- Black, C. B., Duensing, T. D., Trinkle, L. S., Dunlay, R. T. Cell-based screening using high-throughput flow cytometry. Assay Drug Dev Technol. 9 (1), 13-20 (2011).
- Safford, H. R., Bischel, H. N. Flow cytometry applications in water treatment, distribution, and reuse: A review. Water Res. 151, 110-133 (2019).
- Kobayashi, H., et al. Precise analysis of single small extracellular vesicles using flow cytometry. Sci Rep. 14 (1), 7465 (2024).
- Welsh, J. A., et al. A compendium of single extracellular vesicle flow cytometry. J Extracell Vesicles. 12 (2), e12299 (2023).
- Carnino, J. M., Lee, H. Extracellular vesicles in respiratory disease. Adv Clin Chem. 108, 105-127 (2022).
- Sun, Y., Sethu, P. Low-stress microfluidic density-gradient centrifugation for blood cell sorting. Biomed Microdevices. 20 (3), 77 (2018).
- Strachan, B. C., Xia, H., Voros, E., Gifford, S. C., Shevkoplyas, S. S. Improved expansion of T cells in culture when isolated with an equipment-free, high-throughput, flow-through microfluidic module versus traditional density gradient centrifugation. Cytotherapy. 21 (2), 234-245 (2019).
- Malvezzi, H., Sharma, R., Agarwal, A., Abuzenadah, A. M., Abu-Elmagd, M. Sperm quality after density gradient centrifugation with three commercially available media: a controlled trial. Reprod Biol Endocrinol. 12, 121 (2014).
- Sun, Y., Sethu, P. Microfluidic adaptation of density-gradient centrifugation for isolation of particles and cells. Bioengineering (Basel). 4 (3), 67 (2017).
- Karttunen, J., et al. Precipitation-based extracellular vesicle isolation from rat plasma co-precipitate vesicle-free microRNAs. J Extracell Vesicles. 8 (1), 1555410 (2019).
- De Sousa, K. P., et al. Isolation and characterization of extracellular vesicles and future directions in diagnosis and therapy. Wiley Interdiscip Rev Nanomed Nanobiotechnol. 15 (1), e1835 (2023).
- Sidhom, K., Obi, P. O., Saleem, A. A review of exosomal isolation methods: Is size exclusion chromatography the best option. Int J Mol Sci. 21 (18), 6466 (2020).
- Coughlan, C., et al. Exosome isolation by ultracentrifugation and precipitation and techniques for downstream analyses. Curr Protoc Cell Biol. 88 (1), e110 (2020).
- Konoshenko, M. Y., et al. Isolation of extracellular vesicles from biological fluids via the aggregation-precipitation approach for downstream miRNAs detection. Diagnostics (Basel). 11 (3), 384 (2021).
- Drozdz, A., et al. Low-vacuum filtration as an alternative extracellular vesicle concentration method: A comparison with ultracentrifugation and differential centrifugation. Pharmaceutics. 12 (9), 872 (2020).
- Akbar, A., Malekian, F., Baghban, N., Kodam, S. P., Ullah, M. Methodologies to isolate and purify clinical grade extracellular vesicles for medical applications. Cells. 11 (2), 186 (2022).
- Chen, J., et al. Review on strategies and technologies for exosome isolation and purification. Front Bioeng Biotechnol. 9, 811971 (2021).
- Helling, A., et al. Investigation of microbial cell deformability by filter cake compressibility using ultrafiltration membranes. Colloids Surf B Biointerfaces. 185, 110626 (2020).
- Bagci, C., et al. Overview of extracellular vesicle characterization techniques and introduction to combined reflectance and fluorescence confocal microscopy to distinguish extracellular vesicle subpopulations. Neurophotonics. 9 (2), 021903 (2022).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750 (2018).
- van Maanen, J. C., et al. A combined western and bead-based multiplex platform to characterize extracellular vesicles. Tissue Eng Part C Methods. 29 (11), 493-504 (2023).
- Xu, D., et al. MicroRNAs in extracellular vesicles: Sorting mechanisms, diagnostic value, isolation, and detection technology. Front Bioeng Biotechnol. 10, 948959 (2022).
- Lee, H., He, X., Le, T., Carnino, J. M., Jin, Y. Single-step RT-qPCR for detection of extracellular vesicle microRNAs in vivo: a time- and cost-effective method. Am J Physiol Lung Cell Mol Physiol. 318 (4), L742-L749 (2020).
- Kim, J. A., et al. Small RNA sequencing of circulating small extracellular vesicles microRNAs in patients with amyotrophic lateral sclerosis. Sci Rep. 13 (1), 5528 (2023).
- Fan, Y., et al. Differential proteomics argues against a general role for CD9, CD81 or CD63 in the sorting of proteins into extracellular vesicles. J Extracell Vesicles. 12 (8), e12352 (2023).
- Silva, A. M., et al. Quantification of protein cargo loading into engineered extracellular vesicles at single-vesicle and single-molecule resolution. J Extracell Vesicles. 10 (10), e12130 (2021).
- Salunkhe, S., Basak Dheeraj, M., Chitkara, D., Mittal, A. Surface functionalization of exosomes for target-specific delivery and in vivo imaging, tracking: Strategies and significance. J Control Release. 326, 599-614 (2020).
- Silva, A. K. A., et al. Development of extracellular vesicle-based medicinal products: A position paper of the group "Extracellular Vesicle translatiOn to clinicaL perspectiVEs - EVOLVE France". Adv Drug Deliv Rev. 179, 114001 (2021).
- Gurunathan, S., Kang, M. H., Jeyaraj, M., Qasim, M., Kim, J. H. Review of the isolation, characterization, biological function, and multifarious therapeutic approaches of exosomes. Cells. 8 (4), 307 (2019).
- Zhang, Y., et al. Exosome: A review of its classification, isolation techniques, storage, diagnostic and targeted therapy applications. Int J Nanomedicine. 15, 6917-6934 (2020).
- Colombo, M., Raposo, G., Thery, C. Biogenesis, secretion, and intercellular interactions of exosomes and other extracellular vesicles. Annu Rev Cell Dev Biol. 30, 255-289 (2014).
- Pisitkun, T., Shen, R. F., Knepper, M. A. Identification and proteomic profiling of exosomes in human urine. Proc Natl Acad Sci U S A. 101 (36), 13368-13373 (2004).
- Zaborowski, M. P., Balaj, L., Breakefield, X. O., Lai, C. P. Extracellular vesicles: Composition, biological relevance, and methods of study. Bioscience. 65 (8), 783-797 (2015).
- Kowkabany, G., Bao, Y. Nanoparticle tracking analysis: An effective tool to characterize extracellular vesicles. Molecules. 29 (19), 4672 (2024).
- Li, H. Metastatic characteristics of SY86B human gastric carcinoma in athymic nude mice. Zhonghua Zhong Liu Za Zhi. 10 (6), 421-423 (1988).
- Zhao, Z., Wijerathne, H., Godwin, A. K., Soper, S. A. Isolation and analysis methods of extracellular vesicles (EVs). Extracell Vesicles Circ Nucl Acids. 2, 80-103 (2021).
- Gandham, S., et al. Technologies and standardization in research on extracellular vesicles. Trends Biotechnol. 38 (10), 1066-1098 (2020).
- van der Vlist, E. J., Nolte-'t Hoen, E. N., Stoorvogel, W., Arkesteijn, G. J., Wauben, M. H. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nat Protoc. 7 (7), 1311-1326 (2012).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. J Extracell Vesicles. 4, 25530 (2015).
- Breitwieser, K., et al. Detailed characterization of small extracellular vesicles from different cell types based on tetraspanin composition by ExoView R100 platform. Int J Mol Sci. 23 (15), 8544 (2022).
- An, H. J., Cho, H. K., Song, D. H., Kee, C. Quantitative analysis of exosomes in the aqueous humor of Korean patients with pseudoexfoliation glaucoma. Sci Rep. 12 (1), 12875 (2022).
- Hefley, B. S., McKay, T. B., Hutcheon, A. E. K., Ciolino, J. B., Karamichos, D. Corneal epithelial-stromal constructs to study differences associated with diabetes mellitus. Exp Eye Res. 248, 110100 (2024).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon