Method Article
生体分子の局所柔軟性を特徴付けるための単一分子からの時間分解蛍光異方性
要約
ここでは、共焦点顕微鏡モードで単一分子レベルでの時間分解蛍光異方性を使用して、生体分子の局所的な柔軟性とダイナミクスを研究するためのプロトコルを紹介します。
要約
共焦点顕微鏡を使用して、FoxP1転写因子のデオキシリボ核酸(DNA)結合フォークヘッド(FKH)ドメインの局所的な柔軟性とダイナミクスを調査するために、共焦点顕微鏡を使用して単一分子レベルで時間分解蛍光異方性を実施するためのプロトコルについて説明します。FoxP1は、3次元ドメインスワッピング(3D-DS)メカニズムを通じて二量体化し、DNAの有無にかかわらず無秩序な中間体を形成します。3D-DSは天然変性領域を含んでいるため、その振る舞いを理解することは、FoxP1の構造的および機能的特性を解明するために重要です。単一システイン標識FoxP1を使用して、動的異方性光分布分析(daPDA)および時間分解異方性バースト分散分析(traBVA)アプローチを適用して、単一分子蛍光異方性(smFA)実験を実施して、局所的な柔軟性とダイナミクスを調査しました。このプロトコルは、smFA測定の詳細なステップバイステップガイドを提供し、時間分解分析、分散、および確率分布手法を強調して、さまざまなタイムスケールにわたる構造ダイナミクスをキャプチャします。このアプローチにより、ダイナミクスと不均一性をFoxP1の二量体化とDNA結合に関連付けることができ、この転写因子を特徴付ける複雑な作用メカニズムが浮き彫りになりました。
概要
生体分子の機能活性は、その分子柔軟性と構造ダイナミクスに依存する1,2,3。当然のことながら、生体分子は、急速な動きから、その機能に影響を与える長期的な構造変化まで、一定の熱変動を経験します(図1)4。生体分子では、局所的な骨格運動が、酵素のヒンジ曲げやモータータンパク質の大幅な構造変化など、より大規模な地球規模の運動に寄与します。核磁気共鳴(NMR)5、X線結晶構造解析6、極低温電子顕微鏡(クライオEM)7などの構造決定法により、さまざまな生体分子の複数の立体配座が明らかになりました。それにもかかわらず、局所的な揺らぎを生体分子の大きなコンフォメーションダイナミクスと結びつけ、その機能における役割を解明することは、ほとんど解明されていません。ダイナミクスと構造を関連付けることは、特に天然変性タンパク質(IDP)8,9,10にとって困難な場合があります。構造化タンパク質とは異なり、IDPは安定した三次構造を維持しません。それどころか、それらは同様の自由エネルギー準位で広範なコンフォメーション変化を受け、広範囲の生物学的活動を可能にする11,12。
タンパク質の分子柔軟性を探ることにより、タンパク質のコンフォメーションダイナミクスを調査するために、いくつかの実験的アプローチが採用されてきた1,13,14,15,16。その中でも、NMRは、数十ピコ秒から数時間12までのさまざまな時間スケールで原子レベルの分解能を提供できることで際立っています。しかし、高分子の柔軟性を決定することは、自由度が高いことや大型のタンパク質の場合、依然として困難です。したがって、NMRはしばしば約100 kDaの生体分子の研究に限定されます17。
IDPのような非常に動的なタンパク質の構造の複雑さを考えると、その機能を理解するために局所的および長距離の立体配座空間を探索するための追加の方法論的進歩が開発されました11。1分子マルチパラメータ蛍光分光法(smMFS)18,19,20,21,22は、生体分子に関する広範な情報を提供し、その機能、立体配座ダイナミクス、結合状態、および化学量論に関する重要な洞察を提供します。しかし、生体分子から得られる膨大な量の構造データを解釈することは困難であり、分子動力学、蛍光色素の挙動、分子の複雑な挙動などの要因がデータ解析をさらに複雑にする可能性があります23,24,25,26,27,28。
私たちは、生体分子の骨格に沿った局所的および全体的なダイナミクスを評価するための堅牢な方法として、単一分子蛍光異方性(smFA)を採用しています(図1A)。蛍光異方性は、Perrin29によって最初に記述され、Weber30,31によってバイオ分析ツール32として導入されましたが、後に時間分解蛍光技術の出現と検出器の感度の向上により、1分子研究に適応されました33,34,35,36,37.smFAは、ピコ秒から数時間までの幅広い時間スケールにまたがり、単一分子のフェルスター共鳴エネルギー移動(smFRET)実験から得られたデータを補完する38。
smFAは、生体分子動力学に関する重要な情報を抽出するために、さまざまな形式で視覚化できます(図1B)。時間分解蛍光異方性減衰は、ピコ秒からナノ秒の時間スケール39,40のダイナミクスを捉える1次元ヒストグラムです。蛍光寿命と個々の分子の異方性を相関させる2次元単一分子ヒストグラムは、共焦点実験における異方性状態の不均一性を明らかにし、観察時間内の潜在的なダイナミクスへの視覚的な洞察を提供することができる(~ms)41,42。サブミリ秒ダイナミクスの研究には、動的異方性光子分布解析(daPDA)を使用でき、時間分解異方性バースト分散解析(traBVA)は、ミリ秒43付近の特定のダイナミクスを確認するための堅牢な方法を提供します(図1B)。
これらの方法は、偏光分解蛍光相関分光法(pFCS)などの従来のツールを補完するもので、スペクトルは44,45,46,47です。全体として、smFA用の複数のデータ解析ツールは、適切なキャリブレーションが考慮されれば、局所的および全体的なコンフォメーション変化の同定を容易にします。
ここでは、smFAを適用して、ヒトFoxP1転写因子48,49,50,51のDNA結合を研究します。このタンパク質は、そのポリペプチド鎖の天然変性のためにドメインスワップダイマーを採用しており、これはタンパク質の四次状態とDNAの存在によって特に影響を受けます。BODIPY-FLで標識するために異なる単一システイン変異体を作製し、smFA実験を行い、daPDAとtrBVAaを採用しました。このアプローチにより、ダイナミクスと不均一性をFoxP1の二量体化とDNA結合に関連付けることができ、この転写因子を特徴付ける複雑な作用機序が浮き彫りになりました。
プロトコル
注:適切な蛍光色素の選択は、smFA実験に不可欠です。生体分子は、利用可能な反応基に応じて、タンパク質中のアミノ酸または核酸中のヌクレオチド塩基を蛍光マーカーで修飾することにより、部位特異的な位置で標識することができます。有機色素52の中で、Alexa Fluor、Cy、BODIPY、およびJanelia Farmsファミリーは、その長い蛍光寿命、光安定性、および高い量子収率のおかげで、smFAの最も人気のある選択肢です。BODIPY-FLは、蛍光寿命が長く、量子収率が優れており、接続リンカーが短いため、よく好まれます。さらに、代替蛍光色素は、バルク技術が好まれる薬物スクリーニングで一般的に使用されます53。キメラ蛍光タンパク質は、生細胞の異方性実験やイメージングにも使用できますが、ダイナミックレンジが小さいという制限があります。
1. バッファーの調製
注:実験室での実験を行うときは、手袋、目の保護ゴーグル、実験室用コートを着用してください。
- 標準緩衝液(20 mM 4-(2-ヒドロキシエチル)-1-ピペラジンエタンスルホン酸[HEPES]、pH 7.8、150 mM NaCl):2.38 gのHEPES、4.38 gのNaClを400 mLの超純水に溶解し、pHを7.8に調整し、最終容量を500 mLにします。
- 溶解バッファー(20 mM HEPES、pH 7.8、150 mM NaCl、0.1 mM フェニルメチルスルホニルフッ化物[PMSF]、10 μg/mL DNAse):2.38 gのHEPES、4.38 gのNaCl、8.71 mgのPMSFと最終濃度10 μg/mLのDNAseを400 mLの超純水に溶解し、pHを7.8に調整し、最終容量を500 mLにします。
- 平衡化バッファー(50 mM リン酸緩衝生理食塩水 [PBS]、150 mM NaCl、10 mM イミダゾール、pH 7.4):5.05 g の Na2HPO4、0.85 g の NaH2PO4、4.38 g の NaCl、0.34 g のイミダゾールを 400 mL の超純水に溶解し、pH を 7.4 に調整し、最終容量を 500 mL にします。
- 洗浄バッファー(50 mM PBS、150 mM NaCl、および30 mM Imidazole、pH 7.4):5.05 gのNa2HPO4、0.85 gのNaH2PO4、4.38 gのNaCl、および1.02 gのイミダゾールを400 mLの超純水に溶解し、pHを7.4に調整し、最終容量を500 mLにします。
- 溶出バッファー(50 mM PBS、150 mM NaCl、および250 mMイミダゾール、pH 7.4):5.05 gのNa2HPO4、0.85 gのNaH2PO4、4.38 gのNaCl、8.5 gのイミダゾールを400 mLの超純水に溶解し、pHを7.4に調整し、最終容量を500 mLにします。
- PBS緩衝液(50 mMリン酸ナトリウム緩衝生理食塩水、150 mM NaCl、pH 7.4)。400mLの超純水にNa2HPO4 5.05 g、NaH2PO4 0.85 g、NaCl 4.38 gを溶解し、pHを7.4に調整し、最終容量を500 mLにします。
- 0.22 μmのポアサイズフィルターを使用して溶液をろ過し、すべてのバッファーを滅菌し、バッファーを室温(RT)で保存します。
- チャコールフィルターを使用して標準バッファーをろ過し、単一分子を取り込みます。
2. 蛍光プローブ
- BODIPY-FL:BODIPY-FLを含む5mgのバイアルを溶解し、1.29mLの最終容量の新しく開封したジメチルスルホキシド(DMSO)を加えて、10 mMのBODIPY-FLの最終濃度を得ます。
- ローダミン110:ローダミン110の最終容量1 mLを加えて3.67 mgのローダミン110を溶解し、最終濃度の10 mMローダミン110を得ます。
注意: 蛍光プローブは、光にさらされるときは避ける必要があります。必ず感光性チューブ(琥珀色)を使用し、アルミホイルで包んでください。DMSOがすでに開封されている場合は、保存期間を延ばすためにデシケーターに保管する必要があります。 - 調製した蛍光プローブの少量アリコートを調製し、さらに使用するまで-20°Cで保存します。
注意: ラベリング効率を向上させるために、凍結融解サイクルを避けてください。
3. キャリブレーション測定
- 4つの検出チャネルと2つのパルスレーザー(青485nmおよび赤640nm)54を備えた自作の偏光分解セットアップでマルチパラメータ蛍光検出(MFD)実験を実行します。ただし、異方性測定には、青色レーザーと2つの検出器チャンネルを使用します。
- 検出器チャネル(平行および垂直)と青色レーザー出力をオンにします。
- レーザー出力が60μWに設定されていることを確認し、時間相関のある単一光子計数(TCSPC)ソフトウェア36 コントロールパネルを開きます。
- 100 nM Rhodamine 110 1 μL と 49 μL の蒸留水を混合して、最終濃度 2 nM Rhodamine 110 を得ます。キャリブレーション測定のために、カバーガラスの中央に50 μLの2 nM Rhodamine 110を加えます。
- 次に、顕微鏡の対物レンズの上に浸漬液(油または水のいずれかですが、同じ屈折率を共有するように注意してください)を一滴加えて、顕微鏡の解像力を高めます。
- 対物レンズの上にカバーガラスを置き、水滴が顕微鏡の対物レンズの中心にあることを確認します。
- イメージプレーンが溶液の内側、ガラス表面より上に来るように調整します。
- ノブを調整して2番目の明るい焦点を見つけ、1.5ポイントに回します。レーザーをガラスと液体の界面に集束させます。
- フォトンカウントレートを監視しながらピンホール位置(70μm)を調整することで、検出されるフォトンの数を最大化します。
- タイムタグ付き時間解決 (TTTR または T3) モード パネルでソフトウェアを開き、[ スタート ] ボタンをクリックします。120秒間のカウントレートを記録し、取得ソフトウェアを使用して Rhodamine 110.ptu ファイル形式を保存します。この取得時間は、濃度(2 nM)を考慮すると十分です。
- バックグラウンド測定の場合は、カバーガラスの中央に50 μLの蒸留水を加え、手順3.5〜3.10を繰り返します。ただし、フォトン カウント レートを 300 秒間記録し、 water.ptu ファイルを保存します。
- その他のバックグラウンド測定では、カバーガラスの中央に50 μLの標準バッファーを加え、手順3.5〜3.10を繰り返します。フォトンカウントレートを300秒間記録し、 標準のbuffer.ptu ファイルを保存します。次に、バースト積分蛍光寿命(BIFL)解析ソフトウェアを使用してデータを解析します。
4. キャリブレーションとデータ解析
- BIFLソフトウェアを開き、自動ウィンドウから [設定の確認 ]をクリックします。次に、[ ファイルからパラメータを取得]をクリックし、[ OK]をクリックします。
- 分析する測定については、 データパス配列 をクリックして 選択します 。
- 次に、Green scatter などの配列の水測定 water.ptu ファイルを読み込みます。同様に、緑の背景に標準の buffer.ptu ファイルを選択します。[太い緑] で [Rhodamine 110.ptu] を選択します。
- single molecule選択パラメータの下で、[ Next]をクリックし、[ Adjust ]をクリックして新しいポップアップウィンドウを表示します。 [しきい値 ] をクリックして光子間到着時間を変更し、平均光子間到着時間 (dt) に 1 分子イベント時間を選択します。次に、 min. # をクリックして 1 分子イベントあたりの最小光子数を選択し、 Return をクリックしてポップアップウィンドウを閉じます。次に、[ OK] をクリックします。
- 次に、[ カラー フィット パラメーター ] をクリックして、蛍光減衰パラメーターで生成される色である Green の初期蛍光寿命を調整します。 [Prompt] と [Delay] の値を調整するには、[ From ] と [To ] の値を変更します。次に、[ 戻る]をクリックしてポップアップウィンドウを閉じます。次に、[ OK] をクリックします。
- 「保存」をクリックして ASCII ファイルを処理し、選択したフォルダに保存します。次に、ChiSurf22、Photon Distribution Analysis55,56、またはBurst Variance Analysis57,58を使用して、時間分解蛍光異方性崩壊のデータを処理します。
注:ChiSurf、PDA、およびBVAの使用方法に関する例示的なデータと段階的な説明は、それぞれ github.com/Fluorescence-Tools/chisurf、github.com/Fluorescence-Tools/tttrlib、www.mpc.hhu.de/en/software/mfd-fcs-and-mfis、および github.com/SMB-Lab/feda_tools で入手できます。実験データはZenodo(10.5281 / zenodo.13371503)で入手できます。
5. FoxP1タンパク質調製
- 組換えFoxP1細菌の過剰発現
- クイックチェンジ部位特異的ポリメラーゼ連鎖反応(PCR)突然変異誘発後に 大腸菌 C41細菌細胞への形質転換を行います。
- LB培地とオートクレーブを準備して滅菌します。
- 5 μL(100 μg/mL)アンピシリンを含むLB培地5 mLに添加して、単一の 大腸菌 C41コロニーを事前に接種します。インキュベーターシェーカーで37°Cで一晩インキュベートします。
- 翌日、500mLのLB培地に一晩のプレ接種物を1:500の比率で事前に添加した抗生物質を加えて、大規模な細菌培養物に接種します。
- 600 nmでの培養物の吸光度を測定することにより、培養物の成長をモニターします。
- 光学密度が ~0.5-0.7 に達したら、最終濃度の 0.5 mM イソプロピル-β-d-1-チオガラクトピラノシド (IPTG) を添加してタンパク質発現を誘導し、培養物をインキュベーターシェーカーで一晩 15 °C に保ちます。
- 1.4〜1.6の範囲の光学密度(600 nm)を達成した後、3000 g で4°Cで20分間遠心分離することにより、細菌細胞を回収します。 上清を捨て、使用するまでペレットを-20°Cで保管します。
- 組換えFoxP1精製
- 大 腸菌 C41細胞を、超音波処理、液体均質化、フレンチプレスなどの任意の溶解方法を使用して溶解バッファーを添加することにより溶解します。
- ライセートを14000 g で4°Cで10分間遠心分離します。
- His6タグ付きタンパク質については、Ni2+-NTAアフィニティーカラムを洗浄し、高速タンパク質液体クロマトグラフィー(FPLC)を使用してNiSO4 と平衡化します。
- His6タグ付きFoxP1タンパク質を平衡化したNi2+-NTAアフィニティーカラムにロードします。
- Ni2+-NTAカラムからFoxP1タンパク質を溶出するには、溶出バッファーの線形グラジエントを使用します。
- タンパク質が溶出されたら、バッファー交換のための透析を行います。イミダゾールを含まない平衡化バッファーにタンパク質を添加し、マグネチックスターラーを使用して4°Cで一晩透析します。
注:タンパク質が別の方法を使用して精製されている場合は、それに応じてバッファー交換を行います。 - His6タグを除去するには、タバコエッチングウイルス(TEV)プロテアーゼ(TEV:FoxP1の1:100の比率)を透析に一晩加えます。
注:彼の6タグ消化は、オンカラムTEVプロテアーゼ消化を追加することでも実行できます。このステップにより、目的のタンパク質の収量が向上します。 - 翌日、5.2.3 から 5.2.5 の手順を繰り返します。ただし、今回は、溶出バッファーではなく洗浄バッファーでタンパク質を溶出します。
- PBSバッファーを添加してタンパク質を適切な量に濃縮し、タンパク質の吸光係数を考慮して280 nmの吸光度を使用してタンパク質を定量します。
- 組換えFoxP1ラベリング
- 50-100 μM FoxP1タンパク質を、ジチオスレイトール(DTT)またはトリス(2-カルボキシエチル)ホスフィン(TCEP)のいずれかが10倍モル過剰に含んでいる状態で、最終容量500 μLの室温で30分間インキュベートします。
- PD 10 脱塩カラムを使用してバッファー交換を行います。PD10 カラムをカラムアダプターを使用して 50 mL 遠心チューブに入れます。
- 5 mLのPBSバッファーを加えてPD10カラムを平衡化し、1000 × g で2分間遠心分離します。この手順を 3 回繰り返します。
- PD10 カラムを新しい 50 mL チューブに保管し、2 mL の PBS バッファーをカラムに加えます。次に、ステップ1のFoxP1タンパク質500μLをカラムに加えます。1000 × g を2分間遠心分離してタンパク質を溶出し、タンパク質を回収します。
- タンパク質を超遠心フィルター(10 kDa MWCO)に添加して濃縮し、7500 × g で10分間遠心分離し、溶出液を回収します。
- タンパク質濃度を測定し、タンパク質中のシステイン濃度が30%相当のBODIPYを添加し、4°Cで2時間ローテーターに置きます。
- 緩衝液交換を行い、ステップ5.3.2〜5.3.5で説明したようにタンパク質を濃縮します。
- 分光光度計またはBradford法やLowry法などの比色アッセイを使用してタンパク質濃度を確認し、500nmの吸光度で色素の濃度を確認します。
- 次の式を使用して、ラベル付けの程度を測定します。
(A染料/ε染料)/(タンパク質の分子量/ミリグラム/ミリリットルあたり)=染料のモル/タンパク質のモル
ここで、ADye およびεDye は、最大吸光度波長における染料の吸光度値およびモル吸光係数であり、それぞれ59である。280 nmで測定した場合、タンパク質の実際の吸光度は次の式で補正する必要があります。
Aタンパク質= A 280-(A染料・CF280)
ここで、CF280 は、280 nm59 での固有吸光度を考慮した、特定の色素の補正係数です。
注:システイン修飾タンパク質の発現と精製に焦点を当てていますが、システイン残基のタンパク質への戦略的な導入には慎重な検討が必要です。
6. 顕微鏡サンプルチャンバーの準備
- 495 μLの超純水と5 μL(0.01% v/v最終濃度)のTween-20(非イオン性界面活性剤)をチャンバースライドのウェルに加え、混合します。RTで30分以上インキュベートします。
- チャンバーを超純水で2回やさしく洗い、乾燥させます。
注:チャンバーは実験に使用する準備ができています。
7. 1分子蛍光異方性実験
- まず、smFA装置を校正します。ラマン散乱光を測定するか、寿命が短く適切な発光スペクトルを持つ色素(エリスロシンやマラカイトグリーンなど)に飽和ヨウ化カリウムKI溶液でさらに消光された色素の蛍光を測定することにより、装置の応答関数を決定します。
- バックグラウンド蛍光を補正するには、バッファー溶液の測定を行います。高速回転する色素からデータを取得することにより、平行検出器と垂直検出器(Gファクター)の相対感度を較正します。ここでは、2nMローダミン110を用いて検出器60を較正した。
- モノマーFoxP1のsmFA実験では、100 pM BODIPY標識FoxP1および100 nM非標識FoxP1を、チャンバースライド中の標準バッファー500 μL(20 mM HEPES、20 mM NaCl、pH 7.8)に加えます。
- DNAおよび単量体FoxP1を用いた滴定アッセイでは、400 nM DNAを100 pM BODIPY標識/100 nM非標識タンパク質混合物に加え、室温で10分間インキュベートします。
- 二量体FoxP1のsmFA実験では、標準バッファー中の100 pMのBODIPY標識FoxP1に500 nMの非標識FoxP1を添加します。次に、37°Cで30分間インキュベートします。
- DNAと二量体FoxP1を用いた滴定アッセイでは、2000 nM DNAを100 pM BODIPY標識タンパク質/500 nM非標識タンパク質ミックスに加え、室温で10分間インキュベートします。
- 測定を開始し、BIFLソフトウェアで30秒レコードのバースト量が60〜90の間であることを確認します。サンプルがより濃縮されている場合は、その値に達するまで希釈します。このステップにより、単一分子条件が保証されます。
- DNAの非存在または存在下で、単量体のFoxP1または二量体のFoxP1のいずれかの各測定を少なくとも4時間開始します。
- セクション 4 で説明したように、smFA 測定値を分析します。
結果
蛍光異方性は、蛍光色素の吸収と発光双極子モーメントの相対的な配向から生じます。蛍光色素が偏光にさらされると、入射光の電場ベクトルに整列した吸収遷移モーメントを持つ蛍光色素が優先的に励起されます(光選択)。その結果、励起状態の集団は部分的に配向するようになり、励起された分子のかなりの割合が、偏光励起光61の電場ベクトルと整列した遷移モーメントを有する。蛍光色素は、ブラウン運動により回転します。したがって、発光遷移モーメントも回転し、蛍光異方性に時間依存性になります。この効果は、蛍光分子の回転運動の測定、結合イベントの検出、蛍光色素の環境の特性評価、分子動力学の捕捉に使用できます。
単一分子実験は、サンプルの不均一性を決定するための独自の態勢を整えています。単一分子の感度と蛍光異方性を利用することで、マルチパラメータ蛍光分光法に別の次元が追加されます。一般的な単一分子共焦点顕微鏡(図2)20,21では、蛍光異方性は、パルスレーザーを使用する場合、強度ベースまたは時間分解によって決定できます。
共焦点顕微鏡62における高開口対物レンズの脱分極効果を考慮するために、時間分解異方性35,63の適切な形態は、次式で与えられる。
 (1)
(1)
ここで、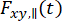 および
および は、波長Xでの励起後のy番目の検出チャネルにおける時間分解蛍光強度であり、平行および垂直偏光L1およびL2についてであり、これらの測定で使用される高い開口数(NA)対物レンズによる平行信号と垂直信号との間の混合を説明する因子である35,62、64.色素の平行、
は、波長Xでの励起後のy番目の検出チャネルにおける時間分解蛍光強度であり、平行および垂直偏光L1およびL2についてであり、これらの測定で使用される高い開口数(NA)対物レンズによる平行信号と垂直信号との間の混合を説明する因子である35,62、64.色素の平行、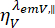 、および垂直の検出チャネル(
、および垂直の検出チャネル(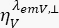 )の検出効率の違いは、検出効率の比率
)の検出効率の違いは、検出効率の比率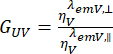 で補正されます。GUV は G ファクターとも呼ばれます。
で補正されます。GUV は G ファクターとも呼ばれます。
時間分解蛍光異方性は、多指数関数的崩壊を使用してモデル化でき、蛍光色素がより大きな生体分子に結合していることを説明できます。
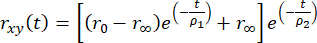 , (2)
, (2)
ここで、r0 は蛍光色素依存の基本異方性 (通常は r0 = 0.38)、 r∞ は残留異方性、ρ1 と ρ2 はそれぞれ速い回転相関時間 (蛍光色素分子の局所的な運動) と遅い (高分子の全体的な運動) 回転相関時間です。
単一分子異方性測定(図2)では、バースト積分蛍光寿命(BIFL)分析33,35を使用して個々のエミッターを識別するために、光子到着時間が記録される。光子間の到着時間(Δt)は、移動平均を使用して平滑化され、視覚化を支援するためにプロットされます。これらの時間のヒストグラムには、背景から発生する光子の平均と標準偏差を決定するために、半ガウス分布が適合されます。標準偏差の倍数に設定された任意のしきい値を使用して、各バーストの最初と最後の光子を識別しながら、個々のイベントを除外します。次いで、各バースト内の光子は、式1および2を使用して、または最尤推定器35を介して、時間分解および強度ベースの定常状態蛍光異方性を計算することを含む、さらなる分析のために統合される。単一分子イベントにおける光子の数が限られているため、最尤推定量は単一の指数成分のみを考慮し、これ以上は説明しません。
単一分子イベントの 2 次元ヒストグラムでは、平均蛍光寿命 (τ) と異方性 (rxy) をペリンの式29,61 で関連付けて、平均回転時間 (ρ) を求めることができます。
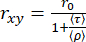 (3)
(3)
特定のρ 値は、異なるバーストの光子が結合された時間分解蛍光異方性崩壊に統合される「サブアンサンブル」(se)解析によって、より高い確実性で取得できます。これは、式2のパラメータを実験的減衰(seTRFA)に最適化することで解析できます。時間分解異方性は、ns時間枠内で発生する蛍光の放出内の生体分子の回転運動(局所的および全体的)に関連する不均一性とダイナミクスを解くことができます。
単一分子イベント内のダイナミクス(サブミリ秒スケール)を検出するために、時間分解異方性バースト分散解析(traBVA)57を導入した。traBVAでは、Mi連続光子セグメントを含む光子バーストの場合、バーストの過剰異方性分散(s2)は次式で表されます
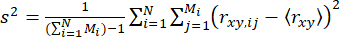 . (4)
. (4)
単一の異方性状態の場合、分散σ2 は、ショットノイズ65 (sn: √N、ここで N は光子の数)のみから生じます
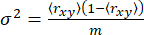 (5)
(5)
ここで、m はバースト内の光子の数です。したがって、異方性のさらなる分散を特定するために、コンフォメーションの不均一性による過剰な異方性分散(S2)を式4と式5の差として定義できます。
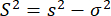 (6)
(6)
個々の分子の観察内で発生するダイナミクスを捉え、分散近似を考慮するために、動的異方性光分布解析(daPDA)55,56を使用することができます。daPDAでは、蛍光強度は、二項分布として表される条件付き確率(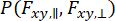 )に従ってモデル化されます。
)に従ってモデル化されます。
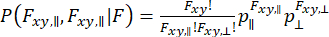 (7)
(7)
一緒に、ポアソン分布に従うバックグラウンドカウント率の推定値
 (8)
(8)
ここで、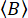 は、設定された時間枠あたりの背景フォトンの平均数です。平行および垂直のバックグラウンドカウント(
は、設定された時間枠あたりの背景フォトンの平均数です。平行および垂直のバックグラウンドカウント(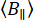 と
と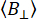 )は、バッファサンプルを基準として使用して測定できます。実験的に決定された蛍光異方性は、速度論的変化を含む偏光チャネルあたりの蛍光強度分布で性能指数χ2を最小化することによって最適化されます。
)は、バッファサンプルを基準として使用して測定できます。実験的に決定された蛍光異方性は、速度論的変化を含む偏光チャネルあたりの蛍光強度分布で性能指数χ2を最小化することによって最適化されます。
提供される分析ルーチンとデータ表現は、収集されたデータを解釈するための包括的なアプローチを提供します。このプロトコルは、ナノ秒からミリ秒までの異方性変化を捉えることに限定される共焦点測定に主に焦点を当てているが、より長い時間スケールで蛍光異方性を監視するために全反射顕微鏡を採用することが可能であり、時系列分析66を可能にする。単一分子共焦点測定では、観察されたアンサンブルのユニークなフィンガープリントを作成する多次元ヒストグラムの使用を強調します。選択した集団から再構築された時間分解蛍光減衰は、ナノ秒スケールで蛍光異方性の進化を追跡できます(図3)。光子分布解析55,56およびバースト分散解析(BVA)57,58も、時間分解崩壊と多次元ヒストグラムとの間の中間時間スケールでのダイナミクスを捉えることができる。このプロトコルは、ナノ秒からミリ秒のタイムスケールをブリッジすることができるパルス励起67,68の有無にかかわらず、偏光蛍光相関分光法(FCS)の使用をカバーしていないが、同じデータをFCS69を計算するために使用することができるが、これは提示されたプロトコルの範囲外である。このような実験を行う場合は、サンプル測定時間を長くすることをお勧めします。
このアプローチは、ヒトのFoxPタンパク質のような複雑なシステムに適用されており、その作用機序に関与する運動に関する貴重な洞察を提供しています。FoxPタンパク質は、脳や肺の発達など、いくつかの生理学的側面に関与する転写因子です。重要なことに、異なる突然変異がこれらのタンパク質の機能を損なうと認識されている70,71。FoxP1のDNA結合ドメインをモデルとして、異なる単一システイン変異体を作製し、運動のトラッカーとしてBODIPY-FL色素を導入しました(図4A)。実際、このタンパク質の主要な構造調節因子としての二量体化とDNA結合の影響を評価しました。smFAアプローチを使用して、2D-smFAプロットを生成し、単量体および二量体条件の各変異体に対してtraBVAおよびdaPDAを作成しました。研究された単一の変異体の1つの例を示します(図4)。異方性挙動は、高回転相関時間と低回転相関時間を決定する点ですべての変異体で類似しており、したがって、推定、無秩序、および折り畳まれたアンサンブルを決定します。それでも、各アンサンブルのフラクションと動力学の点ですべての変異体で非常に不均一であり、二量体化とDNA結合によって影響を受ける異なる秩序から無秩序への移行変化を証明し、鎖に沿った構造ダイナミクスの高解像度での記述を示しています(図5)。
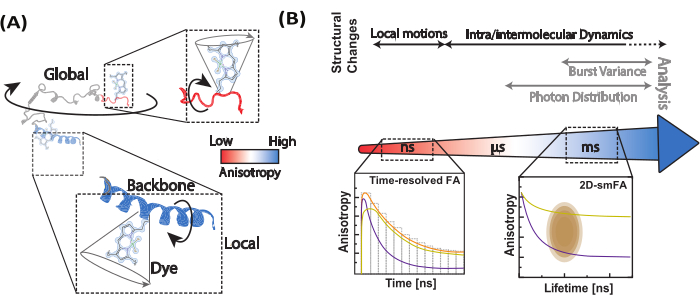
図1:生体分子のダイナミックレンジと蛍光異方性法。 (A)目的の生体分子の骨格に沿ったさまざまな位置につながれた小さな蛍光色素の異方性は、局所的な構造ダイナミクスを知らせます。(B)蛍光強度減衰(時間分解蛍光異方性、FA)および共焦点単一分子顕微鏡データの単一分子ヒストグラムによってプローブされた時間スケール。 この図の拡大版を表示するには、ここをクリックしてください。
図2:1分子蛍光異方性データの登録と処理。 (A)自由に拡散する分子を、直線偏光レーザー(我々の場合は青色)を1本搭載した共焦点1分子顕微鏡で解析します。蛍光発光(この場合は緑色)は、ビーム偏光子が信号を2つの偏光(平行、  、励起源に対して垂直
、励起源に対して垂直  )に分割した後、2つの検出器によって検出されます。(B)検出された各光子は、マイクロ時間、マクロ時間、チャネルタイプの3つのパラメータによって特徴付けられます。データは、Time-Tagged Time-Resolved(TTTR)フォーマット72で格納される。(C)個々の分子のバーストを選択し、処理して、観察された各分子の蛍光異方性を含む蛍光パラメータを抽出します。(D)データは、蛍光異方性対蛍光寿命の2次元プロットや時間分解異方性減衰など、複数の方法で表されます。これらの表現により、蛍光寿命、回転相関時間、およびシステムの不均一性を視覚的および定量的に決定できます。 この図の拡大版を表示するには、ここをクリックしてください。
)に分割した後、2つの検出器によって検出されます。(B)検出された各光子は、マイクロ時間、マクロ時間、チャネルタイプの3つのパラメータによって特徴付けられます。データは、Time-Tagged Time-Resolved(TTTR)フォーマット72で格納される。(C)個々の分子のバーストを選択し、処理して、観察された各分子の蛍光異方性を含む蛍光パラメータを抽出します。(D)データは、蛍光異方性対蛍光寿命の2次元プロットや時間分解異方性減衰など、複数の方法で表されます。これらの表現により、蛍光寿命、回転相関時間、およびシステムの不均一性を視覚的および定量的に決定できます。 この図の拡大版を表示するには、ここをクリックしてください。
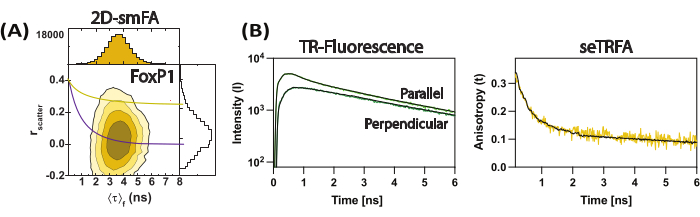
図3:FoxP1ドメインスワップダイマーの代表データ。 (A)蛍光異方性(r散乱)と分子あたりの平均蛍光寿命との相関を等高線プロットとして。分子のアンサンブル平均の代表として、2つの回転成分に対する1つのペリン方程式を重ね合わせたもので、ρ1 と ρ2 がそれぞれ 0.2 ns と 8.5 ns であることを考慮したものです。(B)サブアンサンブル時間分解蛍光減衰を使用して、サンプルの時間分解蛍光異方性を計算します。式2と適合すると、蛍光異方性の局所成分と全体成分が分離されました。 この図の拡大版を表示するには、ここをクリックしてください。
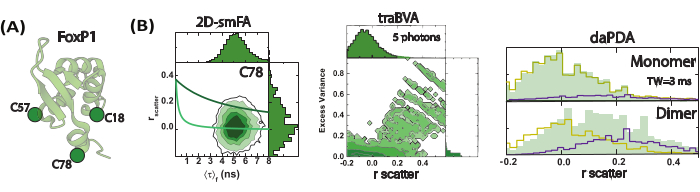
図4:単一分子蛍光異方性(smFA)を使用してモニターされたサブミリ秒のFoxP1ダイナミクス。 (A)単量体のFoxP1構造の漫画表現。(B)2次元ヒストグラムは、動的不均一性を示し、時間分解蛍光異方性によって特定された2つの異なる回転相関時間を明らかにします。時間分解異方性バースト分散解析(traBVA)は、大きな異方性を示す過剰分散(式6)を持つイベントの小さなサブセットを明らかにします。Photon Distribution Analysis(PDA)を用いた定量的動的異方性解析により、このプロセスの為替レートをさらに抽出します。 この図の拡大版を表示するには、ここをクリックしてください。
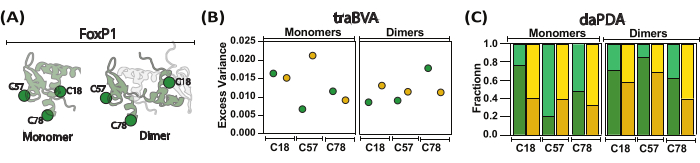
図5:二量体化中のFoxP1の局所的および全球的な動きのスクリーニング (A)漫画の表現は、単量体のFoxP1とその二量体形態を比較します。(B)単量体および二量体条件下での位置ごとの平均過剰分散が示されており、過剰分散が大きいほど異方性の変化が有意であることを示しています。(C)光子分布解析(PDA)を用いた動的異方性解析は、DNAの不在(緑)と存在(黄)における集団分率(暗い色で高い異方性、明るい色で低い異方性)を決定するのに役立ちます。このアプローチでは、ローカルな行動とグローバルな行動の間の遷移について、示されていない率を推定し、FoxP1が部分的な展開を遂げていることを明らかにしました。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
単一分子蛍光異方性実験では、選択した蛍光色素の光物理的特性を慎重に検討することが重要です。これらの特性には、検出システムと一致する必要がある発光波長と、利用可能なパルスレーザーと互換性がある必要がある励起波長が含まれます。ダイナミックレンジを最適化するには、蛍光色素分子の蛍光寿命が分子の回転拡散時間に対して長い必要があります。これは、回転ダイナミクスと、目的の生体分子に対する蛍光色素の双極子の結合/配向を追跡するために重要です。さらに、輝度、光安定性、および量子収率は、安定した信号対雑音比で強力な信号を生成するために不可欠です。これらの理由から、BODIPY-FLはいくつかの研究39,40,42で蛍光色素として選択されています。
生体分子の骨格ダイナミクスをスクリーニングするには、多くの場合、タンパク質の標識が必要であり、通常は部位特異的な標識によって達成されます。これは通常、標的化学修飾のための残留物を導入することによって行われます。最も一般的なアプローチは、目的の位置にシステインを導入し、そのチオール側鎖をマレイミドやヨードアセアミドなどの試薬で選択的に修飾することです。あまり一般的ではありませんが、ハロゲン化ベンジルとブロモメチルケトンはチオエーテル結合を形成するために使用されます。他のアミノ酸側鎖も標的とすることができますが、タンパク質に豊富に存在することはあまり一般的ではありません。しかし、非天然アミノ酸のような代替的なアプローチも使用することができる73。研究対象の生体分子との干渉を最小限に抑えるためには、標識のための適切な部位選択が重要であり、適切な制御を実施する必要があります。例えば、標識分子を結合アッセイに使用する場合、補完的な標識フリー法により、蛍光色素が結合親和性に影響を与えないことを確認すべきです。
適切なサンプルを特定し、最適な標識戦略を実施した後、次のステップは、共焦点顕微鏡が単一分子実験用に適切に位置合わせされ、キャリブレーションされていることを確認することです。このプロトコルでは、さらなる分析に必要な因子を決定する方法について説明します。装置の校正が完了したら、次のステップはサンプルを測定し、データを処理して、検出された光子からできるだけ多くの情報を抽出することです。 図2に示すように、マイクロタイム、マクロタイム、チャンネルタイプなどの主要なパラメータは、一般的なTCSPCエレクトロニクスを使用してさらに解析および視覚化するために使用できます。
近年の単一分子蛍光分光法の進歩は、生体分子の不均一なアンサンブルからの構造情報の研究に広く利用することができます。しかし、蛍光異方性によって得られる知見を活用している研究は比較的少なく、生体分子の構造ダイナミクスを導き出すためには完全なタンパク質モデルが必要です。そのため、いくつかの転写因子のドメイン間相互作用やタンパク質間相互作用のダイナミクスを解明することは困難です。
結論として、単一分子蛍光異方性実験は、その機能を理解するために重要な生体分子骨格の局所的および全体的な運動に関する補完的な情報を提供します。
開示事項
すべての著者は、この記事の内容と競合する金銭的利益がないことを宣言します。
謝辞
この研究は、FONDECYT GRANTS 11200729 と FONDEQUIP EQM200202 to E.M.、NIH R15CA280699 R01GM151334、および NSF CAREER MCB 1749778 awards to HS の支援を受けました。NKは、クレムソン大学のポスドクフェローシッププログラムからの支援を認めました。
資料
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
参考文献
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. Principles of Protein X-Ray Crystallography. , Springer, Verlag. New York, NY. (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701(2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. aM. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , Elsevier. Cambridge, MA. (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. aM. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676(2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854(2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724(2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. Fluorescence Correlation Spectroscopy: Theory and Applications. , Springer. Berlin, Heidelberg. (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375(2020).
- Takahashi, H., Takahashi, K., Liu, F. -C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , Springer. New York, NY. (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302(2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623(2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116(2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. Principles of Fluorescence Spectroscopy. , Springer. New York, NY. (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1(2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077(2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004(2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. Time tagged time-resolved fluorescence data collection in life sciences. , https://www.picoquant.com/images/uploads/page/files/14528/technote_tttr.pdf (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved