Method Article
Anisotropia de fluorescência resolvida no tempo de moléculas únicas para caracterizar a flexibilidade local em biomoléculas
Neste Artigo
Resumo
Aqui, apresentamos o protocolo para estudar a flexibilidade local e a dinâmica de biomoléculas usando anisotropia de fluorescência resolvida no tempo no nível de molécula única no modo de microscopia confocal.
Resumo
Descrevemos um protocolo para conduzir anisotropia de fluorescência resolvida no tempo no nível de molécula única usando microscopia confocal para investigar a flexibilidade local e a dinâmica do domínio forkhead (FKH) de ligação ao ácido desoxirribonucléico (DNA) do fator de transcrição FoxP1. FoxP1 dimeriza através de um mecanismo de troca de domínio tridimensional (3D-DS), formando um intermediário desordenado com ou sem DNA. Como o 3D-DS envolve uma região intrinsecamente desordenada, entender seu comportamento é crucial para elucidar as propriedades estruturais e funcionais do FoxP1. Usando um FoxP1 marcado com cisteína única, conduzimos experimentos de anisotropia de fluorescência de molécula única (smFA), aplicando a Análise de Distribuição de Fótons de anisotropia dinâmica (daPDA) e a Análise de Variância de Explosão de anisotropia resolvida no tempo (traBVA) para sondar a flexibilidade e a dinâmica local. Este protocolo fornece um guia detalhado e passo a passo para medições de smFA, enfatizando análises resolvidas no tempo, variância e técnicas de distribuição de probabilidade para capturar a dinâmica estrutural em diferentes escalas de tempo. Essa abordagem nos permitiu relacionar a dinâmica e a heterogeneidade à dimerização de FoxP1 e à ligação ao DNA, destacando o complexo mecanismo de ação que caracteriza esse fator de transcrição.
Introdução
A atividade funcional das biomoléculas depende de sua flexibilidade molecular e dinâmica estrutural 1,2,3. Naturalmente, as biomoléculas experimentam flutuações térmicas constantes, variando de movimentos rápidos a mudanças conformacionais de longo prazo que influenciam sua função (Figura 1)4. Em biomoléculas, os movimentos locais da espinha dorsal contribuem para movimentos globais em larga escala, incluindo dobradiça em enzimas e mudanças conformacionais significativas nas proteínas motoras. Métodos de determinação de estrutura, como ressonância magnética nuclear (RMN)5, cristalografia de raios-X6 e microscopia eletrônica criogênica (crio-EM)7, revelaram múltiplas conformações em várias biomoléculas. No entanto, conectar as flutuações locais à grande dinâmica conformacional das biomoléculas e seu papel na função são praticamente inexplorados. Relacionar dinâmica e estrutura pode ser desafiador, especialmente para proteínas intrinsecamente desordenadas (IDPs)8,9,10. Ao contrário das proteínas estruturadas, as IDPs não mantêm uma estrutura terciária estável. Em vez disso, eles sofrem extensas mudanças conformacionais com níveis de energia livre semelhantes, permitindo uma ampla gama de atividades biológicas11,12.
Várias abordagens experimentais têm sido empregadas para investigar a dinâmica conformacional de proteínas, sondando sua flexibilidade molecular 1,13,14,15,16. Entre eles, a RMN se destaca por sua capacidade de fornecer resolução em nível atômico em várias escalas de tempo, de dezenas de picossegundos a várias horas12. No entanto, determinar a flexibilidade macromolecular permanece um desafio devido aos altos graus de liberdade e para proteínas de grande porte; assim, a RMN é frequentemente limitada ao estudo de biomoléculas de cerca de 100 kDa17.
Dada a complexidade estrutural de proteínas altamente dinâmicas como IDPs, avanços metodológicos adicionais foram desenvolvidos para explorar o espaço conformacional local e de longo alcance para entender sua função11. A espectroscopia de fluorescência multiparâmetro de molécula única (smMFS) 18 , 19 , 20 , 21 , 22 oferece informações abrangentes sobre biomoléculas, fornecendo informações cruciais sobre sua função, dinâmica conformacional, estados de ligação e estequiometria. No entanto, interpretar a grande quantidade de dados estruturais obtidos de biomoléculas é um desafio, e fatores como dinâmica molecular, comportamento do fluoróforo e o comportamento complexo das moléculas podem complicar ainda mais a análise dos dados 23,24,25,26,27,28.
Empregamos anisotropia de fluorescência de molécula única (smFA) como um método robusto para avaliar a dinâmica local e global ao longo da espinha dorsal das biomoléculas (Figura 1A). A anisotropia de fluorescência, descrita pela primeira vez por Perrin29 e introduzida por Weber30 , 31 como uma ferramenta bioanalítica32 , foi posteriormente adaptada para estudos de molécula única com o advento de técnicas de fluorescência resolvidas no tempo e o incremento da sensibilidade dos detectores33 , 34 , 35 , 36 , 37. O smFA abrange uma ampla gama de escalas de tempo - de picossegundos a várias horas - e complementa os dados obtidos de experimentos de transferência de energia de ressonância de Förster (smFRET) de molécula única38.
O smFA pode ser visualizado em vários formatos para extrair informações críticas sobre a dinâmica biomolecular (Figura 1B). Os decaimentos da anisotropia de fluorescência resolvidos no tempo são histogramas unidimensionais que capturam a dinâmica na escala de tempo de picossegundos a nanossegundos39,40. Histogramas bidimensionais de molécula única, que correlacionam o tempo de vida da fluorescência com a anisotropia para moléculas individuais, podem revelar a heterogeneidade do estado de anisotropia e fornecer insights visuais sobre a dinâmica potencial dentro do tempo de observação em experimentos confocais (~ ms) 41 , 42 . Para estudar a dinâmica de submilissegundos, a análise de distribuição de fótons de anisotropia dinâmica (daPDA) pode ser usada, enquanto a Análise de Variância de Explosão de anisotropia resolvida no tempo (traBVA) oferece um método robusto para confirmar dinâmicas específicas em torno de milissegundos43 (Figura 1B).
Esses métodos complementam ferramentas mais tradicionais, como a espectroscopia de correlação de fluorescência resolvida por polarização (pFCS), que possui um espectro mais amplo 44,45,46,47. No geral, várias ferramentas de análise de dados para smFA facilitam a identificação de mudanças conformacionais locais e globais, desde que a calibração adequada seja considerada.
Aqui, aplicamos smFA para estudar a ligação ao DNA do fator de transcrição FoxP1 humano 48,49,50,51. Esta proteína adota dímero trocado de domínio devido à natureza intrinsecamente desordenada de sua cadeia polipeptídica, que é notavelmente afetada dependendo do estado quaternário da proteína e da presença de DNA. Geramos diferentes mutantes de cisteína única para marcar com BODIPY-FL, realizamos experimentos de smFA e empregamos daPDA e trBVAa. Essa abordagem nos permitiu relacionar a dinâmica e a heterogeneidade à dimerização de FoxP1 e à ligação ao DNA, destacando o complexo mecanismo de ação que caracteriza esse fator de transcrição.
Protocolo
NOTA: Selecionar o fluoróforo adequado é essencial para experimentos de smFA. As biomoléculas podem ser marcadas em posições específicas do local, modificando aminoácidos em proteínas ou bases de nucleotídeos em ácidos nucléicos com marcadores fluorescentes, dependendo dos grupos reativos disponíveis. Entre os corantes orgânicos52, as famílias Alexa Fluor, Cy, BODIPY e Janelia Farms são as escolhas mais populares para smFA, graças à sua longa vida útil de fluorescência, fotoestabilidade e altos rendimentos quânticos. O BODIPY-FL é frequentemente favorecido por sua vida útil prolongada de fluorescência, rendimento quântico superior e ligante de conexão curto. Além disso, fluoróforos alternativos são comumente usados na triagem de drogas, onde as técnicas em massa são preferidas53. As proteínas fluorescentes quiméricas também podem ser usadas para experimentos e imagens de anisotropia de células vivas, embora haja uma limitação de uma faixa dinâmica mais baixa.
1. Preparação do tampão
NOTA: Use luvas, óculos de proteção para os olhos e um jaleco de laboratório ao fazer experimentos de laboratório.
- Tampão padrão (20 mM de ácido 4- (2-hidroxietil)-1-piperazineetanossulfônico [HEPES], pH 7,8, 150 mM de NaCl): Dissolva 2,38 g de HEPES, 4,38 g de NaCl em 400 mL de água ultrapura, ajuste o pH para 7,8 e faça o volume final para 500 mL.
- Tampão de lise (HEPES 20 mM, pH 7,8, NaCl 150 mM, fluoreto de fenilmetilsulfonil 0,1 mM [PMSF], 10 μg / mL DNAse): Dissolva 2,38 g de HEPES, 4,38 g de NaCl e 8,71 mg de PMSF e uma concentração final de 10 μg / mL DNAse em 400 mL de água ultrapura, ajuste o pH para 7,8 e faça o volume final para 500 mL.
- Tampão de equilíbrio (50 mM de solução salina tamponada com fosfato [PBS], 150 mM de NaCl e 10 mM de Imidazol; pH 7,4): Dissolver 5,05 g de Na2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl e 0,34 g de Imidazol em 400 mL de água ultrapura, ajustar o pH para 7,4 e fazer o volume final para 500 mL.
- Tampão de lavagem (50 mM PBS, 150 mM NaCl e 30 mM Imidazol; pH 7,4): Dissolva 5,05 g de Na2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl e 1,02 g de Imidazol em 400 mL de água ultrapura, ajuste o pH para 7,4 e faça o volume final para 500 mL.
- Tampão de eluição (50 mM PBS, 150 mM NaCl e 250 mM Imidazol; pH 7,4): Dissolver 5,05 g de Na2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl e 8,5 g de Imidazol em 400 mL de água ultrapura, ajustar o pH para 7,4 e fazer o volume final para 500 mL.
- Tampão PBS (solução salina tamponada com fosfato de sódio 50 mM, NaCl 150 mM, pH 7,4). Dissolva 5,05 g de Na2HPO4, 0,85 g de NaH2PO4 e 4,38 g de NaCl em 400 mL de água ultrapura, ajuste o pH para 7,4 e faça o volume final para 500 mL.
- Esterilize todos os tampões filtrando a solução usando filtros de tamanho de poro de 0,22 μm e armazene os tampões à temperatura ambiente (RT).
- Filtre o tampão padrão usando um filtro de carvão para aquisição de molécula única.
2. Sondas fluorescentes
- BODIPY-FL: Dissolva o frasco de 5 mg contendo BODIPY-FL adicionando um volume final de 1,29 mL de dimetilsulfóxido (DMSO) recém-aberto para obter a concentração final de 10 mM BODIPY-FL.
- Rodamina 110: Dissolva 3,67 mg de Rodamina 110 adicionando um volume final de 1 mL de DMSO recém-aberto para obter a concentração final de 10 mM de Rodamina 110.
NOTA: As sondas fluorescentes precisam ser evitadas quando expostas à luz. Sempre use um tubo sensível à luz (cor âmbar) e envolva-o com papel alumínio. Se o DMSO já estiver aberto, ele deve ser mantido no dessecador para aumentar sua vida útil. - Preparar alíquotas de pequeno volume da sonda fluorescente preparada e conservá-las a -20 °C até nova utilização.
NOTA: Evite ciclos de congelamento e descongelamento para melhorar sua eficiência de rotulagem.
3. Medições de calibração
- Realize experimentos de detecção de fluorescência multiparâmetro (MFD) em uma configuração resolvida por polarização caseira com 4 canais de detecção e 2 lasers pulsados (azul 485 nm e vermelho 640 nm) 54 . No entanto, para medições de anisotropia, use o laser azul e dois canais detectores.
- Ligue os canais do detector (paralelo e perpendicular) e a potência do laser azul.
- Certifique-se de que a potência do laser esteja definida para 60 μW e abra o painel de controle do software de contagem de fótons únicos correlacionada ao tempo (TCSPC)36 .
- Misture 1 μL de 100 nM de Rodamina 110 e 49 μL de água destilada para fazer uma concentração final de 2 nM de Rodamina 110. Adicione 50 μL de rodamina 110 2 nM ao centro da lamínula para medições de calibração.
- Em seguida, adicione uma gota de líquido de imersão (óleo ou água, mas tomando cuidado para compartilhar o mesmo índice de refração) em cima da lente objetiva do microscópio para aumentar o poder de resolução do microscópio.
- Coloque a lamínula em cima da lente objetiva e certifique-se de que a gota de água esteja no centro da objetiva do microscópio.
- Ajuste o plano da imagem para ficar dentro da solução e acima da superfície do vidro.
- Ajuste o botão para encontrar o segundo ponto focal brilhante e gire para um ponto e meio. Focalizar o laser na interface do vidro e do líquido.
- Maximize o número de fótons detectados ajustando a posição do orifício (70 μm) enquanto monitora a taxa de contagem de fótons.
- Abra o software no painel do modo TTTR ou T3 (time-tagged time-resolved (TTTR) e clique no botão Iniciar . Registre a taxa de contagem por 120 s e salve o formato de arquivo Rhodamine 110.ptu usando o software de aquisição. Esse tempo de aquisição deve ser suficiente considerando a concentração (2 nM).
- Para medições de fundo, adicione 50 μL de água destilada ao centro da lamínula e repita os passos 3.5-3.10. No entanto, registre a taxa de contagem de fótons por 300 s e salve o arquivo water.ptu .
- Para outras medições de fundo, adicione 50 μL de tampão padrão ao centro da lamínula e repita as etapas 3.5 a 3.10. Registre a taxa de contagem de fótons por 300 s e salve o arquivo buffer.ptu padrão . Em seguida, analise os dados usando o software de análise de tempo de vida de fluorescência de integração de ruptura (BIFL).
4. Calibração e análise de dados
- Abra o software BIFL e clique em confirmar a configuração na janela automática. Em seguida, clique em obter parâmetros do arquivo e, em seguida, em OK.
- Para que a medição seja analisada, clique na matriz de caminho de dados e selecione-a .
- Em seguida, carregue o arquivo water.ptu de medição de água para matrizes como dispersão verde. Da mesma forma, selecione o arquivo buffer.ptu padrão para Plano de fundo verde. Para Verde grosso, selecione Rodamina 110.ptu.
- Nos parâmetros de seleção de molécula única, clique em Avançar e, em seguida, em Ajustar para exibir uma nova janela pop-up. Clique em limite para alterar o tempo de chegada entre fótons e selecione o tempo de evento de molécula única para o tempo médio de chegada entre fótons (dt). Em seguida, clique em min. # para selecionar o número mínimo de fótons por evento de molécula única e, em seguida, feche a janela pop-up clicando em Return. Em seguida, clique em OK.
- Em seguida, clique em Parâmetros de ajuste de cor para ajustar o tempo de vida inicial da fluorescência para Verde, cores geradas com parâmetros de decaimento de fluorescência. Ajuste os valores de Prompt e Atraso modificando os valores de De e Para. Em seguida, feche a janela pop-up clicando em Retornar. Em seguida, clique em OK.
- Clique em Salvar para processar os arquivos ASCII e salvá-los em uma pasta selecionada. Em seguida, processe os dados para o decaimento da anisotropia de fluorescência resolvido no tempo usando ChiSurf22, Análise de Distribuição de Fótons 55,56 ou Análise de Variância de Explosão57,58.
NOTA: Dados exemplares e descrições passo a passo sobre como usar ChiSurf, PDA e BVA estão disponíveis em github.com/Fluorescence-Tools/chisurf, github.com/Fluorescence-Tools/tttrlib, www.mpc.hhu.de/en/software/mfd-fcs-and-mfis e github.com/SMB-Lab/feda_tools, respectivamente. Os dados experimentais estão disponíveis em Zenodo (10.5281 / zenodo.13371503).
5. Preparação da proteína FoxP1
- Superexpressão bacteriana recombinante de FoxP1
- Realize a transformação em células bacterianas de E. coli C41 após mutagênese de reação em cadeia da polimerase (PCR) dirigida ao local de mudança rápida.
- Prepare o meio LB e a autoclave para esterilizá-lo.
- Pré-inocular uma única colônia de E. coli C41 adicionando 5 mL de meio LB contendo 5 μL (100 μg / mL) de ampicilina. Deixe incubar a 37 °C durante a noite em um agitador de incubadora.
- No dia seguinte, inocule a cultura bacteriana em larga escala adicionando pré-inóculo durante a noite em 500 mL de meio LB com antibiótico pré-adicionado na proporção de 1:500.
- Monitore o crescimento da cultura medindo a absorbância da cultura a 600 nm.
- Quando a densidade óptica atingir entre ~ 0,5-0,7, induza a expressão da proteína adicionando uma concentração final de 0,5 mM de isopropil-β-d-1-tiogalactopiranosídeo (IPTG) e mantenha a cultura a 15 ° C em um agitador de incubadora durante a noite.
- Depois de atingir uma densidade óptica (600 nm) na faixa de 1,4-1,6, colher as células bacterianas por centrifugação a 3000 g por 20 min a 4 ° C. Rejeitar o sobrenadante e conservar o pellet a -20 °C até à utilização.
- Purificação recombinante de FoxP1
- Lise as células C41 de E. coli adicionando tampão de lise usando qualquer método de lise, como sonicação, homogeneização líquida, prensa francesa, etc.
- Centrifugar o lisado a 14000 g durante 10 min a 4 °C.
- Para proteínas marcadas com His6, lave a coluna de afinidade Ni2+-NTA e equilibre com NiSO4 usando cromatografia líquida de proteína rápida (FPLC).
- Carregue a proteína FoxP1 marcada com His6 na coluna de afinidade Ni2+-NTA equilibrada.
- Eluir a proteína FoxP1 da coluna Ni2+-NTA usando um gradiente linear do tampão de eluição.
- Depois que a proteína for eluída, faça diálise para troca de tampão. Adicione a proteína a um tampão de equilíbrio sem imidazol e dialise durante a noite usando um agitador magnético a 4 ° C.
NOTA: Se a proteína for purificada usando um método diferente, faça a troca de tampão de acordo. - Para remover a etiqueta His6, adicione a protease do vírus da corrosão do tabaco (TEV) (proporção de 1:100 de TEV:FoxP1) à diálise durante a noite.
NOTA: Sua digestão de6 tags também pode ser realizada adicionando digestão de protease TEV na coluna. Esta etapa melhoraria o rendimento da proteína de interesse. - No dia seguinte, repita as etapas de 5.2.3-5.2.5. No entanto, desta vez, elui a proteína no tampão de lavagem em vez do tampão de eluição.
- Concentrar a proteína num volume adequado adicionando tampão PBS e quantificar a proteína utilizando a absorvância a 280 nm, tendo em conta o coeficiente de extinção da proteína.
- Marcação FoxP1 recombinante
- Incubar 50-100 μM de proteína FoxP1 com um excesso molar de 10 vezes de ditiotreitol (DTT) ou tris (2-carboxietil) fosfina (TCEP) com um volume final de 500 μL em RT por 30 min.
- Execute a troca de buffer usando colunas de dessalinização PD 10. Coloque a coluna PD10 em um tubo de centrífuga de 50 mL usando um adaptador de coluna.
- Equilibre a coluna PD10 adicionando 5 mL de tampão PBS e centrifugue a 1000 × g por 2 min. Repita esta etapa 3 vezes.
- Mantenha a coluna PD10 em um tubo novo de 50 mL e adicione 2 mL de tampão PBS na coluna. Em seguida, adicione 500 μL de proteína FoxP1 da etapa 1 na coluna. Eluir a proteína centrifugando 1000 × g durante 2 min e recolher a proteína.
- Concentrar a proteína adicionando-a em filtros ultracentrífugos (10 kDa MWCO), centrifugar a 7500 × g durante 10 min e recolher a eluição.
- Meça a concentração de proteína, adicione BODIPY com 30% equivalente à concentração de cisteína na proteína e coloque-a em um rotador por 2 h a 4 ° C.
- Realize a troca de tampão e concentre a proteína conforme discutido nas etapas 5.3.2-5.3.5.
- Verificar a concentração de proteínas por meio de um espectrofotómetro ou de um ensaio colorimétrico, como o método de Bradford ou Lowry, e a concentração do corante por absorvância a 500 nm.
- Meça o grau de rotulagem usando a seguinte fórmula:
(Um corante/ε corante)/(peso molecular da proteína/miligrama de proteína por mililitro) = moles de corante/moles de proteína
Onde ADye e εDye são o valor de absorbância do corante e o coeficiente de extinção molar do corante no comprimento de onda máximo de absorbância, respectivamente59. Quando medida a 280 nm, a absorvância real da proteína deve ser corrigida pela seguinte fórmula:
Umaproteína = A280-(ADye· CF280)
Em que CF280 é o fator de correção para o corante especificado, tendo em conta a sua absorvância intrínseca a 280 nm59.
NOTA: Enquanto nos concentramos na expressão e purificação de uma proteína modificada por cisteína, deve-se considerar cuidadosamente a introdução estratégica de resíduos de cisteína na proteína.
6. Preparação da câmara de amostra do microscópio
- Adicione 495 μL de água ultrapura e 5 μL (concentração final de 0,01% v / v) de Tween-20 (surfactante não iônico) ao poço de uma lâmina de câmara e misture. Incube por mais de 30 min em RT.
- Lave a câmara suavemente com água ultrapura duas vezes e seque-a.
NOTA: A câmara está pronta para uso no experimento.
7. Experimento de anisotropia de fluorescência de molécula única
- Primeiro, calibre o instrumento smFA. Determine a função de resposta do instrumento, medindo a luz espalhada Raman ou medindo a fluorescência de um corante com vida útil curta e espectro de emissão apropriado (por exemplo, eritrosina ou verde de malaquita) adicionalmente extinto por uma solução saturada de iodeto de potássio, KI.
- Para corrigir a fluorescência de fundo, faça medições de uma solução tampão. Calibre a sensibilidade relativa dos detectores paralelos e perpendiculares (fator G) adquirindo dados de um corante de rotação rápida. Aqui, 2 nM Rodamina 110 foi usado para calibrar os detectores60.
- Para experimentos de smFA em FoxP1 monomérico, adicione 100 pM de FoxP1 marcado com BODIPY e 100 nM de FoxP1 não marcado a 500 μL de tampão padrão (20 mM HEPES, 20 mM NaCl, pH 7,8) em uma lâmina de câmara.
- Para o ensaio de titulação com DNA e FoxP1 monomérico, adicione 400 nM de DNA a uma mistura de proteínas 100 pM marcada com BODIPY / 100nM não marcada e incube em RT por 10 min.
- Para experimentos de smFA em FoxP1 dimérico, adicione 500 nM de FoxP1 não marcado a um FoxP1 de 100 pM de BODIPY no buffer padrão. Em seguida, incubar a 37 °C durante 30 min.
- Para o ensaio de titulação com ADN e FoxP1 dimérico, adicionar 2000 nM de ADN a uma mistura de proteínas 100 pM marcada com BODIPY/500 nM não marcada e incubar a RT durante 10 min.
- Inicie as medições e verifique no software BIFL se a quantidade de rajadas em um registro de 30 s está entre 60-90. Se a amostra estiver mais concentrada, dilua-a até atingir esse valor. Esta etapa garante condições de molécula única.
- Iniciar o smFA de cada medição para FoxP1 monomérico ou FoxP1 dimérico na ausência ou presença de ADN durante, pelo menos, 4 h.
- Analise as medições de smFA conforme discutido na seção 4.
Resultados
A anisotropia de fluorescência surge da orientação relativa da absorção do fluoróforo e dos momentos de dipolo de emissão. Quando os fluoróforos são expostos à luz polarizada, os fluoróforos com momentos de transição de absorção alinhados com o vetor do campo elétrico da luz incidente são preferencialmente excitados (fotosseleção). Consequentemente, a população do estado excitado torna-se parcialmente orientada, com uma fração significativa das moléculas excitadas tendo seus momentos de transição alinhados com o vetor do campo elétrico da luz excitante polarizada61. Os fluoróforos giram devido ao seu movimento browniano. Assim, o momento de transição de emissão também gira, resultando em dependência de tempo na anisotropia de fluorescência. Esse efeito pode ser usado para medir os movimentos rotacionais de moléculas fluorescentes, detectar eventos de ligação, caracterizar o ambiente do fluoróforo e capturar a dinâmica molecular.
Experimentos de molécula única são posicionados de forma única para determinar a heterogeneidade da amostra. Aproveitar a sensibilidade de uma única molécula e a anisotropia de fluorescência adiciona outra dimensionalidade à espectroscopia de fluorescência multiparâmetro. Em um microscópio confocal típico de molécula única (Figura 2) 20 , 21 , a anisotropia de fluorescência pode ser determinada por meio de intensidade ou resolvida no tempo quando lasers pulsados são usados.
Para considerar os efeitos despolarizantes da objetiva de alta abertura numérica em um microscópio confocal62, a forma adequada da anisotropia resolvida no tempo35,63 é dada por
 (1)
(1)
onde 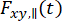 e
e  são a intensidade de fluorescência resolvida no tempo no canal de detecção y-ésimo após excitação no comprimento de onda x, para a polarização paralela e perpendicular l1 e l2 e são fatores que descrevem a mistura entre os sinais paralelos e perpendiculares devido à alta abertura numérica (NA) objetiva usada nessas medições35,62, 64. As diferenças nas eficiências de detecção do canal de detecção paralelo,
são a intensidade de fluorescência resolvida no tempo no canal de detecção y-ésimo após excitação no comprimento de onda x, para a polarização paralela e perpendicular l1 e l2 e são fatores que descrevem a mistura entre os sinais paralelos e perpendiculares devido à alta abertura numérica (NA) objetiva usada nessas medições35,62, 64. As diferenças nas eficiências de detecção do canal de detecção paralelo, 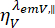 , e perpendicular,
, e perpendicular, 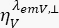 , para o corante são corrigidas com a razão das eficiências de detecção,
, para o corante são corrigidas com a razão das eficiências de detecção, 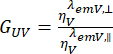 . O G UV também é conhecido como fator G.
. O G UV também é conhecido como fator G.
A anisotropia de fluorescência resolvida no tempo pode ser modelada usando um decaimento multiexponencial para explicar o fluoróforo sendo ligado a uma biomolécula maior como
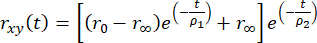 , (2)
, (2)
onde r0 é a anisotropia fundamental dependente do fluoróforo (tipicamente r0 = 0,38), r∞ é a anisotropia residual e ρ1 e ρ2 são tempos de correlação rotacional rápidos (movimentos locais do fluoróforo) e lentos (movimento global da macromolécula), respectivamente.
Nas medições de anisotropia de molécula única (Figura 2), os tempos de chegada dos fótons são registrados para identificar emissores individuais usando a análise de tempo de vida de fluorescência integrada à explosão (BIFL)33,35. Os tempos de chegada entre fótons (Δt) são suavizados usando uma média móvel e, em seguida, plotados para ajudar na visualização. O histograma desses tempos é equipado com um meio gaussiano para determinar a média e o desvio padrão dos fótons originados do fundo. Um limite arbitrário, definido em múltiplos do desvio padrão, é usado para filtrar eventos individuais enquanto identifica o primeiro e o último fóton em cada rajada. Os fótons dentro de cada explosão são então integrados para análise posterior, que inclui o cálculo da anisotropia de fluorescência em estado estacionário resolvida no tempo e baseada em intensidade usando as equações 1 e 2 ou por meio de um estimador de máxima verossimilhança35. Devido ao número limitado de fótons em eventos de molécula única, o estimador de máxima verossimilhança considera apenas um único componente exponencial e não será discutido mais adiante.
Em um histograma bidimensional de eventos de molécula única, o tempo médio de vida de fluorescência (τ) e a anisotropia (rxy) podem ser relacionados pela equação de Perrin29,61 para obter (ρ) como um tempo médio de rotação.
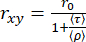 (3)
(3)
Valores específicos de ρ podem ser obtidos com maior certeza por análise de "subconjunto" (se), onde os fótons de diferentes rajadas são integrados em um decaimento combinado de anisotropia de fluorescência resolvido no tempo que pode ser analisado otimizando os parâmetros da equação 2 para o decaimento experimental (seTRFA). A anisotropia resolvida no tempo pode resolver a heterogeneidade e a dinâmica associadas aos movimentos rotacionais (locais e globais) das biomoléculas dentro da emissão de fluorescência que ocorre dentro do período de tempo ns.
Para detectar a dinâmica dentro de eventos de molécula única (na escala de submilissegundos), introduzimos a Análise de Variância de Explosão de anisotropia resolvida no tempo (traBVA) 57 . Em traBVA, para uma explosão de fótons contendo Mi segmentos de fótons consecutivos, a variância de anisotropia excessiva (s2) para rajadas é
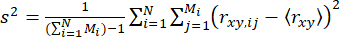 . (4)
. (4)
Para um único estado anisotrópico, a variância σ2 surge apenas do ruído de disparo65 (sn: √N, onde N é o número de fótons)
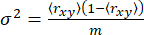 (5)
(5)
onde m é o número de fótons em uma explosão. Portanto, para identificar variância adicional na anisotropia, podemos definir o excesso de variância de anisotropia (S2) devido à heterogeneidade conformacional como a diferença entre as equações 4 e 5.
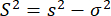 (6)
(6)
Para capturar a dinâmica que ocorre na observação de moléculas individuais e considerar a aproximação da variância, a Análise de Distribuição de Fótons de anisotropia dinâmica (daPDA) 55 , 56 pode ser usada . Em daPDA, a intensidade de fluorescência é modelada seguindo uma probabilidade condicional (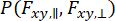 ) expressa como uma distribuição binomial.
) expressa como uma distribuição binomial.
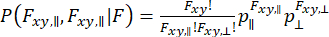 (7)
(7)
Juntos, com uma estimativa da taxa de contagem de antecedentes que segue uma distribuição de Poisson
 (8)
(8)
onde 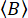 é o número médio de fótons de fundo por janela de tempo definida. As contagens de fundo paralelas e perpendiculares
é o número médio de fótons de fundo por janela de tempo definida. As contagens de fundo paralelas e perpendiculares 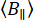 e
e 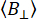 , podem ser medidas usando amostras de buffer como referência. A anisotropia de fluorescência determinada experimentalmente é otimizada minimizando uma figura de mérito χ2 com uma distribuição de intensidade de fluorescência por canal de polarização que pode incluir mudanças cinéticas.
, podem ser medidas usando amostras de buffer como referência. A anisotropia de fluorescência determinada experimentalmente é otimizada minimizando uma figura de mérito χ2 com uma distribuição de intensidade de fluorescência por canal de polarização que pode incluir mudanças cinéticas.
As rotinas de análise e representações de dados fornecidas oferecem uma abordagem abrangente para interpretar os dados coletados. Embora este protocolo se concentre principalmente em medições confocais, que são limitadas na captura de mudanças de anisotropia de nanossegundos a milissegundos, é possível adotar um microscópio de reflexão interna total para monitorar a anisotropia de fluorescência em escalas de tempo mais longas, permitindo a análise de séries temporais66. Para medições confocais de molécula única, destacamos o uso de histogramas multidimensionais que criam uma impressão digital única do conjunto observado. Os decaimentos de fluorescência resolvidos no tempo, reconstruídos a partir de populações selecionadas, podem rastrear a evolução da anisotropia de fluorescência na escala de nanossegundos (Figura 3). A análise de distribuição de fótons55,56 e a análise de variância de explosão (BVA)57,58 também podem capturar a dinâmica em escalas de tempo intermediárias entre decaimentos resolvidos no tempo e histogramas multidimensionais. Embora este protocolo não cubra o uso de espectroscopia de correlação de fluorescência de polarização (FCS), com ou sem excitação pulsada67,68, que pode fazer a ponte entre as escalas de tempo de nanossegundos a milissegundos, os mesmos dados podem ser usados para calcular o FCS69, embora isso esteja fora do escopo do protocolo apresentado. Se tais experimentos forem realizados, recomenda-se um tempo de medição de amostra mais longo.
Essa abordagem foi aplicada a um sistema complexo como as proteínas FoxP humanas, fornecendo informações valiosas sobre os movimentos envolvidos em seu mecanismo de ação. As proteínas FoxP são fatores de transcrição envolvidos em vários aspectos fisiológicos, como desenvolvimento cerebral e pulmonar; É importante ressaltar que diferentes mutações foram reconhecidas como prejudicando a função dessas proteínas 70,71. Usando o domínio de ligação ao DNA de FoxP1 como modelo, geramos diferentes mutantes de cisteína única para introduzir um corante BODIPY-FL como rastreador de movimentos ( Figura 4A ). De fato, avaliamos o efeito da dimerização e da ligação ao DNA como principais reguladores estruturais dessa proteína. Usando a abordagem smFA, geramos gráficos 2D-smFA e fizemos traBVA e daPDA para cada mutante em condições monoméricas e diméricas. Mostramos um exemplo de um dos mutantes individuais estudados (Figura 4). O comportamento da anisotropia é semelhante em todos os mutantes em termos de determinação de tempos de correlação rotacional altos e baixos e, portanto, conjuntos presumíveis, desordenados e dobrados. Ainda assim, também é altamente heterogêneo em todos os mutantes em termos de fração e cinética de cada conjunto, evidenciando diferentes mudanças de transição ordem-desordem influenciadas pela dimerização e pela ligação ao DNA, e mostra a descrição em alta resolução da dinâmica estrutural ao longo da cadeia (Figura 5).
Figura 1: Faixa dinâmica de biomoléculas e métodos de anisotropia de fluorescência. (A) A anisotropia de pequenos fluoróforos amarrados a várias posições ao longo da espinha dorsal da biomolécula de interesse informa a dinâmica estrutural local. (B) Escalas de tempo sondadas por decaimentos de intensidade de fluorescência (anisotropia de fluorescência resolvida no tempo, FA) e histogramas de molécula única de dados de microscópio confocal de molécula única. Clique aqui para ver uma versão maior desta figura.
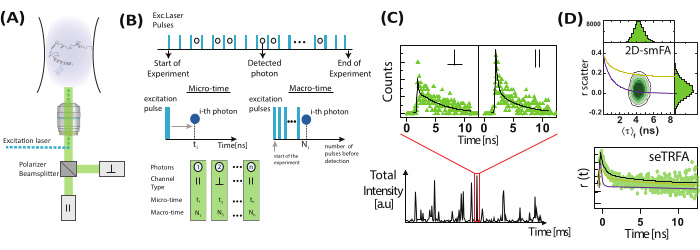
Figura 2: Registro e processamento de dados de anisotropia de fluorescência de molécula única. (A) As moléculas de difusão livre são analisadas usando um microscópio confocal de molécula única equipado com um único laser de excitação polarizado linearmente (azul em nosso caso). A emissão de fluorescência (verde no nosso caso) é detectada por dois detectores depois que um polarizador de feixe divide o sinal em duas polarizações (paralela  e perpendicular
e perpendicular  à fonte de excitação). (B) Cada fóton detectado é caracterizado por três parâmetros: tempo micro, tempo macro e tipo de canal. Os dados são armazenados em um formato TTTR (Time-Tagged Time-Resolved (TTTR)72. (C) Explosões de moléculas individuais são selecionadas e processadas para extrair parâmetros de fluorescência, incluindo anisotropia de fluorescência para cada molécula observada. (D) Os dados são representados de várias maneiras, incluindo gráficos bidimensionais de anisotropia de fluorescência versus tempo de vida de fluorescência e decaimentos de anisotropia resolvidos no tempo. Essas representações permitem a determinação visual e quantitativa do tempo de vida da fluorescência, tempos de correlação rotacional e heterogeneidade do sistema. Clique aqui para ver uma versão maior desta figura.
à fonte de excitação). (B) Cada fóton detectado é caracterizado por três parâmetros: tempo micro, tempo macro e tipo de canal. Os dados são armazenados em um formato TTTR (Time-Tagged Time-Resolved (TTTR)72. (C) Explosões de moléculas individuais são selecionadas e processadas para extrair parâmetros de fluorescência, incluindo anisotropia de fluorescência para cada molécula observada. (D) Os dados são representados de várias maneiras, incluindo gráficos bidimensionais de anisotropia de fluorescência versus tempo de vida de fluorescência e decaimentos de anisotropia resolvidos no tempo. Essas representações permitem a determinação visual e quantitativa do tempo de vida da fluorescência, tempos de correlação rotacional e heterogeneidade do sistema. Clique aqui para ver uma versão maior desta figura.
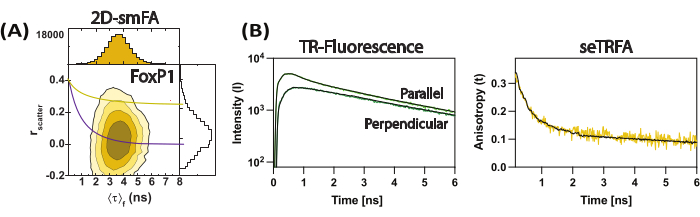
Figura 3: Dados representativos para dímero trocado de domínio FoxP1. (A) Correlação da anisotropia de fluorescência (dispersão r) com o tempo de vida médio de fluorescência por molécula como um gráfico de contorno. Sobreposição de uma única equação de Perrin para dois componentes rotacionais como representante da média do conjunto da molécula, considerando ρ1 e ρ2 de 0,2 ns e 8,5 ns, respectivamente. (B) Os decaimentos de fluorescência resolvidos no tempo do subconjunto são usados para calcular a anisotropia de fluorescência resolvida no tempo da amostra. O ajuste à equação 2 resolveu os componentes locais e globais da anisotropia de fluorescência. Clique aqui para ver uma versão maior desta figura.
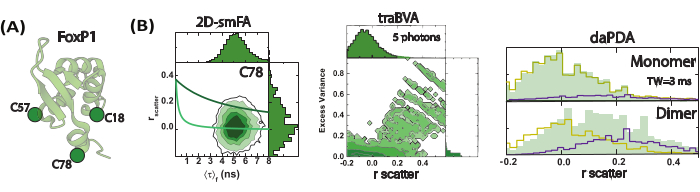
Figura 4: Dinâmica de FoxP1 em submilissegundos monitorada usando anisotropia de fluorescência de molécula única (smFA). (A) Uma representação de desenho animado da estrutura monomérica de FoxP1. (B) Um histograma bidimensional ilustra a heterogeneidade dinâmica, revelando dois tempos de correlação rotacional distintos identificados por meio da anisotropia de fluorescência resolvida no tempo. A Análise de Variância de Explosão de anisotropia resolvida no tempo (traBVA) revela um pequeno subconjunto de eventos com excesso de variância (Eq. 6) que exibem grande anisotropia. A análise quantitativa de anisotropia dinâmica usando a Análise de Distribuição de Fótons (PDA) extrai ainda mais as taxas de câmbio para este processo. Clique aqui para ver uma versão maior desta figura.
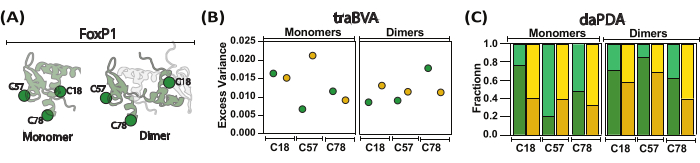
Figura 5: Triagem dos movimentos locais e globais de FoxP1 durante a dimerização. (A) Uma representação de desenho animado compara o FoxP1 monomérico com sua forma dimérica. (B) A variância média do excesso por local em condições monoméricas e diméricas é mostrada, com maior variância excessiva indicando mudanças mais significativas na anisotropia. (C) A análise dinâmica de anisotropia usando a Análise de Distribuição de Fótons (PDA) ajuda a determinar frações populacionais (alta anisotropia em cores escuras e baixa anisotropia em cores claras) na ausência (verde) e presença (amarelo) de DNA. Nessa abordagem, foram estimadas taxas (não mostradas) para transições entre comportamentos locais e globais, revelando que o FoxP1 sofre desdobramento parcial. Clique aqui para ver uma versão maior desta figura.
Discussão
Para experimentos de anisotropia de fluorescência de molécula única, é crucial considerar cuidadosamente as propriedades fotofísicas do fluoróforo escolhido. Essas propriedades incluem o comprimento de onda de emissão, que deve estar alinhado com o sistema de detecção, e o comprimento de onda de excitação, que deve ser compatível com os lasers pulsados disponíveis. Para otimizar a faixa dinâmica, o fluoróforo deve ter uma longa vida útil de fluorescência em relação ao tempo de difusão rotacional da molécula. Isso é fundamental para rastrear a dinâmica rotacional e a ligação/orientação do dipolo do fluoróforo em relação à biomolécula de interesse. Além disso, brilho, fotoestabilidade e rendimento quântico são essenciais para produzir sinais fortes com uma relação sinal-ruído estável. Por essas razões, o BODIPY-FL foi escolhido como fluoróforo em vários estudos 39,40,42.
A triagem da dinâmica da espinha dorsal das biomoléculas geralmente requer a rotulagem de proteínas, normalmente obtida por meio da rotulagem específica do local. Isso geralmente é feito introduzindo um resíduo para modificação química direcionada. A abordagem mais comum é a introdução de cisteínas em posições de interesse, onde suas cadeias laterais de tiol podem ser modificadas seletivamente com reagentes como maleimidas ou iodoacetamidas. Menos comumente, haletos benzílicos e bromometilcetonas são usados para formar ligações tioéter. Outras cadeias laterais de aminoácidos também podem ser direcionadas, mas sua abundância em proteínas é menos comumente usada. No entanto, abordagens alternativas, como aminoácidos não naturais, também podem ser usadas73. A seleção adequada do local para marcação é crucial para minimizar a interferência com a biomolécula em estudo, e os controles apropriados devem estar em vigor. Por exemplo, se a molécula marcada for usada em ensaios de ligação, métodos complementares sem marcação devem verificar se os fluoróforos não afetam a afinidade de ligação.
Depois de identificar a amostra apropriada e implementar a estratégia de rotulagem ideal, o próximo passo é garantir que o microscópio confocal esteja devidamente alinhado e calibrado para experimentos de molécula única. O protocolo descreve como determinar o fator necessário para análise posterior. Uma vez que o instrumento é calibrado, o próximo passo é medir a amostra e processar os dados para extrair o máximo de informações possível dos fótons detectados. Os parâmetros-chave, como micro-tempo, macro-tempo e tipo de canal, conforme mostrado na Figura 2, podem ser usados para análise e visualização adicionais usando a eletrônica típica do TCSPC.
Avanços recentes na espectroscopia de fluorescência de molécula única podem ser amplamente utilizados para estudar informações estruturais dos conjuntos heterogêneos de biomoléculas. No entanto, relativamente poucos estudos aproveitam os insights fornecidos pela anisotropia de fluorescência, e um modelo completo de proteína é necessário para derivar a dinâmica estrutural das biomoléculas. Portanto, desvendar a dinâmica das interações interdomínio e proteína-proteína de vários fatores de transcrição é um desafio.
Em conclusão, os experimentos de anisotropia de fluorescência de molécula única oferecem informações complementares sobre os movimentos locais e globais da estrutura biomolecular, que são críticas para a compreensão de sua função.
Divulgações
Todos os autores declaram não ter interesses financeiros conflitantes com o conteúdo deste artigo.
Agradecimentos
Este trabalho foi apoiado pelas bolsas FONDECYT 11200729 e FONDEQUIP EQM200202 para E.M., NIH R15CA280699 R01GM151334 e NSF CAREER MCB 1749778 prêmios para HS. NK reconheceu o apoio do programa de bolsas de pós-doutorado da Clemson University.
Materiais
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
Referências
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. Principles of Protein X-Ray Crystallography. , Springer, Verlag. New York, NY. (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701(2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. aM. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , Elsevier. Cambridge, MA. (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. aM. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676(2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854(2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724(2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. Fluorescence Correlation Spectroscopy: Theory and Applications. , Springer. Berlin, Heidelberg. (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375(2020).
- Takahashi, H., Takahashi, K., Liu, F. -C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , Springer. New York, NY. (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302(2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623(2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116(2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. Principles of Fluorescence Spectroscopy. , Springer. New York, NY. (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1(2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077(2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004(2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. Time tagged time-resolved fluorescence data collection in life sciences. , https://www.picoquant.com/images/uploads/page/files/14528/technote_tttr.pdf (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados