Method Article
Anisotropía de fluorescencia resuelta en el tiempo a partir de moléculas individuales para caracterizar la flexibilidad local en biomoléculas
En este artículo
Resumen
Aquí, presentamos el protocolo para estudiar la flexibilidad y dinámica local de biomoléculas utilizando anisotropía de fluorescencia resuelta en el tiempo a nivel de una sola molécula en modo de microscopía confocal.
Resumen
Describimos un protocolo para llevar a cabo la anisotropía de fluorescencia resuelta en el tiempo a nivel de una sola molécula utilizando microscopía confocal para investigar la flexibilidad local y la dinámica del dominio forkhead (FKH) de unión al ácido desoxirribonucleico (ADN) del factor de transcripción FoxP1. FoxP1 se dimeriza a través de un mecanismo tridimensional de intercambio de dominios (3D-DS), formando un intermediario desordenado con o sin ADN. Dado que 3D-DS involucra una región intrínsecamente desordenada, comprender su comportamiento es crucial para dilucidar las propiedades estructurales y funcionales de FoxP1. Utilizando un FoxP1 marcado con una sola cisteína, llevamos a cabo experimentos de anisotropía de fluorescencia de una sola molécula (smFA), aplicando enfoques de análisis de distribución de fotones de anisotropía dinámica (daPDA) y análisis de varianza de ráfaga de anisotropía resuelta en el tiempo (traBVA) para sondear la flexibilidad y la dinámica locales. Este protocolo proporciona una guía detallada paso a paso para las mediciones de smFA, haciendo hincapié en los análisis resueltos en el tiempo, la varianza y las técnicas de distribución de probabilidad para capturar la dinámica estructural en diferentes escalas de tiempo. Este enfoque nos permitió relacionar la dinámica y la heterogeneidad con la dimerización de FoxP1 y la unión al ADN, destacando el complejo mecanismo de acción que caracteriza a este factor de transcripción.
Introducción
La actividad funcional de las biomoléculas depende de su flexibilidad molecular y dinámica estructural 1,2,3. Naturalmente, las biomoléculas experimentan fluctuaciones térmicas constantes, que van desde movimientos rápidos hasta cambios conformacionales a largo plazo que influyen en su función (Figura 1)4. En las biomoléculas, los movimientos locales de la columna vertebral contribuyen a movimientos globales a mayor escala, incluida la flexión de la bisagra en las enzimas y cambios conformacionales significativos en las proteínas motoras. Los métodos de determinación de estructuras, como la resonancia magnética nuclear (RMN)5, la cristalografía de rayos X6 y la microscopía electrónica criogénica (crio-EM)7, han revelado múltiples conformaciones en varias biomoléculas. Sin embargo, la conexión de las fluctuaciones locales con la gran dinámica conformacional de las biomoléculas y su papel en la función está en su mayoría inexplorada. Relacionar la dinámica y la estructura puede ser un desafío, especialmente para las proteínas intrínsecamente desordenadas (IDP)8,9,10. A diferencia de las proteínas estructuradas, las IDP no mantienen una estructura terciaria estable. En cambio, experimentan extensos cambios conformacionales con niveles similares de energía libre, lo que permite una amplia gama de actividades biológicas11,12.
Se han empleado varios enfoques experimentales para investigar la dinámica conformacional de las proteínas mediante el sondeo de su flexibilidad molecular 1,13,14,15,16. Entre ellas, la RMN destaca por su capacidad para proporcionar una resolución a nivel atómico en varias escalas de tiempo, desde decenas de picosegundos hasta varias horas12. Sin embargo, la determinación de la flexibilidad macromolecular sigue siendo un desafío debido a los altos grados de libertad y para las proteínas de gran tamaño; por lo tanto, la RMN a menudo se limita al estudio de biomoléculas de alrededor de 100 kDa17.
Dada la complejidad estructural de proteínas altamente dinámicas como las IDP, se han desarrollado avances metodológicos adicionales para explorar el espacio conformacional local y de largo alcance para comprender su función11. La espectroscopia de fluorescencia multiparamétrica de una sola molécula (smMFS)18,19,20,21,22 ofrece una amplia información sobre las biomoléculas, proporcionando información crucial sobre su función, dinámica conformacional, estados de unión y estequiometría. Sin embargo, interpretar la gran cantidad de datos estructurales obtenidos de las biomoléculas es un desafío, y factores como la dinámica molecular, el comportamiento de los fluoróforos y el comportamiento complejo de las moléculas pueden complicar aún más el análisis de datos 23,24,25,26,27,28.
Empleamos la anisotropía de fluorescencia de una sola molécula (smFA) como un método robusto para evaluar la dinámica local y global a lo largo de la columna vertebral de las biomoléculas (Figura 1A). La anisotropía de fluorescencia, descrita por primera vez por Perrin29 e introducida por Weber30,31 como herramienta bioanalítica32, se adaptó posteriormente para estudios de moléculas individuales con el advenimiento de las técnicas de fluorescencia resueltas en el tiempo y el incremento de la sensibilidad de los detectores 33,34,35,36,37. smFA abarca una amplia gama de escalas de tiempo, desde picosegundos hasta varias horas, y complementa los datos obtenidos de los experimentos de transferencia de energía por resonancia de Förster (smFRET) de una sola molécula38.
La smFA se puede visualizar en varios formatos para extraer información crítica sobre la dinámica biomolecular (Figura 1B). Las desintegraciones de anisotropía de fluorescencia resueltas en el tiempo son histogramas unidimensionales que capturan la dinámica en la escala de tiempo de picosegundos a nanosegundos39,40. Los histogramas bidimensionales de una sola molécula, que correlacionan la vida útil de la fluorescencia con la anisotropía de moléculas individuales, pueden revelar la heterogeneidad del estado de anisotropía y proporcionar información visual sobre la dinámica potencial dentro del tiempo de observación en experimentos confocales (~ms)41,42. Para estudiar la dinámica de submilisegundos, se puede utilizar el análisis de distribución dinámica de fotones de anisotropía (daPDA), mientras que el análisis de varianza de ráfagas de anisotropía resuelto en el tiempo (traBVA) ofrece un método robusto para confirmar la dinámica específica alrededor de milisegundos43 (Figura 1B).
Estos métodos complementan herramientas más tradicionales, como la espectroscopia de correlación de fluorescencia resuelta por polarización (pFCS), que tiene un espectro más amplio 44,45,46,47. En general, las múltiples herramientas de análisis de datos para smFA facilitan la identificación de cambios conformacionales locales y globales, siempre que se considere una calibración adecuada.
Aquí, aplicamos smFA para estudiar la unión al ADN del factor de transcripción FoxP1 humano 48,49,50,51. Esta proteína adopta un dímero de intercambio de dominio debido a la naturaleza intrínsecamente desordenada de su cadena polipeptídica, que se ve notablemente afectada dependiendo del estado cuaternario de la proteína y la presencia de ADN. Generamos diferentes mutantes de una sola cisteína para marcarlos con BODIPY-FL, realizamos experimentos con smFA y empleamos daPDA y trBVAa. Este enfoque nos permitió relacionar la dinámica y la heterogeneidad con la dimerización de FoxP1 y la unión al ADN, destacando el complejo mecanismo de acción que caracteriza a este factor de transcripción.
Protocolo
NOTA: La selección del fluoróforo adecuado es esencial para los experimentos de smFA. Las biomoléculas se pueden marcar en posiciones específicas del sitio, ya sea modificando aminoácidos en proteínas o bases de nucleótidos en ácidos nucleicos con marcadores fluorescentes, dependiendo de los grupos reactivos disponibles. Entre los tintes orgánicos52, las familias Alexa Fluor, Cy, BODIPY y Janelia Farms son las opciones más populares para la smFA, gracias a su larga vida útil de fluorescencia, fotoestabilidad y altos rendimientos cuánticos. BODIPY-FL a menudo se ve favorecido por su vida útil de fluorescencia prolongada, rendimiento cuántico superior y enlace de conexión corto. Además, los fluoróforos alternativos se utilizan comúnmente en el cribado de fármacos, donde se prefieren las técnicas a granel53. Las proteínas fluorescentes quiméricas también se pueden utilizar para experimentos de anisotropía y obtención de imágenes de células vivas, aunque existe la limitación de un rango dinámico más bajo.
1. Preparación del tampón
NOTA: Use guantes, gafas de protección para los ojos y una bata de laboratorio mientras realiza experimentos de laboratorio.
- Tampón estándar (20 mM de ácido 4-(2-hidroxietil)-1-piperazineetanosulfónico [HEPES], pH 7,8, 150 mM de NaCl): Disuelva 2,38 g de HEPES, 4,38 g de NaCl en 400 mL de agua ultrapura, ajuste el pH a 7,8 y haga el volumen final a 500 mL.
- Tampón de lisis (20 mM de HEPES, pH 7,8, 150 mM de NaCl, 0,1 mM de fluoruro de fenilmetilsulfonilo [PMSF], 10 μg/mL de ADNasa): Disuelva 2,38 g de HEPES, 4,38 g de NaCl y 8,71 mg de PMSF y una concentración final de 10 μg/mL de ADNasa en 400 mL de agua ultrapura, ajuste el pH a 7,8 y haga el volumen final a 500 mL.
- Tampón de equilibrio (50 mM de solución salina tamponada con fosfato [PBS], 150 mM de NaCl y 10 mM de imidazol; pH 7,4): Disolver 5,05 g de Na2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl y 0,34 g de Imidazol en 400 mL de agua ultrapura, ajustar el pH a 7,4 y hacer el volumen final a 500 mL.
- Tampón de lavado (50 mM de PBS, 150 mM de NaCl y 30 mM de Imidazol; pH 7,4): Disolver 5,05 g deNa2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl y 1,02 g de Imidazol en 400 mL de agua ultrapura, ajustar el pH a 7,4 y hacer el volumen final a 500 mL.
- Tampón de elución (50 mM de PBS, 150 mM de NaCl y 250 mM de Imidazol; pH 7,4): Disolver 5,05 g deNa2HPO4, 0,85 g de NaH2PO4, 4,38 g de NaCl y 8,5 g de Imidazol en 400 mL de agua ultrapura, ajustar el pH a 7,4 y hacer el volumen final a 500 mL.
- Tampón PBS (50 mM de solución salina tamponada con fosfato de sodio, 150 mM de NaCl, pH 7,4). Disuelva 5,05 g de Na2HPO4, 0,85 g de NaH2PO4 y 4,38 g de NaCl en 400 ml de agua ultrapura, ajuste el pH a 7,4 y haga el volumen final a 500 mL.
- Esterilice todos los tampones filtrando la solución con filtros de tamaño de poro de 0,22 μm y almacene los tampones a temperatura ambiente (RT).
- Filtre el tampón estándar utilizando un filtro de carbón para la adquisición de una sola molécula.
2. Sondas fluorescentes
- BODIPY-FL: Disuelva el vial de 5 mg que contiene BODIPY-FL añadiendo un volumen final de 1,29 mL de dimetilsulfóxido (DMSO) recién abierto para obtener la concentración final de 10 mM DE BODIPY-FL.
- Rodamina 110: Disuelva 3,67 mg de Rhodamine 110 añadiendo un volumen final de 1 mL de DMSO recién abierto para obtener la concentración final de 10 mM de Rhodamine 110.
NOTA: Las sondas fluorescentes deben evitarse cuando se exponen a la luz. Utilice siempre un tubo sensible a la luz (color ámbar) y envuélvalo con papel de aluminio. Si el DMSO ya está abierto, debe mantenerse en el desecador para aumentar su vida útil. - Prepare alícuotas de pequeño volumen de la sonda fluorescente preparada y guárdela a -20 °C hasta su uso posterior.
NOTA: Evite los ciclos de congelación-descongelación para mejorar su eficiencia de etiquetado.
3. Mediciones de calibración
- Realice experimentos de detección de fluorescencia (MFD) multiparamétricos en una configuración de resolución de polarización construida en casa con 4 canales de detección y 2 láseres pulsados (azul 485 nm y rojo 640 nm)54. Sin embargo, para las mediciones de anisotropía, utilice el láser azul y dos canales detectores.
- Encienda los canales del detector (paralelo y perpendicular) y la potencia del láser azul.
- Asegúrese de que la potencia del láser esté configurada en 60 μW y abra el panel de control del software de conteo de fotones individuales correlacionado con el tiempo (TCSPC)36 .
- Mezclar 1 μL de 100 nM de Rhodamine 110 y 49 μL de agua destilada para obtener una concentración final de 2 nM de Rhodamine 110. Agregue 50 μL de 2 nM de Rhodamine 110 al centro del cubreobjetos para las mediciones de calibración.
- A continuación, agregue una gota de líquido de inmersión (ya sea aceite o agua, pero teniendo cuidado de compartir el mismo índice de refracción) sobre la lente del objetivo del microscopio para aumentar el poder de resolución del microscopio.
- Coloque la cubierta de vidrio encima de la lente del objetivo y asegúrese de que la gota de agua esté en el centro del objetivo del microscopio.
- Ajuste el plano de la imagen para que esté dentro de la solución y por encima de la superficie del vidrio.
- Ajuste la perilla para encontrar el segundo punto focal brillante y gírelo a un punto y medio. Enfoque el láser en la interfaz del vidrio y el líquido.
- Maximice el número de fotones detectados ajustando la posición del agujero de alfiler (70 μm) mientras monitorea la tasa de recuento de fotones.
- Abra el software en el panel de modo de resolución temporal con etiqueta de tiempo (TTTR o T3) y haga clic en el botón Inicio . Registre la tasa de conteo durante 120 s y guarde el formato de archivo Rhodamine 110.ptu utilizando el software de adquisición. Este tiempo de adquisición debería ser suficiente teniendo en cuenta la concentración (2 nM).
- Para las mediciones de fondo, agregue 50 μL de agua destilada al centro del cubreobjetos y repita los pasos 3.5-3.10. Sin embargo, registre la tasa de recuento de fotones durante 300 s y guarde el archivo water.ptu .
- Para otras mediciones de fondo, agregue 50 μL de tampón estándar al centro del cubreobjetos y repita los pasos 3.5-3.10. Registre la tasa de recuento de fotones durante 300 s y guarde el archivo buffer.ptu estándar . A continuación, analice los datos utilizando el software de análisis de la vida útil de la fluorescencia por integración de ráfagas (BIFL).
4. Calibración y análisis de datos
- Abra el software BIFL y haga clic en confirmar la configuración desde la ventana automática. A continuación, haga clic en obtener parámetros del archivo, luego en Aceptar.
- Para analizar la medición, haga clic en matriz de ruta de datos y selecciónela .
- A continuación, cargue el archivo water.ptu de medición de agua para matrices como Dispersión verde. Del mismo modo, seleccione el archivo buffer.ptu estándar para Fondo verde. En Verde espeso, seleccione Rhodamine 110.ptu.
- En los parámetros de selección de una sola molécula, haga clic en Siguiente y, a continuación, en Ajustar para mostrar una nueva ventana emergente. Haga clic en umbral para cambiar el tiempo de llegada entre fotones y seleccione el tiempo de evento de una sola molécula para el tiempo medio de llegada entre fotones (dt). A continuación, haga clic en min. # para seleccionar el número mínimo de fotones por evento de molécula individual, luego cierre la ventana emergente haciendo clic en Retorno. A continuación, haga clic en Aceptar.
- A continuación, haga clic en Parámetros de ajuste de color para ajustar la duración inicial de la fluorescencia para Verde, colores que se generan con parámetros de decaimiento de fluorescencia. Ajuste los valores de Solicitud y Retardo modificando los valores Desde y Hasta . A continuación, cierre la ventana emergente haciendo clic en Retorno. A continuación, haga clic en Aceptar.
- Haga clic en Guardar para procesar los archivos ASCII y guardarlos en una carpeta seleccionada. A continuación, procese los datos para la desintegración de la anisotropía de fluorescencia resuelta en el tiempo utilizando ChiSurf22, Análisis de distribución de fotones55,56 o Análisis de varianza de ráfaga57,58.
NOTA: Datos ejemplares y descripciones paso a paso sobre cómo usar ChiSurf, PDA y BVA están disponibles en github.com/Fluorescence-Tools/chisurf, github.com/Fluorescence-Tools/tttrlib, www.mpc.hhu.de/en/software/mfd-fcs-and-mfis y github.com/SMB-Lab/feda_tools, respectivamente. Los datos experimentales están disponibles en Zenodo (10.5281/zenodo.13371503).
5. Preparación de proteínas FoxP1
- Sobreexpresión bacteriana recombinante de FoxP1
- Realizar la transformación en células bacterianas C41 de E. coli después de la mutagénesis de la reacción en cadena de la polimerasa (PCR) dirigida al sitio de cambio rápido.
- Prepare el medio LB y el autoclave para esterilizarlo.
- Preinocular una sola colonia de E. coli C41 añadiéndola a 5 mL de medio LB que contenga 5 μL (100 μg/mL) de ampicilina. Deje incubar a 37 °C durante la noche en un agitador de incubadora.
- Al día siguiente, inocule el cultivo bacteriano a gran escala añadiendo pre-inóculo durante la noche en 500 mL de medio LB con antibiótico pre-añadido con una proporción de 1:500.
- Monitoree el crecimiento del cultivo midiendo la absorbancia del cultivo a 600 nm.
- Cuando la densidad óptica alcance entre ~0,5-0,7, induzca la expresión de la proteína añadiendo una concentración final de 0,5 mM de isopropil-β-d-1-tiogalactopiranósido (IPTG) y mantenga el cultivo a 15 °C en un agitador de incubadora durante la noche.
- Después de alcanzar una densidad óptica (600 nm) en el rango de 1,4-1,6, recoja las células bacterianas por centrifugación a 3000 g durante 20 min a 4 °C. Deseche el sobrenadante y almacene el pellet a -20 °C hasta su uso.
- Purificación recombinante de FoxP1
- Lisar las células de E. coli C41 añadiendo tampón de lisis utilizando cualquier método de lisis como la sonicación, la homogeneización de líquidos, la prensa francesa, etc.
- Centrifugar el lisado a 14000 g durante 10 min a 4 °C.
- En el caso de las proteínas marcadas con His6, lavar la columna de afinidad de Ni2+-NTA y equilibrarla con NiSO4 mediante cromatografía líquida rápida de proteínas (FPLC).
- Cargue la proteína FoxP1 marcada con His6 en la columna de afinidad Ni2+-NTA equilibrada.
- Eluir la proteína FoxP1 de la columna de Ni2+-NTA utilizando un gradiente lineal del tampón de elución.
- Después de eluir la proteína, realice la diálisis para el intercambio de tampones. Agregue proteínas a un tampón de equilibrio sin imidazol y dialice durante la noche usando un agitador magnético a 4 °C.
NOTA: Si la proteína se purifica utilizando un método diferente, realice el intercambio de tampón en consecuencia. - Para eliminar la etiquetaHis 6, agregue la proteasa del virus del grabado del tabaco (TEV) (proporción 1:100 de TEV:FoxP1) a la diálisis durante la noche.
NOTA: Su digestión de6 etiquetas también se puede realizar mediante la adición de digestión de proteasa TEV en columna. Este paso mejoraría el rendimiento de la proteína de interés. - Al día siguiente, repita los pasos de 5.2.3 a 5.2.5. Sin embargo, esta vez, eluya la proteína en el tampón de lavado en lugar del tampón de elución.
- Concentrar la proteína a un volumen adecuado añadiendo tampón PBS y cuantificar la proteína utilizando la absorbancia a 280 nm, considerando el coeficiente de extinción de la proteína.
- Etiquetado recombinante FoxP1
- Incubar 50-100 μM de proteína FoxP1 con un exceso molar de 10 veces de ditiotreitol (DTT) o tris (2-carboxietil)fosfina (TCEP) con un volumen final de 500 μL a RT durante 30 min.
- Realice el intercambio de búferes utilizando columnas de desalinización PD 10. Coloque la columna PD10 en un tubo de centrífuga de 50 ml utilizando un adaptador de columna.
- Equilibre la columna PD10 añadiendo 5 mL de tampón PBS y centrífuga a 1000 × g durante 2 min. Repita este paso 3 veces.
- Mantenga la columna PD10 en un tubo nuevo de 50 mL y agregue 2 mL de tampón PBS en la columna. A continuación, añada 500 μL de proteína FoxP1 del paso 1 en la columna. Eluir la proteína centrifugando 1000 × g durante 2 min y recoger la proteína.
- Concentrar la proteína añadiéndola en filtros ultracentrífugos (MWCO de 10 kDa), centrifugar a 7500 × g durante 10 min y recoger el eluido.
- Mida la concentración de proteína, agregue BODIPY con un 30% equivalente a la concentración de cisteína en la proteína y colóquelo en un rotador durante 2 h a 4 °C.
- Realice el intercambio de tampones y concentre la proteína como se describe en los pasos 5.3.2-5.3.5.
- Compruebe la concentración de proteínas mediante un espectrofotómetro o un ensayo colorimétrico, como el método de Bradford o Lowry, y la concentración del colorante por absorbancia a 500 nm.
- Mida el grado de etiquetado utilizando la siguiente fórmula:
(Un colorante/ε colorante)/(peso molecular de la proteína/miligramo de proteína por mililitro) = moles de tinte/moles de proteína
DondeUn colorante y εcolorante son el valor de absorbancia del colorante y el coeficiente de extinción molar del colorante a la longitud de onda de absorbancia máxima, respectivamente59. Cuando se mide a 280 nm, la absorbancia real de la proteína debe corregirse mediante la siguiente fórmula:
Unaproteína = A280-(Uncolorante· CF280)
Donde CF280 es el factor de corrección para el colorante especificado, considerando su absorbancia intrínseca a 280 nm59.
NOTA: Si bien nos centramos en la expresión y purificación de una proteína modificada con cisteína, se debe considerar cuidadosamente la introducción estratégica de residuos de cisteína en la proteína.
6. Preparación de la cámara de muestras del microscopio
- Agregue 495 μL de agua ultrapura y 5 μL (0.01% v/v concentración final) de Tween-20 (surfactante no iónico) al pocillo de una cámara y mezcle. Incubar más de 30 min en RT.
- Lave la cámara suavemente con agua ultrapura dos veces y séquela.
NOTA: La cámara está lista para ser utilizada para el experimento.
7. Experimento de anisotropía de fluorescencia de una sola molécula
- En primer lugar, calibrar el instrumento smFA. Determine la función de respuesta del instrumento, ya sea midiendo la luz dispersa Raman o midiendo la fluorescencia de un colorante con una vida útil corta y un espectro de emisión apropiado (por ejemplo, eritrosina o verde de malaquita) extinguido adicionalmente por una solución saturada de yoduro de potasio, KI.
- Para corregir la fluorescencia de fondo, realice mediciones de una solución tampón. Calibre la sensibilidad relativa de los detectores paralelos y perpendiculares (factor G) mediante la adquisición de datos de un tinte de rotación rápida. Aquí, se utilizó rodamina 110 de 2 nM para calibrar los detectores60.
- Para experimentos de smFA en FoxP1 monomérico, agregue 100 pM de FoxP1 marcado con BODIPY y 100 nM de FoxP1 sin marcar a 500 μL de tampón estándar (20 mM de HEPES, 20 mM de NaCl, pH 7,8) en un portaobjetos de cámara.
- Para el ensayo de valoración con ADN y FoxP1 monomérico, agregue 400 nM de ADN a 100 pM de mezcla de proteínas marcadas con BODIPY/100 nM sin marcar e incube en RT durante 10 min.
- Para experimentos smFA en FoxP1 dimérico, agregue 500 nM de FoxP1 sin marcar a un FoxP1 de 100 pM marcado con BODIPY en el búfer estándar. A continuación, incubar a 37 °C durante 30 min.
- Para el ensayo de valoración con ADN y FoxP1 dimérico, agregue 2000 nM de ADN a 100 pM de mezcla de proteínas marcadas con BODIPY/500 nM sin marcar e incube en RT durante 10 min.
- Inicie las mediciones y compruebe en el software BIFL que la cantidad de ráfagas en un registro de 30 s está entre 60 y 90. Si la muestra está más concentrada, diluirla hasta que alcance ese valor. Este paso asegura condiciones de una sola molécula.
- Comience cada medición de FoxP1 monomérico o FoxP1 dimérico en ausencia o presencia de ADN durante al menos 4 h.
- Analice las mediciones de smFA como se discutió en la sección 4.
Resultados
La anisotropía de fluorescencia surge de la orientación relativa de la absorción del fluoróforo y los momentos dipolares de emisión. Cuando los fluoróforos se exponen a luz polarizada, los fluoróforos con momentos de transición de absorción alineados con el vector de campo eléctrico de la luz incidente se excitan preferentemente (fotoselección). En consecuencia, la población de estado excitado se orienta parcialmente, con una fracción significativa de las moléculas excitadas que tienen sus momentos de transición alineados con el vector de campo eléctrico de la luz excitante polarizada61. Los fluoróforos rotan debido a su movimiento browniano. Por lo tanto, el momento de transición de la emisión también gira, lo que resulta en una dependencia del tiempo de la anisotropía de fluorescencia. Este efecto se puede utilizar para medir los movimientos de rotación de las moléculas fluorescentes, detectar eventos de unión, caracterizar el entorno del fluoróforo y capturar la dinámica molecular.
Los experimentos de una sola molécula están preparados de manera única para determinar la heterogeneidad de la muestra. Aprovechando la sensibilidad de una sola molécula y la anisotropía de fluorescencia, se añade otra dimensionalidad a la espectroscopia de fluorescencia multiparamétrica. En un microscopio confocal típico de una sola molécula (Figura 2)20,21, la anisotropía de fluorescencia se puede determinar mediante la intensidad o la resolución temporal cuando se utilizan láseres pulsados.
Para considerar los efectos despolarizantes del objetivo de apertura numérica alta en un microscopioconfocal 62, la forma adecuada de la anisotropía resuelta en el tiempo35,63 viene dada por
 (1)
(1)
donde 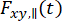 y
y  son la intensidad de fluorescencia resuelta en el tiempo en el canal de detección y-ésimo después de la excitación en la longitud de onda x, para la polarización paralela y perpendicular l1 y l2 y son factores que describen la mezcla entre las señales paralelas y perpendiculares debido al objetivo de alta apertura numérica (NA) utilizado en estas mediciones35,62, Artículo 64. Las diferencias en las eficiencias de detección del canal de detección paralelo,
son la intensidad de fluorescencia resuelta en el tiempo en el canal de detección y-ésimo después de la excitación en la longitud de onda x, para la polarización paralela y perpendicular l1 y l2 y son factores que describen la mezcla entre las señales paralelas y perpendiculares debido al objetivo de alta apertura numérica (NA) utilizado en estas mediciones35,62, Artículo 64. Las diferencias en las eficiencias de detección del canal de detección paralelo, 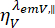 , y perpendicular,
, y perpendicular, 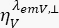 , para el colorante se corrigen con la relación de las eficiencias de detección,
, para el colorante se corrigen con la relación de las eficiencias de detección, 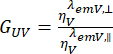 . El G UV también se conoce como factor G.
. El G UV también se conoce como factor G.
La anisotropía de fluorescencia resuelta en el tiempo se puede modelar utilizando una desintegración multiexponencial para explicar que el fluoróforo se une a una biomolécula más grande como
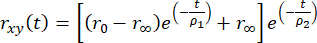 , (2)
, (2)
donde r0 es la anisotropía fundamental dependiente del fluoróforo (típicamente r0 = 0,38), r∞ es la anisotropía residual, y ρ1 y ρ2 son tiempos de correlación rotacional rápidos (movimientos locales del fluoróforo) y lentos (movimiento global de la macromolécula), respectivamente.
En las mediciones de anisotropía de una sola molécula (Figura 2), los tiempos de llegada de los fotones se registran para identificar emisores individuales mediante el análisis de vida útil de fluorescencia integrada en ráfagas (BIFL) 33,35. Los tiempos de llegada entre fotones (Δt) se suavizan utilizando un promedio móvil y luego se trazan para ayudar a la visualización. El histograma de estos tiempos está equipado con un medio gaussiano para determinar la media y la desviación estándar de los fotones que se originan en el fondo. Se utiliza un umbral arbitrario, establecido en múltiplos de la desviación estándar, para filtrar eventos individuales mientras se identifican el primer y el último fotón en cada ráfaga. A continuación, los fotones dentro de cada ráfaga se integran para su posterior análisis, que incluye el cálculo de la anisotropía de fluorescencia en estado estacionario resuelta en el tiempo y basada en la intensidad utilizando las ecuaciones 1 y 2 o mediante un estimador de máxima verosimilitud35. Debido al número limitado de fotones en eventos de una sola molécula, el estimador de máxima verosimilitud considera solo un solo componente exponencial y no se discutirá más a fondo.
En un histograma bidimensional de eventos de una sola molécula, el tiempo de vida medio de la fluorescencia (τ) y la anisotropía (rxy) se pueden relacionar mediante la ecuación de Perrin29,61 para obtener (ρ) como un tiempo de rotación promedio.
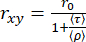 (3)
(3)
Los valores específicos de ρ se pueden obtener con mayor certeza mediante un análisis de "subconjunto" (se) donde los fotones de diferentes ráfagas se integran en una desintegración combinada de anisotropía de fluorescencia resuelta en el tiempo que se puede analizar optimizando los parámetros de la ecuación 2 para la desintegración experimental (seTRFA). La anisotropía resuelta en el tiempo puede resolver la heterogeneidad y la dinámica asociadas con los movimientos de rotación (locales y globales) de las biomoléculas dentro de la emisión de fluorescencia que ocurre dentro del marco de tiempo ns.
Para detectar la dinámica dentro de eventos de una sola molécula (en la escala de submilisegundos), introdujimos el análisis de varianza de ráfaga de anisotropía resuelto en el tiempo (traBVA)57. En traBVA, para una ráfaga de fotones que contiene Mi segmentos de fotones consecutivos, la varianza del exceso de anisotropía (s2) para las ráfagas es
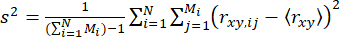 . (4)
. (4)
Para un solo estado anisotrópico, la varianza σ2 surge únicamente del ruido de disparo65 (sn: √N, donde N es el número de fotones)
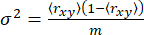 (5)
(5)
donde m es el número de fotones en una ráfaga. Por lo tanto, para identificar la varianza adicional en la anisotropía, podemos definir el exceso de varianza de anisotropía (S2) debido a la heterogeneidad conformacional como la diferencia entre las ecuaciones 4 y 5.
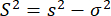 (6)
(6)
Para capturar la dinámica que ocurre dentro de la observación de moléculas individuales y considerar la aproximación de la varianza, se puede utilizar el Análisis de Distribución de Fotones de Anisotropía Dinámica (daPDA)55,56. En daPDA, la intensidad de fluorescencia se modela siguiendo una probabilidad condicional (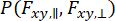 ) expresada como una distribución binomial.
) expresada como una distribución binomial.
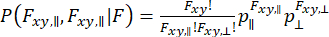 (7)
(7)
Junto con una estimación de la tasa de recuento de fondo que sigue una distribución de Poisson
 (8)
(8)
donde 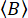 es el número promedio de fotones de fondo por ventana de tiempo establecida. Los recuentos de fondo paralelos y perpendiculares,
es el número promedio de fotones de fondo por ventana de tiempo establecida. Los recuentos de fondo paralelos y perpendiculares, 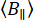 y
y 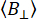 , se pueden medir utilizando muestras de tampón como referencia. La anisotropía de fluorescencia determinada experimentalmente se optimiza minimizando una figura de mérito χ2 con una distribución de intensidad de fluorescencia por canal de polarización que puede incluir cambios cinéticos.
, se pueden medir utilizando muestras de tampón como referencia. La anisotropía de fluorescencia determinada experimentalmente se optimiza minimizando una figura de mérito χ2 con una distribución de intensidad de fluorescencia por canal de polarización que puede incluir cambios cinéticos.
Las rutinas de análisis y las representaciones de datos proporcionadas ofrecen un enfoque integral para interpretar los datos recopilados. Aunque este protocolo se centra principalmente en las mediciones confocales, que están limitadas para capturar los cambios de anisotropía de nanosegundos a milisegundos, es posible adoptar un microscopio de reflexión interna total para monitorear la anisotropía de fluorescencia en escalas de tiempo más largas, lo que permite el análisis de series de tiempo66. Para las mediciones confocales de una sola molécula, destacamos el uso de histogramas multidimensionales que crean una huella digital única del conjunto observado. Las desintegraciones de fluorescencia resueltas en el tiempo, reconstruidas a partir de poblaciones seleccionadas, pueden rastrear la evolución de la anisotropía de fluorescencia a escala de nanosegundos (Figura 3). El análisis de distribución de fotones55,56 y el análisis de varianza de ráfagas (BVA)57,58 también pueden capturar la dinámica en escalas de tiempo intermedias entre decaciones resueltas en el tiempo e histogramas multidimensionales. Si bien este protocolo no cubre el uso de la espectroscopia de correlación de fluorescencia de polarización (FCS), con o sin excitación pulsada67,68, que puede unir las escalas de tiempo de nanosegundos a milisegundos, los mismos datos se pueden usar para calcular FCS69, aunque esto queda fuera del alcance del protocolo presentado. Si se llevan a cabo tales experimentos, se recomienda un tiempo de medición de la muestra más largo.
Este enfoque se ha aplicado a un sistema complejo como las proteínas FoxP humanas, proporcionando información valiosa sobre los movimientos involucrados en su mecanismo de acción. Las proteínas FoxP son factores de transcripción implicados en varios aspectos fisiológicos como el desarrollo cerebral y pulmonar; Es importante destacar que se han reconocido diferentes mutaciones que perjudican la función de estas proteínas70,71. Utilizando el dominio de unión al ADN de FoxP1 como modelo, generamos diferentes mutantes de cisteína única para introducir un colorante BODIPY-FL como rastreador de movimientos (Figura 4A). De hecho, evaluamos el efecto de la dimerización y la unión al ADN como principales reguladores estructurales de esta proteína. Utilizando el enfoque smFA, generamos gráficos 2D-smFA y creamos traBVA y daPDA para cada mutante en condiciones monoméricas y diméricas. Mostramos un ejemplo de uno de los mutantes individuales estudiados (Figura 4). El comportamiento de la anisotropía es similar en todos los mutantes en cuanto a la determinación de tiempos de correlación rotacional altos y bajos y, por lo tanto, conjuntos presumibles, desordenados y plegados. Sin embargo, también es muy heterogéneo en todos los mutantes en cuanto a la fracción y cinética de cada conjunto, evidenciando diferentes cambios de transición de orden a desorden influenciados por la dimerización y la unión al ADN, y muestra la descripción a alta resolución de la dinámica estructural a lo largo de la cadena (Figura 5).
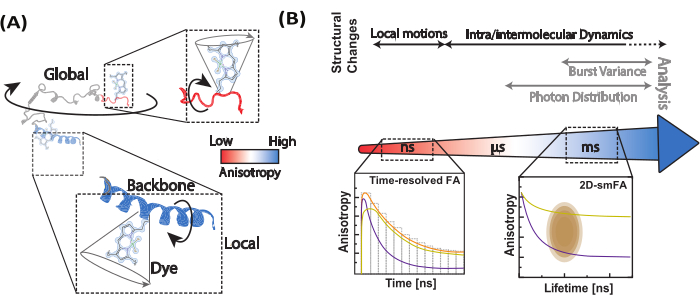
Figura 1: Rango dinámico de biomoléculas y métodos de anisotropía de fluorescencia. (A) La anisotropía de pequeños fluoróforos unidos a varias posiciones a lo largo de la columna vertebral de la biomolécula de interés informa la dinámica estructural local. (B) Escalas de tiempo sondeadas por decaimientos de intensidad de fluorescencia (anisotropía de fluorescencia resuelta en el tiempo, FA) e histogramas de una sola molécula de datos de microscopio confocal de una sola molécula. Haga clic aquí para ver una versión más grande de esta figura.
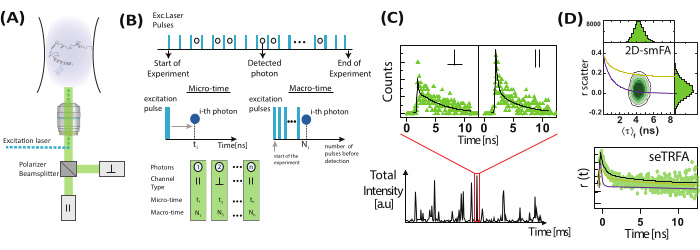
Figura 2: Registro y procesamiento de datos de anisotropía de fluorescencia de una sola molécula. (A) Las moléculas de difusión libre se analizan utilizando un microscopio confocal de una sola molécula equipado con un solo láser de excitación polarizado linealmente (azul en nuestro caso). La emisión de fluorescencia (verde en nuestro caso) es detectada por dos detectores después de que un polarizador de haz divide la señal en dos polarizaciones (paralela  , , y perpendicular,
, , y perpendicular,  , a la fuente de excitación). (B) Cada fotón detectado se caracteriza por tres parámetros: micro tiempo, macro tiempo y tipo de canal. Los datos se almacenan en un formato de tiempo resuelto con etiqueta de tiempo (TTTR)72. (C) Se seleccionan y procesan ráfagas de moléculas individuales para extraer los parámetros de fluorescencia, incluida la anisotropía de fluorescencia para cada molécula observada. (D) Los datos se representan de múltiples maneras, incluidos gráficos bidimensionales de anisotropía de fluorescencia frente a la vida útil de la fluorescencia y desintegraciones de anisotropía resueltas en el tiempo. Estas representaciones permiten la determinación visual y cuantitativa de los tiempos de vida de fluorescencia, los tiempos de correlación rotacional y la heterogeneidad del sistema. Haga clic aquí para ver una versión más grande de esta figura.
, a la fuente de excitación). (B) Cada fotón detectado se caracteriza por tres parámetros: micro tiempo, macro tiempo y tipo de canal. Los datos se almacenan en un formato de tiempo resuelto con etiqueta de tiempo (TTTR)72. (C) Se seleccionan y procesan ráfagas de moléculas individuales para extraer los parámetros de fluorescencia, incluida la anisotropía de fluorescencia para cada molécula observada. (D) Los datos se representan de múltiples maneras, incluidos gráficos bidimensionales de anisotropía de fluorescencia frente a la vida útil de la fluorescencia y desintegraciones de anisotropía resueltas en el tiempo. Estas representaciones permiten la determinación visual y cuantitativa de los tiempos de vida de fluorescencia, los tiempos de correlación rotacional y la heterogeneidad del sistema. Haga clic aquí para ver una versión más grande de esta figura.
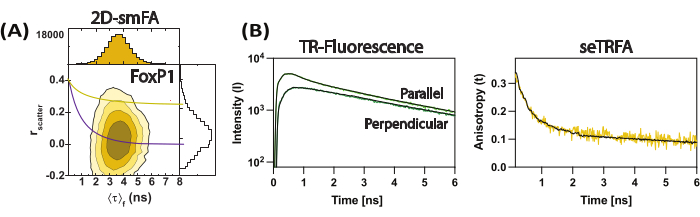
Figura 3: Datos representativos del dímero de intercambio de dominio FoxP1. (A) Correlación de la anisotropía de fluorescencia (dispersión r) contra el tiempo de vida medio de fluorescencia por molécula como un gráfico de contorno. Superposición de una sola ecuación de Perrin para dos componentes rotacionales como representante del promedio del conjunto de la molécula, considerando ρ1 y ρ 2 de 0,2 ns y 8,5 ns, respectivamente. (B) Las desintegraciones de fluorescencia resueltas en el tiempo de subconjuntos se utilizan para calcular la anisotropía de fluorescencia resuelta en el tiempo de la muestra. El ajuste con la ecuación 2 resolvió los componentes locales y globales de la anisotropía de fluorescencia. Haga clic aquí para ver una versión más grande de esta figura.
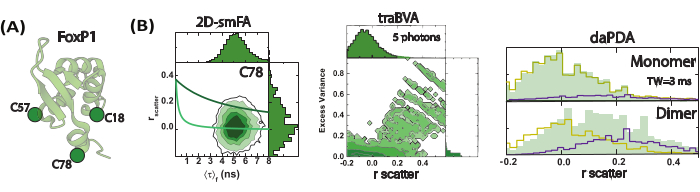
Figura 4: Dinámica de FoxP1 en submilisegundos monitoreada utilizando anisotropía de fluorescencia de una sola molécula (smFA). (A) Una representación de dibujos animados de la estructura monomérica de FoxP1. (B) Un histograma bidimensional ilustra la heterogeneidad dinámica, revelando dos tiempos de correlación rotacional distintos identificados a través de la anisotropía de fluorescencia resuelta en el tiempo. El análisis de varianza de ráfaga de anisotropía resuelta en el tiempo (traBVA) descubre un pequeño subconjunto de eventos con exceso de varianza (Ec. 6) que exhiben una gran anisotropía. El análisis cuantitativo de anisotropía dinámica mediante el análisis de distribución de fotones (PDA) extrae aún más los tipos de cambio para este proceso. Haga clic aquí para ver una versión más grande de esta figura.
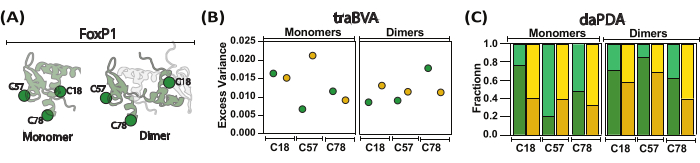
Figura 5: Cribado de los movimientos locales y globales de FoxP1 durante la dimerización. (A) Una representación de dibujos animados compara el FoxP1 monomérico con su forma dimérica. (B) Se muestra el exceso de varianza media por ubicación en condiciones monoméricas y diméricas, con un mayor exceso de varianza que indica cambios más significativos en la anisotropía. (C) El análisis dinámico de anisotropía mediante el análisis de distribución de fotones (PDA) ayuda a determinar las fracciones de población (alta anisotropía en colores oscuros y baja anisotropía en colores claros) en ausencia (verde) y presencia (amarillo) de ADN. En este enfoque, se estimaron las tasas (no mostradas) para las transiciones entre comportamientos locales y globales, revelando que FoxP1 experimenta un despliegue parcial. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Para los experimentos de anisotropía de fluorescencia de una sola molécula, es crucial considerar cuidadosamente las propiedades fotofísicas del fluoróforo elegido. Estas propiedades incluyen la longitud de onda de emisión, que debe alinearse con el sistema de detección, y la longitud de onda de excitación, que debe ser compatible con los láseres pulsados disponibles. Para optimizar el rango dinámico, el fluoróforo debe tener una larga vida útil de fluorescencia en relación con el tiempo de difusión rotacional de la molécula. Esto es fundamental para el seguimiento de la dinámica de rotación y el enlace/orientación del dipolo del fluoróforo en relación con la biomolécula de interés. Además, el brillo, la fotoestabilidad y el rendimiento cuántico son esenciales para producir señales fuertes con una relación señal-ruido estable. Por estas razones, BODIPY-FL ha sido elegido como fluoróforo en varios estudios 39,40,42.
El cribado de la dinámica de la columna vertebral de las biomoléculas a menudo requiere el etiquetado de proteínas, que normalmente se logra mediante el etiquetado específico del sitio. Por lo general, esto se hace mediante la introducción de un residuo para la modificación química específica. El enfoque más común es la introducción de cisteínas en posiciones de interés, donde sus cadenas laterales de tiol pueden modificarse selectivamente con reactivos como maleimidas o yodoacetamidas. Con menos frecuencia, los haluros bencílicos y las cetonas bromometilcetonas se utilizan para formar enlaces tioéter. También se pueden atacar otras cadenas laterales de aminoácidos, pero su abundancia en proteínas se usa con menos frecuencia. Sin embargo, también se pueden utilizar enfoques alternativos, como los aminoácidos no naturales73. La selección adecuada del sitio para el etiquetado es crucial para minimizar la interferencia con la biomolécula en estudio, y se deben implementar controles adecuados. Por ejemplo, si la molécula marcada se utiliza en ensayos de unión, los métodos complementarios sin marcaje deben verificar que los fluoróforos no afecten a la afinidad de unión.
Después de identificar la muestra adecuada e implementar la estrategia de etiquetado óptima, el siguiente paso es asegurarse de que el microscopio confocal esté correctamente alineado y calibrado para experimentos de una sola molécula. El protocolo describe cómo determinar el factor requerido para un análisis posterior. Una vez calibrado el instrumento, el siguiente paso es medir la muestra y procesar los datos para extraer la mayor cantidad de información posible de los fotones detectados. Los parámetros clave, como el microtiempo, el macrotiempo y el tipo de canal, como se muestra en la Figura 2, se pueden utilizar para un análisis y visualización posteriores utilizando la electrónica típica de TCSPC.
Los avances recientes en la espectroscopia de fluorescencia de una sola molécula se pueden utilizar ampliamente para estudiar la información estructural de los conjuntos heterogéneos de biomoléculas. Sin embargo, relativamente pocos estudios aprovechan los conocimientos proporcionados por la anisotropía de fluorescencia, y se requiere un modelo completo de proteínas para derivar la dinámica estructural de las biomoléculas. Por lo tanto, desentrañar la dinámica de las interacciones entre dominios y proteínas-proteínas de varios factores de transcripción es un desafío.
En conclusión, los experimentos de anisotropía de fluorescencia de una sola molécula ofrecen información complementaria sobre los movimientos locales y globales de la columna vertebral biomolecular, que son fundamentales para comprender su función.
Divulgaciones
Todos los autores declaran que no tienen intereses financieros contrapuestos con el contenido de este artículo.
Agradecimientos
Este trabajo contó con el apoyo de las becas FONDECYT 11200729 y FONDEQUIP EQM200202 a los premios E.M., NIH R15CA280699 R01GM151334 y NSF CAREER MCB 1749778 a HS. NK reconoció el apoyo del programa de becas postdoctorales de la Universidad de Clemson.
Materiales
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
Referencias
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. . Principles of Protein X-Ray Crystallography. , (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701 (2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. a. M. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. a. M. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676 (2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854 (2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724 (2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. . Fluorescence Correlation Spectroscopy: Theory and Applications. , (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375 (2020).
- Takahashi, H., Takahashi, K., Liu, F. -. C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302 (2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623 (2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116 (2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. , (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1 (2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077 (2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004 (2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -. J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -. O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. . Time tagged time-resolved fluorescence data collection in life sciences. , (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados