Method Article
Anisotropia di fluorescenza risolta nel tempo da singole molecole per la caratterizzazione della flessibilità locale nelle biomolecole
In questo articolo
Riepilogo
Qui, presentiamo il protocollo per studiare la flessibilità locale e la dinamica delle biomolecole utilizzando l'anisotropia di fluorescenza risolta nel tempo a livello di singola molecola in modalità microscopia confocale.
Abstract
Descriviamo un protocollo per condurre anisotropia di fluorescenza risolta nel tempo a livello di singola molecola utilizzando la microscopia confocale per studiare la flessibilità locale e la dinamica del dominio FKH (Forkhead) legante l'acido desossiribonucleico (DNA) del fattore di trascrizione FoxP1. FoxP1 dimerizza attraverso un meccanismo di scambio di domini tridimensionale (3D-DS), formando un intermedio disordinato con o senza DNA. Poiché il 3D-DS coinvolge una regione intrinsecamente disordinata, comprenderne il comportamento è fondamentale per chiarire le proprietà strutturali e funzionali di FoxP1. Utilizzando un FoxP1 marcato con cisteina singola, abbiamo condotto esperimenti di anisotropia a fluorescenza a singola molecola (smFA), applicando gli approcci di analisi della distribuzione dei fotoni di anisotropia dinamica (daPDA) e di analisi della varianza burst di anisotropia risolta nel tempo (traBVA) per sondare la flessibilità e la dinamica locale. Questo protocollo fornisce una guida dettagliata e passo dopo passo per le misure di smFA, sottolineando le analisi risolte nel tempo, la varianza e le tecniche di distribuzione di probabilità per catturare le dinamiche strutturali su diverse scale temporali. Questo approccio ci ha permesso di correlare la dinamica e l'eterogeneità alla dimerizzazione e al legame del DNA di FoxP1, evidenziando il complesso meccanismo d'azione che caratterizza questo fattore di trascrizione.
Introduzione
L'attività funzionale delle biomolecole dipende dalla loro flessibilità molecolare e dalla dinamica strutturale 1,2,3. Naturalmente, le biomolecole subiscono fluttuazioni termiche costanti, che vanno da movimenti rapidi a cambiamenti conformazionali a lungo termine che influenzano la loro funzione (Figura 1)4. Nelle biomolecole, i movimenti locali della spina dorsale contribuiscono a movimenti globali su larga scala, tra cui la flessione della cerniera negli enzimi e significativi cambiamenti conformazionali nelle proteine motorie. I metodi di determinazione della struttura come la risonanza magnetica nucleare (NMR)5, la cristallografia a raggi X6 e la microscopia elettronica criogenica (cryo-EM)7 hanno rivelato conformazioni multiple in varie biomolecole. Tuttavia, il collegamento delle fluttuazioni locali alle grandi dinamiche conformazionali delle biomolecole e il loro ruolo nella funzione sono per lo più inesplorati. Mettere in relazione dinamica e struttura può essere difficile, soprattutto per le proteine intrinsecamente disordinate (IDPs)8,9,10. A differenza delle proteine strutturate, le IDP non mantengono una struttura terziaria stabile. Invece, subiscono ampi cambiamenti conformazionali con livelli di energia libera simili, consentendo un'ampia gamma di attività biologiche11,12.
Diversi approcci sperimentali sono stati impiegati per studiare la dinamica conformazionale delle proteine sondando la loro flessibilità molecolare 1,13,14,15,16. Tra queste, la risonanza magnetica nucleare si distingue per la sua capacità di fornire una risoluzione a livello atomico su varie scale temporali, da decine di picosecondi a diverse ore12. Tuttavia, la determinazione della flessibilità macromolecolare rimane impegnativa a causa degli elevati gradi di libertà e per le proteine di grandi dimensioni; pertanto, la risonanza magnetica nucleare è spesso limitata allo studio di biomolecole di circa 100 kDa17.
Data la complessità strutturale di proteine altamente dinamiche come le IDP, sono stati sviluppati ulteriori progressi metodologici per esplorare lo spazio conformazionale locale e a lungo raggio per comprendere la loro funzione11. La spettroscopia di fluorescenza multiparametrica a singola molecola (smMFS)18,19,20,21,22 offre ampie informazioni sulle biomolecole, fornendo informazioni cruciali sulla loro funzione, dinamica conformazionale, stati di legame e stechiometria. Tuttavia, l'interpretazione della grande quantità di dati strutturali ottenuti dalle biomolecole è impegnativa e fattori come la dinamica molecolare, il comportamento dei fluorofori e il comportamento complesso delle molecole possono complicare ulteriormente l'analisi dei dati 23,24,25,26,27,28.
Utilizziamo l'anisotropia di fluorescenza a singola molecola (smFA) come metodo robusto per valutare le dinamiche locali e globali lungo la spina dorsale delle biomolecole (Figura 1A). L'anisotropia della fluorescenza, descritta per la prima volta da Perrin29 e introdotta da Weber 30,31 come strumento bioanalitico32, è stata successivamente adattata per studi su singole molecole con l'avvento di tecniche di fluorescenza risolte nel tempo e l'aumento della sensibilità dei rivelatori 33,34,35,36,37. smFA copre un'ampia gamma di scale temporali - da picosecondi a diverse ore - e integra i dati ottenuti dagli esperimenti di trasferimento di energia di risonanza di risonanza di Förster a singola molecola (smFRET)38.
smFA può essere visualizzato in vari formati per estrarre informazioni critiche sulla dinamica biomolecolare (Figura 1B). I decadimenti dell'anisotropia della fluorescenza risolta nel tempo sono istogrammi unidimensionali che catturano la dinamica sulla scala temporale da picosecondo a nanosecondo39,40. Gli istogrammi bidimensionali a singola molecola, che correlano il tempo di vita della fluorescenza con l'anisotropia per le singole molecole, possono rivelare l'eterogeneità dello stato di anisotropia e fornire informazioni visive sulle potenziali dinamiche entro il tempo di osservazione negli esperimenti confocali (~ms)41,42. Per lo studio della dinamica sub-millisecondo, è possibile utilizzare l'analisi dinamica della distribuzione dei fotoni di anisotropia (daPDA), mentre l'analisi della varianza di burst con anisotropia risolta nel tempo (traBVA) offre un metodo robusto per confermare dinamiche specifiche intorno ai millisecondi43 (Figura 1B).
Questi metodi integrano strumenti più tradizionali, come la spettroscopia di correlazione della fluorescenza risolta in polarizzazione (pFCS), che ha uno spettro più ampio 44,45,46,47. Nel complesso, diversi strumenti di analisi dei dati per la smFA facilitano l'identificazione dei cambiamenti conformazionali locali e globali, a condizione che venga presa in considerazione un'adeguata calibrazione.
Qui, applichiamo smFA per studiare il legame al DNA del fattore di trascrizione umano FoxP1 48,49,50,51. Questa proteina adotta un dimero scambiato di dominio a causa della natura intrinsecamente disordinata della sua catena polipeptidica, che è notevolmente influenzata a seconda dello stato quaternario della proteina e della presenza di DNA. Abbiamo generato diversi mutanti a singola cisteina da marcare con BODIPY-FL, eseguito esperimenti con smFA e impiegato daPDA e trBVAa. Questo approccio ci ha permesso di correlare la dinamica e l'eterogeneità alla dimerizzazione di FoxP1 e al legame con il DNA, evidenziando il complesso meccanismo d'azione che caratterizza questo fattore di trascrizione.
Protocollo
NOTA: La selezione del fluoroforo appropriato è essenziale per gli esperimenti con smFA. Le biomolecole possono essere marcate in posizioni sito-specifiche modificando gli amminoacidi nelle proteine o le basi nucleotidiche negli acidi nucleici con marcatori fluorescenti, a seconda dei gruppi reattivi disponibili. Tra i coloranti organici52, le famiglie Alexa Fluor, Cy, BODIPY e Janelia Farms sono le scelte più popolari per l'smFA, grazie alla loro lunga durata di fluorescenza, alla fotostabilità e alle elevate rese quantiche. BODIPY-FL è spesso favorito per la sua durata di fluorescenza prolungata, la resa quantica superiore e il linker di collegamento corto. Inoltre, i fluorofori alternativi sono comunemente usati nello screening farmacologico in cui sono preferite le tecniche di massa53. Le proteine fluorescenti chimeriche possono essere utilizzate anche per esperimenti di anisotropia su cellule vive e per l'imaging, sebbene vi sia una limitazione di un intervallo dinamico inferiore.
1. Preparazione del tampone
NOTA: Indossare guanti, occhiali protettivi per gli occhi e un camice da laboratorio durante gli esperimenti di laboratorio.
- Tampone standard (20 mM di acido 4- (2-idrossietil)-1-piperazineetansolfonato [HEPES], pH 7,8, 150 mM di NaCl): sciogliere 2,38 g di HEPES, 4,38 g di NaCl in 400 mL di acqua ultrapura, regolare il pH a 7,8 e portare il volume finale a 500 mL.
- Tampone di lisi (20 mM di HEPES, pH 7,8, 150 mM di NaCl, 0,1 mM di fenilmetilsulfonilfluoruro [PMSF], 10 μg/mL di DNAsi): Sciogliere 2,38 g di HEPES, 4,38 g di NaCl e 8,71 mg di PMSF e una concentrazione finale di 10 μg/mL di DNAsi in 400 mL di acqua ultrapura, regolare il pH a 7,8 e portare il volume finale a 500 mL.
- Tampone di equilibrazione (50 mM di soluzione salina tamponata con fosfato [PBS], 150 mM di NaCl e 10 mM di imidazolo; pH 7,4): sciogliere 5,05 g di Na2HPO4, 0,85 g di NaH2PO4, 4,38 g di NaCl e 0,34 g di imidazolo in 400 mL di acqua ultrapura, regolare il pH a 7,4 e portare il volume finale a 500 mL.
- Tampone di lavaggio (50 mM PBS, 150 mM NaCl e 30 mM Imidazolo; pH 7,4): sciogliere 5,05 g di Na2HPO4, 0,85 g di NaH2PO4, 4,38 g di NaCl e 1,02 g di imidazolo in 400 mL di acqua ultrapura, regolare il pH a 7,4 e portare il volume finale a 500 mL.
- Tampone di eluizione (50 mM PBS, 150 mM NaCl e 250 mM Imidazolo; pH 7,4): sciogliere 5,05 g di Na2HPO4, 0,85 g di NaH2PO4, 4,38 g di NaCl e 8,5 g di Imidazolo in 400 mL di acqua ultrapura, regolare il pH a 7,4 e portare il volume finale a 500 mL.
- tampone PBS (50 mM di soluzione salina tamponata con fosfato di sodio, 150 mM di NaCl, pH 7,4). Sciogliere 5,05 g di Na2HPO4, 0,85 g di NaH2PO4 e 4,38 g di NaCl in 400 ml di acqua ultrapura, regolare il pH a 7,4 e portare il volume finale a 500 ml.
- Sterilizzare tutti i tamponi filtrando la soluzione utilizzando filtri con pori di 0,22 μm e conservare i tamponi a temperatura ambiente (RT).
- Filtrare il tampone standard utilizzando un filtro a carbone per l'acquisizione di singole molecole.
2. Sonde fluorescenti
- BODIPY-FL: Sciogliere il flaconcino da 5 mg contenente BODIPY-FL aggiungendo un volume finale di 1,29 mL di dimetilsolfossido (DMSO) appena aperto per ottenere la concentrazione finale di 10 mM BODIPY-FL.
- Rodamina 110: Sciogliere 3,67 mg di Rodamina 110 aggiungendo un volume finale di 1 mL di DMSO appena aperto per ottenere la concentrazione finale di 10 mM di Rodamina 110.
NOTA: Le sonde fluorescenti devono essere evitate se esposte alla luce. Utilizzare sempre un tubo sensibile alla luce (colore ambra) e avvolgerlo con un foglio di alluminio. Se il DMSO è già aperto, deve essere conservato nell'essiccatore per aumentarne la durata. - Preparare aliquote di piccolo volume della sonda fluorescente preparata e conservarla a -20 °C fino a nuovo utilizzo.
NOTA: Evitare cicli di congelamento-scongelamento per migliorarne l'efficienza di etichettatura.
3. Misure di taratura
- Esegui esperimenti di rilevamento della fluorescenza multiparametrica (MFD) su una configurazione risolta a polarizzazione domestica con 4 canali di rilevamento e 2 laser pulsati (blu 485 nm e rosso 640 nm)54. Tuttavia, per le misure di anisotropia, utilizzare il laser blu e due canali del rivelatore.
- Attivare i canali del rivelatore (parallelo e perpendicolare) e l'alimentazione del laser blu.
- Assicurarsi che la potenza del laser sia impostata su 60 μW e aprire il pannello di controllo del software TCSPC (Time-Related Single Photon Counting)36 .
- Mescolare 1 μL di 100 nM di Rodamina 110 e 49 μL di acqua distillata per ottenere una concentrazione finale di 2 nM di Rodamina 110. Aggiungere 50 μl di 2 nM di rodamina 110 al centro del vetro di copertura per le misure di calibrazione.
- Quindi, aggiungere una goccia di liquido di immersione (olio o acqua, ma facendo attenzione a condividere lo stesso indice di rifrazione) sopra la lente dell'obiettivo del microscopio per aumentare il potere risolutivo del microscopio.
- Posizionare il vetro di copertura sopra la lente dell'obiettivo e assicurarsi che la goccia d'acqua sia al centro dell'obiettivo del microscopio.
- Regolare il piano dell'immagine in modo che sia all'interno della soluzione e sopra la superficie del vetro.
- Regolare la manopola per trovare il secondo punto focale luminoso e ruotarla su un punto e mezzo. Focalizzare il laser sull'interfaccia tra il vetro e il liquido.
- Massimizza il numero di fotoni rilevati regolando la posizione del foro stenopeico (70 μm) e monitorando la velocità di conteggio dei fotoni.
- Aprire il software nel pannello della modalità TTTR o T3 (Time-Tagged Time-Solved) e fare clic sul pulsante Start . Registra la velocità di conteggio per 120 s e salva il formato del file Rhodamine 110.ptu utilizzando il software di acquisizione. Questo tempo di acquisizione dovrebbe essere sufficiente considerando la concentrazione (2 nM).
- Per le misurazioni di fondo, aggiungere 50 μL di acqua distillata al centro del vetro di copertura e ripetere i passaggi 3.5-3.10. Tuttavia, registrare la velocità di conteggio dei fotoni per 300 s e salvare il file water.ptu .
- Per altre misure di fondo, aggiungere 50 μl di tampone standard al centro del vetro di copertura e ripetere i passaggi 3.5-3.10. Registra la velocità di conteggio dei fotoni per 300 s e salva il file buffer.ptu standard . Quindi, analizza i dati utilizzando il software di analisi della durata della fluorescenza con integrazione burst (BIFL).
4. Calibrazione e analisi dei dati
- Apri il software BIFL e clicca su conferma la configurazione dalla finestra automatica. Quindi, fai clic su Ottieni parametri da file, quindi su OK.
- Per analizzare la misurazione, fare clic sull'array del percorso dati e selezionarlo .
- Quindi, caricare il file water.ptu per la misurazione dell'acqua per array come Green scatter. Allo stesso modo, seleziona il file buffer.ptu standard per Sfondo verde. Per Verde denso, selezionare Rodamina 110.ptu.
- Sotto i parametri di selezione della singola molecola, fare clic su Avanti, quindi su Regola per visualizzare una nuova finestra popup. Fare clic su soglia per modificare il tempo di arrivo tra fotoni e selezionare il tempo dell'evento a singola molecola per il tempo medio di arrivo tra fotoni (dt). Quindi, fai clic su # per selezionare il numero minimo di fotoni per singolo evento molecola, quindi chiudi la finestra pop-up facendo clic su Invio. Quindi, fare clic su OK.
- Quindi, fai clic su Parametri di adattamento del colore per regolare la durata iniziale della fluorescenza per il verde, colori generati con i parametri di decadimento della fluorescenza. Regolare i valori di Prompt e Delay modificando i valori Da e A. Quindi chiudi la finestra pop-up facendo clic su Invio. Quindi, fare clic su OK.
- Fare clic su Salva per elaborare i file ASCII e salvarli in una cartella selezionata. Quindi, elabora i dati per il decadimento dell'anisotropia di fluorescenza risolta nel tempo utilizzando ChiSurf22, Photon Distribution Analysis55,56 o Burst Variance Analysis57,58.
NOTA: Dati esemplari e descrizioni dettagliate su come utilizzare ChiSurf, PDA e BVA sono disponibili rispettivamente a github.com/Fluorescence-Tools/chisurf, github.com/Fluorescence-Tools/tttrlib, www.mpc.hhu.de/en/software/mfd-fcs-and-mfis e github.com/SMB-Lab/feda_tools. I dati sperimentali sono disponibili presso Zenodo (10.5281/zenodo.13371503).
5. Preparato proteico FoxP1
- Sovraespressione batterica di FoxP1 ricombinante
- Eseguire la trasformazione in cellule batteriche C41 di E. coli dopo la mutagenesi della reazione a catena della polimerasi (PCR) diretta dal sito a cambio rapido.
- Preparare il terreno LB e l'autoclave per sterilizzarlo.
- Pre-inoculare una singola colonia di E. coli C41 aggiungendola a 5 ml di terreno LB contenente 5 μL (100 μg/mL) di ampicillina. Lasciare incubare a 37 °C per una notte su un agitatore per incubatrice.
- Il giorno successivo, inoculare la coltura batterica su larga scala aggiungendo durante la notte il pre-inoculo in 500 ml di terreno LB con antibiotico pre-aggiunto con un rapporto di 1:500.
- Monitorare la crescita della coltura misurando l'assorbanza della coltura a 600 nm.
- Quando la densità ottica raggiunge un valore compreso tra ~0,5-0,7, indurre l'espressione proteica aggiungendo una concentrazione finale di 0,5 mM di isopropil-β-d-1-tiogalattopiranoside (IPTG) e mantenere la coltura a 15 °C su un agitatore incubatore per una notte.
- Dopo aver raggiunto una densità ottica (600 nm) nell'intervallo 1,4-1,6, raccogliere le cellule batteriche mediante centrifugazione a 3000 g per 20 minuti a 4 °C. Eliminare il surnatante e conservare il pellet a -20 °C fino al momento dell'uso.
- Purificazione di FoxP1 ricombinante
- Lisi le cellule di E. coli C41 aggiungendo tampone di lisi utilizzando qualsiasi metodo di lisi come sonicazione, omogeneizzazione di liquidi, pressa francese, ecc.
- Centrifugare il lisato a 14000 g per 10 min a 4 °C.
- Per le proteine con 6 marcaturedi His lavare la colonna di affinità Ni2+-NTA ed equilibrare con NiSO4 utilizzando la cromatografia liquida proteica veloce (FPLC).
- Caricare la proteinaFoxP1 con 6 marcature His nella colonna di affinità Ni2+-NTA bilanciata.
- Eluire la proteina FoxP1 dalla colonna Ni2+-NTA utilizzando un gradiente lineare del tampone di eluizione.
- Dopo che la proteina è stata eluita, eseguire la dialisi per lo scambio del tampone. Aggiungere le proteine in un tampone di equilibrazione senza imidazolo e dializzare per una notte utilizzando un agitatore magnetico a 4 °C.
NOTA: Se la proteina viene purificata con un metodo diverso, eseguire lo scambio del tampone di conseguenza. - Per rimuovere l'His6-tag, aggiungere la proteasi del virus dell'incisione del tabacco (TEV) (rapporto 1:100 di TEV:FoxP1) alla dialisi durante la notte.
NOTA: La sua digestione a6 tag può essere eseguita anche aggiungendo la digestione della proteasi TEV su colonna. Questo passaggio migliorerebbe la resa della proteina di interesse. - Il giorno successivo, ripetere i passaggi da 5.2.3 a 5.2.5. Tuttavia, questa volta, eluire la proteina nel tampone di lavaggio piuttosto che nel tampone di eluizione.
- Concentrare la proteina a un volume appropriato aggiungendo un tampone PBS e quantificare la proteina utilizzando l'assorbanza a 280 nm, considerando il coefficiente di estinzione della proteina.
- Marcatura FoxP1 ricombinante
- Incubare 50-100 μM di proteina FoxP1 con un eccesso molare di 10 volte di ditiotreitolo (DTT) o tris (2-carbossietil)fosfina (TCEP) con un volume finale di 500 μl a RT per 30 minuti.
- Eseguire la sostituzione del buffer utilizzando le colonne di desalinizzazione PD 10. Posizionare la colonna PD10 in una provetta da centrifuga da 50 mL utilizzando un adattatore per colonna.
- Equilibrare la colonna PD10 aggiungendo 5 mL di tampone PBS e centrifugare a 1000 × g per 2 minuti. Ripeti questo passaggio 3 volte.
- Conservare la colonna PD10 in una provetta fresca da 50 mL e aggiungere 2 mL di tampone PBS nella colonna. Quindi, aggiungere 500 μL di proteina FoxP1 dalla fase 1 nella colonna. Eluire la proteina centrifugando 1000 × g per 2 minuti e raccogliere la proteina.
- Concentrare la proteina aggiungendola in filtri ultra centrifughi (10 kDa MWCO), centrifugare a 7500 × g per 10 minuti e raccogliere l'eluizione.
- Misurare la concentrazione proteica, aggiungere BODIPY con il 30% equivalente di concentrazione di cisteina nella proteina e posizionarlo su un rotatore per 2 ore a 4 °C.
- Eseguire lo scambio di tamponi e concentrare la proteina come discusso nei passaggi 5.3.2-5.3.5.
- Controllare la concentrazione proteica utilizzando uno spettrofotometro o un saggio colorimetrico, come il metodo Bradford o Lowry, e la concentrazione del colorante per assorbanza a 500 nm.
- Misurare il grado di etichettatura utilizzando la seguente formula:
(ADye/εDye)/(peso molecolare della proteina/milligrammo di proteina per millilitro) = moli di colorante/moli di proteina
Dove ADye e εDye sono rispettivamente il valore di assorbanza del colorante e il coefficiente di estinzione molare del colorante alla massima lunghezza d'onda di assorbanza. Se misurata a 280 nm, l'assorbanza reale della proteina deve essere corretta con la seguente formula:
Aproteina = A280-(AColorante· CF280)
Dove CF280 è il fattore di correzione per il colorante specificato, considerando la sua assorbanza intrinseca a 280 nm59.
NOTA: Mentre ci concentriamo sull'espressione e la purificazione di una proteina modificata con cisteina, è necessario prestare particolare attenzione all'introduzione strategica di residui di cisteina nella proteina.
6. Preparazione della camera del campione al microscopio
- Aggiungere 495 μl di acqua ultrapura e 5 μl (concentrazione finale 0,01% v/v) di Tween-20 (tensioattivo non ionico) nel pozzetto di un vetrino da camera e mescolare. Incubare per oltre 30 minuti a RT.
- Lavare delicatamente la camera con acqua ultrapura due volte e asciugarla.
NOTA: La camera è pronta per l'uso per l'esperimento.
7. Esperimento di anisotropia a fluorescenza a singola molecola
- Innanzitutto, calibrare lo strumento smFA. Determinare la funzione di risposta dello strumento, misurando la luce diffusa Raman o misurando la fluorescenza di un colorante con una breve durata e uno spettro di emissione appropriato (ad esempio, eritrosina o verde di malachite) ulteriormente spento da una soluzione satura di ioduro di potassio, KI.
- Per correggere la fluorescenza di fondo, eseguire le misurazioni di una soluzione tampone. Calibrare la sensibilità relativa dei rivelatori paralleli e perpendicolari (fattore G) acquisendo dati da un colorante a rotazione rapida. Qui, 2 nM di rodamina 110 sono stati utilizzati per calibrare i rivelatori60.
- Per gli esperimenti smFA su FoxP1 monomerico, aggiungere 100 pM di FoxP1 marcato con BODIPY e 100 nM di FoxP1 non marcato a 500 μL di tampone standard (20 mM HEPES, 20 mM NaCl, pH 7,8) in un vetrino da camera.
- Per il saggio di titolazione con DNA e FoxP1 monomerico, aggiungere 400 nM di DNA a 100 pM di miscela proteica marcata con BODIPY/100nM non marcata e incubare a RT per 10 minuti.
- Per gli esperimenti smFA su FoxP1 dimerorico, aggiungere 500 nM di FoxP1 non marcato a 100 pM di FoxP1 marcato con BODIPY nel tampone standard. Quindi, incubare a 37 °C per 30 minuti.
- Per il saggio di titolazione con DNA e FoxP1 dimerico, aggiungere 2000 nM di DNA a 100 pM di miscela di proteine marcate con BODIPY/500 nM non marcate e incubare a RT per 10 minuti.
- Avviare le misurazioni e verificare nel software BIFL che la quantità di raffiche in un record di 30 s sia compresa tra 60 e 90. Se il campione è più concentrato, diluirlo fino a raggiungere tale valore. Questo passaggio garantisce le condizioni di una singola molecola.
- Iniziare l'smFA ad ogni misurazione per FoxP1 monomerico o FoxP1 dimerico in assenza o presenza di DNA per almeno 4 ore.
- Analizzare le misure di smFA come discusso nella sezione 4.
Risultati
L'anisotropia della fluorescenza deriva dall'orientamento relativo dell'assorbimento del fluoroforo e dai momenti di dipolo di emissione. Quando i fluorofori sono esposti alla luce polarizzata, i fluorofori con momenti di transizione di assorbimento allineati con il vettore del campo elettrico della luce incidente sono eccitati preferenzialmente (fotoselezione). Di conseguenza, la popolazione dello stato eccitato diventa parzialmente orientata, con una frazione significativa delle molecole eccitate che hanno i loro momenti di transizione allineati con il vettore del campo elettrico della luce eccitante polarizzata61. I fluorofori ruotano a causa del loro moto browniano. Pertanto, anche il momento di transizione dell'emissione ruota, con conseguente dipendenza dal tempo dall'anisotropia della fluorescenza. Questo effetto può essere utilizzato per misurare i movimenti rotatori delle molecole fluorescenti, rilevare eventi di legame, caratterizzare l'ambiente del fluoroforo e catturare le dinamiche molecolari.
Gli esperimenti su singole molecole sono in grado di determinare l'eterogeneità del campione. Sfruttando la sensibilità di una singola molecola e l'anisotropia della fluorescenza, si aggiunge un'altra dimensionalità alla spettroscopia di fluorescenza multiparametrica. In un tipico microscopio confocale a singola molecola (Figura 2)20,21, l'anisotropia della fluorescenza può essere determinata tramite l'intensità o risolta nel tempo quando si utilizzano laser pulsati.
Per considerare gli effetti depolarizzanti dell'obiettivo ad alta apertura numerica in un microscopio confocale62, la forma corretta dell'anisotropia risolta nel tempo35,63 è data da
 (1)
(1)
dove 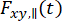 e
e  sono l'intensità di fluorescenza risolta nel tempo nel canale di rivelazione Y-esimo dopo l'eccitazione alla lunghezza d'onda x, per la polarizzazione parallela e perpendicolare L1 e L2 e sono fattori che descrivono la miscelazione tra i segnali paralleli e perpendicolari a causa dell'obiettivo ad alta apertura numerica (NA) utilizzato in queste misure35,62, 64. Le differenze nelle efficienze di rilevamento del canale di rilevamento parallelo,
sono l'intensità di fluorescenza risolta nel tempo nel canale di rivelazione Y-esimo dopo l'eccitazione alla lunghezza d'onda x, per la polarizzazione parallela e perpendicolare L1 e L2 e sono fattori che descrivono la miscelazione tra i segnali paralleli e perpendicolari a causa dell'obiettivo ad alta apertura numerica (NA) utilizzato in queste misure35,62, 64. Le differenze nelle efficienze di rilevamento del canale di rilevamento parallelo, 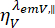 e perpendicolare,
e perpendicolare, 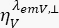 , per il colorante sono corrette con il rapporto delle efficienze di rilevamento,
, per il colorante sono corrette con il rapporto delle efficienze di rilevamento, 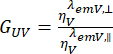 . Il GUV è anche indicato come fattore G.
. Il GUV è anche indicato come fattore G.
L'anisotropia di fluorescenza risolta nel tempo può essere modellata utilizzando un decadimento multiesponenziale per tenere conto del fatto che il fluoroforo è legato a una biomolecola più grande come
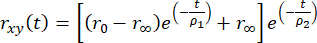 , (2)
, (2)
dove r0 è l'anisotropia fondamentale fluoroforo dipendente (tipicamente r0 = 0,38), r∞ è l'anisotropia residua e ρ1 e ρ2 sono rispettivamente tempi di correlazione rotazionale veloci (movimenti locali del fluoroforo) e lenti (moto globale della macromolecola).
Nelle misure di anisotropia di singole molecole (Figura 2), i tempi di arrivo dei fotoni vengono registrati per identificare i singoli emettitori utilizzando l'analisi BIFL (Burst-Integrated Fluorescence Lifetime)33,35. I tempi di arrivo interfotone (Δt) vengono smussati utilizzando una media corrente e quindi tracciati per facilitare la visualizzazione. L'istogramma di questi tempi è dotato di una mezza gaussiana per determinare la media e la deviazione standard dei fotoni provenienti dallo sfondo. Una soglia arbitraria, fissata a multipli della deviazione standard, viene utilizzata per filtrare i singoli eventi e identificare il primo e l'ultimo fotone in ogni lampo. I fotoni all'interno di ogni lampo vengono quindi integrati per ulteriori analisi, che includono il calcolo dell'anisotropia di fluorescenza allo stato stazionario risolta nel tempo e basata sull'intensità utilizzando le equazioni 1 e 2 o tramite uno stimatore di massima verosimiglianza35. A causa del numero limitato di fotoni negli eventi a singola molecola, lo stimatore di massima verosimiglianza considera solo una singola componente esponenziale e non sarà ulteriormente discusso.
In un istogramma bidimensionale di eventi di singole molecole, il tempo medio di fluorescenza (τ) e l'anisotropia (rxy) possono essere correlati dall'equazione di Perrin29,61 per ottenere (ρ) come tempo di rotazione medio.
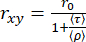 (3)
(3)
Valori specifici di ρ possono essere ottenuti con maggiore certezza mediante analisi "sub-ensemble" (se) in cui i fotoni di diversi burst sono integrati in un decadimento combinato di anisotropia di fluorescenza risolto nel tempo che può essere analizzato ottimizzando i parametri dell'equazione 2 al decadimento sperimentale (seTRFA). L'anisotropia risolta nel tempo può risolvere l'eterogeneità e la dinamica associate ai moti rotazionali (locali e globali) delle biomolecole all'interno dell'emissione di fluorescenza che si verifica entro l'intervallo di tempo ns.
Per rilevare le dinamiche all'interno di eventi di singole molecole (su scala submillisecondo), abbiamo introdotto l'analisi della varianza del burst dell'anisotropia risolta nel tempo (traBVA)57. In traBVA, per un lampo di fotoni contenente Mi segmenti di fotoni consecutivi, l'eccesso di varianza di anisotropia (s2) per i lampi è
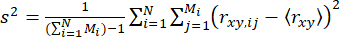 . (4)
. (4)
Per un singolo stato anisotropo, la varianza σ2 deriva esclusivamente dal rumore di sparo65 (sn: √N, dove N è il numero di fotoni)
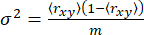 (5)
(5)
dove m è il numero di fotoni in un burst. Quindi, per identificare una varianza aggiuntiva nell'anisotropia, possiamo definire l'eccesso di varianza anisotropia (S2) dovuto all'eterogeneità conformazionale come la differenza tra le equazioni 4 e 5.
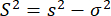 (6)
(6)
Per catturare le dinamiche che si verificano all'interno dell'osservazione di singole molecole e considerare l'approssimazione della varianza, è possibile utilizzare l'analisi dinamica della distribuzione dei fotoni (daPDA)55,56. Nei daPDA, l'intensità della fluorescenza è modellata seguendo una probabilità condizionata (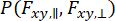 ) espressa come distribuzione binomiale.
) espressa come distribuzione binomiale.
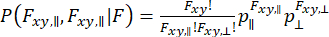 (7)
(7)
Insieme, con una stima del tasso di conteggio di fondo che segue una distribuzione di Poisson
 (8)
(8)
dove  è il numero medio di fotoni di sfondo per una finestra temporale impostata. I conteggi di fondo paralleli
è il numero medio di fotoni di sfondo per una finestra temporale impostata. I conteggi di fondo paralleli 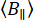 e perpendicolari e
e perpendicolari e 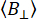 , possono essere misurati utilizzando campioni tampone come riferimento. L'anisotropia di fluorescenza determinata sperimentalmente è ottimizzata minimizzando una cifra di merito χ2 con una distribuzione dell'intensità di fluorescenza per canale di polarizzazione che può includere cambiamenti cinetici.
, possono essere misurati utilizzando campioni tampone come riferimento. L'anisotropia di fluorescenza determinata sperimentalmente è ottimizzata minimizzando una cifra di merito χ2 con una distribuzione dell'intensità di fluorescenza per canale di polarizzazione che può includere cambiamenti cinetici.
Le routine di analisi e le rappresentazioni dei dati fornite offrono un approccio completo all'interpretazione dei dati raccolti. Sebbene questo protocollo si concentri principalmente sulle misure confocali, che sono limitate nel catturare le variazioni di anisotropia da nanosecondi a millisecondi, è possibile adottare un microscopio a riflessione interna totale per monitorare l'anisotropia di fluorescenza su scale temporali più lunghe, consentendo l'analisi delle serie temporali66. Per le misure confocali a singola molecola, evidenziamo l'uso di istogrammi multidimensionali che creano un'impronta digitale unica dell'insieme osservato. I decadimenti di fluorescenza risolti nel tempo, ricostruiti da popolazioni selezionate, possono tracciare l'evoluzione dell'anisotropia di fluorescenza su scala di nanosecondi (Figura 3). L'analisi della distribuzione dei fotoni 55,56 e l'analisi della varianza del burst (BVA)57,58 possono anche catturare dinamiche su scale temporali intermedie tra decadimenti risolti nel tempo e istogrammi multidimensionali. Sebbene questo protocollo non copra l'uso della spettroscopia di correlazione della fluorescenza di polarizzazione (FCS), con o senza eccitazione pulsata67,68, che può colmare le scale temporali da nanosecondi a millisecondi, gli stessi dati possono essere utilizzati per calcolare FCS69, sebbene ciò esuli dall'ambito del protocollo presentato. Se si intraprendono tali esperimenti, si consiglia di prolungare il tempo di misurazione del campione.
Questo approccio è stato applicato a un sistema complesso come le proteine FoxP umane, fornendo preziose informazioni sui movimenti coinvolti nel loro meccanismo d'azione. Le proteine FoxP sono fattori di trascrizione coinvolti in diversi aspetti fisiologici come lo sviluppo cerebrale e polmonare; È importante sottolineare che diverse mutazioni sono state riconosciute come compromettenti la funzione di queste proteine70,71. Utilizzando il dominio di legame al DNA di FoxP1 come modello, abbiamo generato diversi mutanti a cisteina singola per introdurre un colorante BODIPY-FL come tracker per i movimenti (Figura 4A). Infatti, abbiamo valutato l'effetto della dimerizzazione e del legame al DNA come principali regolatori strutturali di questa proteina. Utilizzando l'approccio smFA, abbiamo generato grafici 2D-smFA e realizzato traBVA e daPDA per ciascun mutante in condizioni monomeriche e dimeriche. Mostriamo un esempio di uno dei singoli mutanti studiati (Figura 4). Il comportamento dell'anisotropia è simile in tutti i mutanti in termini di determinazione di tempi di correlazione rotazionale alti e bassi e, quindi, di insiemi presumibili, disordinati e ripiegati. Tuttavia, è anche altamente eterogeneo in tutti i mutanti in termini di frazione e cinetica di ciascun insieme, evidenziando diversi cambiamenti di transizione da ordine a disordine influenzati dalla dimerizzazione e dal legame con il DNA, e mostra la descrizione ad alta risoluzione delle dinamiche strutturali lungo la catena (Figura 5).
Figura 1: Gamma dinamica di biomolecole e metodi di anisotropia a fluorescenza. (A) L'anisotropia di piccoli fluorofori legati a varie posizioni lungo la spina dorsale della biomolecola di interesse informa le dinamiche strutturali locali. (B) Scale temporali sondate dai decadimenti dell'intensità della fluorescenza (anisotropia della fluorescenza risolta nel tempo, FA) e istogrammi a singola molecola di dati confocali al microscopio a singola molecola. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 2: Registrazione ed elaborazione dei dati sull'anisotropia di fluorescenza a singola molecola. (A) Le molecole a diffusione libera vengono analizzate utilizzando un microscopio confocale a singola molecola dotato di un singolo laser di eccitazione polarizzato linearmente (blu nel nostro caso). L'emissione di fluorescenza (verde nel nostro caso) viene rilevata da due rivelatori dopo che un polarizzatore di fascio divide il segnale in due polarizzazioni (parallela,  , e perpendicolare,
, e perpendicolare,  , alla sorgente di eccitazione). (B) Ogni fotone rilevato è caratterizzato da tre parametri: micro tempo, macro tempo e tipo di canale. I dati vengono memorizzati in un formato TTTR (Time-Tagged Time-Solved)72. (C) Esplosioni di singole molecole vengono selezionate ed elaborate per estrarre i parametri di fluorescenza, inclusa l'anisotropia di fluorescenza per ogni molecola osservata. (D) I dati sono rappresentati in diversi modi, tra cui grafici bidimensionali dell'anisotropia della fluorescenza rispetto alla durata della fluorescenza e ai decadimenti dell'anisotropia risolti nel tempo. Queste rappresentazioni consentono la determinazione sia visiva che quantitativa della durata della fluorescenza, dei tempi di correlazione rotazionale e dell'eterogeneità del sistema. Clicca qui per visualizzare una versione più grande di questa figura.
, alla sorgente di eccitazione). (B) Ogni fotone rilevato è caratterizzato da tre parametri: micro tempo, macro tempo e tipo di canale. I dati vengono memorizzati in un formato TTTR (Time-Tagged Time-Solved)72. (C) Esplosioni di singole molecole vengono selezionate ed elaborate per estrarre i parametri di fluorescenza, inclusa l'anisotropia di fluorescenza per ogni molecola osservata. (D) I dati sono rappresentati in diversi modi, tra cui grafici bidimensionali dell'anisotropia della fluorescenza rispetto alla durata della fluorescenza e ai decadimenti dell'anisotropia risolti nel tempo. Queste rappresentazioni consentono la determinazione sia visiva che quantitativa della durata della fluorescenza, dei tempi di correlazione rotazionale e dell'eterogeneità del sistema. Clicca qui per visualizzare una versione più grande di questa figura.
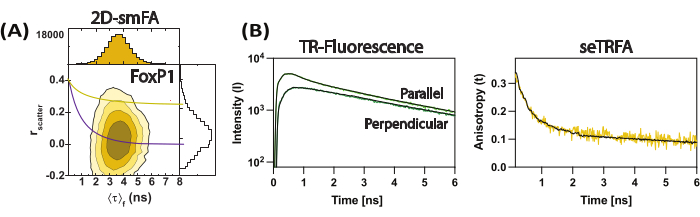
Figura 3: Dati rappresentativi per il dimero con scambio di dominio FoxP1. (A) Correlazione dell'anisotropia della fluorescenza (scatter r) rispetto al tempo medio di fluorescenza per molecola come grafico di contorno. Sovrapposizione di una singola equazione di Perrin per due componenti rotazionali come rappresentante della media d'insieme della molecola, considerando ρ1 e ρ2 di 0,2 ns e 8,5 ns, rispettivamente. (B) I decadimenti di fluorescenza risolti nel tempo del sottoinsieme vengono utilizzati per calcolare l'anisotropia di fluorescenza risolta nel tempo del campione. L'adattamento con l'equazione 2 ha risolto le componenti locali e globali dell'anisotropia di fluorescenza. Clicca qui per visualizzare una versione più grande di questa figura.
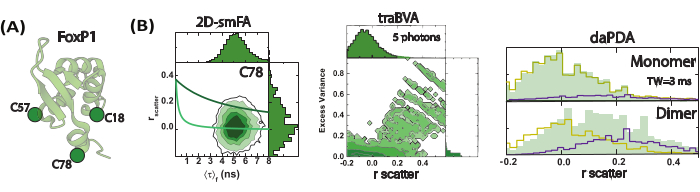
Figura 4: Dinamica di FoxP1 inferiore al millisecondo monitorata utilizzando l'anisotropia di fluorescenza a singola molecola (smFA). (A) Una rappresentazione a fumetti della struttura monomerica di FoxP1. (B) Un istogramma bidimensionale illustra l'eterogeneità dinamica, rivelando due distinti tempi di correlazione rotazionale identificati attraverso l'anisotropia di fluorescenza risolta nel tempo. L'analisi della varianza burst risolta nel tempo (traBVA) scopre un piccolo sottoinsieme di eventi con varianza eccessiva (Eq. 6) che mostrano una grande anisotropia. L'analisi quantitativa dell'anisotropia dinamica mediante l'analisi della distribuzione dei fotoni (PDA) estrae ulteriormente i tassi di cambio per questo processo. Clicca qui per visualizzare una versione più grande di questa figura.
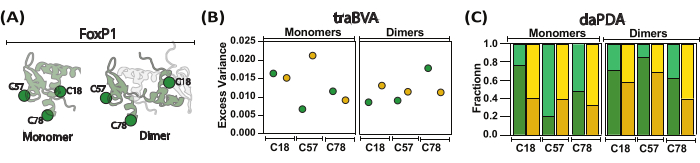
Figura 5: Screening dei movimenti locali e globali di FoxP1 durante la dimerizzazione. (A) Una rappresentazione in cartone animato confronta il monomerico FoxP1 con la sua forma dimerica. (B) Viene mostrata l'eccesso di varianza media per posizione in condizioni monomeriche e dimeriche, con un eccesso di varianza maggiore che indica cambiamenti più significativi nell'anisotropia. (C) L'analisi dell'anisotropia dinamica mediante l'analisi della distribuzione dei fotoni (PDA) aiuta a determinare le frazioni di popolazione (alta anisotropia nei colori scuri e bassa anisotropia nei colori chiari) in assenza (verde) e presenza (giallo) di DNA. In questo approccio, sono stati stimati i tassi (non mostrati) per le transizioni tra comportamenti locali e globali, rivelando che FoxP1 subisce un parziale dispiegamento. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Per gli esperimenti di anisotropia di fluorescenza a singola molecola, è fondamentale considerare attentamente le proprietà fotofisiche del fluoroforo scelto. Queste proprietà includono la lunghezza d'onda di emissione, che deve allinearsi con il sistema di rilevamento, e la lunghezza d'onda di eccitazione, che deve essere compatibile con i laser pulsati disponibili. Per ottimizzare l'intervallo dinamico, il fluoroforo dovrebbe avere una lunga durata di fluorescenza rispetto al tempo di diffusione rotazionale della molecola. Questo è fondamentale per tracciare la dinamica rotazionale e il collegamento/orientamento del dipolo del fluoroforo rispetto alla biomolecola di interesse. Inoltre, la luminosità, la fotostabilità e la resa quantica sono essenziali per produrre segnali forti con un rapporto segnale/rumore stabile. Per questi motivi, BODIPY-FL è stato scelto come fluoroforo in diversi studi 39,40,42.
Lo screening delle dinamiche della spina dorsale delle biomolecole richiede spesso la marcatura delle proteine, tipicamente ottenuta attraverso la marcatura sito-specifica. Questo di solito viene fatto introducendo un residuo per una modifica chimica mirata. L'approccio più comune consiste nell'introdurre cisteine in posizioni di interesse, dove le loro catene laterali tioliche possono essere modificate selettivamente con reagenti come maleimmidi o iodoacetammidi. Meno comunemente, gli alogenuri benzilici e i bromometilchetoni vengono utilizzati per formare legami tioetere. Altre catene laterali di amminoacidi possono anche essere prese di mira, ma la loro abbondanza nelle proteine è meno comunemente usata. Tuttavia, possono essere utilizzati anche approcci alternativi, come gli amminoacidi innaturali73. La corretta selezione del sito per l'etichettatura è fondamentale per ridurre al minimo l'interferenza con la biomolecola in studio e devono essere in atto controlli appropriati. Ad esempio, se la molecola marcata viene utilizzata nei saggi di legame, i metodi complementari senza marcatura dovrebbero verificare che i fluorofori non influiscano sull'affinità di legame.
Dopo aver identificato il campione appropriato e aver implementato la strategia di etichettatura ottimale, il passo successivo consiste nel garantire che il microscopio confocale sia correttamente allineato e calibrato per gli esperimenti su singole molecole. Il protocollo descrive come determinare il fattore richiesto per ulteriori analisi. Una volta calibrato lo strumento, il passaggio successivo consiste nel misurare il campione ed elaborare i dati per estrarre quante più informazioni possibili dai fotoni rilevati. I parametri chiave, come il micro-tempo, il macro-tempo e il tipo di canale, come mostrato nella Figura 2, possono essere utilizzati per ulteriori analisi e visualizzazioni utilizzando l'elettronica tipica del TCSPC.
I recenti progressi nella spettroscopia di fluorescenza a singola molecola possono essere ampiamente utilizzati per studiare le informazioni strutturali provenienti da insiemi eterogenei di biomolecole. Tuttavia, relativamente pochi studi sfruttano le intuizioni fornite dall'anisotropia della fluorescenza ed è necessario un modello proteico completo per derivare la dinamica strutturale delle biomolecole. Pertanto, è difficile svelare le dinamiche dell'interdominio e delle interazioni proteina-proteina di diversi fattori di trascrizione.
In conclusione, gli esperimenti di anisotropia a fluorescenza a singola molecola offrono informazioni complementari sui movimenti locali e globali della spina dorsale biomolecolare, che sono fondamentali per comprenderne la funzione.
Divulgazioni
Tutti gli autori dichiarano di non avere interessi finanziari concorrenti con i contenuti di questo articolo.
Riconoscimenti
Questo lavoro è stato supportato dalle sovvenzioni FONDECYT 11200729 e FONDEQUIP EQM200202 a E.M., NIH R15CA280699 R01GM151334 e NSF CAREER MCB 1749778 premi a HS. NK ha riconosciuto il sostegno del programma di borse di studio post-dottorato della Clemson University.
Materiali
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
Riferimenti
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. Principles of Protein X-Ray Crystallography. , Springer, Verlag. New York, NY. (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701(2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. aM. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , Elsevier. Cambridge, MA. (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. aM. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676(2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854(2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724(2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. Fluorescence Correlation Spectroscopy: Theory and Applications. , Springer. Berlin, Heidelberg. (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375(2020).
- Takahashi, H., Takahashi, K., Liu, F. -C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , Springer. New York, NY. (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302(2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623(2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116(2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. Principles of Fluorescence Spectroscopy. , Springer. New York, NY. (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1(2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077(2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004(2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. Time tagged time-resolved fluorescence data collection in life sciences. , https://www.picoquant.com/images/uploads/page/files/14528/technote_tttr.pdf (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon