Method Article
Zeitaufgelöste Fluoreszenzanisotropie von einzelnen Molekülen zur Charakterisierung der lokalen Flexibilität in Biomolekülen
In diesem Artikel
Zusammenfassung
Hier stellen wir das Protokoll vor, um die lokale Flexibilität und Dynamik von Biomolekülen unter Verwendung von zeitaufgelöster Fluoreszenzanisotropie auf Einzelmolekülebene im konfokalen Mikroskopiemodus zu untersuchen.
Zusammenfassung
Wir beschreiben ein Protokoll zur Durchführung zeitaufgelöster Fluoreszenzanisotropie auf Einzelmolekülebene mittels konfokaler Mikroskopie, um die lokale Flexibilität und Dynamik der Desoxyribonukleinsäure (DNA)-bindenden Forkhead (FKH)-Domäne des FoxP1-Transkriptionsfaktors zu untersuchen. FoxP1 dimerisiert durch einen dreidimensionalen Domänentauschmechanismus (3D-DS) und bildet ein ungeordnetes Zwischenprodukt mit oder ohne DNA. Da es sich bei 3D-DS um eine intrinsisch ungeordnete Region handelt, ist das Verständnis ihres Verhaltens entscheidend für die Aufklärung der strukturellen und funktionellen Eigenschaften von FoxP1. Unter Verwendung eines Einzelcystein-markierten FoxP1 führten wir Einzelmolekül-Fluoreszenzanisotropie-Experimente (smFA) durch, wobei wir die dynamische Anisotropie-Photonenverteilungsanalyse (daPDA) und die zeitaufgelöste Anisotropie-Burst-Varianz-Analyse (traBVA) anwandten, um die lokale Flexibilität und Dynamik zu untersuchen. Dieses Protokoll bietet eine detaillierte Schritt-für-Schritt-Anleitung für smFA-Messungen, wobei der Schwerpunkt auf zeitaufgelösten Analysen, Varianz- und Wahrscheinlichkeitsverteilungstechniken liegt, um die strukturelle Dynamik über verschiedene Zeitskalen hinweg zu erfassen. Dieser Ansatz ermöglichte es uns, Dynamik und Heterogenität mit der Dimerisierung und DNA-Bindung von FoxP1 in Beziehung zu setzen, was den komplexen Wirkmechanismus hervorhebt, der diesen Transkriptionsfaktor charakterisiert.
Einleitung
Die funktionelle Aktivität von Biomolekülen hängt von ihrer molekularen Flexibilität und Strukturdynamik ab 1,2,3. Naturgemäß unterliegen Biomoleküle ständigen thermischen Schwankungen, die von schnellen Bewegungen bis hin zu langfristigen Konformationsänderungen reichen, die ihre Funktion beeinflussen (Abbildung 1)4. In Biomolekülen tragen lokale Rückgratbewegungen zu größeren globalen Bewegungen bei, einschließlich der Scharnierbiegung in Enzymen und signifikanten Konformationsänderungen in Motorproteinen. Methoden zur Strukturbestimmung wie die Kernspinresonanz (NMR)5, die Röntgenkristallographie6 und die kryogene Elektronenmikroskopie (Kryo-EM)7 haben mehrere Konformationen in verschiedenen Biomolekülen aufgedeckt. Dennoch ist der Zusammenhang zwischen den lokalen Fluktuationen und der großen Konformationsdynamik von Biomolekülen und ihrer Rolle in der Funktion weitgehend unerforscht. Die Beziehung zwischen Dynamik und Struktur kann eine Herausforderung darstellen, insbesondere bei intrinsisch ungeordneten Proteinen (IDPs)8,9,10. Im Gegensatz zu strukturierten Proteinen behalten IDPs keine stabile Tertiärstruktur bei. Stattdessen durchlaufen sie umfangreiche Konformationsänderungen mit ähnlichen freien Energieniveaus, was ein breites Spektrum biologischer Aktivitäten ermöglicht 11,12.
Mehrere experimentelle Ansätze wurden verwendet, um die Konformationsdynamik von Proteinen zu untersuchen, indem ihre molekulare Flexibilität untersuchtwurde 1,13,14,15,16. Unter diesen zeichnet sich die NMR durch ihre Fähigkeit aus, eine Auflösung auf atomarer Ebene über verschiedene Zeitskalen zu liefern, von Dutzenden von Pikosekunden bis zu mehreren Stunden12. Die Bestimmung der makromolekularen Flexibilität bleibt jedoch aufgrund der hohen Freiheitsgrade und für großformatige Proteine eine Herausforderung; daher ist die NMR oft auf die Untersuchung von Biomolekülen von etwa 100 kDabeschränkt 17.
Angesichts der strukturellen Komplexität hochdynamischer Proteine wie IDPs wurden zusätzliche methodische Fortschritte entwickelt, um den lokalen und langreichweitigen Konformationsraum zu erforschen und ihre Funktion zu verstehen11. Die Einzelmolekül-Multiparameter-Fluoreszenzspektroskopie (smMFS)18,19,20,21,22 bietet umfangreiche Informationen über Biomoleküle und liefert wichtige Einblicke in ihre Funktion, Konformationsdynamik, Bindungszustände und Stöchiometrie. Die Interpretation der riesigen Menge an Strukturdaten, die aus Biomolekülen gewonnen werden, ist jedoch eine Herausforderung, und Faktoren wie die Molekulardynamik, das Verhalten von Fluorophoren und das komplexe Verhalten von Molekülen können die Datenanalyse weiter erschweren 23,24,25,26,27,28.
Wir verwenden die Einzelmolekül-Fluoreszenzanisotropie (smFA) als robuste Methode zur Bewertung der lokalen und globalen Dynamik entlang des Rückgrats von Biomolekülen (Abbildung 1A). Die Fluoreszenzanisotropie, die zuerst von Perrin29 beschrieben und von Weber30,31 als bioanalytisches Werkzeug32 eingeführt wurde, wurde später mit dem Aufkommen zeitaufgelöster Fluoreszenztechniken und der Erhöhung der Empfindlichkeit der Detektoren für Einzelmolekülstudien angepasst 33,34,35,36,37. smFA deckt einen breiten Bereich von Zeitskalen ab - von Pikosekunden bis zu mehreren Stunden - und ergänzt die Daten aus Einzelmolekül-Experimenten zum Förster-Resonanz-Energietransfer (smFRET)38.
smFA kann in verschiedenen Formaten visualisiert werden, um wichtige Informationen über die biomolekulare Dynamik zu extrahieren (Abbildung 1B). Zeitaufgelöste Fluoreszenz-Anisotropie-Zerfälle sind eindimensionale Histogramme, die die Dynamik auf der Zeitskala von Pikosekunden bis Nanosekunden erfassen39,40. Zweidimensionale Einzelmolekül-Histogramme, die die Fluoreszenzlebensdauer mit der Anisotropie für einzelne Moleküle korrelieren, können die Heterogenität des Anisotropiezustands aufzeigen und visuelle Einblicke in die potenzielle Dynamik innerhalb der Beobachtungszeit in konfokalen Experimenten (~ms) geben41,42. Für die Untersuchung der Dynamik im Sub-Millisekunden-Bereich kann die dynamische Anisotropie-Photonenverteilungsanalyse (daPDA) verwendet werden, während die zeitaufgelöste Anisotropie-Burst-Varianz-Analyse (traBVA) eine robuste Methode zur Bestätigung spezifischer Dynamiken um Millisekunden43 bietet (Abbildung 1B).
Diese Methoden ergänzen traditionellere Werkzeuge, wie z. B. die polarisationsaufgelöste Fluoreszenzkorrelationsspektroskopie (pFCS), die ein breiteres Spektrum aufweist 44,45,46,47. Insgesamt erleichtern mehrere Datenanalysewerkzeuge für smFA die Identifizierung lokaler und globaler Konformationsänderungen, vorausgesetzt, dass eine ordnungsgemäße Kalibrierung in Betracht gezogen wird.
Hier wenden wir smFA an, um die DNA-Bindung des humanen FoxP1-Transkriptionsfaktors 48,49,50,51 zu untersuchen. Dieses Protein nimmt domänentauschtes Dimer aufgrund der intrinsisch ungeordneten Natur seiner Polypeptidkette an, die in Abhängigkeit vom quartären Zustand des Proteins und dem Vorhandensein von DNA besonders beeinflusst wird. Wir erzeugten verschiedene Single-Cystein-Mutanten zur Markierung mit BODIPY-FL, führten smFA-Experimente durch und setzten daPDA und trBVAa ein. Dieser Ansatz ermöglichte es uns, Dynamik und Heterogenität mit der FoxP1-Dimerisierung und der DNA-Bindung in Verbindung zu bringen, was den komplexen Wirkmechanismus hervorhebt, der diesen Transkriptionsfaktor charakterisiert.
Protokoll
HINWEIS: Die Auswahl des richtigen Fluorophors ist für smFA-Experimente unerlässlich. Biomoleküle können an ortsspezifischen Positionen markiert werden, indem entweder Aminosäuren in Proteinen oder Nukleotidbasen in Nukleinsäuren mit Fluoreszenzmarkern modifiziert werden, abhängig von den verfügbaren reaktiven Gruppen. Unter den organischen Farbstoffen52 sind die Familien Alexa Fluor, Cy, BODIPY und Janelia Farms dank ihrer langen Fluoreszenzlebensdauer, Photostabilität und hohen Quantenausbeute die beliebteste Wahl für smFA. BODIPY-FL wird oft wegen seiner verlängerten Fluoreszenzlebensdauer, seiner überlegenen Quantenausbeute und seines kurzen Verbindungslinkers bevorzugt. Darüber hinaus werden alternative Fluorophore häufig im Wirkstoffscreening eingesetzt, wo Bulk-Techniken bevorzugt werden53. Chimere fluoreszierende Proteine können auch für Anisotropie-Experimente und Bildgebung von lebenden Zellen verwendet werden, obwohl es eine Einschränkung durch einen geringeren Dynamikbereich gibt.
1. Vorbereitung des Puffers
HINWEIS: Tragen Sie bei Laborexperimenten Handschuhe, eine Augenschutzbrille und einen Laborkittel.
- Standardpuffer (20 mM 4-(2-Hydroxyethyl)-1-piperazinethensulfonsäure [HEPES], pH 7,8, 150 mM NaCl): 2,38 g HEPES, 4,38 g NaCl in 400 mL Reinstwasser auflösen, den pH-Wert auf 7,8 einstellen und das Endvolumen auf 500 mL erhöhen.
- Lysepuffer (20 mM HEPES, pH 7,8, 150 mM NaCl, 0,1 mM Phenylmethylsulfonylfluorid [PMSF], 10 μg/ml DNAse): 2,38 g HEPES, 4,38 g NaCl und 8,71 mg PMSF und eine Endkonzentration von 10 μg/ml DNAse in 400 mL Reinstwasser auflösen, den pH-Wert auf 7,8 einstellen und das Endvolumen auf 500 mL erhöhen.
- Äquilibrierungspuffer (50 mM phosphatgepufferte Kochsalzlösung [PBS], 150 mM NaCl und 10 mM Imidazol; pH 7,4): 5,05 g Na2HPO4, 0,85 g NaH2PO4, 4,38 g NaCl und 0,34 g Imidazol in 400 mL Reinstwasser auflösen, den pH-Wert auf 7,4 einstellen und das endgültige Volumen auf 500 ml erhöhen.
- Waschpuffer (50 mM PBS, 150 mM NaCl und 30 mM Imidazol; pH 7,4): 5,05 g Na2HPO4, 0,85 g NaH2PO4, 4,38 g NaCl und 1,02 g Imidazol in 400 mL Reinstwasser auflösen, den pH-Wert auf 7,4 einstellen und das Endvolumen auf 500 mL erhöhen.
- Elutionspuffer (50 mM PBS, 150 mM NaCl und 250 mM Imidazol; pH 7,4): 5,05 g Na2HPO4, 0,85 g NaH2PO4, 4,38 g NaCl und 8,5 g Imidazol in 400 mL Reinstwasser auflösen, den pH-Wert auf 7,4 einstellen und das Endvolumen auf 500 mL erhöhen.
- PBS-Puffer (50 mM Natriumphosphat gepufferte Kochsalzlösung, 150 mM NaCl, pH 7,4). Lösen Sie 5,05 g Na2HPO4, 0,85 g NaH2PO4 und 4,38 g NaCl in 400 mL Reinstwasser, stellen Sie den pH-Wert auf 7,4 ein und stellen Sie das endgültige Volumen auf 500 mL ein.
- Sterilisieren Sie alle Puffer, indem Sie die Lösung mit Filtern mit einer Porengröße von 0,22 μm filtrieren und die Puffer bei Raumtemperatur (RT) lagern.
- Filtrieren Sie den Standardpuffer mit einem Aktivkohlefilter für die Einzelmolekülaufnahme.
2. Fluoreszierende Sonden
- BODIPY-FL: 5 mg Durchstechflasche mit BODIPY-FL durch Zugabe eines Endvolumens von 1,29 mL frisch geöffnetem Dimethylsulfoxid (DMSO) auflösen, um die Endkonzentration von 10 mM BODIPY-FL zu erhalten.
- Rhodamin 110: Lösen Sie 3,67 mg Rhodamin 110 auf, indem Sie ein Endvolumen von 1 ml frisch geöffnetem DMSO hinzufügen, um die endgültige Konzentration von 10 mM Rhodamin 110 zu erhalten.
HINWEIS: Fluoreszierende Sonden müssen vermieden werden, wenn sie Licht ausgesetzt werden. Verwenden Sie immer eine lichtempfindliche Tube (bernsteinfarben) und wickeln Sie sie mit Alufolie ein. Wenn DMSO bereits geöffnet ist, muss es im Exsikkator aufbewahrt werden, um die Haltbarkeit zu erhöhen. - Bereiten Sie kleinvolumige Aliquots der vorbereiteten Fluoreszenzsonde vor und lagern Sie sie bis zur weiteren Verwendung bei -20 °C.
HINWEIS: Vermeiden Sie Gefrier-Tau-Zyklen, um die Etikettiereffizienz zu verbessern.
3. Kalibrierungsmessungen
- Durchführung von Multiparameter-Fluoreszenzdetektionsexperimenten (MFD) auf einem selbst entwickelten polarisationsaufgelösten Aufbau mit 4 Detektionskanälen und 2 gepulsten Lasern (blau 485 nm und rot 640 nm)54. Für Anisotropiemessungen verwenden Sie jedoch den blauen Laser und zwei Detektorkanäle.
- Schalten Sie die Detektorkanäle (parallel und senkrecht) und die blaue Laserleistung ein.
- Stellen Sie sicher, dass die Laserleistung auf 60 μW eingestellt ist, und öffnen Sie das Bedienfeld der Software36 für die zeitkorrelierte Einzelphotonenzählung (TCSPC).
- Mischen Sie 1 μl 100 nM Rhodamin 110 und 49 μl destilliertes Wasser, um eine Endkonzentration von 2 nM Rhodamin 110 zu erreichen. Geben Sie 50 μl 2 nM Rhodamin 110 in die Mitte des Deckglases für Kalibriermessungen.
- Geben Sie als Nächstes einen Tropfen Tauchflüssigkeit (entweder Öl oder Wasser, aber achten Sie darauf, dass sie den gleichen Brechungsindex haben) auf die Objektivlinse des Mikroskops, um das Auflösungsvermögen des Mikroskops zu erhöhen.
- Platzieren Sie das Deckglas auf der Objektivlinse und stellen Sie sicher, dass sich der Wassertropfen in der Mitte des Mikroskopobjektivs befindet.
- Stellen Sie die Bildebene so ein, dass sie sich innerhalb der Lösung und über der Glasoberfläche befindet.
- Stellen Sie den Knopf ein, um den zweiten hellen Fokuspunkt zu finden, und drehen Sie ihn auf eineinhalb Punkte. Fokussieren Sie den Laser auf die Grenzfläche von Glas und Flüssigkeit.
- Maximieren Sie die Anzahl der detektierten Photonen, indem Sie die Position der Lochblende (70 μm) anpassen und gleichzeitig die Photonenzählrate überwachen.
- Öffnen Sie die Software im Bereich für den zeitaufgelösten Modus (TTTR oder T3) und klicken Sie auf die Schaltfläche Start . Zeichnen Sie die Zählrate für 120 s auf und speichern Sie das Dateiformat Rhodamin 110.ptu mit der Erfassungssoftware. Diese Erfassungszeit sollte unter Berücksichtigung der Konzentration (2 nM) ausreichend sein.
- Für Hintergrundmessungen geben Sie 50 μl destilliertes Wasser in die Mitte des Deckglases und wiederholen Sie die Schritte 3.5-3.10. Zeichnen Sie jedoch die Photonenzählrate für 300 s auf, und speichern Sie die Datei water.ptu .
- Für andere Hintergrundmessungen geben Sie 50 μl Standardpuffer in die Mitte des Deckglases und wiederholen Sie die Schritte 3.5-3.10. Zeichnen Sie die Photonenzählrate für 300 s auf und speichern Sie die Standarddatei buffer.ptu . Analysieren Sie dann die Daten mit einer BIFL-Analysesoftware (Burst Integration Fluorescence Lifetime).
4. Kalibrierung und Datenanalyse
- Öffnen Sie die BIFL-Software und klicken Sie im automatischen Fenster auf Einrichtung bestätigen . Klicken Sie anschließend auf Parameter aus Datei abrufen und dann auf OK.
- Damit die Messung analysiert werden soll, klicken Sie auf Datenpfad-Array und wählen Sie es aus .
- Laden Sie als Nächstes die Datei water.ptu water measurement für Arrays wie Green scatter. Wählen Sie auf ähnliche Weise die Standarddatei buffer.ptu für den grünen Hintergrund aus. Wählen Sie für Grün dick die Option Rhodamin 110.ptu aus.
- Klicken Sie unter den Parametern für die Auswahl einzelner Moleküle auf Weiter und dann auf Anpassen, um ein neues Popup-Fenster anzuzeigen. Klicken Sie auf den Schwellenwert, um die Ankunftszeit zwischen den Photonen zu ändern, und wählen Sie die Ereigniszeit eines einzelnen Moleküls für die mittlere Ankunftszeit zwischen den Photonen (dt) aus. Klicken Sie anschließend auf min. #, um die minimale Anzahl von Photonen pro einzelnem Molekülereignis auszuwählen, und schließen Sie dann das Popup-Fenster, indem Sie auf Return klicken. Klicken Sie dann auf OK.
- Klicken Sie anschließend auf Farbanpassungsparameter, um die anfängliche Fluoreszenzlebensdauer für Grün anzupassen, also Farben, die mit Fluoreszenzzerfallsparametern generiert werden. Passen Sie die Eingabeaufforderungs- und Verzögerungswerte an, indem Sie die Werte für "Von" und "Bis" ändern. Schließen Sie dann das Popup-Fenster, indem Sie auf Return klicken. Klicken Sie dann auf OK.
- Klicken Sie auf Speichern, um die ASCII-Dateien zu verarbeiten und in einem ausgewählten Ordner zu speichern. Verarbeiten Sie dann die Daten für den zeitaufgelösten Fluoreszenz-Anisotropie-Zerfall mit ChiSurf22, Photonenverteilungsanalyse 55,56 oder Burst-Varianz-Analyse57,58.
HINWEIS: Beispielhafte Daten und Schritt-für-Schritt-Beschreibungen zur Verwendung von ChiSurf, PDA und BVA finden Sie unter github.com/Fluorescence-Tools/chisurf, github.com/Fluorescence-Tools/tttrlib, www.mpc.hhu.de/en/software/mfd-fcs-and-mfis bzw. github.com/SMB-Lab/feda_tools. Die experimentellen Daten sind bei Zenodo (10.5281/zenodo.13371503) verfügbar.
5. FoxP1 Protein-Zubereitung
- Rekombinante bakterielle Überexpression von FoxP1
- Durchführung der Umwandlung in E. coli C41-Bakterienzellen nach einer an die Schnellwechselstelle gerichteten Polymerase-Kettenreaktion (PCR)-Mutagenese.
- Bereiten Sie das LB-Medium vor und stellen Sie es in den Autoklaven, um es zu sterilisieren.
- Eine einzelne E. coli C41-Kolonie wird vorinokuliert, indem 5 ml LB-Medium mit 5 μl (100 μg/ml) Ampicillin zugegeben werden. Über Nacht bei 37 °C auf einem Inkubatorschüttler inkubieren lassen.
- Am nächsten Tag inokulieren Sie eine großflächige Bakterienkultur, indem Sie über Nacht Prä-Inokulum in 500 ml LB-Medien mit einem zuvor hinzugefügten Antibiotikum im Verhältnis 1:500 geben.
- Überwachen Sie das Wachstum der Kultur, indem Sie die Absorption der Kultur bei 600 nm messen.
- Wenn die optische Dichte zwischen ~0,5 und 0,7 liegt, induzieren Sie die Proteinexpression, indem Sie eine Endkonzentration von 0,5 mM Isopropyl-β-d-1-thiogalactopyranosid (IPTG) hinzufügen und die Kultur über Nacht auf einem Inkubatorschüttler bei 15 °C halten.
- Nach Erreichen einer optischen Dichte (600 nm) im Bereich von 1,4-1,6 werden die Bakterienzellen durch Zentrifugation bei 3000 g für 20 min bei 4 °C geerntet. Entsorgen Sie den Überstand und lagern Sie das Pellet bis zur Verwendung bei -20 °C.
- Rekombinante FoxP1-Aufreinigung
- Lyse von E. coli C41-Zellen durch Zugabe von Lysepuffer unter Verwendung beliebiger Lysemethoden wie Beschallung, Flüssigkeitshomogenisierung, French Press usw.
- Das Lysat wird bei 14000 g 10 min bei 4 °C zentrifugiert.
- FürHis-6-markierte Proteine waschen Sie die Ni2+-NTA-Affinitätssäule und äquilibrieren Sie mit NiSO4 unter Verwendung der schnellen Proteinflüssigkeitschromatographie (FPLC).
- Laden Sie das His 6-markierte FoxP1-Protein in die äquilibrierte Ni2+-NTA-Affinitätsspalte.
- Eluieren Sie das FoxP1-Protein aus der Ni2+-NTA-Säule unter Verwendung eines linearen Gradienten des Elutionspuffers.
- Nachdem das Protein eluiert wurde, führen Sie eine Dialyse für den Pufferaustausch durch. Protein in einen Äquilibrierungspuffer ohne Imidazol geben und über Nacht mit einem Magnetrührer bei 4 °C dialysieren.
HINWEIS: Wenn das Protein mit einer anderen Methode aufgereinigt wird, führen Sie den Pufferaustausch entsprechend durch. - Um denHis-6-Tag zu entfernen, geben Sie der Dialyse über Nacht eine Tabakätzvirus-Protease (TEV-Protease (1:100 Verhältnis von TEV:FoxP1).
HINWEIS: Sein 6-Tag-Aufschluss kann auch durch Zugabe eines TEV-Proteaseaufschlusses auf der Säule durchgeführt werden. Dieser Schritt würde die Ausbeute des interessierenden Proteins verbessern. - Wiederholen Sie am nächsten Tag die Schritte von 5.2.3-5.2.5. Dieses Mal eluieren Sie das Protein jedoch im Waschpuffer und nicht im Elutionspuffer.
- Konzentrieren Sie das Protein auf ein geeignetes Volumen, indem Sie PBS-Puffer hinzufügen, und quantifizieren Sie das Protein durch Absorption bei 280 nm unter Berücksichtigung des Extinktionskoeffizienten des Proteins.
- Rekombinante FoxP1-Beschriftung
- Inkubieren Sie 50-100 μ0 μM FoxP1-Protein mit einem 10-fachen molaren Überschuss an Dithiothreitol (DTT) oder Tris (2-carboxyethyl)phosphin (TCEP) mit einem Endvolumen von 500 μl bei RT für 30 min.
- Führen Sie den Pufferaustausch mit PD 10-Entsalzungssäulen durch. Setzen Sie die PD10-Säule mit einem Säulenadapter in ein 50-ml-Zentrifugenröhrchen ein.
- Äquilibrieren Sie die PD10-Säule durch Zugabe von 5 ml PBS-Puffer und zentrifugieren Sie sie 2 Minuten lang bei 1000 × g . Wiederholen Sie diesen Schritt 3 Mal.
- Bewahren Sie die PD10-Säule in einem frischen 50-ml-Röhrchen auf und geben Sie 2 ml PBS-Puffer in die Säule. Geben Sie dann 500 μl FoxP1-Protein aus Schritt 1 in die Säule. Eluieren Sie das Protein, indem Sie 1000 × g für 2 min zentrifugieren und sammeln Sie das Protein.
- Konzentrieren Sie das Protein, indem Sie es in Ultrazentrifugalfilter (10 kDa MWCO) geben, zentrifugieren Sie 10 Minuten lang bei 7500 × g und sammeln Sie das Eluieren.
- Messen Sie die Proteinkonzentration, fügen Sie BODIPY mit 30 % der Cysteinkonzentration im Protein hinzu und legen Sie es für 2 Stunden bei 4 °C auf einen Rotator.
- Führen Sie einen Pufferaustausch durch und konzentrieren Sie das Protein, wie in den Schritten 5.3.2 bis 5.3.5 beschrieben.
- Überprüfen Sie die Proteinkonzentration mit einem Spektralphotometer oder einem kolorimetrischen Assay, wie z. B. der Bradford- oder Lowry-Methode, und die Konzentration des Farbstoffs durch Absorption bei 500 nm.
- Messen Sie den Grad der Beschriftung mit der folgenden Formel:
(A Farbstoff/εFarbstoff)/(Molekulargewicht des Proteins/Milligramm Protein pro Milliliter) = Mol Farbstoff/Mol Protein
Dabei sind ADye und εDye der Absorptionswert des Farbstoffs und der molare Extinktionskoeffizient des Farbstoffs bei der maximalen Absorptionswellenlänge59. Bei Messung bei 280 nm muss die tatsächliche Absorption des Proteins nach folgender Formel korrigiert werden:
EinProtein = A280-(AFarbstoff· CF280)
Dabei ist CF280 der Korrekturfaktor für den angegebenen Farbstoff unter Berücksichtigung seiner intrinsischen Absorption bei 280 nm59.
HINWEIS: Während wir uns auf die Expression und Reinigung eines Cystein-modifizierten Proteins konzentrieren, muss die strategische Einführung von Cysteinresten in das Protein sorgfältig in Betracht gezogen werden.
6. Vorbereitung der Probenkammer des Mikroskops
- 495 μl Reinstwasser und 5 μl (0,01 % v/v Endkonzentration) Tween-20 (nichtionisches Tensid) in die Vertiefung eines Kammerobjektträgers geben und mischen. Über 30 Minuten bei RT inkubieren.
- Waschen Sie die Kammer zweimal vorsichtig mit Reinstwasser und trocknen Sie sie ab.
HINWEIS: Die Kammer ist bereit für das Experiment.
7. Einzelmolekül-Fluoreszenz-Anisotropie-Experiment
- Kalibrieren Sie zunächst das smFA-Instrument. Bestimmen Sie die Ansprechfunktion des Geräts, entweder durch Messung des Raman-Streulichts oder durch Messung der Fluoreszenz eines Farbstoffs mit kurzer Lebensdauer und entsprechendem Emissionsspektrum (z. B. Erythrosin oder Malachitgrün), der zusätzlich durch eine gesättigte Kaliumiodid-Lösung, KI, abgeschreckt wird.
- Um die Hintergrundfluoreszenz zu korrigieren, führen Sie Messungen mit einer Pufferlösung durch. Kalibrieren Sie die relative Empfindlichkeit der parallelen und senkrechten Detektoren (G-Faktor), indem Sie Daten von einem schnell rotierenden Farbstoff erfassen. Hier wurde 2 nM Rhodamin 110 zur Kalibrierung der Detektoren60 verwendet.
- Für smFA-Experimente mit monomerem FoxP1 geben Sie 100 pM BODIPY-markiertes FoxP1 und 100 nM unmarkiertes FoxP1 zu 500 μl Standardpuffer (20 mM HEPES, 20 mM NaCl, pH 7,8) in einen Kammerobjektträger.
- Für den Titrationsassay mit DNA und monomerem FoxP1 werden 400 nM DNA zu einem 100 pM BODIPY-markierten/100 nM unmarkierten Proteinmix hinzugefügt und 10 Minuten lang bei RT inkubiert.
- Für smFA-Experimente mit dimerem FoxP1 fügen Sie 500 nM unmarkiertes FoxP1 zu einem 100 pM BODIPY-markierten FoxP1 im Standardpuffer hinzu. Anschließend 30 min bei 37 °C inkubieren.
- Für den Titrationsassay mit DNA und dimerem FoxP1 werden 2000 nM DNA zu einem 100 pM BODIPY-markierten/500 nM unmarkierten Proteinmix hinzugefügt und 10 Minuten lang bei RT inkubiert.
- Starten Sie die Messungen und überprüfen Sie in der BIFL-Software, ob die Anzahl der Bursts in einer 30 s-Aufzeichnung zwischen 60 und 90 liegt. Wenn die Probe konzentrierter ist, verdünnen Sie sie, bis sie diesen Wert erreicht. Dieser Schritt stellt Einzelmolekülbedingungen sicher.
- Beginnen Sie mit jeder smFA-Messung für monomeres FoxP1 oder dimeres FoxP1 in Abwesenheit oder Vorhandensein von DNA für mindestens 4 Stunden.
- Analysieren Sie die smFA-Messungen, wie in Abschnitt 4 beschrieben.
Ergebnisse
Die Fluoreszenzanisotropie ergibt sich aus der relativen Ausrichtung der Absorption des Fluorophors und der Emissionsdipolmomente. Wenn Fluorophore polarisiertem Licht ausgesetzt werden, werden Fluorophore mit Absorptionsübergangsmomenten, die auf den elektrischen Feldvektor des einfallenden Lichts ausgerichtet sind, bevorzugt angeregt (Photoselektion). Folglich wird die Population des angeregten Zustands teilweise orientiert, wobei die Übergangsmomente eines signifikanten Teils der angeregten Moleküle mit dem elektrischen Feldvektor des polarisierten aufregenden Lichts61 ausgerichtet sind. Fluorophore rotieren aufgrund ihrer Brownschen Bewegung. Somit dreht sich auch das Emissionsübergangsmoment, was zu einer Zeitabhängigkeit von der Fluoreszenzanisotropie führt. Dieser Effekt kann genutzt werden, um die Rotationsbewegungen fluoreszierender Moleküle zu messen, Bindungsereignisse zu erkennen, die Umgebung des Fluorophors zu charakterisieren und die Molekulardynamik zu erfassen.
Einzelmolekülexperimente sind einzigartig positioniert, um die Heterogenität der Probe zu bestimmen. Durch die Nutzung der Einzelmolekülempfindlichkeit und der Fluoreszenzanisotropie wird die Multiparameter-Fluoreszenzspektroskopie um eine weitere Dimensionalität erweitert. In einem typischen konfokalen Einzelmolekülmikroskop (Abbildung 2)20,21 kann die Fluoreszenzanisotropie durch den Einsatz von gepulsten Lasern intensitätsbasiert oder zeitaufgelöst bestimmt werden.
Um die depolarisierenden Effekte des Objektivs mit hoher numerischer Apertur in einem Konfokalmikroskop62 zu betrachten, ist die richtige Form der zeitaufgelösten Anisotropie35, 63 gegeben durch
 (1)
(1)
wobei 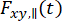 und
und  die zeitaufgelöste Fluoreszenzintensität im y-ten Detektionskanal nach Anregung bei der Wellenlänge X für die parallele und senkrechte Polarisation L1 und L2 sind und Faktoren sind, die die Mischung zwischen den parallelen und senkrechten Signalen aufgrund des bei diesen Messungen verwendeten Objektivs mit hoher numerischer Apertur (NA) beschreiben35,62, 64. Urheberrecht Unterschiede in der Detektionseffizienz des parallelen
die zeitaufgelöste Fluoreszenzintensität im y-ten Detektionskanal nach Anregung bei der Wellenlänge X für die parallele und senkrechte Polarisation L1 und L2 sind und Faktoren sind, die die Mischung zwischen den parallelen und senkrechten Signalen aufgrund des bei diesen Messungen verwendeten Objektivs mit hoher numerischer Apertur (NA) beschreiben35,62, 64. Urheberrecht Unterschiede in der Detektionseffizienz des parallelen 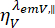 und senkrechten Detektionskanals
und senkrechten Detektionskanals 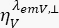 für den Farbstoff werden mit dem Verhältnis der Detektionseffizienzen korrigiert.
für den Farbstoff werden mit dem Verhältnis der Detektionseffizienzen korrigiert. 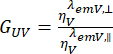 Der G-UV wird auch als G-Faktor bezeichnet.
Der G-UV wird auch als G-Faktor bezeichnet.
Die zeitaufgelöste Fluoreszenzanisotropie kann mit Hilfe eines multiexponentiellen Zerfalls modelliert werden, um zu erklären, dass das Fluorophor an ein größeres Biomolekül gebunden ist
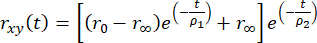 , (2)
, (2)
wobei R0 die Fluorophor-abhängige fundamentale Anisotropie ist (typischerweise R0 = 0,38), R∞ die Restanisotropie ist und ρ1 und ρ2 schnelle (lokale Bewegungen des Fluorophors) bzw. langsame (globale Bewegung des Makromoleküls) Rotationskorrelationszeiten sind.
Bei Einzelmolekül-Anisotropiemessungen (Abbildung 2) werden Photonenankunftszeiten aufgezeichnet, um einzelne Emitter mit Hilfe der burst-integrierten Fluoreszenzlebensdaueranalyse (BIFL) zu identifizieren33,35. Die Ankunftszeiten zwischen den Photonen (Δt) werden anhand eines gleitenden Durchschnitts geglättet und dann zur besseren Visualisierung grafisch dargestellt. Das Histogramm dieser Zeiten ist mit einem halben Gauß versehen, um den Mittelwert und die Standardabweichung der Photonen zu bestimmen, die aus dem Hintergrund stammen. Ein willkürlicher Schwellenwert, der auf ein Vielfaches der Standardabweichung festgelegt ist, wird verwendet, um einzelne Ereignisse herauszufiltern und gleichzeitig das erste und letzte Photon in jedem Ausbruch zu identifizieren. Photonen in jedem Burst werden dann für die weitere Analyse integriert, die die Berechnung der zeitaufgelösten und intensitätsbasierten Fluoreszenzanisotropie im stationären Zustand unter Verwendung der Gleichungen 1 und 2 oder über einen Maximum-Likelihood-Schätzer35 umfasst. Aufgrund der begrenzten Anzahl von Photonen in Einzelmolekülereignissen berücksichtigt der Maximum-Likelihood-Schätzer nur eine einzige exponentielle Komponente und wird nicht weiter diskutiert.
In einem zweidimensionalen Histogramm von Einzelmolekülereignissen können die mittlere Fluoreszenzlebensdauer (τ) und die Anisotropie (rxy) durch die Perrin-Gleichung29,61 in Beziehung gesetzt werden, um (ρ) als mittlere Rotationszeit zu erhalten.
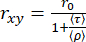 (3)
(3)
Spezifische ρ-Werte können mit höherer Sicherheit durch eine "Sub-Ensemble"-Analyse (se) erhalten werden, bei der die Photonen verschiedener Bursts in einen kombinierten zeitaufgelösten Fluoreszenz-Anisotropie-Zerfall integriert werden, der durch Optimierung der Parameter von Gleichung 2 zum experimentellen Zerfall (seTRFA) analysiert werden kann. Zeitaufgelöste Anisotropie kann die Heterogenität und Dynamik auflösen, die mit Rotationsbewegungen (lokal und global) der Biomoleküle innerhalb der Fluoreszenzemission verbunden sind, die innerhalb des ns-Zeitrahmens auftritt.
Um die Dynamik innerhalb einzelner Molekülereignisse (auf der Submillisekundenskala) zu detektieren, haben wir die zeitaufgelöste Anisotropie-Burst-Varianz-Analyse (traBVA)57 eingeführt. In traBVA beträgt für einen Photonenausbruch, der Mi aufeinanderfolgende Photonensegmente enthält, die überschüssige Anisotropievarianz (s2) für Ausbrüche
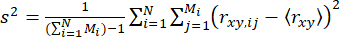 . (4)
. (4)
Für einen einzelnen anisotropen Zustand ergibt sich die Varianz σ2 ausschließlich aus dem Schrotrauschen65 (sn: √N, wobei N die Anzahl der Photonen ist)
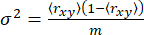 (5)
(5)
Dabei ist m die Anzahl der Photonen in einem Ausbruch. Um eine zusätzliche Varianz in der Anisotropie zu identifizieren, können wir daher die überschüssige Anisotropievarianz (S2) aufgrund der Konformationsheterogenität als Differenz zwischen den Gleichungen 4 und 5 definieren.
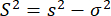 (6)
(6)
Um die Dynamik, die bei der Beobachtung einzelner Moleküle auftritt, zu erfassen und die Varianzapproximation zu berücksichtigen, kann die dynamische Anisotropie-Photonenverteilungsanalyse (daPDA)55,56 verwendet werden. In der daPDA wird die Fluoreszenzintensität modelliert, indem einer bedingten Wahrscheinlichkeit (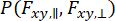 ) gefolgt wird, die als Binomialverteilung ausgedrückt wird.
) gefolgt wird, die als Binomialverteilung ausgedrückt wird.
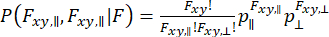 (7)
(7)
Zusammen mit einer Schätzung der Hintergrundzählrate, die einer Poisson-Verteilung folgt
 (8)
(8)
wobei 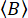 die durchschnittliche Anzahl der Hintergrundphotonen pro eingestelltem Zeitfenster ist. Die parallelen und senkrechten Hintergrundzählungen
die durchschnittliche Anzahl der Hintergrundphotonen pro eingestelltem Zeitfenster ist. Die parallelen und senkrechten Hintergrundzählungen 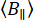 und
und 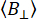 können anhand von Pufferproben als Referenz gemessen werden. Die experimentell ermittelte Fluoreszenzanisotropie wird durch Minimierung einer Gütezahl χ2 mit einer Fluoreszenzintensitätsverteilung pro Polarisationskanal optimiert, die kinetische Änderungen einschließen kann.
können anhand von Pufferproben als Referenz gemessen werden. Die experimentell ermittelte Fluoreszenzanisotropie wird durch Minimierung einer Gütezahl χ2 mit einer Fluoreszenzintensitätsverteilung pro Polarisationskanal optimiert, die kinetische Änderungen einschließen kann.
Die bereitgestellten Analyseroutinen und Datenrepräsentationen bieten einen umfassenden Ansatz zur Interpretation der gesammelten Daten. Obwohl sich dieses Protokoll in erster Linie auf konfokale Messungen konzentriert, die bei der Erfassung von Anisotropieänderungen von Nanosekunden bis Millisekunden begrenzt sind, ist es möglich, ein Totalreflexionsmikroskop einzusetzen, um die Fluoreszenzanisotropie über längere Zeitskalen zu überwachen, was eine Zeitreihenanalyse ermöglicht66. Für konfokale Einzelmolekülmessungen heben wir die Verwendung von mehrdimensionalen Histogrammen hervor, die einen einzigartigen Fingerabdruck des beobachteten Ensembles erstellen. Zeitaufgelöste Fluoreszenzzerfälle, die aus ausgewählten Populationen rekonstruiert werden, können die Entwicklung der Fluoreszenzanisotropie auf der Nanosekundenskala verfolgen (Abbildung 3). Die Photonenverteilungsanalyse55,56 und die Burst-Varianz-Analyse (BVA)57,58 können auch die Dynamik auf mittleren Zeitskalen zwischen zeitaufgelösten Zerfällen und mehrdimensionalen Histogrammen erfassen. Obwohl dieses Protokoll die Verwendung der Polarisationsfluoreszenz-Korrelationsspektroskopie (FCS) mit oder ohne gepulste Anregung67,68, die die Zeitskalen von Nanosekunden bis Millisekunden überbrücken kann, nicht abdeckt, können dieselben Daten zur Berechnung von FCS69 verwendet werden, obwohl dies außerhalb des Rahmens des vorgestellten Protokolls liegt. Wenn solche Versuche durchgeführt werden, wird eine längere Zeit für die Probenmessung empfohlen.
Dieser Ansatz wurde auf ein komplexes System wie die menschlichen FoxP-Proteine angewendet und liefert wertvolle Einblicke in die Bewegungen, die an ihrem Wirkmechanismus beteiligt sind. FoxP-Proteine sind Transkriptionsfaktoren, die an verschiedenen physiologischen Aspekten wie der Entwicklung von Gehirn und Lunge beteiligt sind. Wichtig ist, dass verschiedene Mutationen erkannt wurden, die die Funktion dieser Proteine beeinträchtigen 70,71. Unter Verwendung der DNA-Bindungsdomäne von FoxP1 als Modell erzeugten wir verschiedene Einzelcystein-Mutanten, um einen BODIPY-FL-Farbstoff als Tracker für Bewegungen einzuführen (Abbildung 4A). Tatsächlich haben wir den Effekt der Dimerisierung und der DNA-Bindung als wichtige strukturelle Regulatoren dieses Proteins untersucht. Mit dem smFA-Ansatz generierten wir 2D-smFA-Plots und stellten traBVA und daPDA für jede Mutante unter monomeren und dimeren Bedingungen her. Wir zeigen ein Beispiel für eine der untersuchten Einzelmutanten (Abbildung 4). Das Anisotropieverhalten ist bei allen Mutanten ähnlich, was die Bestimmung von hohen und niedrigen Rotationskorrelationszeiten und damit von vermuteten, ungeordneten und gefalteten Ensembles betrifft. Dennoch ist es in allen Mutanten sehr heterogen in Bezug auf die Fraktion und Kinetik jedes Ensembles, was unterschiedliche Änderungen des Übergangs von Ordnung zu Unordnung zeigt, die durch die Dimerisierung und die DNA-Bindung beeinflusst werden, und zeigt die Beschreibung der strukturellen Dynamik entlang der Kette mit hoher Auflösung (Abbildung 5).
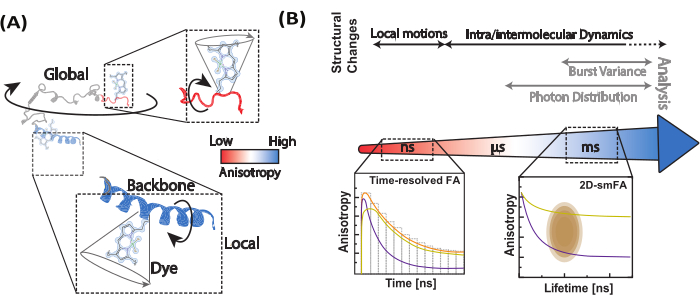
Abbildung 1: Dynamischer Bereich von Biomolekülen und Fluoreszenzanisotropiemethoden. (A) Die Anisotropie kleiner Fluorophore, die an verschiedenen Positionen entlang des Rückgrats des interessierenden Biomoleküls gebunden sind, informiert über die lokale Strukturdynamik. (B) Zeitskalen, die durch Fluoreszenzintensitätszerfälle (zeitaufgelöste Fluoreszenzanisotropie, FA) und Einzelmolekül-Histogramme von konfokalen Einzelmolekülmikroskopdaten untersucht werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
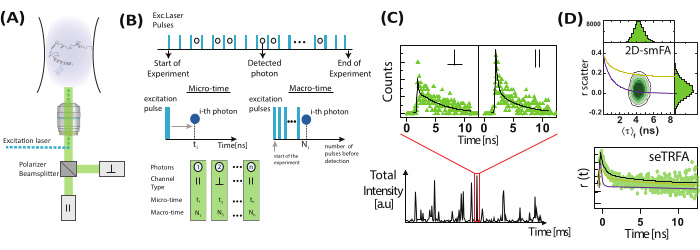
Abbildung 2: Registrierung und Verarbeitung von Einzelmolekül-Fluoreszenz-Anisotropie-Daten. (A) Frei diffundierende Moleküle werden mit einem konfokalen Einzelmolekülmikroskop analysiert, das mit einem einzelnen linear polarisierten Anregungslaser (in unserem Fall blau) ausgestattet ist. Die Fluoreszenzemission (in unserem Fall grün) wird von zwei Detektoren detektiert, nachdem ein Strahlpolarisator das Signal in zwei Polarisationen (parallel  und senkrecht
und senkrecht  zur Anregungsquelle) aufgeteilt hat. (B) Jedes detektierte Photon wird durch drei Parameter charakterisiert: Mikrozeit, Makrozeit und Kanaltyp. Die Daten werden im TTTR-Format (Time-Tagged Time-Resolved)72 gespeichert. (C) Bursts einzelner Moleküle werden ausgewählt und verarbeitet, um Fluoreszenzparameter, einschließlich der Fluoreszenzanisotropie, für jedes beobachtete Molekül zu extrahieren. (D) Die Daten werden auf verschiedene Weise dargestellt, einschließlich zweidimensionaler Diagramme der Fluoreszenzanisotropie im Vergleich zur Fluoreszenzlebensdauer und zeitaufgelöster Anisotropiezerfälle. Diese Darstellungen ermöglichen sowohl die visuelle als auch die quantitative Bestimmung der Fluoreszenzlebensdauer, der Rotationskorrelationszeiten und der Systemheterogenität. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
zur Anregungsquelle) aufgeteilt hat. (B) Jedes detektierte Photon wird durch drei Parameter charakterisiert: Mikrozeit, Makrozeit und Kanaltyp. Die Daten werden im TTTR-Format (Time-Tagged Time-Resolved)72 gespeichert. (C) Bursts einzelner Moleküle werden ausgewählt und verarbeitet, um Fluoreszenzparameter, einschließlich der Fluoreszenzanisotropie, für jedes beobachtete Molekül zu extrahieren. (D) Die Daten werden auf verschiedene Weise dargestellt, einschließlich zweidimensionaler Diagramme der Fluoreszenzanisotropie im Vergleich zur Fluoreszenzlebensdauer und zeitaufgelöster Anisotropiezerfälle. Diese Darstellungen ermöglichen sowohl die visuelle als auch die quantitative Bestimmung der Fluoreszenzlebensdauer, der Rotationskorrelationszeiten und der Systemheterogenität. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
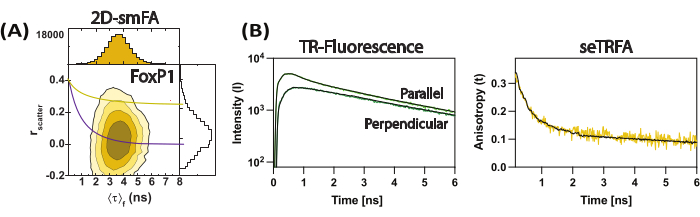
Abbildung 3: Repräsentative Daten für das FoxP1-Domain-Swapped-Dimer. (A) Korrelation der Fluoreszenzanisotropie (r-Streuung) mit der mittleren Fluoreszenzlebensdauer pro Molekül als Konturdiagramm. Überlagerung einer einzelnen Perrin-Gleichung für zwei Rotationskomponenten als Repräsentant des Ensemble-Mittelwerts des Moleküls unter Berücksichtigung von ρ1 und ρ2 von 0,2 ns bzw. 8,5 ns. (B) Zeitaufgelöste Fluoreszenzzerfälle unter dem Ensemble werden verwendet, um die zeitaufgelöste Fluoreszenzanisotropie der Probe zu berechnen. Die Anpassung an Gleichung 2 löste die lokalen und globalen Komponenten der Fluoreszenzanisotropie auf. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
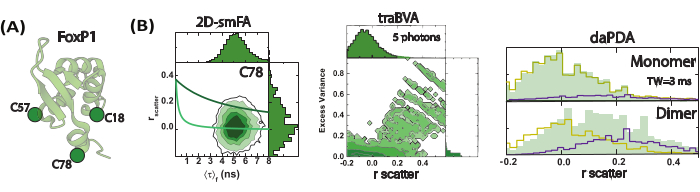
Abbildung 4: FoxP1-Dynamik im Sub-Millisekunden-Bereich, überwacht mit Hilfe der Einzelmolekül-Fluoreszenzanisotropie (smFA). (A) Eine Cartoon-Darstellung der monomeren FoxP1-Struktur. (B) Ein zweidimensionales Histogramm veranschaulicht die dynamische Heterogenität und zeigt zwei unterschiedliche Rotationskorrelationszeiten, die durch zeitaufgelöste Fluoreszenzanisotropie identifiziert wurden. Die zeitaufgelöste Anisotropie-Burst-Varianz-Analyse (traBVA) deckt eine kleine Teilmenge von Ereignissen mit übermäßiger Varianz (Gl. 6) auf, die eine große Anisotropie aufweisen. Die quantitative dynamische Anisotropieanalyse mit Hilfe der Photonenverteilungsanalyse (PDA) extrahiert die Austauschraten für diesen Prozess weiter. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
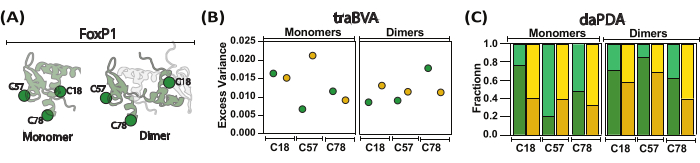
Abbildung 5: Screening der lokalen und globalen Bewegungen von FoxP1 während der Dimerisierung. (A) Eine Cartoon-Darstellung vergleicht das monomere FoxP1 mit seiner dimeren Form. (B) Die mittlere überschüssige Varianz pro Standort unter monomeren und dimeren Bedingungen wird gezeigt, wobei eine größere übermäßige Varianz auf signifikantere Änderungen der Anisotropie hinweist. (C) Die dynamische Anisotropieanalyse mit Hilfe der Photonenverteilungsanalyse (PDA) hilft bei der Bestimmung von Populationsfraktionen (hohe Anisotropie in dunklen Farben und niedrige Anisotropie in heller Farbe) in Abwesenheit (grün) und Vorhandensein (gelb) von DNA. In diesem Ansatz wurden Raten (nicht gezeigt) für Übergänge zwischen lokalem und globalem Verhalten geschätzt, was zeigt, dass FoxP1 eine partielle Entfaltung durchläuft. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Für Einzelmolekül-Fluoreszenz-Anisotropie-Experimente ist es entscheidend, die photophysikalischen Eigenschaften des gewählten Fluorophors sorgfältig zu berücksichtigen. Zu diesen Eigenschaften gehören die Emissionswellenlänge, die mit dem Detektionssystem übereinstimmen muss, und die Anregungswellenlänge, die mit den verfügbaren gepulsten Lasern kompatibel sein sollte. Um den Dynamikbereich zu optimieren, sollte das Fluorophor eine lange Fluoreszenzlebensdauer relativ zur Rotationsdiffusionszeit des Moleküls aufweisen. Dies ist entscheidend für die Verfolgung der Rotationsdynamik und der Kopplung/Ausrichtung des Dipols des Fluorophors relativ zum interessierenden Biomolekül. Darüber hinaus sind Helligkeit, Photostabilität und Quantenausbeute unerlässlich, um starke Signale mit einem stabilen Signal-Rausch-Verhältnis zu erzeugen. Aus diesen Gründen wurde BODIPY-FL in mehreren Studien als Fluorophor gewählt 39,40,42.
Das Screening der Rückgratdynamik von Biomolekülen erfordert häufig eine Proteinmarkierung, die in der Regel durch ortsspezifische Markierung erreicht wird. Dies geschieht in der Regel durch das Einbringen eines Rückstands zur gezielten chemischen Modifikation. Der gebräuchlichste Ansatz ist die Einführung von Cysteinen an interessanten Positionen, wo ihre Thiolseitenketten selektiv mit Reagenzien wie Maleimiden oder Jodacetamiden modifiziert werden können. Seltener werden benzylische Halogenide und Brommethylketone verwendet, um Thioetherbindungen zu bilden. Andere Aminosäure-Seitenketten können ebenfalls ins Visier genommen werden, aber ihre Häufigkeit in Proteinen wird weniger häufig genutzt. Es können aber auch alternative Ansätze, wie z.B. unnatürliche Aminosäuren, verwendet werden73. Die richtige Auswahl der Stelle für die Markierung ist entscheidend, um Interferenzen mit dem untersuchten Biomolekül zu minimieren, und es müssen geeignete Kontrollen vorhanden sein. Wenn das markierte Molekül beispielsweise in Bindungsassays verwendet wird, sollten komplementäre markierungsfreie Methoden sicherstellen, dass die Fluorophore die Bindungsaffinität nicht beeinflussen.
Nach der Identifizierung der geeigneten Probe und der Implementierung der optimalen Markierungsstrategie besteht der nächste Schritt darin, sicherzustellen, dass das konfokale Mikroskop für Einzelmolekülexperimente richtig ausgerichtet und kalibriert ist. Das Protokoll beschreibt, wie der erforderliche Faktor für die weitere Analyse bestimmt wird. Sobald das Instrument kalibriert ist, besteht der nächste Schritt darin, die Probe zu vermessen und die Daten zu verarbeiten, um so viele Informationen wie möglich aus den detektierten Photonen zu extrahieren. Die Schlüsselparameter wie Mikrozeit, Makrozeit und Kanaltyp, wie in Abbildung 2 gezeigt, können für die weitere Analyse und Visualisierung mit typischer TCSPC-Elektronik verwendet werden.
Jüngste Fortschritte in der Einzelmolekül-Fluoreszenzspektroskopie können umfassend genutzt werden, um strukturelle Informationen aus den heterogenen Ensembles von Biomolekülen zu untersuchen. Relativ wenige Studien nutzen jedoch die Erkenntnisse der Fluoreszenzanisotropie, und ein vollständiges Proteinmodell ist erforderlich, um die strukturelle Dynamik von Biomolekülen abzuleiten. Daher ist es eine Herausforderung, die Dynamik der Interdomänen- und Protein-Protein-Wechselwirkungen verschiedener Transkriptionsfaktoren zu entschlüsseln.
Zusammenfassend lässt sich sagen, dass Einzelmolekül-Fluoreszenz-Anisotropie-Experimente komplementäre Informationen über die lokalen und globalen Bewegungen des biomolekularen Rückgrats liefern, die für das Verständnis seiner Funktion entscheidend sind.
Offenlegungen
Alle Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen mit den Inhalten dieses Artikels haben.
Danksagungen
Diese Arbeit wurde durch FONDECYT-Zuschüsse 11200729 und FONDEQUIP-EQM200202 an E.M., NIH R15CA280699 R01GM151334 und NSF CAREER MCB 1749778 Auszeichnungen an HS unterstützt. NK bedankte sich für die Unterstützung durch das Postdoc-Stipendienprogramm der Clemson University.
Materialien
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 488 | ThermoFisher Scienctific | A20100 | https://www.thermofisher.com/order/catalog/product/A20100?SID=srch-srp-A20100 |
| Amicon Ultra Centrifugal Filter, 10 kDa MWCO | Millipore Sigma | UFC901008 | https://www.sigmaaldrich.com/US/en/product/mm/ufc9010 |
| Ampicillin sodium salt | Millipore Sigma | A0166-5G | https://www.sigmaaldrich.com/US/en/product/sigma/a0166 |
| BODIP FL Maleimide (BODIPY FL N-(2-Aminoethyl))Maleimide) | ThermoFisher Scienctific | B10250 | https://www.thermofisher.com/order/catalog/product/B10250?SID=srch-srp-B10250 |
| Disposable PD 10 Desalting Columns | Millipore Sigma | GE17-0851-01 | https://www.sigmaaldrich.com/US/en/product/sigma/ge17085101 |
| Dithiothreitol | Millipore Sigma | 10197777001 | https://www.sigmaaldrich.com/US/en/product/roche/dttro |
| DMSO, Anhydrous | ThermoFisher Scienctific | D12345 | https://www.thermofisher.com/order/catalog/product/D12345?SID=srch-srp-D12345 |
| DNAse | Millipore Sigma | 10104159001 | https://www.sigmaaldrich.com/US/en/product/roche/10104159001 |
| E. coli C41 bacterial cells | Invitrogen | ||
| Foresigh Nuvi Ni-Charged IMAC, 5 mL column | Bio-Rad | 12004037 | https://www.bio-rad.com/en-us/sku/12004037-foresight-nuvia-ni-charged-imac-5-ml-column?ID=12004037 |
| HEPES | Millipore Sigma | 7365-45-9 | https://www.sigmaaldrich.com/US/en/product/sigma/h3375 |
| Imidazole | Millipore Sigma | 288-32-4 | https://www.sigmaaldrich.com/US/en/product/sigma/i5513 |
| IPTG | Millipore Sigma | I6758-1G | https://www.sigmaaldrich.com/US/en/product/sial/i6758 |
| MCE Membrane Filter, 0.22 μm Pore Size | Millipore Sigma | GSWP02500 | https://www.sigmaaldrich.com/US/en/product/mm/gswp02500 |
| NaCl | Millipore Sigma | 7647-14-5 | https://www.sigmaaldrich.com/US/en/product/sigma/s3014 |
| Nunc Lab-Tek II Chamber Slide System | ThermoFisher Scienctific | 154534 | https://www.thermofisher.com/order/catalog/product/154534 |
| OverExpress C41(DE3) Chemically Competent Cells | Millipore Sigma | CMC0017-20X40UL | https://www.sigmaaldrich.com/US/en/product/sigma/cmc0017 |
| PMSF | Millipore Sigma | 329-98-6 | https://www.sigmaaldrich.com/US/en/product/sigma/78830 |
| Rhodamine 110 | ThermoFisher Scienctific | 419075000 | https://www.thermofisher.com/order/catalog/product/419075000?SID=srch-hj-419075000 |
| Sodium phosphate dibasic | Millipore Sigma | 7558-79-4 | https://www.sigmaaldrich.com/US/en/product/sigma/s3264 |
| Sodium phosphate monobasic dihydrate | Millipore Sigma | 13472-35-0 | https://www.sigmaaldrich.com/US/en/product/sigma/71505 |
| TCEP, Hydrochloride, Reagent Grade | Millipore Sigma | 580567-5GM | https://www.sigmaaldrich.com/US/en/product/mm/580567 |
| Tween 20 | Millipore Sigma | 11332465001 | https://www.sigmaaldrich.com/US/en/product/roche/11332465001 |
Referenzen
- Cooper, A. Thermodynamic fluctuations in protein molecules. Proc Natl Acad Sci U S A. 73 (8), 2740-2741 (1976).
- Volozhin, S. I. Inguinal hernia in cryptorchism. Khirurgiia (Mosk). 450 (7), 69-71 (1975).
- Henzler-Wildman, K. A., et al. Intrinsic motions along an enzymatic reaction trajectory. Nature. 450 (7171), 838-844 (2007).
- Karplus, M., Mccammon, J. A. The internal dynamics of globular-proteins. CRC Crit Rev Biochem. 9 (4), 293-349 (1981).
- Ishima, R., Torchia, D. A. Protein dynamics from nmr. Nat Struct Biol. 7 (9), 740-743 (2000).
- Fernandez, F. J., Querol-Garcia, J., Navas-Yuste, S., Martino, F., Vega, M. C. X-ray crystallography for macromolecular complexes. Adv Exp Med Biol. 3234, 125-140 (2024).
- Bai, X. C., Mcmullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends Biochem Sci. 40 (1), 49-57 (2015).
- Wright, P. E., Dyson, H. J. Intrinsically unstructured proteins: Re-assessing the protein structure-function paradigm. J Mol Biol. 293 (2), 321-331 (1999).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Intrinsically unstructured proteins and their functions. Nat Rev Mol Cell Biol. 6 (3), 197-208 (2005).
- Csermely, P., Palotai, R., Nussinov, R. Induced fit, conformational selection and independent dynamic segments: An extended view of binding events. Trends Biochem Sci. 35 (10), 539-546 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Drenth, J. Principles of Protein X-Ray Crystallography. , Springer, Verlag. New York, NY. (2007).
- Loeb, H. Systematic diagnosis of chronic diarrhea in infants and small children. Kinderarztl Prax. 44 (1), 36-41 (1976).
- Ellaway, J. I. J., et al. Identifying protein conformational states in the protein data bank: Toward unlocking the potential of integrative dynamics studies. Struct Dyn. 11 (3), 034701(2024).
- Berjanskii, M. V., Wishart, D. S. A simple method to predict protein flexibility using secondary chemical shifts. J Am Chem Soc. 127 (43), 14970-14971 (2005).
- Puthenveetil, R., Vinogradova, O. Solution NMR: A powerful tool for structural and functional studies of membrane proteins in reconstituted environments. J Biol Chem. 294 (44), 15914-15931 (2019).
- Wozniak, A. K., Schroder, G. F., Grubmuller, H., Seidel, C. A., Oesterhelt, F. Single-molecule fret measures bends and kinks in DNA. Proc Natl Acad Sci U S A. 105 (47), 18337-18342 (2008).
- Weidtkamp-Peters, S., et al. Multiparameter fluorescence image spectroscopy to study molecular interactions. Photochem Photobiol Sci. 8 (4), 470-480 (2009).
- Sisamakis, E., Valeri, A., Kalinin, S., Rothwell, P. J., Seidel, C. A. M. Accurate single-molecule FRET studies using multiparameter fluorescence detection. Methods Enzymol. 475, 455-514 (2010).
- Kalinin, S., et al. A toolkit and benchmark study for fret-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1225 (2012).
- Peulen, T. O., Opanasyuk, O., Seidel, C. aM. Combining graphical and analytical methods with molecular simulations to analyze time-resolved fret measurements of labeled macromolecules accurately. J Phys Chem B. 121 (35), 8211-8241 (2017).
- Hamilton, G. L., Sanabria, H. Multiparameter Fluorescence Spectroscopy of Single Molecules. Spectroscopy and Dynamics of Single Molecules. , Elsevier. Cambridge, MA. (2019).
- Kolimi, N., et al. Out-of-equilibrium biophysical chemistry: The case for multidimensional, integrated single-molecule approaches. J Phys Chem B. 125 (37), 10404-10418 (2021).
- Felekyan, S., Sanabria, H., Kalinin, S., Kuhnemuth, R., Seidel, C. A. Analyzing forster resonance energy transfer with fluctuation algorithms. Methods Enzymol. 519, 39-85 (2013).
- Medina, E. D. R. L., Sanabria, H. Unraveling protein's structural dynamics: From configurational dynamics to ensemble switching guides functional mesoscale assemblies. Curr Opin Struct Biol. 66, 129-138 (2021).
- Aznauryan, M., et al. Comprehensive structural and dynamical view of an unfolded protein from the combination of single-molecule FRET, NMR, and SAXS. Proc Natl Acad Sci U S A. 113 (37), E5389-E5398 (2016).
- Gomes, G. W., et al. Conformational ensembles of an intrinsically disordered protein consistent with NMR, SAXS, and single-molecule fret. J Am Chem Soc. 142 (37), 15697-15710 (2020).
- Perrin, F. Polarisation de la lumière de fluorescence. Vie moyenne des molécules dans l'etat excité. J phys radium. 7 (12), 390-401 (1926).
- Weber, G. Polarization of the fluorescence of macromolecules. I. Theory and experimental method. Biochem J. 51 (2), 145-155 (1952).
- Weber, G. Polarization of the fluorescence of macromolecules. II. Fluorescent conjugates of ovalbumin and bovine serum albumin. Biochem J. 51 (2), 155-167 (1952).
- Jameson, D. M., Ross, J. A. Fluorescence polarization/anisotropy in diagnostics and imaging. Chem Rev. 110 (5), 2685-2708 (2010).
- Eggeling, C., Fries, J. R., Brand, L., Gunther, R., Seidel, C. A. Monitoring conformational dynamics of a single molecule by selective fluorescence spectroscopy. Proc Natl Acad Sci U S A. 95 (4), 1556-1561 (1998).
- Fries, J. R., Brand, L., Eggeling, C., Köllner, M., Seidel, C. aM. Quantitative identification of different single molecules by selective time-resolved confocal fluorescence spectroscopy. Journal of Physical Chemistry A. 102 (33), 6601-6613 (1998).
- Schaffer, J., et al. Identification of single molecules in aqueous solution by time-resolved fluorescence anisotropy. Journal of Physical Chemistry A. 103 (3), 331-336 (1999).
- Widengren, J., et al. Single-molecule detection and identification of multiple species by multiparameter fluorescence detection. Anal Chem. 78 (6), 2039-2050 (2006).
- Gradinaru, C. C., Marushchak, D. O., Samim, M., Krull, U. J. Fluorescence anisotropy: From single molecules to live cells. Analyst. 135 (3), 452-459 (2010).
- Mazal, H., Haran, G. Single-molecule fret methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Tsytlonok, M., et al. Specific conformational dynamics and expansion underpin a multi-step mechanism for specific binding of p27 with cdk2/cyclin a. J Mol Biol. 432 (9), 2998-3017 (2020).
- Tsytlonok, M., et al. Dynamic anticipation by cdk2/cyclin a-bound p27 mediates signal integration in cell cycle regulation. Nat Commun. 10 (1), 1676(2019).
- Cruz, P., et al. Domain tethering impacts dimerization and DNA-mediated allostery in the human transcription factor foxp1. J Chem Phys. 158 (19), (2023).
- Kolimi, N., et al. DNA controls the dimerization of the human foxp1 forkhead domain. Cell Rep Phys Sci. 5 (3), 101854(2024).
- Yanez Orozco, I. S., et al. Identifying weak interdomain interactions that stabilize the supertertiary structure of the n-terminal tandem PDZ domains of PSD-95. Nat Commun. 9 (1), 3724(2018).
- Elson, E. L., Magde, D. Fluorescence correlation spectroscopy. I. Conceptual basis and theory. Biopolymers. 13 (1), 1-27 (1974).
- Rigler, R., Elson, E. S. Fluorescence Correlation Spectroscopy: Theory and Applications. , Springer. Berlin, Heidelberg. (2012).
- Aragon, S. R., Pecora, R. Fluorescence correlation spectroscopy as a probe of molecular-dynamics. J Chem Phys. 64 (4), 1791-1803 (1976).
- Mockel, C., et al. Integrated NMR, fluorescence, and molecular dynamics benchmark study of protein mechanics and hydrodynamics. J Phys Chem B. 123 (7), 1453-1480 (2019).
- Shu, W., Yang, H., Zhang, L., Lu, M. M., Morrisey, E. E. Characterization of a new subfamily of winged-helix/forkhead (Fox) genes that are expressed in the lung and act as transcriptional repressors. J Biol Chem. 276 (29), 27488-27497 (2001).
- Co, M., Anderson, A. G., Konopka, G. Foxp transcription factors in vertebrate brain development, function, and disorders. Wiley Interdiscip Rev Dev Biol. 9 (5), e375(2020).
- Takahashi, H., Takahashi, K., Liu, F. -C. FOXP Genes, Neural Development, Speech and Language Disorders. Forkhead Transcription Factors: Vital Elements in Biology and Medicine. , Springer. New York, NY. (2009).
- Wang, B., Lin, D., Li, C., Tucker, P. Multiple domains define the expression and regulatory properties of foxp1 forkhead transcriptional repressors. J Biol Chem. 278 (27), 24259-24268 (2003).
- Leake, M. C., Quinn, S. D. A guide to small fluorescent probes for single-molecule biophysics. Chem Phys Rev. 4 (1), 011302(2023).
- Zhang, H., Wu, Q., Berezin, M. Y. Fluorescence anisotropy (polarization): From drug screening to precision medicine. Expert Opin Drug Discov. 10 (11), 1145-1161 (2015).
- Ma, J., et al. High precision fret at single-molecule level for biomolecule structure determination. J Vis Exp. (123), e55623(2017).
- Antonik, M., Felekyan, S., Gaiduk, A., Seidel, C. A. Separating structural heterogeneities from stochastic variations in fluorescence resonance energy transfer distributions via photon distribution analysis. J Phys Chem B. 110 (13), 6970-6978 (2006).
- Mcgeer, P. L., Mcgeer, E. G. Enzymes associated with the metabolism of catecholamines, acetylcholine and gaba in human controls and patients with parkinson's disease and huntington's chorea. J Neurochem. 26 (1), 65-76 (1976).
- Terterov, I., Nettels, D., Makarov, D. E., Hofmann, H. Time-resolved burst variance analysis. Biophys Rep (N Y). 3 (3), 100116(2023).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule fret experiments with burst variance analysis. Biophys J. 100 (6), 1568-1577 (2011).
- Haugland, R. P. The Handbook: A Guide to Fluorescent Probes and Labeling Technologies. Molecular Probes. , (2005).
- Medina, E., et al. Intrinsically disordered regions of the DNA-binding domain of human foxp1 facilitate domain swapping. J Mol Biol. 432 (19), 5411-5429 (2020).
- Lakowicz, J. R. Principles of Fluorescence Spectroscopy. , Springer. New York, NY. (2007).
- Koshioka, M., Sasaki, K., Masuhara, H. Time-dependent fluorescence depolarization analysis in three-dimensional microspectroscopy. Appl Spectrosc. 49 (2), 224-228 (1995).
- Kudryavtsev, V., et al. Combining mfd and pie for accurate single-pair forster resonance energy transfer measurements. Chemphyschem. 13 (4), 1060-1078 (2012).
- Van Zanten, T. S., Greeshma, P. S., Mayor, S. Quantitative fluorescence emission anisotropy microscopy for implementing homo-fluorescence resonance energy transfer measurements in living cells. Mol Biol Cell. 34 (6), tp1(2023).
- Laine, R. F., Jacquemet, G., Krull, A. Imaging in focus: An introduction to denoising bioimages in the era of deep learning. Int J Biochem Cell Biol. 140, 106077(2021).
- Strohl, F., Wong, H. H. W., Holt, C. E., Kaminski, C. F. Total internal reflection fluorescence anisotropy imaging microscopy: Setup, calibration, and data processing for protein polymerization measurements in living cells. Methods Appl Fluoresc. 6 (1), 014004(2017).
- Loman, A., Gregor, I., Stutz, C., Mund, M., Enderlein, J. Measuring rotational diffusion of macromolecules by fluorescence correlation spectroscopy. Photochem Photobiol Sci. 9 (5), 627-636 (2010).
- Böhmer, M., Wahl, M., Rahn, H. -J., Erdmann, R., Enderlein, J. Time-resolved fluorescence correlation spectroscopy. Chem Phys Lett. 353 (5-6), 439-445 (2002).
- Peulen, T. -O., et al. Tttrlib: Modular software for integrating fluorescence spectroscopy, imaging, and molecular modeling. arXiv. , (2024).
- Le Fevre, A. K., et al. Foxp1 mutations cause intellectual disability and a recognizable phenotype. Am J Med Genet A. 161A (12), 3166-3175 (2013).
- Hamdan, F. F., et al. De novo mutations in foxp1 in cases with intellectual disability, autism, and language impairment. Am J Hum Genet. 87 (5), 671-678 (2010).
- Wahl, M., Orthaus-Müller, S. Time tagged time-resolved fluorescence data collection in life sciences. , https://www.picoquant.com/images/uploads/page/files/14528/technote_tttr.pdf (2014).
- Lemke, E. A. Site-specific labeling of proteins for single-molecule fret measurements using genetically encoded ketone functionalities. Methods Mol Biol. 751, 3-15 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten