Case Report
ì´ ì œëª©ìž…ë‹ˆë‹¤
Not Published
요약
마ì´í¬ë¡œ : μ ì•°í¼ìƒŒë“œ : & ìƒí‘œ : TESTING ∂ Σ Ω ≈ γ γδ γδ γδ 테스트 ì‹¤ì²´ë„ ì—¬ê¸°ì— ìž…ë ¥
초록
Sample Text
서문
Sample Text
프로토콜
Sample Text
결과
transuroepithelial 호중구 마ì´ê·¸ë ˆì´ì…˜ 분ì„ì€ ë‹¤ì–‘í•œ ìžê·¹ (그림 1B) ë‹˜ì˜ ì§ˆë¬¸ì— ë‹µë³€ ë°°ì–‘ ë°©ê´‘ ìƒí”¼ ì„¸í¬ ì¸µì— ê±¸ì³ ì¸ê°„ì˜ PMN 마ì´ê·¸ë ˆì´ì…˜ì˜ ì–‘ì í‰ê°€ë¥¼ í• ìˆ˜ 있습니다. í”„ë¡œí† ì½œì€ ê°„ë‹¨í•˜ì§€ë§Œ, PMN 마ì´ê·¸ë ˆì´ì…˜ ì˜í–¥ì„ ë¯¸ì¹˜ê³ ê²°ê³¼ì ìœ¼ë¡œì´ ë¶„ì„ì˜ ìž¬í˜„ì„±ì— ì˜í–¥ì„ ë¯¸ì¹ ìˆ˜ìžˆëŠ” ë³€ìˆ˜ì˜ ìˆ«ìžê°€ìžˆë‹¤. ê¸°ìˆ ë° ìƒë¬¼ ë³µì œ 사ì´ì˜ ë³€ë™ì„±ì„ 줄ì´ê¸° 위해 침투성 지ì›ê³¼ PMNì„ ì¤€ë¹„í•˜ëŠ” ë™ì•ˆ 조치를 취해야한다. 예를 들어, 만 투과 충분히 합류 5637 세í¬ì¸µì´ ì‹¤í—˜ì— ì‚¬ìš©ë˜ì–´ì•¼ í•¨ìœ ì§€ì›í•©ë‹ˆë‹¤. 5637 세í¬ì˜ 합류는 ì•¡ì²´ 불 íˆ¬ê³¼ì„±ì„ ì¸¡ì •í•˜ëŠ” 기능 분ì„ì„ ì‚¬ìš©í•˜ì—¬ í‰ê°€ë©ë‹ˆë‹¤. 매체 투과성 지ì›ì„ 통해 ìƒë¶€ ì €ìˆ˜ì§€ í‰í˜•ì„ 추가하면, 5637 ì„¸í¬ ì‹¤í—˜ì„ ìˆ˜í–‰ í• ì¶©ë¶„í•œ 합류하지 않습니다. ìƒë¶€ ì €ìˆ˜ì§€ì— ë³¼ë¥¨ì´ ìœ ì§€ë˜ëŠ” 경우,ë‹¤ìŒ íˆ¬ê³¼ì„± ì§€ì› PMN 마ì´ê·¸ë ˆì´ì…˜ì„ í‰ê°€í•˜ëŠ” ë° ì‚¬ìš©í• ìˆ˜ 있습니다. 우리는 세í¬ì˜ í•©ë¥˜ì— ë”°ë¼ ì†Œí ìƒìŠ¹ì´ ì‹œìŠ¤í…œì— transepithelial ì „ê¸° ì €í•ì„ ì¸¡ì •í•˜ê³ ,ì´ ë°©ë²•ì„ ì„ íƒí•˜ë©´, 치료는 ê·¸ë ‡ì§€ ë©¸ê· ì„¤ì¹˜ë¥¼ 오염ë˜ì§€ ì•Šë„ë¡ì£¼ì˜í•´ì•¼í•œë‹¤. ì¹ ì¼ íŒŒì¢… 후 5637 세í¬ì˜ 합류는 세í¬ì˜ 통과 수와 투과 지ì›ì— ì”¨ì•—ì„ í’ˆê³ ì„¸í¬ì˜ 수 등 여러 ìš”ì¸ì— ì˜í•´ ì˜í–¥ì„ë°›ì„ ìˆ˜ìžˆë‹¤. ë˜í•œ, 5637 세í¬ê°€ 파종하는 ë™ì•ˆ 거꾸로 ìœ„ì¹˜ì— íˆ¬ê³¼ì„± 지ì›ì„ 배양하는 ì‹œê°„ì€ 16 시간 (그림 1A)를 초과하지 않아야합니다. 최ì ì˜ ìž¬í˜„ì„±ì„ ìœ„í•´, í”„ë¡œí† ì½œì€ ì •í™•í•˜ê²Œ ë”°ë¼ì•¼í•œë‹¤. 마지막으로, íˆ¬ê³¼ì„±ì´ í•©ë¥˜ 5637 세í¬ì¸µ 1-2 ì¼ ì´ë‚´ì— 사용하시기 ë°”ëžë‹ˆë‹¤ í•¨ìœ ì§€ì›í•˜ê³ , 지ì›ì˜ 세í¬ë§‰ì€ 5637 세í¬ì˜ 하나 성장하는 ë™ì•ˆì´ë‚˜ transuroepithelial 호중구 마ì´ê·¸ë ˆì´ì…˜ 분ì„하는 ë™ì•ˆ ì ‘ì´‰í•´ì„œëŠ” 안ë©ë‹ˆë‹¤.
5637 세í¬ë¿ë§Œ 아니ë¼, 변화는 PMN 준비 ì¤‘ì— ë„ìž… í• ìˆ˜ 있습니다. ì´ ìˆ«ìžê°€ 개별ì—ì„œ 개별로 다릅니다 있지만, PMNì„ ë¶„ë¦¬ 위ì—ì„œ 설명한 í”„ë¡œí† ì½œì„ ì‚¬ìš©í•˜ì—¬, ì¸ê°„ì˜ í˜ˆì•¡ 1 ㎖는 ì¼ë°˜ì 으로 6 10 PMNì„ ì–»ì„ ìˆ˜ 있습니다. ê°œì¸ ê¸°ì¦ìžì˜ í˜ˆì•¡ì˜ ì „í˜•ì ì¸ ìˆ˜ìœ¨ì´ ì•Œë ¤ì§€ë©´, 격리 í”„ë¡œí† ì½œì„ ë”°ë¼ ìƒí•˜ë¡œ ì¡°ì •í•˜ê±°ë‚˜ í• ìˆ˜ 있습니다. ê±´ê°•ì— í•´ë¡œìš´ 아프거나 ê°œì¸ì˜ PMNì€ í”¼í•´ì•¼í•œë‹¤, ë° ìƒë¬¼ í•™ì 관찰 결과를 재현 í• ìˆ˜ 있는지 확ì¸í•˜ê¸° 위해 ë³µì œì— ëŒ€í•´ 서로 다른 PMN 기ì¦ìžë¥¼ 사용해야합니다. PMNì„ ë¶„ë¦¬í•˜ëŠ” ë™ì•ˆ 활성화를 피하기 위해 부드럽게 ë¬´ê· ì²˜ë¦¬ë˜ì–´ì•¼í•œë‹¤. PMN í•œ 번 몸ì—ì„œ ì œê±° 오랜 기간 ë™ì•ˆ ìƒì¡´í•˜ì§€ 않는 마지막으로, 실험 ì ˆì°¨ì˜ íƒ€ì´ë°ì´ 중요합니다. 우리는 분리 ì ˆì°¨ë¥¼ 완료 1 시간 ì´ë‚´ PMN를 사용합니다. ê¸°ìˆ ìµœì†Œ 3 ë³µì œëŠ” ê° ìƒë¬¼ ë³µì œì— í¬í•¨ë˜ì–´ì•¼í•œë‹¤, ì´ëŸ¬í•œ ê³ ë ¤ 사í•ì„ ê³ ë ¤.
1 시간 후 하부 ì €ìˆ˜ì§€ PMNì˜ ìˆ˜ëŠ” 10 6 ìž…ë ¥ PMNë¡œ ì •ê·œí™” 그림 2ì— í‘œì‹œë©ë‹ˆë‹¤. ë˜í•œ, PMN 번호는 ìƒë¬¼ í•™ì ë³µì œ 사ì´ì˜ 변화를 ì¤„ì¼ ìˆ˜ 있습니다 ìž…ë ¥ PMNì— ì •ìƒí™” ì´í›„ 내부 í†µì œì— ë¹„êµ ë 수있다. 세부 사í•ì—주ì˜ë¥¼ 위ì—ì„œ 설명한 í”„ë¡œí† ì½œì„ ì¤€ìˆ˜ 박테리아 (그림 2A) ë° í™”í•™ 주성 물질 (그림 2B) ë“±ì˜ ìžê·¹ì— 대한 ì‘답으로 PMN 마ì´ê·¸ë ˆì´ì…˜ ì—´ê±° í• ìˆ˜ 있습니다.
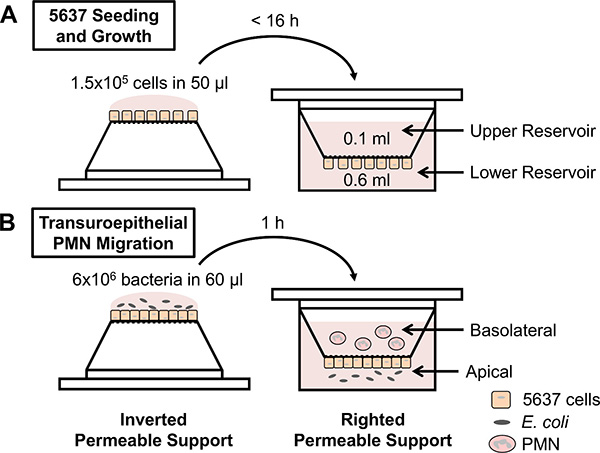
그림 1. 실험 ì„¤ê³„ì˜ ê°œëžµë„. (A) 5637 ë°©ê´‘ ìƒí”¼ 세í¬ê°€ 거꾸로 투과성 지ì›ì— ì”¨ì•—ì„ í’ˆê³ ìžˆìœ¼ë©°, 지ì›ì€ 24 - ì›° í”Œë ˆì´íŠ¸ì— ê³ ì¹˜ê²Œë˜ê³ , 세í¬ëŠ” 합류로 ì„±ìž¥í•˜ê³ ìžˆìŠµë‹ˆë‹¤. (B) 투과 ì €ë…ì„ ë¨¹ë‹¤í•©ë¥˜ 5637 세í¬ë¥¼ í¬í•¨í•˜ëŠ” í¬íŠ¸ëŠ” E.ì— ê±°ê¾¸ë¡œ ê°ì—¼ ìƒí”¼ ì¸µì˜ ê¼ëŒ€ê¸°ë©´ì— ëŒ€ìž¥ê· . ë˜í•œ, chemoattractantsì´ ë‚®ì€ ì €ìˆ˜ì§€ì— ë°°ì¹˜ í• ìˆ˜ 있습니다. 투과성 지ì›ì€ ë‚®ì€ ë¶€ì°© í”Œë ˆì´íŠ¸ë¡œ ê³ ì¹˜ê²Œë˜ë©°, ê°“ ê³ ë¦½ ëœ ì¸ê°„ì˜ PMNì€ ìƒë¶€ ì €ìˆ˜ì§€ (ìƒí”¼ ì¸µì˜ ê¸°ì € ì¸¡ë©´ì„ ë‚˜íƒ€ë‚´ëŠ”)ì— ì ìš©ë©ë‹ˆë‹¤. PMNì€ ìƒí”¼ì— ê±¸ì³ ë§ˆì´ê·¸ë ˆì´ì…˜ hemacytometer를 사용하여 하부 ì €ìˆ˜ì§€ì—ì„œ ì—´ê±°ë©ë‹ˆë‹¤.
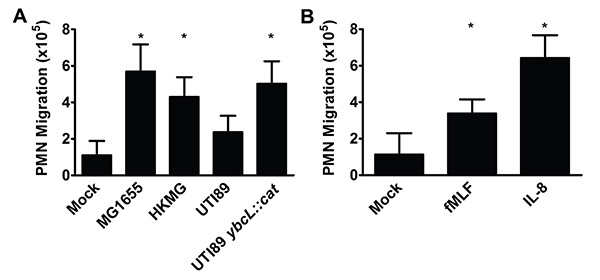
그림 2. PMN는 다양한 ìžê·¹ì— 대한 ì‘답으로 ë°©ê´‘ ìƒí”¼ë¥¼ 통해 마ì´ê·¸ë ˆì´ì…˜ í• ìˆ˜ 있습니다. 비병ì›ì„± E.와 (A) ê°ì—¼ ëŒ€ìž¥ê· ê· ì£¼ MG1655ì€ MG1655 (HKMG) ë˜ëŠ” UPEC ëŒì—°ë³€ì´ UTI89 ybcLì„ ì—´ ì‚´í•´ :: ê³ ì–‘ì´ ê°œì›”ë³´ë‹¤ 훨씬 ë” PMN 마ì´ê·¸ë ˆì´ì…˜ì„ ì´ëŒì–´ì•¼ìƒ 형 UPEC 변형 UTI89와 CK ê°ì—¼ ë˜ëŠ” ê°ì—¼, ë°©ê´‘ì—¼ (*, P <0.001) 분리. (B) ëª¨ì˜ ì¹˜ë£Œ (*, P <0.001)보다 훨씬 ë” PMN ì´ì£¼ 하부 ì €ìˆ˜ì§€ ê²°ê³¼ fMLF (100 NM) ë˜ëŠ” IL-8 (100 NG / ML)ì˜ ì¶”ê°€. ë°ì´í„° ë³µì œ 최소 3 ìƒë¬¼ì˜ í‰ê· ê³¼ 표준 편차를 나타냅니다. 통계ì 으로 ìœ ì˜ í•œ ì°¨ì´ëŠ” ì§ì´ í•™ìƒì˜ t 테스트를 사용하여 ì¸¡ì • 하였다.
토론
공개
íë¡œ
감사의 말
자료
| Name | Company | Catalog Number | Comments |
| (3-Glycidyloxypropyl)trimethoxysilane | Sigma | 440167 | GOPS |
| 0.25% Trypsin-EDTA (1X) | Gibco | 25200-056 | |
| 4-Dodecylbenzenesulfonic acid | Sigma | 44198 | DBSA |
| 96-well plate | Falcon | 353075 | |
| Acetone | Technic | 530 | |
| Acrylic resin | Fischer scientific | NC1455685 | |
| agarose | Sigma | A9539 | |
| autoclave | Tuttnauer | 3150 EL | |
| AZ 10XT | Microchemicals | Positive photoresist | |
| AZ 826 MIF Developer | Merck | 10056124960 | Metal-ion-free developer for the negative photoresist |
| AZ Developer | Merck | 10054224960 | Metal-ion-free developer for the positive photoresist |
| AZ nLof 2070 | Microchemicals | Negative photoresist | |
| Buprenorphine | Axience | ||
| Carprofen | Rimadyl | ||
| Centrifuge Sorvall Legend X1R | Thermo Scientific | 75004260 | |
| CMOS camera Prime 95B | Photometrics | ||
| CO2 incubator HERAcell 150i | Thermo scientific | ||
| DAC board | National Instruments | USB 6259 | |
| Déco spray Pébéo | Cultura | 3167860937307 | Black acrylic paint |
| Dextran Texas Red 70.000 | Thermofisher | D1830 | |
| Die bonding paste "Epinal" | Hitachi | EN-4900GC | Silver paste |
| Dimethyl sulfoxide | Sigma | D2438 | |
| Dispensing machine | Tianhao | TH-2004C | |
| Dulbecco’s Modified Eagle’s Medium + GlutaMAX™-I | Gibco | 10567-014 | |
| Dulbecco's Modified Eagle's Medium | Sigma | D6429 | |
| Egg incubator COUVAD'OR 160 | lafermedemanon.com | ||
| Ethylene glycol | Carl Roth | 6881.1 | |
| Fertilized eggs of Japanese quail | Japocaille | ||
| Fetal Bovine Serum | VWR | S181BH | |
| Flask | Greiner | 658170 | |
| Fluorescence macroscope | Leica MZFLIII | ||
| Gl261 | DSMZ | ACC 802 | |
| Gold pellets - Dia 3 mm x 6 mm th | Neyco | ||
| Handheld automated cell counter | Millipore | PHCC00000 | |
| Heating and drying oven | Memmert | UF110 | |
| Hexadimethrine Bromide Sequa-brene | Sigma | S2667 | |
| hot plate Delta 6 HP 350 | Süss Microtec | ||
| Illumination system pE-4000 | CoolLed | ||
| Infrared tunable femtosecond laser (Maï-Taï) | Spectra Physics (USA) | ||
| Ionomycin calcium salt | Sigma | I3909 | |
| Kapton tape SCOTCH 92 33x19 | 3M | Polyimide protection tape | |
| Lab made pulse generator | |||
| Labcoter 2 Parylene Deposition system PDS 2010 | SCS | ||
| Lenti-X 293 T cell line | Takara Bio | 63218 | HEK 293T-derived cell line optimized for lentivirus production |
| Lenti-X GoStix Plus | Takara Bio | 631280 | Quantitative lentiviral titer test |
| Mask aligner MJB4 | Süss Microtec | ||
| Micro-90 Concentrated cleaning solution | International Products | M9050-12 | |
| Microscope slides 76 x 52 x 1 mm | Marienfeld | 1100420 | |
| Needles 30G | BD Microlance 3 | 304000 | |
| PalmSens4 potentiostat | PalmSens | ||
| parylene-c : dichloro-p-cyclophane | SCS | 300073 | |
| PCB Processing Tanks | Mega Electronics | PA104 | |
| PEDOT:PSS Clevios PH 1000 | Heraeus | ||
| penicillin / streptomycin | Gibco | 15140-122 | |
| Petri dish | Falcon | 351029 | |
| pGP-CMV-GCaMP6f | Addgene | 40755 | plasmid |
| Phosphate Buffer Saline solution | Thermofisher | D8537 | |
| Plasma treatment system PE-100 | Plasma Etch | ||
| PlasmaLab 80 Reactive Ion Etcher | Oxford Instruments | ||
| Plastic mask | Selba | ||
| Plastic weigh boat 64 x 51 x 19 mm | VWR | 10770-454 | |
| Poly-dimethylsiloxane: SYLGARD 184 Silicone Elastomer Kit | Dow chemicals | 1673921 | |
| Polyimide copper film 60 µm (Kapton) | Goodfellow | IM301522 | |
| Propan-2-ol | Technic | 574 | |
| Protolaser S | LPKF | ||
| puromycin | Gibco | A11103 | |
| Round cover glass 5 mm diameter | Fischer scientific | 50-949-439 | |
| Scepter Sensors - 60 µm | Millipore | PHCC60050 | |
| Silicone adhesive Kwik-Sil | World Precision Instruments | ||
| spin coater | Süss Microtec | ||
| Spin Coater | Laurell | WS-650 | |
| Super glue | Office depot | ||
| tetracycline-free fœtal bovine Serum | Takara Bio | 631105 | |
| Thermal evaporator Auto 500 | Boc Edwards | ||
| Two-photon microscope | Zeiss LSM 7MP | ||
| U87-MG | ATCC | HTB-14 | Human glioblastoma cells |
| Ultrasonic cleaner | VWR | ||
| Vortex VTX-3000L | LMS | VTX100323410 | |
| Xfect single shots reagent | Takara Bio | 631447 | Transfection reagent |
참고문헌
- Mukund, K., Subramaniam, S. Skeletal muscle: A review of molecular structure and function. in health and disease. Wiley Interdiscip Rev Syst Biol Med. 12 (1), 1462 (2020).
- Feige, P., Brun, C. E., Ritso, M., Rudnicki, M. A. Orienting muscle stem cells for regeneration in homeostasis, aging, and disease. Cell Stem Cell. 23 (5), 653-664 (2018).
- Mauro, A. Satellite cell of skeletal muscle fibers. J Biophys Biochem Cytol. 9 (2), 493-495 (1961).
- Seale, P., et al. Pax7 is required for the specification of myogenic satellite cells. Cell. 102 (6), 777-786 (2000).
- Fuchs, E., Blau, H. M. Tissue stem cells: Architects of their niches. Cell Stem Cell. 27 (4), 532-556 (2020).
- Hernández-hernández, J. M., et al. The myogenic regulatory factors, determinants of muscle development, cell identity and regeneration. Semin Cell Dev Biol. 72, 10-18 (2017).
- Zammit, P. S. Function of the myogenic regulatory factors Myf5, MyoD, Myogenin and MRF4 in skeletal muscle, satellite cells and regenerative myogenesis. Semin Cell Dev Biol. 72. , 19-32 (2017).
- Sabourin, L. A. The molecular regulation of myogenesis. Clin Genet. 1 (1), 16-25 (2000).
- Cooper, R. N., et al. In vivo satellite cell activation via Myf5 and MyoD in regenerating mouse skeletal muscle. J Cell Sci. 112 (17), 2895-2901 (1999).
- Rudnicki, M. A., Jaenisch, R. The MyoD family of transcription factors and skeletal myogenesis. Bioessays. 17 (3), 203-209 (1995).
- Braun, T., Arnold, H. H. Inactivation of Myf-6 and Myf-5 genes in mice leads to alterations in skeletal muscle development. EMBO J. 14 (6), 1176-1186 (1995).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Braun, T., et al. MyoD expression marks the onset of skeletal myogenesis in Myf-5 mutant mice. Development. 120 (11), 3083-3092 (1994).
- Rudnicki, M. A., et al. MyoD or Myf-5 is required for the formation of skeletal muscle. Cell. 75 (7), 1351-1359 (1993).
- Montarras, D., et al. Developmental biology: Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Sacco, A., Doyonnas, R., Kraft, P., Vitorovic, S., Blau, H. M. Self-renewal and expansion of single transplanted muscle stem cells. Nature. 456 (7221), 502-506 (2008).
- Cerletti, M., et al. Highly efficient, functional engraftment of skeletal muscle stem cells in dystrophic muscles. Cell. 134 (1), 37-47 (2008).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nat Protoc. 10 (10), 1612-1624 (2015).
- Porpiglia, E., et al. High-resolution myogenic lineage mapping by single-cell mass cytometry. Nat Cell Biol. 19 (5), 558-567 (2017).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry Part A. 81 (7), 552-566 (2012).
- Hartmann, F. J., et al. . Mass Cytometry: Methods and Protocols. , (2019).
- Devine, R. D., Behbehani, G. K. Use of the pyrimidine analog, 5-iodo-2'-deoxyuridine (IdU) with cell cycle markers to establish cell cycle phases in a mass cytometry platform. J Vis Exp. 176, 60556 (2021).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Nag, A. C., Foster, J. D. Myogenesis in adult mammalian skeletal muscle in vitro. J Anat. 132 (Pt. 1, 1-18 (1981).
- Le Moigne, A., et al. Characterization of myogenesis from adult satellite cells cultured in vitro). Int J Dev Biol. 34, 171-180 (1990).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Chu, C., Cogswell, J., Kohtz, D. S. MyoD functions as a transcriptional repressor in proliferating myoblasts. J Biol Chem. 272 (6), 3145-3148 (1997).
- Shah, B., Hyde-Dunn, J., Jones, G. E. Proliferation of murine myoblasts as measured by bromodeoxyuridine incorporation. Methods in Mol Biol. 75, 349-355 (1997).
- Springer, M. L., Blau, H. M. High-efficiency retroviral infection of primary myoblasts. Somat Cell Mol Genet. 23 (3), 203-209 (1997).
- Rando, T. A., Blau, H. M. Primary mouse myoblast purification, characterization, and transplantation for cell-mediated gene therapy. J Cell Biol. 125 (6), 1275-1287 (1994).
- Springer, M. L., Rando, T. A., Blau, H. M. Gene delivery to muscle. Curr Protoc Hum Genet. Chapter 13, Unit13.4. , (2002).
- Cull-Candy, S. G., Fohlman, J., Gustavsson, D., Lullmann-Rauch, R., Thesleff, S. The effects of taipoxin and notexin on the function and fine structure of the murine neuromuscular junction. Neuroscience. 1 (3), 175-180 (1976).
- Francis, B., John, T. R., Seebart, C., Kaiser, New toxins from the venom of the common tiger snake (Notechis scutatus scutatus). Toxicon. 29 (1), 85-96 (1991).
- Navarro, K. a. e. l. a. L., Monika Huss, ., Smith, J. e. n. n. i. f. e. r. C., Patrick Sharp, . . James O Marx, Cholawat Pacharinsak, Mouse Anesthesia: The Art and Science, ILAR Journal. 62, 1-2 (2021).
- Langford, D., Bailey, A., Chanda, M., et al. Coding of facial expressions of pain in the laboratory mouse. Nat Methods. 7, 447 (2010).
- Matsumiya LC, ., Sorge RE, . Sotocinal SG, Tabaka JM, Wieskopf JS, Zaloum A, King OD, Mogil JS. Using the Mouse Grimace Scale to reevaluate the efficacy of postoperative analgesics in laboratory mice. J Am Assoc Lab Anim Sci. 2012 (1), 42-49 (2012).
- Gonzalez, V. D., et al. High-grade serous ovarian tumor cells modulate NK cell function to create an immune-tolerant microenvironment. Cell Rep. 36 (9), 109632 (2021).
- Delgado-Gonzalez, A., et al. Measuring trogocytosis between ovarian tumor and natural killer cells. STAR Protoc. 3 (2), 101425 (2022).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry Part A. 83 (5), 483-494 (2013).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. J Vis Exp. 69, (2012).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample preparation for mass cytometry analysis. J Vis Exp. 122, 54394 (2017).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Curr Protoc Cytom. Chapter 10., Uni10.17. , (2010).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry Part A. 81 (6), 467-475 (2012).
- Kimball, A. K., et al. A beginner's guide to analyzing and visualizing mass cytometry data. J Immunol. 200 (1), 3-22 (2018).
- Weber, L. M., Robinson, M. D. Comparison of clustering methods for high-dimensional single-cell flow and mass cytometry data. Cytometry Part A. 89 (12), 1084-1096 (2016).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nat Methods. 13 (6), 493-496 (2016).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Anal Chem. 80 (7), 2539-2547 (2008).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nat Commun. 12 (1), 692 (2021).
- de Morree, A., et al. Staufen1 inhibits MyoD translation to actively maintain muscle stem cell quiescence. Proc Natl Acad Sci U S A. 114 (43), E8996-E9005 (2017).
- Luo, D., et al. Deltex2 represses MyoD expression and inhibits myogenic differentiation by acting as a negative regulator of Jmjd1c. Proc Natl Acad Sci U S A. 114 (15), E3071-E3080 (2017).
- Wersto, R. P., et al. Doublet discrimination in DNA cell-cycle analysis. Cytometry. 46 (5), 296-306 (2001).
- Porpiglia, E., Blau, H. M. Plasticity of muscle stem cells in homeostasis and aging. Curr Opin Genet Dev. 77, 101999 (2022).
- Porpiglia, E., et al. Elevated CD47 is a hallmark of dysfunctional aged muscle stem cells that can be targeted to augment regeneration. Cell Stem Cell. 29 (12), 1653-1668 (2022).
- Brunet, A., Goodell, M. A., Rando, T. A. Ageing and rejuvenation of tissue stem cells and their niches. Nat Rev Mol Cell Biol. 24 (1), 45-62 (2022).
- Danielli, S. G., et al. Single-cell profiling of alveolar rhabdomyosarcoma reveals RAS pathway inhibitors as cell-fate hijackers with therapeutic relevance. Sci Adv. 9 (6), (2023).
- de Morree, A., Rando, T. A. Regulation of adult stem cell quiescence and its functions in the maintenance of tissue integrity. Nat Rev Mol Cell Biol. 24 (5), 334-354 (2023).
- Yucel, N., et al. Glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell function. Cell Rep. 27 (13), 3939-3955 (2019).
- Tierney, M. T., Sacco, A. Inducing and evaluating skeletal muscle injury by notexin and barium chloride. Methods Mol Biol. 1460, 53-60 (2016).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), (2016).
- Call, J. A., Lowe, D. A. Eccentric contraction-induced muscle injury: Reproducible, quantitative, physiological models to impair skeletal muscle's capacity to generate force. Methods Mol Biol. 1460, 3-18 (2016).
- Garry, G. A., Antony, M. L., Garry, D. J. Cardiotoxin Induced Injury and Skeletal Muscle Regeneration. Methods Mol Biol. 1460, 61-71 (2016).
- Le, G., Kyba Lowe, D. A., M, Freeze injury of the tibialis anterior muscle. Methods Mol Biol. 1460, 33-41 (2016).
- Borok, M., et al. Progressive and coordinated mobilization of the skeletal muscle niche throughout tissue repair revealed by single-cell proteomic analysis. Cells. 10 (4), (2021).
- Petrilli, L. L., et al. High-dimensional single-cell quantitative profiling of skeletal muscle cell population dynamics during regeneration. Cells. 9 (7), 1723 (2020).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Mol Cell. 74 (3), 609-621 (2019).
- Hartmann, F. J., et al. Scalable conjugation and characterization of immunoglobulins with stable mass isotope reporters for single-cell mass cytometry analysis. Methods Mol Biol. , 55-81 (1989).
- Frimand, Z., Das Barman, ., Kjær, S., R, T., Porpiglia, E., de Morrée, A. Isolation of quiescent stem cell populations from individual skeletal muscles. J Vis Exp. 190, 64557 (2022).
- Krutzik, P. O., Nolan, G. P. Intracellular phospho-protein staining techniques for flow cytometry: monitoring single cell signaling events. Cytometry A. 55 (2), 61-70 (2003).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat Biotechnol. 30 (9), 858-867 (2012).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Curr Protoc Immunol. Chapter. 8, 11-18 (2012).
- Krutzik, P. O., Clutter, M. R., Nolan, G. P. Coordinate analysis of murine immune cell surface markers and intracellular phosphoproteins by flow cytometry. J Immunol. 175 (4), 2357-2365 (2005).
- Krutzik, P. O., Irish, J. M., Nolan, G. P., Perez, O. D. Analysis of protein phosphorylation and cellular signaling events by flow cytometry: techniques and clinical applications. Clin Immunol. 110 (3), 206-221 (2004).
- Han, G., Spitzer, M. H., Bendall, S. C., Fantl, W. J., Nolan, G. P. Metal-isotope-tagged monoclonal antibodies for high-dimensional mass cytometry. Nat Protoc. 13 (10), 2121-2148 (2018).
- Chevrier, S., et al. Compensation of signal spillover in suspension and imaging mass cytometry. Cell Syst. 6 (5), 612-620 (2018).
- Bjornson, Z. B., Nolan, G. P., Fantl, W. J. Single-cell mass cytometry for analysis of immune system functional states. Curr Opin Immunol. 25 (4), 484-494 (2013).
- Kalina, T., Lundsten, K., Engel, P. Relevance of antibody validation for flow cytometry. Cytometry A. 97 (2), 126-136 (2020).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. J Immunol Methods. 243 (1-2), 77-97 (2000).
- Roederer, M. Spectral compensation for flow cytometry: visualization artifacts, limitations, and caveats. Cytometry. 45 (3), 194-205 (2001).
- Tung, J. W., Parks, D. R., Moore, W. A., Herzenberg, L. A., Herzenberg, L. A. New approaches to fluorescence compensation and visualization of FACS data. Clin Immunol. 110 (3), 277-283 (2004).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eur J Immunol. 51 (12), 2708-3145 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유