Method Article
튜브에 칩 형식을 사용 하 여 산발적 인 가족 용 대 환자에 있는 유전 변이 대 한 디지털 중 합 효소 연쇄 반응 분석 결과
요약
디지털 연쇄 반응 (PCR) 단일 뉴클레오티드 이체와 DNA 복사 번호 이체의 고감도 검출을 위한 유용한 도구입니다. 여기, 칩에 튜브 형식으로 디지털 PCR를 사용 하 여 인간 게놈에 있는 드문 이체를 측정 하기 위한 주요 고려 사항을 설명 합니다.
초록
인간 유전 변이의 정량 분석은 심각한 의료 조건, 종양 등의 분자 특성을 이해 하는 데 중요 합니다. 디지털 중 합 효소 연쇄 반응 (PCR) DNA 복사 번호 이체의 정확한 정량화를 사용 하기 때문에 그들은 약물 내성 돌연변이 등 희귀 유전자 변이 검출을 위한 필수적인 도구가 되고있다. 그것은 것으로 예상 된다 디지털 PCR (dPCR)를 사용 하 여 분자 진단 임상 연습에서 사용할 수 있는 가까운 미래에; 따라서, 인간의 유전 물질과 dPCR를 효율적으로 수행 하는 방법 뜨거운 주제가입니다. 여기, 용 대 대장균 (APC) 신체적인 mosaicism 8 dPCR 반응을 동시에 실시 될 수 있는 칩에 튜브 형식으로 dPCR를 사용 하 여 검색 하는 방법을 소개 합니다. 채우고는 및 밀봉 칩에 반응 혼합물 때 주의 해야 합니다. 이 문서에서는 오버 및 긍정적인 파티션의 과소를 방지 하는 방법을 보여 줍니다. 또한, 우리는 특정 증폭을 확인 하는 데 사용 될 수 있는 칩에는 파티션에서 dPCR 제품을 수집 하기 위한 간단한 절차를 제시. 우리는이 방법을 보고서는 dPCR 유전자 연구에서 칩--튜브 방법으로 홍보 도움이 될 것입니다 바랍니다.
서문
정량 PCR (정량) 단일 뉴클레오티드 변종 (SNVs)를 포함 한 인간의 유전 변이 그리고 DNA 복사 번호 유사 (CNVs) 척도를 자주 사용 됩니다. 정량, 중 합 효소 반응 기존의 끝점 PCR에서 같은 방식으로 각 튜브에서 수행 됩니다 그리고 모든 열 주기 후 튜브에서 증폭 신호 획득. 대조적으로,이 보고서에 끝점 dPCR를 의미 하는 dPCR에 PCR 혼합에 로드 많은 현미경 챔버, 파티션 되 나, DNA 템플릿은 제시 또는 제한 희석 및 모든 파티션에 포함 된 PCR 혼합으로 평가에 결 석 음수 또는 완전 한 PCR 후 긍정적인. 부정적인 파티션에 있는 DNA는 서식 파일이 없습니다는 추정 하기 쉬운 반면, 그것 아니다 분명 DNA 서식 파일의 복사본은 긍정적인 파티션에 존재. 따라서, 긍정적인 파티션 서식 파일 DNA의 수는 부정적인 파티션1에서 수 포아송 분포에 따라 견적입니다. dPCR는 더 비싼 및 정량에 비해 작은 동적 범위는 하지만이 메서드는 절대 정량화를 가능 하 게 제공 하는 높은 감도 및 더 큰 정밀2.
DPCR의 주요 응용 프로그램 중 하나입니다 차세대 시퀀싱 (NGS)에 의해 식별 된 이체의 유효성 검사. 희소 한 이체, 의미 낮은 변형 분수의 경우 특히 유효성 검사는 NGS3에서 발생할 수 있는 시퀀싱 오류로 인해 중요 합니다. 동안 생어 시퀀싱 및 정량 SNVs 및 CNVs의 유효성 검사에 대 한 유용한 도구, 그들의 감도 dPCR에 비해 낮은. 따라서, dPCR 기술은 드문 이체 처리 하는 유전자 연구에 대 한 수요 이다. 최근, 약물 저항 같은 암 특성에 관련 된 드문 이체의 액체 생 검에 의해 감지 분자 진단 및 치료4에 화제가 되고있다. DPCR 기술 액체 생 검 연구에 적합 나타나고 개선 동적 범위와 관련 된 고 비용 구현 될 필요는 가까운 장래에, 중요 한 임상 응용을가지고 것으로 예상 된다.
상업적으로 사용할 수 있는 dPCR 기술은 드롭릿 및 칩 기반 플랫폼;으로 대략 분류 될 수 있다 차이점은 어떻게 DNA 템플렛은 분할 된5,,67. 칩에 튜브 형식으로 dPCR에 PCR 혼합 칩에 분할으로 모 세관 작용에 의해 배포 됩니다. 칩 내장 8 튜브 스트립,는 많은 실험실 직원은 이미 잘 알고 있을 것입니다, 그리고, 따라서, 8 개의 샘플 한 시간8에 대우 될 수 있다. 읽기 단계에서 < 96 샘플 칩 및 아니라 각 분할의 전체 이미지를 감지 하 여 치료를 1 시간 걸립니다. 칩에 튜브 형식으로 dPCR는 다른 dPCR 시스템에 비해 높은 처리량, 이후 성과 생산성은 사용자를 위한 주요 장점이 있습니다.
이 보고서에서 산발적 인 가족 용 대 환자의 APC 유전자의 신체적인 mosaicism 대표적인 경우로 사용 되 고 dPCR 그리고 NGS의 결과 비교. 이 보고서의 주요 목적은 명확 하 게 계량 칩에 튜브 형식으로 dPCR를 사용 하 여 단일 뉴클레오티드 변종 이다. 우리는이 보고서가 채택 하는 그들의 자신의 일에 대 한 dPCR 플랫폼에 관심 있는 연구원을 위한 도움이 바랍니다.
프로토콜
연구 설계는 하 마마 츠 대학의과 대학 (G-260-4)의 제도적 검토 보드에 의해 승인 되었다. 서 면된 동의 환자와 그의 부모에서 얻은 했다.
1입니다. 품질 관리 Genomic DNA의
참고: 게놈 DNA (gDNA) 확고 실리 카 멤브레인 기반 DNA 정화 방법을 사용 하 여 말 초 혈액에서 추출 되었다. 사전에 아래 절차, gDNA의 농도 분 광 광도 계를 사용 하 여 결정 했다.
- 10-100 ng / µ L의 농도에서 gDNA 샘플을 준비 합니다.
- 8-튜브 스트립 10 µ L의 DNA 견본 버퍼를 추가 합니다.
- 튜브를 DNA 사다리 또는 gDNA 1 µ L를 추가 합니다. 소용돌이 1 분 미 판 첨부 파일을 사용 하 여 혼합.
- 튜브 로드에 전기 이동 법에 젤 장치와 피 펫 팁 (테이블의 재료)를 계측 하 고 '시작' 버튼을 눌러 실행을 시작 합니다.
참고: 전기 영동 시스템 시작 버튼 클릭 한 번으로 자동으로 작동 합니다. - DNA 샘플 버퍼에 포함 된 낮은 마커는 electropherograms에 올바르게 할당 되어 있는지 확인 합니다. 하지 할당 올바르게 수동으로 'Electroherogram 모드'에서 소프트웨어의 마커를 할당 합니다.
참고: DNA 무결성 번호 (DIN) 자동으로 계산 됩니다. DIN 값 DNA 무결성에서 온다. 높고 낮은 소음 값 매우 온전 하 고 강하게 저하 gDNA를 각각 나타냅니다. - 자동으로 gDNA (> 200 bp)의 농도 계산 하는 소프트웨어의 '지역 모드'에서 gDNA (> 200 bp)의 크기 영역을 지정 합니다.
2. 뇌관 및 프로브 설계
- 다음과 같은 조건에서 개방형 액세스 도구를 사용 하 여 용융 온도 (Tm) 값 계산: 10-25 기초, 50mm 나+/K+, 0.80 mM dNTPs, 및 3 m m Mg2 + (자료 테이블). 증폭 Tm 가치 약 60 ° C와 100-300 bp에 amplicon 길이 대상 대립 유전자를 포함 하는 게놈 지역에 앞으로 역 뇌관 디자인.
참고: oligonucleotides의 농도 표 1 을 참조 하십시오. - 다음 조건에 따라 잠긴된 핵 산 (LNA) 참조 및 변형 대립 유전자에 대 한 조사 디자인: (i) 1-6 Lna는 각 프로브; (ii)는 형광 염료와는 끄는 5'에 있는지-그리고 3'-종점 각 프로브의 각각; (iii) 일치 하 고 일치 하지 않는 프로브 Tm 값 사이의 차이 > 10 ° C; (일치 하 고 일치 하지 않는 프로브 iv) Tm 값은 높고, 뇌관의 보다 낮은 각각 [예를 들어, 앞으로 뇌관: 60.8 ° C, 역방향 뇌관: 59.8 ° C, 참조 대립 유전자 프로브 (일치/불일치): 62.6/45.3 ° C, 변형 대립 유전자 프로브 (일치/불일치): 61.7/50.5 ° C]. Tm 값 또한 단계 2.1에 사용 되는 오픈 액세스 도구를 사용 하 여 계산 했다.
- 설계 뇌관 및 프로브 > 0.1% [예를 들어, 단일 염기 다형성 데이터베이스 (dbSNP)] 데이터베이스에서 결정의 주파수를 가진 단 하나 뉴클레오티드 동 질 다 상 포함 하지 않는 확인 하십시오.
3. 디지털 PCR
- 뇌관, 프로브, 그리고 테 버퍼와 함께 표 1 에 설명 된 농도에서 gDNA 재고 솔루션을 준비 하 고-20에서 그들을 저장-4 ° c.
-
표 1에 설명 된 대로 최종 농도에 15 µ L의 전체 볼륨에 실 온에서 시 약을 혼합.
- No-템플릿 컨트롤 (NTC)에 gDNA 대신 증류수 3 µ L를 추가 합니다.
- 3.5 x pipetting 오류에 대 한 계정에 3 중에서 PCR 혼합물의 금액을 준비 합니다.
- PCR 혼합물을 아래로 그것을 혼합 플라스틱
- 8-튜브 스트립에 내장 된 칩에 로딩 플랫폼을 설정 하 고 오토로더에서 그들을 설정 합니다. 칩 및 로딩 플랫폼 사이의 연락처를 확인 합니다.
- 로드 슬라이더는 플랫폼에 맞는 로더의 스 토퍼와 슬라이더를 누르고 있습니다.
- (그림 1A) 슬라이더의 끝 근처 플랫폼에 PCR 혼합물의 15 µ L 플라스틱
- (약 1 분)에 대 한 로더 버튼을 누르면 로더를 실행 합니다.
참고: 아니다 문제가 경우 적은 양의 PCR 혼합 플랫폼에 남아. 그것은 순차적으로 로더를 다시 실행할 수 있습니다. - 칩-에-한-튜브 씰링 증강의 측면 슬롯에 PCR 혼합으로 가득 설정 합니다. 하지 휴식 칩 (그림 1B)를 슬라이드 뚜껑과 최고 뚜껑의 가장자리를 밀어.
- 씰링 증강 (대략 2 분)을 실행 합니다. 불완전 한 씰링 긍정적인 신호 (그림 1C 및 1d)의 교차 오염 때문에 액체의 웅덩이 여전히 표시 되어 순차적으로 1 분 동안 봉인 증강을 다시 실행 합니다.
- 석유 기반 시 약, 관에 유체 씰링의 230 µ L를 추가 합니다.
참고: 칩 지금 액체에 몰두 한다. - 열 cycler에 튜브를 설정 합니다. (약 2 시간)에 대 한 표 2 에 설명 된 대로 열 cycler를 실행 합니다. 긍정적인 파티션 (그림 1E)의 고르지 못한 배급 경우 온도 또는 PCR의 기간을 조정 합니다.
참고: 그것은 설정 것이 좋습니다 램프 속도 약 1 ° C/s. - 초기 소음을 줄이기 위해 적어도 15 분 동안 열 cycler에 튜브를 둡니다.
참고: 튜브 남겨질 수 있습니다 하룻밤 뿐만. 형광 신호도 다음 날 발견 했다. - 탐지 지에 튜브를 설정 하 고는 지 그에 6 mL 증류수를 부 어. 공기 방울 튜브 내외에 표시 되는 경우 (그림 1 층 및 1 G) 선택을 취소 합니다.
- 검출기에는 지 그를 로드 하 고 Fluorescene, 실험 및 샘플/NTC 탭 (자료 테이블)의 선택 후 소프트웨어의 '실행' 버튼 클릭으로 실행 합니다.
참고: 기본 강도 8 샘플의 이중 컬러 감지 약 4-5 분 걸립니다. - 위치 그림, 히스토그램 및 2D 점도 확인 합니다.
참고: iIf 부정적인의 형광 강도 높은, 30-70의 범위 내에서 떨어지는 소프트웨어의 설정 버튼 클릭으로 강도 조정. - 다음 수식을 사용 하 여 variant 대립 유전자 분수 (VAF) 계산.
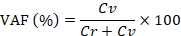
여기, 이력서 및 Cr에는 variant 및 참조 대립 유전자의 복사본 수를은 하는 것이 각각입니다.
참고: 그들은 푸아송 통계 소프트웨어 프로그램에서 자동으로 계산 됩니다.
4입니다. PCR 제품의 수집
- 튜브에서 유체 씰링을 제거 합니다.
- 튜브를 100 µ L의 TE 버퍼를 추가 합니다. 30 s 그리고 짧게 원심 분리기 튜브에 대 한 적극적으로 소용돌이.
- 테 솔루션 다른 튜브에 전송 하 고 30 µ L 3 M 나트륨 아세테이트, 에탄올의 200 µ L를 추가 합니다.
- 하룻밤-20 ° C에서 솔루션을 냉각 한다.
- 16000 x g 에서 30 분에 대 한 솔루션을 원심 고는 상쾌한을 제거 합니다.
- 튜브와 소용돌이를 80% 에탄올의 300 µ L를 추가 합니다.
- 16000 x g 에서 15 분에 대 한 솔루션을 원심 고는 상쾌한을 제거 합니다. 5 분 동안 건조 하 침전을 수 있습니다.
- 1-5 µ L의 TE 버퍼에 침전 공기 후 해산.
- 필요한 경우, 1 단계에 관하여 전기 악기를 사용 하 여 제품 크기를 평가 합니다. 높은 감도 (자료 테이블)에 대 한 젤 장치 솔루션의 1 µ L을 로드 합니다.
결과
우리는 위에서 설명한 절차에 따라 산발적 인 가족 용 대 환자의 APC 유전자의 신체적인 mosaicism 검토. 이전 연구, APC c.834 + 2T > 생어를 사용 하 여 그들의 부모에는 환자만 C 돌연변이 발견 시퀀싱 및 NGS9. 뇌관의 대표적인 결과에 사용 하는 프로브 디자인은 표 3에 표시 됩니다. 게놈 DNA는 proband, 부모, 그리고 6 명의 건강 한 기증자의 혈액에서 추출 되었다. 6.7-7.9 (그림 2 및 표 4)에 9 gDNA 샘플의 DIN 값입니다. 식구 들과 16 진수 사용 되었다 형광 염료로 C와 T 대립 유전자에 대 한 각각. 위치 플롯에 녹색과 노란색 점이 각각 팸 양수와 16 진수-긍정적인 파티션을 나타냅니다 (그림 3A). 블루 관광 명소 팸와 16 진수에 대 한 부정적인 파티션을 나타냅니다. 검은 배경 반응 혼합물 추가 되지 않은 파티션에 해당 합니다. 동안 몇 가지 긍정적인 파티션을 C 대립 유전자의 proband의 아버지 나 NTC에 검색 된, T의 후자, 긍정적인 파티션에 대립 유전자 검색 되지 했다 많은 긍정적인 파티션은 C와 T 대립 유전자의 proband에서 발견 했다. C와 T 대립 유전자 사이 신호 분리 2 차원 (2 차원) scatterplot에 명확 했다 그리고 16 진수와 식구 들의 가장 긍정적인 신호 (그림 3B) 서로 독점 했다. proband의 VAF는 13.2%, 비슷한 NGS (12.7%)를 사용 하 여 결정 (표 4)입니다. Proband의 부모와 건강 한 기증자의 VAFs < 0.1%는 이었다. APC c.834 + 2T에 대 한 검출 한계 > C 돌연변이 총 gDNA10의 10-11 ng와 반복된 측정에 대 한 표준 편차 x 3를 사용 하 여 0.3% 이기 위하여 견적 되었다.
APC c.834 + 2T에 대 한 dPCR 분석 결과 amplicon 크기 > C 123 이기 위하여 예언 되었다 bp. 확인 하 고 dPCR 제품 단일 밴드로 증폭 되었다, dPCR 제품 assayed 칩에서 수집 되었다. 전기 이동 법을 사용 하 여, 단일 밴드 감지 명확 하 게, 그리고 제품 크기 예측 (그림 4)와 일치 했다.
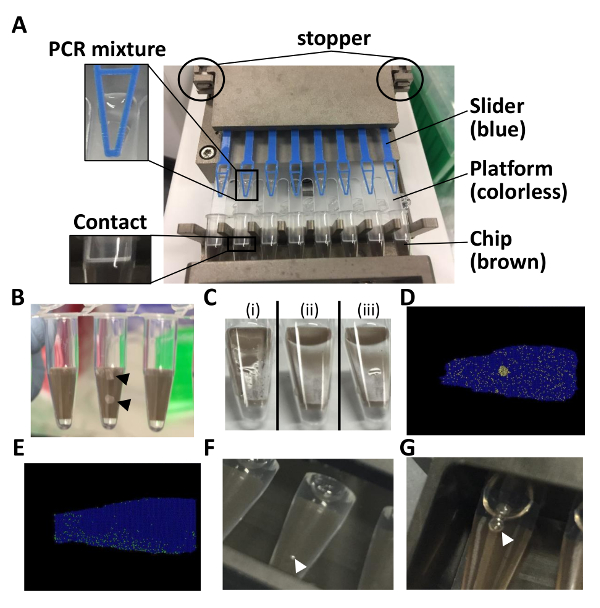
그림 1 : 포인트 칩에서 튜브 형식으로 dPCR 분석 결과 대 한 참고를. (A)이이 패널 오토로더에서 8 튜브 스트립을 설정 하는 방법을 보여 줍니다. (B)이이 패널 깨진된 칩을 보여줍니다. (C)이이 패널 칩 표면 (i) 후 로딩 및 (ii) 씰링 방법을 보여 줍니다. (3) 액체의 웅덩이 불완전 한 봉인 경우 표시 됩니다. (D)이이 패널 교차 오염을 경우 위치 플롯을 표시합니다. (E)이이 패널 고르지 않은 분포의 경우 위치 플롯을 표시합니다. (F)이이 패널 쇼 어떻게 물 튜브 표면에 거품 공기. (G)이이 패널 튜브 내 기포를 보여줍니다. 전체 그림을 통해 흑인과 백인 화살촉 깨진된 칩 조각 나타내고 기포, 각각 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
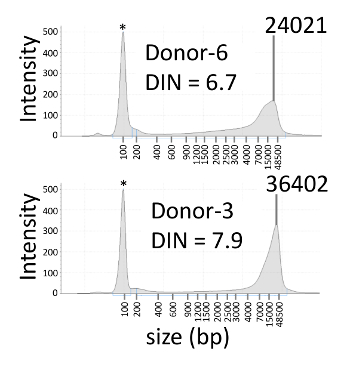
그림 2 : 혈액 gDNA의 대표 electropherograms. 6.7와 7.9로 DIN 혈액 gDNA의 Electropherograms는 각각 상단 및 하단 패널에 표시 됩니다. 별표는 낮은 마커를 나타냅니다. DIN: DNA 무결성 번호입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
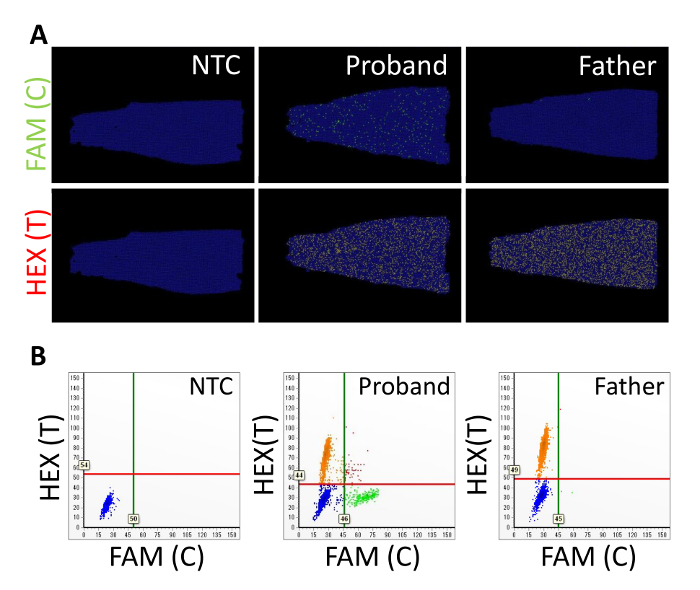
그림 3 : 칩에 튜브 형식으로 dPCR를 사용 하 여 APC 신체적인 mosaicism의 대표적인 결과. (A) 위치 플롯 및 없음-템플릿 컨트롤 (NTC), proband, 그리고 아버지의 (B) scatterplots 여기에 표시 됩니다. 16 진수 및 팸 참조 및 변형 대립 유전자에 각각 해당합니다. 수직 및 수평 라인 각각 16 진수 및 팸 농도의 임계값을 나타냅니다. 이 그림 Kahyo 그 외 여러분 에서 수정 되었습니다. 10. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
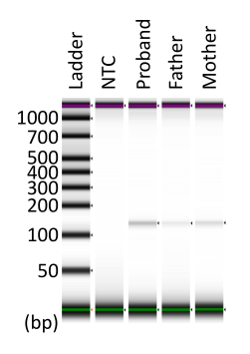
그림 4 : Assayed 칩에서 dPCR 제품의 컬렉션. 수집 및 집중 dPCR 제품 전기 이동 법을 복종 되었다. 예측된 대상 크기는 123 혈압. 이 그림 Kahyo 그 외 여러분 에서 수정 되었습니다. 10. NTC: 없음 템플릿 컨트롤. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 시 약 | 농도 재고 솔루션의 | 볼륨 (μ) | 최종 농도 | 권장 최종 농도 |
| 증류수 DNase/RNase-무료 | - | 1.75 | - | - |
| PCR 마스터 믹스 x 2 | - | 7.5 | - | - |
| LNA 주요 대립 유전자에 대 한 조사 | 2 Μ M | 0.5 | 66.7 nM | 3.33-100 nM |
| LNA 사소한 대립 유전자에 대 한 조사 | 2 Μ M | 0.5 | 66.7 nM | 3.33-100 nM |
| 앞으로 뇌관 | 1 Μ M | 0.5 | 33.3 nM | 3.33-50.0 nM |
| 반전 뇌관 | 1 Μ M | 0.5 | 33.3 nM | 3.33-50.0 nM |
| dPCR 솔루션 x 20 | - | 0.75 | - | - |
| gDNA | 3.33 ng/μ | 3 | 666 pg/μ | 20 세/μ-2000 pg/μ1 |
표 1: dPCR 분석 결과 대 한 사용 하는 시 약. 1 예상된 정밀도 및 감도에 따라.
| 단계 | 온도 | 시간 | 사이클 |
| 초기 변성 | 95 ° C | 5 m | 1 |
| 변성 어 닐 링 및 확장 | 95 ° C 58 ° C | 50 s 90 s | 42 |
| 마지막 확장 | 70 ° C | 5 m | 1 |
표 2: 열 cycler 조건입니다.
| 이름 | 시퀀스1 | Tm 값2 |
| 앞으로 뇌관 | 5'-GGTCAAGGAGTGGGAGAAATC-3 | 60.8 ° C |
| 반전 뇌관 | 5'-TCTTAGAACCATCTTGCTTCATACT-3 | 59.8 ° C |
| LNA T 대립 유전자에 대 한 조사 | 5'-16 진수-ATTT [A] CCTGACCA-IBFQ-3' | 일치: 62.6 ° C 일치 하지 않는: 45.3 ° C |
| LNA C 대립 유전자에 대 한 조사 | 5'-FAM-TTT [G] CCTGACC-IBFQ-3' | 일치: 61.7 ° C 일치 하지 않는: 50.5 ° C |
표 3: 뇌관 및 프로브 디자인. 1 밑줄 및 괄호 문자가 각각 LNA 및 대상된 변형입니다. 2 프로토콜의 2 단계에 따라 계산합니다.
| NGS3 | dPCR4 | |||||||
| 샘플1 | 조직 | 입력 DNA (ng) | DIN2 | VAF (%) | 변형 (복사본) | 참조 (복사본) | VAF (%) | |
| 평균 | RSD | |||||||
| NTC | - | - | - | - | 1.23 | 0 | N/A | N/A |
| Proband | 혈액 | 11.3 | 7.6 | 12.7 | 379 | 2.48 x 103 | 13.2 | 0.0353 |
| 아버지 | 혈액 | 10.3 | 7.7 | 0.0167 | 1.08 | 3.42 x 103 | 0.0341 | 0.439 |
| 어머니 | 혈액 | 10.8 | 7.6 | 0.0136 | 0.956 | 3.04 x 103 | 0.0313 | 0.637 |
| 기증자-1 | 혈액 | 11.7 | 7.3 | - | 1.64 | 3.01 x 103 | 0.0543 | 0.414 |
| 기증자-2 | 혈액 | 10.5 | 7.6 | - | 1.06 | 2.90 x 103 | 0.0406 | 0.539 |
| 기증자-3 | 혈액 | 17.6 | 7.9 | - | 4.24 | 5.65 x 103 | 0.0713 | 0.783 |
| 기증자-4 | 혈액 | 12.3 | 7.6 | - | 2.38 | 4.16 x 103 | 0.0666 | 0.557 |
| 기증자-5 | 혈액 | 19.2 | 7 | - | 1.48 | 6.79 x 103 | 0.0213 | 0.571 |
| 기증자-6 | 혈액 | 14.4 | 6.7 | - | 3.44 | 4.67 x 103 | 0.0728 | 0.729 |
표 4:에 대 한 시험의 결과 dPCR의 APC 신체적인 mosaicism. 1 NTC: 없음 템플릿 컨트롤; 기증자: 건강 한 기증자입니다. 2 DIN: DNA 무결성 번호입니다. 3 이와 이즈미 외. 9, 흠 게놈 var. 2 15057 (2015). 4 실험 triplicate와 독립적으로 반복 3 x; 실행 했다 데이터; 평균 값으로 표현 됩니다. RSD: 상대 표준 편차; N/A: 해당 없음. Kahyo 그 외 여러분 에서 테이블 수정 되었습니다. 10.
토론
DIN 값은 gDNA의 고급 저하 낮은 품질 데이터에 발생할 수 있습니다 때문에 정량화 또는 시퀀싱 하기 전에 손상 된 DNA (예를 들면, 포 르 말린 고정 파라핀 끼워 넣어진 조직 으로부터 gDNA)을 평가 하기 위해 자주 사용 됩니다. DIN 평가, 따라서 되고있다 인간 gDNA11의 품질 관리에 중요 한 단계. 대표적인 실험에 사용 된 gDNA 혈액에서 자사의 추출 후 4-11 년 4 ° C에서 저장 했다, 이후 DIN 값 dPCR 분석 결과 전에 품질 관리에 대 한 결정 했다. 이 단계는 특히 좋습니다 자료 손상 된 것으로 의심 되는 경우. GDNA의 농도 또한 전기 이동 법에 의해 결정 되었다. DPCR의 동적 범위를 없기 때문에 정량의 제한 때문에 관해서는 넓은 gDNA 농도의 측정 성공적인 dPCR 분석 결과 대 한 중요 한 단계 이다. DNA dPCR 분석 결과에서 변형 및 참조 대립 유전자의 총 복사본 수 상관은 입력 금액으로 (R-제곱 = 0.935; 표 4), 전기 이동 법은 dPCR 분석 결과 전에 gDNA 농도 측정 하는 데 유용 합니다.
로드 및 프로세스를 씰링 성공적 결과 대 한 중요 한 단계가 있습니다. 그것은 플랫폼에 PCR 혼합물의 적절 한 설치를 확인 하 여 칩과 플랫폼 (그림 1A) 사이의 접촉 되도록 중요 합니다. 슬라이더에서 PCR 혼합물의 돌출 칩. 에 잘못 된 메일을 발생할 수 있습니다. 위치에 긍정적이 고 부정적인 파티션의 분포를 확인 하는 것은 DNA 템플렛은 임의로 챔버1분할 포아송 통계에서 가정 때문에 정확한 분석을 위해 필요한 또한. 대표 결과에 긍정적인 파티션은 칩 (그림 3)를 통해 배포 했다. 이 결과 포아송 통계에 대 한 요구 사항을 충족 하 고 로드 하 고 절차를 밀봉 했다 효과적인 반영 합니다. 씰링 증강에 튜브를 설정할 경우 칩 수 깨진 최고 뚜껑의 중앙 부분을 너무 강하게 누르면 (그림 1B). 이 방지 하려면 최고 뚜껑의 가장자리를 밀어 부드럽게. 긍정적인 파티션 (그림 1의D)의 인공 클러스터 경우 복사본 수 파티션 사이 교차 오염 때문과 대 수 있습니다. 액체의 물 웅덩이 표면 (그림 1C)에 표시 되 면 봉인 절차 개선 되어야 합니다 (예:순차적으로 1 분 동안 봉인 증강을 다시 실행 하 여). 긍정적인 파티션 (그림 1E)의 고르지 못한 배급이 있는 경우에, 복사 수 부족 증폭 또는 씰링 액체 및 물 오염 과소평가 될 수 있습니다. 이 경우 [예를들면, 온도 또는 PCR (단계 3.11 프로토콜)의 기간을 조정 또는 씰링 액체와 지 그에 부 어 물이 깨끗 하 여] PCR 상태를 조정 한다. 형광 신호는 증류수에 8 튜브 스트립에서 검색 됩니다. 기포 관 표면에 준수, 신호 검출 (그림 1 층) 방해할 수 가능성이 있다. 따라서, 거품이 미세 피 펫 팁 등의 도구를 사용 하 여 허가 한다. 또한, 지 그 (그림 1G)에 설정 하는 경우 튜브 안에 공기 방울 형태 수도 있습니다. 튜브 내부 거품 칩을 충당 하기 위해 충분히 큰 경우에, 그들은 삭제도 한다. 그들은 실 온에서 몇 분 동안 남아 거품 수 있습니다 선택을 취소 합니다.
일부 긍정적인 파티션은 NTC에서 검출 될 수 있습니다. DNA 템플렛의 오염을 방지 하려면 벤치 깨끗 한 유지 하 고 만약에 가능 하다 면, 팬 필터 유닛와 깨끗 한 공간을 만들. DNA 템플렛의 오염이 의심 되 면 철저 하 게 공간 청소 및/또는 사용된 시 약을 삭제. 대조적으로, 참조 대립 유전자의 긍정적인 파티션을 gDNA 존재 검색 되지 않습니다 경우 gDNA, 뇌관, 및 프로브 농도 재확인. 특히, 뇌관은 기존 PCR (표 1)12에 비해 대표적인 실험에서 낮은 농도에서 사용 되었다. 그것은 또한 기존의 PCR에 변경 거의 경사로 속도 확인 하는 중요 한입니다.
DPCR 칩에서 튜브 형식으로 고유 하 게 보편적인 8 튜브 스트립8,13안에 PCR 혼합물의 분할 이다. PCR 튜브로 분할 챔버의 이동이이 시스템에서 필요 하지 않습니다 따라서 오염 위험을 감소. 거기에 또한 이점을 칩에 튜브 형식; dPCR 시스템 그것은 적어도 5 h 드롭릿 기반 dPCR14에 분석 실험의 동일한 수를 완료 하는 동안 해당 dPCR 시스템에서 96 분석 완료 < 4 h 걸립니다. 또한, 범용 8 튜브 스트립 다른 칩 기반 dPCR15, 평면 블록 열 cycler는 달리 기존의 열 cycler의 사용을 수 있습니다. 또한, 축적 된 지식과 기존의 PCR 위한 시 약 dPCR 시스템에 적용할 수 있습니다. 이 dPCR의 애플 리 케이 션을 확장에 대 한 도움이 될 것입니다.
그 dPCR는 몇 가지 제한이 주목 한다. 칩에 튜브 형식으로 dPCR에 칩의 파티션 번호 다른 dPCR 플랫폼에 비해 상대적으로 낮은입니다. 추가 개선 칩 소자에 파티션 수에 따라 동적 범위를 증가 해야 합니다. 보편적인 튜브의 내부 공간 제한 때문에, 칩 소자에 대 한 획기적인 디자인 파티션 수를 증가 해야 합니다. 나중에 전체 dPCR 절차의 자동화 크게 훨씬 더 신뢰할 수 있는 결과에, 인간의 오류를 줄일 것 이다. 성격 때문에 그것의 높은 처리량 및 높은 감도, 칩에서 튜브 형식으로 dPCR 액체 biopsies을 환경 DNA에 대 한 샘플의 수를 치료에 적용 될 예정 이다.
공개
저자는 공개 없다.
감사의 말
이 연구는 일본 사회에는 프로 모션의 과학 (JSP) (No. 15 K 08397)에서 토모 Kahyo에 Scientific Research (C)에 대 한 특정 및 의료 연구 및 개발 (아메드) (No. 927960719), 일본 기관에서 교부 금에 의해 지원 되었다 탐색적 연구 (제 16 K 15256), 관리 비용 보조금 (No. 1019253), 그리고 흡연 연구 재단에서 국립 대학 개혁의 추진에 대 한 인센티브 특별 예산 관련된 프로젝트에 대 한 지출에 대 한 특정 Haruhiko Sugimura입니다. 저자는 그들의 임상 지원 박사 이와 이즈미와 박사 Kurachi을 감사합니다. Funders 원고 준비에 아무런 역할을 했다. 그림 3, 그림 4, 표 4 적응 그리고 Kahyo 그 외 여러분 에 의해 기사에서 증 쇄 10, Elsevier에서 허가입니다.
자료
| Name | Company | Catalog Number | Comments |
| QIAamp DNA Blood Maxi Kit | Qiagen | 51194 | Before Protocol 1: Extraction of genomic DNA |
| NanoDrop 1000 | ThermoFisher SCIENTIFIC | ND-8000 | Before Protocol 1: Spectrophotometer |
| 8-strip tube | Agilent Technologies | 401428 | Protocol 1 |
| Genomic DNA sample buffer | Agilent Technologies | 5067-5366 | Protocol 1: A component of Genomic DNA Screen Tape Assay |
| DNA ladder | Agilent Technologies | 5067-5366 | Protocol 1: A component of Genomic DNA Screen Tape Assay |
| MS3 Basic Small Shaker | IKA | 3617000 | Protocol 1: Vortex mixer |
| Genomic DNA ScreenTape | Agilent Technologies | 5067-5365 | Protocol 1: Gel device |
| 2200 TapeStation system | Agilent Technologies | G2965AA | Protocol 1: Electrophoresis instrument |
| TapeStation Analysis Software | Agilent Technologies | Bundled with G2965AA | Protocol 1: Analysis software |
| DNA oligo primers | IDT | Custom order | Protocol 2 |
| LNA probes | IDT | Custom order | Protocol 2 |
| Software tool | IDT | Web site: http://biophysics.idtdna.com/ | Protocol 2 |
| dbSNP database | NCBI | Web site: https://www.ncbi.nlm.nih.gov/projects/SNP/ | Protocol 2 |
| TE buffer | ThermoFisher SCIENTIFIC | 12090015 | Protocol 3 |
| DNase/RNase-Free Distilled Water | ThermoFisher SCIENTIFIC | 10977015 | Protocol 3 (Table 1) |
| Clarity Digital PCR Probe Mastermix | JN Medsys | 12013 | Protocol 3 (Table 1): 2xPCR Master Mix A component of #10011 |
| Clarity Sealing Fluid | JN Medsys | 12005 | Protocol 3: Sealing fluid A component of #10011 |
| Clarity JN Solution | JN Medsys | 12006 | Protocol 3 (Table 1): 20xdPCR solution A component of #10011 |
| Clarity Tube-strip | JN Medsys | 12007 | Protocol 3: Chip-in-a-tube A component of #10011 |
| Clarity Sample Loading Kit | JN Medsys | 12008 | Protocol 3: Loading platform and slider A component of #10011 |
| Clarity Auto Loader | JN Medsys | 11002 | Protocol 3: Auto loader A component of #10001 |
| Clarity Sealing Enhancer | JN Medsys | 11003 | Protocol 3: Sealing enhancer A component of #10001 |
| Clarity Reader | JN Medsys | 11004 | Protocol 3: Reader A component of #10001 |
| Life Eco Thermal Cycler | Bioer Technology | TC-96GHbC | Protocol 3: Thermal cycler |
| Clarity Software | JN Medsys | Bundled with #10001 | Protocol 3: Analysis software |
| High Sensitivity D1000 screen tape | Agilent Technologies | 5067-5584 | Protocol 4: Gel device for high sensitivity |
참고문헌
- Majumdar, N., Wessel, T., Marks, J. Digital PCR modeling for maximal sensitivity, dynamic range and measurement precision. PLoS One. 10 (3), 0118833 (2015).
- Huggett, J. F., et al. The digital MIQE guidelines: Minimum Information for Publication of Quantitative Digital PCR Experiments. Clinical Chemistry. 59 (6), 892-902 (2013).
- Chen, L., Liu, P., Evans, T. C., Ettwiller, L. M. DNA damage is a pervasive cause of sequencing errors, directly confounding variant identification. Science. 355 (6326), 752-756 (2017).
- Alix-Panabières, C., Pantel, K. Clinical Applications of Circulating Tumor Cells and Circulating Tumor DNA as Liquid Biopsy. Cancer Discovery. 6 (5), 479-491 (2016).
- Kiss, M. M., et al. High-throughput quantitative polymerase chain reaction in picoliter droplets. Analytical Chemistry. 80 (23), 8975-8981 (2008).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Analytical Chemistry. 83 (22), 8604-8610 (2011).
- Ottesen, E. A., Hong, J. W., Quake, S. R., Leadbetter, J. R. Microfluidic digital PCR enables multigene analysis of individual environmental bacteria. Science. 314 (5804), 1464-1467 (2006).
- Low, H., Chan, S. J., Soo, G. H., Ling, B., Tan, E. L. Clarity™ digital PCR system: a novel platform for absolute quantification of nucleic acids. Analytical and Bioanalytical Chemistry. 409 (7), 1869-1875 (2017).
- Iwaizumi, M., et al. A novel APC mosaicism in a patient with familial adenomatous polyposis. Human Genome Variation. 2, 15057 (2015).
- Kahyo, T., et al. Application of digital PCR with chip-in-a-tube format to analyze Adenomatous polyposis coli (APC) somatic mosaicism. Clinica Chimica Acta. 475, 91-96 (2017).
- Kanai, Y., et al. The Japanese Society of Pathology Guidelines on the handling of pathological tissue samples for genomic research: Standard operating procedures based on empirical analyses. Pathology International. 68 (2), 63-90 (2018).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Cao, L., et al. Advances in digital polymerase chain reaction (dPCR) and its emerging biomedical applications. Biosensors and Bioelectronics. 90, 459-474 (2017).
- . Bio-Rad Laboratories Available from: https://www.bio-rad.com/webroot/web/pdf/Isr/literature/Bulletin_6311.pdf (2018)
- . Thermo Fisher Scientific Available from: https://www.garvan.org.au/research/capabilities/molecular-genetics/documents/qs-3d-user-guide.pdf (2013)
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유