JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
침입 식물에 대한 컴퓨터 비전 기반 바이오매스 추정
요약
우리는 무인 항공기(UAV) 원격 감지에서 얻은 데이터를 활용하여 바이오매스를 평가하고 침입종의 공간 분포를 캡처하는 침입 식물 바이오매스 추정 방법에 대한 자세한 절차를 보고합니다. 이 접근 방식은 침입 식물에 대한 위험 평가 및 조기 경고를 수행하는 데 매우 유용한 것으로 입증되었습니다.
초록
우리는 UAV 원격 감지 및 컴퓨터 비전을 기반으로 침입 식물의 바이오매스를 추정하는 방법의 자세한 단계를 보고합니다. 연구 영역에서 샘플을 수집하기 위해 샘플링 점을 무작위화하기 위해 샘플 정사각형 어셈블리를 준비했습니다. 드론과 카메라를 이용한 무인 항공 카메라 시스템을 구축하여 자동 항법을 통해 연구 지역의 연속 RGB 이미지를 획득했습니다. 촬영이 완료된 후 샘플 프레임의 지상 바이오매스를 수집하고 모든 서신에 라벨을 붙이고 포장했습니다. 샘플 데이터를 처리하고 항공 이미지를 280 x 280 픽셀의 작은 이미지로 분할하여 이미지 데이터 세트를 만들었습니다. 심층 컨볼루션 신경망을 사용하여 연구 지역에서 Mikania micrantha 의 분포를 매핑하고 식생 지수를 얻었습니다. 수집된 유기체는 건조되었고, 건조 중량은 실측 생물량으로 기록되었습니다. 침입 식물 바이오매스 회귀 모델은 샘플 이미지에서 식생 지수를 독립 변수로 추출하고 이를 종속 변수로 실측 바이오매스와 통합하여 K-최근접 이웃 회귀(KNNR)를 사용하여 구성되었습니다. 그 결과 침입 식물의 바이오매스를 정확하게 예측할 수 있음을 보여주었습니다. 이미지 순회를 통해 침입 식물 바이오매스의 정확한 공간 분포 맵이 생성되어 침입 식물의 영향을 받는 고위험 지역을 정확하게 식별할 수 있었습니다. 요약하면, 이 연구는 무인 항공기 원격 감지와 기계 학습 기술을 결합하여 침입 식물 바이오매스를 추정할 수 있는 잠재력을 보여줍니다. 침입 식물의 실시간 모니터링을 위한 새로운 기술 및 방법 연구에 크게 기여하고 있으며 지역 규모에서 지능형 모니터링 및 위험 평가를 위한 기술 지원을 제공합니다.
서문
이 프로토콜에서 제안된 UAV 원격 감지 및 컴퓨터 비전을 기반으로 한 침습적 바이오매스 추정 방법은 침입 유기체의 분포를 반영하고 침습적 생물학적 위험의 정도를 예측할 수 있습니다. 침입 유기체의 분포 및 생물량에 대한 추정치는 이러한 유기체의 예방 및 통제에 매우 중요합니다. 외래 식물이 침입하면 생태계를 손상시키고 막대한 경제적 손실을 초래할 수 있습니다. 침입 식물을 빠르고 정확하게 식별하고 주요 침입 식물 바이오매스를 추정하는 것은 침입 식물 모니터링 및 제어의 주요 과제입니다. 이 프로토콜에서는 Mikania micrantha 를 예로 들어 무인 항공 원격 감지 및 컴퓨터 비전을 기반으로 한 침입 식물 바이오매스 추정 방법을 탐색하여 침입 식물의 생태 연구를 위한 새로운 접근 방식과 방법을 제공하고 침입 식물의 생태 연구 및 관리를 촉진합니다.
현재 Mikania micrantha 의 바이오매스 측정은 주로 수동 샘플링으로 수행됩니다1. 전통적인 바이오매스 측정 방법은 많은 인력과 물적 자원이 필요하며, 이는 비효율적이고 지형에 의해 제한됩니다. Mikania micrantha 의 지역 바이오매스 추정 요구를 충족시키는 것은 어렵습니다.이 프로토콜 사용의 주요 이점은 지역의 샘플링 제한을 고려하지 않고 수동 조사의 필요성을 제거하는 방식으로 지역 침입 식물 바이오매스 및 침입 식물의 공간 분포를 정량화하는 방법을 제공한다는 것입니다.
UAV 원격 감지 기술은 식물 바이오매스 추정에서 확실한 결과를 얻었으며 농업 2,3,4,5,6,7, 임업 8,9,10,11 및 초원 12,13,14에서 널리 사용되었습니다.. UAV 원격 감지 기술은 저비용, 고효율, 고정밀 및 유연한 작동(15,16)의 장점이 있어 연구 영역에서 원격 감지 이미지 데이터를 효율적으로 얻을 수 있습니다. 그런 다음, 원격 탐사 이미지의 질감 특징과 식생 지수를 추출하여 넓은 지역의 식물 바이오매스를 추정하기 위한 데이터를 지원합니다. 현재의 식물 바이오매스 추정 방법은 주로 파라메트릭 모델과 비파라메트릭 모델로 분류된다17. 기계 학습 알고리즘의 개발로 더 높은 정확도의 비모수적 기계 학습 모델이 식물 바이오매스의 원격 감지 추정에 널리 사용되었습니다. Chen et al.18은 MLR(Mixed Logistic Regression), KNNR 및 RFR(Random Forest Regression)을 사용하여 윈난성 산림의 지상 바이오매스를 추정했습니다. 그들은 기계 학습 모델, 특히 KNNR 및 RFR이 MLR에 비해 우수한 결과를 가져왔다는 결론을 내렸습니다. Yan et al.19은 다양한 변수 세트를 사용하여 아열대림 바이오매스 추정의 정확성을 평가하기 위해 RFR 및 XGBR(Extreme Gradient Boosting) 회귀 모델을 사용했습니다. Tian et al.20은 11개의 기계 학습 모델을 활용하여 Beibuwan Bay의 다양한 맹그로브 숲 종의 지상 생물량을 추정했습니다. 연구진은 XGBR 방법이 맹그로브 숲의 지상 바이오매스를 결정하는 데 더 효과적이라는 것을 발견했다. 인간-기계 원격 탐사를 사용한 식물 바이오매스 추정은 잘 확립된 관행이지만, 침입 식물 Mikania micrantha의 바이오매스 추정을 위한 UAV 사용은 아직 국내외적으로 보고된 적이 없습니다. 이 접근법은 침입 식물, 특히 Mikania micrantha에 대한 이전의 모든 바이오매스 추정 방법과 근본적으로 다릅니다.
요약하자면, UAV 원격 감지는 고해상도, 고효율 및 저렴한 비용의 장점이 있습니다. 원격 탐사 이미지의 특징 변수 추출에서 식생 지수와 결합된 텍스처 특징은 더 나은 회귀 예측 성능을 얻을 수 있습니다. 비모수 모델은 식물 바이오매스 추정에서 모수적 모델보다 더 정확한 회귀 모델을 얻을 수 있습니다. 따라서 침입 식물과 그 바이오매스의 null 분포를 정확하게 계산하기 위해 UAV 및 컴퓨터 비전을 사용한 원격 감지에 의존하는 침입 식물 바이오매스 실험에 대해 다음과 같은 요약된 절차를 제안합니다.
프로토콜
1. 데이터셋 준비
- 연구 대상 선택
- Mikania micrantha 또는 기타 침입 식물과 같은 옵션을 고려하여 실험 연구의 초점에 따라 테스트 샘플을 선택합니다.
- UAV 이미지 수집
- 연구 영역의 크기에 따라 0.5m*0.5m 크기와 수량 25-50의 정사각형 플라스틱 프레임을 준비합니다.
- 충분한 수의 바이오매스 샘플을 사용하여 연구 지역의 토양 샘플링 위치를 결정하기 위해 무작위 샘플링 접근 방식을 사용합니다. 샘플 프레임을 식물 위에 수평으로 배치하여 각 식물 사이에 최소 2m의 이격 거리로 식물을 완전히 둘러쌉니다.
- 그림 1과 같이 드론과 카메라를 사용하여 UAV 원격 감지 촬영 시스템을 형성합니다.
- UAV를 사용하여 지정된 연구 영역 내의 경로를 플롯합니다. 경로 계획 설정은 그림 2에 나와 있습니다.
- 방향 및 측면 겹침 비율을 70%로 설정하고, 2초의 균일한 시간 간격으로 사진을 캡처하고, 카메라 각도를 지면과 수직으로 90°로 유지하고, 카메라 고도를 30m로 배치합니다. 그 결과 그림 3과 같이 8256 x 5504 픽셀 크기의 단일 이미지 해상도로 연구 영역의 연속적인 가시 이미지 데이터가 생성되었습니다.
- 바이오매스 추정을 위해 Python 소프트웨어를 사용하여 후속 처리를 위해 항공 이미지를 저장합니다.
- 지상 바이오매스 수집
- 드론 데이터 수집을 완료한 후 각 샘플 플롯 내에서 Mikania micrantha 의 지상 바이오매스를 수동으로 수집합니다. 가방에 넣고 그에 따라 각 가방에 라벨을 붙입니다.
- Mikania micrantha를 수집할 때 표본 조사구가 움직이지 않도록 하십시오. 먼저 표본 플롯의 안쪽 가장자리를 따라 Mikania micrantha를 자릅니다.
- 그런 다음 Mikania micrantha 의 뿌리 줄기를 바닥에서 자릅니다. 섞여 있는 흙, 바위 또는 기타 식물을 제거하십시오. 마지막으로, 샘플을 봉지에 넣고 라벨을 붙입니다.
- 1.3.1단계에서 수집된 침입 식물 샘플을 실험실로 가져옵니다. 수집된 모든 샘플을 자연 건조하여 대부분의 수분을 증발시킵니다.
- 공기 건조된 샘플에서 수분을 더 제거하려면 오븐을 사용하십시오. 온도를 55°C로 설정합니다. 샘플을 72시간 동안 건조시킨 다음 전자 저울에서 각 샘플의 무게를 측정하고 바이오매스 데이터를 그램(g) 단위로 기록합니다.
- 전자 저울을 방해받지 않는 환경에 놓고 칭량, 교정 및 칭량을 계속하십시오. Mikania micrantha 가방을 전자 저울에 놓고 판독값이 안정화될 때까지 기다렸다가 판독값을 기록합니다.
- 질량이 더 이상 변하지 않을 때까지 매시간 Mikania micrantha 의 무게를 측정하고 판독값에서 백의 무게를 뺀 값을 해당 샘플의 측정된 질량으로 기록합니다. 아래 공식을 사용하여 침입 식물의 지상 바이오매스를 계산하십시오.
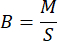
여기서 B 는 Mikania micrantha 의 바이오매스를 평방 미터당 그램(g/m2) 단위로 나타내고, M은 그램(g)으로 측정된 Mikania micrantha의 무게이며, S는 평방 미터 (m2) 단위의 표본 플롯 면적에 해당합니다.
- 드론 데이터 수집을 완료한 후 각 샘플 플롯 내에서 Mikania micrantha 의 지상 바이오매스를 수동으로 수집합니다. 가방에 넣고 그에 따라 각 가방에 라벨을 붙입니다.
- 데이터셋 만들기
- 원본 UAV 이미지에서 샘플 이미지에 해당하는 RGB 이미지를 추출합니다. Python 프로그래밍을 사용하여 280 × 280 픽셀의 그리드로 나눕니다(부록: 그림 1).
- Python 프로그래밍을 사용하여 원시 이미지 데이터를 샘플 이미지와 동일한 크기의 더 작은 이미지로 분할합니다. 분할을 위해 슬라이딩 윈도우 방법을 사용하여 수평 및 수직 단계를 280픽셀로 설정합니다.
- 1.4.2단계에서 분할된 작은 이미지에서 880개의 침입 식물 이미지와 1500개의 배경 이미지를 무작위로 선택하여 데이터 세트를 만듭니다. 그런 다음 이 데이터 세트를 6:2:2 비율로 훈련, 검증 및 테스트 세트로 분할합니다(Supplementray 그림 2).
2. Mikania micrantha의 식별
- 소프트웨어 준비 중
- 아나콘다 공식 웹사이트(https://www.anaconda.com/)로 이동하여 아나콘다를 다운로드하여 설치합니다. 그런 다음 PyCharm 웹 사이트(https://www.jetbrains.com/pycharm/)로 이동하여 PyCharm IDE를 다운로드합니다.
- Conda 환경 만들기.
- Anaconda를 설치한 후 Anaconda Prompt 명령줄을 연 다음 conda create -n pytorch python==3.8 을 입력하여 새 Conda 환경을 만듭니다. 환경을 만든 후 conda info --envs 를 입력하여 pytorch 환경이 있는지 확인합니다.
- Anaconda Prompt를 열고 conda activate pytorch를 입력하여 pytorch 환경을 활성화합니다. nvidia-smi를 입력하여 현재(Compute Unified Device Architecture) CUDA 버전을 확인합니다. 그런 다음 conda install pytorch==1.8.1 torchvision==0.9.1 torchaudio==0.8.1 cudatoolkit=11.0 -c pytorch 명령을 실행하여 PyTorch 버전 1.8.1을 설치합니다.
- 모델 인식을 위한 실행
참고: PyTorch를 사용하여 Mikania micrantha 이 논문에 사용된 인식 모델입니다. 사용된 네트워크 모델은 ResNet101입니다21, 아키텍처에서 원본 종이와 일관성을 유지합니다. 카모마일 인식에 대한 요구 사항을 충족하기 위해 네트워크의 출력 섹션이 수정되었습니다.- 이미지를 전처리하여 모델 입력을 준비합니다. 이미지 크기를 280 x 280 픽셀에서 224 x 224 픽셀로 조정하고 다음 코드를 사용하여 모델의 크기 요구 사항을 충족하도록 정규화합니다.
transform = 변환. 작성([
변환. 크기 조정((224, 224)),
변환. ToTensor()를 호출합니다.
변환. 정규화([0.485, 0.456, 0.406], [0.229, 0.224, 0.225])]) - 컨벌루션 신경망을 사용하여 이미지 특징 추출을 수행하고 차원을 줄일 수 있습니다.
- 먼저, self.conv1을 통해 초기 특징 추출을 위한 컨벌루션 계층을 초기화합니다. 이 컨볼루셔널 레이어를 사용하면 원본 이미지가 초기 특징을 추출하기 위한 self.in_channel개의 채널이 있는 특징 맵으로 컨볼루션됩니다(보충 그림 3A).
참고: 고급 특징은 잔차 가공 패스에 대한 컨볼루션 연산에서 추출됩니다. 이러한 계층은 일련의 잔차 블록으로 구성된 _make_layer 함수를 호출하여 생성됩니다. 각 잔차 블록은 컨볼루션(convolution), 배치 정규화(batch normalization) 및 활성화 함수(activation function)로 구성되어 정교한 특징을 점진적으로 추출합니다(보충 그림 3B). - 계층의 기능을 사용하여 1x1 컨볼루션을 통해 차원 축소를 위한 채널 번호를 수정합니다. 이 작업은 중요한 기능을 유지하면서 계산 부하를 줄입니다(보충 그림 3C).
참고: 전반적으로 ResNet101은 다양한 컨볼루셔널 레이어를 사용하여 기능 추출을 수행하며, 잔차 블록 내의 1x1 컨볼루셔널 레이어를 통해 차원 축소가 이루어집니다. 이 접근 방식을 사용하면 신경망이 특징을 더 깊이 학습하고 기울기 소실 문제를 피할 수 있으므로, 복잡한 작업을 위해 영상 특징을 더 효율적으로 학습하고 표현할 수 있습니다. - 컨벌루션(convolutions)과 풀링(pooling) 연산 후에는 고품질 특징을 완전 연결 계층에 입력합니다.
참고: ResNet 아키텍처에서 특징 추출은 컨볼루셔널 계층에서 발생합니다. 이러한 특징은 이후에 분류를 위해 완전히 연결된(FC) 계층으로 전송됩니다(보충 그림 4). self.avgpool(x) 연산은 적응 형 평균 풀링을 수행하여 텐서를 고정 된 크기로 재구성합니다. torch.flatten(x, 1) 연산은 텐서를 1차원 벡터로 확산시키고, self.fc(x) 는 완전 연결 계층을 평면 벡터에 적용하여 궁극적으로 분류의 마지막 단계 역할을 합니다. 이 프로세스는 추출된 특징을 컨볼루션 계층을 통해 효과적으로 전달하여 완전 연결 계층을 통해 분류에 적합한 형식으로 변환합니다. - Softmax 함수를 사용하여 세 가지 분류 요구 사항에 따라 최종 출력값을 얻을 수 있습니다.
- 먼저, self.conv1을 통해 초기 특징 추출을 위한 컨벌루션 계층을 초기화합니다. 이 컨볼루셔널 레이어를 사용하면 원본 이미지가 초기 특징을 추출하기 위한 self.in_channel개의 채널이 있는 특징 맵으로 컨볼루션됩니다(보충 그림 3A).
- 1.4단계의 데이터 세트를 사용하여 다중 클래스 인식 모델을 학습합니다. 반복 횟수를 200으로 설정하고 초기 학습률을 0.0001로 설정합니다. 배치 크기가 64인 경우 10회 반복할 때마다 학습률을 1/3씩 줄입니다. 각 반복 후에 최적의 모델 매개변수를 자동으로 저장합니다(보충 그림 5).
- 꼼꼼하게 훈련된 인식 모델을 사용하고 식별을 위해 1.2.2단계부터 원본 이미지를 체계적으로 탐색합니다.
- 수평 및 수직 단계를 280픽셀로 정확하게 구성하여 연구 영역 경계 내에 침입성 식물군의 존재를 강조하는 포괄적인 분포 지도를 생성합니다. 선택한 결과를 그림 4와 같이 시각적으로 표시합니다.
참고: 초기 이미지는 더 작은 청크로 분할하고, 훈련된 딥 러닝 모델을 사용하여 각 청크를 분류하고, 결과를 출력 이미지로 결합하여 전처리됩니다. 청크가 침입 식물로 분류되는 경우 출력 이미지의 해당 위치는 255로 설정됩니다. 결과 출력 이미지는 회색조 이미지 파일로 저장됩니다. 구체적인 구현 코드는 보충 그림 6에 나와 있습니다.
- 수평 및 수직 단계를 280픽셀로 정확하게 구성하여 연구 영역 경계 내에 침입성 식물군의 존재를 강조하는 포괄적인 분포 지도를 생성합니다. 선택한 결과를 그림 4와 같이 시각적으로 표시합니다.
- 이미지를 전처리하여 모델 입력을 준비합니다. 이미지 크기를 280 x 280 픽셀에서 224 x 224 픽셀로 조정하고 다음 코드를 사용하여 모델의 크기 요구 사항을 충족하도록 정규화합니다.
3. 침입성 식물 바이오매스의 추정
- RandomResizedCrop 및 RandomHorizontalFlip 함수(보충 그림 7)로 간단한 데이터 증강을 수행하여 1.2단계에서 생성된 이미지 세트를 확장하고 바이오매스 추정에 일반적으로 사용되는 6가지 식생 지수(RBRI, GBRI, GRRI, RGRI, NGBDI 및 NGRDI)를 추출합니다. 이러한 지수에 대한 계산 공식에 대해서는 표 1을 참조하십시오.
- 모델의 출력을 사용하여 K-최근접 이웃 회귀(KNNR)22 모델을 생성하여 침입 식물의 바이오매스를 정확하게 추정할 수 있습니다. 추출된 식생 지수를 추정 모델의 입력값으로 사용합니다.
- 결정 계수 R2 및 RMSE(Root Mean Square Error)23 을 사용하여 다음과 같이 계산되는 모델의 정확도를 평가합니다.
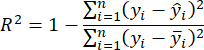
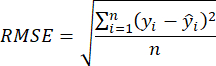
참고: KNNR(K-Nearest Neighbor Regression) 알고리즘은 회귀 문제를 해결하는 데 사용되는 비모수적 기계 학습 기술입니다. 기본 개념은 입력 샘플 거리를 기반으로 기능 공간에서 가장 가까운 K 이웃을 결정하여 결과를 예측하는 것입니다. KNNR 사용의 주요 이점은 단순성과 이해의 용이성이며 교육 단계가 필요하지 않다는 것입니다. 또한 KNNR은 데이터 분포에 대해 과도한 가정을 하지 않습니다. KNNR은 연속적인 객관적 변수를 예측하고 침입 식물의 바이오매스를 정확하게 평가하기 위해 회귀 문제에 적용할 수 있습니다. - 3.2단계에서 선택한 지상 바이오매스 추정 모델을 사용하고 2.3.4단계의 침입 식물 분포 맵을 280픽셀의 수평 및 수직 보폭으로 스캔합니다.
결과
우리는 컴퓨터에서 프로그래밍 방식으로 구현되는 침입 식물 추정을 위한 컴퓨터 비전 기반 방법의 대표적인 결과를 보여줍니다. 이 실험에서는 Mikania micrantha 를 연구 주제로 사용하여 자연 서식지에서 침입 식물의 공간 분포를 평가하고 바이오매스를 추정했습니다. 우리는 드론 카메라 시스템을 활용하여 연구 현장의 이미지를 획득했으며, 그 중 일부는 그림 3에 전...
토론
UAV 원격 감지 및 컴퓨터 비전을 사용하여 침입 식물의 바이오매스를 추정하는 실험의 자세한 단계를 제시합니다. 이 계약의 주요 프로세스와 단계는 그림 7에 나와 있습니다. 적절한 시료 품질은 프로그램에서 가장 중요하고 어려운 측면 중 하나입니다. 이러한 중요성은 다른 식물 바이오매스 추정 실험뿐만 아니라 모든 침입 식물에도 적용된다24.
공개
저자는 밝힐 것이 없습니다.
감사의 말
저자는 이 연구를 지원해준 중국농업과학원(Chinese Academy of Agricultural Sciences)과 광시대학교(Guangxi University)에 감사를 표한다. 이 연구는 중국 국가 핵심 R&D 프로그램(2022YFC2601500 및 2022YFC2601504), 중국 국가자연과학재단(32272633), 선전 과학기술프로그램(KCXFZ20230731093259009)의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| DSLR camera | Nikon | D850 | Sensor type: CMOS; Maximum number of pixels: 46.89 million; Effective number of pixels: 45.75 million; Maximum resolution 8256 x 5504. |
| GPU - Graphics Processing Unit | NVIDIA | RTX3090 | |
| Hexacopter | DJI | M600PRO | Horizontal flight: 65 km/h (no wind environment); Maximum flight load: 6000 g |
| PyCharm | Python IDE | 2023.1 | |
| Python | Python | 3.8.0 | |
| Pytorch | Pytorch | 1.8.1 |
참고문헌
- Lian, J. Y., et al. Influence of obligate parasite Cuscuta campestris on the community of its host Mikania micrantha. Weed Research. 46, 441-443 (2006).
- Yu, X., Bao, Q. Aboveground biomass estimation of potato from UAV multispectral imagery. Remote Sensing. 54 (4), 96-99 (2023).
- Guo, T. C., Wang, Y. H., Feng, W. Research on wheat yield estimation based on multimodal data fusion from unmanned aircraft platforms. Acta Agronomica Sinica. 48 (7), 15 (2022).
- Shao, G. M., et al. Estimation of transpiration coefficient and aboveground biomass in maize using time-series UAV multispectral imagery. The Crop Journal. 10 (5), 1376-1385 (2022).
- Jiang, Q., et al. UAV-based biomass estimation for rice-combining spectral, TIN-based structural and meteorological features. Remote Sensing. 11 (7), 890 (2019).
- Fei, S. P., et al. UAV-based multi-sensor data fusion and machine learning algorithm for yield prediction in wheat. Precision Agriculture. 24, 187-212 (2023).
- Shu, S., et al. Aboveground biomass estimation of rice based on unmanned aerial vehicle imagery. Fujian Journal of Agricultural Sciences. 37 (7), 9 (2022).
- Wu, X., et al. UAV LiDAR-based biomass estimation of individual trees. Fujian Journal of Agricultural Sciences. 22 (34), 15028-15035 (2022).
- Yang, X., Zan, M., Munire, M. Estimation of above ground biomass of Populus euphratica forest using UAV and satellite remote sensing. Transactions of the Chinese Society of Agricultural Engineering. 37 (1), 7 (2021).
- Li, B., Liu, K. Forest biomass estimation based on UAV optical remote sensing. Forest Engineering. 5, 38 (2022).
- Li, Z., Zan, Q., Yang, Q., Zhu, D., Chen, Y., Yu, S. Remote estimation of mangrove aboveground carbon stock at the species level using a low-cost unmanned aerial vehicle system. Remote Sensing. 11 (9), 1018 (2019).
- Luo, S., et al. Fusion of airborne LiDAR data and hyperspectral imagery for aboveground and belowground forest biomass estimation. Ecological Indicators. 73, 378-387 (2017).
- Li, S., et al. Research of grassland aboveground biomass inversion based on UAV and satellite remoting sensing. Remote Sensing Technology and Application. 1, 037 (2022).
- Wengert, M., et al. Multisite and multitemporal grassland yield estimation using UAV-borne hyperspectral data. Remote Sensing. 14 (9), 2068 (2022).
- Li, Y., et al. The effect of season on Spartina alterniflora identification and monitoring. Frontiers in Environmental Science. 10, 1044839 (2022).
- Wang, F., et al. Estimation of above-ground biomass of winter wheat based on consumer-grade multi-spectral UAV. Remote Sensing. 14 (5), 1251 (2022).
- Lu, N., et al. Improved estimation of aboveground biomass in wheat from RGB imagery and point cloud data acquired with a low-cost unmanned aerial vehicle system. Plant Methods. 15 (1), 17 (2019).
- Chen, H., et al. Mapping forest aboveground biomass with MODIS and Fengyun-3C VIRR imageries in Yunnan Province, Southwest China using linear regression, K-nearest neighbor and random. Remote Sensing. 14 (21), 5456 (2022).
- Yan, M., et al. Biomass estimation of subtropical arboreal forest at single tree scale based on feature fusion of airborne LiDAR data and aerial images. Sustainability. 15 (2), 1676 (2023).
- Tian, Y. C., et al. Aboveground mangrove biomass estimation in Beibu Gulf using machine learning and UAV remote sensing. Science of the Total Environment. 781, 146816 (2021).
- Shrivastava, A., et al. Beyond skip connections: Top-down modulation for object detection. arXiv. , (2016).
- Belkasim, S. O., Shridhar, M., Ahmadi, M. Pattern classification using an efficient KNNR. Pattern Recognition. 25 (10), 1269-1274 (1992).
- Joel, S., Jose Luis, A., Shawn, C. K. Farming and earth observation: Sentinel-2 data to estimate within-field wheat grain yield. International Journal of Applied Earth Observation and Geoinformation. 107, 102697 (2022).
- Tian, L., et al. Review of remote sensing-based methods for forest aboveground biomass estimation: Progress, challenges, and prospects. Forests. 14 (6), 1086 (2023).
- Wei, X. Biomass estimation: A remote sensing approach. Geography Compass. 4 (11), 1635-1647 (2010).
- Débora, B., et al. New methodology for intertidal seaweed biomass estimation using multispectral data obtained with unoccupied aerial vehicles. Remote Sensing. 15 (13), 3359 (2023).
- Zhang, J. Y., et al. Unmanned aerial system-based wheat biomass estimation using multispectral, structural and meteorological data. Agriculture. 13 (8), 1621 (2023).
- Shen, H., et al. Influence of the obligate parasite Cuscuta campestris on growth and biomass allocation of its host Mikania micrantha. Journal of Experimental Botany. 56 (415), 1277-1284 (2005).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유