Method Article
메탄-산소 카운터 그래디언트에서 메탄영양생물을 배양하기 위한 아가로스 기반 모델 생태계
요약
호기성 메탄 산화 박테리아의 자연 서식지에서 발견되는 메탄-산소 카운터 구배를 재현하는 간단한 모델 생태계를 준비하기 위한 프로토콜이 설명되어 있으며, 이를 통해 공간적으로 해결된 맥락에서 박테리아의 생리학을 연구할 수 있습니다. 아가로스 기반 모델 에코시스템과 함께 사용하기 위한 일반적인 생화학 분석에 대한 수정 사항도 설명되어 있습니다.
초록
메탄영양생물(methanotrophs)로 알려진 호기성 메탄 산화 박테리아는 생지화학적 순환에서 중요한 역할을 합니다. 메타영양생물은 토양과 퇴적물에서 발견되는 메탄-산소 카운터 구배 내에서 특정 환경적 틈새를 차지하며, 이는 개인 및 커뮤니티 수준에서 그들의 행동에 영향을 미칩니다. 그러나 이러한 온실 가스 완화 미생물의 생리학을 연구하는 기존 방법은 종종 균질 플랑크톤 배양을 사용하며, 이는 환경에서 발견되는 공간적, 화학적 구배를 정확하게 나타내지 않습니다. 이것은 이 박테리아가 현장에서 어떻게 행동하는지에 대한 과학자들의 이해를 방해합니다. 여기에서는 그래디언트 시린지(gradient syringe)라고 하는 간단하고 저렴한 모델 생태계에 대해 설명하며, 이는 반고체 아가로스를 사용하여 메탄영양생물의 자연 서식지의 특징적인 가파른 메탄-산소 카운터 그래디언트를 재현합니다. 그래디언트 시린지를 사용하면 메탄영양 균주를 배양하고 환경 시료에서 혼합 메탄 산화 컨소시엄을 강화할 수 있어 공간적으로 분해된 컨텍스트에서만 볼 수 있는 표현형을 드러낼 수 있습니다. 이 프로토콜은 또한 반고체 아가로스 매트릭스와 호환되도록 수정된 다양한 생화학적 분석을 보고하며, 이는 다른 아가로스 기반 시스템 내에서 미생물을 배양하는 연구자에게 유용할 수 있습니다.
서문
무산소-산소 계면에 사는 미생물은 종종 중요한 생태학적 역할을 수행합니다1. 한 가지 예는 호기성 메탄 산화 박테리아(methanotrophs)로, 토양과 퇴적물에서 메탄과 산소의 반대 구배에 존재한다2. 이 미생물은 환경에 존재하는 가스 구배를 이용할 수 있는 고유한 대사 및 생리학적 특성을 가지고 있으며 수십 년 동안 지속적인 연구의 대상이었습니다 3,4,5. 현재 메탄영양생물 및 메탄 산화 군집에 대해 발표된 대부분의 연구는 자연 미생물 서식지에 내재된 공간적, 화학적 구배를 포착하지 못하는 균질한 플랑크톤 배양에 대한 연구를 기반으로 합니다. 이러한 한계는 미생물 생리학에 대한 이해와 게놈 정보를 표현형 형질에 연결하는 능력을 방해합니다.
이 프로토콜은 Methylomonas sp. 균주 LW13과 같은 특정 메탄영양체와 환경 토양 샘플에서 직접 메탄 산화 군집을 연구하기 위한 재현 가능한 조건을 생성하는 간단한 실험실 기반 모델 에코시스템을 보고합니다. 중요한 것은, 그래디언트 주사기에서 배양하면 균질한 플랑크톤 배양6에는 존재하지 않는 역그래디언트 특이적 표현형이 생성되어 메탄영양 생리학의 새로운 측면을 밝힐 수 있는 시스템의 능력을 강조한다는 것입니다. 이전에 발표된 모델 생태계 7,8,9에서 영감을 받은 그래디언트 시린지는 이 접근 방식을 사용하여 배양된 미생물에서 화학 및 분자 정보를 수집하는 데 사용할 수 있는 간소화된 방법입니다.
유전학, 화학 및 분자 분석을 위해 보고된 절차는 반고체 아가로스 매트릭스 내에서 성장한 미생물 배양에서 신뢰할 수 있도록 수정되었습니다. 이러한 절차는 또한 박테리아 연질 한천 수영 분석에 사용되는 것과 같은 다른 반고체 아가로스 기반 시스템에서 성장한 박테리아를 분석하는 데 유용할 수 있습니다. 이러한 분석을 공간적으로 해결된 상황에 적용하면 생태학적으로 더 관련성이 높은 환경에서 미생물 생명을 연구할 수 있는 새로운 길이 열릴 수 있습니다.
프로토콜
연구에 사용된 시약 및 장비의 세부 사항은 재료 표에 나열되어 있습니다.
1. 그래디언트 주사기의 준비 및 압출
알림: 그래디언트 주사기 준비는 멸균 기술을 사용하여 수행해야 합니다.
- 플레이트에서 갓 자란 Methylomonas sp. LW13의 여러 콜로니를 사용하여 18mm x 150mm 유리관에 6mL의 질산염 무기염(NMS) 배지를 접종합니다. 혈청 스토퍼와 알루미늄 압착 씰로 튜브를 밀봉하고 주사기를 사용하여 공기 중 메탄의 50%(v/v) 메탄이 최종 대기에 추가됩니다. 이 플랑크톤 액체 배양액을 실온에서 200rpm으로 탁해질 때까지(약 1일) 흔듭니다.
- 액체 배양액을 1:10으로 신선한 배지로 통과시킵니다. 메탄영양생물의 액체 배양을 log-phase growth (OD600 of ~0.5)로 계속 성장시키고 OD600 =1.0으로 조정합니다.
- 함께 제공된 플런저를 제거하고 멸균 용기에 보관하여 주사기를 준비합니다. 멸균 PTFE 필터 팁을 주사기에 부착하고 팁이 아래를 향하도록 하여 표준 시험관 랙에 넣습니다.
- 각 10mL 주사기에 대해 1.2단계의 세포 1mL를 NMS 5mL 및 용융 아가로스 4mL(0.5% m/v, 55°C로 냉각)와 완전히 혼합합니다. 이러한 부피는 여러 주사기를 병렬로 채우도록 확장할 수 있습니다.
- 혈청학적 피펫을 천천히 붓거나 사용하여 혼합물을 각 주사기에 최대 8mL 표시까지 추가합니다. 주사기 내의 아가로스가 응고되도록 두고(~15분) 멸균 20mm 고무 부틸 마개로 캡을 씌웁니다. 실험실 테이프를 사용하여 스토퍼를 주사기에 고정하고 주사기 내용물로 라벨을 붙입니다.
- 주사기 헤드스페이스에 메탄을 추가하려면 대형(60mL) 주사기에 100% CH4 를 채우고 멸균 바늘(23G)에 연결된 PTFE 필터 팁(0.2μm, 25mm)을 부착합니다. 큰 주사기로 고무 마개를 뚫고 마개를 통해 두 번째 멸균 바늘을 뚫어 가스 배출구를 만듭니다.
- 대형 주사기의 플런저를 눌러 20mL의 100% CH4 가 헤드 공간을 통해 플러시되도록 하고, 대형 주사기에 1-2mL의 CH4 가 남아 있을 때 출구 바늘을 제거하여 출구 바늘을 통한 산소 역류를 방지합니다.
- 18°C에서 주사기를 배양하고 매일 1.6 및 1.7 단계를 반복하여 메탄을 보충합니다.
- 아가로스를 압출하려면 PTFE 필터 팁을 멸균 23G 바늘로 교체하고 고무 마개를 제공된 주사기 플런저로 교체합니다. 플런저를 천천히 눌러 1mL 단위로 별도의 멸균 1.5mL 미세 원심분리기 튜브에 분주합니다.
2. 카운터 그래디언트 가스 농도 측정
- 용존 산소 구배 측정
- 면도날을 사용하여 PTFE 필터 가까이에 있는 아가로스 충전 주사기(1.1-1.8단계에 따라 준비)의 너비를 자릅니다. 열린 끝부분이 전극 끝을 향하도록 Clark형 미세전극 방향의 주사기 펌프에 열린 주사기를 고정합니다.
- 주사기 펌프의 설정을 조정하여 주사기를 1mL/min(0.6cm/min)의 속도로 미세 전극 쪽으로 이동합니다. 주사기 펌프가 움직이기 시작하는 즉시 Unisense Logger 소프트웨어에서 용존 산소 측정값을 기록하기 시작합니다.
- 메탄 구배 측정
- 압출 직전에 주사기 필터 팁을 23G 바늘에 연결된 단방향 꼭지로 교체하고 고무 마개를 주사기 플런저로 빠르게 교체합니다. 8개의 1mL 아가로스 분취액을 추가하여 배출된 12mL 기밀 바이알을 분리하고 샘플이 실온에서 1시간 동안 평형을 이루도록 합니다.
- 바이알을 깨서 열고 즉시 다시 밀봉하거나 바늘을 뚫고 빠르게 제거하여 샘플 바이알을 대기압과 평형화합니다. 기밀 주사기를 사용하여 화염 이온화 감지(GC-FID) 기능이 있는 가스 크로마토그래프에 500μL의 바이알 헤드스페이스를 주입합니다. CH4 표준물질에서 파생된 검량선을 생성하여 피크 면적(pA*min)을 μmol/L로 변환합니다.
3. 그래디언트 주사기의 세포 계수
- 유세포 분석
- 1.9단계에서 설명한 대로 야생형 또는 돌연변이 LW13을 접종한 그래디언트 주사기에서 1mL의 아가로스 세그먼트를 압출합니다. 또한 무세포, 멸균 주사기에서 아가로스를 음성 대조군으로 준비하고 압출합니다.
- 0.75mL의 0.85%(m/v) NaCl을 물에 넣고 모든 압출된 아가로스 샘플에 넣고 볼텍싱으로 균질화합니다. 또한 100μL를 새 마이크로 원심분리 튜브에 옮기고 900μL의 염 용액을 추가하여 샘플을 1:10으로 희석합니다.
- SYTO9와 프로피듐 요오드화물 염색제의 1:1 혼합물 3μL를 첨가한 다음 어두운 곳에서 실온에서 15분 동안 배양합니다. 아가로스의 mL당 세포를 측정하려면 마이크로스피어 카운팅 비드 현탁액을 수조에서 5분 동안 초음파 처리합니다. 그런 다음 유세포 분석 전에 각 샘플에 10μL의 현탁액을 추가합니다.
- 다음 매개변수를 사용하여 유세포 분석기10,11로 시료 분석: 녹색 형광 트리거링, 10 μL/s의 유속 및 1,000 particles/s 미만의 입자 분석 속도.
- SSC 와 비교 FITC 점도표는 cell-free 대조군 샘플과 접종된 아가로스 샘플 사이의 점을 그려 배경 아가로스 입자를 배제하는 "박테리아 이벤트" 전압 게이트를 그립니다. 또한 마이크로스피어 카운팅 비드에 대한 전압 게이트를 그리며, 이는 샘플 간에 일관성이 있어야 합니다.
- 그래디언트 주사기 내의 각 아가로스 세그먼트에 있는 세포의 농도를 측정하려면 위 프로토콜의 희석 계수가 17.7275이고 10-6mL 가 마이크로스피어 비드 1개의 부피임을 주의하면서 다음 방정식을 사용하십시오.
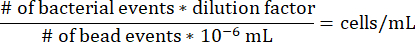
- 그래디언트 주사기 내에서 colony-forming units 계산
- 1mL의 아가로스 세그먼트를 별도의 멸균 2mL 마이크로 원심분리기 튜브로 압출하고 800μL의 NMS를 추가한 다음 10초 동안 소용돌이를 일으켜 피펫팅을 지원합니다.
- 각 웰에 180μL의 NMS를 추가하여 멸균 96웰 플레이트를 준비합니다. 20μL의 희석된 아가로스 샘플을 첫 번째 컬럼의 각 웰에 추가하고 피펫을 추가하여 혼합합니다.
- 다중 채널 피펫을 사용하여 웰의 첫 번째 줄에서 웰의 두 번째 줄로 20μL를 옮기고 피펫팅하여 10번 혼합하여 샘플을 순차적으로 10배 희석합니다. 플레이트의 마지막 행까지 이 과정을 계속합니다.
- NMS 한천 또는 선택한 매체를 포함하는 정사각형 격자 플레이트에 라벨을 부착합니다. 다중 채널 피펫을 사용하여 96웰 플레이트의 컬럼에서 한천 플레이트에 5μL를 찾아냅니다. 한천 플레이트의 크기에 따라 동일한 플레이트에서 여러 기둥을 발견할 수 있습니다.
- 공기 중에서 메탄이 40% 미만인 플레이트를 배양하고 18°C에서 자랍니다. 2-3일 후에 박테리아 콜로니를 세고 밀리리터당 콜로니 형성 단위(CFU/mL)를 결정합니다.
4. 생체 분자 검출 분석
- 다당류 분석
- 1mL의 아가로스 세그먼트를 별도의 2mL 미세 원심분리기 튜브로 압출하고 1mL의 1%(m/v) Na2CO3 용액과 물에 혼합합니다. 5-10분마다 소용돌이를 일으키면서 샘플을 80°C로 30분 동안 가열한 다음 4,000 x g에서 4°C에서 20분 동안 원심분리합니다.
- 100% 에탄올 3권과 결합된 상층액을 채취하고 20°C에서 최소 2시간(또는 하룻밤) 동안 배양합니다.
- 에탄올 침전된 다당류를 4°C에서 30분 동안 16,100 x g 으로 원심분리하여 수집합니다. 상층액을 제거하고 펠릿을 자연 건조시킵니다. 펠릿을 100μL의 탈이온수에 재현탁시킵니다.
- 페놀-황산 비색 분석12,13을 사용하여 각 아가로스 세그먼트의 상대적인 다당류 함량을 측정합니다. 재현탁 추출물 50μL를 150μL의 진한 황산 및 30μL의 5%(v/v) 페놀과 투명한 96웰 플레이트에 담습니다.
- 마이크로플레이트 리더를 사용하여 490nm에서 흡광도를 측정하고 PTFE 필터에 가장 가까운 아가로스 세그먼트의 흡광도에 대한 백분율로 각 아가로스 세그먼트의 상대적 다당류 함량을 계산합니다.
- 단백질 분석
- 아가로스를 별도의 1.5mL 미세 원심분리기 튜브로 압출하고 각 부분 표본 100μL를 유리 시험관으로 옮깁니다.
- BCA 단백질 분석 키트의 시험관 프로토콜을 사용하여 총 단백질 농도를 측정합니다. 멸균 주사기에서 압출된 아가로스를 희석제로 사용하여 알부민 BSA 표준물질을 준비합니다.
- 세포외 DNA 분석
- 아가로스를 별도의 1.5mL 미세 원심분리기 튜브로 압출하고 각각 20μL를 0.2mL 미세 원심분리기 튜브에 전달합니다.
- 제조업체의 프로토콜에 따라 시중에서 판매되는 1x dsDNA 고감도 분석 키트를 사용하여 DNA 농도를 측정합니다.
5. RNA 추출
- RNase가 없는 물 800mL에 CTAB 2.0g, 폴리비닐피롤리돈 2.0g(PVP 40), NaCl 81.8g, Tris-HCl 100mM(pH 8.0) 및 EDTA 20mM을 결합하여 추출 완충액을 준비합니다. 부피를 1L로 올리고 오토클레이브합니다. 4 °C에서 보관하십시오.
- 준비된 추출 버퍼를 분취하고 사용 직전에 최종 농도의 베타-메르캅토에탄올 1%(v/v)를 첨가합니다. 수조 또는 열 블록을 사용하여 버퍼를 65°C로 데웁니다.
- 1.9단계의 절차에 따라 아가로스를 별도의 RNase가 없는 2mL 미세 원심분리기 튜브로 압출하여 각 그래디언트 주사기를 1mL 섹션으로 나눕니다.
- 21,000 x g, 4°C에서 15분 동안 시료를 원심분리하고 상층액을 버리고 시료를 얼음 위에 유지합니다.
- 600 μL의 사전 예열된 추출 완충액을 각 펠릿 1 mL의 압출 아가로스에 추가합니다. 약 200μL의 지르코니아/실리카 비드를 추가하고 비드 비터를 사용하여 30Hz/s에서 3분 동안 샘플을 균질화한 후 중간에 일시 중지하여 샘플을 2분 동안 얼음 위에 놓습니다.
- 15,000 x g, 4°C에서 2분 동안 샘플을 원심분리하여 거품을 줄입니다. 600μL의 클로로포름: 이소아밀 알코올(24:1)을 튜브에 넣고 10초 동안 소용돌이쳐 샘플을 추출합니다.
- 샘플을 15,000 x g, 4°C에서 8분 동안 원심분리합니다. 상부 수성상을 RNase가 없는 새로운 마이크로 원심분리기 튜브로 조심스럽게 옮기고 600μL의 클로로포름: 이소아밀 알코올(24:1)을 전사된 상부에 추가하고 10초 동안 소용돌이를 일으킵니다.
- 15,000 x g, 4°C에서 8분 동안 샘플을 원심분리하고 새로운 상부 수성상을 RNase가 없는 새로운 마이크로 원심분리기 튜브로 옮깁니다. 전사된 상부에 동일한 부피의 이소프로판올을 추가하고 -20°C에서 몇 시간 동안 샘플을 배양합니다. 선택 사항: 시료는 밤새 -20°C에서 방치할 수 있습니다.
- 16,100 x g, 4°C에서 30분 동안 시료를 원심분리하여 RNA 함유 침전물을 수집합니다.
- 상층액을 버리고 RNase가 없는 물과 원심분리기로 만든 차가운 75%(v/v) 에탄올 300μL로 펠릿을 16,100 x g, 4°C에서 5분 동안 세척합니다.
- 5.10단계에 따라 펠릿을 다시 세척하십시오.
- 에탄올 상층액을 제거한 후 펠릿을 15분 동안 자연 건조시킵니다. 펠릿을 RNase가 없는 물 100μL에 녹입니다.
- 제조업체의 프로토콜에 따라 37°C에서 DNase I로 30분 동안 샘플을 처리합니다.
- 300 μL의 산성 페놀 : 클로로포름 : IAA (125 : 24 : 1, pH 4.5)를 첨가하여 DNase I을 비활성화합니다. 10초 동안 소용돌이치고 실온에서 5분 동안 배양합니다.
- 16,100 x g, 4°C에서 5분 동안 원심분리하고 새로운 RNase-free 마이크로 원심분리기 튜브로 옮겨 상부 수성상을 유지합니다.
- 선택 사항: 상부 단계를 원뿔형 튜브로 결합하여 모든 복제 주사기에 걸쳐 동일한 세그먼트의 RNA를 풀합니다.
- RNA 함유 상부상의 부피와 동일한 이소프로판올 1부피를 추가하고 최종 농도 0.8M에 7.5M의 LiCl을 첨가하고 여러 번 반전하여 혼합합니다.
- -20 °C에서 몇 시간 동안 샘플을 배양합니다. 선택 사항: 시료는 밤새 -20°C에서 방치할 수 있습니다.
- 샘플을 16,100 x g, 4°C에서 15분 동안 원심분리하고 상층액을 조심스럽게 버립니다. RNase가 없는 물에 차가운 70%(v/v) 에탄올을 넣고 16,100 x g, 4°C에서 5분 동안 원심분리한 다음 상층액을 제거하여 RNA 함유 펠릿을 두 번 세척합니다.
- 펠릿을 실온에서 10분 동안 자연 건조시키고 50μL의 RNase가 없는 물에 재현탁시킵니다. RNA 샘플을 -80 °C에서 보관합니다.
- RNA 품질에 따라 선택 사항: RNA 정제 키트를 사용하여 작은(<200 nt) RNA를 제거하기 위해 샘플을 재정제할 수 있습니다.
- RNA 샘플에 잔류 DNA가 포함되어 있지 않은지 확인하려면 범용 박테리아 16S rRNA 유전자 프라이머 27F/1492R을 사용하여 PCR 증폭을 위한 템플릿으로 정제된 RNA 1μL를 사용하십시오. 1μL의 게놈 DNA(1:100 희석) 또는 뉴클레아제가 없는 물을 포함하는 두 가지 추가 PCR 반응을 포함하여 각각 양성 및 음성 대조군 역할을 합니다.
- 겔 전기영동14를 사용하여 1% 아가로스 TBE 겔에서 제품을 실행합니다.
참고: DNA 오염이 없는 RNA 샘플은 샘플 레인에 밴드가 없어야 합니다. RNA 샘플에서 DNA 오염이 보이면 5.13단계부터 샘플을 다시 처리합니다. RNA는 다운스트림 분석이 준비되어 있으며 RNA 무결성 번호(RIN)를 측정하여 무결성을 정량화하기 위해 선택적으로 분석할 수 있습니다.
결과
여기에서 그래디언트 주사기 모델 생태계는 단일 균주(메탄영양 Methylomonas sp. 균주 LW13)(그림 1A)를 배양하는 데 사용되었지만, 직접 토양 접종에 의한 메탄 산화 미생물 군집을 농축하는 데에도 사용할 수 있습니다(그림 1B). 메탄-산소 카운터 구배의 존재는 cell-free 및 inoculated syringes의 메탄과 산소 농도를 측정하여 검증되었습니다(그림 1C). LW13 접종 그래디언트 시린지의 경우, 시린지를 세척한 후 1일 이내에 카운터 그래디언트가 형성되었으며, 이는 배양 3일 동안 가파르게 변했습니다. 같은 기간 동안 두 가스 기판이 가장 낮은 농도에 도달하는 동일한 깊이에서 수평 띠가 형성되었습니다(그림 1A). 가파른 가스 구배와 수평 밴드 깊이를 지나는 메탄과 산소의 고갈은 LW13이 메탄을 호기적으로 대사하고 균질 플랑크톤 배양에서 관찰되지 않는 표현형을 생성한다는 것을 보여주었습니다. 이 표현형은 LW136과 동일한 환경 샘플에서 분리 된 다른 메탄 영양 박테리아에 의해서도 생성됩니다. 상이한 메탄영양 균주(methanotrophic strains) 사이에서 수평 띠 발달의 시기와 깊이의 변형 의존적 변화는 수평 띠가 공간적으로 분해된 맥락에서 배양될 때 각 미생물의 특정 행동에 의해 영향을 받는다는 것을 시사했다6.
전체 아가로스 플러그의 세포 수는 유세포 분석 및 콜로니 수(CFU/mL)를 사용하여 측정되었습니다(그림 2A). 이 방법은 이전에 수평 밴드 발달에 영향을 미치는 것으로 나타난 fucose 4-O-acetyltransferase (OAT) 유전자의 결실을 포함하는 LW13의 돌연변이 균주와 야생형 LW13의 세포 분포 및 생존을 비교하는 데 사용되었습니다6. LW13의 ΔOAT 돌연변이는 야생형에 비해 6일 동안 그래디언트 주사기의 전체 성장이 낮았으며, 이는 동일한 균주6의 균질 플랑크톤 배양에서는 관찰되지 않은 효과였다. 돌연변이 균주는 그래디언트 주사기에서 배양할 때 LW13 야생형과 동일한 뚜렷한 수평 띠를 형성하지 않았습니다(그림 2B). 세포 수와 수평 띠 모양은 돌연변이 균주에서 유전자 보완에 따라 야생형과 유사한 수준으로 복원되었습니다. 이러한 결과는 그래디언트 주사기가 메탄-산소 카운터 그래디언트에만 존재하는 특정 표현형에 유전자를 연결하는 데 사용할 수 있음을 보여줍니다.
다양한 유전적, 화학적, 분자적 기법이 반고체 아가로스 매트릭스 내에서 자란 박테리아와 함께 사용하기 위해 적용되었습니다. 그래디언트 주사기 모델 에코시스템은 접종되지 않은 아가로스를 음성 대조군으로 포함하여 표준 생체 분자 정량 분석에 즉시 사용할 수 있습니다. 세포외 고분자 물질 및 생물막에서 일반적으로 발견되는 세 가지 다른 생체 분자, 즉 다당류, 단백질 및 세포외 DNA15 의 농도를 측정했습니다(그림 3). LW13 접종 주사기 분절에서 수평 밴드는 다른 분절보다 훨씬 더 많은 다당류를 가지고 있었으며 단백질이나 세포외 DNA는 크게 증가하지 않았습니다.
RNA-seq는 주사기의 다양한 깊이에서 자라는 LW13의 전사 차이를 측정하는 데 사용되었습니다. CTAB 기반 추출 완충액을 사용한 후 기존 페놀(클로로포름 추출 및 침전 단계)을 사용하여 강력한 RNA 추출을 수행했습니다. RNA-seq 분석의 결과는 나중에 수평 다당류 밴드의 생성과 관련된 유전자를 식별하는 데 사용되었습니다. 이러한 결과는 공간적으로 분해된 모델 생태계를 만드는 데 필수적인 반고체 아가로스가 일반적으로 플랑크톤 및 플레이트 기반 배양에 사용되는 추가 생화학적 분석을 방해하지 않는다는 것을 나타냅니다.
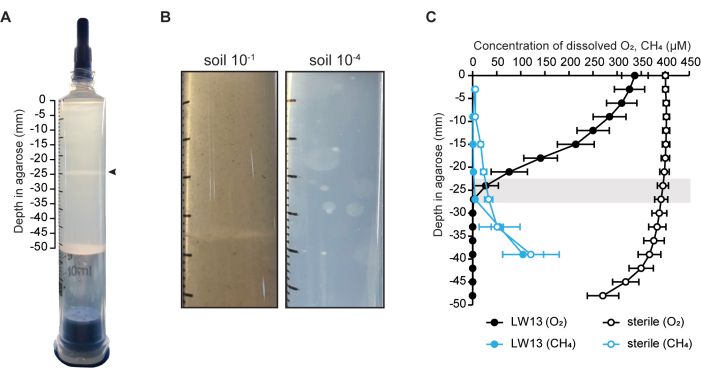
그림 1: 그래디언트 주사기 모델 에코시스템. (A) 메탄영양 Methylomonas sp. LW13을 접종한 그래디언트 주사기. 100% 메탄으로 주사기를 세척한 후 2일 이내에 뚜렷한 수평 띠(화살촉)가 발생합니다. (B) 10-1 및 10-4로 희석한 토양을 접종하고 2주 동안 배양한 그라디언트 주사기의 클로즈업 사진. 더 많은 묽은 토양을 포함하는 그래디언트 주사기는 아가로스 전체에 구형 콜로니를 초래한 반면, 더 농축된 토양 접종자는 뚜렷한 띠를 초래했습니다. (C) 배양 3일 후 LW13 접종 및 멸균 그래디언트 주사기의 메탄-산소 카운터 그래디언트의 특성화. 회색 막대는 다당류 밴드가 위치한 깊이 범위를 나타냅니다. 데이터는 각각 3개의 기술 반복이 있는 3개의 독립적인 실험의 평균 ± SD를 보여줍니다. 패널 (A) 및 (C)는 Beals et al.6에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
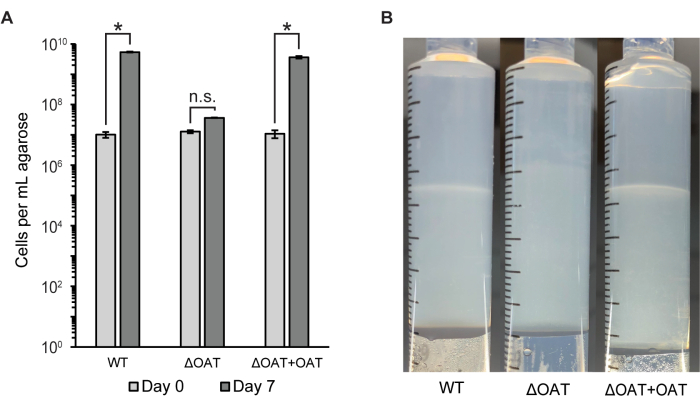
그림 2: 그래디언트 주사기에서 배양 후 LW13 야생형 및 돌연변이의 정량화. (A) 유세포 분석으로 측정한 0일 및 7일에 그래디언트 주사기에서 회수된 mL 압출 아가로스당 LW13 세포의 총 수. ΔOAT는 푸코스 4-O-아세틸전이효소 유전자의 결실을 포함하고 있으며, 이는 다당류 띠의 깊이에 위치한 세포에서 높게 발현되는 것으로 밝혀졌습니다. ΔOAT+OAT는 ΔOAT 게놈의 원위 위치에 삽입된 OAT 유전자를 포함합니다. *, 유의하게 다름(양측 이분산증 t-검정, α = 0.05); NS, 크게 다르지 않습니다. 데이터는 각각 2개의 기술 반복실험이 있는 3개의 독립적인 실험의 평균 ± SD를 보여줍니다. (B) 7일 배양 후 LW13 야생형, ΔOAT 또는 ΔOAT+OAT를 접종한 그래디언트 주사기의 수평 밴드 발달. 이 그림은 Beals et al.6에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
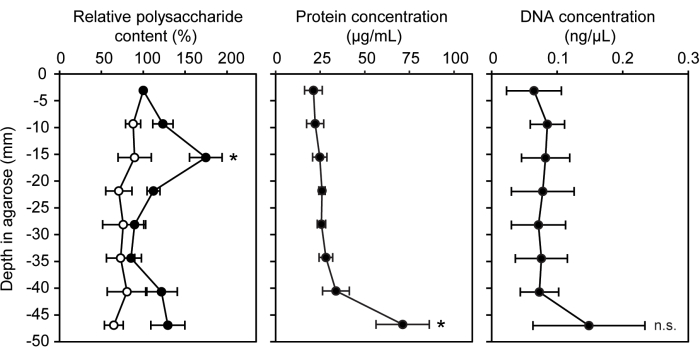
그림 3: 그래디언트 주사기의 증가하는 깊이에서 생체 분자의 정량화. 7일 동안 배양한 LW13 접종 그래디언트 주사기의 8개 섹션에서 상대 다당류 함량(%), 단백질 농도(μg/mL) 및 DNA 농도(ng/μL). 데이터는 각각 3개의 기술 반복실험이 있는 3개의 독립적인 실험의 평균 ± SD를 보여줍니다. 상대적 다당류 함량의 경우, *는 동일한 깊이에서 멸균 대조군과 유의한 차이를 나타냅니다(양측 이종 t-검정, α = 0.05). 단백질 및 DNA 농도의 경우, *는 수평 밴드를 포함하는 섹션과의 유의미한 차이를 나타냅니다(Tukey-Kramer 사후 분석을 사용한 단방향 ANOVA). NS, 중요하지 않습니다. 이 그림은 Beals et al.6에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
메탄영양 배양 방법
메타영양생물은 생리학, 자연 환경에서의 개인 및 공동체 행동, 산업 응용 분야에서 메탄 완화 잠재력을 이해하기 위해 수십 년 동안 연구되어 왔습니다. 이러한 연구를 통해 수행된 연구의 대부분은 공간적 맥락이 손실된 균질한 플랑크톤 배양을 사용하여 수행되었습니다. 그래디언트 시린지 모델 에코시스템은 실험실에서 자연 메탄영양생물 서식지의 메탄-산소 카운터 구배 특성을 복제하기 위해 개발되었으며, 이를 통해 연구원들은 이러한 유기체가 진화한 곳과 더 유사한 환경에서 자라는 메탄영양생물을 연구할 수 있습니다.
지난 30년 동안 연구자들은 다양한 방법을 사용하여 실험실에서 메탄-산소 카운터 그래디언트를 재현했으며, 종종 혼합 메탄 산화 컨소시엄에서 메탄영양체를 분리하고 분류하는 것을 주요 목표로 삼았습니다. 이러한 방법은 메탄과 산소의 반대 챔버를 사용하는 두 가지 접근법으로 나눌 수 있습니다 : 멤브레인(16,17,18)에 상대적으로 방해받지 않는 토양을 부유시키거나 소량의 토양 또는 순수한 박테리아 배양을 아가로스 7,8,19의 최소 매체에 접종합니다. 여기에 설명된 그래디언트 주사기 방법은 Dedysh와 동료9의 주사기 기반 접근법과 Amaral과Knowles8, Schink와 동료7의 이전 연구에서 얻은 메탄영양생물 배양을 결합합니다. 이러한 방법 중 후자는 메탄-산소 카운터 구배에서 메탄영양생물을 배양하기 위한 토대를 마련하고 아가로스 플러그의 양쪽에서 메탄과 산소의 연속적인 흐름을 사용했습니다. 이 방법은 보다 일정한 환경을 제공하지만 실험 설정에 복잡성을 더하고 전용 가스 공급원이 필요합니다.
이와 대조적으로, 여기에 설명된 그래디언트 시린지는 매일 주사기를 세척하여 새로운 메탄을 제공하는 동시에 주사기당 1분도 채 걸리지 않는 공정을 제공하는 동시에 제균 PTFE 필터 팁을 통해 대기 산소에 지속적으로 접근할 수 있습니다. 이 더 간단한 방법은 공간적으로 분해된 맥락에서 메탄영양생물을 연구하기 위해 이 모델 생태계의 광범위한 채택을 가능하게 할 수 있습니다. 설명된 프로토콜은 또한 반고체 아가로스에서 배양된 박테리아에 대해 직접 수행할 수 있는 화학적 및 분자 수준 분석을 자세히 설명합니다. 결과적으로, 분석 전에 아가로스 매트릭스 외부에서 박테리아를 절제하고 배양할 필요가 없어 샘플링 시 가스 구배 조건을 보존할 수 있습니다.
프로토콜에 대한 설명
박테리아는 폴리프로필렌 부피 주사기 내에서 배양되기 때문에 연구원들은 함께 제공되는 주사기 플런저를 사용하여 주사기 배럴에 남아 있는 아가로스 매트릭스의 공간 무결성을 유지하면서 아가로스 플러그를 정확하고 재현성 있게 분할할 수 있습니다. 주사기 특유의 기밀 설계가 없으면 주사기 배럴에서 아가로스 플러그를 제거하고 슬라이스해야 하므로 아가로스 세그먼트의 부피에 불확실성이 발생하고 정량화할 수 없는 양의 메탄과 용존 산소가 대기 중으로 방출됩니다. 멸균 바늘을 통한 아가로스 압출은 시료 준비를 단순화하고 박테리아 세포를 절단하지 않고 압출된 세그먼트를 균질화하는 데 도움이 됩니다. 이 방법을 통해 연구원은 접종된 각 그래디언트 주사기를 최소 8개의 아가로스 세그먼트로 나누고 산소 및 메탄 농도 범위에서 성장하는 메탄영양생물에 대한 병렬 실험을 수행할 수 있습니다.
고다당류 함량의 아가로스에서 RNA 추출을 최적화하는 과정에서 구아니듐 티오시아네이트 및 TRIzol과 같은 일반적인 시약이 아가로스 겔화로 이어져 정제 컬럼을 방해하고 원심분리에 의한 펠릿화에 저항하는 것으로 나타났습니다. 큰 다당류 분자는 핵산을 포획할 수 있는 반면 작은 다당류는 RNA20과 함께 침전될 수 있기 때문에 낮은 RNA 수율과 품질도 우려 사항이었습니다. 대신에, 양이온성 계면활성제(CTAB)를 함유하는 추출 완충액을 사용하였고, 이는 지질막(20)을 가용화시키고; CTAB-핵산 복합체가 형성되는 것을 방지하고 핵산이 침전되도록 허용하지만 용액21에서 다당류를 유지하는 NaCl. RNase는 CTAB 완충액에 β-메르캅토에탄올을 포함시킴으로써 변성시켰다. RNA-seq 실험의 경우, 라이브러리 준비 전에 작은(<200 뉴클레오티드) RNA를 제외하기 위해 선택적 컬럼 기반 정제 단계가 포함되었습니다.
제한 사항 및 고려 사항
NMS 및 아가로스는 메탄영양 박테리아를 배양하기 위한 최소한의 배지 매트릭스를 제공하지만, 여기에 설명된 그래디언트 시린지는 메탄영양 서식지의 가스 그래디언트만을 재현하지만, 이러한 환경에 존재하는 다른 그래디언트(예컨대, 예컨대 미량 금속(22), 염도(23) 또는 기타 영양소(24)는 생성하지 않는다. 이러한 그래디언트는 향후 유사한 시스템에 추가될 수 있습니다. 또한 주사기의 부피(8mL 아가로스)는 주사기당 총 바이오매스를 제한하므로 일부 분석에서는 여러 주사기를 풀링해야 합니다(5.16단계 설명 참조). 휴대용 주사기는 아가로스를 1mL 부분 표본으로 편리하게 분할하지만, 그 크기는 헤드 공간을 약 4mL로 제한하여 배양된 미생물을 위해 저장할 수 있는 벌크 메탄의 양을 제한합니다. 메탄 산화 속도는 호기성 메탄영양 생물25의 성장 속도에 비례하기 때문에, 헤드스페이스 메탄을 매일 보충하는 것이 권장된다. 이로 인해 여전히 메탄이 제한되는 기간이 발생할 수 있지만, 이 기간은 실험실에서 재현할 수 있으며 자연 환경에서 발견되는 상황을 모방할 수 있습니다.
그래디언트 주사기를 사용하는 동안 아가로스 다당류의 존재로 인해 이 시스템에서 성장한 메탄영양 생물을 분석하는 데 사용되는 분석에 대한 일부 조정이 필요합니다. 예를 들어, 소량의 압출된 아가로스를 이송해야 하는 프로토콜은 정확한 피펫팅을 위해 각 희석 간에 철저한 균질화를 통해 여러 희석 단계가 필요합니다. 또한 아가로스 매트릭스에 고유한 다당류가 황산-페놀 시약과 반응하는 다당류 분석과 같은 경우 멸균, 무세포 아가로스 음성 대조군을 포함하는 것이 필수적입니다. 아가로스 가수분해 효소인 β-아가라아제를 포함하여 이러한 문제를 완화하려는 초기 시도는 성공하지 못했고 생물학적 실험에 알려지지 않은 변수를 도입했습니다. 여러 기술 복제, 철저한 희석, 균질화 및 대조군 포함을 사용하여 아가로스 매트릭스에 내재된 대부분의 문제를 완화할 수 있습니다.
응용 프로그램
단일 균주 연구 외에도 그래디언트 주사기는 여러 균주의 공동 배양을 지원할 수 있으며 토양을 순수 박테리아 배양 대신 접종물로 사용할 수 있습니다. 그래디언트 주사기 모델 에코시스템의 간단한 설계는 메탄 대신 H2 또는 CO와 같은 다른 가스 기질을 사용하여 무산소 환경과 산소 환경 사이의 계면에 존재하는 다른 유형의 미생물을 배양할 수 있습니다. 요약하면, 간단하고 공간적으로 분해된 모델 에코시스템을 사용하여 연구자들은 무산소 미생물의 고유한 생리학 및 대사 적응을 연구할 수 있으며 유전자를 유기체 표현형과 연결하는 데 사용할 수 있습니다.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
이 연구는 유타 대학교 화학과와 NSF CAREER Award #2339190의 스타트업 펀딩으로 지원되었습니다. 유익한 토론을 해주신 Puri Lab 회원들에게 감사드립니다. 유세포 분석 실험에 대한 초기 지도를 제공한 Rachel Hurrell(유타 대학교)에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 1% Gas mix analytical standard | Supelco | 22561 | 1% each component in nitrogen: carbon monoxide, carbon dioxide, hydrogen, methane and oxygen |
| 100% Methane | Airgas | ME CP300 | chemically pure grade |
| 15 ppm Gas mix analytical standard | Supelco | 23470-U | 15 ppm each component in nitrogen: methane, ethane, ethylene, acetylene, propane, propylene, propyne, and n-butane |
| 1x Nitrate mineral salts | see CAS numbers below | Dissolve the following in Mili-Q water and autoclave: 0.2 g/L MgSO4·7H2O, 0.2 g/L CaCl2·6H2O, 1 g/L KNO3, and 30 μM LaCl3. Before use, add trace elements to a 1X final concentration and phosphate buffer (pH 6.8) to a final concentration of 5.8 mM. | |
| 23 G needle | BD Biosciences | 305194 | sterile, Luer-Lok |
| 500x Trace elements | see CAS numbers below | Dissolve the following in Milli-Q water: 1.0 g/L Na2-EDTA, 2.0 g/L FeSO4·7H2O, 0.8 g/L ZnSO4·7H2O, 0.03 g/L MnCl2·4H2O, 0.03 g/L H3BO3, 0.2 g/L CoCl2·6H2O, 0.6 g/L CuCl2·2H2O, 0.02 g/L NiCl2·6H2O, and 0.05 g/L Na2MoO·2H2O. | |
| 96 Well plate | CELLTREAT | 229596 | sterile |
| Acid phenol:chloroform:IAA (125:24:1) | Invitrogen | AM9720 | pH 4.5 |
| Agarose | Fisher Scientific | BP160 | molecular biology grade, CAS 9012-36-6 |
| Aluminum crimp seals | VWR | 30618-460 | 20 mm |
| Bead beater | Qiagen | 9003240 | TissueLyser III |
| Butyl rubber stopper | Chemglass Life Science | 50-143-854 | 20 mm, blue |
| Chloroform:isoamyl alcohol (24:1) | Millipore Sigma | 25666 | BioUltra, for molecular biology |
| Clark-type O2 microelectrode | Unisense | OX-500 | |
| DEPC-treated water | Thermo Scientific | R0601 | |
| DNase I (Ambion) | Invitrogen | AM2222 | |
| Flow cytometer | Beckman Coulter | CytoFLEX | |
| Gas chromatograph (flame ionization detection) | Agilent | 6890N | |
| Gastight analytical syringe | Hamilton | 81220 | 1750 TLL |
| Gastight analytical syringe needle | Hamilton | 7729-07 | 22 G, metal hub needle, 2 in, point style 5 |
| Gas-tight vials | Labco | 938W | Exetainer vial: 12 mL, round bottom |
| Glass culture tubes | Bellco Glass | 2048-00150 | 18 x 150 mm |
| LiCl precipitation solution (7.5 M) | Invitrogen | AM9480 | |
| One-way stopcock | VWR | MFLX30600-00 | inlet port: female luer, outlet port: male luer lock |
| Petri dish, square | Fisher Scientific | FB0875711A | 100 x 100 mm |
| Phosphate buffer, 0.2 M (pH 6.8) | see CAS numbers below | Dissolve the following in Milli-Q water and autoclave: 12.24 g/L KH2PO4, 26.29 g/L Na2HPO4 · 7H2O | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| PTFE syringe filter tip | Thermo Scientific | 03-050-469 | hydrophobic, pore size: 0.2 µm, diameter: 4 mm |
| Qubit 1x dsDNA High Sensitivity Assay Kit | Invitrogen | Q33230 | |
| Qubit 4 Fluorometer | Invitrogen | Q33238 | |
| RNA Clean & Concentrator-5 | Zymo Research | R1013 | |
| Serum stopper | Fisher Scientific | 03-340-302 | 20 mm |
| Syringe | BD Biosciences | 302995 | Luer-Lock, 10 mL, single use, sterile |
| Syringe pump | New Era Pump Systems Inc. | 1000-US | NE-1000 one channel programmable |
| SYTO9, propidium iodide, microspheres | Invitrogen | L34856 | LIVE/DEAD BacLight Bacterial Viability Kit |
| Zirconia/silica beads | BioSpec Products | 11079101z | 0.1 mm diameter |
| Chemical reagents | CAS number | ||
| CaCl2·6H2O | 7774-34-7 | ||
| CoCl2·6H2O | 7791-13-1 | ||
| Concentrated sulfuric acid | 7664-93-9 | ||
| CTAB, cetrimonium bromide | 57-09-0 | ||
| CuCl2·2H2O | 10125-13-0 | ||
| Ethanol | 64-17-5 | ||
| FeSO4·7H2O | 7782-63-0 | ||
| H3BO3 | 10043-35-3 | ||
| Isopropanol | 69-63-0 | ||
| KH2PO4 | 7778-77-0 | ||
| KNO3 | 7757-79-1 | ||
| LaCl3 | 10099-58-8 | ||
| MgSO4·7H2O | 10034-99-8 | ||
| MnCl2·4H2O | 13446-34-9 | ||
| Na2CO3, sodium carbonate | 497-19-8 | ||
| Na2-EDTA | 139-33-3 | ||
| Na2HPO4 · 7H2O | 7782-85-6 | ||
| Na2MoO·2H2O | 10102-40-6 | ||
| NaCl, sodium chloride | 7647-14-5 | ||
| NiCl2·6H2O | 7791-20-0 | ||
| Phenol (90% solution in water) | 108-95-2 | ||
| PVP40, polyvinylpyrrolidone | 9003-39-8 | ||
| Tris-HCl | 1185-53-1 | ||
| ZnSO4·7H2O | 7446-20-0 | ||
| β-Mercaptoethanol | 60-24-2 |
참고문헌
- Brune, A., Frenzel, P., Cypionka, H. Life at the oxic-anoxic interface: microbial activities and adaptations. FEMS Microbiol Rev. 24 (5), 691-710 (2000).
- Auman, A. J., Stolyar, S., Costello, A. M., Lidstrom, M. E. Molecular characterization of methanotrophic isolates from freshwater lake sediment. Appl Environ Microbiol. 66 (12), 5259-5266 (2000).
- Whittenbury, R., Phillips, K. C., Wilkinson, J. F. Enrichment, isolation and some properties of methane-utilizing bacteria. J Gen Microbiol. 61 (2), 205-218 (1970).
- Koo, C., Rosenzweig, A. Biochemistry of aerobic biological methane oxidation. Chem Soc Rev. 50 (5), 3424-3436 (2021).
- Strong, P. J., Xie, S., Clarke, W. P. Methane as a resource: Can the methanotrophs add value. Environ Sci Technol. 49 (7), 4001-4018 (2015).
- Beals, D. G., Puri, A. W. Linking methanotroph phenotypes to genotypes using a simple spatially resolved model ecosystem. ISME J. 18 (1), wrae060(2024).
- Bussmann, I., Rahalkar, M., Schink, B. Cultivation of methanotrophic bacteria in opposing gradients of methane and oxygen. FEMS Microbiol. Ecol. 56 (3), 331-344 (2006).
- Amaral, J. A., Knowles, R. Growth of methanotrophs in methane and oxygen counter gradients. FEMS Microbiol Lett. 126 (3), 215-220 (1995).
- Danilova, O. V., et al. A new cell morphotype among methane oxidizers: a spiral-shaped obligately microaerophilic methanotroph from northern low-oxygen environments. ISME J. 10 (11), 2734-2743 (2016).
- Ou, F., McGoverin, C., Swift, S., Vanholsbeeck, F. Absolute bacterial cell enumeration using flow cytometry. J Appl Microbiol. 123 (2), 464-477 (2017).
- Krause, S. M. B., et al. Lanthanide-dependent cross-feeding of methane-derived carbon is linked by microbial community interactions. Proc Natl Acad Sci USA. 114 (2), 358-363 (2017).
- Masuko, T., et al. Carbohydrate analysis by a phenol-sulfuric acid method in microplate format. Anal Biochem. 339 (1), 69-72 (2005).
- Felz, S., Al-Zuhairy, S., Aarstad, O. A., van Loosdrecht, M. C. M., Lin, Y. M. Extraction of structural extracellular polymeric substances from aerobic granular sludge. J Vis Exp. (115), e54534(2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923(2012).
- Costa, O. Y. A., Raaijmakers, J. M., Kuramae, E. E. Microbial extracellular polymeric substances: Ecological function and impact on soil aggregation. Front Microbiol. 9, 1636(2018).
- Sinke, A. J. C., Cottaar, F. H. M., Buis, K., Keizer, P. Methane oxidation by methanotrophs and its effects on the phosphate flux over the sediment-water interface in a eutrophic lake. Microb Ecol. 24 (3), 259-269 (1992).
- Murase, J., Frenzel, P. A methane-driven microbial food web in a wetland rice soil. Environ Microbiol. 9 (12), 3025-3034 (2007).
- Reim, A., Lüke, C., Krause, S., Pratscher, J., Frenzel, P. One millimetre makes the difference: High-resolution analysis of methane-oxidizing bacteria and their specific activity at the oxic-anoxic interface in a flooded paddy soil. ISME J. 6 (11), 2128-2139 (2012).
- Rahalkar, M., Bussmann, I., Schink, B. Methylosoma difficile gen. nov., sp. nov., a novel methanotroph enriched by gradient cultivation from littoral sediment of Lake Constance. Int J Syst Evol Microbiol. 57 (5), 1073-1080 (2007).
- Wang, L., Stegemann, J. P. Extraction of high-quality RNA from polysaccharide matrices using cetlytrimethylammonium bromide. Biomaterials. 31 (7), 1612(2010).
- Liyanage, N. M. N., Chandrasekara, B. C. H. W. M., Bandaranayake, P. C. G. A CTAB protocol for obtaining high-quality total RNA from cinnamon (Cinnamomum zeylanicum Blume). 3 Biotech. 11 (4), 201(2021).
- Semrau, J. D., DiSpirito, A. A., Gu, W., Yoon, S. Metals and methanotrophy. Appl Environ Microbiol. 84 (6), e02289-e02317 (2018).
- Zhang, S., et al. Salinity significantly affects methane oxidation and methanotrophic community in Inner Mongolia lake sediments. Front Microbiol. 13, 1067017(2023).
- Fox, A. L., Trefry, J. H. Nutrient fluxes from recent deposits of fine-grained, organic-rich sediments in a Florida estuary. Front Mar Sci. , (2023).
- He, L., et al. A methanotrophic bacterium to enable methane removal for climate mitigation. Proc Natl Acad Sci. 120 (35), e2310046120(2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유