Method Article
Assembly and Quantification of Co-Cultures Combining Heterotrophic Yeast with Phototrophic Sugar-Secreting Cyanobacteria(종속영양 효모와 광영양성 설탕 분비 시아노박테리아를 결합한 공동 배양의 조립 및 정량화)
* 이 저자들은 동등하게 기여했습니다
요약
이 프로토콜은 광독립영양 설탕 분비 시아노박테리아 및 종속영양 효모를 포함한 공동 배양의 설정 및 정량적 모니터링을 위한 포괄적인 지침을 제공합니다.
초록
지속 가능한 생명 공학에 대한 수요가 증가함에 따라 광 영양 미생물과 종속 영양 파트너 종을 포함하는 혼합 컨소시엄이 태양 기반 바이오 생산의 방법으로 탐구되고 있습니다. 한 가지 접근 방식은 공동 재배된 종속 영양 생물의 대사를 지원하기 위해 유기 탄소를 분비하는 CO2 고정 시아노박테리아를 사용하는 것이며, 이는 다시 탄소를 더 높은 가치의 상품 또는 서비스로 변환합니다. 이 프로토콜에서는 실험자가 자당을 분비하는 시아노박테리아 균주와 모델 효모 종으로 대표되는 곰팡이 파트너를 결합한 공동 배양을 설정하는 데 도움이 되는 기술 설명이 제공됩니다. 이 프로토콜은 공동 배양 확립을 위한 주요 전제 조건을 설명합니다: 배지 조성 정의, 개별 파트너의 성장 특성 모니터링, 동일한 성장 용기에 결합된 여러 종의 혼합 배양 분석. 현미경 검사, 세포 계수기 및 단일 세포 유세포 분석을 포함한 공동 배양 모니터링을 위한 기본 실험실 기술을 요약하고, FAIR(Findable, Accessible, Interoperable, Reusable) 원칙에 따라 원시 유세포 분석 표준(FCS) 파일의 데이터 분석에 사용할 수 있는 비독점 소프트웨어의 예를 제공합니다. 마지막으로, 당을 분비하는 시아노박테리아(cyanobacteria)와 새로운 종속영양 파트너(heterotrophic partner)와의 공동 배양을 확립하려고 할 때 자주 발생하는 병목 현상과 함정에 대한 설명이 포함되어 있습니다. 이 프로토콜은 시아노박테리아(cyanobacterium)와 종속영양 미생물(heterotrophic microbe)을 포함하는 새로운 공동 배양 미생물 쌍을 확립하려는 연구자들에게 리소스를 제공합니다.
서문
최근 몇 년 동안 게놈 도구와 DNA 기술이 급속히 확장됨에 따라 생명공학 노력은 축색 배양에만 집중하는 대신 혼합 미생물 군집을 생물 생산 전략에 실행 가능한 것으로 간주할 수 있게 되었습니다. 미생물 컨소시엄은 단일 종 배양에 비해 전문화 및 분업, 적응성 및 견고성, 기질 활용의 효율성 등 여러 가지 잠재적 이점을 가지고 있습니다1. 그러나 다종 컨소시엄의 예측 가능한 엔지니어링은 종간 상호 작용2에서 나타나는 고차원 행동으로 인한 불확실성으로 인해 복잡합니다. 종간 신호전달과 대사산물 교환은 분업 원칙의 핵심이지만, 컨소시엄 참가자들 사이에 예상치 못한 시너지 효과와 적대감을 초래하기도 한다3. 혼합 미생물 컨소시엄의 잠재력을 최대한 실현할 수 있으려면 "상향식"에서 미생물 상호 작용을 더 잘 특성화하고 이해하는 데 사용할 수 있는 유연한 2 및 3 파트너 공동 배양 플랫폼의 사용을 포함하여 이 분야의 상당한 개발이 필요합니다.
현재 이 분야에서 몇 가지 지배적인 유형의 공동 배양 플랫폼이 사용되고 있으며, 여기에는 무료 보조 영양 파트너 및 광범위한 미생물 종에 일반적으로 유익한 대사 산물을 분비하는 미생물이 포함됩니다. 후자의 범주에서, 시아노박테리아는 쉽게 대사되는 탄수화물의 분비로 이어지는 경로의 도입을 통해 향상된 1차 생산자가 되도록 설계되었으며, 현재 합리적으로 설계된 다양한 컨소시엄에서 탐구되고 있습니다. 간단히 말해서, 이러한 조작된 미생물 군집에서 시아노박테리아 파트너는 빛과CO2 를 주요 입력으로 활용할 수 있으며, 산소 광합성 과정을 통해 이러한 균주는 중앙 탄소당을 공공재로 분비할 수 있습니다. 이러한 조작된 시아노박테리아 균주의 한 종류는 이당류 자당4를 분비하도록 조작된 균주입니다. 이러한 균주는 자당이 많은 종의 중심 탄소 대사에 가까운 대사 산물이며 다양한 환경 비 생물 스트레스에 적응하기 위해 소위 "호환 가능한 용질"로 자주 과도하게 축적되기 때문에 성공을 거두었을 가능성이 높습니다5. 최소한의 유전적 개입은 다양한 시아노박테리아 모델 유기체에서 효율적인 자당 분비로 이어질 수 있습니다4.
자당을 분비하는 시아노박테리아는 합리적으로 설계된 미생물 컨소시엄을 조사하는 데 유용한 플랫폼인데, 그 이유는 다양한 종속영양 종이 자당을 탄소와 에너지의 지배적인 원천으로 대사할 수 있기 때문입니다. 실제로, 많은 실험실에서는 자당 분비 능력을 가진 몇 가지 모델 시아노박테리아를 활용하여 하나 이상의 종속영양 미생물을 포함하고 궁극적으로 유기 탄소 공급 원료를 보충하지 않고 빛과CO2 의 1차 입력에 의해 지원되는 혼합 종 공동 배양 및 컨소시엄을 합리적으로 설계했습니다4. 종속영양 균주는 단순히 시아노박테리아 유래 탄수화물에 의존할 수 있거나, 자당 공급 원료를 더 높은 가치의 바이오 생성물(예: 연료, 폴리머, 안료 등)로 전환하는 데 사용될 수 있습니다. 지속 가능한 생물 생산을 위한 잠재적인 전략이 될 뿐만 아니라, 이러한 단순한 공동 배양은 관련이 없는 미생물 종 간의 창발적 상호 작용을 조사하기 위한 플랫폼으로도 유용할 수 있습니다.
이 동영상 기사는 경량과 무기 탄소 투입량만을 보충함으로써 안정될 수 있는 간단한 미생물 컨소시엄을 설계하기 위한 유연한 플랫폼으로 당을 생산하는 시아노박테리아를 활용하기 위한 방법론과 전제 조건에 초점을 맞춥니다. 확립된 단일 미생물을 포함하는 배양물의 설정 및 모니터링은 대부분 간단하고 광학 밀도(OD) 또는 후방 산란 방법론을 사용하여 쉽게 달성할 수 있지만, 두 개 이상의 유기체가 하나의 용기에 결합되면 실현 가능하지 않습니다. 주된 이유는 이러한 방법이 서로 다른 미생물을 구별하지 못하기 때문에 배양에 대한 전체적인 그림만 제공하고 개별 유기체의 성장을 해결하지 않기 때문입니다. 더욱이, 시아노박테리아는 400-750 nm 범위에서 넓은 흡수 스펙트럼을 가지고 있기 때문에 종속영양 생물의 OD600 을 측정하면 피코시아닌(620 nm에서 흡수)으로 인해 잘못된 결과가 발생할 수 있습니다. 따라서 실험실 내에서 시아노박테리아-종속영양 혼합 군집을 설정하기 위한 특정 프로토콜과 시간 경과에 따른 이러한 컨소시엄의 성능을 분석하기 위한 유용한 일반 프로토콜이 제공됩니다. 프로토콜은 하나 이상의 모델 종속영양 미생물과 자당을 분비하는 시아노박테리아 종의 특정 쌍에 초점을 맞추고 있지만, 이 작업의 목적은 새로운 종 쌍을 설계하고자 하는 연구자에게 리소스를 제공하고 이러한 배양 확립을 위한 최적화 단계를 가속화하는 것입니다. 따라서 종별 프로토콜 외에도 독자의 필요에 따라 정의된 대로 맞춤형 커뮤니티에 맞게 이러한 프로토콜을 조정하고 일반화하는 데 사용할 수 있는 정보와 전략이 포함됩니다.
본원에 기술된 공동배양 플랫폼의 유연성으로 인해, 당-분비 시아노박테리아(cyanobacteria)와의 공동배양에서 이전에 보고된 다수의 상이한 종속영양 종에 대한 프로토콜이 설명된다. 예를 들어, Synechococcus elongatus PCC 7942와 일반적인 실험실 효모 Saccharomyces cerevisiae 의 공동 배양을 위한 단계별 프로토콜이 제공됩니다. 그러나 이 기사에는 Ustilago maydis의 효모 형태를 포함하여 다른 모델 종을 포함하는 공동 배양의 성능을 분석하는 데 적합한 프로토콜도 포함되어 있습니다.
이 기사는 시아노박테리아/종속영양 공동 배양을 확립하고 시간 경과에 따른 이러한 혼합 컨소시엄의 성능에 대한 기본 특성화를 수행하는 데 필요한 핵심 프로토콜 세트에 중점을 둡니다. 특히, 다양한 종에 대한 정확한 인구 조사를 수행하는 데 적합한 단일 세포 유세포 분석 및 입자 계수 방법뿐만 아니라 세포 형태를 평가하기 위한 현미경 접근 방식이 강조됩니다. 이러한 프로토콜은 사용 가능한 요구 사항과 장비에 적응하기 위한 기초 역할을 하기 위한 것입니다. 중요한 것은 실험실 내에서 공동 배양을 수립하고 모니터링하는 데 중요한 기술 노트 및 기타 고려 사항이 제공된다는 것입니다. 마지막으로, Python 패키지를 사용하여 원시 FCS6 파일의 데이터 분석을 위한 비독점적 대안의 예가 포함되어 있습니다. 요약하자면, 목표는 시아노박테리아 기반 공동 배양 기술을 더 많은 과학 청중이 더 쉽게 접근할 수 있도록 하는 것입니다.
프로토콜
참고: 이 프로토콜에는 설탕을 분비하는 S. elongatus 및 종속영양 모델 효모 종의 공동 배양을 설정하고 정량화하는 방법에 대한 자세한 지침이 포함되어 있습니다. 일반적으로 이 프로토콜은 유전자 조작이 가능한 모든 효모 종에 적용할 수 있습니다.
1. 광영양성 시아노박테리아(phototrophic cyanobacteria)와 종속영양 효모(heterotrophic yeast)를 결합한 공동 배양 확립
- 공동 재배를 위한 준비: 배지와 사전 재배
알림: 초순수 여과수를 사용하여 모든 매체 및 스톡 용액을 준비합니다. 모든 유리 제품(계량 비커, 눈금이 매겨진 실린더, 보관 병 및 배양 플라스크)은 세척하고 고압 멸균해야 합니다. 멸균 매체는 실온(RT)에서 보관할 수 있습니다. photoautotrophic 재배의 요구 사항과 호환되는 장치를 사용할 수 있어야 합니다.- 의도한 공동 배양 시작 최소 3-4일 전에 공동 배양 배지를 준비합니다. 대부분의 공동 배양 배지 레시피는 많은 시아노박테리아 모델 종의 일상적인 배양에 사용되는 일반적인 BG-11 배지7 의 변형입니다. 이 프로토콜은 이전에 보고된 CoYBG-11 매체를 예로 사용합니다(8; 표 1 참조). 오토클레이브(121°C, 20분) 또는 필터는 준비된 매체를 적절하게 멸균(0.22μm 공극 크기)합니다.
참고: 다른 종 쌍에 적합한 배지를 조정하는 방법에 대한 제안은 토론 섹션을 참조하십시오. - 분자 시약 준비 : cscB 발현 유도 및 자당 수출 9,10,11을 위해 이소 프로필 -β-티오 갈락 토시드 (IPTG)의 1M 스톡을 준비하십시오.
참고: 공동 배양 중 항생제 사용은 최적이 아니지만 모든 균주에 내성 카세트가 있는 경우 사용할 수 있습니다. 이 경우, 고용된 균주의 필요에 따라 항생제 재고를 준비하십시오. - 시아노박테리움 전배양 준비: 공동 배양을 시작하기 최소 3일 전에 BG-11 배지에서 자라는 시아노박테리아의 설탕 분비 균주의 사전 배양에서 CoYBG-11 배지를 함유한 신선한 배플 플라스크로 세포를 이동합니다(표 1).
참고: 여러 종과 균주의 시아노박테리아가 이제 당을 분비하도록 변형되었습니다. 이 글을 쓰는 시점에서 사용 가능한 균주에 대한 포괄적인 검토는4를 참조하십시오. 이 참고 문헌은 또한 시아노박테리아 공동 배양이 이전에 보고된 종속영양 종(1.1.4 참조)을 나열합니다.- preculture의 OD750을 측정합니다. 방정식 V1 = C2 x V2/C1을 사용하여 0.3(C1)의 목표 OD750을 달성하기 위해 필요한 사전 배양 부피를 계산합니다. 여기서 C2 = 목표 OD, V2 = 목표 부피, V1 = 필요한 사전 배양 부피. 예를 들어, 30 mL 배양액(V2)을 접종하는 경우, 사용하는 액상 배양액의 x량은V1 (mL) = (0.3 x 30)/(측정된 OD750)이다.
참고: 광학 밀도는 세포 밀도의 근사치이며 성장 조건과 세포 형태에 따라 달라집니다. 따라서 연구자들은 공동 배양 성능의 적절한 평가를 위해 광학 밀도를 절대 세포 수와 상관시키는 방법(섹션 2.1, 2.2 및 2.3 참조)을 사용해야 합니다. 시아노박테리아의 경우, 광합성 활성 스펙트럼(즉, 400-700nm) 내의 광 파장은 엽록소 및 기타 색소에 흡수되기 때문에 광학 밀도를 결정하는 데 사용해서는 안 됩니다. 따라서 원적외선 파장(예: 750nm)이 일상적으로 사용됩니다. - 희석된 배양물을 적절한 광배양기에서 배양합니다. 일반적인 배양
모델 시아노박테리아의 조건은 다음과 같습니다: 30°C, 150rpm(25mm 투사), 2% CO2 헤드스페이스, ~200μmol 광자 m-2·s-1 LED 조명, 75% 습도.
참고: 방출되는 자당의 양을 최적화하기 위해 CO2 를 보충하는 것이 적극 권장됩니다. - 시아노박테리아가 기하급수적으로 성장할 수 있도록 공동 배양을 설정하기 전에 최소 3일 동안 매일 이 희석 과정을 반복합니다. 이는 서로 다른 날의 실험 간의 공동 배양 성능의 일관성을 개선하는 데 도움이 됩니다.
- preculture의 OD750을 측정합니다. 방정식 V1 = C2 x V2/C1을 사용하여 0.3(C1)의 목표 OD750을 달성하기 위해 필요한 사전 배양 부피를 계산합니다. 여기서 C2 = 목표 OD, V2 = 목표 부피, V1 = 필요한 사전 배양 부피. 예를 들어, 30 mL 배양액(V2)을 접종하는 경우, 사용하는 액상 배양액의 x량은V1 (mL) = (0.3 x 30)/(측정된 OD750)이다.
- 공동 배양을 시작하기 최소 2일 전에 종속영양 배양을 준비합니다. 언급했듯이, 많은 효모 종과 아종은 이전에 시아 노 박테리아8 , 12 , 13 과 공동 배양에서 성장했습니다.
- 풍부한 배양 배지( 표 1 참조)에서 자라는 종속영양 종의 액체 배양물로부터 1:100 희석(250μL)으로 세포를 250mL 당황한 플라스크에 20g/L 자당이 보충된 50mL의 CoYBG-11 배지로 전달합니다.
- 시아노박테리아 파트너에 사용되는 배양 조건과 동일한 온도에서 밤새 배양합니다.
- 종속영양 균주가 실험적 필요성에 충분히 성장했는지 확인하기 위해 공동 배양 접종 예정 시간 최소 24시간 전에 OD600 을 확인하십시오(1.2 참조): 필요한 경우 기하급수적 성장을 유지하기 위해 새로운 배지로 희석합니다.
- 의도한 공동 배양 시작 최소 3-4일 전에 공동 배양 배지를 준비합니다. 대부분의 공동 배양 배지 레시피는 많은 시아노박테리아 모델 종의 일상적인 배양에 사용되는 일반적인 BG-11 배지7 의 변형입니다. 이 프로토콜은 이전에 보고된 CoYBG-11 매체를 예로 사용합니다(8; 표 1 참조). 오토클레이브(121°C, 20분) 또는 필터는 준비된 매체를 적절하게 멸균(0.22μm 공극 크기)합니다.
- 공동배양 접종 및 유지
참고: 광영양 미생물과 종속영양 미생물 간의 접종 비율에 대한 고려 사항에 대해서는 토론 섹션을 참조하십시오.- 공동 배양의 접종을 위해, 원심 분리에 의해 두 preculture를 모두 풍부하게하십시오.
- 표준 C1 x V1 = C2 x V2 방정식(여기서 C1 = 농축 배양의 OD)을 사용하여 두 종에 대해 원하는 시작 밀도를 달성하기 위해 농축된 세포 현탁액의 부피를 계산합니다. 예를 들어, 시작 OD750이 0.5인 S. elongatus의 25mL 배양물(1.5 OD 축연산 배양에서)과 시작 OD600이 0.05인 S. cerevisiae(0.7 OD 축연산 배양에서)를 만들기 위해서는 위의 방정식에 따라 8.33mL의 S. elongatus 시작 배양액, 1.79mL의 농축 종속영양 배양액이 필요하며, 및 14.88mL의 CoYBG-11 배지.
- 13,000 x g 를 사용하여 실온에서 10분 동안 배양물을 원심분리합니다. 무균 상태에서는 상층액을 디캔팅하고 폐기합니다.
참고: S. cerevisiae 는 S. elongatus 에 비해 더 큰 세포(2x-3x)이므로 상대 원심력(RCF; xg)이 적기 때문에 펠릿에 사용할 수 있지만 상층액이 투명하게 변하도록 주의하십시오. 시아노박테리아 바이오매스를 채취하기 위해 13,000 x g 미만도 사용할 수 있지만 원심분리에 더 오랜 시간이 필요합니다(예: 4,000 x g 를 20분 동안 사용할 수 있음). - 시아노박테리아 펠릿을 25mL의 멸균 CoYBG-11 배지에 재현탁합니다. 이전과 동일한 원심분리 조건을 사용하고, 그 후에는 상층액을 버리고 디캔트합니다.
- S. elongatus에 대해 이 과정을 두 번, S. cerevisiae에 대해 4번 더 반복합니다.
알림: 시아노박테리아와 종속영양체(S. cerevisiae)의 펠릿을 세척하여 잔류 배지 성분을 제거하는 것이 중요합니다. S. cerevisiae 는 이전에 자당이 있는 상태에서 자랐기 때문에 세척 단계를 늘리면 가능한 잔류 당을 제거할 수 있습니다. - OD가 확인되면 멸균 조건에서 250mL 배플 플라스크에 S. elongatus 및 S. cerevisiae 의 부피를 결합하고 CoYBG-11 을 최종 부피 50mL에 추가합니다. 이전에 준비된 1M 용량의 IPTG를 1mM의 최종 농도로 희석합니다(이는 시아노박테리아가 자당을 배지로 내보내도록 유도하기 위한 것입니다). 50mL 배양의 경우 1M IPTG 스톡에서 50μL를 사용합니다.
참고: 배플 플라스크는 시아노박테리아에 대해 더 나은 통기 및 혼합을 제공하기 때문에 일반적으로 더 좋습니다. - 이전 단계의 플라스크를 200μmol 광자·m-2·s-1 LED 조명에 놓고 30°C에서 2% CO2 가 보충되고 150rpm(25mm 투사) 및 75% 습도에서 궤도 흔들림이 있습니다.
참고: 위에서 언급한 조건은 다를 수 있으며 실험 결과에 따라 최적화해야 합니다. - 12시간 또는 24시간마다 멸균 샘플링(1mL)을 통해 배양물의 성장을 모니터링합니다(섹션 2 참조). 선택적으로 샘플을 사용하여 표준 라이브 셀 현미경 검사를 수행하고 적절한 배지에서 효모 세포의 집락 형성 단위(CFU14)를 측정하여 세포 형태 및 적합성을 추적하고 살아있는 종속영양 세포의 양과 오염을 모니터링할 수 있습니다.
참고: CFU는 액체에서 고체 매질로 배양에서 전이할 때 상당한 스트레스를 경험할 수 있는 시아노박테리아 종의 정확한 세포 밀도를 모니터링하는 방법으로 권장되지 않습니다. 따라서 효모 추출물 펩톤 덱스트로스(YEPD)15와 같은 효모 파트너 종의 성장에 적합한 풍부한 배지를 가진 한천 플레이트를 선택해야 합니다.
- 공동 배양의 접종을 위해, 원심 분리에 의해 두 preculture를 모두 풍부하게하십시오.
| 화합물 | BG-11 (농도 mg/L) | CoY BG-11 (농도 mg/L) |
| 재질 보기 NaNO3 | 1500 | 1500 |
| 케이2HPO4 | 40 | 40 |
| 마그네슘4·7H2O | 75 | 75 |
| 염화 2 · 2시간2영형 | 36 | 36 |
| 구연산 | 6 | 6 |
| 구연산 철 암모늄 | 6 | 6 |
| EDTA (디 소듐 염) | 1 | 1 |
| 나2CO3 | 20 | 20 |
| 미량 금속 조성 | ||
| H3보3 | 2.86 | 2.86 |
| ZnSO4·7H2O | 0.222 | 0.222 |
| 공동(3호)2 · 6H2O | 0.0494 | 0.0494 |
| 재질 보기 MnCl2·4H2O | 1.81 | 1.81 |
| 쿠소4 · 5시간2영형 | 0.079 | 0.079 |
| 나무4·2H2O | 0.39 | 0.39 |
| 추가 | ||
| 헵소 | 7160 | 7160 |
| 효모 질소 염기(YNB), 아미노산 없음, 황산암모늄 없음 | – | 3608, 120013년 |
| pH 8.3 적정제 | 코 | 코 |
| KPO3 | – | 118 |
| 설탕 (종속 영양 생물에만 해당) | – | 13690 |
표 1: BG-11 및 CoYBG-11의 매체 조성. CoYBG-11의 효모-질소 염기 농도의 농도는 발표된 자원 8,12,13에서 파생됩니다.
2. 공동 문화의 성장을 모니터링하기 위한 도구 및 방법론
참고: 이 프로토콜은 현미경 검사 및 계수 챔버와 같은 간단하지만 작업 집약적인 기술부터 입자 계수기 및 단일 세포 유세포 분석과 같은 고처리량 응용 분야에 이르기까지 공동 배양 분석 및 모니터링을 위한 지침입니다. 실제 공동 배양과는 별개로, 포괄적인 분석을 위해 단일 미생물의 축삭 배양을 포함하는 것이 좋습니다. 분석의 일반적인 시작점으로 문화권의 OD를 결정합니다. 이 섹션에서는 세포 밀도(즉, OD)의 상대적 측정값을 부피당 세포 수의 절대값으로 변환하는 데 사용할 수 있는 다양한 방법론에 대해 자세히 설명합니다. OD750 측정은 400-700nm 파장의 흡수로 인해 시아노박테리아 배양의 세포 밀도를 측정하는 데 자주 사용되는 반면, OD600 은 종속영양 유기체에 사용됩니다. 두 측정 모두 임의의 단위로 대략적인 안내 값을 제공합니다. 값은 상품마다 크게 다를 수 있습니다.
- 카운팅 챔버를 통한 현미경 정량화
참고: 계수실(즉, Neubauer(개선)/혈구계)는 형태학적으로 쉽게 구별할 수 있는 세포로 구성된 공동 배양의 구성을 결정하는 간단하고 저렴한 수단입니다. i.e.을 셀 모양(그림 1A). 두꺼운 유리 현미경 슬라이드와 커버 유리로 구성되어 있습니다. 커버 유리가 슬라이드 상단에 올바르게 배치되면 새겨진 그리드(그림 1B-디). 그리드의 정의된 영역을 계산하여 현탁액의 세포 농도를 계산할 수 있습니다. 계수 챔버는 다양한 깊이에서 사용할 수 있습니다. 셀 두께에 따라 적절한 챔버 깊이를 선택해야 합니다. 다음과 같은 곰팡이 세포의 경우 U. maydis 또는 S. cerevisiae, 0.1mm의 깊이가 적합합니다. 이 챔버 깊이는 시아노박테리아와 같은 작은 세포에 비해 너무 커서 세포가 챔버에 떠 있습니다. 깊이가 0.02mm인 챔버는 시아노박테리아와 시아노박테리아 및 곰팡이의 공동 배양 모두에 적합합니다.- 계수실과 덮개 유리에 먼지와 세포가 없는지 확인하십시오.
알림: 사용 직전에 70%(v/v) 에탄올과 보푸라기가 없는 티슈로 세척하는 것이 좋습니다. - 커버 유리를 올바르게 배치하려면 약간의 압력으로 두 개의 지지대에 밀어 넣되 파손되지 않도록 주의하십시오.
알림: 이 응용 프로그램에는 특정하고 두꺼운 커버 유리가 필요합니다(챔버 설명서 참조). 커버 유리는 조립 중에 깨지는 경우가 있으므로 교체품을 준비하는 것이 좋습니다. - 커버 유리가 적절하게 배치되면 두 유리 표면 사이의 소위 뉴턴 고리(그림 1B)를 관찰하고 커버 유리가 더 이상 미끄러지지 않는지 확인합니다.
알림: 조립하기 전에 커버 유리에 부드럽게 숨을 들이쉬면 종종 결과가 향상됩니다. - 세포 현탁액을 철저히 혼합하고 세포 현탁액의 몇 마이크로리터(챔버 깊이에 따라 ~2-10 μL)를 챔버 가장자리에 적용하고 모세관 힘을 통해 완전히 채워지도록 합니다. 예를 들어, 큰 정사각형당 20-200개의 세포가 생성되는 등 신뢰할 수 있는 계수가 가능하도록 세포 현탁액을 적절하게 희석합니다.
- 광학 현미경의 적절한(예: 10x) 대물렌즈(명시야 또는 위상차 모드)를 사용하고 계수 챔버 그리드 라인에 초점을 맞춥니다. 방향은 그리드에 의해 제공됩니다. 주어진 셀 크기에 적합한 사각형의 셀 수를 세기 시작합니다.
알림: 휴대용 카운터 또는 계산을 위한 적절한 스마트폰 애플리케이션과 같은 기술 보조 도구를 사용하십시오(예: 일반 "사물 카운터" 또는 매우 유용한 기능을 제공하는 특수 혈구계 앱). 이중 계산을 피하기 위해 그리드 경계에 있는 셀을 계산하는 규칙을 정의합니다. 예를 들어, 각 사각형의 위쪽 및 왼쪽 테두리에 있는 셀은 계산되고 오른쪽 및 아래쪽 테두리에 있는 셀은 제외될 수 있습니다. 대체 규칙이 존재함16. 형광 현미경을 사용하면 자가 형광 또는 형광 마커를 기반으로 세포를 구별할 수도 있습니다. - 예를 들어, 모서리에 있는 4개의 큰 사각형을 모두 세고 나서 각 셀 유형에 대한 평균값을 결정합니다. 이러한 평균값을 사용하여 사용된 정사각형의 면적과 주어진 챔버의 깊이(제조업체 정보 참조)를 사용하여 세포 농도를 계산합니다( 대표 결과 참조).
참고: 셀 크기에 따라 다른 사각형을 사용하여 계산할 수 있습니다. 효모 세포와 시아노박테리아 세포의 경우, 모서리에 있는 4개의 큰 사각형이 잘 작동하고 있습니다(그림 1D, 빨간색 직사각형). - 사용 후에는 샘플의 요구 사항을 고려하여 챔버를 청소합니다(예: 유전자 변형 유기체를 적절하게 비활성화).
- 계수실과 덮개 유리에 먼지와 세포가 없는지 확인하십시오.
- 입자 계수기를 사용한 정량화
참고: 공동 배양에서 세포의 특성에 따라 입자 계수기를 적용하여 파트너의 세포 수를 결정할 수 있습니다. 이 방법을 적용하려면 세포의 크기가 크게 달라야 합니다. 예를 들어, 박테리아 세포와 효모 세포를 구별할 수 있습니다. 컨소시엄의 단일 미생물의 축삭 배양은 결과를 해석할 수 있도록 포함되어야 합니다. 크기가 비슷하거나 파트너가 2개 이상인 세포가 있는 공동 배양은 섹션 2.1 또는 2.3에 설명된 방법론으로 분석해야 합니다.- 준비
- 배양물의 OD를 0.1로 조정하고 총 부피 10mL에서 등장성 측정 버퍼(장치 설명서 참조)로 샘플을 1,000배 희석합니다. 모든 샘플에 대한 기술적 삼중을 준비합니다.
- 실험에 적합한 모세관 공극 크기를 선택합니다.
참고: 기공 크기는 공동 배양에서 가장 작은 세포의 범위 내에 있어야 하지만 더 큰 세포도 포함해야 합니다. 세포 분열로 인해 발생할 수 있는 세포 응집으로 인해 모공이 막힐 수 있으므로 너무 작게 가지 마십시오. 사용 가능한 기공 크기는 다릅니다. 예를 들어, 45μm의 모세관은 직경이 0.7μm인 세포를 감지하기 시작합니다(U. maydis 및 S. cerevisiae 3.5 μm-5.5 μm, S. elongatus/Synechocystis sp. ~1-2.5 μm).
- 입자 계수기(particle counter)에 있는 세포의 정량화(quantification of cells in a particle counter)
- 장치를 시작합니다. 결국 품질 관리를 위한 자체 테스트를 수행하십시오(특정 장치의 설명서 참조).
- 샘플을 샘플 컵에 넣고 뚜껑을 단단히 닫은 다음 컵을 약간 기울여 샘플을 혼합합니다.
알림: 거품과 거품을 피하십시오. 항상 한 번 측정할 때마다 한 컵을 사용하십시오. 컵은 여러 번 세척하고 재사용 할 수 있습니다. 제거할 수 없는 잔여물이 있는 경우 컵을 버리십시오. 샘플이 컵에 더 오래 남아 있으면 측정하기 전에 다시 혼합하십시오. - 세포 응집체가 단일 세포보다 클 수 있기 때문에 0-30μm 범위에서 기록합니다.
참고: 먼저, 공동 배양 파트너의 각 축삭 배양을 사용하여 세포의 크기에 대한 아이디어를 얻고 세포의 크기로 구별할 수 있는지 확인합니다. - 평가 후 동일한 방식으로 공동 배양의 구성을 결정하십시오.
- 준비
- Consortium single-cell 유세포 분석법을 사용한 정량 분석
참고: Single-cell 유세포 분석은 파트너를 크기/광 산란 특성 또는/및 형광으로 구별할 수 있다는 점을 감안할 때 공동 배양에서 개별 파트너의 세포 수를 결정하는 데 사용할 수 있는 고처리량 방법입니다. 중요한 것은 방법 확립을 위해 공동 배양 파트너의 축삭 배양이 세포분석기에서 특성을 분석하고 그에 따라 개체군 게이트를 조정하는 데 필요하다는 것입니다. 형광 리포터를 운반하는 균주의 경우, 형광 리포터가 없는 균주가 음성 대조군으로 필요합니다. 세포 분석기 응용 분야에 대한 기본적인 지식이 있으면 아래 프로토콜을 성공적으로 구현할 수 있습니다17,18,19,20,21. 이 예에서는 3가지 미생물의 인공 혼합물을 분석합니다( 대표적인 성과): Synechocystis (세포질 리포터 fluorophore, mVenus를 발현)와 2 개의 효모는 S. cerevisiae 그리고 U. maydis. 의 정확한 구별을 위해 S. cerevisiae 그리고 U. maydis, 형광 마커는 세포질 녹색 형광 단백질(GFP) (U. maydis eGFP) 및 적색 형광 단백질(RFP) mKate2(S. cerevisiae mKate2)를 사용하여 기본 분자 클로닝 기술을 사용할 수 있습니다. 가능한 경우, 리포터의 게놈 통합이 있는 균주는 연속 선택 압력에 대한 요구 사항을 피하기 위해 선호되어야 합니다.- 시료 전처리
- 분석할 주어진 성장 단계에서 배양물의 OD를 측정합니다.
- 샘플의 OD를 조정하십시오.amp측정에 적합한 범위로 설정합니다. 신선한 배양 배지를 사용하여 시아노박테리아에 대해 약 0.05-0.5의 OD750 을 사용하고 효모에 대해 0.2-1.5의 OD600 을 사용합니다(최종 부피: 최소 500μL). 공동 배양의 경우 ~0.1에서 0.5 사이의 OD750 을 목표로 하고 희석 계수를 기록합니다. 계산을 위해 1.1.3.1단계에 제공된 공식을 적용합니다. 이렇게 하면 10 μL/분의 유속으로 세포 수가 1,000 - 10,000 cells/s 범위로 유지됩니다(6,000 - 60,000 cells/μL에 해당).
참고: 희석을 위해 자가형광 및 입자가 없는 매체 또는 완충액(예: BG-11)을 사용할 수 있습니다. 측정에서 입자로 나타날 수 있는 효모 추출물과 같은 복잡한 성분이 포함된 매체에 주의하십시오. - 각 샘플 셀 현탁액 300μL를 96웰 플레이트(표준 원형 웰, 투명하고 평평한 바닥)의 개별 웰로 옮깁니다.
참고: 또는 플레이트 리더 모드 대신 반자동 시료 모드를 사용하는 경우 0.5-2mL를 형광 활성화 세포 분류(FACS) 튜브로 옮깁니다.
- 세포분석 측정
- 유세포 분석기를 시작하고 제조업체의 지침에 따라 시작 프로그램 및 품질 관리(QC)를 수행합니다.
- 로드(플레이트 로더 또는 반자동 샘플링)하고 샘플을 실행합니다. 각 공동 배양 파트너의 축삭 배양을 사용하여 대조군 샘플부터 시작하여 공동 배양 샘플에서 분리하는 것을 목표로 하기 전에 특성을 기반으로 각 종을 식별합니다.
- 샘플과 관련된 형광 및 산란(예: 전방 산란[FSC] 또는 측면 산란[SSC], 높이[-H] 또는 면적[-A]) 채널에 대한 열린 도트 플롯 및/또는 히스토그램.
- 필요한 세포 집단을 찾고 임계값을 조정하여 배지에서 기술적 노이즈와 작은 입자를 제외합니다.
참고: FSC-width에서 FSC-H 또는 SSC-H 도트 플롯에서 FSC-H에서 세포 집단을 검색하고 박테리아와 같은 작은 세포(일반적으로 1-3μm)에 대해 선형에서 대수로 스케일을 조정하는 것이 좋습니다. FSC-H 또는 SSC-H 채널에서 임계값을 설정합니다. 직경이 1μm 미만인 세포의 경우 더 나은 해상도를 위해 405nm의 보라색 레이저에서 보라색 SSC(VSSC)를 사용합니다. - 관심 있는 각 채널의 게인을 25-2,500 범위로 수동으로 조정합니다. 네거티브 대조군(예: 형광 마커가 없는 경우)이 1 x 102-1 x 103 의 범위에 있고 포지티브 대조군(예: 형광 마커 사용)이 1 x 105-1 x 106 범위에 있도록 게인을 조정하여 최상의 분리를 얻고 1 x 101-1 x 107 범위 내에서 유지됩니다.
참고: QC의 표준 이득도 좋은 참조로 유지할 수 있습니다. - 시료 내 세포 농도를 측정하려면 기록 기능을 사용하여 정의된 시료 부피를 기록합니다(예: 10μL/분의 유속에서 10μL). 나중에, 이 부피에 대해 측정된 개별 세포 수를 기록된 부피로 나누어 각 집단에 대한 μL당 세포 수를 얻습니다.
참고: 배양/샘플의 세포 농도를 계산할 때 초기 희석 계수를 염두에 두십시오.
- 세포 집단의 게이팅/선택
참고: 측정을 실행하는 데 사용되는 장치와 별개로, 분석 및 게이팅은 아래와 같이 세포분석기 운영 소프트웨어 또는 상용 분석 소프트웨어에서 수행할 수 있습니다22. 또한 비독점 소프트웨어를 사용하여 유세포 분석 데이터를 분석하는 것은 FlowCytometryTools와 같은 다양한 오픈 소스 도구를 사용하여 효율적으로 수행할 수 있습니다23 또는 FlowKit, FlowUtils 활용24 Python의 패키지. 오픈 사이언스의 정신에 따라 이 프로토콜은 기본 데이터 탐색을 위해 이러한 패키지를 사용하는 방법을 보여주는 모범적인 JupyterNotebook을 공유합니다. 물론 대체 소프트웨어는 유세포 분석 생물 정보학을 수행하는 데 똑같이 적합할 수 있습니다25.- 점도표에서 세포 주위에 게이트를 그리거나 히스토그램에서 피크 주위에 구분선 또는 선 세그먼트를 설정하여 특정 세포 집단을 선택합니다(그림 4).
참고: 프로그램의 자동 게이트 기능을 참고로 사용할 수 있습니다. - 스펙트럼의 적색 영역에서 광영양 유기체(주로 엽록소에 의해 발생)의 자가형광을 기반으로 광영양 유기체와 종속영양 유기체에 대한 신호를 분리합니다. 예: APC-H 채널의 히스토그램(638nm에서 여기(예: 638nm에서 여기), 660/10nm에서 방출(em.))을 사용하고 오른쪽 피크(광영양 = 엽록소 자가형광 양성 모집단)와 왼쪽 피크(종속영양 = 엽록소 자가형광 음성 모집단) 사이의 수직 구분선을 설정합니다(그림 4A).
참고: 형광 마커는 동일한 배양 내에서 두 개(또는 그 이상)의 광영양 파트너를 구별하는 데 사용할 수도 있습니다(예: Synechocystis의 경우 mVenus). - 형광 마커 또는 산란 특성(가능한 경우 FSC, SSC)에 따라 두 개의 종속영양 유기체를 분리합니다.
참고: 산란 특성에 따른 분리는 예를 들어 세포가 산란에서 눈에 띄는 변화를 초래하는 세포 크기 또는 형태가 다른 경우에만 적용할 수 있습니다(예: Synechocystis: ~1.5-3 μm 직경의 구형 세포 대 U. maydis 길이 10 μm 및 약 1-2 μm 직경26). - 예: "종속영양" 집단만 표시하는 FITC-H 채널(예: 488 nm, em.: 525/40 nm)의 히스토그램을 사용하여 GFP를 포함하는 종속영양 세포(예: U. maydis eGFP)와 GFP가 없는 세포(예: S. cerevisiae RFP/mKate2)를 구별합니다(그림 4B). 또는 "종속영양" 집단만 표시하는 PC5.5-H 채널(예: 561 nm, em.: 710/50 nm)의 히스토그램을 사용하여 RFP를 포함하는 종속영양 세포(예: S. cerevisiae RFP/mKate2)와 RFP가 없는 세포(예: U. maydis GFP)를 구별합니다(그림 4C).
참고: 측정하는 동안 두 개(또는 그 이상)의 서로 다른 세포 유형이 함께 "고정"될 수 있음을 명심하십시오. 이러한 "이중선" 또는 "다중선"은 두 형광 마커의 점도표(예: 광영양체의 이중항 및 GFP 태그가 지정된 종속영양 파트너를 식별하기 위한 FITC-H에 대한 APC-H)에서 쉽게 검출할 수 있습니다. - Python의 기본 데이터 분석은 이 Jupyter(https://git.rwth-aachen.de/computational-life-science/cytoflow)의 지침을 따릅니다.
참고: 이 노트북은 세 가지 단계의 데이터 분석을 지원합니다. i) 데이터 가져오기: FCS 파일을 가져옵니다. ii) 품질 관리 수행: 데이터 무결성 및 이벤트 수를 확인합니다. iii) 데이터 시각화: 히스토그램, 산점도 및 밀도 플롯과 같은 플롯을 사용하여 데이터를 시각화합니다. 또한, 그림 4 를 재현하는 관심 집단을 분리하기 위해 게이팅을 수행하는 방법이 표시됩니다.
장기 프로젝트에서 공동 배양 분석을 용이하게 하기 위해 연구자는 위의 방법을 사용하여 세포 수를 계산하고 OD 값을 절대 세포 수와 비교하는 표준 곡선을 만드는 것이 좋습니다. 광학 밀도는 종, 균주, 성장 조건 및 분광계에 따라 달라질 수 있는 상대적 측정입니다. 따라서 OD 값을 셀 번호로 정확하게 변환하기 위해서는 "사내" 표준 곡선의 생성이 필요합니다.
- 점도표에서 세포 주위에 게이트를 그리거나 히스토그램에서 피크 주위에 구분선 또는 선 세그먼트를 설정하여 특정 세포 집단을 선택합니다(그림 4).
- 시료 전처리
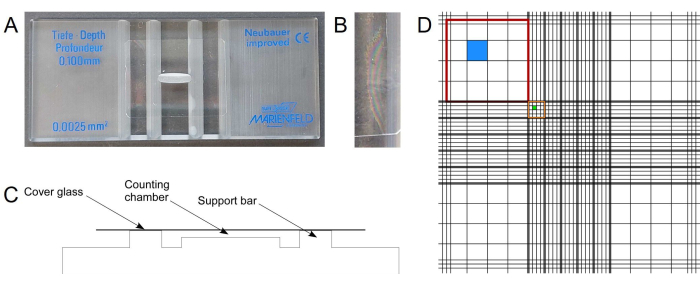
그림 1: 형태학적으로 구별할 수 있는 세포의 현미경 정량화. (A) Neubauer 계수실. (B) 뉴턴의 링은 특수 커버 슬립의 올바른 위치를 나타냅니다. (C) 세포 계수를 위해 정의된 부피의 중앙 공동이 있는 챔버 구조의 개략적 묘사. (D) 묘사된 Neubauer 계수실의 그리드는 1mm2 (빨간색) 크기의 9개의 큰 사각형으로 구성됩니다. 모서리에 있는 4개의 큰 사각형은 16개의 사각형(파란색)으로 더 나뉩니다. 중앙의 큰 사각형은 0.04mm2 (주황색) 크기의 25 개의 그룹 사각형으로 나뉩니다. 각 그룹 사각형은 16개의 가장 작은 사각형(녹색)으로 구성됩니다. 이 수치는 공개적으로 사용 가능한 제조업체의 정보를 기반으로 생성되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
phototrophic S. elongatus 및 종속영양 효모의 공동 배양 확립
우리는 이전에 Synechococcus elongatus PCC 7942와 S. cerevisiae의 다양한 아종을 공동 배양하여 상세한 결과를 보고한 바 있습니다. 이 시아노박테리아/효모 쌍을 사용한 공동 배양 결과에 대한 포괄적인 설명은8을 참조하십시오. 간결성과 정확성을 위해 이러한 결과는 여기에 재현되지 않습니다. 간단히 말해서, 이전 결과는 장기적인 시아노박테리아-효모 공동 배양을 확립하는 데 중요한 여러 고려 사항을 나타냅니다. 주요 관심사는 시아노박테리아가 고정 탄소의 유일한 형태를 제공하는 조건에서 효모가 생존할 수 있는 능력은 효모 균주가 자당을 활용할 수 있는 효율성에 크게 의존한다는 것입니다. 자당을 보다 효율적으로 대사하도록 진화하거나 조작된 S. cerevisiae 균주27,28은 공동 배양 성장 모드로의 전환에서 생존할 가능성이 더 높았고, 더 높은 세포 밀도를 달성했으며, 장기(며칠에서 몇 주) 시아노박테리아 공동 배양 실험에서 더 높은 견고성을 보여주었습니다8. 공동 배양을 접종하는 초기 단계는 효모 생존력에 특히 중요했는데, 이는 배양 희석, 배지 조성 전환 및/또는 더 농축된 탄소원의 회수에 대한 스트레스 때문일 수 있습니다. 따라서 공동 배양 시작 시 더 풍부한 성장 배지에서 최소 탄소 가용성으로 쉽게 전환하기 위한 노력은 실험 성능과 일관성을 향상시킬 수 있습니다(1.2.1.1 단계 참조). 또한, S. cerevisiae는 S. elongatus의 조밀한 배양물에 접종되었을 때 고산소와 일치하는 스트레스 반응을 보였으며, 이는 산소 광합성의 주요 부산물인 O2의 형성과 일치했습니다. 그러므로, 시아노박테리아 파트너의 과다 증식 및/또는 "낮/밤" 빛의 순환을 번갈아 가며 방지하려는 노력은 장기적인 공동 배양에서 S. cerevisiae의 생존 가능성을 실질적으로 연장할 수 있습니다. Discussion section(논의 섹션)에서 axenic control samples와 관련된 공동 배양에서 흔히 발생하는 현상에 대한 추가 요약을 참조하십시오.
다양한 방법론을 사용한 공동 배양의 성장 모니터링
다음 섹션에서는 세 가지 다른 방법을 사용하여 Synechocystis와 두 개의 효모 S. cerevisiae와 U. maydis의 인위적으로 혼합된 삼자 컨소시엄의 예시적인 정량화가 설명됩니다. 혼합물에 대해 단일 배양의 OD750(시아노박테리아의 경우) 및 OD600(종속영양의 경우)을 측정하고 OD 0.1로 조정했습니다. 단일 배양물은 광학 밀도를 사용하여 1:1:1의 비율로 혼합되었습니다(이는 세포 수와 구별되며, 위 참조). 세포분석기에서 효모의 구별을 용이하게 하기 위해, 세포질 mKate2 (유전자형: URA3Δ/pTDH3::mKate2; 균주: S. cerevisiae mKate230)를 구성적으로 생산하는 유전자 변형 S. cerevisiae FY1679-O1B29 및 구조적으로 eGFP를 생산하는 U. maydis 균주 AB3331 (유전자형: pep4Δ/pRpl40::egfp; 균주: U. maydis eGFP32)가 사용되었습니다. 주목할 점은 시아노박테리아 균주에는 노란색 형광 단백질 버전 mVenus(Synechocystis sp. PCC 6803 pSHDY-Pcpc560-mVenus, 균주 Synechocystis mVenus,33)과 유사하며, 또한 광합성 기계의 존재로 인해 전형적인 강한 자가형광을 나타낸다.
계수 챔버를 사용한 현미경 정량화: 3 가지 미생물의 인공 혼합물에 사용되는 모든 세포 유형은 현미경으로 쉽게 구별 할 수 있습니다 (그림 2A) : Synechocystis와 S. cerevisiae 는 직경이 크게 다른 구형 세포로 대표되지만 (Synechocystis : 약 직경 1.5-3 μm, S. cerevisiae : 약 직경 3-6 μm), U. maydis 세포는 길쭉한 시가 모양의 형태와 길이가 최소 10 μm (그림 2A)). 이러한 명확한 형태학적 특성으로 인해 혼합물의 각 파트너를 정확하게 정량화할 수 있습니다. 예시적인 예로, 37, 18, 36 및 21 S. cerevisiae 세포가 각각 4개의 큰 사각형에 계수됩니다(그림 2B). 평균값은 28개의 셀입니다. 사용된 혈구계의 큰 정사각형은1mm2 의 면적을 가지며 챔버 깊이가 0.02mm이기 때문에 0.02μL당 28개의 세포가 생성됩니다. 이는 1,400 cells/μL에 해당하며, 이는 1.4 x 106 cells/mL에 해당합니다. 다른 세포 유형을 세고 그에 따라 농도를 측정했습니다(그림 2C, 표 2).
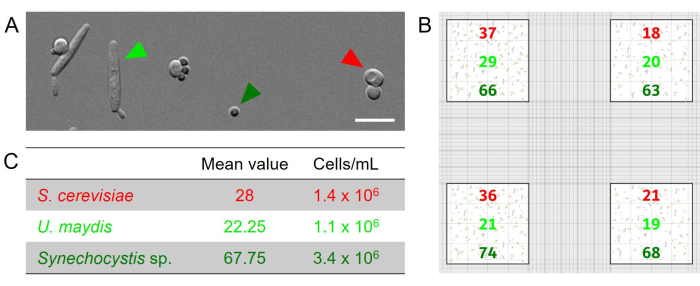
그림 2: Synechocystis와 효모 S. cerevisiae 및 U. maydis로 구성된 삼자 컨소시엄의 현미경 정량화. (A) 표시된 미생물의 인위적으로 결합된 혼합물의 현미경 차등 대비(DIC) 이미지. 모든 종은 형태에 따라 쉽게 구별 할 수 있습니다. 스케일 바 : 10 μm. (B) OD가 0.1 인 axenic cultures를 기반으로 표시된 균주의 인위적으로 조립 된 혼합물에 대한 예시 계산 결과. 그리드 가장자리에 있는 4개의 큰 사각형을 분석했습니다( 그림 1D의 빨간색 표시). (C) 4개의 정사각형에서 계수된 서로 다른 세포의 평균값은 다음 방정식을 사용하여 현탁액 내 세포의 농도를 계산하는 데 사용되었습니다: 평균값/(챔버 깊이[0.02mm] x 계수된 정사각형의 크기[1mm,2] x 1,000). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
입자 계수기를 사용한 정량화: 파티클 카운터는 크기에 따라 현탁액의 파티클 수를 결정합니다. 예제 데이터 세트에서는 45μm 모세관이 있는 입자 계수기가 사용되었습니다. S. cerevisiae 와 U. maydis 세포는 유사한 치수를 나타내기 때문에 입자 계수기에서 구별할 수 없지만 더 작은 Synechocystis 세포는 명확하게 분리할 수 있습니다. 따라서 단일 배양물을 분석하면 두 효양에 대해 3-6μm를 참조하는 동일한 위치에서 피크가 나타납니다(그림 3A). 측정하는 동안 음압이 가해져 세포가 모세혈관으로 들어갑니다. 이것은 장치가 변경에 따라 입자 크기를 결정할 수 있도록 전기 저항을 잠시 변경합니다. 혼합 배양액에서 두 개의 뚜렷한 피크를 검출할 수 있는데, 하나는 더 작은 시아노박테리아 세포와 관련이 있고 다른 하나는 두 효모 종의 결합 분획을 나타냅니다(그림 3B). 중요한 것은 살아있는 세포를 반영하는 피크 외에도 세포 파편과 더 작은 입자에 해당하는 약 1μm(그림 3)에서 추가 피크가 검출되었다는 것입니다. 예상보다 큰 직경에서 나타나는 신호는 세포 응집으로 인해 발생할 수 있습니다. 주목할 점은 피크의 모양이 세포의 균질성을 반영한다는 것입니다: 날카로운 피크는 매우 균질한 세포를 보여주며, 이는 공동 배양에 적합하지 않습니다. 공동 문화에서는 매우 넓은 정점 또는 공동 문화의 파트너가 분명히 크기가 다른 경우 두 개의 정점을 볼 수 있을 것으로 예상됩니다. 종속영양 균류의 축삭 배양의 경우, 봉우리는 중간 너비의 봉우리일 수 있습니다.
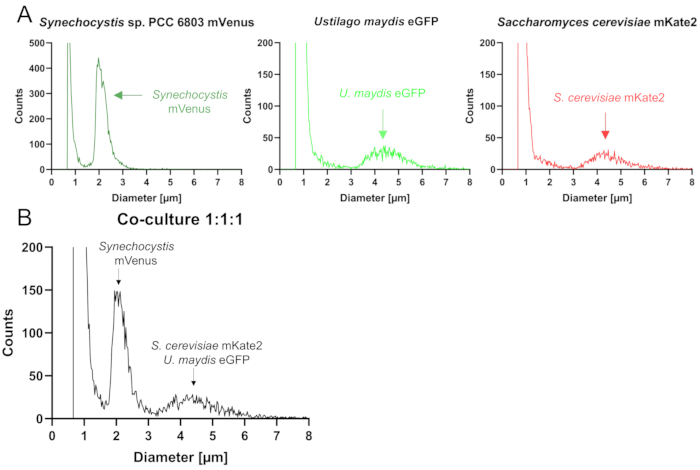
그림 3: 입자 계수기를 사용하여 Synechocystis mVenus와 효모 S. cerevisiae mKate2 및 U. maydis eGFP로 구성된 삼자 컨소시엄의 정량화. (A) 그래프에 표시된 대로 세 가지 균주의 축삭 배양 분석의 시각적 출력(45μm 모세관이 있는 세포 계수기). (B) 동일한 조건을 사용하는 인공 삼자 컨소시엄의 분석을 위한 예시적인 시각적 출력 파일. 입자 계수기는 두 번째 피크로 표시되는 두 효모 종의 구별을 지원하지 않습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
단세포 세포 분석을 사용한 정량화: single-cell 유세포분석기를 사용하면 (자동) 형광 및 광 산란 특성에 따라 다양한 세포 집단을 쉽게 구별할 수 있습니다. 광영양 세포(Synechocystis)는 APC-H 채널에서 측정된 광합성 색소의 적색 자가형광을 기반으로 종속영양 세포(U. maydis 및 S. cerevisiae)와 구별할 수 있습니다(그림 4A). 광영양 세포와 종속영양 세포의 초기 분리를 기반으로 두 종속영양 집단은 FITC-H 채널의 형광 마커 eGFP와 PC5.5 채널의 mKate2를 기반으로 구별할 수 있습니다(그림 4B, C). 세 개체군(FSC-H 및 FSC-Width) 모두의 산란 특성을 보여주는 점도표에서 개체군은 개체군의 일부 미미한 중복만으로 구별할 수 있습니다(그림 4D).
이 방법을 사용하여 희석된 샘플 중 10μL를 10μL/분의 유속으로 분석하여 1분 내에 약 70,000개의 세포를 정량화할 수 있었습니다. 이러한 세포의 약 60%는 Synechocystis (4.23 x 106 cells/mL)에 할당될 수 있었고, 나머지 40%는 U. maydis (1.35 x 106 cells/mL)와 S. cerevisiae (1.36 x 106 cells/mL, 그림 4E, 표 2)에 균등하게 분포되었습니다.
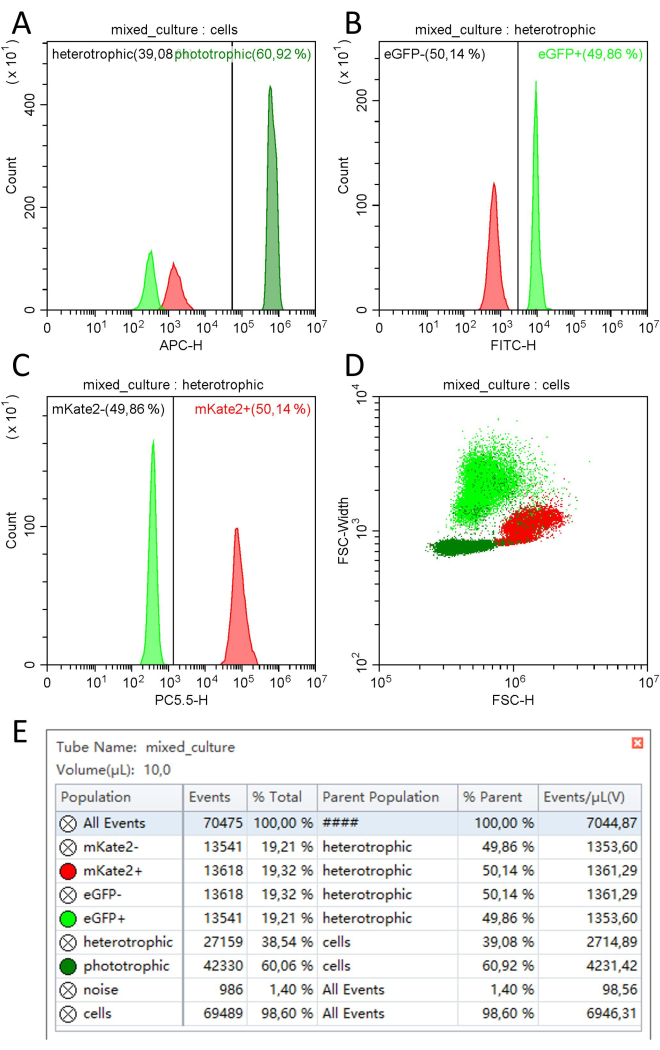
그림 4: 단일 세포 유세포 분석을 사용한 cyanobacterium Synechocystis mVenus와 효모 U. maydis eGFP 및 S. cerevisiae mKate2로 구성된 삼자 컨소시엄의 정량화. APC-H 채널(예: 638 nm, em.: 660/10 nm)의 모든 세포는 자가형광에 따라 광영양 세포와 종속영양 세포를 구별하는 데 사용됩니다. (B) 녹색 형광 특성에 따라 U. maydis eGFP 및 S. cerevisiae mKate2 세포를 구별하는 데 사용되는 모든 종속영양 세포의 FITC-H 채널(예: 488 nm, em.: 525/40 nm)의 이벤트 횟수 및 형광의 히스토그램. (C) 적색 형광 특성을 기반으로 U. maydis eGFP 및 S. cerevisiae mKate2 세포를 구별하는 데 사용되는 모든 종속영양 세포의 PC5.5-H 채널(예: 561nm, em.: 710/50nm)의 이벤트 횟수 및 형광의 히스토그램. (D) 형광 특성이 없는 세포 집단을 식별하는 데 사용되는 FSC-H 채널에 대한 FSC-Width의 산란 신호의 점도표. (E) 사건 수, 백분율, 산술 평균 및 관련 형광 채널의 표준 편차(SD)를 포함한 개체군 통계. 세포의 총 양은 크기 및 형광과 함께 10μL/분의 유속으로 10μL의 부피로 측정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
서로 다른 정량화 방법의 출력을 비교적으로 시각화하기 위해 결정된 세포 수가 다음 두 표(표 2 및 표 3)에 제시되어 있습니다. 앞서 설명한 세 가지 방법을 사용하여 측정된 세포의 최종 농도는 모든 방법에 대해 유사한 범위에 있습니다. 유세포 분석기는 가장 높은 샘플 크기를 제공하고 입자 계수기와 현미경 정량화가 그 뒤를 잇며, 두 방법 간에 약 한 자릿수 감소합니다.
| 공동 문화 1:1:1 | 광도 계 | 세포분석기 | 입자 계수기 | 현미경 카운팅 챔버 | ||||
| 유기체 | 외경750/600 | 세포 수 | 세포/mL | 세포 수 | 세포/mL | 세포 수 | 세포/mL | |
| Saccharomyces cerevisiae mKate2 | 0.0333 | 13,618 | 1.36 엑스 106 | 1,546* | 2.58 x 106* | 112 | 1.40 엑스 106 | |
| Ustilago maydis eGFP | 0.0333 | 13,541 | 1.35 엑스 106 | 89 | 1.11 엑스 106 | |||
| Synechocystis sp. PCC 6803 m금성 | 0.0333 | 42,330 | 4.23 엑스 106 | 3,094 | 5.16 엑스 106 | 271 | 3.39 엑스 106 | |
표 2: 다양한 정량화 방법의 비교: 인공 혼합 배양. U. maydis와 S. cerevisiae는 입자 계수기(*)를 사용하여 구별할 수 없습니다.
| 단일 문화권 | 광도 계 | 세포분석기 | 입자 계수기 | 현미경 카운팅 챔버 | ||||
| 유기체 | 외경750/600 | 세포 수 | 세포/mL | 세포 수 | 세포/mL | 세포 수 | 세포/mL | |
| Saccharomyces cerevisiae mKate2 | 0.1 | 38,936 | 3.89 엑스 106 | 1,928 | 3.21 엑스 106 | 403 | 4.03 엑스 106 | |
| Ustilago maydis eGFP | 0.1 | 36,927 | 3.69 엑스 106 | 2,465 | 4.11 엑스 106 | 307 | 3.07 엑스 106 | |
| Synechocystis sp. PCC 6803 m금성 | 0.1 | 127,864 | 1.28 엑스 107 | 8,186 | 1.36 엑스 107 | 428 | 1.07 엑스 107 | |
표 3: 다양한 정량화 방법 비교: 단일/축성 배양을 사용한 보정.
토론
실험실 맥락에서 단일 축잔 배양에서 미생물을 처리하는 것은 많은 미생물 모델에 대해 수십 년 동안 확립되어 왔습니다. 그러나 자연에서 지배적인 형태의 생명체는 미생물 군집이지만, 단일 재배 용기에서 둘 이상의 파트너를 결합하는 것은 확립되지 않았으며 기존 지식과 방법론의 격차로 인해 문제가 제시됩니다. 또한 세포 간에 창발적 상호 작용과 대사 산물 교환이 발생하여 공동 배양의 운명에 큰 영향을 미치기 때문에 군집에서 세포의 행동을 예측하는 것이 더 어렵습니다34,35. 따라서 공동 배양 확립은 성장 배지 정의 수준, 공통 성장 조건의 식별, 미량 대사 산물/신호의 종간 교환, 시간 경과에 따른 공동 배양 구성을 포함하여 사소한 일이 아닙니다. 종속영양 파트너와 함께 광영양성, 설탕을 분비하는 시아노박테리아의 조립에서 지난 몇 년 동안의 진전은 이제 새로운 공동 배양 쌍의 설계에 유용한 지침을 제공할 수 있는 첫 번째 규칙과 방법론을 추론할 수 있게 되었습니다. 이러한 지식을 바탕으로 이 프로토콜의 첫 번째 부분에서는 자당-분비물, 시아노박테리움 및 하나 이상의 효모를 포함하는 공동 배양 조립에 대한 단계별 지침이 제공됩니다.
관련 없는 미생물 간의 공동 배양을 처음 시도할 때 한 가지 중요한 고려 사항은 둘 이상의 종에 대한 모든 영양 요구 사항을 충족하는 공통 성장 배지의 구성입니다. 공간 제한으로 인해 여기에서 이 프로세스에 대한 완전히 자세한 프로토콜을 제공하는 것은 불가능하며, 경우에 따라 높은 수준의 사용자 지정이 필요할 수도 있지만 대신 다음에서는 염두에 두어야 할 중요한 고려 사항을 간략하게 설명합니다. 한 가지 간단한 초기 접근법은 일반적인 시아노박테리아 성장 배지(예: BG11, 표 1 참조)를 관심 종속영양 종을 위해 구성된 확립된 최소 배지와 비교하는 것입니다. 종속영양 최소 배지 내에 포함된 누락된 성분으로 표준 시아노박테리아 배지를 보충하는 것은 초기 테스트를 위한 좋은 출발점입니다 8,36. 최소 배지에는 종속영양 성장을 위한 중요한 유기 탄소원(예: 1%-4% 포도당)이 보충되는 경우가 많기 때문에 초기 공동 배양 실험에서 달성할 수 있는 것보다 더 높은 종속영양 세포 밀도를 지원하도록 설계되는 경우가 많습니다. 마찬가지로, 일부 일반적인 배지 성분은 시아노박테리아 파트너(예: 시트르산염)가 제공하는 광합성물과는 독립적으로 유기 탄소원으로 작용할 수 있으며, 이는 시아노박테리아/종속영양 공동 배양의 추후 분석을 복잡하게 만들 수 있습니다. 그러므로, 공동 배양 배지를 설계할 때 누락된 원소의 전체 농도로 최소 시아노박테리아 배지를 보완할 필요가 없을 수도 있습니다. 예를 들어, 많은 유기체는 다양한 형태의 환경 질소(예: N2, 질산염, 아질산염, 요소)의 사용 효율성이 다양하며 더 산화된 질소 공급원 중 일부를 완전히 활용하지 못할 수 있습니다. 마찬가지로, 많은 미생물은 필수 비타민(예: 비타민 B12), 보조 인자 또는 필수 아미노산의 보충이 필요할 수 있는데, 이는 이러한 화합물의 직접 합성을 위한 완전한 생합성 경로가 없기 때문입니다. 이러한 이유로, 시아노박테리아 파트너의 확립된 최소 배지(예: BG11)를 종속영양 물질의 잘 정의된 최소 배지(예: 합성 정의[SD])와 함께 단순히 "병합"하는 것으로 시작하는 것이 가장 실용적일 수 있습니다. 불필요한 성분을 반복적으로 제거/감소시키는 후기 사이클을 사용하여 매체를 최적화하고 파트너 중 하나의 성장을 억제할 수 있는 모든 화합물의 풍부함을 감소시킬 수 있습니다. 유용한 시작점은 중성 또는 약간 염기성 pH에서 배지를 완충하는 것인데, 이는 대부분의 시아노박테리아 모델 종에서 선호하는 조건인 경향이 있기 때문입니다.
이 시점에서, 과량의 자당이 공급될 때 새로운 공동 배양 배지에서 지지된 종속영양 생물의 성장에 대한 예비 테스트를 수행하는 것이 종종 도움이 됩니다. 물론, 잠재적인 종속영양 생물을 선택할 때는 시아노박테리아 파트너가 공급할 유기 탄소의 주요 공급원을 이화할 수 있는 것을 선택하는 것이 중요합니다. 많은 조작된 시아노박테리아/종속영양 배양물에서 공급되는 지배적인탄수화물인 자당(sucrose)4은 포도당(glucose)처럼 종속영양 미생물에 의해 보편적으로 사용되는 탄수화물이 아니라는 점에 유의하는 것이 유용하다: 전용 자당 수송체는 종속영양 종에 의해 암호화되어야 하며, 그렇지 않으면 자당을 친화성이 높은 수송체에 의해 종종 인식되는 과당 및 포도당으로 전환하기 위해 세포 외 인버타제(extracellular invertases)가 필요할 수 있다8. 혼합 미생물 군집을 연구하는 여러 실험실에서 일반적으로 보고된 중요한 관찰은 광영양 및 종속영양 파트너 사이에 고차 시너지 효과와 길항작용이 나타난다는 것입니다 4,8,13. 예를 들어, 다른 자연 분비 대사 산물(예: 유기산, 감소된 형태의 질소) 또는 보조 인자(예: 시데로포어)는 축산 대조군에 비해 동일한 배지에서 배양할 때 하나 또는 두 파트너의 더 높은 성장률을 가능하게 할 수 있습니다. 반대로, 광합성 물 분해에 의한 배지의 과산소화와 같은 잠재적으로 유해한 대사 부산물은 한쪽 또는 양쪽 파트너의 종간 성장 억제를 유발하는 것으로 보고되었습니다8. 따라서 axenic control은 유용한 벤치마크를 제공할 수 있지만 이러한 창발적 속성으로 인해 공동 배양 성능이 예상과 다를 수 있습니다.
종속영양 파트너와 S. elongatus의 자당을 배설하는 능력(유도 수준, 사용된 IPTG 농도에 따라)에 따라 두 유기체의 다른 비율을 테스트해야 합니다. 배양의 성공 여부는 주로 종속영양 파트너의 성장을 유지하는 S. elongatus의 능력(즉, 충분한 탄소원을 생산할 수 있는지)에 달려 있습니다. 종속영양 균주의 과다 증식은 일반적으로 공동 배양 배지 조성에 제공된 유기 탄소의 부족으로 인해 제한되지만, 시아노박테리아는 종속영양 균주를 앞지를 수 있으며, 이는 새로운 억제 상호 작용(예: 고산소 상태 8,13)으로 이어질 수 있습니다. 시아노박테리아 효모에 대한 세포 밀도의 적절한 비율을 초기에 결정하려고 할 때, 좋은 근사치 규칙은 시아노박테리아 파트너가 동반되는 종속영양 세포의 동일한 세포 부피를 지원할 수 있다는 것입니다. 진핵 효모는 모델 자당을 분비하는 시아노박테리아보다 상당히 큰 세포 부피를 갖는 경향이 있기 때문에 이는 시아노박테리아 세포의 밀도가 정상 상태에서 상당히 높을 것(예: S. cerevisiae의 경우 50-100배)을 의미할 수 있습니다. 따라서 연구자별 실험실 및 종 조건과 함께 새로운 공동 배양을 설정할 때 좋은 출발점은 시아노박테리아와 효모 모두의 평균 세포 부피를 계산하고(발표된 값을 기반으로 함, 예: B10NUMB3R537 참조) 부피 비율을 추정하는 것입니다. 초기 플라스크는 이 세포의 비율로 파종할 수 있습니다. 용액 공간을 탐색하기 위해 연구자는 시아노박테리아의 농도를 일정하게 유지하고(예: OD750 = 0.3) 효모 세포의 농도를 연구원의 사용 가능한 광영양 배양 공간에서 허용하는 처리량에 따라 대략 한 자릿수씩 증가 또는 감소시키기를 원할 수 있습니다. 물론, 이 부피 측정 '경험 법칙'은 종속영양 파트너가 사용할 수 있는 유기 광합성물(예: 자당)을 분비할 수 있는 속도에 따라 달라집니다. 일단 공동 배양 조건이 확립되면, 시간 경과에 따른 두 파트너의 성장 성과를 주의 깊게 모니터링하면 이상적인 종 비율에 대한 귀중한 데이터를 얻을 수 있으며, 특히 초기 공동 배양이 며칠에서 몇 주 동안 유지될 수 있는 경우 연구자가 공동 배양이 끝날 무렵에 도달한 정상 상태 비율을 식별할 수 있습니다. 이 정보는 후속 실험에서 각 파트너 종에 대한 초기 접종 밀도를 선택할 때 활용하여 배양이 자체 결정된 이상적인 종 비율에 보다 빠르게 도달하는 데 도움이 될 수 있습니다.
프로토콜의 두 번째 부분에서는 공동 배양 분석에 대한 자세한 지침이 제공됩니다. 공동 배양의 신뢰할 수 있는 정량화는 성공적인 구현의 핵심입니다: 광방성, 탄소 분비 파트너의 성장(또는 적어도 대사 활동)은 종속영양 생물을 유지하는 데 필수적입니다. 배양액을 접종하는 데 사용되는 시아노박테리아:종속영양체의 비율이 항상 최적화에 중요한 것은 아니지만, 장기적인 공동 배양은 안정적인 비율로 수렴하는 경향이 있기 때문에 하나 이상의 종의 배양 희석 또는 캡슐화를 통해 파트너의 억제되지 않은 성장을 확인하는 방법을 통합하는 것이 중요할 수 있습니다 36,38,39,40. 위에서 언급한 바와 같이, 시아노박테리아 파트너의 부산물은 고농도(예:O2)에서 종속영양을 억제할 수 있으며, 종속영양 대사의 일부 산물도 시아노박테리아 건강에 해로울 수 있습니다. 대부분의 발표된 공동 배양은 대부분의 모델 시아노박테리아 종보다 성장 속도가 빠른 종속영양 생물을 사용합니다. 그러므로, 종속영양 성장률은 시아노박테리아에 의해 생산된 유기탄소의 공급에 의해 제한되는 경향이 있다. 그럼에도 불구하고, 시간이 지남에 따라 종 풍부도 역학을 결정하는 것은 안정성의 실패를 설명하고 장기 재배에서 강력한 공동 배양을 최적화하는 데 중요한 가치입니다.
카운팅 챔버(counting chambers), 입자 계수기(particle counters) 및 단일 세포 유세포 분석(single-cell flow cytometry)을 사용한 정량화를 위한 프로토콜이 제공됩니다. 그러나 모든 기술은 공동 배양의 특성화를 위한 유용한 도구이지만 전제 조건, 장점 및 제한 사항이 다릅니다. 계수실(counting chamber)은 공동 배양 정량화에 매우 광범위하게 적용할 수 있으며, 배양의 모든 파트너를 세포 모양 또는 기타 특성으로 시각적으로 구별할 수 있는 경우 가능합니다. 가장 큰 장점은 이 장치의 가격이 저렴하여 기본적으로 모든 실험실에서 달성할 수 있다는 것입니다. 서로 다른 파트너의 공동 배양 구성 및 비율에 대한 정보 외에도 세포의 형태 및 적합성에 대한 인상을 얻을 수 있으며 잠재적인 오염 물질을 검출할 수 있습니다. 그러나 계수 챔버의 적용은 또한 시간이 많이 걸리고 작업량이 많으며 낮은 처리량만 지원할 수 있습니다.
입자 계수기와 single-cell 유세포 분석은 모두 높은 처리량과 편리하고 시간을 절약할 수 있는 취급이라는 큰 이점을 제공합니다. cell counter는 co-culture에서 파트너의 세포 크기의 명확한 차이에 의존하는 반면, single-cell cytometry는 형광 표지를 구별할 수도 있으므로 서로 다른 형광 마커를 기반으로 동일한 크기의 두 개 이상의 구성원 또는 동일한 유기체의 두 개의 다른 돌연변이가 있는 co-culture를 정량화할 수 있습니다. S. cerevisiae와 U. maydis를 가진 Synechocystis의 인공 조립체에 대한 제공된 예에서, 세포 내 형광 리포터 mKate2 및 eGFP를 사용하여 두 효모를 구별했습니다. 동일한 형광 리포터를 사용하여 동일한 유기체의 두 유전자 변형 균주(예: S. cerevisiae)를 구별할 수 있으며, 이는 광 산란 특성만으로는 분리할 수 없습니다. 합성 박테리아 컨소시엄(synthetic bacterial consortia)을 추적하기 위해 유사한 전략이 시행되었다41. 세포 유형, 컨소시엄의 파트너 수 및 잠재적인 자가형광 범위에 따라 형광 마커 선택은 특히 시아노박테리아의 자가형광 특성을 고려할 때 스펙트럼 품질의 중복을 피하기 위해 각별한 주의가 필요합니다. 42의
우리는 여기에서 과학 데이터의 저장, 관리 및 공유 측면에서 FAIR 지침 원칙을 준수하는 방법을 옹호합니다. FAIR는 Findable, Accessible, Interoperable, Reusable43의 약자입니다. 이러한 원칙이 널리 수용됨에 따라 생물학 연구의 환경을 변화시키고 데이터 관리 및 사용에 대한 보다 개방적이고 협력적이며 효율적인 접근 방식을 촉진할 것을 약속합니다. 접근성의 중요한 구성 요소는 ARC(Annotated Research Context)44에 데이터를 저장하여 보장할 수 있지만, 오픈 액세스 소프트웨어로 데이터 처리 단계를 재현할 수 있도록 하는 것이 중요합니다. Raw FCS 파일은 유세포 분석 실험45에 대한 데이터와 메타데이터(파장, 필터 등을 포함한 레이저 및 검출기에 대한 정보)를 모두 저장합니다. 정교한 데이터 분석을 허용하는 독점 소프트웨어 외부에서 로드하고 읽을 수 있습니다(예: Python 환경 내에서 원시 데이터로 작업할 수 있는 여러 패키지가 있음). 개발된 패키지는 Python23에서 사용 가능한 많은 패키지 중 하나를 사용하여 독점 소프트웨어 외부에서 세포 분석 데이터가 포함된 원시 .fcs 파일을 로드, 읽기 및 시각화하는 방법을 보여줍니다. 위에서 설명한 것과 같은 개방형 소프트웨어를 사용하면 독점 소프트웨어와 관련된 값비싼 라이선스 비용의 필요성을 없애고, 데이터 처리(예: 변환, 보상, 게이팅)를 완전히 제어할 수 있으며, 추가적으로 여러 도구를 한 곳에 통합할 수 있습니다.
세포 분류기와 함께 단일 세포 세포 분석을 활용하는 것의 중요한 추가 이점은 FACS에 의해 고유한 유형의 세포를 수집할 수 있는 기회입니다. 이는 RNASeq 또는 대사체학(metabolomics)과 같은 공동 배양 내 종 상호 작용에 대한 자세한 통찰력을 얻기 위해 모든 오믹스 기술을 적용하는 데 매우 유용할 수 있습니다. 이러한 장치의 분명한 단점은 가격이 높다는 것이므로 이러한 기계의 경제성과 가용성은 일부 실험실에서 명백한 제한 사항이 될 수 있습니다.
흥미롭게도, 컨소시엄에서 세 파트너의 정량화를 위해 제시된 방법을 직접 비교한 결과, 서로 다른 기술 간에 매우 우수한 일치가 나타났으며, 이는 OD 측정을 기반으로 조립된 혼합물이 설명된 모든 방법에 의해 안정적으로 정량화되었음을 나타냅니다. 흥미롭게도, 시아노박테리아의 총 수는 두 효모 종의 수보다 약 3-4배 더 많았는데, 이는 세포 크기가 더 작기 때문일 수 있는 현상입니다. 이러한 관찰은 OD가 보정 없이 실제 세포 수에 대한 정보를 제공하지 않는다는 일반적인 지식과 잘 일치합니다46. 그러나 효모는 비슷한 수준의 수치를 보였다(표 2 및 표 3). 또한, 단일 배양의 정량화와 비교하면 두 효모 종을 분리하지 않은 입자 계수기를 제외하고는 세 가지 방법 모두 세포 유형을 구별할 수 있는 신뢰할 수 있는 도구임을 보여줍니다.
본질적으로 단일 세포 분석 응용 프로그램은 공동 배양의 구성을 모니터링하는 가장 강력한 도구로, 초당 1,000-10,000개의 세포를 계수할 수 있습니다. 파트너의 수가 두 개 이상으로 증가하거나 파트너의 모양 및/또는 직경이 유사한 경우 특히 그렇습니다. 특히 공동 배양을 모니터링할 수 있는 대안이 많이 있습니다. 예를 들어, 공동 배양에서 형광 표지된 미생물의 성장은 형광 측정 또는 미생물 반응기에 의해 지속적으로 추적될 수 있습니다47. 그러나 이러한 작업은 종종 두 파트너의 공동 배양으로 제한되며 예를 들어 형광 마커를 선택할 때 신중한 실험 설계가 필요합니다. 앰플리콘 염기서열분석(16S rRNA 염기서열분석) 및 정교한 생물정보학(bioinformatics)과 결합된 기타 차세대 염기서열분석 기술은 합성 군집의 특성화를 위한 또 다른 옵션입니다 48,49,50. 이러한 기술은 고처리량 접근 방식에 적합하며 장기 재배, 진화적 질문 또는 여러 미생물 파트너와의 돌연변이 추적에서 확립된 상호 작용을 해결할 수 있습니다.
종합하면, 합리적으로 설계된 단순한 미생물 공동 배양은 자연계를 지배하는 다종 군집 내에서 접근하기가 더 어려울 수 있는 종 간 역학을 조사하기 위한 강력한 "상향식" 접근 방식을 제공합니다 51,52,53. 여기에서는 새로운 시아노박테리아 쌍과 종속영양 파트너의 확립 및 분석을 위해 과학계에서 쉽게 적용할 수 있는 간소화된 프로토콜이 제공됩니다. 인공 미생물 공동 배양에서 얻을 수 있는 잠재적인 근본적인 통찰력을 활용하고 합성으로 설계된 미생물 컨소시엄이 생명 공학 응용 분야에 대한 문헌에서 종종 언급되는 잠재력과 일치할 수 있는지 여부를 결정하기 위해 많은 연구가 필요하다는 것은 분명합니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
이 연구는 Deutsche Forschungsgemeinschaft (DFG, German Research Foundation) - SFB1535 - Project ID 458090666 (프로젝트 B03에서 KS, AM 및 IA까지) 및 주요 연구 계측 장비 INST 208/808-1의 지원을 받았습니다. DCD는 현재 SFB1535의 메르카토르 펠로우로 HHU 뒤셀도르프를 방문하고 있습니다. EJK에 대한 추가 지원은 National Science Foundation Awards #1845463 및 #2334680에서 제공했습니다.
자료
| Name | Company | Catalog Number | Comments |
| 10x Ph1 objective | Carl Zeiss AG | 46 04 01-9904 | |
| 525/40 nm Bandpass Filter | Beckman Coulter, Inc. | A01-1-0051 | FITC filter (in 488 nm laser) |
| 660/10 nm Bandpass Filter | Beckman Coulter, Inc. | A01-1-0055 | APC channel (in 638 nm laser) |
| 710/50 nm Bandpass Filter | Beckman Coulter, Inc. | B71092 | PC5.5 channel (in 561 nm laser) |
| Accuvettes for Multisizer 4e | Beckman Coulter, Inc. | A35473 | Sample cups with lids |
| Aperture tube 30 μM for Multisizer 4e | Beckman Coulter, Inc. | C92760 | Capillary |
| Autoclave | Lab Associates | EQM-ARBP | for sterilisation of material |
| Baffled flasks ROTILABO®, Straight neck, 250 ml | Carl Roth GmbH + Co. KG | LY95.1 | |
| BG11 / CoYBG11 components | different suppliers | see Table 1 | |
| Bottle top sterile filter | Sigmaaldrich | Z222593 | to sterile media if you dont want to use autoclave |
| Capillary 45 µm | Omni Life Science | OLS 5651738 | |
| CASY Cell Counter Model TTC | Schaerfe Systems | ||
| CASY cups | Omni Life Science | OLS 5651794 | |
| CASYclean | Omni Life Science | OLS 5651786 | Cleaning solution |
| CASYton solution | Omni Life Science | OLS 5651808 | Isotonic solution |
| Centrifuge tubes (50 mL) | Sigmaaldrich | CLS430828 | |
| CO2 | BOC | 270182-L | to supply cyanobacteria axenic cultures and co-cultures |
| CONTRAD® 70 | Beckman Coulter, Inc. | 81911 | Deep clean solution |
| Cover glass 20 × 26 x 0.4 mm | VWR International GmbH | 631-1190 | Cover glass for counting chamber |
| Cuvettes | Sarstedt | 67.742 | |
| Cyanobacteria BG-11 Freshwater Solution | Merck KGaA | C3061 | |
| CytExpert Software v. 2.6 | Beckman Coulter, Inc. | Cytometer application software | |
| CytoFLEX Sheath Fluid | Beckman Coulter, Inc. | B51503 | Sheat fluid |
| Cytometer CytoFLEX S | Beckman Coulter, Inc. | BE51180 | |
| DxH Cleaner for Multisizer 4e | Beckman Coulter, Inc. | 628022 | Cleaning solution |
| Eppendorf tube centrifuge (small) | Labstac | CEN16-15 | to centrifuge less than 2mL of culture |
| Ethanol | Carl Roth GmbH + Co. KG | K928.1 | |
| FACS tubes | Beckman Coulter, Inc. | 2523749 | tubes for semi-automatic mode |
| FlowClean Cleaning Agent, 500ml | Beckman Coulter, Inc. | C48093 | Daily clean solution |
| Graduated cylinders | Sigmaaldrich | Z131121 | for preparation of media |
| HEPPSO | Merck KGaA | R426725 | |
| Infors MultiTron photoincubator | Infors AG | white LED light | |
| IPTG | Merck KGaA | I6758 | Prepare aliquots, store at -20°C, thaw and keep on ice |
| Isoton II for Multisizer 4e | Beckman Coulter, Inc. | 8448011 | Isotonic solution |
| Kaluza v. 2.2 | Beckman Coulter, Inc. | B16406 | Cytometry data analysis software |
| KimWipes | VWR International GmbH | 115-2221 | Lint-free tissues |
| KOH | Merck KGaA | 221473 | |
| KPO3 | Noah Chemicals | 7790-53-6 | |
| Measuring beakers | VWR | 213-3747 | |
| Micro test plate, 96 well, slip-on lid, flat base, PS, transparent | SARSTEDT AG & Co. KG | 82.1581.001 | |
| Microscope base | Carl Zeiss AG | 47 09 18-9902/16 | |
| Microsope head | Carl Zeiss AG | 47 30 14 | |
| Milli-Q water purification system | Merck Millipore | C85358 | |
| Mulitisizer 4e particle counter | Beckman Coulter, Inc. | B23005 | Alternative to the CASY Cell Counter |
| Neubauer improved, depth 0.02 mm | Assistent Germany | Counting chamber | |
| Neubauer improved, depth 0.1 mm | Paul Marienfeld GmbH & Co. KG | 640010 | Counting chamber |
| Ready to Use Daily QC Fluorospheres | Beckman Coulter, Inc. | C65719 | Reference for QC |
| SCHOTT bottles | Dutscher | 90347 | for storage |
| Specord®200 Plus Spectrophotometer | Analytic Jena GmbH | OD600/750 | |
| Sucrose | Merck KGaA | 84100 | |
| Table top centrifuge (big) | Labstac | CEN18-06R | collect culture biomass |
| Yeast Nitrogen Base, without amino acids, without ammonium sulfate | Thermo Fisher Scientific Inc. | 11743014 |
참고문헌
- Roell, G. W., Zha, J., Carr, R. R., Koffas, M. A., Fong, S. S., Tang, Y. J. Engineering microbial consortia by division of labor. Microb Cell Factories. 18 (1), 35 (2019).
- Grandel, N. E., Reyes Gamas, K., Bennett, M. R. Control of synthetic microbial consortia in time, space, and composition. Trends Microbiol. 29 (12), 1095-1105 (2021).
- Weiland-Bräuer, N. Friends or Foes-Microbial Interactions in Nature. Biology. 10 (6), (2021).
- Santos-Merino, M., Yun, L., Ducat, D. C. Cyanobacteria as cell factories for the photosynthetic production of sucrose. Front Microbiol. 14, 1126032 (2023).
- Klähn, S., Hagemann, M. Compatible solute biosynthesis in cyanobacteria. Environ Microbiol. 13 (3), 551-562 (2011).
- Dean, P. N., Bagwell, C. B., Lindmo, T., Murphy, R. F., Salzman, G. C. Introduction to flow cytometry data file standard. Cytometry. 11 (3), 321-322 (1990).
- Rippka, R., Stanier, R. Y., Deruelles, J., Herdman, M., Waterbury, J. B. Generic assignments, strain histories and properties of pure cultures of Cyanobacteria. Microbiology. 111 (1), 1-61 (1979).
- Hays, S. G., Yan, L. L. W., Silver, P. A., Ducat, D. C. Synthetic photosynthetic consortia define interactions leading to robustness and photoproduction. J Biol Eng. 11, 4 (2017).
- Ducat, D. C., Avelar-Rivas, J. A., Way, J. C., Silver, P. A. Rerouting carbon flux to enhance photosynthetic productivity. Appl Environ Microbiol. 78 (8), 2660-2668 (2012).
- Thiel, K., et al. Redirecting photosynthetic electron flux in the cyanobacterium Synechocystis sp. PCC 6803 by the deletion of flavodiiron protein Flv3. Microb Cell Factories. 18 (1), 189 (2019).
- Song, K., Tan, X., Liang, Y., Lu, X. The potential of Synechococcus elongatus UTEX 2973 for sugar feedstock production. Appl Microbiol Biotechnol. 100 (18), 7865-7875 (2016).
- Feng, J., et al. Generation and comprehensive analysis of Synechococcus elongatus-Aspergillus nidulans co-culture system for polyketide production. Biotechnol Biofuels Bioprod. 16 (1), 32 (2023).
- Li, T., et al. Mimicking lichens: incorporation of yeast strains together with sucrose-secreting cyanobacteria improves survival, growth, ROS removal, and lipid production in a stable mutualistic co-culture production platform. Biotechnol Biofuels. 10, 55 (2017).
- JoVE Science Education Database. Microbiology. Growth Curves: Generating Growth Curves Using Colony Forming Units and Optical Density Measurements. , (2023).
- . . Yeast Extract-Peptone-Dextrose (YEPD). (9), (2015).
- Zhang, M., et al. Improvement of cell counting method for Neubauer counting chamber. J Clin Lab Anal. 34 (1), e23024 (2020).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies. Eu J Immunol. 47 (10), 1584-1797 (2017).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eu J Immunol. 51 (12), 2708-3145 (2021).
- Shapiro, H. M. . Practical Flow Cytometry. , (2003).
- Tung, J. W., et al. Modern flow cytometry: a practical approach. Clin Lab Med. 27 (3), 453-468 (2007).
- JoVE Science Education Database. . Flow Cytometry. , (2023).
- Cheung, M., Campbell, J. J., Whitby, L., Thomas, R. J., Braybrook, J., Petzing, J. Current trends in flow cytometry automated data analysis software. Cytometry A. 99 (10), 1007-1021 (2021).
- . FlowCytometryTools: Version 0.4.5 Available from: https://pypi.org/project/FlowCytometryTools/ (2021)
- White, S., et al. FlowKit: A Python Toolkit for Integrated Manual and Automated Cytometry Analysis Workflows. Front Immunol. 12, 768541 (2021).
- O'Neill, K., Aghaeepour, N., Spidlen, J., Brinkman, R. Flow cytometry bioinformatics. PLoS Comput Biol. 9 (12), e1003365 (2013).
- Klement, T., Milker, S., Jäger, G., Grande, P. M., Domínguez de María, P., Büchs, P. Biomass pretreatment affects Ustilago maydis in producing itaconic acid. Microb Cell Factories. 11, 43 (2012).
- Koschwanez, J. H., Foster, K. R., Murray, A. W. Sucrose utilization in budding yeast as a model for the origin of undifferentiated multicellularity. PLoS Biol. 9 (8), e1001122 (2011).
- Koschwanez, J. H., Foster, K. R., Murray, A. W. Improved use of a public good selects for the evolution of undifferentiated multicellularity. ELife. 2, e00367 (2013).
- Winston, F., Dollard, C., Ricupero-Hovasse, S. L. Construction of a set of convenient Saccharomyces cerevisiae strains that are isogenic to S288C. Yeast. 11 (1), 53-55 (1995).
- Shcherbo, D., et al. Far-red fluorescent tags for protein imaging in living tissues. Biochem J. 418 (3), 567-574 (2009).
- Brachmann, A., Weinzierl, G., Kämper, J., Kahmann, R. Identification of genes in the bW/bE regulatory cascade in Ustilago maydis. Mol Microbiol. 42 (4), 1047-1063 (2001).
- Cormack, B. P., Valdivia, R. H., Falkow, S. FACS-optimized mutants of the green fluorescent protein (GFP). Gene. 173 (1 Spec), 33-38 (1996).
- Behle, A., Saake, P., Germann, A. T., Dienst, D., Axmann, I. M. Comparative dose-response analysis of inducible promoters in cyanobacteria. ACS Synth Biol. 9 (4), 843-855 (2020).
- Lindemann, S. R., et al. Engineering microbial consortia for controllable outputs. ISME J. 10 (9), 2077-2084 (2016).
- Duncker, K. E., Holmes, Z. A., You, L. Engineered microbial consortia: strategies and applications. Microb Cell Factories. 20 (1), 211 (2021).
- Fedeson, D. T., Saake, P., Calero, P., Nikel, P. I., Ducat, D. C. Biotransformation of 2,4-dinitrotoluene in a phototrophic co-culture of engineered Synechococcus elongatus and Pseudomonas putida. Microb Biotechnol. 13 (4), 997-1011 (2020).
- Milo, R., Jorgensen, P., Moran, U., Weber, G., Springer, M. BioNumbers-the database of key numbers in molecular and cell biology. Nucleic Acids Res. 38 (suppl_1), D750-D753 (2009).
- Tóth, G. S., et al. Photosynthetically produced sucrose by immobilized Synechocystis sp. PCC 6803 drives biotransformation in E. coli. Biotechnol Biofuels Bioprod. 15 (1), 146 (2022).
- Weiss, T. L., Young, E. J., Ducat, D. C. A synthetic, light-driven consortium of cyanobacteria and heterotrophic bacteria enables stable polyhydroxybutyrate production. Metabol Eng. 44, 236-245 (2017).
- Zhang, L., Chen, L., Diao, J., Song, X., Shi, M., Zhang, W. Construction and analysis of an artificial consortium based on the fast-growing cyanobacterium Synechococcus elongatus UTEX 2973 to produce the platform chemical 3-hydroxypropionic acid from CO2. Biotechnol Biofuels. 13, 82 (2020).
- Jorrin, B., et al. Stable, fluorescent markers for tracking synthetic communities and assembly dynamics. Microbiome. 12 (1), 81 (2024).
- Yokoo, R., et al. Live-cell imaging of cyanobacteria. Photosynth Res. 126, 36-46 (2015).
- Wilkinson, M. D., et al. The FAIR guiding principles for scientific data management and stewardship. Sci Data. 3, 160018 (2016).
- Venn, B., et al. Fostering the democratization of research data by using the Annotated Research Context (ARC) as practical implementation. E-Science-Tage 2021: Share Your Research Data. , (2021).
- Ortolani, C. The Cytometric File. Flow Cytometry Today: Everything You Need to Know about Flow Cytometry. , (2022).
- Beal, J., et al. Robust estimation of bacterial cell count from optical density. Communi Biol. 3 (1), 512 (2020).
- Funke, M., et al. Microfluidic biolector-microfluidic bioprocess control in microtiter plates. Biotechnol Bioeng. 107 (3), 497-505 (2010).
- Zhang, P., Spaepen, S., Bai, Y., Hacquard, S., Garrido-Oter, R. Rbec: a tool for analysis of amplicon sequencing data from synthetic microbial communities. ISME Commun. 1 (1), 73 (2021).
- Ezzamouri, B., Shoaie, S., Ledesma-Amaro, R. Synergies of systems biology and synthetic biology in human microbiome studies. Front Microbiol. 12, 681982 (2021).
- Zaramela, L. S., Tjuanta, M., Moyne, O., Neal, M., Zengler, K. synDNA-a synthetic DNA spike-in method for absolute quantification of shotgun metagenomic sequencing. mSystems. 7 (6), e0044722 (2022).
- Bittihn, P., Din, M. O., Tsimring, L. S., Hasty, J. Rational engineering of synthetic microbial systems: from single cells to consortia. Curr Opin Microbiol. 45, 92-99 (2018).
- San León, D., Nogales, J. Toward merging bottom-up and top-down model-based designing of synthetic microbial communities. Curr Opin Microbiol. 69, 102169 (2022).
- Fedeson, D. T., Ducat, D. C. . Symbiotic Interactions of Phototrophic Microbes: Engineering Synthetic Consortia for Biotechnology. Role of Microbial Communities for Sustainability. , (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유