É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Microscopia óptica de dispersão com base em filtros Gabor Two-Dimensional
Neste Artigo
Resumo
Demonstramos um método de microscopia de campo escuro com base em Gabor-como filtragem para medir a dinâmica subcelulares dentro de células vivas único. A técnica é sensível a alterações na estrutura das organelas, tais como a fragmentação mitocondrial.
Resumo
Demonstramos um instrumento microscópicas que podem medir textura subcelulares decorrentes da organela morfologia e organização dentro de células vivas sem mácula. O instrumento proposto se estende a sensibilidade da microscopia óptica rótulo sem a mudanças em nanoescala em organela tamanho e forma e podem ser usados para acelerar o estudo da relação estrutura-função pertencentes a organela dinâmica subjacente processos biológicos fundamentais, como a morte celular programada ou celular diferenciação. O microscópio pode ser facilmente implementado em plataformas de microscopia existentes, e pode, portanto, ser divulgada a laboratórios específicos, onde os cientistas podem implementar e utilizar os métodos propostos com acesso irrestrito.
A técnica proposta é capaz de caracterizar a estrutura subcelular, observando a célula através de duas dimensões filtros ópticos Gabor. Esses filtros podem ser ajustados para sentir com a sensibilidade (10 de nm) em nanoescala, atributos específicos morfológicas relativas ao tamanho e orientação de não-esférica organelas subcelulares. Embora baseado em contraste gerado por espalhamento elástico, a técnica não se baseia em um modelo de espalhamento inverso ou detalhada sobre a teoria Mie para extrair medidas morfométricas. Esta técnica é, portanto, aplicável a não-esférica organelas para a qual uma descrição precisa dispersão teórica não é facilmente determinada, e fornece parâmetros morfométricos distintivo que pode ser obtido dentro de células vivas sem mácula para avaliar a sua função. A técnica é vantajosa em comparação com processamento digital de imagens em que ele atua diretamente no campo do objeto, em vez de transformar a intensidade do objeto é discretizada. Ele não depende de altas taxas de amostragem de imagem e, portanto, pode ser usada para rapidamente tela de atividades morfológicas dentro de centenas de células de cada vez, portanto, facilitando bastante o estudo da estrutura organela organela além da segmentação individual e reconstrução por microscopia de fluorescência confocal de imagens digitais altamente ampliada de campos de vista limitado.
Nesta demonstração vamos mostrar dados de uma diatomácea marinha para ilustrar a metodologia. Nós também mostram os dados preliminares coletados em células vivas para dar uma idéia de como o método pode ser aplicado em um contexto biológicas relevantes.
Protocolo
1. Ficando as células prontas
- As células que foram semeadas no dia anterior necessita ser etiquetado com o verde Mitotracker para imagens de fluorescência das mitocôndrias.
- Remover 100 mM solução estoque de verde Mitotracker em DMSO feitas anteriormente a partir dos 4 ° C freezer, e à temperatura ambiente com a mão. Também retirar células endoteliais aórticas bovinas (BAEC) meio de cultura celular também previamente preparado e quente a 37 ° C no banho de água de bancada.
- Uma vez que o Mitotracker e meio de cultura são aquecidos, colocá-las dentro do capuz certificando-se de esterilizar as mãos com luvas e todas as superfícies externas dos recipientes com solução de etanol 70%. Não acenda a luz capô, como o rótulo fluorescente é sensível à luz e rapidamente photobleach na luz ambiente.
- Tornando a concentração correta para a rotulagem mitocondrial é muito importante. Muito pouco não rotular o efetivamente mitocôndrias, enquanto Mitotracker demais pode ter efeitos tóxicos. A concentração de 100 nM de Mitotracker incubadas por 45 minutos com as células funciona bem. Prepare essa concentração pela adição de 100 L de estoque Mitotracker a 10 mL meio de cultura em um tubo de 15 mL. Isso fará com que a abundância por pelo menos um experimento.
- Substituir o médio já existentes com o meio rotulado por sugando o meio de idade, com uma pipeta Pasteur conectado à linha de vácuo. Logo em seguida adicionar 2 mL do meio rotulados para cada cultura ocupou bem na placa de 6 bem.
- Porque a etiqueta fluorescente é sensível à luz, substituir as células na incubadora rapidamente, sem expor à luz ambiente direta. Cobrindo a placa de 6 bem com as mãos funciona bem para isso. As células vão ficar na incubadora por 45 minutos.
2. Obtendo a configuração óptica pronto
- Enquanto as células esperar na incubadora, temos que ligar a configuração óptica. Na sala de óptica, primeiramente ligue a lâmpada de vapor de mercúrio, seguido pelo computador, o microscópio, as câmeras, eo laser. Em seguida, conecte o dispositivo de microespelhos digitais (DMD) e do difusor spinning.
- Certifique-se que o lançamento está alinhado óptica, olhando através da ocular do microscópio para assegurar que o campo de visão é iluminado pela luz laser.
- Limpe o objetivo de dobrar um pedaço de papel com lentes em uma praça apertados e segure firmemente com uma pinça hemostática. Mergulhe o papel de lente em amônia livre de solução de limpeza de vidro para absorver uma pequena quantidade no papel. Rap do hemostat em sua mão livre várias vezes para remover qualquer excesso. Limpe o objetivo de aplicar um golpe firme, limpa e contínua através da objectiva de uma ponta à outra, passando por cima da lente no meio. Não re-furto ou matagal. Descarte de papel usado.
- Para carregar a amostra, colocar a retícula sobre o objetivo 63x óleo de imersão, largando 02/01 pequenas gotas de óleo de imersão sobre o objetivo, enquanto o objetivo é todo o caminho. Em seguida, coloque a retícula no palco. Em seguida, levantar o objetivo para que o óleo "agarra" a amostra. O foco da amostra na ocular.
- Para alinhar o condensador, ajuste a altura do condensador de modo a que ele está alinhado no centro de iluminação Kohler, concentrando-se na borda hexagonal da parada de campo condensador. Centro do campo o condensador batente sobre o campo de visão, se necessário, ao girar o botão do condensador de centralização. A paragem de abertura do condensador deve ser fechado.
- Inicie o programa IPlab e as configurações de entrada para operar o RoperScientific Cascade 512 câmera. Confirmar que a câmera está ajustada para o modo de transferência de quadro. Iniciar a visualização ao vivo, executando o "Adquirir foco" de comando. Definir o prefixo do índice e localização do arquivo para o qual as imagens serão salvas.
- Inicie o programa RSImage e as configurações de entrada para operar o programa CoolSnap. Modo de clock deve ser definido como normal.
- Inicie o software DMD e coloque o iris de campo escuro no menu script, seguido pelo "Load e Reset" comando e executar o script.
- Enviar a luz para a DMD e Cascade 512 câmera ajustando o microscópio optovar e viewport para LSM. Isto irá enviar a imagem através da DMD e da ótica alinhada, projetando a imagem sobre o CCD DF. Escuro campo de imagem (DF) deve aparecer na visualização ao vivo já em curso em IPlab. Ajustar o foco fino do microscópio, se necessário, para focar a imagem na visualização ao vivo.
- Tirar um instantâneo do campo de visão usando o "adquirir single" comando. Definir o tempo de exposição alto o suficiente para garantir pelo menos 10 mil crimes de sinal na imagem. Após a aquisição, use o "salvar como indexados" comando para salvar a imagem em disco. Esta imagem do graticule mede o tamanho do campo de visão (FOV).
- Agora, mova a amostra de modo que a retícula retículas está além do FOV para que de fundo só é visível. Adquirir uma imagem de fundo do campo em umlongo tempo de exposição suficiente para garantir que pelo menos 5000 pontos de sinal é adquirido. Esta imagem ajudará na subtração de fundo das imagens não filtrada.
3. Carregando o filterbank e usando a configuração de adquirir filtrada fundo de imagens
- Agora, precisamos adquirir Gabor filtrada imagens de fundo. Carregar o filtro de Gabor roteiro bancária para o software de controle de DMD. Executar o script inteiro para o buffer os filtros para a memória onboard do DMD, isso pode levar alguns minutos.
- Uma vez que todo o script é tamponado, podemos agora adquirir imagens filtradas do fundo. Use os marcadores de início e fim dentro do software DMD para instruir o DMD para carregar apenas um conjunto de filtros correspondentes a um filtro de Gabor-like de cada vez, e executar o script. A pré-visualização de imagens ao vivo deve mudar de campo escuro para a imagem filtrada para esse filtro.
- Abra o script de aquisição em IPlab do disco. Ajustar o tempo de exposição para garantir que pelo menos 2.000 acusações de sinal estão sendo adquiridos. Como o script DMD estiver em execução, cancelar a visualização ao vivo em IPlab e executar o script de aquisição. Isto irá automaticamente adquirir, índice e salvar a imagem filtrada para o disco.
- Uma vez que a primeira imagem é adquirida, pare o script em execução no software DMD e excluir os comandos usados a partir do script. Substitua o início e parar de marcadores no início e final do conjunto de filtros que vem. Repita a aquisição em IPlab.
- Repita o passo 3.4 até o filterbank inteira tem sido utilizado e todas as imagens filtradas foram adquiridos e salvos.
4. Chapeamento as células
- Até agora, as células em breve estará pronto para placa para o experimento. Ligue o ferro de solda na bancada do laboratório. Remova a mídia de visualização L15 e calor a 37 ° C. Faça uma estação de trabalho com uma toalha de papel e um Kimwipe. Faça mechas várias rasgando e girando Kimwipes. Os pavios vai ajudar na transferência de fluidos de e para a placa de celular.
- Após isso, precisamos placa da amostra. Usamos o porta-amostras usinadas placa de metal para nossas células, fazendo um "sanduíche lamela" com a placa de metal no meio. Aplique uma camada fina de graxa de vácuo com uma seringa em torno da periferia superior do buraco placa de metal estendendo até a metade para as extremidades dos sulcos de cada lado. Pressione suavemente um não limpo. Uma lamela para a graxa. Virar a placa ao longo e aplique graxa ao redor do buraco. Desligue as luzes da sala.
- Agora temos as células da incubadora, a manipulação do conteúdo incubadora com luvas de borracha nitrílica exame esterilizados com álcool 70%. Remova a placa de células da incubadora, prendendo a respiração enquanto a porta da incubadora está aberta. Tenha cuidado para minimizar a exposição à luz ambiente.
- Retirar as lamelas que serão utilizados para o experimento a partir da placa de seis bem, observando que o lado que estava virado para cima no poço é o lado com células anexado. Secar cuidadosamente a lamínula em ambos os lados até que ele é quase completamente seco, mantendo a leitura de que lado da lamela tem as células. Em seguida, pressione a lamela lateral célula, para baixo, para a placa de metal sobre o buraco untada visualização, para que não se lacunas de ar permanecem dentro da camada de graxa. A graxa deve formar uma vedação estanque para permitir-nos para carregar as células com o meio L15. Uma vez que você esteja certo disto, virar a placa traseira mais.
- Pipetar o meio L15 nas células banhado, forçando o líquido através do sulco entre a lamela superior ea placa de metal. Pipetagem 200 mL de cada vez funciona bem. O primeiro volume da pipeta deve preencher o espaço imprensado entre as lamelas com o líquido estender quase até o sulco do outro lado.
- Pipetar 200 mL de outro meio para as células banhado, mas desta vez, realizar um pavio no bosque opor de modo que o meio flui de um lado para o outro. Esta lava as células e remove todos os traços do meio de idade. Tenha cuidado para evitar que as bolhas se formem dentro do líquido durante esta etapa. Repita esse processo 2-3 vezes usando um novo pavio para cada lavagem.
- Lembre-se que o ferro de solda ligado? Agora é a hora dele ser usado. Virar a placa de cabeça para baixo mais uma vez, apoiando a chapa a partir das bordas para que o líquido é aprisionado no reservatório de células e não pode escorrer. Mergulhe o ferro de solda no copo valap. Isso vai derreter rapidamente alguns dos valap que então se apegam à ponta do ferro de soldar. Cuidadosamente aplicar o valap fundido em torno das bordas da lamínula de fundo (que agora está voltada para cima), usando a ponta do ferro de solda como um aplicador. Continue mergulhando e aplicação até que uma você percorrer todo o caminho em torno do perímetro lamela, selando a lamínula para a placa de metal.
- A lamela inferior tem células crescendo sobre ele, e pode ter resíduo do meio de secas no lado exposto. Limpe todos os resíduos da superfície lamela por balling um Kimwipe e limpandog as lamelas em um único movimento deslizante bem como a limpeza do objetivo. Isso garante que a lamela é o mais limpo no centro, onde ele será visto.
- Desligue o ferro de solda e retornar a placa 6-bem para a incubadora observando a contenção mesmos procedimentos e esterilidade. Tome as células banhado ao laboratório óptica e montar o objectivo, tal como descrito nos passos 2.4 e 2.5.
5. Condução do experimento
- Encontrar um campo bom de células com aparência saudável.
- Adquirir uma imagem de campo escuro do campo de visão. Alinhar o microscópio em contraste de interferência diferencial (DIC) e adquirir uma imagem DIC. Certifique-se que os tempos de exposição são longos o suficiente para garantir que o sinal é adequada.
- Agora temos que adquirir as imagens fluorescentes na câmara outras. Para obter imagens DIC na CoolSnap, usamos um LED azul ligado ao condensador, substituindo-o e removendo-o conforme necessário. Enquanto o microscópio é ainda alinhados em DIC, Enviar a luz para o CoolSnap definindo o optovar microscópio para 1.0x e viewport para binocular 100%. Desviar a imagem do visor da câmera. Coloque o LED sobre o condensador para iluminar o campo e visualizar o FOV em RSimage e ajuste de foco fino, se necessário. Adquirir uma imagem DIC e salvar no disco. Note como o FOV é diferente do obtido a partir da câmera Cascade. Essas imagens terão de ser registrados durante a fase de análise após o experimento.
- Obter uma imagem de fluorescência, ajustando o cubo filtro para o filtercube fluoresceína. Adquirir uma imagem por breves instantes, virando na excitação de fluorescência usando o microscópio e depois desligá-lo, logo que a aquisição seja concluída. Uma vez que focamos a amostra em DIC, a imagem de fluorescência está focada também. Isso economiza tempo de exposição em fluorescência, atrasando assim o fotobranqueamento. Salvar a imagem de fluorescência para o disco.
- Agora temos que adquirir as imagens filtradas. Redefinir o microscópio para campo escuro e reenviar a luz através da porta LSM como em 2.9.
- Executar o script toda filterbank Gabor como em 3,3-3,5. Temos agora completou a aquisição de dados para um ponto no tempo.
6. Mudar o meio para expor as células a estaurosporina (STS), e mantendo a média durante todo o experimento
- Enquanto as células ainda estão no palco e sem perturbar o campo de visão, mude o meio L-15 regular para o mesmo contendo uma solução M 1 da STS feita a partir de uma solução stock 4 mM de STS em DMSO. Use o método wicking descrito nos passos 4,6 para mudar a mídia.
- Agora, repetimos os passos de 5,2-5,6 pontos subseqüentes. Repetimos este processo até que o experimento seja concluída.
- Durante o curso do experimento, mais médio terá de ser adicionado de forma a amostra não desidratar. Isso é realizado por meio de pipetagem para o bosque da placa de celular, sem retirar do palco e sem perturbar o FOV.
7. Resultados representante
Na conclusão do experimento, os dados coletados vão incluir um grande número de imagens filtradas que precisam ser processados para extrair os dados subcelulares estruturais. Dois exemplos são mostrados para um banco de filtro óptico consistindo de 9 Gabor filtros como com o período de filtro S = 0.95μm, Gaussian desvio envelope padrão s = S / 2 = 0,45, e orientações Φ = 0 ° a Φ = 160 º em 20 ° incrementos. (Ver também [1] para mais detalhes).
Exemplo 1: Marine Diatom
Nós aplicada pela primeira vez nossa orientação banco sensíveis filtro para uma amostra de diatomáceas marinhas (Carolina Supply Company Biológicas) com características orientado que eram claramente visíveis em campo escuro (DF) imagem (Fig. 1). As imagens opticamente filtrados são mostrados ao lado da imagem não filtrada da amostra para comparação.
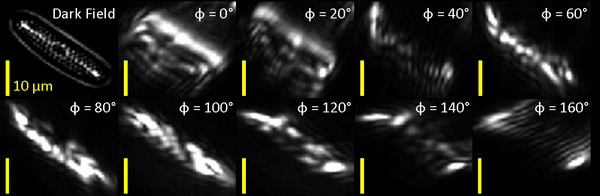
Figura 1: campo escuro (DF) e imagem opticamente filtrada de diatomáceas marinhas. Vamos analisar a diatomácea no canto inferior direito da imagem (seta branca no painel mais à esquerda).
O conjunto de nove Gabor filtrada imagens das diatomáceas foram processadas pixel por pixel de orientação a objetos e redondeza. O processamento consistiu de (1) somando respostas medidas de todas as nove Gabor filtrada imagens em cada pixel para determinar a magnitude total do sinal de resposta, assim, a codificação significado resposta, e (2) encontrar a orientação Gabor filtro, Φ, em que a resposta é maximizada e tendo a proporção desta resposta máxima para a resposta média para todos os ângulos, assim, a codificação em que medida os objetos em cada pixel tem uma orientação preferencial. O grau de orientação está intimamente relacionada com a proporção geométrica da partícula. Na fig. 2B, the de resposta global do pixel para o banco de filtros (parâmetro 1) eo grau de orientação ou relação de aspecto (parâmetro 2) são codificados na saturação de cor e tonalidade, respectivamente. Uma razão de aspecto próxima de 1 (azul) está presente nas áreas em que não há ângulo resposta preferida, enquanto valores maiores (vermelho) indicam áreas em que uma resposta maior ângulo preferido é o presente. Orientação de partículas sub-estrutura é codificado em uma trama quiver (Fig. 2C), onde cada linha de perto concordou com a orientação a objetos subjacentes locais visíveis em campo escuro-não filtrada (Fig. 2A).

Figura 2: A: imagem de campo escuro de diatomáceas. B: image orientação a objetos. Escala de cores indica grau de orientação (relação de aspecto), enquanto o brilho codifica significado do total de resposta do filtro Gabor. C: Orientação de objetos com a intensidade da resposta ≥ 10% do máximo. Segmento de reta indica eixo longitudinal da estrutura correspondente.
Exemplo 2: células apoptóticas
Aqui nós mostramos imagens filtradas de bovinos tratados com células endoteliais estaurosporina (STS), que foram processados da mesma maneira como a diatomácea. Fig. 3 mostra um sem filtro escuro-campo de imagem (DF) das células, juntamente com as nove imagens filtradas em tempo T =- 180 min. antes do tratamento STS.
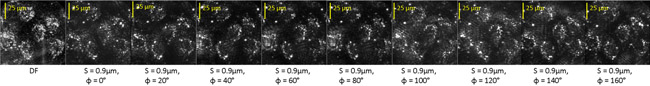
Figura 3: campo escuro (DF) e imagens opticamente filtrada de um campo que contém células vivas várias endoteliais.
As imagens filtradas foram adquiridos a cada 20 minutos por um período de três horas após o tratamento STS. Fig. 4a mostra um mapa proporção das células em função do tempo. Neste caso, a tonalidade da cor representa o grau de orientação (orientedness rotulado) como para a tonalidade de cor na figura. 2b acima. No entanto, o brilho relação de aspecto não foi ponderada pelo médio de resposta do filtro. Ao registrar nossos mapas proporção com imagens de fluorescência da mitocôndria nessas células rotulados (Fig. 4b), determinou-se que a queda de relação medida aspecto foi confinada às regiões celular contendo mitocôndrias e foi concomitante com a fragmentação mitochondrial que poderia ser observada diretamente no as imagens de fluorescência das mesmas células. Fig. 5 mostra gráficos tempo mostrando a mudança na relação de aspecto em função do tempo em células em apoptose. Dentro de cada cela, há uma queda na proporção em T = 60-100 min nas regiões que registram com fluorescentes mitocôndria, mas não em regiões que registram com as áreas de fundo fraca fluorescência.
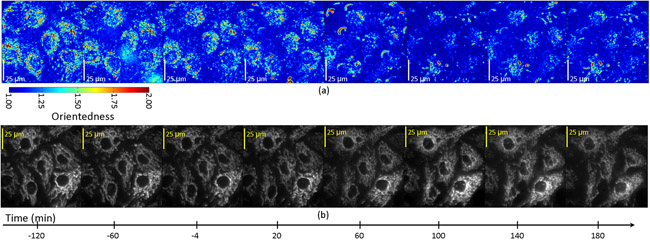
Figura 4: Proporção (a) e de fluorescência (b) imagens de células endoteliais tratadas com o indutor de apoptose, estaurosporina.
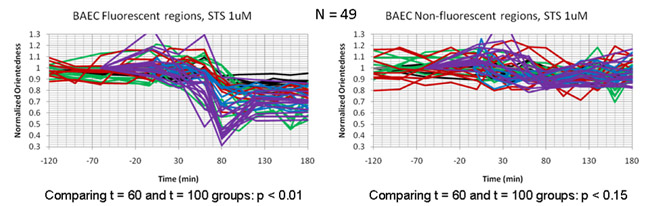
Figura 5: Tempo parcelas comparando a diminuição da proporção de partículas de aspecto (orientedness) em células endoteliais tratadas com estaurosporina. Os traços individuais representam parcelas tempo dentro de células individuais. A queda na orientedness está confinada às regiões das células que se registrar (painel esquerdo) mitocôndrias fluorescentes e está ausente das regiões de fundo restantes fluorescência (direita do painel).
Agora que nós determinamos que a queda proporção corresponde à fragmentação mitocondrial, pode-se induzir a apoptose nessas células, medir a fragmentação usando o nosso método de dispersão óptica sem ter que rotular as células, e estudar o efeito de diferentes condições genéticas e experimentais nesta dinâmica.
Discussão
O método descrito acima produz mapas morfométricos do objeto que pode codificar tamanho de partícula ou orientação, por exemplo. Esta informação estrutural pode ser usado de várias maneiras:
- Ele pode ser usado como uma tela inicial para identificar as regiões de tecidos ou células que são alteradas durante um tratamento específico e, em seguida, aprofundar a análise destas regiões com os ensaios moleculares e bioquímicas.
- Pode ser usado em conjunto com fluorescência para produzir u...
Divulgações
Agradecimentos
O dispositivo de micro-espelho nesta pesquisa foi financiada pela Fundação Whitaker conceder RG-02-0682 a N. Boustany. Trabalhos em curso é financiado pela concessão do NSF-DBI-0852857 para N. Boustany. Pasternack RM foi parcialmente financiado por uma Bolsa de Pós-Graduação Rutgers Presidencial. Gostaríamos também de agradecer ao Dr. E. White para as células iBMK utilizado em nossos estudos e Metaxas Dr. DN para discussão útil sobre óptica estratégias de filtragem.
Materiais
| Name | Company | Catalog Number | Comments |
| DMEM | Invitrogen | Low glucose DMEM | |
| Liebowitz L15 medium | Invitrogen | Without phenol red | |
| L-glutamine | Invitrogen | ||
| Mitotracker Green | Invitrogen | ||
| Bovine Brain Extract | Clonetics | ||
| Fetal Bovine Serum | Gemini Bio Products | ||
| Heparin | Sigma-Aldrich | ||
| Staurosporine | Sigma-Aldrich | ||
| Dymethylsulfoxide | Sigma-Aldrich | ||
| Inverted microscope | Carl Zeiss, Inc. | Axiovert 200M | |
| DMD | Texas Instruments | TI 0.7 XGA DMD 1100 | |
| CCD | Roper Scientific | Cascase 512B | High (16 bit) dynamic range CCD |
| CCD | Roper Scientific | Coolsnap cf |
Referências
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., White, E., Boustany, N. N. Measurement of Subcellular Texture by Optical Gabor-Like Filtering with a Digitial Micromirror Device. Optics Letters. 33 (19), 2209-2211 (2008).
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., Boustany, N. N. Highly sensitive size discrimination of submicron objects using optical Fourier filtering based on two-dimensional Gabor filters. Optics Express. 17 (14), 12001-12012 (2009).
- Zheng, J. -. Y., Pasternack, R. M., Boustany, N. N. Optical scatter imaging with a digital micromirror device. Optics Express. 17 (22), 20401-20414 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados