È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Scatter microscopia ottica Sulla base bidimensionale Filtri di Gabor
In questo articolo
Riepilogo
Noi dimostrare un campo scuro metodo di microscopia in base a Gabor-come filtro per misurare la dinamica sub-cellulari all'interno delle cellule viventi singolo. La tecnica è sensibile alle alterazioni nella struttura degli organelli, quali la frammentazione mitocondriale.
Abstract
Si dimostra uno strumento microscopici in grado di misurare struttura subcellulare derivanti dalla morfologia degli organelli e organizzazione all'interno delle cellule viventi senza macchia. Lo strumento proposto estende la sensibilità di etichetta senza microscopia ottica a cambiamenti nelle dimensioni degli organelli nanoscala e la forma e può essere utilizzato per accelerare lo studio della relazione struttura-funzione degli organelli dinamiche relative alla base dei processi biologici fondamentali, come la morte cellulare programmata o cellulare differenziazione. Il microscopio può essere facilmente implementata su piattaforme microscopia esistenti, e può quindi essere diffusi ai laboratori, dove gli scienziati possono implementare e utilizzare i metodi proposti con accesso illimitato.
La tecnica proposta è in grado di caratterizzare la struttura subcellulare osservando la cellula attraverso filtri ottici bi-dimensionale Gabor. Questi filtri possono essere sintonizzati con senso su scala nanometrica (10 di nm) sensibilità, specifici attributi morfologici riguardanti le dimensioni e l'orientamento di organuli subcellulari non sferiche. Mentre in base al contrasto generato dalla diffusione elastica, la tecnica non si basa su un modello dettagliato scattering inverso o sulla teoria di Mie per estrarre le misurazioni morfometriche. Questa tecnica è quindi applicabile alle obbligazioni extracontrattuali sferiche organelli per i quali una descrizione precisa dispersione teorica non è facilmente dato, e fornisce distintivo parametri morfometrici che possono essere ottenuti all'interno di cellule viventi senza macchia per valutare la loro funzione. La tecnica è vantaggiosa in confronto con l'elaborazione digitale delle immagini, nel senso che opera direttamente sul campo dell'oggetto trasformare piuttosto che l'intensità dell'oggetto discretizzato è. Non si basa su alti tassi di campionamento delle immagini e può quindi essere usata per selezionare rapidamente l'attività morfologica all'interno di centinaia di cellule alla volta, facilitando in tal modo lo studio della struttura degli organelli organello di là di singoli segmentazione e ricostruzione di microscopia confocale di fluorescenza molto ingrandito le immagini digitali del campo di visuale limitato.
In questa dimostrazione si mostrano i dati da una diatomea marina per illustrare la metodologia. Mostriamo anche i dati preliminari raccolti da cellule viventi per dare un'idea di come il metodo può essere applicato in un contesto rilevante biologico.
Protocollo
1. Come le cellule pronte
- Le cellule che sono stati placcati il giorno prima deve essere etichettato con il verde Mitotracker per l'imaging di fluorescenza dei mitocondri.
- Rimuovere 100 soluzione madre di verde Mitotracker mM in DMSO fatte precedentemente dal 4 ° C freezer, e la temperatura ambiente con la mano. Altresì stipulare bovina cellule endoteliali aortiche (BAEC) terreno di coltura cellulare anche precedentemente preparato e caldo a 37 ° C in bagnomaria da banco.
- Una volta che il Mitotracker e mezzo di coltura sono riscaldati, inserire questi nel cappuccio facendo attenzione a sterilizzare le mani guantate e tutte le superfici esterne dei contenitori con una soluzione di etanolo al 70%. Non accendere la luce cofano, come l'etichetta fluorescente è sensibile alla luce e presto photobleach in luce ambiente della stanza.
- Rendere la giusta concentrazione per l'etichettatura mitocondriale è molto importante. Troppo poco non si etichetta il efficacemente mitocondri, mentre Mitotracker troppo può avere effetti tossici. Una concentrazione di 100 nM di Mitotracker incubate per 45 minuti con le cellule funziona bene. Preparare questa concentrazione aggiungendo 100 ml di magazzino Mitotracker terreno di coltura a 10 mL in un tubo da 15 ml. Questo renderà molto per almeno un esperimento.
- Sostituire il supporto esistente con il medium etichettato succhiando il vecchio mezzo con una pipetta Pasteur collegato alla linea del vuoto. Poi subito aggiungere 2 mL del mezzo etichettati per ogni cultura occupato anche nella piastra ben 6.
- Perché l'etichetta fluorescente è sensibile alla luce, sostituire le celle nell'incubatore velocemente senza esporre alla luce artificiale diretta. Che copre i 6 pozzetti con le mani funziona bene per questo. Le cellule resteranno in incubatrice per 45 minuti.
2. Ottenere la configurazione ottica pronto
- Mentre le cellule aspettare nell'incubatrice, dobbiamo accendere la configurazione ottica. Nella sala ottica, primo turno la lampada a vapori di mercurio, seguita dal computer, il microscopio, le telecamere, e il laser. Poi collegare il dispositivo a microspecchio digitale (DMD) e il diffusore di filatura.
- Verificare che il lancio ottico sia allineato guardando attraverso l'oculare microscopio per garantire che il campo visivo è illuminato dalla luce laser.
- Pulire l'obiettivo piegando un pezzo di carta per lenti in una piazza stretta e afferrare saldamente con una hemostat. Immergere la carta lente in ammoniaca senza soluzione di pulizia di vetro di assorbire una piccola quantità nella carta. Il rap hemostat sulla vostra mano libera più volte per rimuovere l'eccesso. Pulire l'obiettivo con fermezza applicando una pulita, strisciare continuo attraverso l'obiettivo da un capo all'altro, passando sopra l'obiettivo al centro. Non re-swipe o macchia. Eliminare carta utilizzata.
- Per caricare il campione, posizionare il reticolo rispetto all'obiettivo 63x ad immersione ad olio di cadere 1-2 gocce di olio immersione rispetto all'obiettivo, mentre l'obiettivo è fino in fondo. Poi posto il reticolo nella fase. Quindi sollevare l'obiettivo in modo che l'olio "afferra" il campione. Messa a fuoco il campione nell'oculare.
- Per allineare il condensatore, regolare l'altezza del condensatore in modo che sia allineato in illuminazione centrale Kohler mettendo a fuoco il bordo esagonale della fermata campo condensatore. Centro del campo condensatore sosta il campo di vista, se necessario, ruotando le manopole condensatore di centraggio. Il diaframma a condensatore deve essere chiusa.
- Avviare il programma IPlab e le impostazioni di ingresso per operare il RoperScientific Cascade 512 telecamera. Verificare che la fotocamera è impostata per inquadrare modalità di trasferimento. Avviare l'anteprima dal vivo eseguendo il comando "Acquisisci focus". Impostare il prefisso indice e la posizione del file in cui le immagini verranno salvate.
- Avviare il programma RSImage e le impostazioni di ingresso per operare il programma CoolSnap. Modalità di clock deve essere impostato su normale.
- Avviare il software DMD e posto l'iride scura campo nel menu script, seguito dal "Load e Reset", il comando ed eseguire lo script.
- Invia la luce per la DMD e Cascade 512 fotocamera impostando il microscopio optovar e finestra da LSM. Questo inviare la foto tramite la DMD e l'ottica allineati, proiettando l'immagine DF sul CCD. Il campo scuro (DF) l'immagine dovrebbe apparire l'anteprima dal vivo già in corso in IPlab. Regolare la messa a fuoco fine del microscopio, se necessario, mettere a fuoco l'immagine sul anteprima dal vivo.
- Prendere una fotografia istantanea del campo visivo usando il comando "acquisire singolo". Impostare il tempo di esposizione abbastanza elevata da garantire almeno 10000 conteggi di segnale nell'immagine. Dopo l'acquisizione, utilizzare il comando "salva come indicizzato" per salvare l'immagine su disco. Questa immagine del reticolo misura la dimensione del campo visivo (FOV).
- Ora, spostare il campione del reticolo in modo che il reticolo è al di là del campo visivo in modo che solo sfondo è visibile. Acquisire un'immagine di sfondo del campo ad unalungo tempo di esposizione sufficiente a garantire che almeno 5000 conti di segnale è acquisito. Questa immagine sarà di aiuto in fondo sottrazione delle immagini non filtrate.
3. Caricamento del filtro a e utilizzando la configurazione per acquisire filtrate-immagini di sfondo
- Ora, abbiamo bisogno di acquisire Gabor filtrata immagini dello sfondo. Caricare il filtro di Gabor script di banca per il software di controllo DMD. Eseguire l'intero script per tamponare i filtri per la memoria a bordo della DMD, questo potrebbe richiedere alcuni minuti.
- Una volta che l'intero script è tamponata, siamo in grado di acquisire immagini filtrate dello sfondo. Utilizzare i marcatori di inizio e fine all'interno del software DMD per istruire il DMD di caricare un solo set di filtri corrispondente ad un Gabor-come filtro alla volta, ed eseguire lo script. L'anteprima immagine dal vivo dovrebbe cambiare da campo scuro per l'immagine filtrata per quel filtro.
- Aprire lo script di acquisizione in IPlab dal disco. Regolare il tempo di esposizione per garantire che almeno 2000 conti di segnale sono in corso di acquisizione. Come lo script DMD è in esecuzione, annullare l'anteprima dal vivo in IPlab ed eseguire lo script di acquisizione. Questo automaticamente acquisire, indicizzare e salvare l'immagine filtrata su disco.
- Una volta che la prima immagine viene acquisita, fermare lo script in esecuzione nel software DMD ed eliminare i comandi usati dallo script. Sostituire l'inizio e marcatori arresto all'inizio e alla fine del set filtro successivo. Ripetere l'acquisizione IPlab.
- Ripetere il punto 3.4 fino a quando il filtro a tutto è stato utilizzato e tutte le immagini filtrate sono stati acquisiti e salvati.
4. Placcatura le cellule
- A questo punto, le cellule saranno presto in grado di piastra per l'esperimento. Collegare il saldatore sul bancone di laboratorio. Rimuovere la L15 media di visualizzazione e di calore a 37 ° C. Fai una stazione di lavoro con un tovagliolo di carta e un Kimwipe. Fare stoppini diversi da strappi e twirling Kimwipes. Gli stoppini aiuterà a trasferire fluido da e per la piastra cellulare.
- Dopo questo, abbiamo bisogno di piastra del campione. Usiamo il porta-campioni in metallo lavorato a piastra nostre cellule, rendendo un "sandwich vetrino" con la piastra di metallo in mezzo. Applicare uno strato sottile di grasso vuoto da una siringa intorno alla periferia superiore del foro piastra metallica si estende per circa metà strada fino ai confini delle scanalature su ogni lato. Premere delicatamente un no pulito. 1 coprioggetto sul grasso. Capovolgere la piastra su e applicare grasso intorno al buco. Spegnere le luci della stanza.
- Ora abbiamo le cellule dal termostato, la gestione dei contenuti incubatore con i guanti di nitrile esame sterilizzati con etanolo al 70%. Rimuovere la piastra di cellule dal termostato, trattenere il respiro mentre la porta è aperta incubatore. Fare attenzione a minimizzare l'esposizione alla luce ambiente.
- Rimuovere il vetrino che verrà utilizzato per l'esperimento di sei pozzetti, facendo notare che il lato che era rivolto verso l'alto nel pozzo è il lato con le cellule allegato. Asciugare con cura il vetrino su entrambi i lati fino a quando è quasi completamente asciutta, tenendo traccia di quale lato del coprioggetto ha le cellule. Quindi premere il, lato cellula coprioggetti verso il basso, nella piastra di metallo unto sopra il foro di visualizzazione, facendo attenzione che non rimangano vuoti d'aria all'interno dello strato di grasso. Il grasso deve formare una guarnizione a tenuta stagna per permettere di caricare le cellule con il mezzo L15. Una volta certi di questo, capovolgere la piastra posteriore sopra.
- Pipetta la L15 media nelle cellule placcato forzando il liquido attraverso il solco tra il vetrino superiore e la piastra metallica. Pipettaggio 200 ul in un momento funziona bene. La pipetta primo volume dovrebbe riempire lo spazio interposto tra i coprioggetti con il liquido si estende quasi fino alla scanalatura sul lato opposto.
- Pipettare altri 200 l di media nelle cellule placcato, ma questa volta, tenere uno stoppino al boschetto si oppongono in modo che il fluido scorre da un lato all'altro. Questa lava le cellule e rimuove ogni traccia del mezzo vecchio. Fare attenzione per evitare la formazione di bolle all'interno del liquido durante questo passaggio. Ripetete questo processo 2-3 volte con un nuovo stoppino per ogni lavaggio.
- Ricorda il saldatore abbiamo inserito? Ora è il momento viene utilizzato. Capovolgere la piastra a testa in giù una volta di più, sostenendo la piastra dai bordi in modo che il liquido è intrappolato nel serbatoio di cellule e non può gocciolare. Immergere il saldatore nel becher valap. Questo sarà presto sciogliere alcuni dei valap che poi aggrapparsi alla punta saldatore. Applicare con attenzione la valap fuso lungo i bordi del coprioggetto fondo (che ora è rivolto verso l'alto) usando la punta saldatore come un applicatore. Continua ad immersione e applicando fino a quando un si va tutto intorno al perimetro coprioggetti, sigillando il coprioggetto sulla lamiera.
- Il fondo ha coprioggetto cellule in crescita su di esso, e possono avere residui dal mezzo a secco sul lato esposto. Pulire eventuali residui dalla superficie del vetrino da balling un Kimwipe e Cleaning il coprioggetto con un unico movimento molto scorrevole come pulire l'obiettivo. Ciò assicura che il vetrino è pulito nel centro dove sarà visualizzato.
- Scollegare il saldatore e ritorno il 6-pozzetti per l'incubatore osservando il contenimento stessi e le procedure di sterilità. Prendere le cellule placcato al laboratorio di ottica e montare sull'obiettivo come descritto nei punti 2.4 e 2.5.
5. Condurre l'esperimento
- Trova un bel campo di un aspetto sano cellule.
- Acquisire un'immagine scura di campo del campo di vista. Allineare il microscopio in contrasto di interferenza differenziale (DIC) e di acquisire una immagine DIC. Assicurarsi che i tempi di esposizione sono abbastanza a lungo per garantire che il segnale sia sufficiente.
- Ora dobbiamo acquisire le immagini fluorescenti sulla fotocamera. Per ottenere immagini DIC sul CoolSnap, usiamo un LED blu attaccato al condensatore, in sostituzione di esso e la rimozione se necessario. Mentre il microscopio è ancora allineata a DIC, Invia la luce alla CoolSnap impostando il optovar microscopio a 1,0 x e finestra al 100% binoculare. Deviare l'immagine dall'oculare alla fotocamera. Posizionare il LED sopra il condensatore ad illuminare il campo e vedere in anteprima il FOV in RSimage e regolare la messa a fuoco fine, se necessario. Acquisire un'immagine DIC e salvarlo su disco. Si noti come il campo visivo è diverso da quello ottenuto dalla fotocamera Cascade. Queste immagini dovranno essere registrati durante la fase di analisi dopo l'esperimento.
- Ottenere un'immagine fluorescenza regolando il cubo filtro alla filtercube fluoresceina. Acquisire immagini con una breve accendere l'eccitazione di fluorescenza utilizzando il microscopio e poi si spegne non appena l'acquisizione è stata completata. Dal momento che ci siamo concentrati il campione in CID, l'immagine di fluorescenza è concentrata pure. Ciò consente di risparmiare sul tempo di esposizione in fluorescenza, rallentando di conseguenza photobleaching. Salvare l'immagine di fluorescenza su disco.
- Ora abbiamo di acquisire le immagini filtrate. Resettare il microscopio a campo scuro e inviare nuovamente la luce attraverso la porta LSM come in 2.9.
- Eseguire l'intero script di filtro a Gabor come in 3,3-3,5. Ora abbiamo completato l'acquisizione di dati per un punto del tempo.
6. Commutazione del mezzo per esporre le cellule a staurosporina (STS), e mantenendo il supporto per tutto l'esperimento
- Mentre le cellule sono ancora sul palco e senza disturbare il campo visivo, passare la regolare L-15 di media per lo stesso contenente una soluzione 1 mM di STS fatto da una soluzione al 4 magazzino mM di STS in DMSO. Utilizzare il metodo traspirazione descritto nei passaggi da 4,6 a cambiare i media.
- Ora, abbiamo ripetere i passaggi 5,2-5,6 per rilevazioni temporali successive. Ripetiamo questo processo fino a quando l'esperimento è stato completato.
- Nel corso dell'esperimento, più a medio dovranno essere aggiunti in modo che il campione non essiccare. Ciò si ottiene pipettando medio nel boschetto della piastra cellulare senza togliere dal palco e senza disturbare il campo visivo.
7. Rappresentante Risultati
Al termine dell'esperimento, i dati raccolti verranno comprende un gran numero di immagini filtrate, che devono essere elaborati per estrarre i dati strutturali subcellulare. Due esempi sono mostrati di una banca filtro ottico composto da 9 Gabor-come filtri con filtro periodo S = 0.95μm, deviazione busta gaussiana standard s = S / 2 = 0.45μm, e gli orientamenti Φ = 0 ° a Φ = 160 ° in 20 ° incrementi. (Vedi anche [1] per maggiori dettagli).
Esempio 1: Marine diatomee
In primo luogo abbiamo applicato la nostra banca sensibile orientamento filtro a un campione di diatomee marine (Supply Company Carolina biologica) con caratteristiche orientate che erano chiaramente visibili in campo scuro (DF) immagini (fig. 1). Le immagini otticamente filtrate sono mostrati a fianco l'immagine filtrata del campione per il confronto.
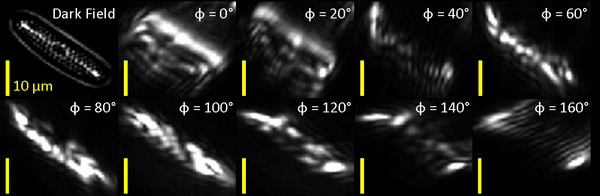
Figura 1: campo scuro (DF) e l'immagine filtrata otticamente di diatomee marine. Analizzeremo le diatomee in basso a destra dell'immagine (freccia bianca nel riquadro più a sinistra).
La serie di nove Gabor filtrata immagini della diatomea sono stati elaborati pixel per pixel per l'orientamento agli oggetti e rotondità. Lavorazione consisteva (1) sommando le risposte misurate di tutti e nove Gabor filtrata immagini a ciascun pixel per determinare la grandezza complessiva della risposta del segnale in tal modo la codifica significato di risposta, e (2) trovare l'orientamento Gabor filtro, Φ, in cui la risposta è massimizzato e prendendo il rapporto di questa risposta massima al di risposta medio per tutti gli angoli così che codifica per la misura in cui gli oggetti per ogni pixel ha un orientamento preferenziale. Il grado di orientamento è strettamente legato al rapporto di aspetto geometrico della particella. Nella fig. 2B, thrisposta e complessiva del pixel al banco di filtri (parametro 1) e il grado di orientamento o rapporto di aspetto (parametro 2) sono codificati nella saturazione del colore e la tonalità, rispettivamente. Un rapporto vicino a 1 (blu) è presente in aree in cui non c'è risposta angolo preferito, mentre i valori più elevati (rosso) indicano le aree in cui una risposta più alto angolo preferito è presente. Particelle orientamento sottostruttura è codificato in una trama faretra (Fig. 2C), dove ogni linea strettamente concordato con l'orientamento di fondo oggetto locale visibile non filtrato a campo scuro (Fig. 2A).

Figura 2: A: immagine campo scuro di diatomee. B: l'orientamento delle immagini di oggetti. Scala di colore indica il grado di orientamento (aspect ratio), mentre la luminosità codifica significato del totale risposta Gabor filtro. C: Orientamento di oggetti con intensità di risposta ≥ 10% del massimo. Segmento di linea indica l'asse lungo della struttura corrispondente.
Esempio 2: le cellule apoptotiche
Qui vi mostriamo le immagini filtrate di bovini cellule endoteliali trattate con staurosporina (STS) che sono stati trattati nello stesso modo come il diatomee. Fig. 3 mostra un non filtrato a campo scuro (DF) l'immagine delle celle con nove immagini filtrate al tempo T =- 180 min. prima del trattamento STS.
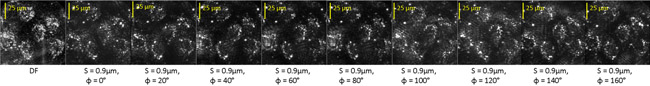
Figura 3: campo scuro (DF) e le immagini otticamente filtrata di un campo che contiene le cellule viventi diversi endoteliali.
Le immagini filtrate sono stati successivamente acquisiti ogni 20 minuti per un periodo di tre ore dopo il trattamento STS. Fig. 4a mostra una mappa formato delle celle in funzione del tempo. In questo caso la tonalità del colore rappresenta il grado di orientamento (Orientazione etichettati) come per la tonalità del colore in fig. 2b di cui sopra. Tuttavia, la luminosità proporzioni non è stata ponderata dalla risposta media del filtro. Registrando le nostre mappe rapporto di aspetto con immagini di fluorescenza dei mitocondri etichettate in queste cellule (Fig. 4b), abbiamo stabilito che la misura goccia proporzioni era confinato alle regioni cellulare contenente mitocondri ed è stata concomitante con la frammentazione mitocondriale che potrebbe essere osservati direttamente in le immagini fluorescenza delle cellule stesse. Fig. 5 mostra trame volta raffigurante il cambiamento di proporzioni in funzione del tempo nelle cellule in fase di apoptosi. All'interno di ogni cella, c'è un calo di proporzioni a T = 60-100 min nelle regioni che registrano con quelle fluorescenti mitocondri, ma non nelle regioni che registrano con le aree di sfondo fioca fluorescenza.
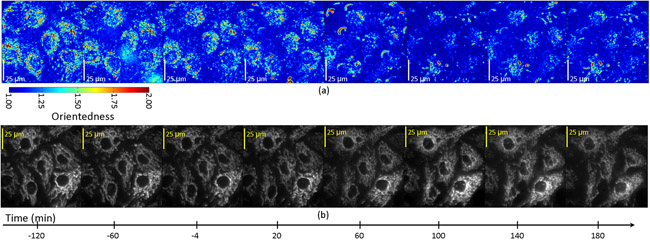
Figura 4: Aspect ratio (a) e di fluorescenza (b) immagini di cellule endoteliali trattate con l'induttore dell'apoptosi, staurosporina.
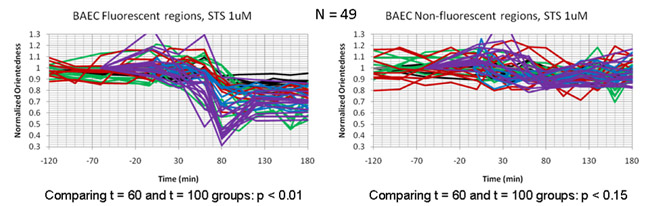
Figura 5: trame Ora confrontando la diminuzione del rapporto di aspetto di particelle (Orientazione) nelle cellule endoteliali trattate con staurosporina. Le tracce individuali rappresentano trame volta all'interno delle cellule singole. Il calo Orientazione si limita alle regioni delle cellule che registri con fluorescenti mitocondri (a sinistra del pannello) ed è assente dalle regioni rimanenti fluorescenza di fondo (a destra del pannello).
Ora che abbiamo determinato che la caduta di proporzioni corrisponde alla frammentazione mitocondriale, siamo in grado di indurre l'apoptosi in queste cellule, misurare la frammentazione utilizzando il nostro metodo ottico dispersione senza dover etichettare le cellule, e studiare l'effetto di differenti condizioni genetiche e sperimentali su questo dinamica.
Discussione
Il metodo sopra descritto produce mappe morfometriche dell'oggetto che possono codificare la dimensione delle particelle o di orientamento per esempio. Queste informazioni strutturali possono essere utilizzati in diversi modi:
- Può essere utilizzato come una schermata iniziale per identificare le regioni tessuti o cellule che sono state modificate nel corso di un trattamento specifico e poi analizzare ulteriormente queste regioni, con specifiche analisi molecolari e biochimici.
- Può essere uti...
Divulgazioni
Riconoscimenti
Il micro-specchio dispositivo in questa ricerca è stato finanziato dalla sovvenzione della Fondazione Whitaker RG-02-0682 a N. Boustany. I lavori in corso è finanziato da concedere NSF-DBI-0852857 a N. Boustany. Pasternack RM è stato parzialmente sostenuto da una borsa di studio Rutgers presidenziale Graduate. Vorremmo anche ringraziare il Dott. E. White per le cellule iBMK utilizzati nei nostri studi e il Dr. Metaxas DN di discussione utile per quanto riguarda le strategie di filtraggio ottico.
Materiali
| Name | Company | Catalog Number | Comments |
| DMEM | Invitrogen | Low glucose DMEM | |
| Liebowitz L15 medium | Invitrogen | Without phenol red | |
| L-glutamine | Invitrogen | ||
| Mitotracker Green | Invitrogen | ||
| Bovine Brain Extract | Clonetics | ||
| Fetal Bovine Serum | Gemini Bio Products | ||
| Heparin | Sigma-Aldrich | ||
| Staurosporine | Sigma-Aldrich | ||
| Dymethylsulfoxide | Sigma-Aldrich | ||
| Inverted microscope | Carl Zeiss, Inc. | Axiovert 200M | |
| DMD | Texas Instruments | TI 0.7 XGA DMD 1100 | |
| CCD | Roper Scientific | Cascase 512B | High (16 bit) dynamic range CCD |
| CCD | Roper Scientific | Coolsnap cf |
Riferimenti
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., White, E., Boustany, N. N. Measurement of Subcellular Texture by Optical Gabor-Like Filtering with a Digitial Micromirror Device. Optics Letters. 33 (19), 2209-2211 (2008).
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., Boustany, N. N. Highly sensitive size discrimination of submicron objects using optical Fourier filtering based on two-dimensional Gabor filters. Optics Express. 17 (14), 12001-12012 (2009).
- Zheng, J. -. Y., Pasternack, R. M., Boustany, N. N. Optical scatter imaging with a digital micromirror device. Optics Express. 17 (22), 20401-20414 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon