É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Bromodeoxiuridina Labeling (BrdU) e separação de células subseqüentes Fluorescence Activated de Cultura independente de Identificação de carbono orgânico dissolvido de degradação bacterioplâncton
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Bacterioplâncton ambientais são incubados com um modelo de carbono orgânico dissolvido composto (DOC) e um reagente rotulagem DNA, bromodeoxiuridina (BrdU). Depois, DOC-degradante células são separadas da comunidade em massa com base em sua incorporação BrdU elevada usando separação de células ativadas por fluorescência (FACS). Estas células são então identificadas por análises subseqüentes molecular.
Resumo
Micróbios são agentes importantes mediando a degradação do carbono dissolvido numerosos orgânico (DOC) substratos em ambientes aquáticos. No entanto, a identificação da taxa de bactérias que transformam a grupos específicos de DOC na natureza representa um desafio técnico.
Aqui nós descrevemos uma abordagem que os casais bromodeoxiuridina incorporação (BrdU), separação de células ativadas por fluorescência (FACS), e 16S rRNA gene baseada em análise molecular que permite que a cultura independente de identificação do bacterioplâncton capazes de degradar compostos DOC uma específicos em ambientes aquáticos. Microcosmos bacterioplâncton triplicado são configurados para receber tanto BrdU e um composto modelo DOC (DOC alterações), ou apenas BrdU (sem adição de controle). Substitutos BrdU as posições de timidina no DNA recentemente sintetizado bacteriana e BrdU marcado DNA pode ser prontamente 1,2 immunodetected. Através de uma de 24 horas de incubação bacterioplâncton, que são capazes de usar o composto adicionado DOC são esperados para ser seletivamente ativado, e, portanto, têm níveis mais elevados de incorporação de BrdU (células HI) que os não-responsivos células nas alterações DOC e células em no- Além controles (células de baixa incorporação de BrdU, células LI). Depois de imunodetecção de fluorescência, as células HI são distinguidos e separados fisicamente das células LI por separação de células ativadas por fluorescência (FACS) 3. Ordenado DOC-responsiva (células HI) são extraídos de DNA e identificados taxonomicamente por meio de 16S rRNA gene subseqüentes análises baseadas incluindo PCR, clonagem e sequenciamento de construção da biblioteca.
Protocolo
1. Processamento de amostra de água
- Filtro de água 10L ambiental através de uma filtros mM de poros de tamanho de membrana para remover partículas grandes e bacteriovores. Recolher o filtrado em um garrafão de água.
- Transferência de 36 ml cada filtrado em 3 tubos estéreis Eppendorf (50 ml) contendo 4 ml de solução recém-preparada de paraformaldeído (PFA; 10%). Incubar por 2 horas em temperatura ambiente para preservar as células. Coletar células para 0,22 mícrons de tamanho de poros filtros de membrana branca por filtração a vácuo. Lavar o filtro, passando de 10 ml tampão fosfato (PBS) através do filtro por filtração a vácuo. Rotular os filtros como controles negativos e armazená-los a -20 ° C.
1,3-1,4 passos são opcionais para o estabelecimento de DOC-limitada condições.
- Adicionar uma mistura de nitrogênio inorgânico e fósforo (5 mM NH 4 Cl, 5 M NaNO 3 e 1 Hm NaH 2 PO 4 a concentração final) para o garrafão.
- Incubar no escuro, na temperatura local por 48 horas, com agitações ocasionais.
2. Estabelecimento microcosmo e incubação
- Preencha cada um dos seis frascos de 1 L de vidro com 800 ml de amostra de água do garrafão de 1,4 passo para estabelecer microcosmos. Adicionar BrdU (10 mM, concentração final) para cada um microcosmo. Misture bem.
- Adicionar 1 ml de solução de modelo composto DOC em três dos microcosmos, estes servirão como alterações DOC. Adicionar 1 ml estéril PBS em três microcosmos restantes, estes servirão como sem adição de controles.
- Incubar todos os microcosmos em uma incubadora shaker e incubar no escuro, na temperatura situ agitando a 100 rpm.
- Coletar 36 ml de amostra de água de cada microcosmo e transferir para 50 ml tubos estéreis Eppendorf em momentos de 0, 8, 16 e 24 horas. Imediatamente adicionar 4 ml de PFA fresco (10%) para tubos de coleta e incubar por 2 horas em temperatura ambiente para preservar as células.
- Filtro preservada células através de 0,22 mícrons de tamanho de poros filtros de membrana de policarbonato. Lavar os filtros com 10 ml de PBS. Proceder de imediato para a próxima etapa ou armazenar os filtros a -20 ° C.
3. Na imunodetecção situ de Incorporação BrdU
- Descongelar os filtros (amostras DOC alterada, sem adição de controles e os controles negativos) em temperatura ambiente.
- Aplicar 1 ml de solução de lisozima [10 mg / ml lisozima clara de ovo em 100 mM Tris, 50 nM EDTA (pH = 8)] 4 para cobrir as células bacterianas no filtro. Incubar à temperatura ambiente por 30 minutos. Lavar o filtro, passando de 10 ml PBS através dela sob sucção.
- Adicionar 1 ml de solução de proteinase K [2 mg / ml proteinase K em 100 mM Tris, 50 nM EDTA (pH = 8)] 4 para cobrir as células bacterianas no filtro. Incubar à temperatura ambiente por 30 minutos. Lavar o filtro, passando de 10 ml PBS através dela sob sucção.
- Adicionar 1 ml solução exonuclease [exonuclease III (50 U / ml) em 5 mM MgCl 2 e 50 mM Tris-HCl] 5 para cobrir as células bacterianas no filtro. Incubar a 37 ° C por 30 minutos. Lavar o filtro, passando de 10 ml PBS através dela sob sucção.
- Montar câmaras quadro selo de incubação (Bio-Rad) de acordo com as instruções do fabricante.
- Fatia do filtro em oitavos utilizando uma lâmina estéril em um álcool limpo superfície seca.
- Utilizando uma pinça, coloque uma oitava seção de um filtro em uma câmara de incubação montadas quadro selo (oito quadros selo câmaras são necessários para cada amostra do filtro). O lado da seção de filtro que contém células deve estar voltado para cima. A umidade na parte de trás do filtro permitirá a vara filtro para o slide. Se o filtro torna-se seca, aplique uma pequena gota (2 L) de dih 2 O no centro da câmara antes de colocar a seção de filtro no slide.
Passos 3,8-3,20 use os reagentes da Roche em Kit Situ Proliferação Celular, FLUOS seguintes procedimentos modificados a partir de instruções do fabricante. Exceto para PBS, todos os reagentes são fornecidos dentro do kit.
- Aplicar tampão de incubação suficiente (0,5% albumina sérica bovina, 0,1% Tween20 em PBS), para cobrir uniformemente toda a seção de filtro na câmara de incubação, sem causar estouro de uma vez o selo é aplicado.
- Coloque uma capa de quadro de poliéster sobre o quadro câmara de incubação. Pressione para baixo para selar o bem câmara de incubação. Evite bolhas de ar acima do filtro. Incubar as câmaras seladas por 10 min em temperatura ambiente no escuro.
- Retire o selo de poliéster e abrir as câmaras de incubação. (Nota: a abertura das câmaras, por vezes, vai puxar para cima o selo moldura do slide Nesse caso, prepare uma nova câmara para as etapas seguintes..)
- Pipeta a solução de incubação de um canto da câmara. Evitar o contacto com o filtro.
- Lavar o filtro com cuidado pipetando 100 mL PBS efora da câmara de 3 vezes.
- Prepare anti-BrdU-FLUOS solução de trabalho, seguindo os passos recomendados pelo fabricante imediatamente antes da utilização.
- Pipetar 120 mL anti-BrdU-FLUOS solução de trabalho para o filtro, tendo o cuidado de cobrir uniformemente toda a superfície.
- Feche a câmara com uma capa de poliéster novo. Evite bolhas de ar no topo do filtro. Incubar a câmara no escuro a 37 ° C por 3 horas. Esta etapa irá rótulo BrdU incorporado DNA in situ com fluoresceína isotiocianato (FITC).
- Remova a tampa de poliéster e abrir a câmara de incubação. Pipeta com a solução anti-BrdU-FLUOS de trabalho. Lavar o filtro 3 vezes com PBS.
- Transferir o filtro da câmara de incubação a uma superfície estéril. Fatia da seção de filtro em pedaços pequenos usando uma lâmina estéril.
- Transferir os pedaços de filtro em 2 tubos de microcentrífuga ml, cada um contendo 1 ml de PBS. Firmemente a tampa do tubo e vedação com parafilme. Incubar a 37 ° C e 200 rpm por 10 min.
- Segura os tubos em um vortex vortexer e à velocidade máxima por 5 minutos. Pipetar o sobrenadante para um tubo de 15 ml estéril Eppendorf. Repita as etapas de incubação e vortex por 5 vezes mais. Combine o sobrenadante no mesmo tubo de 15 ml Eppendorf para cada amostra. Normalmente, 80% de células podem ser recuperados na suspensão.
- Armazenar o sobrenadante com células ressuspenso a 4 ° C. Ordenar dentro de 2 dias.
4. Análise FACS
Um procedimento para um citômetro de fluxo BD FACSAria e software correspondente é descrito aqui.
- Otimizar a configuração do fluxo passos seguintes citómetro recomendado pelo fabricante. Isto implica: ajustando o controle de amplificação para definir os parâmetros necessários e breakoff o ponto doce; otimizando o atraso laser e fatores de escala da área para a pressão de bainha de experiência e otimizar as configurações para FSC e SSC tensões, limiar FSC, FSC escala de fluorescência, fluorescência tensões PMT , etc
- Executar amostras de controlo negativo no citômetro de fluxo (FCM) com base na intensidade de fluorescência FITC e de dispersão lateral (SSC). Aumentar o limite até que não FITC células nos controles negativos podem ser visualizados pelo visor de aquisição FITC-SSC.
- Executar amostras sem adição de controle e examinar o padrão de distribuição de 10.000 células com base em FITC e aquisição SSC. Definir uma porta para colocar todas as células e designá-los como "células de baixa intensidade" (LIs).
- Run-DOC alterado amostras e analisar o padrão de distribuição de 10.000 células com base em FITC e SSC. Algumas células irão aparecer na porta LI predefinido. Definir um outro portão para colocar as células que têm maior intensidade de fluorescência (HIS) do que LIs. Adquirir as estatísticas para ver a abundância relativa de células fechadas.
- Ordenar HI células em tubos de coleta contendo 500 mL PBS em "1 gota purificar" o modo. Terminar a classificação quando o número dos seus atinge 500.000 contagens.
5. Filtro de amplificação por PCR do 16S rRNA genes
Procedimentos filtro de PCR são modificados a partir Kirchman et al 6.
- Filtro de células classificadas em um branco, 0,22 mM de poros de tamanho, 25 mm de diâmetro, filtro de membrana de policarbonato. Corte a borda do filtro que não contém células bacterianas usando uma lâmina estéril. Fatia do filtro em 8 pedaços iguais de tamanho.
- Coloque um pedaço do filtro único em um tubo de reação de PCR, com as células virado do tubo. Adicionar 45 mL de água grau PCR em tubo de reação de PCR. Submergir o filtro totalmente na água.
- Adicione 2 ROCHE illustra PuReTaq Read-To-Go Beads PCR em tubo de reação de PCR, vortex por alguns instantes. Adicione 2 mL cada um dos frente e verso 16S rRNA gene primers (0,4 mM concentração final para cada primer), como 27F e 1492R 7, em cada um dos tubos de reação de PCR.
- (Opcional) Adicione 1μl solução de soro albumina bovina (BSA, a concentração final é 30μg/100 mL) à mistura de reação de PCR para ajudar a inibidores de amplificação adsorver. Se alguém opta por não adicionar BSA, adicione 1μl de água em seu lugar.
- Realizar a amplificação em um termociclador. Um toque para baixo programa de PCR é recomendado, que tem a temperatura de recozimento em seqüência decrescente 62-52 ° C por 1 ° C por ciclo para 11 ciclos, seguidos de 15 ciclos com temperatura de anelamento de 52 ° C. Todos os ciclos incluem desnaturação (a 95 ° C), anelamento (a 62-52 ° C) e extensão (a 72 ° C) medidas de duração 50s. Uma desnaturação 3 min-inicial e etapa de extensão final de 10 min também está incluída no programa de PCR.
- Confirmar a amplificação PCR por eletroforese em um brometo de etídio manchada de 1% agarose gel. Excise o PCR amplicons do gel e limpa com o kit de extração QIAGEN QIAquick gel.
- Realizar dois adicionais amplificações PCR para cada amostra, cada vez que utilizar uma nova secção do filtro. Após PCR gel purificatina, o pool de amplicons da mesma amostra juntos. Amplicons purificados 16S rDNA está agora pronto para uma série de análises moleculares que permitem a identificação taxonômica, como a construção clone biblioteca e seqüenciamento.
6. Resultados representativos:
Resultados representativos são descritos através de um estudo de putrescina de degradação de bactérias como um exemplo. Amostras de água foram coletadas de um site costeira da Geórgia e processadas seguindo os procedimentos descritos acima. Análise FACS revelou que além putrescina induzida desenvolvimento de um grupo de bactérias com intensidade de fluorescência FITC alta, indicando alta taxa de incorporação BrdU (Figura 1). Essas células foram designadas como de alto BrdU incorporação de células (HIS) e era esperado que contêm principalmente putrescina de degradação bactérias. Seu estavam faltando nos controles não-adição, que só continha células com menores níveis de BrdU incorporação (LIs). LIs se esperava que contêm principalmente bacterioplâncton que foram incapazes de usar putrescina acrescentou. Sua foram classificados em tubos separados e depois coletados em filtros de membrana. 16S rRNA gene amplicons foram obtidas para células de alta HI usando filtro de PCR.
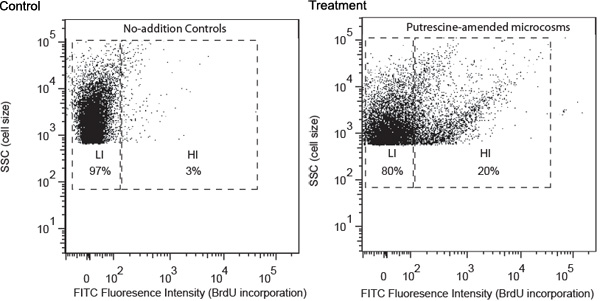
Figura 1. Análises por citometria de fluxo sem adição de controle e modelo composto-alterada (putrescina como um exemplo aqui) amostras coletadas após 24 h de incubação. Análise da distribuição das células foi baseada em (1) intensidade de fluorescência de FITC rotulagem (eixo-x), que é positivamente relacionada com o nível de incorporação de BrdU, e (2) dispersão lateral (SSC, y-eixo), que é positivamente relacionada com a célula tamanho. Notação portão é baseada no nível de BrdU incorporação, (HI, high-BrdU incorporação; LI, baixa incorporação BrdU). A percentagem relativa de HI e células LI são apresentados na portões correspondente.
Access restricted. Please log in or start a trial to view this content.
Discussão
Casais a nossa abordagem incorporação de BrdU, FACS e 16S rDNA para permitir a análise em nível de espécie de identificação do bacterioplâncton que metabolizam os componentes individuais DOC em ambientes aquáticos. O ensaio de incorporação BrdU rótulos células bacterianas com base em atividades metabólicas, que permite a análise somente em bactérias ativas e, portanto, não inclui as células adormecidas. Em nossa abordagem, a incorporação é BrdU em bactérias in situ immunodetected ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Financiamento deste projeto foi fornecido pelo National Science Foundation concede OCE1029607 (para XM) e MCB0702125 (para MAM) eo Gordon e Betty Moore Foundation (a MAM).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments | |
| Nome | Tipo | Companhia | Número de catálogo | Comentários |
| BrdU | Reagente | Sigma | B5002-5G | |
| Lisozima | Reagente | Sigma | L6876-5G | |
| Proteinase K | Reagente | Sigma | P2308-25mg | |
| In Situ Proliferação Celular Kit, FLUOS | Conjunto | Roche | 11810740001 | Consumir mais de Anti-BrdU-FLUOS e tampão de incubação por reação do que o sugerido pelo fabricante. |
| Quadro Seal-Chambers Incubação | Material | Bio-Rad | SLF-1201 | |
| Filtros de membrana de policarbonato (142 mm de diâmetro, 1,0 mM de poros de tamanho) | Material | Millipore | FALP14250 | |
| Filtros de membrana de policarbonato (25 mm de diâmetro, 0,2 m de poros de tamanho) | Material | Millipore | FGLP02500 | |
| illustra PuReTaq Ready-To-Go PCR Beads | Conjunto | GE Healthcare | 27-9559-01 | |
| QIAquick extração de gel kit | Conjunto | QIAGEN | 28704 | |
| FailSafe PCR System | Conjunto | Epicentro | FS99060 |
Referências
- Pernthaler, A., Pernthaler, J., Schattenhofer, M., Amann, R. Identification of DNA-synthesizing bacterial cells in coastal North Sea plankton. Appl. Environ. Microbiol. 68, 5728-5728 (2002).
- Urbach, E., Vergin, K. L., Giovannoni, S. J. Immunochemical detection and isolation of DNA from metabolically active bacteria. Appl. Environ. Microbiol. 65, 1207-12 (1999).
- Mou, X. Z., Hodson, R. E., Moran, M. A. Bacterioplankton assemblages transforming dissolved organic compounds in coastal seawater. Environ. Microbiol. 9, 2025-2025 (2007).
- Hodson, R. E., Dustman, W. A., Garg, R. P., Moran, M. A. In situ PCR for visualization of microscale distribution of specific genes and gene products in prokaryotic communities. Appl. Environ. Microbiol. 61, 4074-4074 (1995).
- Dinjens, W. N. Bromodeoxyuridine (BrdU) immunocytochemistry by exonuclease III (Exo III) digestion. Histochemistry. 98, 199-199 (1992).
- Kirchman, D. L., Yu, L. Y., Fuchs, B. M., Amann, R. Structure of bacterial communities in aquatic systems as revealed by filter PCR. Aquat. Microb. Ecol. 26, 13-13 (2001).
- Delong, E. F., Wickham, G. S., Pace, N. R. Phylogenetic stains: ribosomal RNA-based probes for the identification of single cells. Science. 243, 1360-1360 (1989).
- Artursson, V., Jansson, J. K. Use of bromodeoxyuridine immunocapture to identify active bacteria associated with arbuscular mycorrhizal hyphae. Appl. Environ. Microbiol. 69, 6208-6208 (2003).
- Mou, X. Z. Bacterial carbon processing by generalist species in the coastal ocean. Nature. 451, 708-708 (2008).
- Mou, X. Flow-cytometric cell sorting and subsequent molecular analyses for culture-independent identification of bacterioplankton involved in dimethylsulfoniopropionate transformations. Appl. Environ. Microbiol. 71, 1405-1405 (2005).
- Dean, F. B. Comprehensive human genome amplification using multiple displacement amplification. Proc. Natl. Acad. Sci. U.S.A. 99, 5261-5261 (2002).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados