È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Bromodeossiuridina (BrdU) Etichettatura e successive ordinamento fluorescenza cellulare attivo per la cultura indipendente Identificazione di carbonio organico disciolto-degradanti batterioplancton
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Batterioplancton ambientali vengono incubate con un modello di carbonio organico disciolto (DOC) e un reattivo composto di etichettatura del DNA, bromodeossiuridina (BrdU). In seguito, DOC-degradante celle sono separate dalla comunità di massa in base alla loro elevata incorporazione di BrdU usando fluorescenza separazione delle cellule attivate (FACS). Queste cellule vengono poi identificati da successive analisi molecolari.
Abstract
I microbi sono agenti principali mediare la degradazione di numerosi carbonio organico disciolto (DOC) substrati in ambienti acquatici. Tuttavia, l'identificazione di taxa batterici che trasformano piscine specifiche di DOC in natura rappresenta una sfida tecnica.
Qui si descrive un approccio che le coppie bromodeossiuridina (BrdU) l'incorporazione, fluorescenza cell sorting attivato (FACS) e 16S rRNA gene-based analisi molecolare che consente di cultura indipendente identificazione di batterioplancton in grado di degradare un composto DOC specifico in ambienti acquatici. Microcosmi batterioplancton tre esemplari sono impostati per ricevere sia BrdU e un composto modello DOC (DOC emendamenti), oppure solo BrdU (senza aggiunta di controllo). Sostituti BrdU le posizioni di timidina nel DNA di nuova sintesi batterica e BrdU marcato DNA può essere facilmente immunodetected 1,2. Attraverso una 24-ore di incubazione, batterioplancton che sono in grado di utilizzare il composto aggiunto DOC dovrebbero essere selettivamente attivato, e quindi hanno più alti livelli di incorporazione di BrdU (cellule HI) che non rispondono le cellule negli emendamenti DOC e le cellule nel no- Inoltre controlla (basso cellule incorporazione di BrdU, le cellule LI). Dopo immunolocalizzazione fluorescenza, le cellule si distinguono HI e fisicamente separato dalle cellule LI da fluorescenza separazione delle cellule attivate (FACS) 3. Ordinati DOC-responsive cellule (cellule HI) vengono estratte per il DNA e tassonomicamente individuate attraverso successive rRNA 16S a base genetica tra cui analisi di PCR, la costruzione della libreria clone e sequenziamento.
Protocollo
1. Acqua esempio per l'elaborazione
- Filtrare l'acqua 10L ambientale attraverso 1 micron-pori di dimensioni filtri a membrana per rimuovere le particelle più grandi e bacteriovores. Raccogliere il filtrato l'acqua in una damigiana.
- Trasferire 36 ml ciascuno filtrato in 3 provette sterili Eppendorf (50 ml) contenente 4 ml di soluzione appena preparata paraformaldeide (PFA; 10%). Incubare per 2 ore a temperatura ambiente per preservare le cellule. Raccogliere le cellule su 0,22-micron-pori di dimensioni filtri a membrana bianca per filtrazione a vuoto. Lavare il filtro facendo passare 10 ml di soluzione salina tampone fosfato (PBS) attraverso il filtro per filtrazione a vuoto. Etichetta i filtri come controlli negativi e conservarli a -20 ° C.
Passi 1,3-1,4 sono opzionali per stabilire DOC-limitata condizioni.
- Aggiungere una miscela di azoto inorganico e fosforo (5 mM NH 4 Cl, 5 mM NaNO 3 e 1μM NaH 2 PO 4 concentrazione finale) nella damigiana.
- Incubare al buio a temperatura in situ per 48 ore con agitazioni occasionali.
2. Microcosmo creazione e l'incubazione
- Riempire ogni sei 1-L vasi in vetro con 800 ml di acqua campione dalla damigiana di passo da 1,4 a stabilire microcosmi. Aggiungi BrdU (10 micron, concentrazione finale) per ogni microcosmo. Mescolare bene.
- Aggiungere 1 ml di modello DOC soluzione composta in tre dei microcosmi, queste modifiche serviranno come DOC. Aggiungere 1 ml di PBS sterile nei tre microcosmi restanti, queste serviranno come no-Oltre controlli.
- Incubare tutte le microcosmi in un agitatore incubatore e incubare al buio a temperatura in situ agitando a 100 giri al minuto.
- Raccogliere 36 ml di campione d'acqua da ogni microcosmo e trasferire 50 ml in provette sterili Eppendorf in momenti di 0, 8, 16 e 24 ore. Immediatamente aggiungere 4 ml di fresca PFA (10%) ai tubi di raccolta e incubare per 2 ore a temperatura ambiente per preservare le cellule.
- Filtro conservate cellule attraverso 0.22-micron-pori di dimensioni filtri a membrana in policarbonato. Lavare i filtri con 10 ml di PBS. Procedere immediatamente alla fase successiva o conservare i filtri a -20 ° C.
3. Immunolocalizzazione in situ per incorporazione di BrdU
- Scongelare i filtri (campioni DOC modificato, senza aggiunta controlli e controlli negativi) a temperatura ambiente.
- Applicare 1 ml di soluzione di lisozima [10 mg / ml di lisozima albume in 100 mM Tris, 50 nM EDTA (pH = 8)] 4 per coprire le cellule batteriche sul filtro. Incubare a temperatura ambiente per 30 minuti. Lavare il filtro facendo passare 10 ml di PBS attraverso di essa sotto aspirazione.
- Aggiungere 1 ml di soluzione di proteinasi K [2 mg / ml di proteinasi K in 100 mM Tris, 50 nM EDTA (pH = 8)] 4 per coprire le cellule batteriche sul filtro. Incubare a temperatura ambiente per 30 minuti. Lavare il filtro facendo passare 10 ml di PBS attraverso di essa sotto aspirazione.
- Aggiungere 1 ml di soluzione esonucleasi [esonucleasi III (50 U / ml) in 5 mM MgCl 2 e 50 mM Tris-HCl] 5 per coprire le cellule batteriche sul filtro. Incubare a 37 ° C per 30 minuti. Lavare il filtro facendo passare 10 ml di PBS attraverso di essa sotto aspirazione.
- Montare frame-sigillo camere di incubazione (Bio-Rad) secondo le istruzioni del produttore.
- Tagliate il filtro in ottavi con una lama sterile su un alcool-superficie asciutta e pulita.
- Utilizzando pinze, posto una sezione ottavo di filtro montato in un quadro di tenuta della camera di incubazione (otto frame-sigillo camere sono necessari per ogni campione filtro). Il lato della sezione di filtro che contiene cellule deve essere rivolto verso l'alto. L'umidità nella parte posteriore del filtro permetterà il bastone filtro alla diapositiva. Se il filtro si secchi, applicare una piccola goccia (2 mL) di DIH 2 O nel centro della camera prima di mettere la sezione filtro sul vetrino.
Passi 3,8-3,20 utilizzare i reagenti dalla ROCHE In Situ Kit proliferazione cellulare, FLUOS seguenti procedure modificate dalle istruzioni del produttore. Fatta eccezione per la PBS, tutti i reagenti sono forniti nel kit.
- Applicare tampone abbastanza incubazione (0,5% di siero bovino albume, 0,1% Tween20 in PBS,) per coprire in modo uniforme l'intera sezione del filtro nella camera di incubazione senza causare overflow di una volta la guarnizione viene applicata.
- Inserire una struttura di copertura in poliestere sopra la struttura della camera di incubazione. Premere verso il basso per sigillare ermeticamente la camera di incubazione. Evitare bolle d'aria al di sopra del filtro. Incubare le camere sigillate per 10 minuti a temperatura ambiente al buio.
- Rimuovere il sigillo di poliestere e aprire le camere di incubazione. (Nota: l'apertura delle camere a volte tirerà su il sigillo telaio dalla diapositiva In questo caso, preparare una nuova camera per i seguenti passaggi..)
- Pipettare il buffer di incubazione da un angolo della camera. Evitare il contatto con il filtro.
- Lavare il filtro pipettando gentilmente 100 l di PBS efuori dalla camera di 3 volte.
- Preparare anti-BrdU-FLUOS soluzione di lavoro seguendo la procedura raccomandata dal produttore immediatamente prima dell'uso.
- Pipettare 120 microlitri anti-BrdU-FLUOS soluzione di lavoro sul filtro, avendo cura di coprire uniformemente tutta la superficie.
- Richiudere la camera con una copertura in poliestere nuovo. Evitare bolle d'aria sulla parte superiore del filtro. Incubare la camera al buio a 37 ° C per 3 ore. Questo passo etichetta BrdU DNA incorporato in situ con isotiocianato di fluorescina (FITC).
- Rimuovere la copertura in poliestere e aprire la camera di incubazione. Pipettare il anti-BrdU-FLUOS soluzione di lavoro. Lavare il filtro 3 volte con PBS.
- Trasferire il filtro dalla camera di incubazione per una superficie sterile. Tagliate la sezione filtro in piccoli pezzi con una lama sterile.
- Trasferire il filtro in 2 pezzi microprovette ml, contenenti ciascuno 1 ml di PBS. Strettamente tappo del tubo e sigillare con parafilm. Incubare a 37 ° C e 200 rpm per 10 min.
- Fissare i tubi in un vortice vortex e alla velocità massima per 5 minuti. Pipetta il sopranatante in una provetta sterile 15 ml Eppendorf. Ripetere le fasi di incubazione e vortex per 5 volte di più. Combina il surnatante nello stesso tubo 15 ml Eppendorf per ogni campione. Tipicamente, l'80% di cellule possono essere recuperati in sospensione.
- Conservare il supernatante con le cellule risospese a 4 ° C. Ordina entro 2 giorni.
4. FACS analisi
Una procedura per un citometro di flusso BD FACSAria e relativo software è descritto qui.
- Ottimizzare l'impostazione delle fasi del citometro a flusso seguito consigliato dal produttore. Ciò comporta: regolando il controllo di amplificazione per impostare i parametri richiesti e breakoff lo sweet spot, ottimizzando il ritardo laser e fattori di scala area per la pressione guaina esperimento e ottimizzare le impostazioni per FSC e tensioni SSC, soglia FSC, FSC scala fluorescenza, fluorescenza tensioni PMT , ecc
- Esegui campioni di controllo negativo sul citometro a flusso (FCM) sulla base di intensità di fluorescenza di dispersione FITC e laterali (SSC). Aumentare la soglia fino a quando non FITC cellule nei controlli negativi possono essere visualizzati dal FITC-SSC visualizzazione acquisizione.
- Eseguire senza aggiunta campioni di controllo e esaminare il modello di distribuzione di 10.000 celle in base a FITC e acquisizione SSC. Definire un cancello di racchiudere tutte le cellule e designare come "celle a bassa intensità" (LIS).
- Esegui DOC-modificato campioni ed esaminare il modello di distribuzione di 10.000 celle in base a FITC e SSC. Alcune cellule apparirà nel cancello preset LI. Definire un altro cancello per racchiudere le cellule che hanno una maggiore intensità di fluorescenza (HIS) di LIS. Acquisire le statistiche per vedere la relativa abbondanza di cellule gated.
- Celle di tipo HI in provette contenenti 500 microlitri PBS a "purificare 1 goccia" modalità. Termina ordinamento quando il numero dei suoi raggiunge 500.000 conta.
5. Filtro amplificazione di geni rRNA 16S
Filtro procedure di PCR vengono modificati da Kirchman et al 6.
- Filtro cellule ordinati su un bianco, 0,22 micron-pori di dimensioni, 25 mm di diametro, in policarbonato membrana del filtro. Tagliare il bordo del filtro che contiene le cellule batteriche con una lama sterile. Tagliate il filtro in 8 pezzi uguali dimensioni.
- Posizionare un pezzo unico filtro in un tubo di reazione di PCR, con le cellule rivolti verso l'interno del tubo. Aggiungere 45 microlitri di acqua grado di PCR nel tubo di reazione PCR. Immergere il filtro completamente in acqua.
- Aggiungere 2 ROCHE illustrazioni PuReTaq Read-To-Go PCR Beads nel tubo di reazione PCR, mescolare brevemente nel vortex. Aggiungere 2 microlitri ciascuno degli avanti e indietro primer gene rRNA 16S (0,4 microM concentrazione finale per ciascun primer), come 27F e 1492R 7, in ciascuna delle provette di reazione della PCR.
- (Facoltativo) Aggiungere 1ml soluzione di albumina sierica bovina (BSA, concentrazione finale è 30μg/100 microlitri) alla miscela di reazione PCR per aiutare inibitori dell'amplificazione assorbire. Se uno sceglie di non aggiungere BSA, aggiungere 1ml di acqua, invece.
- Eseguire la PCR su un termociclatore. Un tocco giù programma PCR si raccomanda, che è la temperatura di ricottura sequenzialmente decrescenti 62-52 ° C di 1 ° C per ciclo per 11 cicli, seguiti da 15 cicli con temperatura di annealing di 52 ° C. Tutti i cicli sono denaturazione (a 95 ° C), ricottura (a 62 a 52 ° C), ed estensione (a 72 ° C) le misure di durata 50 anni. Un primo 3-min di denaturazione e una finale di 10 minuti passo estensione è inoltre inclusa nel programma PCR.
- Conferma PCR mediante elettroforesi su un bromuro macchiato etidio 1% gel. Dell'accisa gli ampliconi della PCR dal gel e pulire con l'estrazione QIAGEN QIAquick kit gel.
- Eseguire due ulteriori amplificazioni PCR per ogni campione, ogni volta che utilizzano una nuova sezione filtro. Dopo la PCR gel purificatiin poi, la piscina ampliconi del campione stesso insieme. Purificata 16S rDNA ampliconi sono ora pronti per una serie di analisi molecolari che permettono l'identificazione tassonomica, come la costruzione clone biblioteca e sequenziamento.
6. Rappresentante dei risultati:
I risultati rappresentativi sono descritte utilizzando uno studio di putrescina-degradante batteri come esempio. Campioni di acqua sono stati raccolti in un sito costiero della Georgia e trattati seguendo le procedure sopra descritte. FACS analisi ha rivelato che oltre putrescina indotto lo sviluppo di un gruppo di batteri ad alta intensità FITC fluorescenza, indicando alto tasso di incorporazione di BrdU (Figura 1). Queste cellule sono state designate come high-BrdU-incorporazione cellule (HIS) e ci si aspettava che contengono principalmente putrescina-degradanti batteri. Il suo mancavano nel no-aggiunta controlli, che conteneva solo le cellule con livelli inferiori di incorporazione di BrdU (LIS). LI ci si aspettava che contengono principalmente batterioplancton che sono stati in grado di utilizzare putrescina aggiunto. I suoi sono stati ordinati in tubi separati e poi raccolto su filtri a membrana. Ampliconi gene 16S rRNA sono stati ottenuti per le celle ad alta HI con filtro PCR.
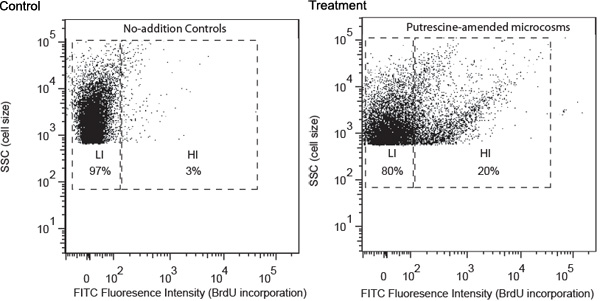
Figura 1. Analisi citofluorimetrica la mancata aggiunta di controllo e il modello-compound-modificato (putrescina ad esempio qui) i campioni raccolti dopo 24 ore di incubazione. Analisi della distribuzione delle cellule si è basata su (1) intensità di fluorescenza del FITC etichettatura (asse x), che è positivamente correlata al livello di incorporazione di BrdU, e (2) side scatter (SSC, asse y), che è positivamente correlato alla cella dimensioni. Notazione cancello si basa sul livello di incorporazione di BrdU, (HI, high-BrdU-incorporazione, LI, basso BrdU-incorporazione). La percentuale relativa di HI e cellule LI sono riportati nella cancelli corrispondente.
Access restricted. Please log in or start a trial to view this content.
Discussione
Il nostro approccio coppie BrdU incorporazione, FACS analisi e 16S rDNA per consentire l'identificazione delle specie a livello di batterioplancton che metabolizzano i singoli componenti DOC in ambienti acquatici. Il saggio di incorporazione di BrdU etichette cellule batteriche sulla base di attività metabolica, che permette l'analisi solo sui batteri attivi e quindi non include le cellule dormienti. Nel nostro approccio, incorporazione di BrdU è nei batteri in situ immunodetected e che hanno...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Il finanziamento di questo progetto è stato fornito dal National Science Foundation concede OCE1029607 (a XM) e MCB0702125 (a MAM) e Gordon and Betty Moore Foundation (a MAM).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments | |
| Nome | Tipo | Azienda | Numero di catalogo | Commenti |
| BrdU | Reagente | Sigma | B5002-5G | |
| Lisozima | Reagente | Sigma | L6876-5G | |
| Proteinasi K | Reagente | Sigma | P2308-25mg | |
| In Situ Kit proliferazione delle cellule, FLUOS | Kit | Roche | 11810740001 | Consumano più di Anti-BrdU-FLUOS e buffer di incubazione per reazione rispetto a quanto suggerito dal produttore. |
| Frame-Seal incubazione Chambers | Materiale | Bio-Rad | SLF-1201 | |
| I filtri a membrana in policarbonato (142 mm di diametro, 1,0 micron-poro-size) | Materiale | Millipore | FALP14250 | |
| I filtri a membrana in policarbonato (25-mm di diametro, 0,2 micron a poro-size) | Materiale | Millipore | FGLP02500 | |
| illustrazioni PuReTaq Ready-To-Go PCR Beads | Kit | GE Healthcare | 27-9559-01 | |
| QIAquick gel kit di estrazione | Kit | QIAGEN | 28704 | |
| FailSafe PCR System | Kit | Epicentre | FS99060 |
Riferimenti
- Pernthaler, A., Pernthaler, J., Schattenhofer, M., Amann, R. Identification of DNA-synthesizing bacterial cells in coastal North Sea plankton. Appl. Environ. Microbiol. 68, 5728-5728 (2002).
- Urbach, E., Vergin, K. L., Giovannoni, S. J. Immunochemical detection and isolation of DNA from metabolically active bacteria. Appl. Environ. Microbiol. 65, 1207-12 (1999).
- Mou, X. Z., Hodson, R. E., Moran, M. A. Bacterioplankton assemblages transforming dissolved organic compounds in coastal seawater. Environ. Microbiol. 9, 2025-2025 (2007).
- Hodson, R. E., Dustman, W. A., Garg, R. P., Moran, M. A. In situ PCR for visualization of microscale distribution of specific genes and gene products in prokaryotic communities. Appl. Environ. Microbiol. 61, 4074-4074 (1995).
- Dinjens, W. N. Bromodeoxyuridine (BrdU) immunocytochemistry by exonuclease III (Exo III) digestion. Histochemistry. 98, 199-199 (1992).
- Kirchman, D. L., Yu, L. Y., Fuchs, B. M., Amann, R. Structure of bacterial communities in aquatic systems as revealed by filter PCR. Aquat. Microb. Ecol. 26, 13-13 (2001).
- Delong, E. F., Wickham, G. S., Pace, N. R. Phylogenetic stains: ribosomal RNA-based probes for the identification of single cells. Science. 243, 1360-1360 (1989).
- Artursson, V., Jansson, J. K. Use of bromodeoxyuridine immunocapture to identify active bacteria associated with arbuscular mycorrhizal hyphae. Appl. Environ. Microbiol. 69, 6208-6208 (2003).
- Mou, X. Z. Bacterial carbon processing by generalist species in the coastal ocean. Nature. 451, 708-708 (2008).
- Mou, X. Flow-cytometric cell sorting and subsequent molecular analyses for culture-independent identification of bacterioplankton involved in dimethylsulfoniopropionate transformations. Appl. Environ. Microbiol. 71, 1405-1405 (2005).
- Dean, F. B. Comprehensive human genome amplification using multiple displacement amplification. Proc. Natl. Acad. Sci. U.S.A. 99, 5261-5261 (2002).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon