Method Article
Um Método de Extração Fracionada Simples para a Análise Abrangente de Metabolitos, Lípidos e Proteínas de uma única amostra
Neste Artigo
Resumo
É apresentado um protocolo para extração abrangente de lipídios, metabólitos e proteínas de tecidos biológicos usando uma amostra.
Resumo
A compreensão de sistemas biológicos complexos requer a medição, análise e integração de múltiplas classes de compostos da célula viva, geralmente determinadas por medidas transcriptômicas, proteômicas, metabolômicas e lipidômicas. Neste protocolo, introduzimos um método simples para a extração reprodutível de metabólitos, lipídios e proteínas a partir de tecidos biológicos usando uma alíquota única por amostra. O método de extração é baseado em um sistema de metil terc -butil éter: metanol: água para separação líquida: líquida de metabolitos hidrofóbicos e polares em duas fases não miscíveis, juntamente com a precipitação de proteínas e outras macromoléculas como uma pelota sólida. Este método, portanto, fornece três frações diferentes de composição molecular específica, que são totalmente compatíveis com tecnologias comuns de alto teor de débito, tais como cromatografia líquida (LC) ou cromatografia gasosa (GC) acoplada a espectrômetros de massa. Mesmo que o método fosse initDesenvolvido para a análise de diferentes amostras de tecidos vegetais, provou ser totalmente compatível para a extração e análise de amostras biológicas de sistemas tão diversos como algas, insetos e tecidos de mamíferos e culturas celulares.
Introdução
A biologia dos sistemas, que surgiu em meados do século passado 1 e foi avançada pela análise em larga escala dos conjuntos de dados genômicos e transcriptômicos 2 , 3 , tornou-se uma abordagem nova e indispensável para a análise de sistemas biológicos complexos 4 , 5 . O objetivo principal da biologia de sistemas é decifrar as interações e dependências dos componentes em sistemas biológicos e para colmatar o vínculo entre genótipos, sua realização, transformações moleculares e fenótipos resultantes. Por conseguinte, a integração de conjuntos de dados abrangentes, produzidos pelas várias abordagens analíticas de grande escala, a saber, genômica, transcriptômica, metabolômica, lipidômica e proteômica e sua análise computacional tornou-se um pré-requisito para a descrição e compreensão de sistemas biológicos complexos.
Bas Com base na enorme diversidade química e na complexidade dos componentes biológicos em qualquer sistema vivo, a produção de conjuntos de dados "omísticos" amplos e abrangentes depende fortemente da qualidade do método de extração aplicada 9 . Além da qualidade do método de extração, a economia do método é importante; Isso significa que seria desejável obter a maior quantidade de informações moleculares a partir da menor entrada de amostra possível. Muitas vezes, as quantidades de amostra podem ser limitantes e, portanto, é altamente desejável fazer uso de um método de extração, que pode derivar tantas classes moleculares de uma única extração de uma determinada amostra. Isto significa que, em vez de utilizar vários métodos de extração especializados para a extração de diferentes classes de compostos de diferentes alíquotas de amostra da mesma amostra, emprega-se um método de extração seqüencial, que fracciona os constituintes moleculares de uma única alíquota em diferentes frações moleculares.
_content "> O método comum empregado para esses métodos de extração fracionada baseia-se no método de extração de lipídios de duas fases de Folch et al., Desenvolvido em 1957 10. Este método é baseado em uma divisão de clorofórmio / metanol / água de metabolitos polares e hidrofóbicos e foi Destinada a limpar e descomplicar amostras para análises de lipídios de alta qualidade. Com a evolução da biologia de sistemas multi-sistemas, o método de Folch foi ampliado, por etapas, melhorando-o para a partição da amostra de proteínas e metabolitos polares e lipídios para Metabolomics e lipidomics baseados em cromatografia gasosa e líquida de compostos polares e hidrofóbicos, além da proteômica líquida baseada em cromatografia 11 , 12 , 13 , 14. Infelizmente, todos esses métodos dependem de um método de extração baseado em clorofórmio, que não só Leva ao não desejadoA proteína do grânulo de proteína como uma fase intermédia entre a fase polar e lipídica, mas que também é um solvente indesejável a partir de uma perspectiva quimica verde 15 , 16 . No entanto, o solvente éter metil terc -butílico (MTBE) supera ambos os problemas acima mencionados e é uma substituição adequada para o clorofórmio. Com base nesses requisitos, decidimos estabelecer um método de extração de MTBE: metanol: à base de água, que atinja todas as especificações acima mencionadas e, portanto, funciona como um ponto de partida ideal para análises multi-omáticas abrangentes 16 .Este protocolo orienta o usuário passo a passo através do fluxo de trabalho simples, rápido e reprodutível da preparação da amostra, incluindo a solução de problemas comumente observados. Além disso, apresentaremos brevemente dados analíticos exemplares de cromatografia líquida de ultra-desempenho - espectrometria de massa (UPLC-MS) -basEd lipidomics, metabolomics e proteomics a partir de amostras de tecido vegetal. Mesmo que os exemplos fornecidos sejam derivados de 50 mg de uma amostra de tecido de folha de Arabidopsis thaliana , este protocolo foi utilizado para várias outras amostras e tecidos biológicos, incluindo algas 17 , 18 , insetos 19 e células de mamíferos, órgãos e tecido 20 , 21 , 22 . O escopo do protocolo de extração apresentado é fornecer uma descrição clara e detalhada do manuseio da amostra de pré-extração e do próprio procedimento de extração. Embora forneçamos três exemplos breves de aplicação analítica, informações detalhadas sobre o tratamento de dados pré e pós-analítico podem ser obtidas em nossas publicações anteriores 16 , 23 , 24 , 25 , 26 .
Protocolo
Atenção : O metanol (MeOH) e o éter metil terc -butílico (MTBE), utilizados durante a extração, são inflamáveis e podem ter, em exposição prolongada e / ou contato, irritação respiratória, ocular ou cutânea. Por favor, lembre-os com cuidado somente em uma chaminé e use os procedimentos de segurança adequados durante a extração (bata de laboratório, óculos de segurança, luvas, etc. ). O nitrogênio líquido e o gelo seco, usados em várias etapas deste protocolo, podem causar queimaduras graves por contato prolongado com a pele. Manuseie-os com cuidado usando luvas e óculos de proteção. Os usuários podem usar diferentes produtos químicos, reagentes ou padrões internos para análises de amostras, algumas das quais podem ser tóxicas. Por favor, examine as folhas de dados de segurança química relevantes para todo o material utilizado.
1. Coleta e colheita de amostras biológicas
- Prepare tubos de colheita rotulados.
NOTA: Aqui, colete amostras biológicas em rotulado, 2 mL, fundo redondo e seguro.K tubos de microcentrífuga contendo duas bolas metálicas de 5 mm de diâmetro para o homogeneizador de tecido. - Prepare um engo de nitrogênio líquido cheio.
- Colhe a amostra biológica e encaixe congelar o tecido em nitrogênio líquido. Execute este passo o mais rápido possível dentro de alguns segundos para evitar mudanças metabólicas induzidas por feridas.
Nota: Para fins de demonstração, use folhas de roseta de Arabidopsis thaliana (Col-0) de tipo selvagem de 30 dias cultivadas em solo em condições de longo dia. - Mantenha as amostras colhidas em gelo seco para quebras de curto prazo ou guarde-as a -80 ° C por períodos mais longos.
2. Moagem e ruptura dos tecidos
- Pré-arrefecer os suportes do tubo do homogeneizador de tecido em nitrogênio líquido durante pelo menos 10 min. Se um homogeneizador de tecido não estiver disponível, use argamassas e pilões limpos e pré-arrefecidos.
- Pegue as amostras do nitrogênio líquido, gelo seco ou congelador de -80 ° C e coloque-as no pré-arrefecidoSuportes de tubos.
- Coloque rapidamente os suportes do tubo no homogeneizador de tecido.
- Moer o material biológico em um pó fino e homogêneo. Use 20 Hz durante 1 min para as folhas.
Nota; O tempo e a velocidade de homogeneização podem ser variados dependendo do tecido, certifique-se de que a amostra é homogeneizada em pó fino e que este pó é mantido congelado em cada passo da homogeneização. - Pegue as amostras biológicas dos suportes dos tubos e mantenha-os congelados até extração.
3. Pesagem de tecidos
- Use um balanço analítico com precisão suficiente para as quantidades de amostra necessárias.
- Prepare um tubo de microcentrífuga com retenção segura com fundo redondo de 2 mL rotulado.
- Pré-arrefecer os tubos e espátulas em nitrogênio líquido.
- Alíquota a quantidade necessária de tecido em pó no tubo de microcentrífuga de segurança segura de 2 mL.
Cuidado: Evite qualquer descongelamento do material da planta minimizando o tempo necessário paraOitenta das amostras. - Retorne as amostras aliquotadas imediatamente após a pesagem no nitrogênio líquido.
- Registre para cada amostra o peso exato. Use 10-50 mg ± 10% para a maioria dos tecidos vegetais.
- Armazene as amostras alíquotas a -80 ° C até extração adicional.
4. Configuração do Reagente
- Utilizar uma mistura de extracção de éter metil- terc -butílico (MTBE) / metanol (MeOH).
- Para a preparação de 100 mL de solvente de extração, adicione 75 mL de MTBE a 25 mL de MeOH para formar uma mistura de MTBE: MeOH (3: 1, vol / vol).
- Adicione padrões internos para a normalização pós-análise de acordo com as necessidades analíticas. Tipicamente, adicione 50 μL de 1,2-diheptadecanoil- sn -glicero-3-fosfocolina (1 mg / mL em clorofórmio) como padrão interno para a análise lipídica baseada em UPLC-MS, ao mesmo tempo em que adiciona 50 μL de sorbitol 13C ( 1 mg / mL em água) como padrões internos para a análise baseada em GC-MS de primáriasMetabolitos. Os padrões internos para análise de metabólitos baseados em UPLC-MS são 50 μL de corticosterona (1 mg / mL em metanol) e 25 μL de ampicilina (1 mg / mL em metanol).
- Transfira o solvente para uma garrafa de vidro limpa que foi enxaguada com mistura MTBE: MeOH.
- Armazene a mistura de extração por até 1 semana a 4 ° C.
NOTA: Não armazene a mistura de extração por períodos mais longos para manter resultados reprodutíveis.
- Para induzir a separação de fases, use água (H 2 O) / metanol (MeOH).
- Para a preparação de 100 ml de H2O: MeOH, adicionar 75 mL de H2O a 25 mL de MeOH para preparar H2O: MeOH (3: 1, vol / vol).
- Transfira o solvente para uma garrafa de vidro limpa que foi enxaguada com mistura de H 2 O: MeOH. Este solvente pode ser armazenado durante várias semanas à temperatura ambiente.
5. Extração de amostras
- Pré-arrefecer a extração mIxture (MTBE: MeOH, 3: 1, vol / vol) a -20 ° C utilizando um sistema de arrefecimento líquido ou um congelador de -20 ° C.
- Retire as amostras alíquotas uma a uma e adicione 1 mL da mistura de extração pré-arrefecida a cada tubo de amostra. Cuidado: Execute este passo rapidamente devido à baixa viscosidade do MTBE.
- Misture imediatamente em um misturador de vórtice até o tecido ser bem homogeneizado dentro da mistura de extração.
NOTA: Este passo é muito importante, uma vez que é necessário precipitar as proteínas e inativa suas atividades enzimáticas. - Incubar todas as amostras em um agitador orbital a 100 rpm durante 45 min a 4 ° C.
- Sonicate as amostras por 15 minutos num banho de sonicação arrefecido com gelo.
6. Fracionamento por Separação de Fases
- Adicionar 650 μL de H 2 O: MeOH (3: 1, vol / vol) a cada tubo de amostra.
- Misture bem por vortexing por 1 min.
- Centrifugue as amostras a uma velocidade de 20 000 x g durante 5 minutos a 4 ° C.
NOTA: Após este passo, existem duas fases líquidas imiscíveis com uma pelota sólida na parte inferior do tubo.
Cuidado: Manuseie os tubos com cuidado para evitar a mistura das duas fases líquidas e evite interromper o sedimento precipitado.
7. Alíquota de Fracções Polares e Hidrofóbicas
- Transfira 500 μL do solvente da fase superior, contendo lipídios, para um tubo de microcentrífuga de 1,5 mL etiquetado.
NOTA: As amostras de lípidos aliquotadas podem ser concentradas diretamente para análise UPLC-MS imediata (passo 8.1) ou armazenadas durante várias semanas a -80 ° C. - Uma vez que os 500 μL são removidos da amostra, remova a fase lipídica restante usando uma pipeta de 200 μL.
- Transfira 400 μL do solvente da fase inferior (metabolitos polares e semi-polares) para um tubo de microcentrífuga de 1,5 mL etiquetado. As amostras polares aliquotadas podem ser concentradas diretamente para análise UPLC-MS imediata (etapa 8.2) ou armazenadas paraVárias semanas a -80 ° C.
- Pegue uma alíquota adicional de 200 μL para realizar análises adicionais, por exemplo, análise de metabólitos à base de cromatografia em gases como descrito de forma precoce 16 .
- Remova o restante da fase aquosa pipeteando o excesso de volume.
- Lave a proteína obtida, o amido, o sedimento de parede celular com 500 μL de metanol, agitando-o completamente durante 1 min.
- Centrifugar as amostras a uma velocidade de 10 000 x g durante 5 min a 4 ° C.
- Execute a extração e a digestão de proteínas (passo 11) ou a análise de parede de amido / célula conforme descrito anteriormente 16 .
NOTA: Se não for utilizado imediatamente, estes pellets podem ser armazenados por várias semanas a -80 ° C.
8. Concentração e Armazenamento de Frações
- Evaporar o solvente das amostras de lípidos (do passo 7.1) num concentrador de vácuo sem aquecimento (durante 1-2 horas) ou de preferência usar um nitrEvaporador de fluxo de ogen para evitar modificações oxidativas dos lipídios.
NOTA: As amostras secas devem ser analisadas imediatamente. Para armazenamento, deixe amostras na solução MTBE, idealmente em frascos de vidro (passo 7.1). - Evaporar o solvente das amostras aquosas (do passo 7.3 ou 7.4) durante a noite num concentrador de vácuo sem aquecimento. NOTA: as amostras secas podem ser armazenadas durante várias semanas a -80 ° C antes da análise.
9. Análise de lipídios usando UPLC-MS 24
- Re-suspender as frações lipídicas secas (do passo 8.1) em 400 μL de acetonitrilo: 2-propanol (7: 3, vol / vol).
- Transfira líquido suficiente para frascos de vidro e tampa firmemente.
- Coloque os frascos de vidro em um amostrador automático arrefecido (4 ° C).
- Injecte 2 μL por amostra e separe os lípidos em uma coluna C 8 de fase reversa (RP) mantida a 60 ° C usando um sistema UPLC com um caudal de 400 μL / min.
- Use o celularFases descritas na Tabela 1 para a separação cromatográfica.
- Adquirir os espectros de massa em modo de ionização positiva e negativa usando um instrumento MS adequado que cobre a faixa de massa entre 150 e 1.500 m / z.
10. Análise de metabólitos polares e semi-polares usando UPLC-MS 25 .
- Re-suspender a fase polar (do passo 8.2) em 200 μL de metanol UPLC: água (1: 1, vol / vol).
- Transfira líquido suficiente para frascos de vidro e tampa firmemente.
- Coloque os frascos de vidro em um amostrador automático arrefecido (4 ° C).
- Injecte 2 μL de cada amostra e separe os metabolitos numa coluna RP C18 mantida a 40 ° C utilizando um sistema UPLC com um caudal de 400 μL / min.
- Use as fases móveis para a separação cromatográfica com os parâmetros indicados na Tabela 2 .
- Adquirir espectros de massa de varredura completa em modo de ionização positiva e negativa nosUm espectrômetro de massa adequado que cobre uma faixa de massa entre 50 e 1.500 m / z.
11. Extração de Proteínas, Digestão e Análise 16
- Re-suspenda a proteína lavada / amido / massa de parede celular (do passo 7.8) em 200 μL do tampão de extração de proteína de escolha. Nota: Utilizamos tampão de ureia / tioureia (ureia 5 M, 2 mM de tioureia, 15 mM de DTT, 2% de CHAPS e protease e inibidores de fosfatase) 27 .
- Sonicate as amostras por 10 minutos em um banho sônico arrefecido com gelo.
- Incubar amostras por 30 min em um agitador orbital (100 rpm) à temperatura ambiente.
- Centrifugar as proteínas dissolvidas a 10 000 x g durante 5 min.
- Recolher o sobrenadante de proteína em um novo tubo.
- Determine a concentração de proteína do sobrenadante coletado 28 .
- Digerir 50 μg de proteína em solução com um protocolo de escolha. Tipicamente, use o acordo de mistura Trypsin / Lys-CDe acordo com o manual de instruções.
- Após a digestão, realize a dessalinização dos péptidos antes da espectrometria de massa usando dicas de estágio C 18 e eluir os péptidos digeridos 29 .
- Concentre as amostras até perto da secura em um concentrador de vácuo sem aquecimento.
- Re-suspender as amostras em tampão de carga apropriado (por exemplo, acetonitrilo a 5%, ácido fórmico a 0,5%) e analisar as misturas de péptidos por LC-MS / MS usando um espectrômetro de massa de alta resolução conectado a um sistema nano LC.
NOTA: No exemplar conjunto de dados de proteômica apresentado neste protocolo, usamos um gradiente conforme descrito na Tabela 3 . - Defina o espectrômetro de massa, utilizando uma estratégia de 15 principais, onde uma varredura completa (FS) foi seguida por até 15 exames MS / MS dependentes de dados, para os seguintes parâmetros: O FS estava na faixa de massa 200 - 2.000 m / z em Uma resolução de 70.000 com um valor alvo de 3x10 6 íons. Obtenha as varreduras MS / MS dependentes de dados pelo nível superiorDissociação colisional rgy (HCD). Defina os valores alvo para os íons MS / MS para 1e 5 , com um tempo máximo de preenchimento de íons de 50 ms, uma janela de isolamento de 4.0 m / z, energia de colisão normalizada (NCE) de 30% e uma relação de enxofre de 1%. Meça os íons MS / MS em uma resolução 17.500 e a exclusão dinâmica foi definida para 60 s.
Resultados
Conjuntos de dados abrangentes multi-omics são inestimáveis para a compreensão de sistemas biológicos complexos. A estratégia para uma experiência biológica bem sucedida geralmente começa a partir de um projeto experimental significativo, configuração e desempenho do experimento, seguido de coleta de amostras, extração, aquisição de dados analíticos, processamento de dados brutos, análise de dados estatísticos, identificação de metabolito relevante e interpretação de dados biológicos Incluindo mapeamento e visualização de via ( Figura 1 ).
No protocolo de extração introduzido aqui, nos concentramos nas etapas de coleta, controle e extração de amostra, que são retratadas na visão detalhada do fluxo de trabalho na Figura 2 . Para fins de demonstração, selecionou-se 50 mg de tecido de Arabidopsis. Este material foi colhido, moído e extraído antes de sujeitá-lo a trêsPlataformas UPLC-MS analíticas exemplares, fornecendo dados que podem ser utilizados para análises lipidômicas, metabolômicas e proteômicas direcionadas e não segmentadas. As Figuras 2 e 6 incluem, adicionalmente, imagens representativas de como, em condições padrão, o solvente de extração deve ser semelhante. Além disso, são apresentados exemplos de amostras contendo quantidades excessivas de macromoléculas precipitadas (proteínas e amido) e amostras com homogeneização inapropriada da amostra ( Figura 3 ). A solução de problemas para esses dois problemas comuns é dada em breve na Figura 3, mas também é discutida com mais detalhes em nossa publicação anterior 16 .
As figuras 4 e 5 descrevem exemplos de três cromatogramas analíticos derivados do lípido, o polar / semMetabolos i-polares e análises de proteínas. Os lipídios, que foram retirados da fase MTBE superior ( Figura 2 ), foram analisados por cromatografia líquida ultra-performance de fase reversa (RP) C 8 acoplada a espectrometria de massa de alta resolução. Os lipídios podem ser adquiridos usando modos de ionização MS positivos e negativos ( Figura 4 , painel superior) 16 , 24 .
Os metabolitos primários e secundários polares e semi-polares foram analisados a partir da fase polar (água / metanol) ( Figura 2 ) pela fase reversa (RP) C 18 UPLC-MS 25 . O método ilustrado, utilizando a cromatografia de fase reversa, é altamente compatível com a análise de metabolitos semi-polares (nomeadamente metabolitos do metabolismo secundário da planta), que podem ser analisados usando modos de ionização positiva e negativa na MS( Figura 4 , painel inferior) 16 . Mais metabolitos hidrofílicos desta fração (açúcares, aminoácidos polares, etc.), que não apresentam boa retenção no material de fase reversa, podem ser analisados por outros métodos analíticos, tais como GC-MS 16 ou cromatografia líquida de interação hidrófila 30 .
As proteínas, que foram recuperadas do sedimento sólido no fundo do tubo de extração ( Figura 2 ), foram digeridas em solução e analisadas usando a arma de tiro LC-MS ( Figura 5 ), enquanto o protocolo para extração de amido e parede celular O material pode ser obtido no nosso protocolo 16 anteriormente publicado.
Em resumo, mais de 200 espécies lipídicas, 50 metabolitos semi-polares anotados e vários milhares de proteídiosNs podem ser rotineiramente identificados a partir de amostras do tipo usado em nosso exemplo. Além disso, o método mostrou ampla aplicabilidade usando diferentes tecidos, órgãos e material de cultura de células ( Figura 6 ).

Figura 1: Fluxo de trabalho geral para Análise Omics não segmentada de grande escala. Clique aqui para ver uma versão maior desta figura.

Figura 2 : Fluxo de trabalho de preparação e extração de amostras para análise de lipídios, metabólitos e proteínas a partir de uma única alíquota de uma amostra biológica.Clique aqui para ver uma versão maior desta figura.
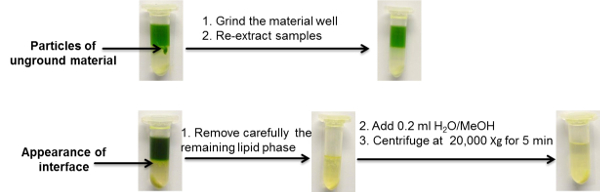
Figura 3 : Exemplos ilustrativos de problemas comumente observados usando protocolos de extração de partição em duas fases. Clique aqui para ver uma versão maior desta figura.
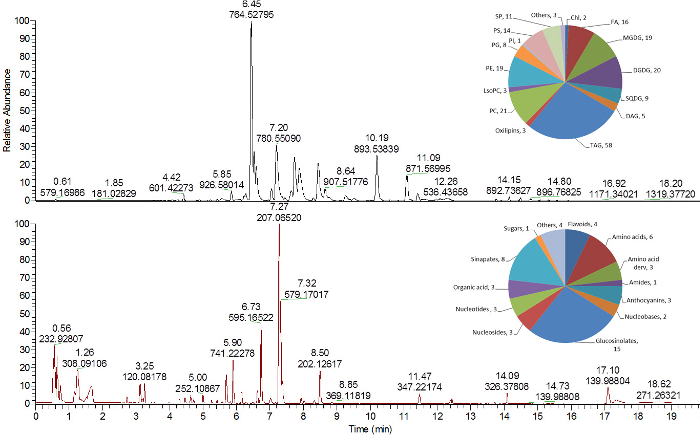
Figura 4 : Cromatogramas representativos de metabólitos lipídicos e semi-polares de extractos de folhas de Arabidopsis Thaliana . Cromatograma de pico baseS de lipídios (painel superior) e metabolitos semi-polares (painel inferior) analisados em modo de ionização positiva 16 . Os gráficos de torta no canto superior direito de cada cromatograma mostram o número de lipídios e metabólitos identificados atribuídos a diferentes classes químicas. Chl, clorofilas; DAG, diacilglicérido; DGDG, digalactosildiacilglicerol; FA, ácido gordo; LysoPC, lisofosfatidilcolina; MGDG, monogalactosildildacilglicerol; PC, fosfatidilcolina; PE, fosfatidiletanolamina; PG, fosfatidilglicerol; PI, fosfatidilinositol; PS, fosfatidilserina; SP, esfingolípido; SQDG, sulfoquinovosyldiacilglicerol; TAG, triacilglicerido. Clique aqui para ver uma versão maior desta figura.
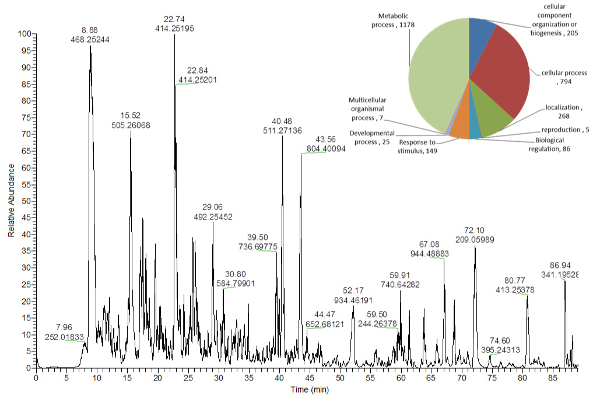
Figura 5 Trong: Cromatograma de Pepitas de Pepitas Representativos de extratos de folhas de Arabidopsis Thaliana .
O gráfico de pizza mostra que no canto superior direito indica o número de proteínas identificadas atribuídas a diferentes processos biológicos 16 . Clique aqui para ver uma versão maior desta figura.

Figura 6 : Exemplos de extração representativa de vários tipos de tecidos de Arabidopsis thaliana de tipo selvagem .
Todas as amostras foram extraídas utilizando 50 mg de peso fresco dos tecidos indicados."> Clique aqui para ver uma versão maior desta figura.
| Tempo (min) | % Buffer A para Buffer B | |
| 0 a 1 min | 45% A | Tampão A: Acetato de NH4 a 1% 1M, ácido acético a 0,1% em água de grau MSLC |
| 1 a 4 minutos | Gradiente linear de 45% A a 25% A | Tampão B: 1% de acetato de NH4 1 M, ácido acético a 0,1% em acetonitrilo / isopropanol 7: 3, (v: v) |
| 4 a 12 min | Gradiente linear de 25% A a 11% A | Caudal 400 μL / min |
| 12 a 15 minutos | Gradiente linear de 11% A a 0% A | Volume de injeção 2 μL |
| 15 a 19,5 min | Lavar a coluna por 4,5 minutos com 0% A | |
| 19,50 a 19,51 min | Retornar para 45% A | |
| 19,51 a 24 min | Equilibre com 45% A |
Tabela 1: Parâmetros de Gradiente para Separação RP-UPLC de Lípidos. RP, fase reversa. UPLC, cromatografia líquida ultra-performance.
| Tempo (min) | % Buffer A para Buffer B | |
| 0 a 1 min | 99% A | Tampão A: 0,1% de ácido fórmico em água de grau UPLC |
| 1 a 11 min | Gradiente linear de 99% A a 60% A | Tampão B: ácido fórmico a 0,1% em acetonitrilo UPLC grad |
| 11 a 13 min | Gradiente linear de 60% A a 30% A | Caudal 4001; L / min |
| 13 a 15 min | Gradiente linear de 30% A a 1% A | Volume de injeção 2 μL |
| 15 a 16 minutos | Lave a coluna durante 1 min com 1% de A | |
| 16 a 17 min | Gradiente linear de 1% A a 99% A | |
| 17 a 20 min | Equilibre-se durante 3 min a 99% A |
Tabela 2: Parâmetros de gradiente para separação RP-UPLC de metabólitos polares e semi-polares. RP, fase reversa. UPLC, cromatografia líquida ultra-performance.
| Tempo (min) | % Buffer B para Buffer A | |
| 0 a 5 minutos | Gradiente linear de 0 a 10% | Tampão A: 0,1% de ácido fórmico em água de grau UPLC |
| 5 a 80 min | Gradiente linear de 10% a 40% | Tampão B: ácido fórmico a 0,1% em acetonitrilo com grau UPLC a 60% |
| 80 a 85 min | Gradiente linear de 40% a 60% | Caudal 300 nL / min |
| 85 a 86 min | Gradiente linear de 60% a 95% | Volume de injeção 5 μL |
| 86 a 91 min | Coloque a coluna durante 5 min com 95% | |
| 91 a 92 min. | Gradiente linear de 95% a 0% | |
| 93 a 110 min | Equilibre a coluna durante 17 min a 0% |
Tabela 3: Parâmetros de Gradiente para Separação Nano-LC de Peptídeos. LC, cromatografia líquida.
Discussão
Neste artigo, descrevemos e ilustramos um protocolo de extração simples e altamente aplicável para análise lipídica, metabolômica e proteômica abrangente a partir de uma única amostra de folha de 50 mg. O método foi anteriormente utilizado em vários estudos, que foram publicados em diversos artigos 17 , 18 , 19 , 20 , 21 , 22 , 23 , 24 , 25 , 26 , 31 , 32 , 33 , 34 , 35 , 36 , 37 E provou, além de seu straight-forwardFluxo de trabalho e alta aplicabilidade para ser robusto e reprodutível.
As aplicações fornecidas aqui mostram alguns métodos de rotina para a triagem inicial de uma amostra biológica complexa. Estes conjuntos de dados metabolômicos e lipidômicos de larga escala podem fornecer informações abrangentes sobre as mudanças amplas ou específicas no metabolismo do sistema biológico analisado, enquanto os dados obtidos a partir da análise de proteínas fornecem informações sobre a quantitativa (abundância) e qualitativa (modificações ) Mudanças de enzimas, proteínas estruturais ou fatores de transcrição (TFs) que controlam funções e máquinas celulares. Conseqüentemente, os dados de informação de integração podem revelar informações iniciais sobre possíveis mudanças induzidas por perturbações genéticas ou bióticas e / ou abióticas de um sistema biológico, ao elucidar as mudanças moleculares de diversas moléculas associadas a caminhos metabólicos específicos ou processos celulares.
Claro, A longo prazo, é bastante essencial, para uma análise bem sucedida da biologia dos sistemas, maximizar o número de entidades moleculares analisadas e anotadas, permitindo o monitoramento das funções e atividades celulares o mais completamente possível. Para este fim, as frações obtidas podem ser aplicadas adicionalmente a diversos métodos analíticos, visando outros compostos ou classes compostos ( Figura 4 ).
Dito isto, deve-se mencionar que a estratégia de análise global dos dados obtidos pode ser seguida por duas estratégias diferentes: por um lado, enfatizamos a elucidação das funções celulares pela quantificação de compostos conhecidos. Por outro lado, muitos dos metabolitos e lipídios medidos ainda não são conhecidos ou anotados. Contudo, as medições de compostos não anotadas também contêm muitas informações significativas, que, podem ser utilizadas por métodos estatísticos para a classificação ou discriminação bGrupos ou tratamentos 20 , 21 , 22 .
Ainda assim, esses compostos desconhecidos, especialmente os que são relevantes para classificação em grupo ou servindo como biomarcadores, precisam ser identificados. Este processo de identificação é, infelizmente, bastante tedioso e não pode ser alcançado sem medidas ou estratégias analíticas adicionais 38 . Como pode ser visto na Figura 4 , o número de compostos não anotados é bastante elevado (na verdade, a grande maioria). No entanto, como mencionado acima, esses picos cromatográficos podem ser tratados dentro da análise de dados e, portanto, as entidades significativamente afetadas podem ser elucidadas e submetidas a novas estratégias de identificação.
Em resumo, podemos concluir que o protocolo aqui introduzido oferece várias vantagens para a biologia de sistemas experimentais, bem como para aplicação estatística clássicaAções.
Primeiro, uma vez que todas as frações são extraídas de uma única amostra, a variação entre os diferentes conjuntos de dados experimentais (lipídios, metabolitos, proteínas) é significativamente reduzida, uma vez que cada conjunto de dados é derivado da mesma alíquota de amostra. Isto conduz claramente à maior comparabilidade dos resultados obtidos.
Em segundo lugar, o método é facilmente escalável e, portanto, altamente compatível com quantidades de amostra pequenas a grandes. Nós usamos rotineiramente 10-100 mg de amostras de tecido, mas estudos lipídicos bem sucedidos também foram realizados em apenas 20 sementes de Arabidopsis 31 . Especialmente, a compatibilidade com pequenas quantidades de amostra torna este método aplicável se quantidades limitadas de tecidos ou amostras biológicas estiverem disponíveis. Ainda assim, mesmo que haja material de amostra suficiente, o método aqui apresentado oferece a vantagem de utilizar essas amostras em um maior número de repetições experimentais em vez de vocêCante-os para diferentes procedimentos de extração. Isso permite uma análise de dados estatísticos melhor e mais refinada.
Em terceiro lugar, uma vez que o método é baseado em um fracionamento líquido-líquido de moléculas polares e não-polares, fornece, em contraste com métodos simples de extração em uma fase ( por exemplo, extrações de metanol), um passo de descombinação significativo no procedimento. Este desmatamento eficiente da amostra leva a uma purificação parcial das frações individuais devido à separação de moléculas que interferem quimicamente umas das outras. Por conseguinte, o processo de divisão química não só proporciona uma vantagem prática para a alíquota sistemática das amostras extraídas em diferentes classes químicas, mas também melhora as medidas analíticas individuais, uma vez que remove compostos contaminantes das diferentes frações. Claramente, podemos observar que especialmente os lipídios, que são divididos na fase orgânica e que geralmente afetam negativamente aAnálise cromatográfica de compostos polares, estará quase completamente ausente da fração polar. O mesmo é verdade para a análise dos lípidos hidrófobos, que serão esgotados dos compostos polares. Além da purificação de compostos polares e não polares uns dos outros, esvaziamos e colecionamos proteínas e outras macro-moléculas da amostra, que não só fornece uma fração separada, que pode ser utilizada para análises de proteína, amido e parede celular 16 , mas também Leva a uma amostra mais limpa dentro das frações individuais. Isto é especialmente relevante, uma vez que se sabe que a presença de macromoléculas grandes leva a danos ou, pelo menos, a vida útil mais curta das colunas analíticas.
Por último, mas não menos importante, o método de extração MTBE descrito, que depende do solvente de substituição de clorofórmio menos perigoso e mais favorável 15 , já foi mostrado por vários estudos do nosso grupo, para ser amplamente aplicávelIcability para diferentes amostras biológicas de plantas 16 , algas 17 , 18 , moscas 19, mas também vários tecidos, órgãos ou células de mamíferos 20 , 21 , 22 .
Divulgações
Os autores não têm nada a revelar
Agradecimentos
O MS é apoiado por uma bolsa de doutorado completa do programa GERLS-DAAD. Gostaríamos de agradecer ao Dr. Andrew Wiszniewski pela prova de leitura e comentário sobre o manuscrito. Estamos muito gratos a todos os membros do laboratório Giavalisco no Instituto Max-Planck de Molecular Plant Physiology, Golm, na Alemanha por sua ajuda.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents and standards | |||
| Ampicillin | Sigma Aldrich | A9393-5G | Internal standard for metabolites |
| Corticosterone | Sigma Aldrich | 27840-500MG | Internal standard for metabolites, HPLC grade |
| 13C Sorbitol | Sigma Aldrich | 605514 | Internal standard for metabolites, ISOTEC® Stable Isotopes |
| 1,2-diheptadecanoyl-sn-glycero-3-phosphocholine (17:0 PC) | Avanti Polar Lipids | 850360P | Internal standard for lipids |
| Methanol (MeOH) | Biosolve Chemicals | 13684102 | ULC-MS grade |
| Water | Biosolve Chemicals | 23214102 | ULC-MS grade |
| Methyl tert-butyl ether (MTBE) | Biosolve Chemicals | 13890602 | HPLC grade |
| Trypsin/Lys-C mix | Promega | V5072 | Enzymatic digestion of proteins |
| Equipment | |||
| Balance | Sartorius Corporation | 14 557 572 | |
| Tissue grinding mixer mill | Retsch, Mixer Mill MM 300 | 20.746.0001 | |
| Mortar and pestle | Sigma Aldrich | Z247464-1EA | |
| Vortex mixer | Vortex-Genie 2, Model G560 | SI-0236 | |
| Vacuum concentrator | Scan Speed Maxi Vac Alpha Evaporators | 7.008.500.002 | |
| 2 mL Safe-lock microcentrifuge tubes | Eppendorf | 30120094 | Used for sample extarction |
| 1.5 mL Safe-lock microcentrifuge tubes | Eppendorf | 30120086 | Used for fractions |
| Shaker | Eppendorf Thermomixer 5436 | 2050-100-05 | |
| Sonicator | USC 300 TH | 142-0084 | |
| Refrigerated microcentrifuge | Eppendorf, model 5427R | 22620701 | |
| UPLC system | Waters Acquity UPLC system (Waters, Machester, UK) | ||
| MS system | Exactive, Orbitrap-type, MS (Exactive, Thermo-Fisher, Bremen, Germany). | ||
| Reversed Phase (RP) Bridged Ethyl Hybrid (BEH) C8 column (100 mm × 2.1 mm containing 1.7 μm diameter particles) | Waters, Machester, UK | 186002878 | Analysis of lipids |
| RP High Strength Silica (HSS) T3 column (100 mm × 2.1 mm containing 1.8 μm diameter particles) | Waters, Machester, UK | 186003539 | Analysis of metabolites |
| Q ExactivePlus high resolution mass spectrometer connected to an EASY-nLC 1000 system | Thermo-Fisher, Bremen, Germany | Analysis of peptides |
Referências
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Yang, M. Q., et al. The emerging genomics and systems biology research lead to systems genomics studies. BMC Genomics. 15, 1 (2014).
- Sheth, B. P., Thaker, V. S. Plant systems biology: insights, advances and challenges. Planta. 240 (1), 33-54 (2014).
- Ideker, T., Galitski, T., Hood, L. A new approach to decoding life: Systems biology. Annu Rev Genom Hum G. 2, 343-372 (2001).
- Somvanshi, P. R., Venkatesh, K. V. A conceptual review on systems biology in health and diseases: from biological networks to modern therapeutics. Syst Synth Biol. 8 (1), 99-116 (2014).
- Oliver, S. G., Winson, M. K., Kell, D. B., Baganz, F. Systematic functional analysis of the yeast genome. Trends Biotechnol. 16 (9), 373-378 (1998).
- Tweeddale, H., Notley-McRobb, L., Ferenci, T. Effect of slow growth on metabolism of Escherichia coli, as revealed by global metabolite pool ("metabolome") analysis. J Bacteriol. 180 (19), 5109-5116 (1998).
- Kim, H. K., Verpoorte, R. Sample preparation for plant metabolomics. Phytochemical analysis : PCA. 21 (1), 4-13 (2010).
- Folch, J., Lees, M., Stanley, G. H. S. A Simple Method for the Isolation and Purification of Total Lipides from Animal Tissues. J Biol Chem. 226 (1), 497-509 (1957).
- Weckwerth, W., Wenzel, K., Fiehn, O. Process for the integrated extraction, identification and quantification of metabolites, proteins and RNA to reveal their co-regulation in biochemical networks. Proteomics. 4 (1), 78-83 (2004).
- Wienkoop, S., et al. Integration of metabolomic and proteomic phenotypes: analysis of data covariance dissects starch and RFO metabolism from low and high temperature compensation response in Arabidopsis thaliana. Mol Cell Proteomics. 7 (9), 1725-1736 (2008).
- Valledor, L., et al. A universal protocol for the combined isolation of metabolites, DNA, long RNAs, small RNAs, and proteins from plants and microorganisms. Plant J. 79 (1), 173-180 (2014).
- Nakayasu, E. S., et al. MPLEx: a Robust and Universal Protocol for Single-Sample Integrative Proteomic, Metabolomic, and Lipidomic Analyses. mSystems. 1 (3), (2016).
- Alfonsi, K., et al. Green chemistry tools to influence a medicinal chemistry and research chemistry based organisation. Green Chem. 10 (1), 31-36 (2008).
- Salem, M. A., Juppner, J., Bajdzienko, K., Giavalisco, P. Protocol: a fast, comprehensive and reproducible one-step extraction method for the rapid preparation of polar and semi-polar metabolites, lipids, proteins, starch and cell wall polymers from a single sample. Plant Methods. 12, 45 (2016).
- Hemme, D., et al. Systems-wide analysis of acclimation responses to long-term heat stress and recovery in the photosynthetic model organism Chlamydomonas reinhardtii. Plant Cell. 26 (11), 4270-4297 (2014).
- Sharma, D. K., et al. UPLC-MS analysis of Chlamydomonas reinhardtii and Scenedesmus obliquus lipid extracts and their possible metabolic roles. J Appl Phycol. 27 (3), 1149-1159 (2015).
- Delgado, R., Munoz, Y., Pena-Cortes, H., Giavalisco, P., Bacigalupo, J. Diacylglycerol activates the light-dependent channel TRP in the photosensitive microvilli of Drosophila melanogaster photoreceptors. J Neurosci. 34 (19), 6679-6686 (2014).
- Bozek, K., et al. Organization and evolution of brain lipidome revealed by large-scale analysis of human, chimpanzee, macaque, and mouse tissues. Neuron. 85 (4), 695-702 (2015).
- Khrameeva, E. E., et al. Neanderthal ancestry drives evolution of lipid catabolism in contemporary Europeans. Nat Commun. 5, 3584 (2014).
- Bozek, K., et al. Exceptional evolutionary divergence of human muscle and brain metabolomes parallels human cognitive and physical uniqueness. PLoS Biol. 12 (5), 1001871 (2014).
- Krueger, S., Steinhauser, D., Lisec, J., Giavalisco, P. Analysis of subcellular metabolite distributions within Arabidopsis thaliana leaf tissue: a primer for subcellular metabolomics. Methods Mol Biol. 1062, 575-596 (2014).
- Hummel, J., et al. Ultra performance liquid chromatography and high resolution mass spectrometry for the analysis of plant lipids. FrontPlant Sci. 2, 54 (2011).
- Giavalisco, P., et al. Elemental formula annotation of polar and lipophilic metabolites using (13) C, (15) N and (34) S isotope labelling, in combination with high-resolution mass spectrometry. Plant J. 68 (2), 364-376 (2011).
- Caldana, C., et al. Systemic analysis of inducible target of rapamycin mutants reveal a general metabolic switch controlling growth in Arabidopsis thaliana. Plant J. 73 (6), 897-909 (2013).
- Giavalisco, P., et al. Proteome analysis of Arabidopsis thaliana by two-dimensional gel electrophoresis and matrix-assisted laser desorption/ionisation-time of flight mass spectrometry. Proteomics. 5 (7), 1902-1913 (2005).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature protocols. 2 (8), 1896-1906 (2007).
- Spagou, K., et al. Hydrophilic interaction chromatography coupled to MS for metabonomic/metabolomic studies. J Sep Sci. 33 (6-7), 716-727 (2010).
- Hauck, O. K., et al. Uric acid accumulation in an Arabidopsis urate oxidase mutant impairs seedling establishment by blocking peroxisome maintenance. Plant Cell. 26 (7), 3090-3100 (2014).
- Avin-Wittenberg, T., et al. Global analysis of the role of autophagy in cellular metabolism and energy homeostasis in Arabidopsis seedlings under carbon starvation. Plant Cell. 27 (2), 306-322 (2015).
- Watanabe, M., et al. Comprehensive dissection of spatiotemporal metabolic shifts in primary, secondary, and lipid metabolism during developmental senescence in Arabidopsis. Plant physiol. 162 (3), 1290-1310 (2013).
- Lambers, H., et al. Proteaceae from severely phosphorus-impoverished soils extensively replace phospholipids with galactolipids and sulfolipids during leaf development to achieve a high photosynthetic phosphorus-use-efficiency. New Phytol. 196 (4), 1098-1108 (2012).
- Degenkolbe, T., et al. Differential remodeling of the lipidome during cold acclimation in natural accessions of Arabidopsis thaliana. Plant J. 70, 972-982 (2012).
- Bromke, M. A., et al. Liquid chromatography high-resolution mass spectrometry for fatty acid profiling. Plant J. 81 (3), 529-536 (2015).
- Li, N., et al. FAX1, a novel membrane protein mediating plastid fatty acid export. PLoS Biol. 13 (2), 1002053 (2015).
- Kueger, S., Steinhauser, D., Willmitzer, L., Giavalisco, P. High-resolution plant metabolomics: from mass spectral features to metabolites and from whole-cell analysis to subcellular metabolite distributions. Plant J. 70 (1), 39-50 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados