É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um ensaio simples repórter baseado em fluorescência para identificar componentes celulares necessários para toxina ricina uma cadeia (RTA) o tráfico de levedura
Neste Artigo
Resumo
O manuscrito, descreveremos o uso de um ensaio de repórter de fluorescência baseada em fermento para identificar componentes celulares envolvidos no tráfico e matar processos dos citotóxicos uma subunidade da ricina de toxina da planta (RTA).
Resumo
Bacteriana e planta A / toxinas B exploram os caminhos naturais de tráfico em células eucarióticas para alcançar seu alvo (s) intracelular no citosol e, finalmente, matar. Tal A / toxinas B consistem geralmente em um enzimaticamente ativo Asubunit (por exemplo, toxina ricina um (RTA)) e uma ou mais células vinculação Bsubunit(s), que é responsáveis por toxina vinculação específica de receptores de superfície da pilha. Nosso conhecimento atual de como A / B toxinas são capazes de intoxicante eficientemente células ajudados cientistas para entender os mecanismos celulares fundamentais, como a endocitose e proteína intracelular em células eucarióticas superiores. Do ponto de vista médico, é igualmente importante identificar as rotas de tráfico de toxina principais para encontrar soluções de tratamento adequado para pacientes ou eventualmente desenvolver aplicações terapêuticas baseadas em toxina para terapia do câncer.
Desde análises de todo o genoma da / toxina B tráfico em células de mamíferos é complexo, demorado e caro, vários estudos na / transporte de toxina B foram executadas no organismo modelo levedura Saccharomyces cerevisiae. Apesar de ser menos complexos, fundamentais processos celulares em leveduras e células eucarióticas superiores são semelhantes e muitas vezes os resultados obtidos no fermento podem ser transferidos para a situação dos mamíferos.
Aqui, descrevemos um ensaio rápido e fácil de usar do repórter para analisar o tráfico intracelular de RTA em levedura. Uma vantagem essencial do novo ensaio é a oportunidade de investigar não só RTA retrô-translocação do retículo endoplasmático (ER) no citosol, mas prefiro endocitose e toxina retrógrada de transporte da membrana plasmática no pronto-socorro. O ensaio faz uso de um repórter do que permite a medição indirecta de toxicidade de RTA através de emissão de fluorescência da proteína verde fluorescente (GFP) após a tradução na vivo . Desde que o RTA eficientemente impede a iniciação da biossíntese de proteínas por depurinação do rRNA 28S, este ensaio permite a identificação de proteínas de célula de acolhimento envolvidas no transporte intracelular de RTA através da detecção de alterações em emissão de fluorescência.
Introdução
Pacientes que sofrem de infecções pela toxina produzindo bactérias representam um fardo de médico e financeiro grave para cada sistema de saúde social, em particular desde tratamentos terapêuticos eficientes ainda são em grande parte faltando. Para desenvolver novas estratégias terapêuticas, os mecanismos complexos de intoxicação do clinicamente relevante A / toxinas B como toxina da cólera, toxina Shiga ou ricina precisam ser completamente compreendido a nível molecular, baseado no romance poderosos ensaios que devem ser implementadas.
Nos últimos anos, vários estudos tentaram analisar A / transporte de toxina B em leveduras e células de mamíferos usando métodos demorados e onerosos como toxinas radioactivas rotulagem1,2 , bem como a triagem de siRNA-baseado aproxima-se3. Em alguns casos, toxina tráfico tem sido visualizada na vivo por microscopia de fluorescência após acoplamento químico e/ou genético de subunidades de toxina individual com fluorophores, pontos quânticos ou proteínas fluorescentes4,5. Infelizmente, tais modificações muitas vezes levam a inativas e/ou alteradas propriedades naturais das toxinas. Outra maneira elegante de responder indiretamente uma grande variedade de questões científicas é a utilização de sistemas de repórter baseados em enzimas como lacZ, luciferase, ou proteínas fluorescentes (por exemplo, GFP ou Discosoma sp. (proteína fluorescente vermelha dsRed)).
Neste manuscrito, um simples protocolo é descrito que identifica celulares componentes necessários para o transporte intracelular de RTA extracelularmente aplicada no S. cerevisiae. Desse modo, um plasmídeo de fluorescência-repórter contendo um sinal de N-terminal ER-importação seguido de GFP funciona como um sensor de biossíntese de proteínas, que mede indiretamente a inibição da tradução de proteínas mediada por RTA por GFP fluorescência emissão após in vivo tradução6. No caso que endocitose RTA e/ou tráfico intracelular é negativa (ou positiva) afetado em um mutante de levedura especial exclusão em relação ao tipo selvagem, isto pode ser detectado através de um aumento (ou diminuição) na GFP de emissão de fluorescência6.
Até agora, todos os métodos de análise de transporte de RTA em células de levedura foram restringidos ao processo de translocação retrô ER-para-citosol de RTA. Em um sistema artificial, RTA contendo um sinal de importação ER é expressa de um promotor inducible, resultando em um fenótipo suicida1,7. Embora a célula de ligação B-subunidade de ricina desapareceu da mesma forma na instalação experimental descrita neste manuscrito e, assim, não totalmente representa a situação natural de ricina holotoxin intoxicação8, transporte de toxina do plasma membrana através do aparelho de Golgi ao pronto-socorro pode ser imitada estreitamente com este ensaio de romance. Curiosamente, os resultados preliminares obtidos no estudo piloto indicam que as vias de tráfico usadas pelo RTA revelam semelhanças marcantes com a via de intoxicação de ricina holotoxin.
Em resumo, o método descrito pode ser usado para determinar o papel específico das proteínas celulares selecionadas em endocitose RTA e do tráfico de levedura. Além disso, esta configuração experimental pode ser facilmente adaptada para outra Ribossoma inactivar as toxinas produzidas e secretadas de vários espécies de leveduras e bactérias como zymocin ou toxina Shiga.
Protocolo
Nota: Uma visão geral do fluxo de trabalho geral experimental é descrita na Figura 1.
Cuidado: RTA é altamente tóxico para os seres humanos. É necessário permissão do laboratório de segurança S2 (nível de biossegurança 2 equivalente). Por favor, use luvas durante todo o experimento.
1. heteróloga expressão de RTA com sua Tag em Escherichia coli
- Transforme células de Escherichia coli com o expressão do plasmídeo pET24-RTA(seu) 6 ou o vetor vazio pET24a(+) usando o padrão electroporation protocolos9,10. Células contendo o plasmídeo vazio servem como um controle negativo.
- Após a seleção de clones positivos em placas LB (100 µ g/mL) contendo canamicina, inocular as células contendo o plasmídeo de expressão RTA ou o vetor vazio no meio dekan 5ml LB (LB médio com 100 µ g/mL de canamicina) e incubar a 37 ° C e 220 rpm por 24 h.
- 1 LB Lkan média com a pré-cultura 5 mL de suplemento e incubar as células a 37 ° C e 220 rpm até células têm alcançado uma OD600 de 0.8-1.0 (aproximadamente 3-4 h). Depois disso, reduza a temperatura de cultura a 28 ° C e prepare um 1 M de solução-mãe de isopropil-β-D-1-thiogalactopyranoside (IPTG) em H2O.
- Induzi a expressão de RTA da e. coli pela adição de IPTG para uma concentração final de 1 mM.
- Depois de 3,5 h de 28 ° c e 220 rpm, com colheita de células por centrifugação a 10.000 x g e 4 ° C por 10 min, lave o pellet duas vezes com 5 mL de tampão de ligação (500 mM NaCl, imidazol 10 mM e 20 mM KH2PO4 pH = 7,4) em 10.000 x g e 4 ° C por 10 min e ressuspender pelota em 5 mL de tampão de ligação.
Nota: O protocolo pode ser pausado, nesta fase, e as células podem ser armazenadas a 80 ° C por vários dias.
2. purificação de RTA com sua Tag através de cromatografia de afinidade
- Proceda à sonicação células no gelo usando o seguinte protocolo: 15 pulso s (20 mícrons), 30 s de pausa. Repita essa etapa cinco vezes.
- Centrifugue a célula lisada em 21.000 x g e 4 ° C por 15 min e filtrar o sobrenadante através de um sistema de filtro de seringa estéril (tamanho de poro de 0,2 µm).
Nota: Pelotas de célula de amostras com sucesso lisadas mostram bordas transparentes. - Usar um sistema de purificação automatizados, equipado com um 5 mL Ni2 +-com base em coluna de afinidade para purificar a fração RTA com sua Tag do filtrado estéril Escherichia coli sobrenadante. Em geral, use uma velocidade de eluição de 1 mL/min e fixe o sistema de purificação de todo para impedir a ligação da toxina não-eficiente e perda da atividade da toxina.
Nota: Parâmetros para a purificação de RTA eficiente estão listados na tabela 1. Veja também Becker et al. para mais informações9.- Brevemente, equilibrar a coluna de afinidade com 20 mL de tampão de ligação para remover o buffer de armazenamento. Aplica o sobrenadante filtrado estéril para a coluna de afinidade, usando uma seringa.
- Lave a coluna com 25-35 mL de tampão de ligação para remover as proteínas desvinculadas da coluna. Executar a etapa de lavagem até absorção UV em 280 nm aproxima-se o valor inicial de UV.
- Eluir acoplada fração RTA em 20-35 mL de tampão de eluição (imidazol 500 mM, 500 mM de NaCl, 20mm KH2PO4, pH = 7,2) e manter a amostra em gelo (Figura 2A e 2B figura).
Nota: A eluição da fração RTA é marcada pelo aumento da absorção de UV. Por favor note para coletar exclusivamente esta fração para evitar a contaminação com proteínas acopladas inespecíficas. - Usar 20 µ l de eluted amostras para realizar11 de coloração de azul de Coomassie ou Western blot análise12,13 (opcional). Use primário antisua anticorpos (1:1, 000) e secundária anti-mouse-IgG-HRP (01:10, 000) para detectar RTA e verificar a pureza da amostra (Figura 3).
- Do desalt eluted fração RTA em uma coluna do desalting 5ml e equilibrar a amostra em sorbitol de 0,8 M.
- Substituir a coluna de afinidade do sistema de purificação por coluna do desalting e primeiro equilibrar a coluna com 20 mL de sorbitol de 0,8 M.
- Aplica a fração RTA eluted à coluna através de uma seringa. Lavar a coluna com 100 mL de sorbitol 0,8 M e eluir a fração RTA demolhada em um tubo de 15 mL, assim como a absorção de UV começa a aumentar (Figura 2).
- Armazenar a fração RTA eluted a 4 ° C.
Nota: Pare diretamente amostragem de fração RTA quando aumenta a condutância para evitar a contaminação de sal. Parâmetros para o procedimento do desalting estão listados na tabela 1.
- Eluted fração RTA a 10.000 x g e 4 ° C por 30-180 min usando uma coluna de rotação de corte 10 kDa até um volume final de 1 a 2 mL de concentrar e armazenar a amostra a 4 ° C por 3-4 semanas.
Cuidado: Não congele a amostra desde congelamento leva a uma perda completa da atividade de RTA. - Determine a concentração de proteína usando um kit de determinação de proteína convencional. Concentração de proteína deve ser na faixa de 1-1,5 mg/mL.
Nota: RTA tende a precipitar-se a concentração de proteínas é muito alta (> 5 mg/mL).
3. transformação e remoção da parede celular de levedura
- Transforme mutantes de exclusão de levedura selvagem-tipo ou selecionado com o GFP repórter do plasmídeo pRS315-K28SP- GFP6 usando acetato de lítio padrão a transformação métodos14. Incubar as células no abandono leucina (d/s) placas de glicose (glicose 2%, ágar 1,5%, 0,5% de sulfato de amónio, 0,17% fermento nitrogênio Base (YNB) e mistura de 0,087% d/s sem leucina) a 30 ° C, por 2-3 dias para a seleção de clones positivos.
- Escolha 3 fermento diferentes clones de cada prato e inocular os clones em 100 mL de meio de rafinose de d/s leucina (rafinose de 2%, 0,5% de sulfato de amónio, 0,17% YNB e misture sem leucina e 0,087% d/s) a 220 rpm e 30 ° C a OD600 = 1,0-2,0 (2-4 x 107 células/mL).
Nota: O crescimento de células das diferentes exclusão cepas de leveduras é diferente. Monitor OD600 através de um espectrofotômetro. - Para calcular valores de600 OD, diluir as amostras para OD600 = 0.1-0.3 (1:5 a 01:10 diluições) e medir OD600 em um espectrofotômetro. Como referência, use H2O, complementado com a quantidade correspondente de meio de raffionose de d/s leucina.
- Depois passo acabamento 3.5, misture 4 µ l da cultura spheroplasted 50 mL com tampão de spheroplasting µ l 496 (aproximadamente 2 × 106 spheroplasts) e centrifugar durante 10 min a 400 g de x.
- Ressuspender pelota em 10 mL de H2O destilado, amostra de vórtice para 30 s e a placa para fora 10 µ l da amostra em placas de glicose de d/s leucina (2% de glicose, ágar 1,5%, 0,5% de sulfato de amónio, 0,17% YNB e mistura de 0,087% d/s sem leucina).
- Incubar as células durante 3 dias a 30 ° C. Conte o número total de colônias de células crescidas na placa. Para a avaliação de dados, use apenas amostras com eficiência superior a 98% (total número de colônia de célula < 40 colônias/placa).
4. GFP repórter ensaio medição em placas de 96 poços
- Semente as spheroplasts de células de levedura obtidos na etapa 3.7 em placas de 96 poços (200 µ l/poço).
- Adicionar 70 meio de rafinose d e/s de µ l de leucina estabilizado, contendo controle negativo (eluato de um Ni2 +-afinidade purificada lisado celular de células induzida por IPTG Escherichia coli expressando o vetor vazio) ou purificada RTA em concentração final de 5 µM (RTA correspondente a 160 g/L RTA) em cada poço.
- Imediatamente adicione 30 µ l estabilizado galactose solução (30% de galactose, sorbitol 0,8 M) para induzir a expressão de GFP e posteriormente iniciar a medição.
Nota: Realizar no mínimo 3 repetições técnicas por experiência e 3 biológicos Replica por levedura. - Depois de terminar a preparação da amostra (etapas 4.1-4.3), coloquei a placa de 96 poços em um leitor de fluorescência e iniciar a medição. Use o filtro de 475/509 nm conjunto necessário para a deteção de fluorescência de GFP. Realize medições a 30 ° C, 120 rpm e com um tremendo diâmetro de 1 mm sobre uma janela de tempo de 20 h (medição em intervalos de 10 min).
Nota: O conjunto de filtro GFP é normalmente disponível em todos os sistemas do leitor. Temperatura, janela de tempo, medindo intervalos e a concentração de RTA pode ser ajustada para as próprias necessidades. Resultados representativos são mostrados na Figura 4. - Opcional: Use controles internos adicionais na medição para controle de qualidade. Prepare um controlo negativo, acrescentando meio rafinose d e/s de leucina estabilizado de µ l de 30 em vez de 30% de galactose (sem indução de GFP). Além disso, adicione 70 µ l de solução de G418 de sorbitol estabilizado de 0,8 M (300 µ g/mL). O inibidor da tradução de proteínas G418 serve como controle positivo para inibição da proteína que impede a expressão de GFP no fermento.
- Calcular a fluorescência de GFP relativa por cento por ponto de tempo de 20 h, de acordo com a seguinte equação (ver Figura 4A):
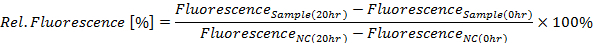 onde NC é a do controlo negativo
onde NC é a do controlo negativo
- Em alternativa, determinar a fluorescência de GFP relativa para cada ponto de medição (neste caso 10 min, ver também passo 4.4) usando a equação acima. Como mostrado na Figura 4B, crie um gráfico borrando as intensidades de fluorescência de GFP (eixo y) ao longo do tempo (eixo x).
Resultados
O fluxo de trabalho geral do protocolo descrito neste manuscrito é ilustrado na Figura 1, aproximadamente, resumindo os passos simples para purificação de RTA bem sucedida e o experimento de ensaio de repórter GFP subsequente. Uma descrição mais detalhada de cada etapa individual pode ser encontrada no protocolo. A Figura 2 ilustra o resultado esperado de uma purificação de RTA bem sucedido por cromatografia de afinidade ...
Discussão
Ao executar o protocolo acima, recomendamos as seguintes sugestões para alcançar um resultado bem sucedido do experimento.
Para a expressão da proteína heteróloga, é importante para não exceder a concentração de IPTG de 1 mM. Concentrações de IPTG > 1mm inibir induzida pelo promotor expressão RTA e chumbo para reduzir a produção de toxina. Além disso, as células não devem ser cultivadas em temperaturas acima de 28 ° C para evitar a formação de corpo de inclusão, dobrad...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Partes deste estudo foram gentilmente com uma subvenção da Deutsche Forschungsgemeinschaft (SFB 1027, A6).
Materiais
| Name | Company | Catalog Number | Comments |
| Bacterial and yeast strains | |||
| E. coli BL21 DE3(Gold) | Aligent Technologies | 230130 | |
| S. cerevisiae BY4742 | Euroscarf | Y10000 | |
| S. cerevisiae BY4742 deletion mutants | Dharmacon | YSC1054 | whole collection |
| Name | Company | Catalog Number | Comments |
| Plasmids used in this protocol | |||
| pET24a(+) (Novagen) | Millipore | 69772-3 | |
| pET-RTA(His6) | Becker et al. (2016)3 | ||
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Zymolyase 20T | USBio | Z1000.250 | lytic enzyme for cell wall removal |
| LB broth medium | Thermo Scientific | 10855021 | 15 g agar was added for plate production |
| YNB | Thermo Scientific | DF0335-15-9 | |
| Ammonium sulfate | Sigma-Aldrich | A4418-100G | |
| Yeast drop-out mix supplemts without leucine | Sigma-Aldrich | Y1376-20G | |
| Agar | Sigma-Aldrich | 05040-100G | |
| D-glucose | Sigma-Aldrich | G8270-100G | |
| DTT | Sigma-Aldrich | 10197777001 | |
| D-raffinose | Sigma-Aldrich | 83400-25G | |
| D-sorbitol | Sigma-Aldrich | S1876-1KG | |
| D-galactose | Sigma-Aldrich | G0750-10MG | |
| G418 | Thermo Scientific | 11811031 | |
| IPTG | Sigma-Aldrich | I6758-1G | |
| Imidazole | Roth | 3899.1 | |
| PAGE ruler prestained | Fermentas | 26616 | protein ladder used for Western analysis |
| Name | Company | Catalog Number | Comments |
| Material for RTA purification, desalting and reader measurements | |||
| Spectrophotometer Ultrospec 2100 pro | Amersham | ||
| Soniprep 150 | MSE | old model, other models available | |
| Fluoroskan Ascent | Thermo Scientific | 5210470 | old model, not available anymore |
| ÄKTAPurifier | Thermo Scientific | 28406266 | Product is discontinued and replaced |
| HisTRAP HP column | GE Healthcare | 17-5248-02 | |
| HiTRAP desalting column | GE Healthcare | 11-0003-29 | |
| Midisart sterile filter | Sartorius | 16534K | 0.2 µm pore size |
| BCA protein assay kit | Pierce | 23225 | |
| 660 nm assay kit | Thermo Scientific | 22660 | |
| 96 well plates | Thermo Scientific | 260860 | |
| Name | Company | Catalog Number | Comments |
| Antibodies (optional) | |||
| Anti-Tetra-His | Qiagen | 34670 | primary antibody; 1:1,000 dilution |
| Anti-mouse-HRP | Sigma-Aldrich | A9044-2ML | secondary antibody, 1:10,000 dilution |
Referências
- Li, S., et al. Folding-competent and folding-defective forms of ricin A chain have different fates after retrotranslocation from the endoplasmic reticulum. Mol Biol Cell. 21 (15), 2543-2554 (2010).
- Li, S., Spooner, R. A., Hampton, R. Y., Lord, J. M., Roberts, L. M. Cytosolic entry of Shiga-like toxin a chain from the yeast endoplasmic reticulum requires catalytically active Hrd1p. PLoS One. 7 (7), e41119 (2012).
- Moreau, D., et al. Genome-wide RNAi screens identify genes required for Ricin and PE intoxications. Dev Cell. 21 (2), 231-244 (2011).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312 (5771), 217-224 (2006).
- Majoul, I. V., Bastiaens, P. I., Soling, H. D. Transport of an external Lys-Asp-Glu-Leu (KDEL) protein from the plasma membrane to the endoplasmic reticulum: studies with cholera toxin in Vero cells. J Cell Biol. 133 (4), 777-789 (1996).
- Becker, B., Schnoder, T., Schmitt, M. J. Yeast Reporter Assay to Identify Cellular Components of Ricin Toxin A Chain Trafficking. Toxins (Basel). 8 (12), (2016).
- Li, X. P., Baricevic, M., Saidasan, H., Tumer, N. E. Ribosome depurination is not sufficient for ricin-mediated cell death in Saccharomyces cerevisiae. Infect Immun. 75 (1), 417-428 (2007).
- Lord, J. M., Roberts, L. M., Robertus, J. D. Ricin: structure, mode of action, and some current applications. Faseb J. 8 (2), 201-208 (1994).
- Becker, B., Schmitt, M. J. Adapting yeast as model to study ricin toxin a uptake and trafficking. Toxins (Basel). 3 (7), 834-847 (2011).
- Seidman, C. E., Struhl, K., Sheen, J., Jessen, T. Introduction of plasmid DNA into cells. Curr Protoc Mol Biol. , (2001).
- Brunelle, J. L., Green, R. Coomassie blue staining. Methods Enzymol. 541, 161-167 (2014).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. 1979. Biotechnology. 24, 145-149 (1992).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Vitetta, E. S., Yen, N. Expression and functional properties of genetically engineered ricin B chain lacking galactose-binding activity. Biochim Biophys Acta. 1049 (2), 151-157 (1990).
- Wales, R., Roberts, L. M., Lord, J. M. Addition of an endoplasmic reticulum retrieval sequence to ricin A chain significantly increases its cytotoxicity to mammalian cells. J Biol Chem. 268 (32), 23986-23990 (1993).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat Methods. 5 (8), 711-718 (2008).
- Jablonowski, D., Schaffrath, R. Zymocin, a composite chitinase and tRNase killer toxin from yeast. Biochem Soc Trans. 35 (Pt 6), 1533-1537 (2007).
- Jablonowski, D., Schaffrath, R. Saccharomyces cerevisiae RNA polymerase II is affected by Kluyveromyces lactis zymocin. J Biol Chem. 277 (29), 26276-26280 (2002).
- Jablonowski, D., Frohloff, F., Fichtner, L., Stark, M. J., Schaffrath, R. Kluyveromyces lactis zymocin mode of action is linked to RNA polymerase II function via Elongator. Mol Microbiol. 42 (4), 1095-1105 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados