É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uma sonda de força direta para medir a integração mecânica entre o núcleo e o citoesqueleto
Neste Artigo
Resumo
Neste protocolo, descrevemos um método de micropipeta para aplicar directamente uma força controlada para o núcleo de uma célula viva. Este ensaio permite interrogatório das propriedades mecânicas nucleares na célula viva, aderente.
Resumo
As propriedades mecânicas do núcleo determinam sua resposta às forças mecânicas geradas nas células. Porque o núcleo é molecularmente contínuo com o citoesqueleto, métodos são necessários para sondar seu comportamento mecânico em células aderentes. Aqui, vamos discutir a sonda de força direta (DFP) como uma ferramenta para aplicar a força diretamente para o núcleo em uma célula aderente viva. Atribuímos uma micropipeta estreita à superfície nuclear com sucção. A micropipeta é traduzida longe do núcleo, o que faz com que o núcleo se deforma e traduzir. Quando a força restauradora é igual à força de sucção, o núcleo desconecta e elasticamente, relaxa. Porque o pressão de sucção é precisamente conhecido, é conhecida a força na superfície nuclear. Este método revelou-se que as forças de nano-escala são suficientes para deformar-se e traduzir o núcleo nas células aderentes e identificaram elementos do citoesqueleto que permitem que o núcleo resistir às forças. O DFP pode ser usado para dissecar as contribuições dos componentes celulares e nucleares para propriedades mecânicas nucleares em células vivas.
Introdução
Patologias como câncer envolvem alterações à forma nuclear e estrutura1,2, que geralmente são acompanhados de um amolecimento do núcleo3,4. Nuclear resistência à deformação mecânica tem sido geralmente caracterizada pela aplicação de uma força de núcleos isolados5.
O núcleo nas células molecularmente está ligado ao citoesqueleto pelo vinculador de Nucleoskeleton e citoesqueleto (LINC) complexo6,7,8,9. Como resultado, o núcleo mecanicamente é integrado com o citoesqueleto e, através de aderências célula-substrato, a matriz extracelular. Mecanicamente, sondar o núcleo no interior das células aderentes pode fornecer insights sobre essa integração mecânica. Métodos para manipular os núcleos em células vivas incluem micropipeta aspiração10,11e microscopia de força atômica a12,13,14. Recentemente descrevemos uma sonda de força direta (DFP) que se aplica forças mecânicas diretamente no núcleo em uma vida aderentes célula15.
Aqui, podemos descrever o procedimento para usar um sistema de microinjeção que está geralmente disponível em instalações de microscopia para aplicar uma força mecânica de nano-escala conhecida, diretamente para o núcleo em uma célula aderente. Um femtotip (0,5 µm ponta de micropipeta de diâmetro) é montado e ligado ao sistema de microinjeção por um tubo. A ponta, posicionada em um ângulo de 45° em relação à superfície do prato, cultura é abaixada até adjacente à superfície nuclear. O tubo é então desligado e aberto para a atmosfera, o que cria uma pressão negativa de sucção na superfície nuclear e lacra a ponta da micropipeta contra a superfície nuclear. Através da tradução da ponta da micropipeta, o núcleo é deformado e eventualmente (dependendo da magnitude da força aplicada), extraído da micropipeta. Este desprendimento ocorre quando as forças de restaura (oposição), exercidas pelo núcleo e célula, igual a força de sucção aplicada pela micropipeta. Análise pode ser realizada medindo-se o deslocamento do núcleo, a estirpe de comprimento (equação 1), ou a tensão da área (figura 1A).
Protocolo
1. preparar células para a imagem latente
Nota: A sonda de força direta (DFP) pode ser usada para qualquer tipo de células aderentes. Aqui, fibroblastos de rato do NIH 3T3 são usados como a linha celular do modelo para este protocolo.
- Células de fibroblastos cultura NIH 3T3 em modificado águia de Dulbecco médio (DMEM) suplementado com 10% de soro de bovino doador e 1% penicilina-estreptomicina em um fundo de vidro 35mm prato até confluência desejada. Manter as células em 37 ° C e 5% de CO2.
- Não se esqueça de revestir todos os pratos de fundo de vidro 35mm com 5 µ g/mL de fibronectina (ou proteína de ECM semelhante), antes da semeadura das células 3T3 NIH para a imagem latente.
Nota: As células devem ser totalmente espalhadas e aderente sobre o prato para o experimento. Não existem quaisquer restrições em termos de confluência para o método DFP trabalhar.
- Não se esqueça de revestir todos os pratos de fundo de vidro 35mm com 5 µ g/mL de fibronectina (ou proteína de ECM semelhante), antes da semeadura das células 3T3 NIH para a imagem latente.
- Imediatamente antes do experimento, as células de duas lavagens com PBS, seguido por uma simples lavagem com meio de cultura completo.
- Adicione 3 mL de meio de cultura completo para o prato fundo de vidro.
2. microscopia e aquisição de imagem
Nota: Um invertido fluorescência microscópio (ou equivalente) com micromanipulador instalado para o lado do braço, de acordo com as recomendações do fabricante. O microscópio também deve ser equipado com uma câmara ambiental para manter a temperatura a 37 ° C e o nível de CO2 em 5%. Também é necessário um micromanipulador e microinjector anexado ao microscópio. Um óleo de imersão x 40 / 1,3 at ou 60 x / 1.49 at (ou objectivos equivalentes) são recomendados para os experimentos. O microscópio deve ser montado sobre uma mesa de isolamento de vibração.
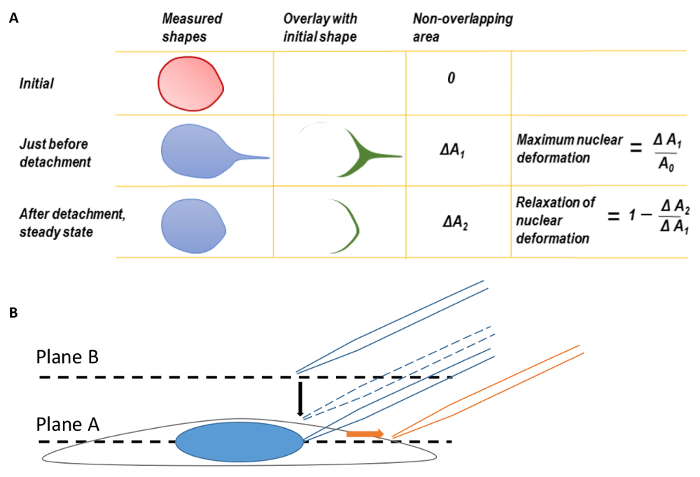
Figura 1 . Deformação nuclear e microscópio com foco
R. máxima deformação nuclear e relaxamento da deformação nuclear. Antes de calcular a máxima deformação nuclear, as bordas traseiras das formas nucleares foram primeiro coincidiu para corrigir para a tradução do núcleo deformado. A forma do núcleo no momento do desprendimento de ponta micropipeta era revestida na forma nuclear inicial antes de puxar. A diferença de área entre as duas formas foi medida como Δum1. A máxima deformação nuclear foi definida como ΔA1 dividido pela área nuclear original. Da mesma forma, um segundo parâmetro, ΔA2, podem ser definidas por sobrepondo a forma nuclear final estado estacionário após descolamento micropipeta na forma nuclear original. B. a célula no avião A concentrar-se e mova o plano focal até avião B para encontrar a ponta da micropipeta. Durante a imagem latente, a micropipeta foi traduzida para a direita (sentido da seta laranja). Esta figura foi modificada de Eelam et al. 15. clique aqui para ver uma versão maior desta figura.
- Ligue o microinjector acordo com o protocolo do fabricante.
- Usando um gotas de óleo de imersão, aplica uma gota de óleo de imersão em cima da lente objetiva.
- Fixar o prato firmemente no suporte do prato e carregar o suporte para o palco.
Nota: As células devem ser mantidas a 37 ° C e 5% CO2 durante todo o experimento. - Ajuste a altura do objectivo de colocar as células em foco (A do avião, figura 1B).
- Mova o estágio de microscópio para encontrar uma célula de interesse.
- Gire o joystick no micromanipulador para mover o titular pipeta para a posição superior. Carrega a micropipeta de ponta de diâmetro de 0,5 µm no suporte pipeta.
- Para evitar aderência de célula para a micropipeta, pré-tratamento a ponta da micropipeta com 0,3 mg/mL solução de PLL-g-PEG por 1h à temperatura ambiente. Teste de aderência tocando a micropipeta para o núcleo sem qualquer pressão de sucção, e então traduzir a micropipeta longe do núcleo. A ausência de adesão pode ser discernida por falta completa de deformação nuclear e tradução.
Nota: Por favor siga sugestões de fabrico para abrir o pacote.
- Para evitar aderência de célula para a micropipeta, pré-tratamento a ponta da micropipeta com 0,3 mg/mL solução de PLL-g-PEG por 1h à temperatura ambiente. Teste de aderência tocando a micropipeta para o núcleo sem qualquer pressão de sucção, e então traduzir a micropipeta longe do núcleo. A ausência de adesão pode ser discernida por falta completa de deformação nuclear e tradução.
- Elevar o plano focal objectivo acima A do avião e o topo da célula para o plano B, ajustando o controle fino (figura 1B, consulte a etapa 2.4).
- Conjunto o micromanipulador para controle de grosseira . Traga lentamente a micropipeta até avião B observando para a silhueta da micropipeta, até a micropipeta totalmente entra em foco.
- Uma vez que a ponta da micropipeta está em foco, defina o micromanipulador para controle fino .
- Baixe o objectivo para o plano equatorial da célula (avião A, figura 1B) e inferior a micropipeta para cerca de 15 µm acima plano A (figura 1B, micropipeta tracejada).
- Ajustar a pressão de compensação (Pc) sobre o microinjector a pressão desejada; Aguarde alguns segundos até a pressão estabilizar.
Nota: O ponto de ajuste de pressão ideal depende de tipo de célula e os objetivos específicos do experimento. Para a maioria dos casos, 300 hPa seria um bom ponto de partida. - Certifique-se de que a micropipeta não está entupida usando a configuração de limpeza no painel micromanipulador e verificar para certificar-se de bolhas de ar emergem da ponta da micropipeta.
- Introduza a ponta dentro da célula, diminuindo gradualmente a micropipeta, até que a ponta é levemente tocar a superfície nuclear.
Nota: Ao descer a micropipeta, a silhueta da ponta da micropipeta se tornará clara como entra em foco. Antes a micropipeta toca o núcleo, aumentar o foco da objetivo e alinhe a ponta da micropipeta com núcleo (mesma coordenada x-y, z-plano superior). Retornar o foco para o plano equatorial do núcleo (A de avião, figura 1B) e gradualmente diminuir a ponta da micropipeta. - Crie um selo entre a ponta da micropipeta e a membrana nuclear, desconectar o tubo de alimentação de pressão do sistema de microinjeção, abrindo assim a extremidade do tubo micropipeta para a atmosfera. Esta etapa cria uma pressão negativa igual a Pc na superfície nuclear.
- Adquira imagens com o software de coleta de imagem de microscópios. Configure um avi-aquisição (vídeo) ou nd-aquisição do software de coleta de imagem (imagens).
Nota: Para qualquer imagem adquirir o software, criar imagens de vídeo em tempo real ou aquisições de imagem de lapso de tempo com um curto intervalo de tempo. - Alternar para o fluorescente correspondente canal de imagem (ou seja, as boas práticas agrícolas, RFP, etc.) e começar a imagem latente.
- Definição da palavra a ponta da micropipeta longe do corpo da célula (para a direita, figura 1B) até que o núcleo se desprende da micropipeta.
Nota: Puxe a ponta ao longo de x-direção positiva (à direita no campo de visão). A taxa de tração pode ser programada e controlada por computador ou o joystick pode ser movido manualmente. Não encontramos qualquer correlação entre a tirar taxa e deformação nuclear15 sugerindo uma resposta principalmente elástica para forçar.
3. análise de dados
- Realizar análise de imagem com qualquer software de processamento de imagem básico disponível. O grau de deformação nuclear pode ser quantificado pela estirpe de comprimento (Ɛ) ou a tensão da área (figura 1A). Quantificar a estirpe de comprimento usando a equação 1, onde L e L0 representam os comprimentos do núcleo na deformação máxima e a posição inicial, respectivamente.
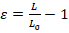 (Equação 1)
(Equação 1)
Resultados
Figura 2A mostra a força de um núcleo de fibroblasto NIH 3T3 rato. Como a ponta da micropipeta é traduzida para a direita, o núcleo deforma e eventualmente desanexa da ponta da micropipeta. A estirpe do comprimento do núcleo é vista a aumentar com o aumento da força de sucção (Figura 2B). Na extremidade dianteira do núcleo (micropipeta puxando a borda) forma uma protusão nuclear e a borda direita é deslocada de sua po...
Discussão
Medindo a integração mecânica do núcleo com o citoesqueleto é um desafio para métodos mais atuais, como micropipeta aspiração16, pois eles exigem ou núcleos isolados (onde o núcleo é dissociado do citoesqueleto) ou núcleos em células suspensos (onde forças extracelulares, como forças de tração, estão ausentes). Força aplicou-se para o núcleo aplicando tensão biaxial de aderente de células a uma membrana17,,18; no enta...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pelo NIH R01 EB014869.
Materiais
| Name | Company | Catalog Number | Comments |
| FluoroDish | WPI | FD35 | |
| SYTO 59 | ThermoFisher Scientific | S11341 | |
| Femtotips | Eppendorf | 930000043 | |
| InjectMan NI2 | Eppendorf | NA | discontinued, current equivalent model: InjectMan 4 |
| FemtoJet | Eppendorf | NA | Current model FemtoJet 4i |
| Plan Fluor oil immersion 40x | Nikon | NA | |
| Apo TIRF oil immersion 60x | Nikon | NA | |
| Donor Bovine Serum (DBS) | ThermoFisher Scientific | 16030074 | NIH 3T3 serum |
| Dulbecco's Modification of Eagle's (DMEM) | Mediatech cellgro | MT10013CVRF | NIH 3T3 medium |
| Penicillin-Streptomycin | Mediatech | MT30004CIRF | NIH 3T3 medium supplement |
| Immersion Oil Type LDF Non-Fluorescing | Nikon | 77007 | Immersion oil for objective lens |
Referências
- Chow, K. H., Factor, R. E., Ullman, K. S. The nuclear envelope environment and its cancer connections. Nature Reviews Cancer. 12 (3), 196-209 (2012).
- Zink, D., Fischer, A. H., Nickerson, J. A. Nuclear structure in cancer cells. Nature Reviews Cancer. 4 (9), 677-687 (2004).
- Bank, E. M., Gruenbaum, Y. The nuclear lamina and heterochromatin: a complex relationship. Biochemical Society Transactions. 39 (6), 1705-1709 (2011).
- Lammerding, J., et al. Lamins A and C but not lamin B1 regulate nuclear mechanics. Journal of Biological Chemistry. 281 (35), 25768-25780 (2006).
- Dahl, K. N., Engler, A. J., Pajerowski, J. D., Discher, D. E. Power-law rheology of isolated nuclei with deformation mapping of nuclear substructures. Biophysical Journal. 89 (4), 2855-2864 (2005).
- Crisp, M., et al. Coupling of the nucleus and cytoplasm: role of the LINC complex. Journal of Cell Biology. 172 (1), 41-53 (2006).
- Sosa, B. A., Rothballer, A., Kutay, U., Schwartz, T. U. LINC complexes form by binding of three KASH peptides to domain interfaces of trimeric SUN proteins. Cell. 149 (5), 1035-1047 (2012).
- Tapley, E. C., Starr, D. A. Connecting the nucleus to the cytoskeleton by SUN-KASH bridges across the nuclear envelope. Current Opinion in Cell Biology. 25 (1), 57-62 (2013).
- Arsenovic, P. T., et al. Nesprin-2G, a Component of the Nuclear LINC Complex, Is Subject to Myosin-Dependent Tension. Biophysical Journal. 110 (1), 34-43 (2016).
- Rowat, A. C., Lammerding, J., Ipsen, J. H. Mechanical properties of the cell nucleus and the effect of emerin deficiency. Biophysical Journal. 91 (12), 4649-4664 (2006).
- Rowat, A. C., Foster, L. J., Nielsen, M. M., Weiss, M., Ipsen, J. H. Characterization of the elastic properties of the nuclear envelope. Journal of the Royal Society Interface. 2 (2), 63-69 (2005).
- Pagliara, S., et al. Auxetic nuclei in embryonic stem cells exiting pluripotency. Nature Materials. 13 (6), 638-644 (2014).
- Liu, H., et al. In situ mechanical characterization of the cell nucleus by atomic force microscopy. ACS Nanotechnology. 8 (4), 3821-3828 (2014).
- Krause, M., Te Riet, J., Wolf, K. Probing the compressibility of tumor cell nuclei by combined atomic force-confocal microscopy. Physical Biology. 10 (6), 065002 (2013).
- Neelam, S., et al. Direct force probe reveals the mechanics of nuclear homeostasis in the mammalian cell. Proceedings of the National Academy of Sciences of the United States of America. 112 (18), 5720-5725 (2015).
- Pajerowski, J. D., Dahl, K. N., Zhong, F. L., Sammak, P. J., Discher, D. E. Physical plasticity of the nucleus in stem cell differentiation. Proceedings of the National Academy of Sciences of the United States of America. 104 (40), 15619-15624 (2007).
- Lammerding, J., et al. Lamin A/C deficiency causes defective nuclear mechanics and mechanotransduction. Journal of Clinical Investigation. 113 (3), 370-378 (2004).
- Chancellor, T. J., Lee, J., Thodeti, C. K., Lele, T. Actomyosin tension exerted on the nucleus through nesprin-1 connections influences endothelial cell adhesion, migration, and cyclic strain-induced reorientation. Biophysical Journal. 99 (1), 115-123 (2010).
- Neelam, S., Dickinson, R. B., Lele, T. P. New approaches for understanding the nuclear force balance in living, adherent cells. Methods. 94, 27-32 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados