È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una sonda di forza diretta per la misura di integrazione meccanica tra il nucleo e il citoscheletro
In questo articolo
Riepilogo
In questo protocollo, descriviamo un metodo micropipetta per applicare direttamente una forza controllata al nucleo in una cellula vivente. Questo test permette di interrogatorio di proprietà meccaniche nucleari nella cellula viva, aderente.
Abstract
Le proprietà meccaniche del nucleo determinano la sua risposta a forze meccaniche generate nelle cellule. Poiché il nucleo è molecolarmente continuo con il citoscheletro, metodi sono necessari per sondare il suo comportamento meccanico in cellule aderenti. Qui, discutiamo la sonda di forza diretta (DFP) come strumento per applicare la forza direttamente al nucleo in una cellula aderente vivente. Noi attribuiamo una micropipetta stretta per la superficie nucleare con aspirazione. La micropipetta è traslata lontano il nucleo, che fa sì che il nucleo di deformare e tradurre. Quando la forza di ripristino è uguale alla forza di aspirazione, il nucleo si stacca e rilassa elasticamente. Poiché la pressione di aspirazione è noto con precisione, la forza sulla superficie nucleare è noto. Questo metodo ha rivelato che le forze su scala nanometrica sono sufficienti per deformare e tradurre il nucleo in cellule aderenti e identificati gli elementi del citoscheletro che consentono il nucleo resistere alle forze. La DFP può essere utilizzata per sezionare i contributi di componenti cellulari e nucleare nucleare proprietà meccaniche in cellule viventi.
Introduzione
Patologie come il cancro coinvolgono alterazioni nucleare forma e struttura1,2, che sono generalmente accompagnati da un 'addolcimento' del nucleo3,4. Nucleare resistenza alla deformazione meccanica è stata caratterizzata generalmente applicando una forza di nuclei isolati5.
Il nucleo in cellule è molecolarmente collegato al citoscheletro dal Linker di Nucleoskeleton e citoscheletro (LINC) complesso6,7,8,9. Di conseguenza, il nucleo è integrato meccanicamente con il citoscheletro e, attraverso le adesioni cellula-substrato, la matrice extracellulare. Meccanicamente sondando il nucleo all'interno di cellule aderenti può fornire la comprensione in questa integrazione meccanica. Metodi per manipolare i nuclei in cellule viventi includono micropipetta aspirazione10,11e a forza atomica microscopia12,13,14. Recentemente abbiamo descritto una sonda di forza diretta (DFP) che si applica forze meccaniche direttamente sul nucleo in un vivente cellulari aderenti15.
Qui, descriviamo la procedura per l'utilizzo di un sistema di microiniezione che è comunemente disponibile in strutture di microscopia di applicare una forza meccanica di nano-scala nota, direttamente al nucleo in una cellula aderente. Un femtotip (punta della micropipetta diametro di 0,5 µm) è montato e collegato al sistema di microiniezione di un tubo. La punta, posizionata ad un angolo di 45° rispetto alla superficie della piastra di coltura, si abbassa fino a adiacente alla superficie nucleare. Il tubo viene quindi disconnesso e aperto all'atmosfera, che crea una pressione negativa di aspirazione sulla superficie nucleare e sigilla la punta della micropipetta contro la superficie nucleare. Attraverso la traduzione della punta della micropipetta, il nucleo è deformato e alla fine (a seconda della grandezza della forza applicata), staccato dalla micropipetta. Questo distacco si verifica quando le forze antagoniste (resiste), esercitate dal nucleo e cella, uguale la forza di aspirazione applicata dalla micropipetta. Analisi può essere effettuata misurando lo spostamento del nucleo, il ceppo di lunghezza (equazione 1), o il ceppo di zona (Figura 1A).
Protocollo
1. preparare le celle per l'Imaging
Nota: La sonda di forza diretta (DFP) può essere utilizzata per qualsiasi tipo di cellula aderente. Qui, fibroblasti NIH 3T3 del mouse sono usati come la linea cellulare modello per questo protocollo.
- Cellule di cultura NIH 3T3 del fibroblasto dell'Aquila per volta di Dulbecco Medium (DMEM) completati con 10% siero bovino di donatore e 1% penicillina-streptomicina su un fondo di 35 mm vetro piatto fino al confluency di desiderata. Mantenere le cellule a 37 ° C e 5% CO2.
- Assicurarsi di ricoprire tutti i piatti di fondo di vetro 35mm con 5 µ g/mL di fibronectina (o proteina simile ECM), prima della semina di cellule NIH 3T3 per l'imaging.
Nota: Le cellule devono essere pienamente diffuso e aderente sul piatto per l'esperimento. Non ci sono vincoli in termini di confluenza per utilizzare il metodo DFP.
- Assicurarsi di ricoprire tutti i piatti di fondo di vetro 35mm con 5 µ g/mL di fibronectina (o proteina simile ECM), prima della semina di cellule NIH 3T3 per l'imaging.
- Immediatamente prima dell'esperimento, lavare le cellule due volte con PBS, seguita da un lavaggio singolo con il mezzo di crescita completa.
- Aggiungere 3 mL di terreno di crescita completa il piatto di fondo di vetro.
2. microscopia e acquisizione di immagini
Nota: Un invertito fluorescenza Microscopio (o equivalente) con micromanipolatore installato per il braccio di lato, secondo le raccomandazioni del produttore. Il microscopio deve anche essere dotato di una camera climatica per mantenere la temperatura a 37 ° C e il livello di CO2 al 5%. È necessario anche un micromanipolatore e microinjector collegato al microscopio. Un olio immersione 40x / 1.3 NA o 60x / 1,49 NA (o obiettivi equivalenti) sono raccomandati per gli esperimenti. Il microscopio deve essere montato su un tavolo di isolamento delle vibrazioni.
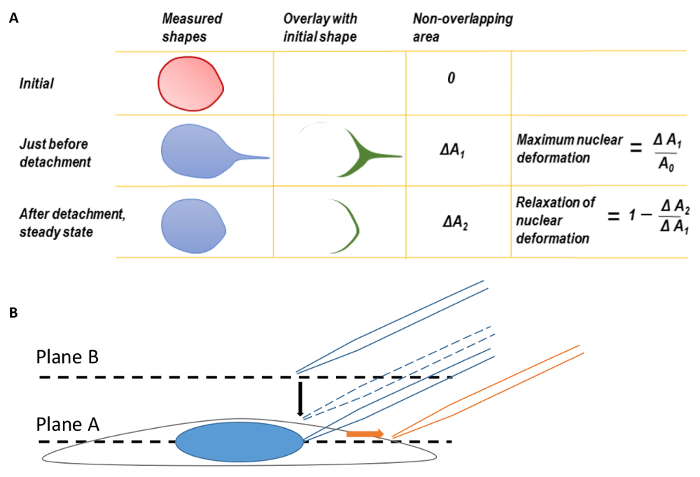
Figura 1 . Microscopio di messa a fuoco e di deformazione nucleare
A. massima deformazione nucleare e rilassamento della deformazione nucleare. Prima di calcolare la deformazione massima nucleare, i bordi posteriori delle forme nucleare erano in primo luogo ha coinciso per correggere per la traduzione del nucleo deformato. La forma del nucleo al momento del distacco di punta della micropipetta è stata sovrapposta la forma nucleare iniziale prima di tirare. La differenza nell'area tra le due forme è stata misurata come Δun1. La massima deformazione nucleare è stata definita come ΔA1 diviso per l'area nucleare originale. Allo stesso modo, un secondo parametro, ΔA2, può essere definito mediante la sovrapposizione della forma nucleare finale allo stato stazionario dopo distacco micropipetta della forma nucleare originale. B. lo stato attivo della cella al piano A e quindi spostare il piano focale fino a piano B per trovare la punta di una micropipetta. Durante la formazione immagine, la micropipetta è stato tradotto a destra (direzione della freccia arancione). Questa figura è stata modificata da Neelam et al. 15. Clicca qui per visualizzare una versione più grande di questa figura.
- Accendere il microinjector secondo il protocollo del produttore.
- Con un contagocce di olio di immersione, applicare una sola goccia di olio per immersione in cima la lente dell'obiettivo.
- Fissare saldamente il piatto nel titolare del piatto e caricare il titolare di piatto sul palco.
Nota: Le celle devono essere mantenute a 37 ° C e 5% di CO2 in tutto l'esperimento. - Regolare l'altezza dell'obiettivo di portare le cellule a fuoco (aereo A, Figura 1B).
- Spostare il tavolino del microscopio per trovare una cella di interesse.
- Ruotare il joystick sul micromanipolatore per spostare il supporto di dispensare alla posizione superiore. Caricare la micropipetta di punta di diametro 0,5 µm al supporto per dispensare.
- Per evitare l'adesione delle cellule per la micropipetta, pre-trattare la punta della micropipetta con 0,3 mg/mL di soluzione di PLL-g-PEG per 1 h a temperatura ambiente. Test per adesione toccando la micropipetta al nucleo senza alcuna pressione di aspirazione, e quindi tradurre la micropipetta dal nucleo. L'assenza di adesione possa essere individuata da una completa mancanza di deformazione nucleare e traduzione.
Nota: Si prega di seguire suggerimenti di fabbricazione per aprire il pacchetto.
- Per evitare l'adesione delle cellule per la micropipetta, pre-trattare la punta della micropipetta con 0,3 mg/mL di soluzione di PLL-g-PEG per 1 h a temperatura ambiente. Test per adesione toccando la micropipetta al nucleo senza alcuna pressione di aspirazione, e quindi tradurre la micropipetta dal nucleo. L'assenza di adesione possa essere individuata da una completa mancanza di deformazione nucleare e traduzione.
- Sollevare il piano focale obiettivo sopra A aereo e parte superiore della cella per piano B regolando il controllo fine (Figura 1B, vedere il punto 2.4).
- Impostare il micromanipolatore controllo grossolana . Portare lentamente la micropipetta giù per piano B guardando per la silhouette della micropipetta, fino a quando la micropipetta viene completamente messa a fuoco.
- Una volta che la punta della micropipetta è a fuoco, impostare il micromanipolatore controllo bene .
- Abbassare l'obiettivo per il piano equatoriale della cellula (aereo A, Figura 1B) e abbassare la micropipetta a circa 15 µm sopra piano A (Figura 1B, micropipetta tratteggiata).
- Impostare la pressione di compensazione (Pc) su microinjector alla pressione desiderata; attendere alcuni secondi per la pressione si stabilizzi.
Nota: Il set point di pressione ottimale dipende dal tipo di cellula sia gli obiettivi specifici dell'esperimento. Per la maggior parte dei casi, 300 hPa sarebbe un buon punto di partenza. - Garantire che la micropipetta non sia intasata utilizzando l'impostazione pulite sul pannello micromanipolatore e controllo per assicurarsi che le bolle d'aria emergere dalla punta della micropipetta.
- Inserire la punta nella cella abbassando gradualmente la micropipetta fino a quando la punta è leggermente toccando la superficie nucleare.
Nota: Quando si abbassa la micropipetta, la silhouette della punta della micropipetta diventerà chiara come essa viene messa a fuoco. Prima la micropipetta tocca il nucleo, sollevare l'obiettivo messa a fuoco e allineare la punta della micropipetta con nucleo (stessa coordinata x-y, z-piano superiore). Tornare il fuoco al piano equatoriale del nucleo (aereo A, Figura 1B) e abbassare gradualmente la punta della micropipetta. - Creare una guarnizione tra la punta della micropipetta e la membrana nucleare scollegando il tubo di alimentazione di pressione dal sistema di microiniezione, aprendo così l'estremità del tubo micropipetta all'atmosfera. Questo passaggio crea una pressione negativa uguale a Pc sulla superficie nucleare.
- Acquisire immagini con il software di raccolta immagini di microscopi. Impostare un'acquisizione avi (video) o nd-acquisizione (immagini) del software di raccolta di immagine.
Nota: Per qualsiasi imaging l'acquisizione di software, è istituito da formazione immagine video in tempo reale o acquisizioni di time-lapse immagine con un intervallo di tempo breve. - Attiva/disattiva per le fluorescenti corrispondente canale di imaging (cioè, GFP, RFP, ecc.) e iniziare la formazione immagine.
- Tradurre la punta della micropipetta lontano dal corpo della cella (a destra, Figura 1B) finché il nucleo non si stacca dalla micropipetta.
Nota: Tirare la punta lungo la direzione x positiva (a destra nel campo visivo). Il tasso di trazione può essere programmato e controllato da computer o il joystick può essere spostato manualmente. Non abbiamo trovato alcuna correlazione tra la trazione tasso e deformazione nucleare15 suggerendo una risposta prevalentemente elastica per forzare.
3. analisi dei dati
- Eseguire l'analisi di immagine con qualsiasi software di elaborazione delle immagini base disponibile. L'entità della deformazione nucleare può essere quantificata dal ceppo di lunghezza (Ɛ) o il ceppo di zona (Figura 1A). Quantificare il ceppo di lunghezza utilizzando l'equazione 1, dove L e L0 rappresentano le lunghezze del nucleo a deformazione massima e la posizione iniziale, rispettivamente.
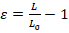 (Equazione 1)
(Equazione 1)
Risultati
La Figura 2A Mostra la forzatura di un nucleo di fibroblasto NIH 3T3 del mouse. Come la punta di una micropipetta è tradotto a destra, il nucleo si deforma e alla fine si stacca dalla punta della micropipetta. Il ceppo di lunghezza del nucleo è visto ad aumentare con l'aumento della forza di aspirazione (Figura 2B). Il bordo anteriore del nucleo (micropipetta tirando bordo) forma una protrusione nucleare ed il bordo d'uscita è...
Discussione
L'integrazione meccanica del nucleo con il citoscheletro di misura è una sfida per i metodi più attuali, ad esempio micropipetta aspirazione16, perché richiedono entrambi nuclei isolati (dove il nucleo è disaccoppiato dal citoscheletro) o nuclei in cellule sospese (dove le forze extracellulari, quali le forze di trazione, sono assenti). Vigore è stato applicato al nucleo applicando deformazione biassiale a aderente celle a una membrana17,18...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da NIH R01 EB014869.
Materiali
| Name | Company | Catalog Number | Comments |
| FluoroDish | WPI | FD35 | |
| SYTO 59 | ThermoFisher Scientific | S11341 | |
| Femtotips | Eppendorf | 930000043 | |
| InjectMan NI2 | Eppendorf | NA | discontinued, current equivalent model: InjectMan 4 |
| FemtoJet | Eppendorf | NA | Current model FemtoJet 4i |
| Plan Fluor oil immersion 40x | Nikon | NA | |
| Apo TIRF oil immersion 60x | Nikon | NA | |
| Donor Bovine Serum (DBS) | ThermoFisher Scientific | 16030074 | NIH 3T3 serum |
| Dulbecco's Modification of Eagle's (DMEM) | Mediatech cellgro | MT10013CVRF | NIH 3T3 medium |
| Penicillin-Streptomycin | Mediatech | MT30004CIRF | NIH 3T3 medium supplement |
| Immersion Oil Type LDF Non-Fluorescing | Nikon | 77007 | Immersion oil for objective lens |
Riferimenti
- Chow, K. H., Factor, R. E., Ullman, K. S. The nuclear envelope environment and its cancer connections. Nature Reviews Cancer. 12 (3), 196-209 (2012).
- Zink, D., Fischer, A. H., Nickerson, J. A. Nuclear structure in cancer cells. Nature Reviews Cancer. 4 (9), 677-687 (2004).
- Bank, E. M., Gruenbaum, Y. The nuclear lamina and heterochromatin: a complex relationship. Biochemical Society Transactions. 39 (6), 1705-1709 (2011).
- Lammerding, J., et al. Lamins A and C but not lamin B1 regulate nuclear mechanics. Journal of Biological Chemistry. 281 (35), 25768-25780 (2006).
- Dahl, K. N., Engler, A. J., Pajerowski, J. D., Discher, D. E. Power-law rheology of isolated nuclei with deformation mapping of nuclear substructures. Biophysical Journal. 89 (4), 2855-2864 (2005).
- Crisp, M., et al. Coupling of the nucleus and cytoplasm: role of the LINC complex. Journal of Cell Biology. 172 (1), 41-53 (2006).
- Sosa, B. A., Rothballer, A., Kutay, U., Schwartz, T. U. LINC complexes form by binding of three KASH peptides to domain interfaces of trimeric SUN proteins. Cell. 149 (5), 1035-1047 (2012).
- Tapley, E. C., Starr, D. A. Connecting the nucleus to the cytoskeleton by SUN-KASH bridges across the nuclear envelope. Current Opinion in Cell Biology. 25 (1), 57-62 (2013).
- Arsenovic, P. T., et al. Nesprin-2G, a Component of the Nuclear LINC Complex, Is Subject to Myosin-Dependent Tension. Biophysical Journal. 110 (1), 34-43 (2016).
- Rowat, A. C., Lammerding, J., Ipsen, J. H. Mechanical properties of the cell nucleus and the effect of emerin deficiency. Biophysical Journal. 91 (12), 4649-4664 (2006).
- Rowat, A. C., Foster, L. J., Nielsen, M. M., Weiss, M., Ipsen, J. H. Characterization of the elastic properties of the nuclear envelope. Journal of the Royal Society Interface. 2 (2), 63-69 (2005).
- Pagliara, S., et al. Auxetic nuclei in embryonic stem cells exiting pluripotency. Nature Materials. 13 (6), 638-644 (2014).
- Liu, H., et al. In situ mechanical characterization of the cell nucleus by atomic force microscopy. ACS Nanotechnology. 8 (4), 3821-3828 (2014).
- Krause, M., Te Riet, J., Wolf, K. Probing the compressibility of tumor cell nuclei by combined atomic force-confocal microscopy. Physical Biology. 10 (6), 065002 (2013).
- Neelam, S., et al. Direct force probe reveals the mechanics of nuclear homeostasis in the mammalian cell. Proceedings of the National Academy of Sciences of the United States of America. 112 (18), 5720-5725 (2015).
- Pajerowski, J. D., Dahl, K. N., Zhong, F. L., Sammak, P. J., Discher, D. E. Physical plasticity of the nucleus in stem cell differentiation. Proceedings of the National Academy of Sciences of the United States of America. 104 (40), 15619-15624 (2007).
- Lammerding, J., et al. Lamin A/C deficiency causes defective nuclear mechanics and mechanotransduction. Journal of Clinical Investigation. 113 (3), 370-378 (2004).
- Chancellor, T. J., Lee, J., Thodeti, C. K., Lele, T. Actomyosin tension exerted on the nucleus through nesprin-1 connections influences endothelial cell adhesion, migration, and cyclic strain-induced reorientation. Biophysical Journal. 99 (1), 115-123 (2010).
- Neelam, S., Dickinson, R. B., Lele, T. P. New approaches for understanding the nuclear force balance in living, adherent cells. Methods. 94, 27-32 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon