É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação da Saúde Mitocondrial em Fibroblastos Associados ao Câncer Isolados de Esferoides de Tumores Pulmonares Multicelulares 3D
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Esferoides tumorais 3D multicelulares foram preparados com células de adenocarcinoma pulmonar, fibroblastos e monócitos, seguidos pelo isolamento de fibroblastos associados ao câncer (CAFs) desses esferoides. CAFs isolados foram comparados com fibroblastos normais para avaliar a saúde mitocondrial estudando o potencial transmembranar mitocondrial, espécies reativas de oxigênio e atividades enzimáticas.
Resumo
Os fibroblastos associados ao câncer (CAFs) estão entre as células estromais mais abundantes presentes no microambiente tumoral, facilitando o crescimento e a progressão do tumor. A complexidade dentro do microambiente tumoral, incluindo secretoma tumoral, inflamação de baixo grau, hipóxia e desequilíbrio redox, promove a interação heterotípica e permite a transformação de fibroblastos residentes inativos para se tornarem CAFs ativos. Os CAFs distinguem-se metabolicamente dos fibroblastos normais (NFs), pois são mais glicoliticamente ativos, produzem níveis mais altos de espécies reativas de oxigênio (ROS) e superexpressam o exportador de lactato MCT-4, levando à abertura do poro de transição de permeabilidade mitocondrial (MPTP). Aqui um método foi descrito para analisar a saúde mitocondrial de CAFs ativados isolados dos esferoides tumorais 3D multicelulares compostos por células de adenocarcinoma pulmonar humano (A549), monócitos humanos (THP-1) e células de fibroblastos pulmonares humanos (MRC5). Os esferoides tumorais foram desintegrados em diferentes intervalos de tempo e, por meio da triagem celular ativada por magnetismo, os CAFs foram isolados. O potencial de membrana mitocondrial dos CAFs foi avaliado por meio do corante JC-1, da produção de ROS pela coloração de diacetato de 2',7'-diclorodihidrofluoresceína (DCFDA) e da atividade enzimática nos CAFs isolados. A análise da saúde mitocondrial de CAFs isolados fornece uma melhor compreensão do efeito Warburg reverso e também pode ser aplicada para estudar as consequências das alterações mitocondriais do CAF, como fluxos metabólicos e os mecanismos regulatórios correspondentes na heterogeneidade do câncer de pulmão. Assim, o presente estudo defende a compreensão das interações tumor-estroma na saúde mitocondrial. Ele forneceria uma plataforma para verificar os candidatos a medicamentos específicos mitocondriais quanto às suas eficácias contra CAFs como terapêuticas potenciais no microambiente tumoral, evitando assim o envolvimento do CAF na progressão do câncer de pulmão.
Introdução
Os tumores sólidos são compostos por populações celulares heterogêneas que são guiadas pelo microambiente tumoral (EMT), no entanto, a origem da maioria das células ainda não foi descoberta. Principalmente células estromais e imunes (fibroblastos, células endoteliais, monócitos, macrófagos, células dendríticas, células B, células T e seus subconjuntos) refletem a heterogeneidade tumoral em câncer de pulmão, mama, renal e outros cânceres sólidos 1,2,3. Compreender a origem de cada subtipo e seu potencial de transdiferenciação é de extrema necessidade para o desenvolvimento de terapias avançadas contra esses tipos de câncer. A análise dessa população celular diversificada em biópsias humanas apresenta-se com diversos desafios devido ao tipo de tumor, localização, estágio, limitação da quantidade de amostra e variabilidades específicas do paciente4. Assim, é necessário um modelo experimental, que não seja apenas confiável, mas também possa simular a condição tumoral in vivo, mostrando-se ideal para o estudo do crosstalk tumor-estroma e seu envolvimento na fisiopatologia da doença.
As culturas tridimensionais (3D) de tumores multicelulares esferoides (MCTS) são um sistema modelo in vitro vantajoso de tumores devido à sua semelhança com contrapartes naturais. O MCTS pode replicar melhor aspectos de tumores sólidos do que os modelos de cultura de células 2D, incluindo sua arquitetura espacial, respostas fisiológicas, liberação de mediadores solúveis, padrões de expressão gênica e mecanismos de resistência a medicamentos. Além disso, uma das principais vantagens do MCTS é que ele pode ser usado para estudar a heterogeneidade tumoral e o microambiente tumoral (EMT). O método hanging-drop é a ferramenta mais comumente empregada para desenvolver e analisar o MCTS5. Neste método, as diferentes células com meios são suspensas na forma de gotículas, o que permite o seu crescimento de forma agregada 3D coerente e é de fácil acesso para exame. A técnica é simples; não requer muitas células e elimina a necessidade de um substrato especializado como a agarose para o desenvolvimento esferoide6. Uma vantagem adicional deste método reside na reprodutibilidade de sua técnica. Além disso, esse método também tem sido utilizado para co-cultivar populações de células mistas, como células endoteliais e células tumorais, para simular a angiogênese tumoral precoce7.
Neste estudo, esferoides de tumores pulmonares 3D multicelulares foram preparados com células de adenocarcinoma pulmonar, fibroblastos e monócitos usando o método de gota suspensa que imita o microambiente do tumor pulmonar. Em seguida, a população de fibroblastos associados ao câncer (CAF) foi isolada para investigar a saúde mitocondrial. A ideia principal por trás do desenvolvimento desses esferoides é isolar os CAFs, pois o crosstalk entre as células em esferoides poderia transformar os fibroblastos em um estado CAF ativado semelhante a um miofibroblasto. Em segundo lugar, este estudo também pode descrever como a produção aberrante de ERO e a disfunção mitocondrial impulsionam os fibroblastos normais em direção ao fenótipo CAF mais agressivo. Verificou-se que fibroblastos montados dentro de esferoides tumorais ganharam características miofibroblásticas com aumento da atividade das EROs e indução da expressão gênica metabólica. Este protocolo destaca a importância do microambiente tumoral na ativação da CAF e pode ser um excelente modelo para geração in vitro e estudo das características fenotípicas da CAF.
Protocolo
1. Cultura celular
- Cultura de adenocarcinoma pulmonar humano da linhagem celular A549 e linhagem celular monocítica humana THP-1 em meio RPMI1640 suplementado com 10% de FBS e 1% de penicilina-estreptomicina a 37 °C em uma câmara umidificada com 5% de CO2.
- Cultura de células fibroblásticas pulmonares humanas MRC-5 em meio DMEM suplementadas com FBS a 10% e solução de penicilina-estreptomicina a 37 °C em câmara umidificada com CO2 a 5%.
2. Preparação de esferoides tumorais multicelulares utilizando linhagem celular de adenocarcinoma pulmonar A549, fibroblastos MRC5 e monócitos THP-1
NOTA: Os esferoides 3D tumorigênicos e não tumorigênicos multicelulares foram preparados pelo método da gota suspensa em um prato de cultura celular de 90 mm. Uma descrição detalhada do desenvolvimento desses esferoides é dada abaixo. Todos os reagentes da cultura celular, como meio completo, PBS e solução de tripsina-EDTA a 0,25%, devem ser pré-aquecidos a 37 °C antes do uso, salvo indicação em contrário.
- Preparação da suspensão celular
- Cultivar células aderentes A549 e MRC5 (5 x 106 células cada) em meio de crescimento completo DMEM (meio águia modificado de Dulbecco [DMEM] + 10% de soro fetal bovino [FBS] + 1% de penicilina-estreptomicina) em frascos T25 a 37 °C em câmara umidificada com 5% de CO2.
- Para as células THP-1, cultivar a suspensão celular (5 x 106 células) em meio de crescimento completo (RPMI1640 + 10% FBS + 1% penicilina-estreptomicina) em frascos T25. Para um melhor crescimento, colocar os frascos T25 a 37 °C numa câmara humidificada com 5% de CO2 em posição de pé.
- Após 3 dias, com 80%-85% de confluência, lavar as células A549 e MRC5 com PBS livre de cálcio e magnésio (solução salina tampão fosfato) adicionando 1 mL de PBS (25-30 °C) em cada frasco por 1 min e aspirando-o.
- Colher as células A549 e MRC5 do balão incubando-as com 500 μL de solução de tripsina-EDTA a 0,25% durante 5 min a 37 °C numa incubadora humidificada com 5% de CO2. Imediatamente após, adicione 4 mL de meio de crescimento completo para inativar a tripsina.
- Recolher a suspensão celular do balão T25 num tubo de 15 ml e repeltá-la a 125 x g durante 5 minutos. Remova o sobrenadante e ressuspenda as células em 5 mL de meio de crescimento completo.
- Estabelecimento de esferoides tumorais multicelulares
NOTA: Todas as etapas da formação de esferoides tumorais devem ser realizadas dentro do gabinete de biossegurança, a fim de manter condições estéreis. O volume máximo da suspensão celular foi padronizado como uma gota de 25 μL para preparar uma gota de tal forma que ela não caia durante a inversão das tampas.- Conte os números de células A549, MRC5 e THP-1 usando um contador de células. Para cada gotícula esferoide (25 μL) mantenha os seguintes números celulares: 5.000 células A549, 4.000 células MRC5 e 1.000 células THP-1, seguindo o protocolo descrito por Arora et al.7. Calcule os números de células de acordo para um volume de 1 mL.
NOTA: Os esferoides foram inicialmente preparados com três contagens de células diferentes por esferoide (ou seja, 5000, 8000 e 10.000). Além disso, diferentes proporções celulares de células tumorais/fibroblastos/monócitos (1:1:1, 2:2:1, 4:2:1, 5:2:1 e 5:4:1) foram verificadas. Finalmente, uma formação esferoide multicelular 3D bem-sucedida foi observada com uma proporção de 5:4:1 e foi usada para o estudo. A concentração de fibroblastos foi aumentada com base em sua proporção no microambiente tumoral, o que aumentou ainda mais a rigidez dos esferoides tumorais. O procedimento detalhado foi relatado em publicação recente de Arora et al.7. - Preparar a suspensão celular numa proporção de 5 (A549, 2 x 10 5 células/ml) para 4 (MRC5, 1,6 x 10 5 células/ml) para 1 (THP-1,5 x 104 células/ml) e completar o volume para 1 ml com DMEM completo.
- Coloque uma gota de 25 μL de mistura de suspensão celular na tampa de um prato de cultura de 90 mm (aproximadamente 50 gotas/prato de 90 mm). Encha o fundo do prato de cultura de 90 mm com 10 mL de água estéril.
- Inverta cuidadosamente a tampa sobre a câmara de hidratação cheia de água e coloque o prato em uma incubadora de cultura de células por 3 dias.
- Monitore os esferoides sob o microscópio com uma ampliação de 10x no dia 4. Para adquirir imagens, ligue o interruptor de energia, coloque o prato de 60 mm cuidadosamente no palco e selecione a ampliação (10x). Ajuste as lentes e examine as células para analisar a agregação e proliferação celular. Pressione os botões Congelar e Salvar fornecidos no microscópio para capturar a imagem.
- Altere o meio de crescimento no dia 4 aspirando cuidadosamente 20 μL de meio de cada gota e substituindo-o por um meio de crescimento completo fresco.
- Conte os números de células A549, MRC5 e THP-1 usando um contador de células. Para cada gotícula esferoide (25 μL) mantenha os seguintes números celulares: 5.000 células A549, 4.000 células MRC5 e 1.000 células THP-1, seguindo o protocolo descrito por Arora et al.7. Calcule os números de células de acordo para um volume de 1 mL.
3. Análise de mortos-vivos de esferoides tumorais
- Nos dias 7 e 10, inverta cuidadosamente o prato de 90 mm no armário de biossegurança e use uma pipeta de 200 μL para coletar esferoides de cada gota. Colete cinco esferoides cada um em um tubo de 1,5 mL.
- Adicionar 500 μL de 1x PBS ao tubo de 1,5 mL contendo esferoides e centrífuga a 125 x g por 5 min. Descarte o sobrenadante cuidadosamente e ressuspenda os esferoides em 200 μL de 1x PBS. Não pipetar rigorosamente para evitar a desintegração esferoide.
- Pipetar os esferoides utilizando uma pipeta de 200 μL numa placa de 60 mm para coloração de calceína-AM e iodeto de propídio.
- Colocar 5 μL de solução de calceína-AM de 1 μM e 5 μL de solução de iodeto de propídio a 2 mg/mL nos esferoides. Incubar por 10 min. Após a conclusão do período de incubação, lave os esferoides suavemente com 1x PBS duas vezes.
- Coloque a placa de 60 mm contendo esferoides sob o microscópio invertido fluorescente, observe e capture imagens com ampliação de 10x, selecionando a opção de fluorescência no software do microscópio e selecionando o FITC (canal de fluorescência verde; excitação 490 nm, emissão 515 nm) para calceína e canal vermelho Texas (TXR; excitação 535 nm, emissão 617 nm).
- Para adquirir a imagem, ligue o Ctr Adv, que é o switch 1, seguido de ligar a CPU. Aguarde até que o sistema de software inicialize. Quando inicializado, coloque o prato de 60 mm cuidadosamente no palco e selecione a ampliação (10x) e os canais fluorescentes (FITC, TXR). Ajuste as lentes e digitalize a imagem.
- Para visualizar a imagem no sistema, selecione a opção Live e visualize a imagem. Ajuste a intensidade da fluorescência para otimização apropriada. Salve a imagem clicando no botão Salvar .
NOTA: Nesta fase, os esferoides serão visíveis a olho nu. Sob o microscópio de luz, os esferoides aparecerão como esferas redondas e rígidas com ampliação de 10x. Vários esferoides podem ser coletados de uma só vez usando uma pipeta de 200 μL.
4. Desintegração e suspensão celular de esferoides tumorais
- Coletar 200 esferoides tumorais cada um nos dias 7 e 10, usando uma pipeta de 1 mL em um tubo de 15 mL.
NOTA: Antes da recolha, verifique cuidadosamente a forma e a forma de um único esferoide depois de o transferir para uma lâmina microscópica ou para uma placa de 30 mm e observe ao microscópio. - Pellet os esferoides por centrifugação a 125 x g durante 5 min. Aspirar o sobrenadante cuidadosamente sem perturbar os esferoides tumorais.
- Lave cuidadosamente os esferoides com 200 μL de PBS, centrifugar a 125 x g durante 5 min e eliminar cautelosamente o sobrenadante.
- Para a desintegração esferoide, adicionar 400 μL de solução de tripsina-EDTA a 0,25% e manter a 37 °C durante 10 minutos. Realize pipetagem vigorosa para a completa desintegração dos esferoides.
- Neutralizar a tripsina adicionando 1 mL de meio de crescimento DMEM completo. Centrifugar a 125 x g durante 5 min e eliminar cuidadosamente o sobrenadante.
- Ressuscite o pellet em 1 mL de meio DMEM completo e conte o número total de células.
5. Isolamento de fibroblastos associados ao câncer (CAF) através de microesferas
- Para o isolamento de CAFs dos esferoides tumorais, ressuspender 1 x 10 7 células em 80 μL de tampão de classificação de células ativadas magnéticas a frio (MACS) (PBS a pH7,2 contendo 0,5% de albumina sérica bovina [BSA] e ácido etilenodiamina tetraacético [EDTA] a 2 mM).
- Incubar a suspensão celular (contendo 1 x 107 células) com 20 μL de microesferas antifibroblásticas. Misture bem batendo suavemente no tubo e incube por 30 minutos à temperatura ambiente.
- Lavar as células com 1 mL de tampão MACS frio, centrifugar a 125 x g por 5 min e aspirar o sobrenadante. Ressuspenda as células em 500 μL de buffer MACS.
- Para a separação celular à base de grânulos magnéticos, prepare a coluna MACS enxaguando-a com 3 mL de tampão MACS.
- Coloque a suspensão celular na coluna seguida da coleta de fluxo contendo a população celular não rotulada.
- Lave a coluna três vezes com 3 mL de tampão MACS. Remova a coluna do separador e coloque-a no tubo de coleta.
- Colete as células marcadas com microesferas antifibroblastos adicionando 5 mL de tampão MACS e empurrando firmemente o êmbolo para a coluna.
- Centrifugar as células marcadas a 125 x g durante 5 minutos. Prossiga com os fibroblastos isolados para aplicações a jusante.
6. Análise baseada em citometria de fluxo da expressão de ACTA2 em CAFs isolados
- Conte o número de CAFs isolados e use aproximadamente 6 x 104 células. Lave as células uma vez com PBS, centrifugar a 125 x g por 5 min e aspirar o sobrenadante.
- Adicionar 100 μL de tampão de permeabilização celular (PBS + 0,5% BSA + 0,3% v/v Triton X-100) e incubar as células a 4 °C por 30 min.
- Vórtice as células intermitentemente para manter uma única suspensão celular. Centrifugar e ressuspender as células em 100 μL de tampão de permeabilização celular.
- Adicionar 2 μL de anticorpo α-SMA humano conjugado com APC e incubar a 4 °C durante 45 min. Após a incubação, adicione 1 mL de tampão de permeabilização e centrifugar a 125 x g por 5 min para remover o excesso de anticorpo.
- Ressuspender o pellet em 400 μL de tampão de permeabilização para análise citométrica de fluxo. Adquira um total de 10.000 eventos de cada amostra em um citômetro de fluxo. Selecione as células positivas para ACTA2 depois de distinguir as populações de células com base em sua dispersão frontal e lateral, selecionando a população singleta seguida da seleção das células positivas para ACTA2 que aparecem como um único pico no histograma de parâmetro único.
7. Coloração JC-1 para determinar o potencial de membrana mitocondrial
- Contar o número de CAFs isolados e utilizar aproximadamente 6 x 104 células para coloração de iodeto de 5,5,6,6'-tetracloro-1,1,1',3,3' tetraetilbenzimi-dazoilcarbocianina (JC-1) utilizando citometria de fluxo.
- Lave bem as células com PBS, centrifugar a 125 x g por 5 min, aspirar o sobrenadante e adicionar 100 μL de tampão de coloração celular.
- Adicionar o corante JC-1 a uma concentração de trabalho de 2 μM e incubar à temperatura ambiente durante 30 min. No final da incubação, lavar as células com PBS, centrifugar a 125 x g por 5 min e ressuspender em um volume final de 400 μL.
- Adquira um total de 20.000 eventos de cada amostra em um citômetro de fluxo. Quantifique o potencial da membrana mitocondrial medindo os agregados JC-1 deslocados para o vermelho no canal FL-2 e os monômeros deslocados para o verde no canal FL-1.
8. Coloração DCFDA para estimar os níveis de espécies reativas celulares de oxigênio (ROS)
- Use os esferoides inteiros (50 números), bem como os CAFs isolados (6 x 104 células) dos esferoides para coloração de diacetato de 2',7'-diclorodihidrofluoresceína (DCFDA).
- Lave bem as células com PBS, centrifugar a 125 x g por 5 min, aspirar o sobrenadante e adicionar 100 μL de tampão de coloração celular.
- Adicionar o corante DCFDA a uma concentração de trabalho de 1 μM e incubar à temperatura ambiente durante 30 min. Lavar as células duas vezes com PBS, centrifugar a 125 x g durante 5 min e, em seguida, ressuspender num volume final de 400 μL.
- Adquira um total de 20.000 eventos de cada amostra em um citômetro de fluxo. Avalie os níveis de ROS medindo a fluorescência no dia 7 e no dia 10 para os esferoides, bem como os CAFs isolados.
9. Análise RT-qPCR de marcadores CAF e genes glicolíticos
- Extraia o RNA dos CAFs classificados usando um kit de lise de célula única seguindo o protocolo do fabricante. Prepare cDNA a partir de 100 ng de RNA usando um kit de síntese de cDNA seguindo as diretrizes do fabricante.
- Realizar RT-qPCR para analisar a expressão relativa dos marcadores CAF (ACTA2 8 e COL1A29) e do gene glicolítico (GLUT1 10 e MCT4 11). Realizar análise de curva de fusão após a extensão final para garantir a especificidade dos produtos. Normalize os dados usando a expressão de GAPDH como o gene de referência. As sequências de primer utilizadas para RT-qPCR estão listadas na Tabela Suplementar 1.
10. Extração e quantificação da proteína celular a partir de CAFs
NOTA: Execute todas as etapas de extração de proteínas no gelo para evitar a degradação da proteína.
- Resuspender aproximadamente 4 x 106 células CAF em 100 μL de tampão de lise RIPA gelado (contendo 30 mM HEPES, 150 mM NaCl, 1% NP-40, 0,1% SDS e 0,5% desoxicolato de sódio com 5 mM de coquetel de inibidor de protease e fosfatase Halt e 5 mM de EDTA em pH 7,4).
- Vórtice rigorosamente para lise celular adequada. Sonicate a suspensão celular na frequência de 20 Hz, amplitude de 20% para 15 s e pulso de 3x.
- Após a sonicação, centrifugar o extrato de proteína a 13.000 x g por 15 min a 4 °C. Transfira o sobrenadante para um tubo de 1,5 mL pré-resfriado. Realize o ensaio de proteína BCA para quantificar a proteína.
11. Análise espectrofotométrica das atividades enzimáticas em CAFs
NOTA: As seguintes atividades enzimáticas são analisadas em CAFs derivados de esferoides tumorais.
- Medição da atividade da succinato desidrogenase
- Para avaliar a atividade da succinato desidrogenase (SDH) em CAFs, preparar tampão SHE com sacarose 250 mM, ácido 4-(2-hidroxietil)-1-piperazineetanossulfônico (HEPES) 1 mM e 1 mM de etilenoglicol-bis(éter β-aminoetílico)-N,N,N′,N′-tetraacético (EGTA) e ajustar o pH para 7,3. Adicionar 0,4 mM de metossulfato de fenazina (PMS), 0,2 mM 2,6-dicloroindofenol (DCPIP), 50 mM MgCl2, 0,02% Triton X-100 e 1 mM de cianeto no tampão SHE e manter a 37 °C.
- Extraia a proteína celular dos CAFs conforme descrito na etapa 10 (2 x 106 células). Em cada poço de uma placa de 96 poços, incubar 100 μL de 0,1 mg de proteína celular com 100 μL de mistura de reação SHE a 37 °C por 15 min.
- Para iniciar a atividade enzimática, adicione succinato 12 mM10. Calcular a actividade enzimática em nM/min/ml medindo as alterações na absorbância a 600 nm.
- O fator de conversão para DCIP é de 0,0215 A/nM com base em seu coeficiente de extinção molar12. Calcule a atividade relativa dos DSS usando a fórmula mencionada:
Atividade relativa (nM/min/mL/enzima) =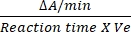 X X
X X  V
V
Onde ΔA/min = taxa de reação enzimática (A inicial- A final)/(tempofinal- A/mininicial), Ve = volume da amostra e V = volume da reação.
- Estimativa da atividade da citocromo c oxidase
- Para avaliar a atividade da citocromo c oxidase (COX) em CAFs, adicionar 0,2 mg/mL de proteína celular em uma solução tampão de reação KME contendo tampão KME (125 mM KCl, 20 mM 3-(N-morpholino)ácido sulfônico de propano [MOPS] e 1 mM EGTA, pH 7,4), 0,02 % Triton X-100 e 5 mM de ascorbato de sódio.
- Num tubo separado, misturar o citocromo c cardíaco de cavalo 50 μM com ascorbato de sódio 5 mM e incubar a 37 °C durante 5 min13. Após a incubação, adicionar 20 μL de citocromo c reduzido a 500 μL de solução tampão de reação KME contendo proteína celular.
- Calcular a actividade enzimática em unidades/μL medindo as alterações na absorvância a 550 nm. A absorção do citocromo c a 550 nm muda com o seu estado de oxidação. A diferença nos coeficientes de extinção (mM) entre citocromo c reduzido e oxidado é de 21,84 a 550 nm14. Calcule a quantidade de atividade enzimática no lisado celular como:
Unidades/μL =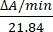
Onde ΔA/min = A/minamostra-A/minem branco e 21,84 = ΔӑmM entre citocromo c oxidado e citocromo c reduzido a 550 nm
- Avaliação da atividade da lactato desidrogenase
- Realize um ensaio de lactato desidrogenase (LDH) usando um kit de ensaio de células de lactato desidrogenase seguindo as instruções do fabricante.
- Adicionar o reagente de teste LDH (10 μL) a 0,5 mg/mL de proteínas celulares e incubar por 5 min a 37 °C. No final da incubação, medir a absorvância a 45 nm a cada 1 min durante 8 min.
- Preparar os padrões NADH para detecção colorimétrica usando 0 (em branco), 2,5, 5, 7,5, 10 e 12,5 nM/poço de NADH seguido pela adição de reagente de teste LDH a um volume final de 50 μL.
- Plote as diferenças de absorvância entre Tinicial e Tfinal para a curva padrão NADH para determinar a quantidade de geração de NADH. Avalie a atividade da LDH usando a fórmula mencionada:
Atividade LDH =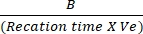 X fator de diluição da amostra
X fator de diluição da amostra
Onde B = quantidade (nmole) de NADH gerada entre T inicial e T final, tempo de reação = Tfinal - Tinicial (min) e Ve = volume da amostra (mL) adicionado ao poço.
Resultados
A Figura 1 mostra o desenvolvimento de esferoides tumorais multicelulares utilizando três populações celulares diferentes - A549 (adenocarcinoma pulmonar), MRC-5 (fibroblastos) e THP-1 (monócitos) - pelo método da queda suspensa, conforme observado no dia 7 e no dia 10 ao microscópio. No 7º dia, os esferoides eram compactos e rígidos, com 260 ± 5,3 μm de diâmetro, e no 10º dia, os esferoides tinham 480 ± 7,5 μm de diâmetro (Figura 1A no painel sup...
Discussão
O presente estudo apresenta o desenvolvimento de esferoides tumorais multicelulares compreendendo células tumorais, população de células estromais (isto é, fibroblastos) e população de células imunes (ou seja, monócitos) usando um método modificado de gota suspensa. Fibroblastos e monócitos/macrófagos estão entre as populações mais significativas que constituem o microambiente tumoral (EMT), e sua presença está frequentemente ligada ao mau prognóstico do paciente16. Quando presen...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Este trabalho foi apoiado pelo SERB-Women Excellence Award Project, Índia (SB/WEA-02/2017) e pelo SERB-Early Career Research Award Project, Índia (ECR/2017/000892) para a DP. Os autores, LA e SR reconhecem o IIT Ropar e o MHRD por suas bolsas de pesquisa. MK reconhece o ICMR por sua bolsa de pesquisa.
Materiais
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC anti-human α-SMA | R&D systems | Cat# IC1420A | |
| Anti-fibroblast microbeads | Miltenyi Biotec | Cat# 130-050-601 | |
| Cell lines | |||
| A549 lung adenocarcinoma cells | NCCS Pune | - | |
| MRC-5 fetal lung fibroblasts | ATCC | CCL-171 | |
| THP-1 Human monocytes | NCCS Pune | - | |
| Chemicals | |||
| BSA | Himedia | Cat# 9048-46-8 | |
| 2,6-dichloroindophenol (DCPIP) | SRL | Cat# 55287 | |
| Calcein-AM | Thermo Fisher Scientific | Cat# C3099 | |
| DAPI | Thermo Fisher Scientific | Cat# D1306 | |
| DCFDA | Sigma | Cat# D6883 | |
| DMEM | Gibco | Cat# 11995073 | |
| DPBS | Gibco | Cat# 14190-144 | |
| EDTA | Thermo fisher scientific | Cat# 17892 | |
| EGTA | SRL | Cat# 62858 | |
| EZcoun Lactate Dehydrogenase Cell Assay Kit | HiMedia | Cat# CCK036 | |
| FBS | Gibco | Cat# 10082147 | |
| Halt Protease and Phosphatase Inhibitor Cocktail (100X) | Thermo Fisher Scientific | Cat# 87786 | |
| HEPES | Thermo Fisher Scientific | Cat# 15630080 | |
| Horse heart Cytochrome c | SRL | Cat# 81551 | |
| Image-iT Red hypoxia reagent | Thermo Fisher Scientific | Cat# H10498 | |
| JC-1 Dye | Thermo Fisher Scientific | Cat# T3168 | |
| KCl | Merck | Cat# P9541 | |
| MgCl2 | Merck | Cat# M8266 | |
| MOPS | Thermo Fisher Scientific | Cat# 69824 | |
| Nacl | Sigma-Aldrich | Cat# S9888 | |
| NADH MB Grade | SRL | Cat# 54941 | |
| NP-40 | Thermo Fisher Scientific | Cat# 85124 | |
| Penicillin/Streptomycin | Gibco | Cat# 15140122 | |
| Phenazine methosulfate (PMS) | SRL | Cat# 55782 | |
| Propidium iodide | Thermo fisher scientific | Cat# P1304MP | |
| RPMI 1640 | Gibco | Cat# 11875093 | |
| Single Cell Lysis Kit | Thermo Fisher Scientific | Cat# 4458235 | |
| Sodium ascorbate | Merck | Cat# A7631 | |
| Sodium cyanide | Sigma | Cat# 205222 | |
| Sodium Deoxycholate | Thermo Fisher Scientific | Cat# 89904 | |
| Sodium dodecyl sulphate | Sigma-Aldrich | Cat# L3771 | |
| Sodium succinate hexahydrate | SRL | Cat# 36313 | |
| Sucrose | Sigma | Cat# S0389 | |
| SuperScript VILO cDNA synthesis kit | Thermo Fisher Scientific | Cat# 11754-050 | |
| Triton X-100 | Sigma | Cat# T8787 | |
| Trypsin 0.25% EDTA | Gibco | Cat# 25200072 | |
| Universal SYBR Green Supermix | BIO-RAD | Cat# 172-5124 | |
| Plasticware | |||
| MACS LS Columns | Miltenyi Biotec | Cat# 130-042-401 | |
| Equipment | |||
| Countess II FL Automated Cell Counter | Thermo Fisher Scientific | Cat# AMQAF1000 | |
| EVOS XL core imaging system | Thermo Fisher Scientific | Serial Number F0518-1727-0191 | |
| LAS X software | Leica Microsystems | ||
| Leica fluorescent inverted microscope | s | DMi8 automated S/N 424150) | |
| Midi MACS separator | Miltenyi Biotec | Cat# 130-042-302 |
Referências
- Kim, N., et al. Single-cell RNA sequencing demonstrates the molecular and cellular reprogramming of metastatic lung adenocarcinoma. Nature Communications. 11 (1), 1-5 (2020).
- Davidson, S., et al. Single-cell RNA sequencing reveals a dynamic stromal niche that supports tumor growth. Cell Reports. 31 (7), 107628 (2020).
- Zhang, Y., et al. Single-cell analyses of renal cell cancers reveal insights into tumor microenvironment, cell of origin, and therapy response. Proceedings of the National Academy of Sciences. 118 (24), (2021).
- Bray, L. J., Hutmacher, D. W., Bock, N. Addressing patient specificity in the engineering of tumor models. Frontiers in Bioengineering and Biotechnology. 7, 217 (2019).
- Timmins, N. E., Nielsen, L. K. Generation of multicellular tumor spheroids by the hanging-drop method. Tissue Engineering. , 141-151 (2007).
- Dituri, F., et al. Complex tumor spheroid formation and one-step cancer-associated fibroblasts purification from hepatocellular carcinoma tissue promoted by inorganic surface topography. Nanomaterials. 11 (12), 3233 (2021).
- Arora, L., et al. Development of a multicellular 3D tumor model to study cellular heterogeneity and plasticity in NSCLC tumor microenvironment. Frontiers in Oncology. 12, 881207 (2022).
- Nurmik, M., Ullmann, P., Rodriguez, F., Haan, S., Letellier, E. In search of definitions: Cancer-associated fibroblasts and their markers. International Journal of Cancer. 146 (4), 895-905 (2020).
- Zhang, Y., et al. HIF-1α is necessary for activation and tumour-promotion effect of cancer-associated fibroblasts in lung cancer. Journal of Cellular and Molecular Medicine. 25 (12), 5457-5469 (2021).
- Bu, L., et al. Biological heterogeneity and versatility of cancer-associated fibroblasts in the tumor microenvironment. Oncogene. 38 (25), 4887-4901 (2019).
- Whitaker-Menezes, D., et al. Evidence for a stromal-epithelial "lactate shuttle" in human tumors: MCT4 is a marker of oxidative stress in cancer-associated fibroblasts. Cell cycle. 10 (11), 1772-1783 (2011).
- Mandujano-Tinoco, E. A., Gallardo-Pérez, J. C., Marín-Hernández, A., Moreno-Sánchez, R., Rodríguez-Enríquez, S. Anti-mitochondrial therapy in human breast cancer multi-cellular spheroids. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research. 1833 (3), 541-551 (2013).
- Bregman, A. A. . Laboratory Investigations in Cell and Molecular Biology. , (2002).
- Berry, E. A., Trumpower, B. L. Simultaneous determination of hemes a, b, and c from pyridine hemochrome spectra. Analytical Biochemistry. 161 (1), 1-15 (1987).
- Avagliano, A., et al. Metabolic reprogramming of cancer associated fibroblasts: the slavery of stromal fibroblasts. BioMed Research International. , (2018).
- Lorusso, G., Rüegg, C. The tumor microenvironment and its contribution to tumor evolution toward metastasis. Histochemistry and Cell Biology. 130 (6), 1091-1103 (2008).
- Liu, T., Zhou, L., Li, D., Andl, T., Zhang, Y. Cancer-associated fibroblasts build and secure the tumor microenvironment. Frontiers in Cell and Developmental Biology. 7, 60 (2019).
- Sebastian, A., et al. Single-cell transcriptomic analysis of tumor-derived fibroblasts and normal tissue-resident fibroblasts reveals fibroblast heterogeneity in breast cancer. Cancers. 12 (5), 1307 (2020).
- Elyada, E., et al. Cross-species single-cell analysis of pancreatic ductal adenocarcinoma reveals antigen-presenting cancer-associated fibroblasts. Cancer Discovery. 9 (8), 1102-1123 (2019).
- Ganguly, D., et al. Cancer-associated fibroblasts: Versatile players in the tumor microenvironment. Cancers. 12 (9), 2652 (2020).
- Harryvan, T. J., Verdegaal, E. M., Hardwick, J. C., Hawinkels, L. J., vander Burg, S. H. Targeting of the cancer-associated fibroblast-T-cell axis in solid malignancies. Journal of Clinical Medicine. 8 (11), 1989 (2019).
- Santi, A., Kugeratski, F. G., Zanivan, S. Cancer associated fibroblasts: the architects of stroma remodeling. Proteomics. 18 (5-6), 1700167 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados