É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Determinando a estabilidade sérica da enzima adenosina desaminase 1 humana
Neste Artigo
Resumo
Neste artigo, detalhamos métodos para caracterizar a capacidade de uma enzima de reter a função quando incubada a 37 ° C no soro humano, uma propriedade farmacológica conhecida como estabilidade sérica. Essa capacidade pode ser um fator chave na previsão do perfil farmacocinético de uma enzima e sua adequação para uso terapêutico.
Resumo
O conceito de estabilidade enzimática é normalmente usado para se referir à termoestabilidade de uma enzima - sua capacidade de reter estrutura e atividade à medida que a temperatura aumenta. Para uma enzima terapêutica, outras medidas de estabilidade também podem ser críticas, particularmente sua capacidade de reter a função no soro humano a 37 ° C, que chamamos de estabilidade sérica. Aqui, descrevemos um ensaio in vitro para avaliar a estabilidade sérica da enzima Homo sapiens adenosina desaminase I (HsADA1) do tipo selvagem usando um procedimento de microplaca baseado em absorbância. Especificamente, este manuscrito descreve a preparação de tampões e reagentes, um método que organiza a coincubação de HsADA1 no soro, um método para analisar as amostras de teste usando um leitor de microplacas e uma análise de acompanhamento para determinar a fração de atividade que uma enzima HsADA1 retém no soro em função do tempo. Discutimos ainda considerações para adaptar este protocolo a outras enzimas, usando um exemplo de uma enzima Homo sapiens quinurenase, para ajudar na adaptação do protocolo a outras enzimas onde a estabilidade sérica é de interesse.
Introdução
O método a seguir permite que um usuário avalie quantitativamente a capacidade de uma enzima de reter sua atividade quando exposta a condições que imitam o que ela encontrará após a injeção intravenosa. O método in vitro imita essas condições in vivo e consiste na incubação da enzima em soro humano agrupado a 37 °C e análises temporais da retenção da atividade enzimática. Referimo-nos à capacidade de uma enzima de reter atividade nessas condições como sua estabilidade sérica, e o método de análise da atividade enzimática aproveita as diferenças na absorbância entre o substrato de uma enzima e o produto resultante. O conceito de estabilidade sérica não é apenas específico da enzima e também tem sido aplicado a várias outras modalidades de tratamento. Por exemplo, a estabilidade sérica de aptâmeros de RNA direcionados às proteínas spike COVID foi previamente avaliada monitorando sua degradação pós-incubação com soro fetal de bezerro1. Os peptídeos antibacterianos também foram avaliados quanto à sua capacidade de suprimir o crescimento bacteriano pós-incubaçãocom soro humano 2.
HsADA1 é uma enzima que catalisa a conversão de adenosina ou 2-desoxiadenosina em inosina ou 2-desoxiinosina, respectivamente3. A adenosina tem um pico de absorção de 260 nm, enquanto a inosina absorve mais forte a 250 nm4. Essa mudança nos picos de absorção pode ser detectada em um leitor de microplacas por uma diminuição na intensidade de absorção a 260 nm quando HsADA1 é adicionado à adenosina. A enzima HsADA1 tem implicações importantes no corpo humano, e sua deficiência causa uma grave imunodeficiência combinada (ADA-SCID)5. O homólogo bovino de HsADA1, BtADA1, pode ser utilizado como uma terapêutica de reposição enzimática para o tratamento de ADA-SCID, e mostramos anteriormente que o HsADA1 selvagem perde sua atividade quando incubado com soro humano combinado, potencialmente dificultando seu uso como terapêutico6. Portanto, selecionamos a enzima HsADA1 do tipo selvagem para demonstrar um procedimento para determinar a estabilidade sérica de uma enzima. Um método de purificação detalhado para HsADA1 foi descrito anteriormente6.
No protocolo a seguir (conforme detalhado na Figura 1), demonstramos como co-incubar HsADA1 do tipo selvagem em soro humano combinado a 37 ° C. Durante esse período, as amostras de teste são coletadas em pontos de tempo definidos e congeladas rapidamente para análise futura. Uma vez que todas as amostras tenham sido coletadas, um ensaio de microplaca é executado em que cada amostra é combinada com o substrato, com a diminuição da absorbância resultante sendo uma correlação para a atividade retida da enzima. Resultados representativos que ilustram a estabilidade sérica de HsADA1 são mostrados e, como essa métrica pode ser relevante para determinar o valor terapêutico potencial de outras enzimas, também discutimos considerações para adaptar este protocolo a uma enzima Homo sapiens quinureninase modificada (HsKYNase) e quaisquer enzimas em geral. A HsKYNase é uma enzima envolvida no metabolismo do triptofano e é capaz de degradar os subprodutos do triptofano quinurenina e hidroxi-quinurenina (OH-Kyn) em ácido antranílico e ácido hidroxi-antranílico (OH-AA), respectivamente. A modulação do metabolismo do triptofano mediada por enzimas pode ser de relevância terapêutica7.
Protocolo
1. Incubação de soro
- Prepare um estoque de 10x de HsADA1 em 1x PBS pH 7,4 (1x PBS) a uma concentração final de 10 μM. Descongele uma alíquota de 15 mL de soro humano agrupado e pré-aqueça a 37 ° C. Pré-aqueça uma alíquota de 50 mL de 1x PBS a 37 °C.
- Preparar as misturas de incubação enzima-soro adicionando 100 μL do stock 10x HsADA1 a 900 μL de soro humano agrupado num tubo de microcentrífuga de baixa ligação, denominado mistura enzima + soro. Em um tubo de microcentrífuga de baixa ligação separado, adicione 100 μL do estoque 10x HsADA1 a 900 μL de 1x PBS para comparação com um controle sem soro, conhecido como mistura de enzima + 1x PBS. A concentração final de HsADA1 em cada mistura será de 1 μM.
- Prepare um controle adicional em um tubo de microcentrífuga de baixa ligação consistindo de 100 μL de 1x PBS misturado com 900 μL de soro humano agrupado. Isso servirá como controle não enzimático para a mistura enzima + soro. Além disso, faça um controle composto por 1 mL de 1x PBS. Isso servirá como controle não enzimático para a mistura enzimática + 1x PBS.
- Alíquota de 100 μL de cada mistura da etapa 1.2 em tubos separados de baixa ligação e diluir 1:1 com 100 μL de glicerol a 30% (v/v) em 1x PBS. Congelar rapidamente com azoto líquido e conservar a -80 °C. Essas amostras serão o ponto de tempo do Dia 0 e a concentração de HsADA1 nelas será de 500 nM. Repetir este passo para as misturas de controlo não enzimáticas a partir do ponto 1.3. Selar todas as enzimas e misturas de controlo com película transparente e colocá-las numa incubadora a 37 °C.
- Após 24 h, alíquota de 100 μL de cada amostra incubada e diluir 1:1 com 100 μL de glicerol a 30% (v/v) em 1x PBS. Congelar rapidamente com azoto líquido e conservar a -80 °C. Essas amostras serão o ponto de tempo do Dia 1. Voltar a colocar todas as misturas enzimáticas e de controlo na incubadora a 37 °C. Repita esta etapa em 72 h e 120 h.
NOTA: As amostras alíquotas não precisam necessariamente ser congeladas rapidamente. Alternativamente, o usuário pode realizar o ensaio baseado em microplacas imediatamente após a coleta de cada amostra, embora curvas padrão frescas e estoques de substrato de adenosina devam ser preparados cada vez que uma amostra é testada.
2. Ensaio baseado em microplacas
- Preparar o leitor de microplacas estabelecendo um protocolo de leitura cinética (figura 2) para medir a absorvância a 260 nm com o intervalo de leitura mais curto durante 30 minutos e pré-aquecer o leitor até um ponto de regulação de 37 °C.
- Descongele todas as amostras coletadas no gelo. Diluir cada amostra descongelada 1:10 usando 1x PBS pré-aquecido a 37 °C como diluente em tubos de microcentrífuga de baixa ligação. A nova concentração de HsADA1 nessas amostras será de 50 nM.
- Faça uma diluição de 10x em 1x PBS da amostra de controle de soro não enzimático também. Além disso, prepare um estoque de adenosina 5 mM adicionando 66,8 mg de adenosina a 50 mL de 1x PBS. Prepare uma diluição de 250 μM de adenosina adicionando 400 μL do estoque de adenosina a 7.600 μL de 1x PBS e pré-aqueça a 37 ° C em uma incubadora.
- Em uma microplaca compatível com UV de 96 poços, adicione 160 μL da diluição de adenosina de 250 μM e 40 μL de cada amostra de proteína diluída em triplicado para um volume total de 200 μL. Isso produzirá uma concentração final de adenosina de 200 μM e uma concentração enzimática final de 10 nM.
- Repetir a diluição para as amostras de controlo adicionando 40 μl do soro diluído ou 1x controlos PBS e 160 μl da diluição de adenosina de 250 μM em triplicata. Isso também produzirá uma concentração final de adenosina de 200 μM, mas sem enzima.
NOTA: Pode ser benéfico adicionar todas as enzimas diluídas e amostras de controle individualmente à microplaca primeiro e, em seguida, adicionar o substrato de adenosina com uma micropipeta de 12 canais para reduzir o tempo total de carregamento. Além disso, placas transparentes aos raios UV devem ser usadas nesta etapa para evitar que a própria placa confunda o sinal de absorbância medido. - Medir a absorvância de cada alvéolo a 260 nm durante 30 min a 37 °C. Uma vez feito isso, exporte os dados para uma planilha para análise posterior.
3. Análise dos dados do leitor de microplacas
- Determine a inclinação da região linear dos dados de absorbância em função do tempo para os primeiros minutos dos dados da microplaca. Para as amostras diluídas em soro, isso correspondeu aos primeiros ~ 120 s. Para as amostras diluídas em PBS 1x, isso correspondeu aos primeiros ~ 270 s. Esta inclinação dos valores de absorbância decrescente terá um valor numérico negativo e terá unidades de variação em unidades de absorbância por segundo (ΔA260/s).
- Subtrair a inclinação do controlo negativo que consiste em soro humano agrupado da inclinação dos dados enzimática + soro. Execute a mesma operação nas misturas de enzima + 1x PBS subtraindo a inclinação do controle 1x PBS.
- Normalize todas as inclinações ajustadas para a inclinação original em t = 0 h para obter a fração de atividade restante usando a Equação 1.
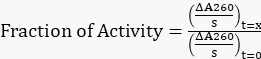 Equação 1
Equação 1 - Plote a fração de atividade restante versus o tempo.
Resultados
As figuras mostram os resultados do ensaio executado quando conduzido com HsADA1 do tipo selvagem. A Figura 3A, B ilustra as curvas de declínio da absorbância a 260 nm das amostras provenientes das misturas 1x PBS/soro-enzima para HsADA1 de tipo selvagem após a adição de adenosina. Essa absorbância em declínio em função dos dados de tempo é o que o usuário pode esperar após a conclusão bem-sucedida do ensaio baseado em microplac...
Discussão
Este protocolo usa a mudança de absorbância à medida que o substrato é convertido no produto para medir a atividade de uma enzima. Como tal, o substrato e o produto devem ter perfis espectrais distintos. Este é o caso da adenosina e da inosina, ambas com perfis espectrais distintos e coeficientes de extinção entre 260-265 nm 6,8,12,13. Este ensaio é ins...
Divulgações
JB e MRJ são inventores de patentes ou pedidos de patentes relacionados às enzimas adenosina desaminase e/ou quinureninase. Todos os outros autores não têm conflitos de interesse a divulgar.
Agradecimentos
Este trabalho foi apoiado pelo National Institutes of Health [1DP2CA280622-01] e financiamento da Biolocity. Agradecemos à Dra. Maria Jennings e Andrea Fox por fornecerem os vetores de expressão HsADA1 e HsKYNase.
Materiais
| Name | Company | Catalog Number | Comments |
| Adenosine | Sigma Aldrich | A9251-25G | 25 g |
| BioTek Synergy HT Microplate Reader | |||
| Eppendorf LoBind Microcentrifuge Tubes: Protein | Fisher Scientific | 13-698-795 | 2 mL |
| Glycerol | Fisher Scientific | G33-4 | 4 L |
| HsKYNase66-W102H-T333N | In-house | ||
| Human Serum, Pooled | MP Biomedicals | 92931149 | 100 mL |
| Hydroxy-kynurenine | Cayman Chemicals | 27778 | |
| Inosine | TCI | I0037 | 25 g |
| PBS, 1x pH 7.4+/- 0.1 | Corning | 21-040-CM | |
| Pyridoxal 5-phosphate monohydrate, 99% | Thermo Scientifc | 228170010 | 1 g |
| UV-STAR MICROPLATE, 96 WELL, COC, F-BOTTOM | Greiner Bio | 655801 | |
| Wildtype Human Adenosine Deaminase 1 | In-house |
Referências
- Valero, J., et al. A serum-stable RNA aptamer specific for SARS-COV-2 neutralizes viral entry. Proc Natl Acad Sci U S A. 118 (50), e2112942118 (2021).
- Iannuzo, N., et al. High-throughput screening identifies synthetic peptides with antibacterial activity against mycobacterium abscessus and serum stability. ACS Omega. 7 (27), 23967-23977 (2022).
- Ma, M. T., Jennings, M. R., Blazeck, J., Lieberman, R. L. Catalytically active holo homo sapiens adenosine deaminase i adopts a closed conformation. Acta Crystallograph Sect D. 78 (1), 91-103 (2022).
- Li, W., et al. Determination of 4 nucleosides via one reference compound in chinese cordyceps by hplc-uv at equal absorption wavelength. Natural Prod Comm. 18 (3), 1934578X231161410 (2023).
- Whitmore, K. V., Gaspar, H. B. Adenosine deaminase deficiency - more than just an immunodeficiency. Front Immunol. 7, 314 (2016).
- Jennings, M. R., et al. Optimized expression and purification of a human adenosine deaminase in E. coli and characterization of its asp8asn variant. Prot Express Purificat. 213, 106362 (2024).
- Blazeck, J., et al. Bypassing evolutionary dead ends and switching the rate-limiting step of a human immunotherapeutic enzyme. Nat Catalysis. 5 (10), 952-967 (2022).
- Lu, J., Grenache, D. G. Development of a rapid, microplate-based kinetic assay for measuring adenosine deaminase activity in body fluids. Clinica Chimica Acta. 413 (19), 1637-1640 (2012).
- Maciel, L. G., Dos Anjos, J. V., Soares, T. A. Fast and low-cost evaluation of hydroxykynurenine activity. MethodsX. 7, 100982 (2020).
- Bokman, A. H., Schweigert, B. S. 3-hydroxyanthranilic acid metabolism. Iv. Spectrophotometric evidence for the formation of an intermediate. Arch Biochem Biophys. 33 (2), 270-276 (1951).
- Tanford, C. . Advances in protein chemistry. 23, 121-282 (1968).
- Tritsch, G. L. Validity of the continuous spectrophotometric assay of kalckar for adenosine deaminase activity. Anal Biochem. 129 (1), 207-209 (1983).
- Gracia, E., et al. The catalytic site structural gate of adenosine deaminase allosterically modulates ligand binding to adenosine receptors. FASEB J. 27 (3), 1048-1061 (2013).
- Kalckar, H. M. Differential spectrophotometry of purine compounds by means of specific enzymes: Iii. Studies of the enzymes of purine metabolism. J Bio Chem. 167 (2), 461-475 (1947).
- Hartwick, R., Jeffries, A., Krstulovic, A., Brown, P. R. An optimized assay for adenosine deaminase using reverse phase high pressure liquid chromatography. J Chromatographic Sci. 16 (9), 427-435 (1978).
- Paul, M. K., Grover, V., Mukhopadhyay, A. K. Merits of hplc-based method over spectrophotometric method for assessing the kinetics and inhibition of mammalian adenosine deaminase. J Chromatography B. 822 (1), 146-153 (2005).
- Ubbink, J. B., Vermaak, W. J. H., Bissbort, S. H. High-performance liquid chromatographic assay of human lymphocyte kynureninase activity levels. J Chromatography B: Biomed Sci Appl. 566 (2), 369-375 (1991).
- Tsentalovich, Y. P., Snytnikova, O. A., Forbes, M. D. E., Chernyak, E. I., Morozov, S. V. Photochemical and thermal reactivity of kynurenine. Exp Eye Res. 83 (6), 1439-1445 (2006).
- Demain, A. L., Vaishnav, P. Production of recombinant proteins by microbes and higher organisms. Biotechnol Adv. 27 (3), 297-306 (2009).
- Gräslund, S., et al. Protein production and purification. Nat Methods. 5 (2), 135-146 (2008).
- Faber, M. S., Whitehead, T. A. Data-driven engineering of protein therapeutics. Curr Opin Biotechnol. 60, 104-110 (2019).
- Das, S., Zhao, L., Elofson, K., Finn, M. G. Enzyme stabilization by virus-like particles. Biochemistry. 59 (31), 2870-2881 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados