Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Терруар Концепция толкуется через виноградной ягоды метаболомики и транскриптомика
* Эти авторы внесли равный вклад
В этой статье
Резюме
В данной статье описывается применение нецелевых метаболомике, транскриптомику и многомерного статистического анализа к виноградным ягодных транскриптов и метаболитов, чтобы получить представление о концепции терруара, то есть, влияние окружающей среды на качество ягодный черт.
Аннотация
Терруар относится к сочетанию факторов внешней среды , которые влияют на характеристики культур , таких как виноградной лозы (Vitis Vinifera) в соответствии с конкретной среды обитания и практики управления. В данной статье показано, как некоторые сигнатуры терруар могут быть обнаружены в ягодного метаболом и транскриптома виноградной лозы сорта Corvina с использованием многомерного статистического анализа. Метод сначала требует соответствующего плана отбора проб. В данном примере, конкретный клон сорта Corvina был выбран, чтобы свести к минимуму генетические различия, и образцы были собраны из семи виноградников, представляющих три различных макро-зоны в течение трех сезонов роста. Нецелевой ЖХ-МС метаболомика подход рекомендуется из-за его высокой чувствительности, сопровождается эффективной обработки данных с использованием программного обеспечения MZmine и стратегию идентификации метаболита, основанный на анализе фрагментации дерева. Комплексный анализ транскриптом может быть достигнуто с использованием микрочиповсодержащие зонды покрытия ~ 99% всех предсказанных генов виноградной лозы, что позволяет одновременный анализ всех дифференциально выраженных генов в контексте различных терруара. И, наконец, многомерный анализ данных на основе проекционных методов могут быть использованы для преодоления сильного марочные конкретного эффекта, что позволяет метаболомика и данные транскриптомика быть интегрированы и детально проанализированы с целью выявления информативных корреляции.
Введение
Анализ данных Крупномасштабное на основе геномов, Транскриптом, протеомов и metabolomes растений обеспечивает беспрецедентное понимание поведения сложных систем, таких как терруара характеристик вина, которые отражают взаимодействие между виноградной лозы растений и окружающей их средой. Поскольку терруар вина могут быть различны, даже если идентичные клоны виноградная лоза выращивается в разных виноградников, анализ геномика мало пользы, так как клональные геномы одинаковы. Вместо этого необходимо посмотреть на корреляции между экспрессией генов и метаболических свойств ягод, которые определяют качество черты вина. Анализ экспрессии генов на уровне транскриптома выгоды от аналогичных химических свойств всех транскриптов, что облегчает количественный анализ путем использования универсальных характеристик, таких как гибридизации с иммобилизованными зондами на микрочипов. В противоположность этому, универсальные аналитические методы в протеомики ай метаболомика являются более сложными из-за огромного физического и химического разнообразия отдельных белков и метаболитов. В случае метаболомике это разнообразие еще более экстремальным, поскольку отдельные метаболиты значительно различаются по размеру, полярности, изобилия и волатильности, поэтому ни один процесс экстракции или аналитический метод предлагает целостный подход.
Среди аналитических платформ, подходящих для нелетучих метаболитов, те, которые основаны на высокоэффективной жидкостной хроматографии в сочетании с масс-спектрометрией (ВЭЖХ-МС) значительно более чувствительны, чем альтернативные варианты, такие как HPLC с ультрафиолетовым или диодных матричных детекторов (ВЭЖХ-УФ, ВЭЖХ-DAD ) или ядерного магнитного резонанса (ЯМР), но количественный анализ с помощью ВЭЖХ-МС могут влиять такие явления, как эффект матрицы и подавления ионов / улучшения 1-3. Исследование таких эффектов при анализе ягод винограда Корвина с помощью ВЭЖХ-МС с использованием источником ионизации электрораспылением (ВЭЖХ-ESI-MS), показал, что сахара и другие молекулы с самыми низкими временами удерживания были сильно занижены, вероятно, также отражающий большое число молекул в этой зоне, и что обилие других молекул могут быть занижены, завышены или не зависит от матричного эффекта , но нормализация данных для матричного эффекта , казалось, имеют ограниченное влияние на общие результаты 4,5. Описанный здесь способ оптимизирован для анализа средней полярности метаболитов, которые накапливаются на высоких уровнях в виноградных ягод во время созревания, и которые могут оказывать значительное влияние терруара. Они включают в себя антоцианы, флавонолы, флаван-3-олы, процианидины, другие флавоноиды, ресвератрол, стильбены, гидроксикоричными кислоты и гидроксибензойной кислоты, которые вместе определяют цвет, вкус и свойства, связанные со здоровьем вин. Другие метаболиты, такие как сахара и алифатических органических кислот, игнорируются, поскольку Количественное определение с помощью ВЭЖХ-МС является ненадежным из-за матрицы эфт и подавление ионных явлений 5. В пределах диапазона полярности , выбранной с помощью этого метода, подход нецелевой в том , что он направлен на обнаружение , как много различных метаболитов максимально 6.
Методы транскриптомика , которые позволяют тысячи виноградной лозы транскриптов, подлежащих мониторингу одновременно облегчается наличием последовательности генома 7,8 полный виноградной лозы. Ранние методы транскриптомика, основанные на высокой пропускной способности кДНК последовательности развивались с появлением следующего поколения последовательности в набор процедур, описанных в совокупности, как РНК-Seq технологии. Такой подход быстро становится методом выбора для транскриптомике исследований. Тем не менее, большая часть литературы на основе микрочипов, которые позволяют тысячи транскриптов быть количественно параллельно с помощью гибридизации, накопила для виноградной лозы. Действительно, прежде чем РНК Seq стала основной технологией, многие специализированные коммерческие микрочипов платформы былиразработаны позволяя виноградная транскриптомный быть осмотрены в мельчайших подробностях. Среди огромного множества платформ, только два позволил геному анализ транскриптом 9. Наиболее эволюционировали массив позволил гибридизацию до 12 независимых выборок на одном устройстве, что позволяет снизить затраты каждого эксперимента. 12 подмассивы каждая из которых содержит 135000 60-мерных зондов, представляющих 29,549 виноградной лозы стенограммы. Это устройство было использовано в большом количестве исследований 10-24. Эти две платформы теперь было прекращено , но новый заказ микрочипов недавно был разработан и представляет собой более недавнее развитие , поскольку она содержит еще большее количество зондов , представляющих дополнительные недавно обнаруженных генов виноградной лозы 25.
Наборы данных большой продажи, произведенные транскриптомика и Метаболомика анализа требуют подходящих статистических методов для анализа данных, включая многомерные методы для определения корреляции между различной формыs данных. Наиболее широко используемые многомерные методы, основанные на проекции, и они могут быть без присмотра, например, анализа главных компонент (PCA), или под контролем, такие как двунаправленного ортогональной проекции на латентные структуры дискриминантный анализ (O2PLS-DA) 26. Протокол, представленные в этой статье используется РСА для анализа поисковых данных и O2PLS-DA для выявления различий между группами образцов.
протокол
1. Выберите соответствующие материалы и Построить план выборочного контроля
- Начать эксперимент по разработке соответствующего плана отбора проб. Там нет общего и универсального подхода поэтому оценить каждый план на индивидуальной основе случая. Убедитесь в том, что план выборочного контроля указывается места отбора проб, времени и точной процедуры отбора образцов. На рисунке 1 плана выборки , используемого в этом исследовании.
Примечание: В данном примере, виноградные ягоды из одного клона (. Виноград культурный резюме Corvina, клон 48) были собраны из семи коммерческих виноградников в трех различных макро-зон в провинции Верона (озеро Гарда, Вальполичелла и Соаве). Главные особенности каждого виноградника приведены в таблице 1. Ягоды были собраны в течение трех сезонов роста (2006, 2007 и 2008) в трех временных точках, соответствующих veraison (началом созревания), в середине созревания плодов и спелых ягод.- Для каждого из присоединений (Vineyard / год / стадии созревания), урожай 30 групп из разных позиций вдоль двух виноградных рядов, рандомизированные высоты и места на заводе.
- Выберите три ягоды случайным образом из каждого кластера, избегая тех, кто видимых повреждений и / или признаков инфекции.
- Повторите шаги 1.1.1 и 1.1.2, чтобы получить три независимых бассейна.
- Удалять семена ягоды и сразу же заморозить околоплодника в жидком азоте.
- Измельчите 10 замороженные ягоды из каждого пула с автоматической мельницы шлифовальный станок, и разделить каждую порошке на две равные части, одна для анализа транскриптомика и один для анализа Метаболомика.
- Хранить порошки при температуре -80 ° С.
| AM | BA | BM | CS | FA | Миннесота | ВЕЧЕРА | |
| Макро-зона | Соаве | Озеро Гарда | Вальполичелла | Озеро Гарда | Вальполичелла | Вальполичелла | Соаве |
| Высота (м) | 250 | 120 | 450 | 100 | 130 | 250 | 130 |
| корневище | 41В | S04 | K5BB | 420A | 420A | K5BB | 41В |
| направление строки | EW | Н.С. | EW | EW | EW | Н.С. | Н.С. |
| система обучения | Накладные система (Пергола) | Накладные система (Пергола) | Вертикальный Shoot позиционирования (Гийо) | Накладные система (Пергола) | OverheОбъявление системы (Пергола) | Вертикальный Shoot позиционирования (Гийо) | Вертикальный Shoot позиционирования (Гийо) |
| Тип почвы | суглинки | суглинок | глиняный | суглинок | Суглинок | Иле суглинок | Суглинок |
| Посадка макета (м) | 3.20 х 1,00 | 4,50 х 0,80 | 4,00 х 1,25 | 3,50 х 1,20 | 3,50 х 0,75 | 2,80 х 1,00 | 1,80 х 0,80 |
| Общая известь% | 3,9 | 19,3 | 18,3 | 14.4 | 31 | 5.9 | 27,9 |
| Активная известь% | 0,5 | 2.6 | 9.4 | 6.3 | 11.3 | 3.1 | 8.3 |
| Песок% | 15 | 47 | 66 | 42 | 29 | 13 | 36 |
| Глина% | 43 | 36 | 21 | 37 | 39 | 67 | 36 |
| Глина% | 42 | 17 | 13 | 21 | 32 | 20 | 28 |
| рН почвы | 8.3 | 7.9 | 7.8 | 8.2 | 8.2 | 7.8 | 7.9 |
| Органические вещества (%) | 2.9 | 2.5 | 2.2 | 1.2 | 2.9 | 1.6 | 2.5 |
| Сменная фосфора (мг / кг) | 26 | 73 | 73 | 68 | 48 | 47 | 64 |
| Сменная калия (мг / кг) | 190 | 376 | 620 | 230 | 168 | 154 | 126 |
| Сменная магния (мг / кг) | 272 | 468 | 848 | 623 | 294 | 293 | 183 |
| Сменная кальция (мг / кг) | 6500 | 5380 | 7358 | 6346 | 4652 | 10055 | 2878 |
| Ягодные восстанавливающими сахарами 2006 | 211,25 ± 1,20 | 176,20 ± 0,42 | 187,40 ± 0,00 | 203,70 ± 1,13 | 212,55 ± 0,64 | 195,20 ± 0,00 | 211,65 ± 0,64 |
| Ягодные восстанавливающими сахарами 2007 | 190,00 ± 1,27 | 165,25 ± 0,49 | 153,00 ± 0,42 | 203,60 ± 0,71 | 210,90 ± 0,71 | 192,25 ± 0,64 | 188,70 ± 1,84 |
| Ягодные восстанавливающими сахарами 2008 | 191,35 ± 0,64 | 178,90 ± 0,57 | 170,05 ± 0,49 | 205,15 ± 1,48 | 188,70 ± 0,57 | 169,35 ± 0,49 | 108,05 ± 1,06 |
| Berry рН 2006 | 3,01 ± 0,01 | 2,96 ± 0,01 | 2,84 ± 0,00 | 2,9 ± 0,00 | 2,98 ± 0,00 | 3,02 ± 0,00 | 3,06 ± 0,01 |
| Berry рН 2007 | 2,97 ± 0,00 | 3,00 ± 0,00 | 2,74 ± 0,00 | 3,07 ± 0,01 | 2,98 ± 0,00 | 2,87 ± 0,01 | 3,09 ± 0,00 |
| Berry рН 2008 | 2,83 ± 0,00 | 3,04 ± 0,01 | 2,71 ± 0,00 | 2,98 ± 0,01 | 2,98 ± 0,00 | 2,82 ± 0,00 | 3.11 ± 0.00 |
| Сбор урожая Дата | 2006 | 2007 | 2008 | ||||
| Veraison | 8-Aug | 18-июля | 12-Aug | ||||
| Середина Созревание | 4-Sep | 8-Aug | 2-Sep | ||||
| созревший | 18-Sep | 29-Aug | 23-Sep |
Таблица 1: Основные особенности каждого виноградника иобразец коллекции даты. м = м, EW = Ешь-запад, NS = Север-Юг. 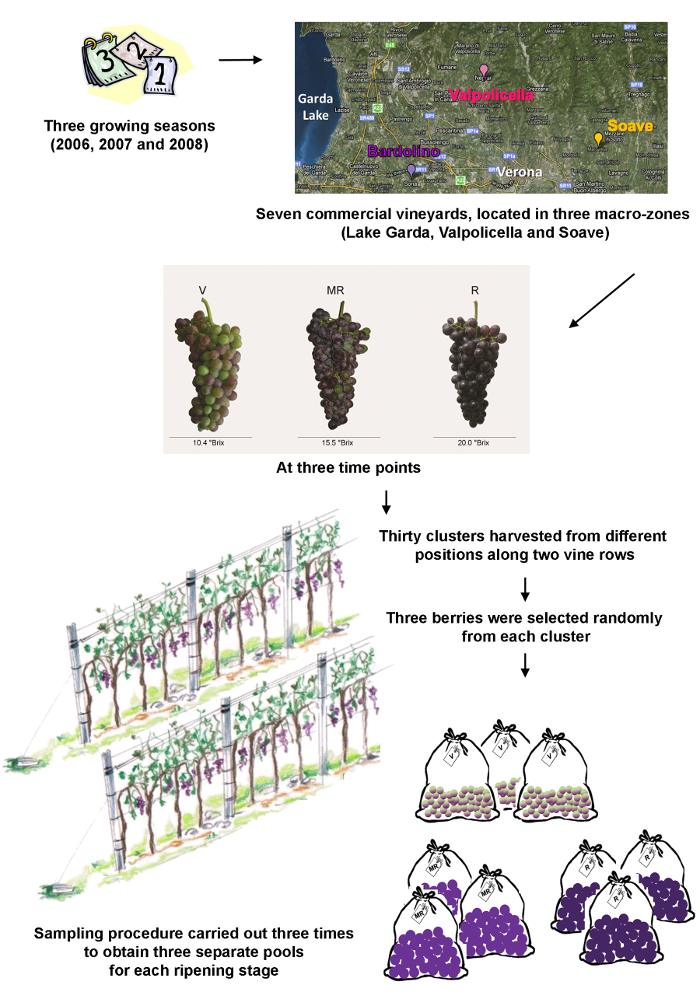
Рисунок 1:. Схематическое представление процедуры отбора образцов Три производства вина макро-зоны расположены в окрестностях города Верона, Венето, Италия. Три временных точек являются veraison (V) , представляющий начало созревания в виноградарстве, в середине созревания (MR) и спелые ягоды (R). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
2. Подготовка Берри порошок экстрактов, анализ Метаболиты и обрабатывать данные
- Подготовка образцов Берри порошок для анализа.
- Готовят метаболические экстракты образцов ягодных при комнатной температуре в трех томах (вес / объем) метанола, подкисленного 0,1% (об / об) в течениемуравьиной кислоты в ультразвуковой ванне при частоте 40 кГц в течение 15 мин. С помощью ЖХ-МС-класса воды и муравьиную кислоту, и ВЭЖХ-метанол марки.
- Центрифуга экстракты при 16000 х г в течение 10 мин при температуре 4 ° С.
- Развести супернатант из шага 2.1.2 в двух объемах (об / об) деионизированной воды и пропускают через фильтр 0,2 мкм.
- Проанализировать ягодных экстрактов с помощью ВЭЖХ-ESI-MS.
- Настройка системы ВЭЖХ-ESI-MS в соответствии с рекомендациями поставщика.
Примечание: В данном примере, установка включала ВЭЖХ систему, оснащенную автосамплера, соединенных в линию с масс-спектрометр с ионной ловушкой и источником ионизации электрораспылением. - Подключение системы ВЭЖХ с применением колонки C18 (7,5 сторожевой х 2,1 мм 2) и колонке с обращенной фазой С18 (150 х 2,1 мм 2, размер частиц 3 мкм).
- Готовят растворители, используемые в качестве подвижной фазы. С помощью 5% (об / об) ацетонитрила в 0,5% (об / об) муравьиной кислоты в воде в качестве растворителя А и 100% ацетонитрила при s-olvent B. Использование ЖХ-МС-класса ацетонитрила, воды и муравьиную кислоту.
- Выполните разделение ВЭЖХ с линейным градиентом растворителей А и В , при постоянной скорости потока 0,2 мл мин -1 с использованием образца вводимый объем 30 мкл.
- Первоначально уравновешивают колонку со 100% А. После растворителя инъекции пробы, устанавливают градиент от 0% до 10% растворител в течение 5 мин, от 10% до 20% растворител в течение 20 мин, от 20% до 25% растворител в 5 мин, и от 25% до 70% растворител в течение 15 мин.
- Анализ каждого образца в двух экземплярах. Перемешайте анализ проб, чтобы избежать инструментов управляемых эффектов. Разрешить 20 минут для повторного уравновешивания с использованием 100% растворителя, между каждым анализом.
- Приобретать масс-спектры в альтернативных отрицательных и положительных режимов ионизации. Для получения результатов , описанных в этом исследовании, задавать параметры , как и в таблице 2. Точная настройка машины зависит от конкретной платформы.
Примечание: В качестве альтернативы, использовать другой костюмспособные методы в зависимости от конкретной платформы.- Во всех случаях, как с любой платформой разделения, убедитесь, что использовать подходящее время повторного установления равновесия, чтобы иметь время удерживания воспроизводимости. Когда число образцов высока (выше десяти выборкам), анализируют их в партиях 9-10 образцов, с программами для очистки хроматографической колонки (медленный градиенты между двумя элюирования растворителей) между каждой партии. Для того, чтобы иметь достаточно времени удержания воспроизводимости, убедитесь , что первый хроматографический анализ каждой партии является пустой анализ (т.е. анализ растворителя).
- Также приобретают масс - спектры в фрагментированных отрицательных и положительных ионов режимов установки опций фрагментации (два ионных предшественников, MS 3). Аннотирования метаболитов анализа фрагментации дерева (MS / MS и MS 3) в соответствии с инструкциями изготовителя.
Примечание: Это особенно хорошо подходит для метаболитов растений, так как они часто гликозилированный, TherEfore первая фрагментация (МС / МС) , как правило, удалить сахарные группы получали свободную агликоновую иона и последующей фрагментации (MS 3) помогает идентифицировать агликоне против подлинной библиотеки стандартов. - Acquire MS / MS и MS 3 спектров в диапазоне m / z 50-1,500 с осколочной амплитудой 1 В. В качестве альтернативы, использовать другие подходящие методы в соответствии с конкретной платформой.
- Для каждого m / z сигнала, сравните MS / MS и MS 3 осколочные моделей и времени удерживания аутентичного библиотеки стандартов в соответствии с инструкциями изготовителя.
Примечание: Многие тип платформ включают в себя программные средства для создания такого рода библиотеки на основе анализа аутентичных стандартов. Это должно идентифицировать многие из сигналов, но не все сигналы будут примеч. - Для остальных анонимных сигналов, сравните MS / MS и MS шаблоны 3 осколочные с опубликованными в литературе, Searcповесят для значений / г м (т.е. цифра "м / з 353" , чтобы искать публикаций , в которых молекулы с упоминаются такой т / г) с любым из общей поисковой системы в свободном доступе, или использовать онлайновые базы данных , такие как MassBank (WWW .massbank.jp / ен / database.html) и Human метаболом Database (www.hmdb.ca/search/spectra?type=ms_search).
- Настройка системы ВЭЖХ-ESI-MS в соответствии с рекомендациями поставщика.
| Масс - спектрометр компоненты | функция | параметры |
| Ионизацией электрораспылением Источник | распыляющего газа | 50 фунтов на квадратный дюйм, 350 ° С |
| Сушка газа | 10 л мин -1 | |
| Ионная ловушка и детектор | сканирование | Режим полного сканирования, 13 000 м / г в секунду, диапазон 50-1,500 м / г |
| Целевая масса | 400 м / г | |
| Столкновение газа | гелий | |
| давление вакуума | 1,4 × 10 -5 мбар | |
| источник Капиллярная | +4000 V | |
| Концевая пластина смещения | -500 V | |
| шумовка | -40 V | |
| выход Cap | -121 V | |
| 1 октября DC | -12 В | |
| 2 октября DC | -1,7 V | |
| Объектив 1 | +5 В | |
| Объектив 2 | +60 V | |
| ICC для положительного режима ионизации | +20,000 | |
| ICC для режима отрицательной ионизации | 7000 |
Таблица 2: Основной набор параметров для получения масс - спектров.
| операция | выбор | функция | параметры | Значения |
| Обнаружение пиков | Массовое обнаружение | центроида | Уровень шума | 3500 |
| Хроматограмма строитель | Самая высокая точка данных | промежуток времени, мин | 0,15 | |
| мин высота | 4000 | |||
| м / г Допуск | 0,3 | |||
| Обнаружение пиков | Пик деконволюции | Локальный поиск минимального | порог Хроматографическое | 70 |
| Поиск минимум в диапазоне RT (мин) | 0:50 | |||
| Минимальная относительная высота | 15% | |||
| Минимальная абсолютная высота | 4000 | |||
| Соотношение Мин пика верхнего края / | 2 | |||
| Диапазон Длительность (мин) | 0-10 | |||
| Изотопы | Изотопные пики группер | - | м / г Допуск | 1.2 |
| допуск RT | 0:50 | |||
| монотонная форма | Нет | |||
| Максимальный заряд | 3 | |||
| представитель изотоп | Нет | |||
| центровка | Регистрация выравнивателя | - | м / г Допуск | 1.2 |
| Вес для т / г | 10 | |||
| Сохранение допустимых значений времени | 0:50 | |||
| Вес для РТ | 5 | |||
| Требовать же состояние заряда | Нет | |||
| Требовать же идентификатор | Нет | |||
| Сравните изотопный шаблон | Нет | |||
| Заполнение промежутка | Пик искатель | - | допуск интенсивности | 20% |
| м / г Допуск | 0,9 | |||
| Сохранение допустимых значений времени | 0:40 | |||
| коррекция RT | Нет | |||
| фильтрация | Дубликат Peak фильтр | - | м / г Допуск | 1.2 |
| допуск RT | 0:30 | |||
| Требовать же идентификационный | Нет |
Таблица 3: Mzmine рабочий процесс с конкретными значениями для обработки отрицательных файлов данных виноградный ягодные LC-MS.
- Обработать LC-MS данных.
- Доступ илизагрузить пакет программного обеспечения для обработки данных, который может извлечь соответствующую информацию из многих сырых хроматограммах и построить матрицу данных, в которой каждый обнаруженный метаболит квантифицируется в каждом образце.
Примечание: шаги Протокола ниже приспособлены для открытого исходного кода программного обеспечения MZmine v2.14 (http://mzmine.sourceforge.net). Это является открытым исходным кодом программное обеспечение для обработки данных масс-спектрометрии, с основным акцентом на данных LC-MS. 27 - Преобразование данных хроматограмме LC-MS в формат NetCDF с использованием программного обеспечения, поставляемого производителем оборудования. Для получения результатов, описанных здесь, используйте V5.2 Bruker Daltonics Esquire и анализ данных V3.2 программного обеспечения и выполнять действия в соответствии с инструкциями изготовителя.
Примечание: Другие преобразователи могут быть использованы (различные изделия свободной преобразователи доступны). - Импорт .cdf файлы в программное обеспечение.
- Внедрить процедуры фильтрации обнаружения пика, выравнивание, заполнение пропусков и пик с PARAMETERS приведены в таблице 3.
- В качестве первого шага, выберите импортированный файл .cdf, а затем перейти к визуализации → TIC / XIC визуализатора. Поместите курсор на основании наименьшего пика на хроматограмме, и принять к сведению о минимальной интенсивности сигнала основного иона. Затем перейдите к Raw методы обнаружения данных → Peak.
- Выберите Mass Обнаружение и заполнить уровень сигнала для обнаружения отдельных ионов для каждого сканирования и создания списка ионов.
Примечание: Проверка алгоритма перед выполнением массового обнаружения - это зависит от масс-спектрометра. В нашем случае мы выбираем центроида. - Выберите хроматограмме Builder и заполнить с уровнем сигнала для подключения точек данных из списка ионов и построить хроматограмма для каждого значения массы. См параметры , указанные в руководстве масс - спектрометра для регулировки Min Временной диапазон и допуск м / г.
- Затем перейдите к пиковым Список методов → Обнаружение пиков хроматограммы → Deconvoлюция. Выберите соответствующий алгоритм (в данном случае локальный минимум поиск), чтобы обеспечить разделение каждой хроматограмме в отдельных пиков.
- Вручную проверить хроматограммы, чтобы выяснить соответствующие значения для следующих параметров. Установите хроматографического порог до 30%, чтобы удалить шум и поиск Минимум в RT диапазоне до 2 (мин), чтобы определить наличие минимальных местных жителей, чтобы различить два пика.
- Установка минимальной относительной высоты в 2,0. Вручную проверьте хроматограмму, чтобы определить минимальную абсолютную высоту сигнала, который соответствует пику, а не на задний план; установить это значение как абсолютная высота (например, 10000 в представленном эксперименте).
- Установка Min Отношение пиковых Top / EDGE до 1,1 изложить минимальное соотношение между верхней и самой низкой интенсивности точечных данных пика, чтобы быть признанным в качестве истинного пика.
- Вручную проверить хроматограммы, чтобы увидеть что минимальная продолжительность различных пиков в используемом хроматographic условия (например, от 0,2 до 2 мин, в зависимости от соединения, в представленных экспериментах), и, следовательно, использовать эти значения как Peak Продолжительность Range (мин), чтобы установить диапазон приемлемой длины пика, как продолжительность.
- Затем перейдите к Peak Список Методы → изотопах → Изотопных Пикс Группировщиком группы изотопов в одном пике, как правило, в наиболее интенсивно. Примечание: Параметры зависят от разрешения масс-спектрометра и воспроизводимости времен удерживания.
- И, наконец, перейти к пиковым Список Методы → Выравнивание → Join Aligner для выравнивания пиков в зависимости от их m / z и время удерживания, используя счет матча.
- После выравнивания пиков, заполнить любые пробелы в данных, нажав на Peak Список Методы → Заполнение зазора → Peak Finder. Наконец, фильтр дублировать точки данных, нажав на Peak Перечислим методы → Фильтрация → Дублировать Peak Filter.
- Экспорт результирующего набора данных в качестве .csv файл.
- Вручную изменить. Расширение CSV на расширение TXT.. Если нет расширений файлов не видны, измените настройки компьютера, чтобы показать расширения файлов. Перейти к File Explorer Функции → View → Advanced Settings и снимите флажок "Скрывать расширения для зарегистрированных типов файлов".
- Импорт .txt файлы в электронную таблицу. Это создает матрицу данных , в которой все обнаруженные метаболиты, распознаваемые времени идентификационный номер, м / г и значение удержания, количественно во всех образцах , с точки зрения их пиковых значений площади.
- Доступ илизагрузить пакет программного обеспечения для обработки данных, который может извлечь соответствующую информацию из многих сырых хроматограммах и построить матрицу данных, в которой каждый обнаруженный метаболит квантифицируется в каждом образце.
3. Подготовка Берри порошок Экстракты для Транскриптом анализа и обработки данных
- Экстракт тотальной РНК из образцов Берри и определить РНК качества.
- Экстракт тотальной РНК из образцов ягодные.
Примечание: В данном примере, патентованного набора и модифицированной методике, которая обеспечивает полное удаление молекул, в томФере с анализом РНК, такие как полисахариды и полифенолы, были использованы. Приведенные ниже инструкции являются специфическими для данного набора 18.- Взвесьте 400 мг ягодного порошка для каждого образца, разделить его на две части и помещают 200 мг каждая в двух микроцентрифужных труб.
- Добавьте 900 мкл лизирующего раствора, содержащего бета-меркаптоэтанола (входящий в комплект) к каждому 200 мг порошка и вихря немедленно и энергично в течение по крайней мере 30 секунд. Нагрева образца при температуре 56 ° С в течение 5 мин при встряхивании 800 оборотов в минуту.
- Центрифуга образцов при максимальной скорости в микроцентрифуге стендовых в течение 10 мин, чтобы осадить клеточный дебрис.
- Пипеткой 700 мкл супернатанта в колонну фильтрации можно найти в комплекте (синий стопорное кольцо), сидящего в пробирку 2 мл. Закройте крышку и центрифуге при максимальной скорости в микроцентрифуге в стендовых в течение 1 мин для удаления остаточного мусора. Повторите этот шаг дважды с использованием той же колонки фильтрации, но свежую пробирку для сбора, в результате чего три ткаждая из которых содержит массовые рассылки ~ 700 мкл осветленного лизата.
- Пипеткой 750 мкл связывающего раствора (поставляется в комплекте) в каждую пробирку осветленной лизата и сразу перемешать с помощью пипетки вверх и вниз, по крайней мере, пять раз. Передача 700 мкл этой смеси в колонке связывания, представленной в комплекте (красный стопорное кольцо), сидящего в пробирку 2 мл. Закройте крышку и центрифуге при максимальной скорости в микроцентрифуге в стендовых в течение 1 мин для связывания РНК.
- Слейте часть проточный, инвертировать пробирку для сбора и нажмите на него кратко на чистую площадку гигроскопической бумаги для слива остаточной жидкости.
- Вернуть колонку в пробирку и пипеткой оставшуюся смесь в ту же колонку и повторяют центрифугирование и декантации шаги. Повторяйте, пока вся смесь не была отфильтрована в тот же красном переплете колонке.
- Теперь следуйте дальнейшим инструкциям в наборе и элюции РНК в 50 мкл буфера для элюции (поставляется в комплекте) и магазин ят при температуре -80 ° C готовый к ступеньках контроля качества.
- Определить количество и чистоту РНК с использованием спектрофотометра. Запишите коэффициенты абсорбции , которые показывают степень загрязнения с белками (A 260/280) и полифенолов / полисахаридами (A 260/230).
Примечание: РНК подходит для микрочипов гибридизации должна набрать не менее 1,8 для обоих коэффициентов. - Определение целостности РНК.
Примечание: Различные системы могут быть использованы. Цифровой приобретатель, который выполняет капиллярного электрофоретического работать в сочетании с флуоресцентным красителем был использован в этом исследовании. РНК подходит для микрочипов гибридизации должна иметь РНК целостности номер (РИН), по меньшей мере, 8.
- Экстракт тотальной РНК из образцов ягодные.
- Подготовка образцов и гибридизацию РНК к пользовательскому Microarray.
- Установите исходное количество тотальной РНК для анализа микрочипов до 200 нг путем разбавления РНК раствора, полученного на стадии 3.1.1.8 деионизованной РНКазы без воды. ДобавитьПодходящее количество РНК в 1,5 мл трубки микроцентрифужных в конечном объеме 1,5 мкл.
- Добавьте 2 мкл разведенной смеси спайка к каждому образцу РНК и следовать инструкциям производителя для синтеза первой нити кДНК, транскрибировать это в кРНК и маркировать кРНК с цианинового 3CTP.
- Очищают меченых кРНК в соответствии с инструкциями изготовителя и элюирования в 30 мкл РНКазы без воды.
- Определение доходности и удельной активности каждого кРНК путем записи трех значений на спектрофотометре: цианиновые 3 концентрация красителя (пмоль мкл -1), чистота РНК (A 260/280) и концентрацию кРНК (нг мкл -1). С помощью формулы, найденные в инструкции по эксплуатации завода-изготовителя для расчета выхода кРНК (мкг) и удельную активность (пмоль Cy3 на мкг кРНК).
Примечание: Рекомендуется выходы и конкретные мероприятия различаются на основе конкретного формата микрочипов. В этом случае исследование формат 44K 4-пакет былвыбран, рекомендуемый выход составлял 1,65, а удельная активность была 9. - Проектирование пользовательских микрочипов с помощью соответствующего программного обеспечения для проектирования зонда.
- Для получения результатов, описанных в этом исследовании, подготовить новый пользовательский микрочип, разработанный на формате 4-пак 44K с помощью веб-приложения для пользовательских микрочипов конструкций и библиотек олигонуклеотидных. Дизайн зондов для соответствия 34,651 целевых стенограммы, в том числе 29971 предсказывал транскриптов из пино нуар V1 массива, 4,500 новый локусов идентифицированы в сортом Пино по транскриптома реконструкции Corvina и 180 частных генов Corvina 25.
ПРИМЕЧАНИЕ: Это включало производство 34,651 специфических 60-мерных зондов, включающий 29798 пино нуар предсказывал транскриптов, 4,392 новый Pinot локусов и 179 Corvina частных генов.
- Для получения результатов, описанных в этом исследовании, подготовить новый пользовательский микрочип, разработанный на формате 4-пак 44K с помощью веб-приложения для пользовательских микрочипов конструкций и библиотек олигонуклеотидных. Дизайн зондов для соответствия 34,651 целевых стенограммы, в том числе 29971 предсказывал транскриптов из пино нуар V1 массива, 4,500 новый локусов идентифицированы в сортом Пино по транскриптома реконструкции Corvina и 180 частных генов Corvina 25.
- Подготовка сборки гибридизацию, основываясь на спецификации формата 4-Pack следующим образом.
- Поместите 1,65 мкг Cy-3 меченой кРНК в конечном объеме 41,8мкл деионизованной РНКазы без воды. Добавьте 11 мкл 10х блокирующего агента и 2,2 мкл 25x фрагментация буфера. Инкубируют при 60 ° С в течение 30 мин в термостате к фрагменту РНК. Остудите на льду в течение 1 мин.
- И, наконец, добавить 55 мкл 2x буфера для гибридизации, хорошо перемешать с помощью пипетки и спином в течение 1 мин при 15500 х г при комнатной температуре. Незамедлительно поместите трубку микроцентрифужных на льду. Используйте немедленно, не храните его.
- Загрузка пользовательских микрочипов следующим образом.
- Загрузите прокладку слайд с этикеткой вверх в основание гибридизации камеры. Медленно загрузить 100 мкл гибридизационного образца, полученного на этапе 3.2.6.2 на каждую прокладку хорошо, дозирования жидкости с кончика пипетки, избегая пузырьков.
- Медленно поместите на заказ микрочипов лицевой стороной вниз, гарантируя, что цифровой штрих-кода лицевой стороной вверх. Убедитесь, что сэндвич пара правильно выровнен. Наконец поместите крышку камеры на гибридизация сэндвичслайды и вручную затяните зажим на камеру. Поверните камеру в собранном для оценки подвижности пузырьков.
- Поместите собранный слайд-камеру в вертелом в гибридизации печь, установленную на 65 ° C. Установите поворотное устройство для вращения при 10 оборотах в минуту. Разрешить гибридизацию проводили в течение 17 ч.
- Продолжайте стирке микрочипов слайд следующим образом.
- Во-первых, подготовить три слайд-окрашивания блюд и заполнить их с соответствующими стиральными буферами: 2 блюда с промывочного буфера 1 при комнатной температуре и 1 блюдо с подогретого (37 ° C) промывочного буфера 2.
- Разберите гибридизационная камеру и удалить бутерброд. С микрочипов слайд-цифровой штрих-код лицевой стороной вверх, погрузить бутерброд в первый слайд-окрашивания блюдо, заполнены промывочного буфера 1 при комнатной температуре и, с помощью чистого пинцета, отделить прокладку от микрочипов слайда. Быстро перевести микрочипов слайд в стойку слайдов и поместить его во второй слайд-окрашивания блюдо, заполнены промывочного буфера 1при комнатной температуре.
- Положите слайд-окрашивания блюдо на магнитной мешалкой и моют 1 мин при умеренном перемешивании. Быстро перенесите слайд стойку в третий слайд-окрашивания блюдо, заполнены предварительно нагретом (37 ° C) промывочного буфера 2 и промыть 1 мин при умеренном перемешивании.
- Медленно снимите стойку с слайд-окрашивания блюдо и осторожно выньте слайд из стойки, избегая капель.
Примечание: Не добавляйте Triton X-102 в промывных буферах и опустить стадию ацетонитрил стирки.
- Храните промытый чип в темноте при комнатной температуре.
- Сканирование микрочипов и извлечение Характерными особенностями.
- Поместите микрочипов слайд в подходящий сканер и сканировать каждый массив, используя значения параметров, рекомендованные в инструкции по эксплуатации завода-изготовителя Microarray в. Для получения результатов, описанных здесь, поместите каждый микрочипов слайд в держатель слайдов, чтобы облегчить процедуру сканирования.
- Импорт вывод .shpподать в соответствующее программное обеспечение, которое способно преобразовывать цифровой сигнал в числовые значения флуоресцентными. Проверьте отчет о контроль качества, чтобы гарантировать, что процедура гибридизация была успешной.
- Для получения результатов , описанных здесь, используйте настройки параметров , рекомендованные в инструкции по эксплуатации программного обеспечения извлечения признаков и проверить отчет контроля качества , чтобы гарантировать , что параметры , перечисленные в таблице 4 находятся в пределах нормального диапазона данного производителя.
| Метрика Имя | Верхний предел | Нижний предел | Описание |
| AnyColorPrcntFeatNonUnif | 1,00 | Не Доступно | Процент функций, которые являются особенностью неоднородности выпадающие в любом канале |
| Предел обнаружения | 2.00 | 0.10 | Среднее плюс 1 стандартное отклонение спайк инов ниже линейного диапазона концентраций |
| absGE1E1aSlope | 1.20 | 0,90 | Абсолютное наклона, пригодный для сигнала от концентрации e1a зондов |
| MedCVProcSignal | 8.00 | Не Доступно | Медиана% CV для сигнала |
| gNegCtrlAveBGSubSig | 5.00 | -10,00 | Среднее значение фона вычитают сигнал всех отрицательных контрольных выступа (BGSubSignal рассчитывается путем вычитания значения под названием BGUsed из функции означают сигнал) |
| gNegCtrlAveNetSig | 40.00 | Не Доступно | Среднее значение чистого сигнала всех отрицательных контролей выступа |
| gNegCtrlSDevBGSubSig | 10.00 | Не Доступно | Стандартное отклонениефон вычитают сигналы всех отрицательных контролей выступа |
| gNonCntrlMedCVProcSignal | 8.00 | Не Доступно | Медиана% CV для сигнала зондов неуправляющая |
| gSpatialDetrendRMSFilter | 15.00 | Не Доступно | Остаточная фона приступе удаления тренда |
Таблица 4: Основные параметры , которые будут проверены , чтобы проверить качество микрочипов гибридизации.
- Процесс микрочипов данных.
- После того, как все микрочипов слайды были отсканированы и контроль качества был оценен положительно, подготовить табуляции данных матрицу путем выбора значения gProcessedSignal из каждого отдельного файла-подмассив итогового, представляющий исходные интенсивности флуоресценции каждого зонда.
- В таблице, нормализовать данные по 75 - й процентиль в пределах каждого массива (значения P) и Расчётногоели среднее значение всех значений Р среди всех различных подмассивов для вычисления отношения R.
- Затем на той же таблице, нормализуют каждое значение gProcessedSignal к отношению R своего собственного подрешетки.
4. Провести детальный статистический анализ метаболомики и транскриптомика данных
- Подготовка статистического анализа программного обеспечения.
Примечание: В этом случае исследование, программного обеспечения, способного выполнять PCA, был использован PLS-DA и O2PLS-DA.- Импорт метаболомики и данные транскриптомика. Перейти к Файл → Создать → обычный проект нового регулярного проекта для импорта матрицы данных, полученных с помощью программного обеспечения MZmine. Затем нажмите на кнопку Edit → Транспонируйте всю матрицу → Дом и назначить соответствующий системе начального и среднего ID → Готово.
- Средний центр данных и масштабировать их, используя шкалу парето. В главном окне перейдите к Edit → M1 и изменить соответствующие парамеОслабляет в зависимости данных. Для получения результатов, описанных здесь, изменить масштаб с единичной дисперсией для пар.
- Масштаб данных транскриптомика с использованием настройки единичной дисперсией.
- Проводят многомерный статистический анализ.
- Реализация PCA , как показано на F igure 2. В этом случае исследование, PCA раскрывает основные различия между образцами, отражающие различные стадии созревания и растущих сезонов.
- В окне рабочего набора, выберите PCA-X в качестве типа модели. Нажмите Autofit. Обратите внимание на R2x (диплом) и Q2 (диплом) значения, поскольку они дают представление о качестве модели.
Примечание: Как правило, чем выше значения, тем лучше модель, но модели с очень высокой R2x (диплом) может чрезмерно соответствовать данным. Как эмпирическому правилу, мы перестаем добавлять главные компоненты, когда значение Q2 (диплом) начинает уменьшаться. - Затем выберите Scores → Scatter, чтобы увидеть график, который показывает возможную группировку образцов.
- Осмотрите PCAОценка участка. Если хорошая модель получается (Q2cum> 0,5), используют одни и те же классы образцов для построения модели O2PLS-DA (этап 4.2.2).
- В окне рабочего набора, выберите PCA-X в качестве типа модели. Нажмите Autofit. Обратите внимание на R2x (диплом) и Q2 (диплом) значения, поскольку они дают представление о качестве модели.
- Построить две модели O2PLS-DA с использованием образцов, классифицированных по макро-зоны и проверки моделей с использованием теста перестановку с 200 перестановок.
- Назначают классы, перейдя в главном окне → New → Как M1 → Наблюдения. Установите нужные классы. Затем измените тип модели от PCA-X для O2PLS-DA. Нажмите Autofit. Убедитесь, что количество компонентов модели PLS-DA такая же, как у O2PLS-DA.
- Для проверки модели O2PLS-DA, перейдите Анализ CV-ANOVA и увидеть правильное значение р. Кроме того, нажмите на новые, как в окне Главная → M2 и изменить тип модели от O2PLS-DA к PLS-DA. Нажмите Autofit.
- Перейти к → Анализ Перестановки и выполнить 200 перестановкам (отмените выбор опции Пересчитать Перестановки).
Примечание: Окончательный вывод показывает окно, в котором значение R2 ShouЛД в целом ударил Y-ось в значениях под 0,4, в то время как Q2 должен ударить отрицательную часть оси ординат. Если значения R2 и / или Q2 являются неправильными, уменьшить число компонентов, как в моделях PLS-DA и O2PLS-DA. - Продолжить, чтобы выбрать M2 в окне проекта и нажмите на Scores → Scatter, чтобы посмотреть сюжет и положение классов образцов. Для того, чтобы наблюдать, что метаболиты характеризуют один или несколько конкретных классов, перейдите на участок / List → Scatter.
- Изменение Выберите тип данных для наблюдения и Нагрузки → Добавить Series и изменить позицию в X-оси и серии в рд (Corr) и Pred Comp в 1 и 2 или дополнительных компонентов, когда присутствует, соответственно.
- С правой кнопка мыши нажата на участке, перейдите к недвижимости → Цвет и выберите По условиям различать символы сюжетных среди молекул и классов. Перейти к Layout → Формат → Участок оси и / или стилей, чтобы изменить сюжет с требуемыми характеристиками.
- На основании результатов метаболомике O2PLS-DA анализа, существующие различия в относительных уровней специфических метаболитов или классов метаболитов в качестве гистограмм.
- На основании результатов анализа транскриптомика O2PLS-DA, извлекать и назначать дифференцированно модулированные генов Джин Онтология (ГО) классификаций 28.
- Выявление взаимосвязи между уровнями метаболита и экспрессии генов вручную.
- Реализация PCA , как показано на F igure 2. В этом случае исследование, PCA раскрывает основные различия между образцами, отражающие различные стадии созревания и растущих сезонов.
Результаты
Конкретное исследование описано в этой статье дали окончательную матрицу данных , содержащую 552 сигналов (m / z признаки) , включая молекулярных ионов плюс их изотопов, аддуктов и некоторых фрагментов, относительно количественно среди 189 образцов (7 виноградников х 3...
Обсуждение
В данной статье описываются метаболомике транскриптомика и протоколы статистического анализа, используемые для интерпретации концепции терруар винограда ягоды. Анализ Метаболомика с помощью ВЭЖХ-ESI-MS достаточно чувствительны, чтобы обнаружить большое количество метаболитов одновр...
Раскрытие информации
The authors have nothing to disclose.
Благодарности
This work benefited from the networking activities coordinated within the EU-funded COST ACTION FA1106 "An integrated systems approach to determine the developmental mechanisms controlling fleshy fruit quality in tomato and grapevine". This work was supported by the 'Completamento del Centro di Genomica Funzionale Vegetale' project funded by the CARIVERONA Bank Foundation and by the 'Valorizzazione dei Principali Vitigni Autoctoni Italiani e dei loro Terroir (Vigneto)' project funded by the Italian Ministry of Agricultural and Forestry Policies. SDS was financed by the Italian Ministry of University and Research FIRB RBFR13GHC5 project "The Epigenomic Plasticity of Grapevine in Genotype per Environment Interactions".
Материалы
| Name | Company | Catalog Number | Comments |
| Mill Grinder | IKA | IKA A11 basic | |
| HPLC Autosampler | Beckman Coulter | - | System Gold 508 Autosampler |
| HPLC System | Beckman Coulter | - | System Gold 127 Solvent Module HPLC |
| C18 Guard Column | Grace | - | Alltima HP C18 (7.5 mm x 2.1 mm; 5 μm) Guard Column |
| C18 Column | Grace | - | Alltima HP C18 (150 mm x 2.1 mm; 3 μm) Column |
| Mass Spectometer | Bruker Daltonics | - | Bruker Esquire 6000; The mass spectometer was equipped with an ESI source and the analyzer was an ion trap. |
| Extraction solvents and HPLC buffers | Sigma | 34966 | Methanol LC-MS grade |
| Sigma | 94318 | Formic acid LC-MS grade | |
| Sigma | 34967 | Acetonitrile LC-MS grade | |
| Sigma | 39253 | Water LC-MS grade | |
| Minisart RC 4 Syringe filters (0.2 μm) | Sartorius | 17764 | |
| Softwares for data collection (a) and processing (b) | Bruker Daltonics | - | Bruker Daltonics Esquire 5.2 Control (a); Esquire 3.2 Data Analysis and MzMine 2.2 softwares (b) |
| Spectrum Plant Total RNA kit | Sigma-Aldrich | STRN250-1KT | For total RNA extractino from grape pericarps |
| Nanodrop 1000 | Thermo Scientific | 1000 | |
| BioAnalyzer 2100 | Agilent Technologies | G2939A | |
| RNA 6000 Nano Reagents | Agilent Technologies | 5067-1511 | |
| RNA Chips | Agilent Technologies | 5067-1511 | |
| Agilent Gene Expression Wash Buffer 1 | Agilent Technologies | 5188-5325 | |
| Agilent Gene Expression Wash Buffer 2 | Agilent Technologies | 5188-5326 | |
| LowInput QuickAmp Labeling kit One-Color | Agilent Technologies | 5190-2305 | |
| Kit RNA Spike In - One-Color | Agilent Technologies | 5188-5282 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-5242 | |
| RNeasy Mini Kit (50) | Qiagen | 74104 | For cRNA Purification |
| Agilent SurePrint HD 4X44K 60-mer Microarray | Agilent Technologies | G2514F-048771 | |
| eArray | Agilent Technologies | - | https://earray.chem.agilent.com/earray/ |
| Gasket slides | Agilent Technologies | G2534-60012 | Enable Agilent SurePrint Microarray 4-array Hybridization |
| Thermostatic bath | Julabo | - | |
| Hybridization Chamber | Agilent Technologies | G2534-60001 | |
| Microarray Hybridization Oven | Agilent Technologies | G2545A | |
| Hybridization Oven Rotator Rack | Agilent Technologies | G2530-60029 | |
| Rotator Rack Conversion Rod | Agilent Technologies | G2530-60030 | |
| Staining kit | Bio-Optica | 10-2000 | Slide-staining dish and Slide rack |
| Magnetic stirrer device | AREX Heating Magnetic Stirrer | F20540163 | |
| Thermostatic Oven | Thermo Scientific | Heraeus - 6030 | |
| Agilent Microarray Scanner | Agilent Technologies | G2565CA | |
| Scanner Carousel, 48-position | Agilent Technologies | G2505-60502 | |
| Slide Holders | Agilent Technologies | G2505-60525 | |
| Feature extraction software v11.5 | Agilent Technologies | - | inside the Agilent Microarray Scanner G2565CA |
| SIMCA + V13 Software | Umetrics |
Ссылки
- Jessome, L. L., Volmer, D. A. Ion suppression: A major concern in mass spectrometry. Lc Gc N Am. 24 (5), 498-510 (2006).
- Kim, H. K., Choi, Y. H., Verpoorte, R. NMR-based plant metabolomics: where do we stand, where do we go?. Trends Biotech. 29 (6), 267-275 (2011).
- Sumner, L. W., Mendes, P., Dixon, R. A. Plant metabolomics: large-scale phytochemistry in the functional genomics era. Phytochem. 62 (6), 817-836 (2003).
- Bottcher, C., von Roepenack-Lahaye, E., Willscher, E., Scheel, D., Clemens, S. Evaluation of matrix effects in metabolite profiling based on capillary liquid chromatography electrospray ionization quadrupole time-of-flight mass spectrometry. Anal Chem. 79 (4), 1507-1513 (2007).
- Toffali, K., et al. Novel aspects of grape berry ripening and post-harvest withering revealed by untargeted LC-ESI-MS metabolomics analysis. Metabolomics. 7 (3), 424-436 (2011).
- Martin, J. C., et al. Can we trust untargeted metabolomics? Results of the metabo-ring initiative, a large-scale, multi-instrument inter-laboratory study. Metabolomics. 11 (4), 807-821 (2015).
- Jaillon, O., et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 449 (7161), 463-467 (2007).
- Velasco, R., et al. A high quality draft consensus sequence of the genome of a heterozygous grapevine variety. Plos One. 2 (12), (2007).
- Tornielli, G. B., Zamboni, A., Zenoni, S., Delledonne, M., Pezzotti, M., Gerós, H., Chaves, M., Delrot, S. Ch. 11. The Biochemestry of the Grape Berry. 11, (2012).
- Anesi, A., et al. Towards a scientific interpretation of the terroir concept: plasticity of the grape berry metabolome. BMC Plant Biol. 15, 1-17 (2015).
- Berdeja, M., et al. Water limitation and rootstock genotype interact to alter grape berry metabolism through transcriptome reprogramming. Hort Res. 2, 1-13 (2015).
- Carbonell-Bejerano, P., et al. Solar ultraviolet radiation is necessary to enhance grapevine fruit ripening transcriptional and phenolic responses. BMC Plant Biol. 14, 1-16 (2014).
- Carbonell-Bejerano, P., et al. Reducing sampling bias in molecular studies of grapevine fruit ripening: transcriptomic assessment of the density sorting method. Theor Exp Plant Phys. 28 (1), 109-129 (2016).
- Carbonell-Bejerano, P., et al. Circadian oscillatory transcriptional programs in grapevine ripening fruits. BMC Plant Biol. 14, 1-15 (2014).
- Cavallini, E., et al. Functional diversification of grapevine MYB5a and MYB5b in the control of flavonoid biosynthesis in a petunia anthocyanin regulatory mutant. Plant & Cell Physiol. 55 (3), 517-534 (2014).
- Cramer, G. R., et al. Transcriptomic analysis of the late stages of grapevine (Vitis vinifera cv. Cabernet Sauvignon) berry ripening reveals significant induction of ethylene signaling and flavor pathways in the skin. BMC Plant Biol. 14, 1-21 (2014).
- Dal Santo, S., et al. The plasticity of the grapevine berry transcriptome. Genome Biol. 14 (6), 1-17 (2013).
- Fasoli, M., et al. The Grapevine Expression Atlas Reveals a Deep Transcriptome Shift Driving the Entire Plant into a Maturation Program. Plant Cell. 24 (9), 3489-3505 (2012).
- Gambino, G., et al. Co-evolution between Grapevine rupestris stem pitting-associated virus and Vitis vinifera L. leads to decreased defence responses and increased transcription of genes related to photosynthesis. J Exp Bot. 63 (16), 5919-5933 (2012).
- Ghan, R., et al. Five omic technologies are concordant in differentiating the biochemical characteristics of the berries of five grapevine (Vitis vinifera L.) cultivars. BMC Genomics. 16 (1), 1-26 (2015).
- Pastore, C., et al. Selective defoliation affects plant growth, fruit transcriptional ripening program and flavonoid metabolism in grapevine. BMC Plant Biol. 13, 1-13 (2013).
- Pastore, C., et al. Increasing the source/sink ratio in Vitis vinifera (cv Sangiovese) induces extensive transcriptome reprogramming and modifies berry ripening. BMC Genomics. 12, 1-23 (2011).
- Rinaldo, A. R., et al. A Grapevine Anthocyanin Acyltransferase, Transcriptionally Regulated by VvMYBA, Can Produce Most Acylated Anthocyanins Present in Grape Skins. Plant Physiol. 169 (3), 1897-1916 (2015).
- Royo, C., et al. Developmental, transcriptome, and genetic alterations associated with parthenocarpy in the grapevine seedless somatic variant Corinto bianco. J Exp Bot. , 259-273 (2015).
- Venturini, L., et al. De novo transcriptome characterization of Vitis vinifera cv. Corvina unveils varietal diversity. BMC Genomics. 14, 1-13 (2013).
- Commisso, M., Strazzer, P., Toffali, K., Stocchero, M., Guzzo, F. Untargeted metabolomics: an emerging approach to determine the composition of herbal products. Comput Struct Biotechnol J. 4, 1-7 (2013).
- Pluskal, T., Castillo, S., Villar-Briones, A., Oresic, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 1-11 (2010).
- Ashburner, M., et al. Gene Ontology: tool for the unification of biology. Nat Genet. 25 (1), 25-29 (2000).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены