Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Das Terroir-Konzept Interpretiert durch Grape Berry Metabolomics und Transcriptomics
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt die Anwendung von ungezielte Metabolomik, Transcriptomics und multivariate statistische Analysen Traubenbeere Transkripte und Metaboliten , um einen Einblick in das Terroir - Konzept zu gewinnen, das heißt, die Auswirkungen der Umwelt auf die Beere Qualitätsmerkmale.
Zusammenfassung
Terroir bezieht sich auf die Kombination von Umweltfaktoren , die die Eigenschaften von Kulturen wie Weinreben (Vitis vinifera) nach bestimmten Habitaten und Managementpraktiken beeinflussen. Dieser Artikel zeigt, wie bestimmte Terroir Signaturen können in der Beere Metabolom und Transkriptom der Weinrebe Sorte Corvina mit multivariaten statistischen Analyse nachgewiesen werden. Das Verfahren erfordert zunächst eine geeignete Stichprobenplan. In dieser Fallstudie wurde ein spezifischer Klon der Corvina Sorte ausgewählten genetischen Unterschiede zu minimieren und Proben von sieben Weinbergen gesammelt wurden, die drei verschiedene Makro Zonen während der drei verschiedenen Wachstumsphasen. Eine ungezielte LC-MS Metabolomics Ansatz wird durch eine effiziente Datenverarbeitung mit MZmine Software und eine Identifizierung von Metaboliten Strategie auf Fragmentierung Baumanalyse basiert aufgrund seiner hohen Empfindlichkeit, begleitet empfohlen. Umfassende Transkriptom-Analyse kann unter Verwendung von Microarrays erreicht werdenenthaltenden Sonden abdeckt ~ 99% aller Weinrebe Gene vorhergesagt wird, die gleichzeitige Analyse aller differentiell exprimierte Gene im Zusammenhang mit verschiedenen terroirs ermöglicht. Schließlich kann der multivariaten Datenanalyse auf Projektionsverfahren auf Basis verwendet werden, um den starken Jahrgang spezifischen Effekt zu überwinden, so dass die Metabolomik und Transkriptom-Daten zu integrieren und im Detail analysiert werden, um informative Korrelationen zu identifizieren.
Einleitung
Groß angelegte Datenanalyse auf der Basis der Genome, Transkriptom, Proteom und Metabolomen von Pflanzen bietet noch nie da gewesenen Einblick in das Verhalten von komplexen Systemen, wie zum Beispiel die Terroir Eigenschaften von Wein, die die Wechselwirkungen zwischen Weinrebe Pflanzen und ihrer Umwelt zu reflektieren. Da das Terroir eines Weines verschieden sein kann, selbst wenn identische Weinrebe Klone in verschiedenen Weinbergen angebaut werden, ist Genomik Analyse von geringem Nutzen, da die klonalen Genome identisch sind. Stattdessen ist es notwendig, auf Korrelationen zwischen Genexpression und den metabolischen Eigenschaften der Beeren zu suchen, die die Qualitätsmerkmale des Weines bestimmen. Die Analyse der Genexpression auf der Ebene der Transkriptom profitiert von den ähnlichen chemischen Eigenschaften aller Transkripte, die durch Ausnutzung universellen Eigenschaften wie die Hybridisierung an immobilisierte Sonden auf Mikroarrays quantitative Analyse ermöglicht. Im Gegensatz dazu universellen analytischen Methoden in der Proteomik einnd Metabolomik sind schwieriger wegen der großen physikalischen und chemischen Vielfalt einzelner Proteine und Metaboliten. Im Falle von Metabolomics diese Vielfalt ist noch extremer, weil einzelne Metaboliten stark in Größe, Polarität, die Fülle und die Volatilität unterscheiden, so dass keine einzige Extraktionsverfahren oder Analysemethode bietet einen ganzheitlichen Ansatz.
Zu den analytischen Plattformen geeignet für nichtflüchtige Stoffwechselprodukte sind solche auf Basis von Hochleistungs-Flüssigkeitschromatographie gekoppelt mit Massenspektrometrie (HPLC-MS) viel empfindlicher als Alternativen wie HPLC mit ultravioletter oder Diodenarray-Detektoren (HPLC-UV, HPLC-DAD ) oder Kernspinresonanz (NMR) Spektroskopie, aber quantitative Analyse durch HPLC-MS kann durch Phänomene wie die Matrixeffekt und Ionenunterdrückungs / Erweiterung 1-3 beeinflußt werden. Die Untersuchung solcher Effekte bei der Analyse von Corvina Traubenbeeren durch HPLC-MS unter Verwendung einer Elektrospray-Ionisations-Quelle (HPLC-ESI-MS) zeigte, dass Zucker und andere Moleküle mit den niedrigsten Retentionszeiten waren stark underreported, wahrscheinlich auch die große Anzahl von Molekülen in dieser Zone reflektiert, und dass die Fülle anderer Moleküle unterschätzt werden könnte, überschätzt bzw. unbeeinflußt von der Matrixeffekt , aber die Datennormalisierung für die Matrix - Effekt schien begrenzte Auswirkungen auf die Gesamtergebnisse 4,5 zu haben. Das hier beschriebene Verfahren ist für die Analyse von mittlerer Polarität Metaboliten optimiert, die während der Reifung, auf einem hohen Niveau in Traubenbeeren anreichern und werden wesentlich durch die terroir beeinflusst. Dazu gehören Anthocyane, Flavonole, Flavans-3-ole, Procyanidine, andere Flavonoide, Resveratrol, Stilbenen, Hydroxyzimtsäuren und Hydroxybenzoesäuren, die zusammen bestimmen die Farbe, Geschmack und gesundheitsbezogenen Eigenschaften von Weinen. Andere Stoffwechselprodukte, wie Zuckern und aliphatischen organischen Säuren, werden ignoriert, da die Quantifizierung durch HPLC-MS unverlässlich ist aufgrund der Matrix effect und Ionensuppression Phänomene 5. Innerhalb der Polaritätsbereich durch diese Methode gewählt, ist der Ansatz ungezielte, dass es zielt darauf ab , so viele verschiedene Stoffwechselprodukte wie möglich 6 zu erkennen.
Transcriptomics Methoden , die Tausende von Weinrebe Transkripten erlauben gleichzeitig überwacht werden sollen , durch die Verfügbarkeit des gesamten Weinrebe Genomsequenz 7,8 erleichtert. Frühe Transkriptomik Methoden basieren auf Hochdurchsatz-cDNA-Sequenzierung haben mit dem Aufkommen der nächsten Generation Sequenzierung in eine Sammlung von Prozeduren gemeinsam beschrieben als RNA-Seq-Technologie entwickelt. Dieser Ansatz wird schnell die Methode der Wahl für Transkriptomik Studien. eine große Menge an Literatur jedoch auf Mikroarrays, die durch Hybridisierung in parallel quantifiziert Tausende von Transkripten ermöglicht werden, hat für Weinrebe akkumuliert. Tatsächlich bevor RNA-Seq eine Mainstream-Technologie wurde, hatte viele engagierte kommerziellen Microarray-Plattformen gewesenentwickelt, mit dem Weinrebe Transkriptom zu sehr detailliert untersucht werden. Unter der Vielfalt von Plattformen, nur zwei erlaubt genomweite Transkriptomanalyse 9. Die entwickelte Anordnung erlaubt die Hybridisierung von bis zu 12 unabhängige Stichproben auf einer einzigen Vorrichtung, wodurch die Kosten für jedes Experiment zu verringern. Die 12 Unterfelder umfasst jeweils 135.000 60-mer-Sonden repräsentieren 29.549 Weinrebe Transkripte. Dieses Gerät wurde von 10-24 in einer großen Anzahl von Studien verwendet. Diese beiden Plattformen sind nun eingestellt , sondern eine neue benutzerdefinierte Microarray wurde vor kurzem entwickelt worden und stellt eine neuere Entwicklung , da sie eine noch größere Anzahl von Sonden enthält , die zusätzliche neu entdeckten Gene Weinrebe 25.
Die groß Verkauf Datensätze von Transcriptomics und Metabolomics-Analyse erfordern geeignete statistische Verfahren zur Datenanalyse produziert, darunter multivariate Techniken Korrelationen zwischen verschiedenen Form zu bestimmen,s von Daten. Die am häufigsten verwendeten Techniken multivariate sind solche auf Projektion basiert, und diese können zu latent Strukturen Diskriminanzanalyse (O2PLS-DA) unüberwachten wie Hauptkomponentenanalyse (PCA), oder überwacht, wie beispielsweise bidirektionale orthogonale Projektion 26. Das Protokoll in diesem Artikel vorgestellt nutzt PCA für die explorative Datenanalyse und O2PLS-DA Unterschiede zwischen den Gruppen von Proben zu identifizieren.
Protokoll
1. Wählen Sie geeignete Materialien und Konstruieren Sie einen Stichprobenplan
- Beginnen Sie mit dem Experiment durch eine geeignete Stichprobenplan zu entwickeln. Es gibt keine allgemeinen und universellen Ansatz so jeden Plan auf einer Basis von Fall zu Fall zu bewerten. Stellen Sie sicher, dass die Stichprobenplan nennt die Probenahme Orte, Zeiten und die genaue Stichprobenverfahren. Siehe Abbildung 1 für den Stichprobenplan in dieser Fallstudie verwendet.
HINWEIS: In dieser Fallstudie, Traubenbeeren von einem einzigen Klon (. Vitis vinifera cv Corvina, Klon 48) wurden in drei verschiedenen Makrozonen in der Provinz Verona (Gardasee, Valpolicella und Soave) von sieben kommerziellen Weinbergen gesammelt. Die wichtigsten Merkmale der einzelnen Weinberg zusammengefasst sind in der Tabelle 1. Beeren wurden während drei Vegetationsperioden gesammelt (2006, 2007 und 2008) zu drei Zeitpunkten, entsprechend veraison (der Beginn der Reifung), Mitte der Reifung und reifen Beeren.- Für jede der Beitritte (vineyard / Jahr / Reifestadium), Ernte 30 Cluster aus verschiedenen Positionen entlang zwei Rebzeilen, mit randomisierten Höhen und Positionen auf der Anlage.
- Wählen drei Beeren zufällig aus jedem Cluster, die mit sichtbarer Schaden und / oder Anzeichen einer Infektion vermieden wird.
- Wiederholen Sie die Schritte 1.1.1 und 1.1.2 drei unabhängige Pools zu erhalten.
- die Beeren und frieren die Perikarp sofort in flüssigem Stickstoff entkernen.
- Crush 10 gefrorene Beeren aus jedem Pool mit einer automatischen Mühle Mühle, und teilen jeden pulverisierte Probe in zwei gleiche Teile, eine für Transkriptomik Analyse und eine für Metabolomics-Analyse.
- Lagern Sie die Pulver bei -80 ° C.
| AM | BA | BM | CS | FA | MN | PM | |
| Makrozone | Soave | Gardasee | Valpolicella | Gardasee | Valpolicella | Valpolicella | Soave |
| Höhe (m) | 250 | 120 | 450 | 100 | 130 | 250 | 130 |
| Unterlagen | 41B | S04 | K5BB | 420A | 420A | K5BB | 41B |
| Zeilenrichtung | EW | NS | EW | EW | EW | NS | NS |
| Das Trainingssystem | Überkopfsystem (Pergola) | Überkopfsystem (Pergola) | Vertikale schießen Positionierung (Guyot) | Überkopfsystem (Pergola) | OverheAnzeige-System (Pergola) | Vertikale schießen Positionierung (Guyot) | Vertikale schießen Positionierung (Guyot) |
| Bodenart | silty Ton | Lehm | Lehm | Lehm | Tonlehm | Schlick-Lehm | Tonlehm |
| Das Einpflanzen Layout (m) | 3,20 x 1,00 | 4,50 x 0,80 | 4,00 x 1,25 | 3,50 x 1,20 | 3.50 x 0.75 | 2,80 x 1,00 | 1,80 x 0,80 |
| Insgesamt Kalk% | 3.9 | 19.3 | 18,3 | 14.4 | 31 | 5.9 | 27.9 |
| Aktive Kalk% | 0,5 | 2.6 | 9.4 | 6.3 | 11.3 | 3.1 | 8.3 |
| Sand% | 15 | 47 | 66 | 42 | 29 | 13 | 36 |
| Loam% | 43 | 36 | 21 | 37 | 39 | 67 | 36 |
| Lehm % | 42 | 17 | 13 | 21 | 32 | 20 | 28 |
| Boden - pH | 8.3 | 7.9 | 7.8 | 8.2 | 8.2 | 7.8 | 7.9 |
| Organische Substanz (%) | 2.9 | 2.5 | 2.2 | 1.2 | 2.9 | 1.6 | 2.5 |
| Auswechselbare Phosphor (mg / kg) | 26 | 73 | 73 | 68 | 48 | 47 | 64 |
| Austauschbares Kalium (mg / kg) | 190 | 376 | 620 | 230 | 168 | 154 | 126 |
| Austauschbares Magnesium (mg / kg) | 272 | 468 | 848 | 623 | 294 | 293 | 183 |
| Auswechselbare Calcium (mg / kg) | 6500 | 5380 | 7358 | 6346 | 4652 | 10055 | 2878 |
| Berry reduzierende Zucker 2006 | 211,25 ± 1,20 | 176,20 ± 0,42 | 187,40 ± 0,00 | 203,70 ± 1,13 | 212,55 ± 0,64 | 195,20 ± 0,00 | 211,65 ± 0,64 |
| Berry reduzierende Zucker 2007 | 190,00 ± 1,27 | 165,25 ± 0,49 | 153,00 ± 0,42 | 203,60 ± 0,71 | 210,90 ± 0,71 | 192,25 ± 0,64 | 188,70 ± 1,84 |
| Berry reduzierende Zucker 2008 | 191,35 ± 0,64 | 178,90 ± 0,57 | 170,05 ± 0,49 | 205,15 ± 1,48 | 188,70 ± 0,57 | 169,35 ± 0,49 | 108,05 ± 1,06 |
| Berry pH 2006 | 3,01 ± 0,01 | 2,96 ± 0,01 | 2,84 ± 0,00 | 2,9 ± 0,00 | 2,98 ± 0,00 | 3,02 ± 0,00 | 3,06 ± 0,01 |
| Berry pH 2007 | 2,97 ± 0,00 | 3,00 ± 0,00 | 2,74 ± 0,00 | 3,07 ± 0,01 | 2,98 ± 0,00 | 2,87 ± 0,01 | 3,09 ± 0,00 |
| Berry pH 2008 | 2,83 ± 0,00 | 3,04 ± 0,01 | 2,71 ± 0,00 | 2,98 ± 0,01 | 2,98 ± 0,00 | 2,82 ± 0,00 | 3,11 ± 0,00 |
| Erntedatum | 2006 | 2007 | 2008 | ||||
| Veraison | 8-Aug | 18-Jul | 12-Aug | ||||
| Mid Ripening | 4-Sep | 8-Aug | 2-Sep | ||||
| Reif | 18-Sep | 29-Aug | 23-Sep |
Tabelle 1: Hauptmerkmale der einzelnen Weinberg undProbensammeltermine. m = Meter, EW = Essen-West, NS = Nord-Süd. 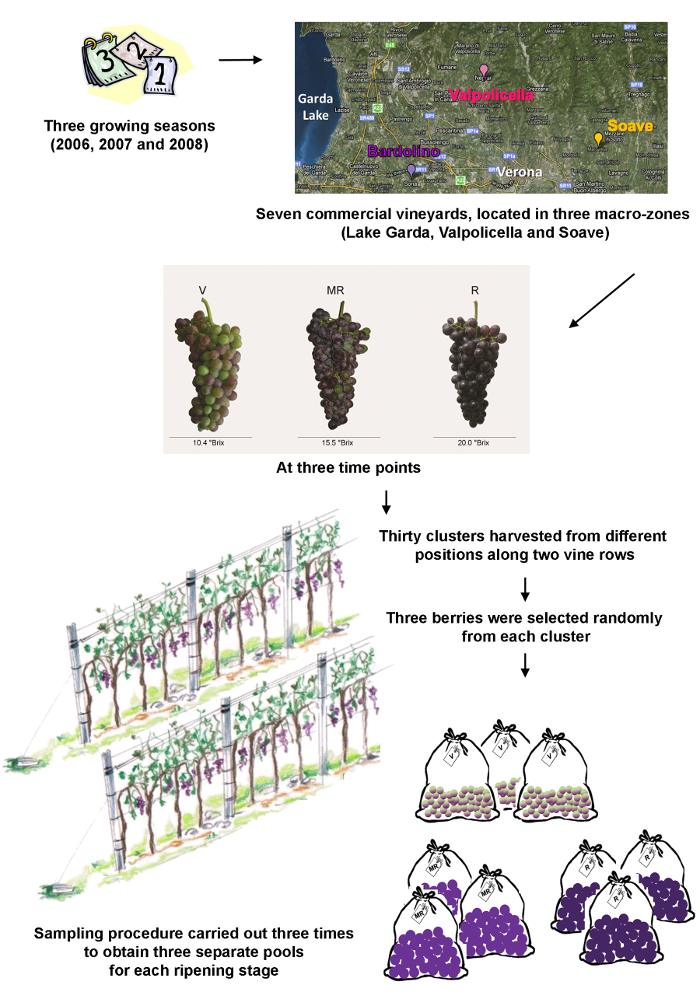
Abb . 1: Schematische Darstellung des Probenahmeverfahren Die drei Weinproduktion Makrozonen werden in der Umgebung der Stadt Verona, Veneto, Italien. Die drei Zeitpunkte sind veraison (V) , die den Ausbruch im Weinbau der Reifung, Mitte der Reifung (MR) und reifen Beeren (R). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
2. Bereiten Sie Berry Powder Auszüge, Analysieren Sie die Metaboliten und die Daten verarbeiten
- Bereiten Sie die Berry Pulverproben für die Analyse.
- Bereiten Sie die metabolischen Extrakte der Beere Proben bei Raumtemperatur in drei Volumina (w / v) Methanol mit 0,1% angesäuert (v / v) fürmic Säure in einem Ultraschallbad bei 40 kHz für 15 min. Verwenden Sie LC-MS-grade Wasser und Ameisensäure und HPLC-Methanol.
- Zentrifugieren der Extrakte bei 16.000 xg für 10 min bei 4 ° C.
- Verdünnen Sie den Überstand aus Schritt 2.1.2 in zwei Bänden (v / v) von entsalztem Wasser und durch ein 0,2-um-Filter.
- Analysieren Sie die Berry Extrakte durch HPLC-ESI-MS.
- Stellen Sie die HPLC-ESI-MS Systems gemäß den Empfehlungen des Lieferanten.
HINWEIS: In dieser Fallstudie umfasste die Einrichtung eines HPLC-Systems mit einem Autosampler ausgestattet, verbunden in-line mit einem Ionenfallen-Massenspektrometer und Elektrospray-Ionisations-Quelle. - Schließen Sie das HPLC - System mit einer C18 - Schutzsäule (7,5 x 2,1 mm 2) und einer Umkehrphasen - C18 - Säule (150 x 2,1 mm 2, Partikelgröße 3 um).
- Bereiten Sie die Lösungsmittel als mobile Phase verwendet. Verwenden von 5% (v / v) Acetonitril, 0,5% (v / v) Ameisensäure in Wasser als Lösungsmittel A und 100% Acetonitril als solvent B. Verwendung LC-MS-Grade Acetonitril, Wasser und Ameisensäure.
- Führen Sie die HPLC - Trennung mit einem linearen Gradienten von Lösungsmittel A und B mit einer konstanten Fließgeschwindigkeit von 0,2 ml min -1 einer Probeninjektionsvolumen von 30 ul verwendet.
- Zunächst äquilibrieren die Säule mit 100% Lösungsmittel A. Probeninjektion folgend, einen Gradienten von 0% bis 10% Lösungsmittel B in 5 min herzustellen, von 10% bis 20% Lösungsmittel B in 20 min, von 20% bis 25% Lösungsmittel B in 5 min, und von 25% bis 70% Lösungsmittel B in 15 min.
- Analysieren Sie jede Probe in zweifacher Ausfertigung. Randomisieren Probenanalyseinstrument getriebene Effekte zu vermeiden. Warten Sie 20 min für die Wiederausgleich mit 100% Lösungsmittel A zwischen jeder Analyse.
- Erwerben Massenspektren in abwechselnden negativen und positiven Ionisierungsmodi. Für die Ergebnisse in dieser Fallstudie beschrieben, stellen Sie die Parameter wie in Tabelle 2. Die genaue Einstellung der Maschine hängt von der jeweiligen Plattform.
HINWEIS: Alternativ können Sie auch andere Anzugkönnen Methoden entsprechend der spezifischen Plattform.- In allen Fällen, wie bei jeder Trennung Plattform, stellen Sie sicher, dass eine geeignete Wieder Equilibrierungszeit zu verwenden, um die Reproduzierbarkeit der Retentionszeit zu haben. Wenn die Anzahl von Abtastwerten hoch ist (über zehn Proben), zu analysieren, sie in Gruppen von 9-10 Proben mit Chromatographiesäule Reinigungsprogramme (langsame Gradienten zwischen den beiden Elutionslösungsmittel) zwischen jeder Charge. Um genügend Reproduzierbarkeit der Retentionszeit haben, sicherzustellen , dass die erste chromatographische Analyse jeder Charge ein Blinduntersuchung (dh Analyse des Lösungsmittels).
- Auch Massenspektren in fragmentierten negativen und positiven Ionen - Modi erwerben Einstellung der Fragmentierung Optionen (zwei Ionenvorstufen, MS 3). Anmerken die Metaboliten durch Fragmentierung Baumanalyse (MS / MS und MS 3) gemäß den Anweisungen des Herstellers.
Hinweis: Dies ist besonders geeignet für die Pflanzenstoffe, weil sie oft glykosyliert sind, therefore die erste Fragmentierung (MS / MS) neigt dazu , die Zuckerreste der freie Aglycon - Ion und anschließender Fragmentierung (MS 3) zu entfernen verlassen hilft , das Aglykon gegen einen authentischen Standards Bibliothek zu identifizieren. - Erwerben Sie MS / MS und MS 3 Spektren im m / z - Bereich 50-1,500 mit einer Fragmentierung Amplitude von 1 V Alternativ können andere geeignete Verfahren entsprechend der spezifischen Plattform.
- Für jedes m / z Signal, vergleichen Sie die MS / MS und MS 3 Fragmentierungsmuster und die Retentionszeiten der authentischen Standards Bibliothek gemäß den Anweisungen des Herstellers.
HINWEIS: Viele Arten von Plattformen umfassen Software Einrichtungen dieser Art von Bibliothek durch die Analyse von authentischen Standards zu bauen. Dies sollte viele der Signale identifizieren, aber nicht alle Signale mit Anmerkungen versehen werden. - Für die verbleibenden anonymen Signale, zu vergleichen , die MS / MS und MS 3 Fragmentierungsmuster mit den in der Literatur, searc veröffentlichtfrei verfügbar, oder Online - Datenbanken verwenden wie MassBank Hing für m / z - Werte (dh Ziffer "m / z 353" für Publikationen zu suchen , in denen solche m / z - Moleküle mit erwähnt werden) mit einem der gemeinsamen Suchmaschine (www .massbank.jp / en / database.html) und der Datenbank (www.hmdb.ca/search/spectra?type=ms_search Human Metabolome).
- Stellen Sie die HPLC-ESI-MS Systems gemäß den Empfehlungen des Lieferanten.
| Massenspektrometer Komponenten | Funktion | Parameter |
| Elektrospray-Ionisations Quelle | Zerstäubertest Gas | 50 psi, 350 ° C |
| Trocknungsgas | 10 L min -1 | |
| Ionenfalle und Detektor | Scan | Voll - Scan - Modus, 13.000 m / z pro Sekunde, der Bereich 50-1,500 m / z |
| Zielmasse | 400 m / z | |
| Kollision Gas | Helium | |
| Vakuum-Druck | 1,4 x 10 -5 mbar | |
| Kapillar-Quelle | 4000 V | |
| Endplatte Offset | -500 V | |
| Schaumlöffel | -40 V | |
| Cap Ausfahrt | -121 V | |
| 1. Oktober DC | -12 V | |
| 2. Oktober DC | -1,7 V | |
| Objektiv 1 | +5 V | |
| Objektiv 2 | +60 V | |
| ICC für positive Ionisierung Modus | 20.000 | |
| ICC für negative Ionisierung Modus | 7.000 |
Tabelle 2: Hauptparametersatz für Massenspektren zu erwerben.
| Betrieb | Auswahl | Funktion | Parameter | Werte |
| Spitzenerfassung | Massendetektion | Centroid | Geräuschpegel | 3500 |
| Chromatogram Bauer | Höchste Datenpunkt | min Zeitspanne | 0,15 | |
| min Höhe | 4000 | |||
| m / z Toleranz | 0,3 | |||
| Spitzenerfassung | Peak-Entfaltungs | Lokale Minimumsuche | Chromatographische Schwelle | 70 |
| Suche Minimum RT (min) | 00.50 | |||
| Minimale relative Höhe | 15% | |||
| Mindest absolute Höhe | 4000 | |||
| Min Verhältnis von Spitzen oben / Rand | 2 | |||
| Dauer (min) | 0-10 | |||
| Isotopes | Isotopen-Peaks Grouper | - | m / z Toleranz | 1.2 |
| RT Toleranz | 00.50 | |||
| monotone Form | Nein | |||
| Der maximale Ausgabeaufschlag | 3 | |||
| Repräsentative Isotop | Nein | |||
| Ausrichtung | Registriert Ausrichter | - | m / z Toleranz | 1.2 |
| Gewicht für m / z | 10 | |||
| Retentionszeit Toleranz | 00.50 | |||
| Gewicht für RT | 5 | |||
| Erfordern gleichen Ladezustand | Nein | |||
| Erfordern gleiche ID | Nein | |||
| Vergleichen Isotopenmuster | Nein | |||
| Eine Lücke stopfen | Peak-Finder | - | Intensity Toleranz | 20% |
| m / z Toleranz | 0,9 | |||
| Retentionszeit Toleranz | 0.40 | |||
| RT Korrektur | Nein | |||
| Filtering | Duplizieren Peak-Filter | - | m / z Toleranz | 1.2 |
| RT Toleranz | 0.30 | |||
| Erfordern gleichen Identifikations | Nein |
Tabelle 3: Mzmine Workflow mit bestimmten Werten negativ LC-MS Traubenbeere Datendateien zu verarbeiten.
- Verarbeiten Sie die LC-MS-Daten.
- Access odereine Datenverarbeitungssoftwarepaket herunterladen, die relevanten Informationen aus vielen rohen Chromatogramme extrahieren und eine Datenmatrix, in dem jeder erfassten Metaboliten in jeder Probe bauen quantifiziert.
HINWEIS: Das Protokoll unten Schritte sind für die Open-Source-Software MZmine v2.14 zugeschnitten (http://mzmine.sourceforge.net). Es ist ein Open-Source - Software für die massenspektrometrische Datenverarbeitung, mit dem Schwerpunkt auf der LC-MS - Daten. 27 - Verwandeln Sie die LC-MS Chromatogrammdaten in netCDF-Format mit der Software vom Gerätehersteller zur Verfügung gestellt. Für die hier beschriebenen Ergebnisse, verwenden Sie die Bruker Daltonics Esquire v5.2 und v3.2 Datenanalyse-Software und Schritte ausführen, gemäß den Anweisungen des Herstellers.
Hinweis: andere Wandler verwendet werden können (verschiedene frei-ware-Wandler zur Verfügung stehen). - Importieren Sie die .cdf Dateien in die Software.
- Implementieren Sie die Spitzenerfassung, Ausrichtung, Spaltfüllung und Spitzenfilterverfahren mit dem parameters in Tabelle 3.
- Als ersten Schritt wählen Sie eine importierte .cdf Datei, um Visualisierung → TIC / XIC Visualizer gehen dann. Setzen Sie den Cursor auf der Basis des kleinsten Peaks im Chromatogramm und zur Kenntnis nehmen über die Mindestintensität Signal der Basis Ion. Dann gehen Sie auf Rohdaten Methoden → Spitzenerfassung.
- Wählen Sie Massenerkennung und füllen Sie den Signalpegel zu erfassen, einzelne Ionen für jeden Scan und schaffen eine Ionen Liste.
HINWEIS: Überprüfen Sie den Algorithmus vor der Massendetektion durchführen - es hängt von dem Massenspektrometer. In unserem Fall wählen wir Centroid. - Wählen Sie Chromatogram Builder und füllen mit dem Signalpegel, um Datenpunkte aus der Ionenliste verbinden und ein Chromatogramm für jeden Massewert aufzubauen. Siehe die angegebenen Parameter in der Bedienungsanleitung des Massenspektrometers die Mindestzeitspanne und m / z Toleranz einzustellen.
- Weiter finden Sie auf Peak List Methoden → Spitzenerfassung → Chromatogram Deconvolution. Wählen Sie den entsprechenden Algorithmus (in diesem Fall Local Minimum Search) die Trennung des Chromatogramms in einzelne Spitzen zu ermöglichen.
- inspizieren Sie manuell die Chromatogramme die entsprechenden Werte für die folgenden Parameter, um herauszufinden. Stellen Sie die chromatographischer Schwelle auf 30%, das Rauschen zu entfernen und Suche Minimum in RT Bereich bis 2 (min) die Anwesenheit von mindestens Einheimischen zu identifizieren zwei Spitzen zu unterscheiden.
- Set Mindest Relative Höhe bei 2,0. inspizieren manuell das Chromatogramm die minimale absolute Höhe des Signals zu identifizieren, die auf einen Spitzenwert und nicht dem Hintergrund entspricht; setzen Sie diesen Wert als absolute Höhe (etwa 10.000 im dargestellten Experiment).
- Set Min Verhältnis von Spitzen Top / Edge 1.1 das Mindestverhältnis zwischen der Spitze zu behaupten und den niedrigsten Datenpunktintensitäten eines Peaks als wahre Spitze erkannt zu werden.
- inspizieren Sie manuell die Chromatogramme zu sehen, welche die Mindestdauer der verschiedenen Spitzen in der verwendeten Chromat istographic Bedingungen (zum Beispiel zwischen 0,2 und 2 min auf der Verbindung abhängig, in den vorgestellten Experimenten) und damit diese Werte als Peak-Dauer (min) den Bereich der zulässigen Spitzenlänge als Dauer einzustellen.
- Dann gehen Sie zu Peak List Methoden → Isotopes → Isotopen-Peaks Grouper zu Gruppe-Isotope in einem Peak, in der Regel in der intensivsten. Hinweis: Die Parameter sind abhängig von der Auflösung des Massenspektrometers und die Reproduzierbarkeit der Retentionszeiten.
- Schließlich gehen Sie zu Peak List Methoden → Ausrichtung → Join Aligner Spitzen auszurichten je nach ihrer m / z und die Retentionszeit durch ein Match - Score.
- Nachdem die Spitzen ausrichten, füllen auf Peak List Methoden → Gap Filling → Peak-Finder alle Datenlücken durch einen Klick. Schließlich Filterdatenpunkte duplizieren, indem Sie auf Peak List Methoden klicken → Filter → Peak-Filter duplizieren.
- Exportieren Sie die resultierende Datenmenge als CS-v Datei.
- Ändern Sie manuell die. Csv Erweiterung auf eine. Txt - Erweiterung. Wenn keine Dateierweiterungen sichtbar sind, die Computereinstellungen ändern Dateierweiterungen zu offenbaren. Go Explorer Optionen → Ansicht → Erweiterte Einstellungen auf Datei und deaktivieren Sie das Feld "Hide Erweiterungen bei bekannten Dateitypen".
- Importieren Sie die TXT - Dateien in eine Tabelle. Dies erzeugt eine Datenmatrix , in der alle nachgewiesenen Metaboliten, erkannt durch eine Identifikationsnummer, m / z - Wert und die Retentionszeit, in allen Proben in Bezug auf ihre Peakflächenwerte quantifiziert.
- Access odereine Datenverarbeitungssoftwarepaket herunterladen, die relevanten Informationen aus vielen rohen Chromatogramme extrahieren und eine Datenmatrix, in dem jeder erfassten Metaboliten in jeder Probe bauen quantifiziert.
3. Bereiten Sie Berry Powder Extrakte für Transkriptom-Analyse und die Daten verarbeiten
- Extrahieren Sie Gesamt-RNA aus den Berry Proben und Bestimmen Sie die RNA-Qualität.
- Extrahieren Sie die Gesamt-RNA aus den Beerenproben.
HINWEIS: In dieser Fallstudie, ein proprietäres Kit und ein modifiziertes Verfahren, das die vollständige Entfernung von Molekülen, die unter gewährleistetfere mit RNA-Analyse, wie Polysaccharide und Polyphenole, verwendet wurden. Die folgenden Anweisungen sind spezifisch für dieses Kit 18.- Man wiegt 400 mg Beerenpulver für jede Probe, teilen sie in zwei Teile und Platz 200 mg jeweils in zwei Mikrozentrifugenröhrchen.
- Hinzufügen, 900 & mgr; l Lyse-Lösung, die β-Mercaptoethanol (im Kit bereitgestellt) zu je 200 mg Pulver und vortex sofort und kräftig für mindestens 30 sec. Die Probe wird bei 56 ° C für 5 Minuten bei 800 Umdrehungen pro Minute schütteln.
- Zentrifugiere die Proben bei maximaler Geschwindigkeit in einer Benchtop-Mikrofuge für 10 min um Zelltrümmer zu pelletieren.
- Pipette 700 ul des Überstands in die Filtrationssäule in dem Kit (blue Haltering) sitzt, die in einer 2 ml Sammelröhrchen. Schließen der Kappe und Zentrifuge bei maximaler Geschwindigkeit in einer Benchtop-Mikrofuge für 1 min Restbruchstücke zu entfernen. Wiederholen Sie diesen Schritt zweimal die gleiche Filtrations Spalte aber einen frischen Sammelröhrchen, die sich in drei tUBEs jeweils ~ 700 & mgr; l des geklärten Lysats.
- Pipettieren 750 ul Bindungslösung (im Kit mitgeliefert) in jedes Röhrchen von geklärten Lysat und mischen sofort durch Pipettieren von oben und unten mindestens fünfmal. Übertragung von 700 & mgr; l dieser Mischung an die Bindungsspalte in dem Kit (rot Haltering) sitzt, die in einer 2 ml Sammelröhrchen. Schließen Sie die Kappe und Zentrifuge bei maximaler Geschwindigkeit in einer Benchtop Mikrofuge für 1 min, um die RNA zu binden.
- Dekantiert die Durchflussfraktion, das gesamte Sammelröhrchen und tippen Sie kurz auf eine saubere Unterlage aus absorbierendem Papier die restliche Flüssigkeit abzulassen.
- Bringen Sie die Spalte an die Sammelröhrchen und pipettieren die verbleibende Mischung in der gleichen Spalte und wiederholen Sie die Zentrifugation und Abgießen. Wiederholen, bis die gesamte Mischung wurde in die gleiche rote Bindung Säule gefiltert.
- Nun folgen Sie den weiteren Anweisungen im Kit und eluieren die RNA in 50 & mgr; l Elutionspuffer (im Kit mitgeliefert) und Speicher it bei -80 ° C bereit für die Qualitätskontrolle Schritte.
- Bestimmen die Menge und Reinheit der RNA unter Verwendung eines Spektrophotometers. Notieren Sie sich die Absorptionsverhältnisse , die den Grad der Kontamination mit Proteinen (A 260/280) und Polyphenole / Polysaccharide (A 260/230) offenbaren.
HINWEIS: RNA geeignet für Microarray-Hybridisierung sollte mindestens 1,8 für beide Verhältnisse punkten. - Bestimmen, um die Integrität der RNA.
HINWEIS: Verschiedene Systeme verwendet werden können. Ein digitaler Erwerber, der eine Kapillar-Elektrophorese-Lauf in Kombination mit einem fluoreszierenden Farbstoff führt wurde in dieser Fallstudie verwendet. RNA geeignet für die Mikroarray-Hybridisierung ist eine RNA Integrity Number (RIN) von mindestens 8 aufweisen.
- Extrahieren Sie die Gesamt-RNA aus den Beerenproben.
- Vorbereitung der Proben und hybridisieren die RNA in eine angepasste Microarray.
- Stellen Sie den Ausgangsbetrag der Gesamt-RNA für Microarray-Analyse zu 200 ng durch die RNA-Lösung in dem Schritt 3.1.1.8 mit entsalztem RNase-freiem Wasser erhalten zu verdünnen. Ergänzen Sie diegeeignete Menge an RNA in ein 1,5 ml Mikrozentrifugen-Röhrchen in einem Endvolumen von 1,5 & mgr; l.
- In 2 ul verdünnt Spike-Mix zu jeder RNA-Probe und folgen Sie den Anweisungen des Herstellers erste cDNA-Strang zu synthetisieren, transkribieren diese in cRNA und beschriften Sie die cRNA mit Cyanin 3CTP.
- Man reinige den markierten cRNA gemäß den Anweisungen des Herstellers und Eluieren in 30 & mgr; l RNase-freiem Wasser.
- Bestimmen Sie die Ausbeute und die spezifische Aktivität jedes cRNA durch Aufzeichnung drei Werte auf einem Spektrophotometer: Cyanin 3 Farbstoffkonzentration (pmol ul -1), RNA Reinheit (A 260/280) und cRNA - Konzentration (ng & mgr; l -1). Verwenden Sie die gefundenen Formeln in der Bedienungsanleitung des Herstellers der cRNA-Ausbeute (ug) und die spezifische Aktivität (pmol Cy3 pro ug cRNA) zu berechnen.
HINWEIS: Empfohlene Ausbeuten und spezifischen Aktivitäten unterscheiden sich auf der Grundlage des spezifischen Mikroarray-Format. In dieser Fallstudie ein 4-Pack 44K-Format wargewählt, war die empfohlene Ausbeute 1,65 und war die spezifische Aktivität 9. - Entwerfen Sie die benutzerdefinierte Microarray mit geeigneter Software für Sondendesign.
- Für die Ergebnisse in dieser Fallstudie beschrieben, bereiten Sie einen neuen benutzerdefinierten Microarray, entworfen auf dem 4-Pack 44K-Format durch eine webbasierte Anwendung für benutzerdefinierte Microarray-Designs und Oligonukleotid-Bibliotheken. Design - Sonden 34.651 Zieltranskripten übereinstimmen, einschließlich 29.971 vorhergesagt Transkripte aus dem Pinot noir V1 Array, 4500 neue Loci in der Sorte Pinot identifiziert durch Corvina Transkriptom Rekonstruktion und 180 privaten Corvina Gene 25.
HINWEIS: Dies beinhaltete die Produktion von 34.651 spezifischen 60-mer-Sonden, bestehend aus 29.798 Pinot noir Transkripte vorhergesagt, 4392 neue Pinot loci und 179 Corvina privaten Gene.
- Für die Ergebnisse in dieser Fallstudie beschrieben, bereiten Sie einen neuen benutzerdefinierten Microarray, entworfen auf dem 4-Pack 44K-Format durch eine webbasierte Anwendung für benutzerdefinierte Microarray-Designs und Oligonukleotid-Bibliotheken. Design - Sonden 34.651 Zieltranskripten übereinstimmen, einschließlich 29.971 vorhergesagt Transkripte aus dem Pinot noir V1 Array, 4500 neue Loci in der Sorte Pinot identifiziert durch Corvina Transkriptom Rekonstruktion und 180 privaten Corvina Gene 25.
- Bereiten Sie die Hybridisierungsanordnung basiert auf 4-Pack-Formatspezifikationen wie folgt.
- Platzieren 1,65 ug Cy3-markierten cRNA in einem Endvolumen von 41,8ul entsalztem RNase-freies Wasser. In 11 ul 10x Blockierungsmittel und 2,2 ul 25x Fragmentation Buffer. bei 60 ° C inkubieren für 30 min in einem thermostatischen Bad RNA zu fragmentieren. Ab sofort kühl auf Eis für 1 Min.
- Schließlich, fügen Sie 55 ul 2x Hybridisierungspuffer, gut mischen durch Pipettieren und Spin für 1 min bei 15.500 × g bei RT. Stellen umgehend Mikrozentrifugenröhrchen auf Eis. Verwenden Sie sofort, es lagern.
- Legen Sie das benutzerdefinierte Microarray wie folgt.
- Legen Sie eine Dichtung Folie mit dem Etikett in die Basis einer Hybridisierungskammer nach oben zeigt. Last langsam 100 ul Hybridisierungsprobe in Schritt erhaltenen gut 3.2.6.2 auf jeder Dichtung, die Flüssigkeit mit der Spitze der Pipette Abgabe vermieden Blasen.
- Langsam setzen die individuelle Microarray nach unten ein, um sicherzustellen, dass die numerische Barcode nach oben zeigt. Stellen Sie sicher, dass das Sandwich-Paar richtig ausgerichtet ist. Schließlich legen die Abdeckung der Hybridisierungskammer auf die SandwichDias und die Klammer auf die Kammer handfest anziehen. Drehen Sie die zusammengebauten Kammer, um die Mobilität von Blasen zu beurteilen.
- Ort versammelt Gleitkammer in einem Bräter in einem Hybridisierungsofen auf 65 ° C. Stellen Sie den Rotator bei 10 Umdrehungen pro Minute dreht. Lassen Sie die Hybridisierung für 17 Stunden, um fortzufahren.
- Fahren Sie mit dem Microarray Objektträger waschen wie folgt.
- Zuerst bereiten drei Einschub färbekasten und füllen sie mit den entsprechenden Waschpuffer: 2 Geschirr mit Waschpuffer 1 bei RT und 1 Schale mit vorgewärmten (37 ° C) Waschpuffer 2.
- Zerlegen Sie die Hybridisierungskammer und entfernen Sie das Sandwich. Mit dem Microarray-Dia numerischen Barcode nach oben, versenken Sie das Sandwich in die erste Einschub Färbetrog gefüllt mit Waschpuffer 1 bei RT und mit Hilfe von sauberen Pinzette, trennen Sie die Dichtung aus dem Microarray-Folie. Schnell übertragen Sie die Microarray-Folie in eine Dia-Rack und legen Sie sie in der zweiten Einschub Färbetrog mit Waschpuffer gefüllt 1bei RT.
- Setzen Sie die Dia-Färbetrog auf einem Magnetrührer und waschen 1 min unter mäßigem Rühren. Schnell übertragen Sie den Schiebegestell in die dritte Einschub Färbekasten gefüllt mit vorgewärmten (37 ° C) Waschpuffer 2 und 1 min unter mäßigem Rühren waschen.
- Entfernen Sie langsam den Ständer aus dem Einschub Färbekasten und vorsichtig die Folie aus dem Rack und vermeidet Tröpfchen zu entfernen.
HINWEIS: Verwenden Sie nicht Triton X-102 zu den Waschpuffer hinzufügen und das Acetonitril Waschschritt auslassen.
- Bewahren Sie die gewaschenen Chip im Dunkeln bei Raumtemperatur.
- Scannen Sie den Microarray und Extrahieren Sie die Auffallende Eigenschaften.
- Platzieren Sie den Microarray-Objektträger in einen geeigneten Scanner und scannen jede Array mit den Parameter empfohlenen Einstellungen in die Anleitung des Microarray-Hersteller. Für die hier beschriebenen Ergebnisse legen jedes Microarray-Folie in eine Dia-Halter den Scanvorgang zu erleichtern.
- Importieren Sie die Ausgabe SHPDatei in einer geeigneten Software, die ein digitales Signal in numerische Werte fluoreszierendes umwandeln kann. Überprüfen Sie die Qualitätskontrolle Bericht, um sicherzustellen, dass das Hybridisierungsverfahren erfolgreich war.
- Für die hier beschriebenen Ergebnisse, verwenden Sie die Parameter - Einstellungen in der Bedienungsanleitung des Merkmalsextraktion Software empfohlen und die Qualitätskontrolle Bericht überprüfen , um sicherzustellen , dass die Parameter in Tabelle 4 im normalen Bereich vom Hersteller angegeben sind.
| Metric - Name | Höchstgrenze | Untere Grenze | Beschreibung |
| AnyColorPrcntFeatNonUnif | 1,00 | N / A | Prozentsatz der Funktionen, die Funktion Uneinheitlichkeit Ausreißer in einem der Kanäle |
| Nachweisgrenze | 2.00 | 0,10 | Durchschnittliche plus 1 Standardabweichung der Spitze ins unterhalb der linearen Konzentrationsbereich |
| absGE1E1aSlope | 1.20 | 0,90 | Absolute der Steigung fit für Signal gegen die Konzentration von E1a-Sonden |
| MedCVProcSignal | 8.00 | N / A | Median% CV für das verarbeitete Signal |
| gNegCtrlAveBGSubSig | 5.00 | -10.00 | Durchschnitt der Hintergrund subtrahiert Signal aller inlier negativen Kontrollen (BGSubSignal durch Substraktion Wert genannt BGUsed aus dem Merkmal bedeuten Signal berechnet wird) |
| gNegCtrlAveNetSig | 40.00 | N / A | Durchschnittliche Netto-Signal aller inlier Negativkontrollen |
| gNegCtrlSDevBGSubSig | 10.00 | N / A | Die Standardabweichung vonHintergrund subtrahiert Signale aller inlier Negativkontrollen |
| gNonCntrlMedCVProcSignal | 8.00 | N / A | Median% CV für das verarbeitete Signal der nicht-Kontrollsonden |
| gSpatialDetrendRMSFilter | 15.00 | N / A | Rest der Hintergrund Detrending fit |
Tabelle 4: Wesentliche Parameter geprüft werden , die Qualität der Mikroarray - Hybridisierung zu überprüfen.
- Verarbeiten Sie die Microarray-Daten.
- Sobald alle Microarray - Slides gescannt wurden und die Qualitätskontrolle positiv bewertet worden, bereiten Sie einen Tab-getrennte Daten-Matrix , die durch die gProcessedSignal Werte von jedem einzelnen Sub - Array - Ergebnis - Datei auswählen, die die rohen Fluoreszenzintensitäten jeder Sonde.
- In einer Tabelle, normalisieren die Daten auf den 75 - Perzentil in jedem Array (P - Werte) und calculaß den Mittelwert aller P - Werte unter allen unterschiedlichen Subarrays das R - Verhältnis zu berechnen.
- Dann wird auf der gleichen Tabellenkalkulationsprogramm , zu normalisieren jedes gProcessedSignal Wert dem Verhältnis R der eigenen Unterarray.
4. Führen Sie die detaillierte statistische Analyse der Metabolomik und Transcriptomics Daten
- Bereiten Sie die statistische Analyse-Software.
HINWEIS: In dieser Fallstudie, eine Software in der Lage PCA auszuführen, PLS-DA und O2PLS-DA verwendet wurde.- Importieren Sie die Metabolomik und Transkriptom-Daten. Gehe zu → New Regular Projekt → Neu Regelmäßige Projekt in Datei die Datenmatrix mit MZmine Software erhalten zu importieren. Klicken Sie dann auf Bearbeiten → die gesamte Matrix → Heim Transponieren und den entsprechenden primären und sekundären ID → Fertigstellen zuweisen.
- Die mittlere Rechenzentrum und skalieren die Pareto-Skala. Gehen Sie im Hauptfenster auf Bearbeiten → M1 und ändern Sie die entsprechende Para-ters die Daten abhängig. Für die hier beschriebenen Ergebnisse, die Skala von Einheitsvarianz zu Par ändern.
- Skalieren Sie die Transkriptom-Daten des Geräts Variance-Einstellung.
- Führen Sie die multivariate statistische Analyse.
- Implementieren Sie die PCA wie in B ild 2 gezeigt. In dieser Fallstudie, PCA zeigt die wichtigsten Unterschiede zwischen den Proben, welche die unterschiedlichen Reifestadien und Vegetationszeiten.
- Im Workset Fenster wählen PCA-X als Modelltyp. Drücken Sie Autofit. Achten Sie auf die R2X (cum) und Q2 (cum) Werte, da sie eine Vorstellung über die Qualität des Modells geben.
HINWEIS: In der Regel, desto höher die Werte, desto besser das Modell, aber Modelle mit sehr hoher R2X (cum) könnten die Daten über passen. Als empirische Regel aufhören Hinzufügen wir Hauptkomponenten, wenn die Q2 (cum) Wert zu sinken beginnt. - Wählen Sie dann Noten → Scatter den Plot zu sehen, die die mögliche Gruppierung der Proben zeigt.
- Überprüfen Sie die PCAScore-Plots. Wenn ein gutes Modell erhalten (Q2cum> 0,5), wobei die gleichen Klassen von Proben verwenden, um ein O2PLS-DA-Modell (Schritt 4.2.2) zu bauen.
- Im Workset Fenster wählen PCA-X als Modelltyp. Drücken Sie Autofit. Achten Sie auf die R2X (cum) und Q2 (cum) Werte, da sie eine Vorstellung über die Qualität des Modells geben.
- Erstellen Sie zwei O2PLS-DA-Modelle, die Proben klassifiziert durch Makro-Zone mit und Validierung der Modelle eine Permutation Test mit 200 Permutationen.
- Ordnen Sie die Klassen von → Neu Als → M1 → Beobachtungen zum Hauptfenster gehen. Legen Sie die gewünschten Klassen. Ändern Sie dann Typ das Modell von PCA-X zu O2PLS-DA. Drücken Sie Autofit. Stellen Sie sicher, dass die Anzahl der Komponenten des PLS-DA-Modell ist die gleiche wie die der O2PLS-DA.
- Um die O2PLS-DA-Modell bestätigen, gehen Sie zu analysieren CV-ANOVA und finden Sie in der rechten p-Wert. Darüber hinaus finden Sie auf New Wie im Heim Fenster → M2 und das Modell Typ von O2PLS-DA zu PLS-DA ändern. Drücken Sie Autofit.
- Gehen → Permutationen zu analysieren und ausführen 200 Permutationen (de-select neu berechnen Permutationen Option).
Hinweis: Die letzte Ausgabe zeigt ein Fenster, in dem der R2-Wert should treffen im Allgemeinen die Y-Achse in Werte unter 0,4, während das Q2 den negativen Teil der Y-Achse treffen sollte. Wenn R2 und / oder Q2-Werte nicht korrekt sind, reduzieren die Anzahl der Komponenten sowohl in PLS-DA und O2PLS-DA-Modelle. - Gehen M2 in Projekt-Fenster auswählen und klicken Sie auf Noten → Scatter die Handlung und die Position der Probe Klassen zu sehen. Zu beobachten, was, Metaboliten charakterisieren eine oder mehrere bestimmte Klassen, gehen Sie auf Plot / Liste → Scatter.
- Ändern Sie den Datentyp auswählen, um Beobachtungen und Beladungen → Hinzufügen Series und ändern Sie den Artikel in der X-Achse und der Serie in pq (korr) und Pred Comp in 1 und 2 oder weitere Komponenten vorhanden, wenn auf.
- Mit der rechten Taste der Maus auf dem Grundstück gedrückt, gehen Sie auf Eigenschaft → Farbe, und wählen Sie durch Begriffe, die Plotsymbole zwischen Molekülen und Klassen zu unterscheiden. Zum Layout → Format Plot → Achse und / oder Stile, die Handlung mit den gewünschten Eigenschaften zu modifizieren.
- Basierend auf den Ergebnissen der metabolomics O2PLS-DA-Analyse, Unterschiede in den relativen Mengen spezifischer Metaboliten oder Klassen von Metaboliten als Histogramme.
- Basierend auf den Ergebnissen der Transkriptomik O2PLS-DA - Analyse, abrufen und weisen unterschiedlich modulierten Gene Gene Ontology (GO) 28 Klassements.
- Identifizieren Sie die Beziehungen zwischen Metabolitenspiegel und Genexpression manuell.
- Implementieren Sie die PCA wie in B ild 2 gezeigt. In dieser Fallstudie, PCA zeigt die wichtigsten Unterschiede zwischen den Proben, welche die unterschiedlichen Reifestadien und Vegetationszeiten.
Ergebnisse
Die Fallstudie in diesem Artikel beschrieben ergab eine endgültige Datenmatrix, die 552 Signale (m / z Merkmale) einschließlich Molekülionen sowie deren Isotope, Addukte und einige Fragmente, relativ unter 189 Proben quantifiziert (7 Weinberge x 3 Reifestufen x 3 Vegetationsperioden x 3 biologischen Replikaten). Die Gesamtzahl für Datenpunkte war daher 104.328. Fragmentation Baumanalyse führte zur Annotation von 282 m / z Merkmale, entsprechend Metaboliten sowie Ad...
Diskussion
Dieser Artikel beschreibt die Metabolomik, verwendet Transcriptomics und statistische Analyseprotokolle die Traubenbeere Terroir-Konzept zu interpretieren. Metabolomics Analyse durch HPLC-ESI-MS ist empfindlich genug, um eine große Anzahl von Metaboliten gleichzeitig zu erfassen, aber relativ Quantifizierung wird durch den Matrixeffekt und Ionenunterdrückungs / Erweiterung betroffen. Allerdings hat sich ein ähnlicher Ansatz bereits verwendet worden , um die Reifung und nach der Ernte Welken von Corvina Beeren, und di...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
This work benefited from the networking activities coordinated within the EU-funded COST ACTION FA1106 "An integrated systems approach to determine the developmental mechanisms controlling fleshy fruit quality in tomato and grapevine". This work was supported by the 'Completamento del Centro di Genomica Funzionale Vegetale' project funded by the CARIVERONA Bank Foundation and by the 'Valorizzazione dei Principali Vitigni Autoctoni Italiani e dei loro Terroir (Vigneto)' project funded by the Italian Ministry of Agricultural and Forestry Policies. SDS was financed by the Italian Ministry of University and Research FIRB RBFR13GHC5 project "The Epigenomic Plasticity of Grapevine in Genotype per Environment Interactions".
Materialien
| Name | Company | Catalog Number | Comments |
| Mill Grinder | IKA | IKA A11 basic | |
| HPLC Autosampler | Beckman Coulter | - | System Gold 508 Autosampler |
| HPLC System | Beckman Coulter | - | System Gold 127 Solvent Module HPLC |
| C18 Guard Column | Grace | - | Alltima HP C18 (7.5 mm x 2.1 mm; 5 μm) Guard Column |
| C18 Column | Grace | - | Alltima HP C18 (150 mm x 2.1 mm; 3 μm) Column |
| Mass Spectometer | Bruker Daltonics | - | Bruker Esquire 6000; The mass spectometer was equipped with an ESI source and the analyzer was an ion trap. |
| Extraction solvents and HPLC buffers | Sigma | 34966 | Methanol LC-MS grade |
| Sigma | 94318 | Formic acid LC-MS grade | |
| Sigma | 34967 | Acetonitrile LC-MS grade | |
| Sigma | 39253 | Water LC-MS grade | |
| Minisart RC 4 Syringe filters (0.2 μm) | Sartorius | 17764 | |
| Softwares for data collection (a) and processing (b) | Bruker Daltonics | - | Bruker Daltonics Esquire 5.2 Control (a); Esquire 3.2 Data Analysis and MzMine 2.2 softwares (b) |
| Spectrum Plant Total RNA kit | Sigma-Aldrich | STRN250-1KT | For total RNA extractino from grape pericarps |
| Nanodrop 1000 | Thermo Scientific | 1000 | |
| BioAnalyzer 2100 | Agilent Technologies | G2939A | |
| RNA 6000 Nano Reagents | Agilent Technologies | 5067-1511 | |
| RNA Chips | Agilent Technologies | 5067-1511 | |
| Agilent Gene Expression Wash Buffer 1 | Agilent Technologies | 5188-5325 | |
| Agilent Gene Expression Wash Buffer 2 | Agilent Technologies | 5188-5326 | |
| LowInput QuickAmp Labeling kit One-Color | Agilent Technologies | 5190-2305 | |
| Kit RNA Spike In - One-Color | Agilent Technologies | 5188-5282 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-5242 | |
| RNeasy Mini Kit (50) | Qiagen | 74104 | For cRNA Purification |
| Agilent SurePrint HD 4X44K 60-mer Microarray | Agilent Technologies | G2514F-048771 | |
| eArray | Agilent Technologies | - | https://earray.chem.agilent.com/earray/ |
| Gasket slides | Agilent Technologies | G2534-60012 | Enable Agilent SurePrint Microarray 4-array Hybridization |
| Thermostatic bath | Julabo | - | |
| Hybridization Chamber | Agilent Technologies | G2534-60001 | |
| Microarray Hybridization Oven | Agilent Technologies | G2545A | |
| Hybridization Oven Rotator Rack | Agilent Technologies | G2530-60029 | |
| Rotator Rack Conversion Rod | Agilent Technologies | G2530-60030 | |
| Staining kit | Bio-Optica | 10-2000 | Slide-staining dish and Slide rack |
| Magnetic stirrer device | AREX Heating Magnetic Stirrer | F20540163 | |
| Thermostatic Oven | Thermo Scientific | Heraeus - 6030 | |
| Agilent Microarray Scanner | Agilent Technologies | G2565CA | |
| Scanner Carousel, 48-position | Agilent Technologies | G2505-60502 | |
| Slide Holders | Agilent Technologies | G2505-60525 | |
| Feature extraction software v11.5 | Agilent Technologies | - | inside the Agilent Microarray Scanner G2565CA |
| SIMCA + V13 Software | Umetrics |
Referenzen
- Jessome, L. L., Volmer, D. A. Ion suppression: A major concern in mass spectrometry. Lc Gc N Am. 24 (5), 498-510 (2006).
- Kim, H. K., Choi, Y. H., Verpoorte, R. NMR-based plant metabolomics: where do we stand, where do we go?. Trends Biotech. 29 (6), 267-275 (2011).
- Sumner, L. W., Mendes, P., Dixon, R. A. Plant metabolomics: large-scale phytochemistry in the functional genomics era. Phytochem. 62 (6), 817-836 (2003).
- Bottcher, C., von Roepenack-Lahaye, E., Willscher, E., Scheel, D., Clemens, S. Evaluation of matrix effects in metabolite profiling based on capillary liquid chromatography electrospray ionization quadrupole time-of-flight mass spectrometry. Anal Chem. 79 (4), 1507-1513 (2007).
- Toffali, K., et al. Novel aspects of grape berry ripening and post-harvest withering revealed by untargeted LC-ESI-MS metabolomics analysis. Metabolomics. 7 (3), 424-436 (2011).
- Martin, J. C., et al. Can we trust untargeted metabolomics? Results of the metabo-ring initiative, a large-scale, multi-instrument inter-laboratory study. Metabolomics. 11 (4), 807-821 (2015).
- Jaillon, O., et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 449 (7161), 463-467 (2007).
- Velasco, R., et al. A high quality draft consensus sequence of the genome of a heterozygous grapevine variety. Plos One. 2 (12), (2007).
- Tornielli, G. B., Zamboni, A., Zenoni, S., Delledonne, M., Pezzotti, M., Gerós, H., Chaves, M., Delrot, S. Ch. 11. The Biochemestry of the Grape Berry. 11, (2012).
- Anesi, A., et al. Towards a scientific interpretation of the terroir concept: plasticity of the grape berry metabolome. BMC Plant Biol. 15, 1-17 (2015).
- Berdeja, M., et al. Water limitation and rootstock genotype interact to alter grape berry metabolism through transcriptome reprogramming. Hort Res. 2, 1-13 (2015).
- Carbonell-Bejerano, P., et al. Solar ultraviolet radiation is necessary to enhance grapevine fruit ripening transcriptional and phenolic responses. BMC Plant Biol. 14, 1-16 (2014).
- Carbonell-Bejerano, P., et al. Reducing sampling bias in molecular studies of grapevine fruit ripening: transcriptomic assessment of the density sorting method. Theor Exp Plant Phys. 28 (1), 109-129 (2016).
- Carbonell-Bejerano, P., et al. Circadian oscillatory transcriptional programs in grapevine ripening fruits. BMC Plant Biol. 14, 1-15 (2014).
- Cavallini, E., et al. Functional diversification of grapevine MYB5a and MYB5b in the control of flavonoid biosynthesis in a petunia anthocyanin regulatory mutant. Plant & Cell Physiol. 55 (3), 517-534 (2014).
- Cramer, G. R., et al. Transcriptomic analysis of the late stages of grapevine (Vitis vinifera cv. Cabernet Sauvignon) berry ripening reveals significant induction of ethylene signaling and flavor pathways in the skin. BMC Plant Biol. 14, 1-21 (2014).
- Dal Santo, S., et al. The plasticity of the grapevine berry transcriptome. Genome Biol. 14 (6), 1-17 (2013).
- Fasoli, M., et al. The Grapevine Expression Atlas Reveals a Deep Transcriptome Shift Driving the Entire Plant into a Maturation Program. Plant Cell. 24 (9), 3489-3505 (2012).
- Gambino, G., et al. Co-evolution between Grapevine rupestris stem pitting-associated virus and Vitis vinifera L. leads to decreased defence responses and increased transcription of genes related to photosynthesis. J Exp Bot. 63 (16), 5919-5933 (2012).
- Ghan, R., et al. Five omic technologies are concordant in differentiating the biochemical characteristics of the berries of five grapevine (Vitis vinifera L.) cultivars. BMC Genomics. 16 (1), 1-26 (2015).
- Pastore, C., et al. Selective defoliation affects plant growth, fruit transcriptional ripening program and flavonoid metabolism in grapevine. BMC Plant Biol. 13, 1-13 (2013).
- Pastore, C., et al. Increasing the source/sink ratio in Vitis vinifera (cv Sangiovese) induces extensive transcriptome reprogramming and modifies berry ripening. BMC Genomics. 12, 1-23 (2011).
- Rinaldo, A. R., et al. A Grapevine Anthocyanin Acyltransferase, Transcriptionally Regulated by VvMYBA, Can Produce Most Acylated Anthocyanins Present in Grape Skins. Plant Physiol. 169 (3), 1897-1916 (2015).
- Royo, C., et al. Developmental, transcriptome, and genetic alterations associated with parthenocarpy in the grapevine seedless somatic variant Corinto bianco. J Exp Bot. , 259-273 (2015).
- Venturini, L., et al. De novo transcriptome characterization of Vitis vinifera cv. Corvina unveils varietal diversity. BMC Genomics. 14, 1-13 (2013).
- Commisso, M., Strazzer, P., Toffali, K., Stocchero, M., Guzzo, F. Untargeted metabolomics: an emerging approach to determine the composition of herbal products. Comput Struct Biotechnol J. 4, 1-7 (2013).
- Pluskal, T., Castillo, S., Villar-Briones, A., Oresic, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 1-11 (2010).
- Ashburner, M., et al. Gene Ontology: tool for the unification of biology. Nat Genet. 25 (1), 25-29 (2000).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten