JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
포도 베리 대사 체학 및 Transcriptomics을 통해 해석 떼루아 개념
* 이 저자들은 동등하게 기여했습니다
요약
이 문서는 즉 떼루아 개념에 대한 통찰력을 얻기 위해 포도 베리 성적 증명서 및 대사에 타겟이 불분명 한 대사 체학, transcriptomics 및 다변량 통계 분석의 응용 프로그램을 설명 베리 품질 특성에 대한 환경의 영향.
초록
떼루아 특히 서식지 및 관리 방법에 따라 같은 포도 (유럽 종 포도)로 작물의 특성에 영향을 미치는 환경 요인의 조합을 의미합니다. 이 문서에서는 특정 떼루아 서명이 다변량 통계 분석을 사용하여 포도의 품종, 조기의 베리 대사 체 및 사체에서 검출 할 수있는 방법을 보여줍니다. 이 방법은 먼저 적절한 샘플링 계획이 필요합니다. 이 사례에서, 조기 품종의 특정 클론 유전 적 차이를 최소화하도록 선택되고, 샘플은 세 가지 성장 계절 동안 세 가지 매크로 영역을 나타내는 일곱 포도에서 수집 하였다. 타겟팅되지 않은 LC-MS의 대사 체학 접근 MZmine 소프트웨어 및 단편화 트리 분석을 기초 대사 식별 전략을 사용하여 효율적으로 데이터 처리를 수반 높은 민감도 때문에 권장된다. 종합 사체 분석은 마이크로 어레이를 사용하여 달성 될 수있다함유 프로브 다른 TERROIRS의 컨텍스트에서 모든 차등 유전자 발현의 동시 분석을 가능 ~ 모든 예측 리세 유전자의 99 %를 커버. 마지막으로, 투영 방법에 기초하여 다변량 데이터 분석은 대사 체학 및 transcriptomics 데이터 통합 정보의 상관 관계를 식별하기 위해 상세하게 분석 될 수 있도록 강력한 빈티지 특정 효과를 극복하기 위해 사용될 수있다.
서문
식물 게놈 전 사체, 프로테옴 및 metabolomes에 기초하여 대규모 데이터 분석은 포도 식물과 그 환경 사이의 상호 작용을 반영하는 와인 떼루아 특성과 같은 복잡한 시스템의 작동에 대한 새로운 통찰력을 제공한다. 동일한 포도 나무의 클론이 다른 포도원에서 재배하는 경우에도 와인의 떼루아 구별 할 수 있기 때문에 클론 게놈이 동일하기 때문에, 게놈 분석은 거의 사용이다. 대신 유전자 발현 와인의 품질 특성을 결정하는 열매의 대사 특성 간의 상관 관계를 볼 필요가있다. 마이크로 어레이에 고정 된 프로브와 같은 하이브리드와 같은 보편적 인 특성을 이용하여 정량 분석을 용이하게 모든 성적 증명서의 유사한 화학적 성질,의 사체 혜택의 수준에서 유전자 발현의 분석. 프로테오믹스 A의 대조적으로, 보편적 인 분석 방법차 대사 체학 때문에 개별 단백질 및 대사 산물의 거대한 물리적, 화학적 다양성을 더 도전입니다. 대사 체학의 경우 개별 대사 산물의 크기, 극성, 풍부하고 변동성에 크게 다르기 때문에 이러한 다양성은 훨씬 더 극단적이다, 그래서 하나의 추출 과정이나 분석 방법 론적 접근 방식을 제공합니다.
비 휘발성 대사 적합한 분석 플랫폼 중, 고성능 액체 크로마토 그래피에 기초하여 그 질량 분석계에 연결된 (HPLC-MS)는 훨씬 더 민감 자외선 또는 다이오드 배열 검출기를 가진 HPLC (HPLC-UV, HPLC-DAD 같은 대안보다 ) 또는 HPLC-MS에 의해 핵 자기 공명 (NMR) 분광법 있지만 정량 분석은 이러한 매트릭스 효과 이온 진압 / 인핸스 1-3 현상에 의해 영향을받을 수있다. (HPLC-E를 전기 분무 이온화 소스를 사용하여 HPLC-MS에 의한 조기는 포도 열매의 분석시 이러한 영향의 조사SI-MS)은 가장 낮은 체류 시간과 당 및 다른 분자 강하게 아마도이 영역에서 분자의 다수를 반영 과소 것으로 나타났다 및 다른 분자의 존재 량 매트릭스 효과에 의해 과대 또는받지 과소 평가 될 수 있다는 하지만, 매트릭스 효과에 대한 데이터 정규화는 전체 결과 4,5에 제한된 영향을 미칠 것 같았다. 본원에 기재된 방법은 숙성 중에 포도 열매에서 높은 수준으로 축적 매체 극성 대사 산물의 분석을 위해 최적화되고, 이는 상당히 떼루아의 영향. 그들은 함께 색상, 맛과 와인의 건강 관련 특성을 결정 안토시아닌, 플라 보놀, flavan-3-OLS, 프로시아니딘, 다른 플라보노이드, 레스베라트롤, 스틸 벤, 히드 록시 산, 하이드 록시 산을 포함한다. HPLC-MS에 의한 정량은 신뢰할 수 있기 때문에 같은 당 및 지방족 유기산과 같은 다른 대사는, 때문에 effec 행렬에 무시됩니다t 이온 억제 현상 (5). 이 방법에 의해 선택된 극성의 범위 내에서 접근 가능 6만큼 다른 대사 물질을 검출하는 것을 목적으로 불특정이다.
포도 나무의 성적 증명서의 수천을 동시에 모니터링 할 수 Transcriptomics 방법은 전체 포도 게놈 서열 7,8의 가용성에 의해 촉진된다. 높은 처리량의 cDNA 서열에 기초하여 조기 transcriptomics 방법 일괄 RNA-SEQ 기술로 설명한 절차의 집합으로 차세대 서열의 출현으로 발전해왔다. 이 방법은 빠르게 transcriptomics 연구에 대한 선택의 방법이되고있다. 그러나, 성적 증명서의 수천 하이브리드에 의해 병렬로 정량화 할 수 있도록 마이크로 어레이를 기반으로 문학의 큰 몸은 포도 나무에 대한 축적하고있다. RNA-서열이 주류 기술이되기 전에 실제로, 많은 전용 상용 마이크로 어레이 플랫폼은 있었다개발 허용 포도 나무의 사체는 훌륭한 세부 사항에 검사합니다. 플랫폼의 광대 한 다양한 가운데, 두 개의 게놈 전체의 전 사체 분석 (9)을 허용했다. 가장 진화 배열 따라서 각 실험의 비용을 절감하는 하나의 장치에서 최대 12 개의 독립적 인 샘플의 혼성화시켰다. 12 서브 어레이는 각각 29,549 포도 나무의 성적을 나타내는 135,000 60 머 프로브를 구성. 이 장치는 시험 10-24의 다수에 사용되고있다. 이 두 플랫폼은 이제 중단되었지만 새 사용자 정의 마이크로 어레이는 최근 설계되고있어 추가 새로 발견 된 포도 나무의 유전자 (25)를 나타내는 프로브의 더 큰 수를 포함로 최근 개발을 나타냅니다.
transcriptomics와 대사 체학 분석에 의해 생성 된 대형 판매 데이터 세트는 다른 형태 사이의 상관 관계를 결정하기 위해 다변량 기법을 포함한 데이터 분석에 적합한 통계 방법을 필요로데이터의. 가장 널리 사용되는 변수 기술들은 투영에 기초하여 해당하며, 여기에는 주성분 분석 (PCA), 또는, 자율 될 수있는 잠재적 인 구조 판별 분석 (O2PLS-DA) (26)에 양방향 정사영 같은 감독. 이 문서에 제시된 프로토콜은 샘플 그룹 간의 차이를 식별하기 위해 탐색 적 데이터 분석 및 O2PLS-DA에 대한 PCA를 사용합니다.
프로토콜
1. 적절한 재료를 선택하고 샘플링 계획 구축
- 적절한 샘플링 계획을 개발하여 실험을 시작합니다. 그래서 경우에 따라 각각의 계획을 평가하고 더 일반적이고 보편적 인 방법은 없습니다. 샘플링 계획은 샘플링 장소, 시간 및 정확한 샘플링 절차를 명시 있는지 확인합니다. 이 사례 연구에 사용 된 표본 추출 계획에 대한 그림 1을 참조하십시오.
참고 :이 사례 연구, 단일 클론 (. 티스 VINIFERA의 이력서, 조기, 복제 48)에서 포도 열매가 베로나의 지방에서 세 가지 매크로 영역 (가르다 호수, Valpolicella 및 소아베)에 칠 상업 포도원에서 수집 하였다. 각 포도원의 주요 기능은 표 1에 요약되어있다. 열매는 veraison (숙성의 시작), 중간은-숙성과 잘 익은 열매에 해당하는 세 가지 시점에서 세 가지 성장 계절 (2006, 2007 및 2008) 동안 수집 하였다.- 가입 나 각 (차량 등록에 대한eyard / 년 / 숙성 단계), 무작위 높이와 공장에 위치 두 개의 포도 나무 행을 따라 다른 위치에서 수확 30 클러스터.
- 눈에 띄는 손상 및 / 또는 감염의 징후와 사람들을 피하고, 각 클러스터에서 무작위로 세 장을 선택합니다.
- 반복은 3 개의 독립적 인 풀을 얻기 위해 1.1.1와 1.1.2 단계를 반복합니다.
- 열매를 Deseed 및 액체 질소에 즉시 과피를 동결.
- 자동 밀 분쇄기와 각 풀에서 10 냉동 된 딸기 크러시, 2 개의 동일한 부품, transcriptomics 분석을위한 하나 대사 체학 분석을위한 일에 각 분말 샘플을 나눕니다.
- -80 ° C에서 분말을 저장합니다.
| 오전 | BA | BM | CS | 파 | MN | 오후 | |
| 매크로 영역 | 소아베 | 가르다 호수 | Valpolicella | 가르다 호수 | Valpolicella | Valpolicella | 소아베 |
| 높이 (m) | (250) | (120) | (450) | (100) | (130) | (250) | (130) |
| 뿌리 줄기 | (41B) | S04 | K5BB | 420A | 420A | K5BB | (41B) |
| 행 방향 | EW | NS | EW | EW | EW | NS | NS |
| 교육 시스템 | 오버 헤드 시스템 (페르 골라) | 오버 헤드 시스템 (페르 골라) | 수직 촬영 위치 결정 (기요) | 오버 헤드 시스템 (페르 골라) | Overhe광고 시스템 (페르 골라) | 수직 촬영 위치 결정 (기요) | 수직 촬영 위치 결정 (기요) |
| 토양 유형 | 미사 점토 | 옥토 | 점토 | 옥토 | 클레이 옥토 | 미사 질 양토 | 식양토 |
| 심기 레이아웃 (m) | 3.20 X 1.00 | 4.50 X 0.80 | 4.00 X 1.25 | 3.50 X 1.20 | 3.50 X 0.75 | 2.80 X 1.00 | 1.80 X 0.80 |
| 총 라임 % | 3.9 | 19.3 | 18.3 | 14.4 | (31) | 5.9 | 27.9 |
| 활성 라임 % | 0.5 | 2.6 | 9.4 | 6.3 | 11.3 | 3.1 | 8.3 |
| 모래 % | (15) | 47 | (66) | (42) | (29) | (13) | (36) |
| 양토의 % | (43) | (36) | (21) | (37) | (39) | 67 | (36) |
| 클레이 % | (42) | (17) | (13) | (21) | (32) | (20) | (28) |
| 토양의 pH | 8.3 | 7.9 | 7.8 | 8.2 | 8.2 | 7.8 | 7.9 |
| 유기물 (%) | 2.9 | 2.5 | 2.2 | 1.2 | 2.9 | 1.6 | 2.5 |
| 교환 인 (㎎ / ㎏) | (26) | 73 | 73 | (68) | (48) | 47 | (64) |
| 교환 칼륨 (㎎ / ㎏) | (190) | 376 | (620) | (230) | (168) | (154) | (126) |
| 교환 마그네슘 (㎎ / ㎏) | (272) | 468 | 848 | (623) | 294 | 293 | 183 |
| 교환 칼슘 (㎎ / ㎏) | 6500 | 5,380 | 7,358 | 6,346 | 4,652 | 10055 | 2,878 |
| 베리 환원당 2006 | 211.25 ± 1.20 | 176.20 ± 0.42 | 187.40 ± 0.00 | 203.70 ± 1.13 | 212.55 ± 0.64 | 195.20 ± 0.00 | 211.65 ± 0.64 |
| 베리 환원당 2007 | 190.00 ± 1.27 | 165.25 ± 0.49 | 153.00 ± 0.42 | 203.60 ± 0.71 | 210.90 ± 0.71 | 192.25 ± 0.64 | (18)8.70 ± 1.84 |
| 베리 환원당 2008 | 191.35 ± 0.64 | 178.90 ± 0.57 | 170.05 ± 0.49 | 205.15 ± 1.48 | 188.70 ± 0.57 | 169.35 ± 0.49 | 108.05 ± 1.06 |
| 베리의 pH 2006 | 3.01 ± 0.01 | 2.96 ± 0.01 | 2.84 ± 0.00 | 2.9 ± 0.00 | 2.98 ± 0.00 | 3.02 ± 0.00 | 3.06 ± 0.01 |
| 베리의 pH 2007 | 2.97 ± 0.00 | 3.00 ± 0.00 | 2.74 ± 0.00 | 3.07 ± 0.01 | 2.98 ± 0.00 | 2.87 ± 0.01 | 3.09 ± 0.00 |
| 베리의 pH 2008 | 2.83 ± 0.00 | 3.04 ± 0.01 | 2.71 ± 0.00 | 2.98 ± 0.01 | 2.98 ± 0.00 | 2.82 ± 0.00 | 3.11 ± 0.00 |
| 수확 날짜 | 2,006 | 2,007 | 2,008 | ||||
| Veraison | 8 8 월 | 18 7 월 | 12 8 월 | ||||
| 중간 숙성 | 4 ~ 9 월 | 8 8 월 | 2 9 월 | ||||
| 익은 | 18 9 월 | 29 8 월 | 23 9 월 |
표 1 : 각 포도원의 주요 기능과샘플 수집 날짜. m = m, EW = NS = 북한 남서부을 먹는다. 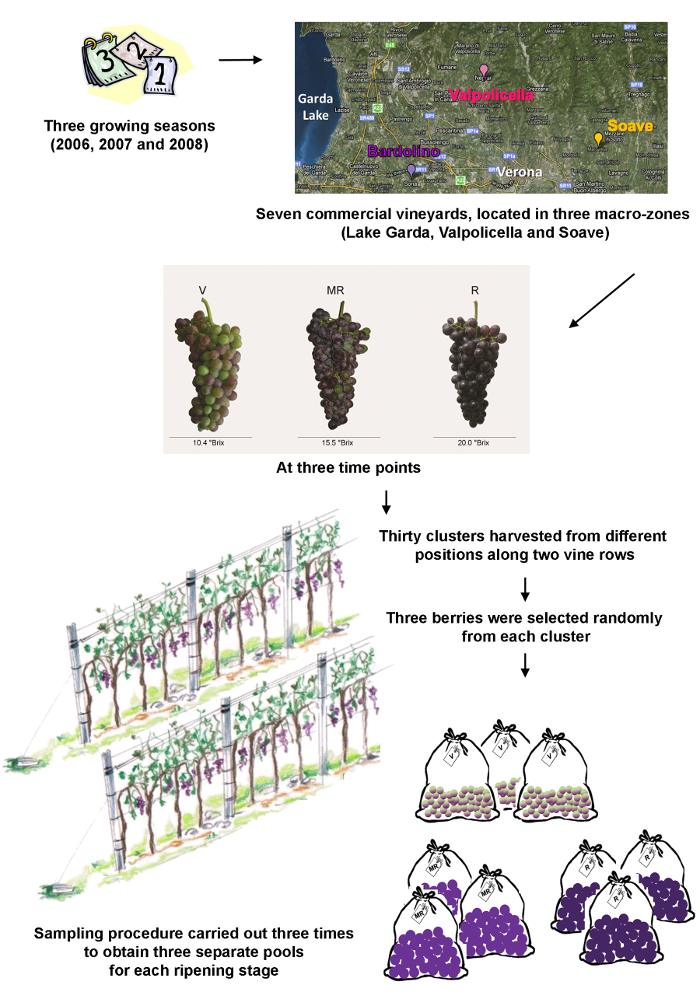
그림 1 :. 샘플링 절차의 도식 표현은 세 가지 와인 생산 매크로 지역은 베로나, 베네토 지역, 이탈리아의 도시 주변에 위치하고 있습니다. 세 시점은 포도 재배에 숙성의 발병을 나타내는 veraison (V)이며, 중간 숙성 (MR)과 잘 익은 열매 (R). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
2. 베리 분말 추출물을 준비, 대사 산물을 분석하고 데이터를 처리
- 분석의 베리 분말 샘플을 준비합니다.
- 세 권 실온에서 베리 샘플의 대사 추출물을 준비한다 (w / v)의 0.1 %로 산성화 메탄올 (v / v)의 대15 분에서 40 kHz의 초음파 조에서 MIC 산. LC-MS-등급의 물과 포름산 및 HPLC 급 메탄올을 사용합니다.
- 4 ℃에서 10 분 동안 16,000 XG에서 추출물을 원심 분리기.
- 탈 이온수 두 개의 볼륨 (v / v)의 단계 2.1.2에서 상층 액을 희석하고 0.2 μm의 필터를 통해 전달합니다.
- HPLC-ESI-MS에 의해 베리 추출물을 분석합니다.
- 공급 업체의 권장 사항에 따라 HPLC-ESI-MS 시스템을 설정합니다.
주 :이 사례에서, 설정은 이온 트랩 질량 분석기 및 전기 분무 이온화 소스 - 라인에 접속 된 오토 샘플러를 구비 한 HPLC 시스템을 구성. - C18 가드 컬럼 (7.5 X 2.1 mm 2) 역상 C18 컬럼 (150 X 2.1 mm 2, 입자 크기 3 μm의)와 HPLC 시스템을 연결합니다.
- 이동상으로 사용되는 용매를 준비한다. S와 같은 용매 A에서 물과 100 % 아세토 니트릴 5 % (v / v) 아세토 니트릴 0.5 % (v / v)의 포름산을 사용하여olvent B. 사용 LC-MS-등급 아세토 니트릴, 물과 포름산.
- 0.2 ㎖의 30 분 -1 μL의 샘플 주입 부피를 이용하여 일정한 유량으로 용매 A 및 B의 선형 구배로 HPLC 분리를 실행.
- 초기 샘플 주입, 25 % 용매 B 20 %에서, 20 분에 20 % 용매 B 10 %에서 5 분, 10 % 용매 B 0 %에서 구배를 확립 후 100 % 용매 A.으로 열 평형 15 분에서 5 분, 25 % 내지 70 % 용매 B.
- 중복 된 각 샘플을 분석 할 수 있습니다. 기기 중심의 효과를 방지하기 위해 시료 분석을 랜덤. 각각의 분석 사이에 100 % 용매 A의 재 평형 20 분을 허용합니다.
- 대체 부정과 긍정적 인 이온화 모드에서 질량 스펙트럼을 획득. 이 사례 연구에 설명 된 결과를 표 2에서와 같이 매개 변수를 설정합니다. 정확한 시스템 설정은 특정 플랫폼에 따라 달라집니다.
참고 : 다른 방법으로, 다른 한 벌을 사용특정 플랫폼에 따라 수있는 방법.- 모든 경우에, 임의의 분리 플랫폼과 같은 체류 시간 재현성을 위해 적절한 재 평형화 시간을 사용하여주십시오. 샘플 수 (열 샘플 이상)이 높은 경우에는, 각 배치 사이에서의 크로마토 그래피 컬럼 세정 프로그램 (두 용출 용매 사이 천천히 구배)으로 시료 9-10 일괄들을 분석한다. 충분한 체류 시간 재현성이 각 배치의 첫 크로마토 그래피 분석 빈 분석 될 수 있도록 (즉, 용매의 분석).
- 또한 조각 옵션 (두 이온 전구체, MS 3)을 설정 조각난 부정과 긍정적 인 이온 모드에서 질량 스펙트럼을 취득. 제조업체의 지침에 따라 분할 트리 분석 (MS / MS 및 MS 3)에 의해 대사 주석.
주 : 그들은 종종 당화되기 때문 THER 식물 대사에 특히 적합는 efore 첫 번째 조각 (MS / MS)가 무료 아글 리콘 이온, 이후 분열 (MS 3)를 떠나 당 부분을 제거하는 경향은 정통 표준 라이브러리에 대해 아글 리콘을 식별하는 데 도움이됩니다. - 또한 1 V.의 분열 진폭으로 m / z 범위 50-1,500에서 MS / MS 및 MS (3) 스펙트럼을 획득, 특정 플랫폼에 따라 다른 적절한 방법을 사용합니다.
- 각 m / z 신호, 제조업체의 지시에 따라 상기 인증 된 표준 라이브러리에 MS / MS 및 MS (3) 분할 패턴 및 체류 시간을 비교한다.
참고 : 플랫폼의 많은 유형은 정통 표준의 분석을 통해 라이브러리의이 종류를 구축하는 소프트웨어 기능을 포함한다. 이것은 많은 신호를 식별한다 아니지만 신호 주석한다. - 나머지 익명 신호를 들면, 문헌 공학자 출판 그와 MS / MS 및 MS (3) 단편화 패턴을 비교할m / z 값 (즉, 숫자 "m / z 353"은 m / z를 언급과 분자하는 출판물을 찾기 위해) 자유롭게 사용할 수있는 일반적인 검색 엔진의와 함께, 또는 MassBank 온라인 데이터베이스를 사용 (WWW에 대한 힝 .massbank.jp / EN / database.html)과 인간의 대사 체 데이터베이스 (www.hmdb.ca/search/spectra?type=ms_search).
- 공급 업체의 권장 사항에 따라 HPLC-ESI-MS 시스템을 설정합니다.
| 질량 분석기 구성 요소 | 기능 | 매개 변수 |
| 전기 분무 이온화 소스 | Nebulizing 가스 | 50 psi의 350 ° C |
| 건조 가스 | 10 L 분 -1 | |
| 이온 트랩 검출기 | 주사 | 초당 전체 스캔 모드, 13,000m / Z, 범위 50-1,500 m / z |
| 대상 질량 | 400m / Z | |
| 충돌 가스 | 헬륨 | |
| 진공 압력 | 1.4 × 10 -5 mbar에서 | |
| 모세관 소스 | 4000 V | |
| 단판 오프셋 | -500 V | |
| 대충 훑어 보는 사람 | -40 V | |
| 캡 종료 | -121 V | |
| 10월 1일 DC | -12 V | |
| 10월 2일 DC | -1.7 V | |
| 렌즈 1 | +5 V | |
| 렌즈 2 | +60 V | |
| 양성 이온화 모드 ICC | 20.000 | |
| 이온화 네가티브 모드 ICC | 7,000 |
표 2 : 질량 스펙트럼을 획득하기위한 주요 매개 변수를 설정합니다.
| 조작 | 선택 | 기능 | 매개 변수 | 값 |
| 피크 검출 | 대량 검출 | 중심 | 소음 수준 | 3,500 |
| 크로마토 그램 빌더 | 최고 데이터 포인트 | 최소 시간 범위 | 0.15 | |
| 최소 높이 | 4,000 | |||
| m / z 허용 | 0.3 | |||
| 피크 검출 | 피크 디콘 볼 루션 | 지역 최소 검색 | 크로마토 그래피 임계 값 | (70) |
| RT 범위에서 검색 최소 (분) | 0시 50분 | |||
| 최소 상대 높이 | 15 % | |||
| 최소 절대 높이 | 4,000 | |||
| 피크 톱 / 날의 최소 비율 | 이 | |||
| 시간 범위 (분) | 0-10 | |||
| 동위 원소 | 동위 원소 봉우리 그룹화 | - | m / z 허용 | 1.2 |
| RT 허용 | 0시 50분 | |||
| 단조 모양 | 아니 | |||
| 최대 충전 | 삼 | |||
| 대표 동위 원소 | 아니 | |||
| 조정 | 정렬 가입 | - | m / z 허용 | 1.2 |
| m / z에 대한 무게 | (10) | |||
| 체류 시간 허용 | 0시 50분 | |||
| RT에 대한 무게 | (5) | |||
| 동일한 충전 상태를 필요 | 아니 | |||
| 동일한 ID를 필요 | 아니 | |||
| 동위 원소 패턴 비교 | 아니 | |||
| 갭 충전 | 피크 찾기 | - | 강도 허용 | 20 % |
| m / z 허용 | 0.9 | |||
| 체류 시간 허용 | 0시 40분 | |||
| RT 보정 | 아니 | |||
| 필터링 | 피크 필터를 중복 | - | m / z 허용 | 1.2 |
| RT 허용 | 0시 반 | |||
| 동일한 식별 필요 | 아니 |
표 3 : 특정 값을 Mzmine 워크 플로우는 부정적인 LC-MS 포도 베리 데이터 파일을 처리합니다.
- 액정-MS 데이터를 처리합니다.
- 액세스 또는많은 원시 크로마토로부터 관련 정보를 추출하고 각각의 검출 된 대사 물질은 각각의 샘플에서 정량화 된 데이터 행렬을 구축 할 수있는 데이터 처리 소프트웨어 패키지를 다운로드.
참고 : 프로토콜 단계는 아래의 오픈 소스 소프트웨어 MZmine의 v2.14 (http://mzmine.sourceforge.net)에 맞게 조정됩니다. 이 LC-MS 데이터 주요 초점, 질량 스펙트럼 데이터 처리를위한 오픈 소스 소프트웨어이다. 27 - 장비 제조자에 의해 제공된 소프트웨어를 사용 netCDF의 형식으로 LC-MS 크로마토 그램 데이터를 변환. 여기에 설명 된 결과를 들어, 브루 커 달 토닉 에스콰이어 V5.2 및 데이터 분석 V3.2의 소프트웨어를 사용하고 제조업체의 지침에 따라 단계를 수행합니다.
참고 : 다른 컨버터를 사용할 수있다 (다양한 무료웨어 컨버터를 사용할 수 있습니다). - 소프트웨어에 .cdf 파일을 가져옵니다.
- paramet와 피크 검출, 정렬, 갭 충진 피크 필터링 절차를 구현ERS는 표 3에보고 하였다.
- 첫 번째 단계로, 다음 시각화 → TIC / XIC 비주얼로 이동 가져온 .cdf 파일을 선택합니다. 크로마토 그램에서 가장 작은 피크의베이스에 커서를 놓고베이스 이온의 최소 강도 신호에 대한 메모를 가져 가라. 그리고 피크 검출 → 원시 데이터 방법으로 이동합니다.
- 질량 감지를 선택하고 각 스캔에 대한 개별 이온을 감지하고 이온 목록을 만들 수있는 신호 레벨을 입력합니다.
주 : 앞서 질량 감지를 수행하는 알고리즘 점검 -은 질량 분석에 의존한다. 우리의 경우, 우리는 무게 중심을 선택합니다. - 크로마토 그램 Builder를 선택하고 이온 목록에서 데이터 포인트를 연결하고 각각의 질량 값에 대한 크로마토 그램을 구축하기 위해 신호 레벨로 입력합니다. 최소 시간 범위 및 m / Z 허용 오차를 조정하는 질량 분석기의 사용 설명서에 지정된 매개 변수를 참조하십시오.
- 다음으로, 피크 검출 → 크로마토 그램 Deconvo → 피크 목록 방법으로 이동lution. 각 피크로 각각의 크로마토 그램의 분리를 할 수 있도록 (이 경우 지역 최소 검색) 적절한 알고리즘을 선택합니다.
- 수동으로 다음 매개 변수에 대한 적절한 값을 알 수 있습니다 크로마토 그램을 검사합니다. 노이즈를 제거하고 두 피크를 구별하는 최소 지역 주민의 존재를 식별하기 위해 2 (분)에 RT 범위에서 최소 검색 30 %로 크로마토 그래피 임계 값을 설정합니다.
- 2.0에서 최소 상대 높이를 설정합니다. 수동 배경 피크에 대응하지 신호의 최소 절대 높이를 식별하기 위해 크로마토 그램을 검사; (예를 들어, 제시된 실험 10000)의 절대 높이로이 값을 설정한다.
- 상부 및 피크의 최하위 데이터 포인트 사이의 최소 강도 비율을 서술하기 위해 1.1 피크 톱 / 에지의 최소 비율을 설정 진정한 피크로서 인식한다.
- 수동으로 다양한 피크의 최소 지속 기간은 사용 chromat에있는 볼 수있는 크로마토 그램을 검사ographic 조건 (예를 들면, 0.2 내지 2 분간, 화합물에 따라 제시된 실험 사이), 그 결과 지속 기간과 같은 허용되는 최대 길이의 범위를 설정하는 피크 시간 범위 (분)으로서 다음 값을 사용한다.
- 그리고 일반적으로 가장 강렬한에서, 하나의 피크에 그룹에 피크 목록 방법 → 동위 원소 → 동위 원소 봉우리 그룹화에 동위 원소 이동합니다. 주 : 파라미터는 질량 분석계의 해상도 및 체류 시간의 재현성에 좌우된다.
- 마지막으로, 경기 점수를 사용하여 자신의 m / z 및 유지 시간에 따라 피크를 정렬하는 피크 목록 방법 → 정렬 → 가입 얼 라이너로 이동합니다.
- 피크를 정렬 한 후, → 피크 Finder를 작성 피크 목록 방법 → 갭을 클릭하여 모든 데이터 간격을 입력합니다. 마지막으로, 필터는 피크 목록 방법 → 여과 → 피크 필터를 중복 클릭하여 데이터 포인트를 중복.
- .cs로 생성 된 데이터 세트를 내 보냅니다V 파일입니다.
- 수동으로 변경.가. TXT 확장자를 csv로 확장합니다. 어떤 파일 확장자를 볼 수없는 경우, 파일 확장자를 나타 내기 위해 컴퓨터 설정을 변경합니다. 탐색기 옵션 →보기 → 고급 설정 파일과 상자 '알려진 파일 형식의 파일 확장명 숨기기'를 체크 해제로 이동합니다.
- 스프레드 시트로이 .txt 파일을 가져옵니다. 이 식별 번호, m / z 값과 유지 시간이 인식하는 모든 검출 된 대사 산물, 이들의 피크 면적 값의 관점에서 모든 샘플에서 정량하는 데이터 매트릭스를 생성한다.
- 액세스 또는많은 원시 크로마토로부터 관련 정보를 추출하고 각각의 검출 된 대사 물질은 각각의 샘플에서 정량화 된 데이터 행렬을 구축 할 수있는 데이터 처리 소프트웨어 패키지를 다운로드.
3. 사체 (Transcriptome) 분석을위한 베리 분말 추출물을 준비하고 데이터를 처리
- 베리 샘플로부터 전체 RNA를 추출하고 RNA 품질을 결정합니다.
- 베리 샘플에서 총 RNA를 추출합니다.
참고 :이 사례 연구, 독점 키트와 그 간 분자의 완전한 제거를 보장하는 수정 과정에서이러한 폴리 사카 라이드 및 폴리 페놀 등의 RNA 분석과 FERE가 사용되었다. 지침은 다음이 키트 18 다릅니다.- 각 샘플 베리 분말 400 mg의 무게를 두 부분으로 나누어 두 가지의 microfuge 튜브에 200 mg을 각각 배치합니다.
- 적어도 30 초 동안 즉시 적극적으로 분말과 소용돌이 각각 200 mg의 (키트에 제공) β-머 캅토 에탄올을 함유하는 분해 용액 900 μl를 추가합니다. 5 분간 800 rpm으로 흔들면서 56 ℃로 샘플을 가열한다.
- 세포 파편 펠렛 10 분 동안 벤치 탑의 microfuge에서 최대 속도로 샘플을 원심 분리기.
- 피펫 2 ml의 포집 관에 장착 키트 (파란색 고정 링)에서 제공하는 여과 컬럼에 뜨는 700 μL. 잔여 이물질을 제거하기 위해 1 분 동안 벤치 탑의 microfuge에서 최대 속도로 모자와 원심 분리기를 닫습니다. 회 삼t 결과 동일한 여과 칼럼 그러나 새로운 포집 관을 사용하여이 단계를 반복ubes 정화 된 해물을 각각 함유 ~ 700 μL.
- 피펫 (750) 명확히 해물의 각 튜브에 (키트에 제공) 결합 솔루션의 μL 아래로 5 배 이상을 피펫 팅에 의해 즉시 섞는다. 2 ml의 포집 관에 장착 키트 (빨간색 고정 링)에서 제공하는 바인딩 열이 혼합물 700 μl를 전송합니다. RNA의 결합 1 분 동안 벤치 탑의 microfuge에서 최대 속도로 모자와 원심 분리기를 닫습니다.
- , 플로우를 통해 부분을 가만히 따르다 포집 관을 반전 잔류 액체를 배출 흡수 종이의 깨끗한 패드에 간단히를 누릅니다.
- 수집 튜브에 열을 반환하고 동일한 컬럼에 남아있는 혼합물을 피펫과 원심 분리 및 경사 단계를 반복합니다. 전체 혼합물 같은 붉은 바인딩 컬럼에 여과 될 때까지 반복합니다.
- 이제 키트의 나머지 지침을 따르과 (키트에 제공) 50 μL 용출 버퍼에 RNA 및 저장 난을 용출품질 관리 단계를위한 준비 -80 ° C에서 t.
- 분광 광도계를 사용하여 RNA의 양과 순도를 결정합니다. 단백질 (A 280 분의 260)과 폴리 페놀 / 폴리 사카 라이드 (A 230분의 260)와 오염의 정도를 나타 흡광도 비율을 기록합니다.
주 : 마이크로 어레이 혼성화에 적합한 RNA 모두 비율 적어도 1.8 득점해야합니다. - RNA의 무결성을 확인합니다.
참고 : 다양한 시스템을 사용할 수 있습니다. 형광 염료와 조합하여 모세관 전기 영동 실행을 수행하는 디지털 취득자이 경우의 연구에 사용 하였다. 마이크로 어레이 혼성화에 적합한 RNA는 8의 RNA 무결성 번호 (RIN)을 가져야한다.
- 베리 샘플에서 총 RNA를 추출합니다.
- 샘플을 준비하고 사용자 정의 마이크로 어레이에 RNA를 하이브리드.
- 탈의 RNase없는 물과 단계 3.1.1.8에서 얻은 RNA 용액을 희석하여 200 NG에 마이크로 어레이 분석을위한 총 RNA의 시작 양을 설정합니다. 추가1.5 μL의 최종 부피 1.5 ml의 마이크로 원심 튜브에 RNA 적당량.
- 각각의 RNA 샘플로 희석 스파이크 믹스 2 μl를 추가하고, 첫번째 가닥의 cDNA를 합성 된 cRNA이 점을 전사 및 시아닌 3CTP로 된 cRNA 라벨 제조업체의 지침을 따르십시오.
- 제조업체의 지침에 따라 표지 된 cRNA를 정화 30 μL의 RNase없는 물에 용출.
- 분광 광도계에 세 개의 값을 기록하여 각각 된 cRNA의 수율 및 특정 활동을 결정합니다 시아닌 3 염료 농도 (㎕를 -1 pmol의), RNA 순도 (A 280 분의 260)과 된 cRNA 농도 (ng의 μL -1). 된 cRNA 수율 (μg의) 및 특정 활동 (μg의의 cRNA를 당 pmol의의를 Cy3)를 계산하는 제조업체의 사용 설명서에있는 수식을 사용합니다.
주 : 추천 수율 특정 활동 특정 마이크로 어레이 포맷에 기초하여 상이하다. 이 사례에서, 4 팩 44K 형식이었다선택 권장 수율은 1.65이었고, 특정 활동 (9)이었다. - 프로브 설계에 적합한 소프트웨어를 사용하여 사용자 마이크로 어레이 디자인.
- 이 사례에서 설명 된 결과를 사용자 마이크로 어레이 디자인 및 라이브러리 올리고 뉴클레오티드 웹 기반 애플리케이션을 이용하여 4- 팩 44K 포맷에 설계된 새로운 맞춤형 마이크로 어레이를 준비한다. 디자인 프로브는 29971이 피노 누아 V1 배열에서 성적 증명서를 예측 포함, 34,651 목표 성적 증명서와 일치하는,, 조기의 사체 재구성 180 개인, 조기 유전자 (25)에 의해 피노 품종에서 확인 된 4,500 새로운 궤적.
참고 :이 29,798 피노 누아는 성적 증명서, 4392 새로운 피노 궤적과 179, 조기 개인 유전자를 예측 포함, 34,651 특정 60 머 프로브의 생산을하고있었습니다.
- 이 사례에서 설명 된 결과를 사용자 마이크로 어레이 디자인 및 라이브러리 올리고 뉴클레오티드 웹 기반 애플리케이션을 이용하여 4- 팩 44K 포맷에 설계된 새로운 맞춤형 마이크로 어레이를 준비한다. 디자인 프로브는 29971이 피노 누아 V1 배열에서 성적 증명서를 예측 포함, 34,651 목표 성적 증명서와 일치하는,, 조기의 사체 재구성 180 개인, 조기 유전자 (25)에 의해 피노 품종에서 확인 된 4,500 새로운 궤적.
- 다음 4 팩 포맷 사양에 기초하여 혼성화 조립체를 준비한다.
- 41.8 최종 부피 Cy3가 표지 된 cRNA를 1.65 μg의 배치탈의 RNase없는 물 μL. 11 배 차단 에이전트의 μl를 2.2 ㎕의 25 배 분할 버퍼를 추가합니다. RNA를 단편화 항온조에서 30 분 동안 60 ℃에서 인큐베이션. 1 분 얼음에 즉시 차가운.
- 마지막으로 2 배 하이브리드 버퍼의 55 μl를 추가 실온에서 15,500 XG에서 1 분 동안 피펫과 스핀으로 잘 섞는다. 즉시 얼음에 microcentrifuge 관을 배치합니다. 를 저장하지 않는, 즉시 사용합니다.
- 다음과 같이 정의 마이크로 어레이를 넣습니다.
- 혼성화 챔버의 기지로 레이블이 위를 향하게하여 가스켓 슬라이드를로드합니다. 천천히 거품을 피하고, 피펫의 끝 액체를 분배는 물론 각 개스킷에 단계 3.2.6.2에서 얻은 하이브리드 샘플 100 μl를로드합니다.
- 천천히 사용자 정의 마이크로 어레이는 숫자 바코드가 직면 보장, 아래로 향하게 놓습니다. 샌드위치 쌍 제대로 정렬되어 있는지 확인합니다. 마지막으로 끼워에 혼성화 챔버의 덮개를 올려슬라이드 챔버 상에 클램프를 손으로 조입니다. 거품의 이동성을 평가하기 위해 조립 된 챔버를 돌립니다.
- 오븐 65 ° C로 설정된 하이브리드의 불고기 조립 슬라이드 챔버를 놓습니다. 10 rpm으로 회전하는 회 전자를 설정합니다. 하이브리드는 17 시간 동안 진행 할 수 있습니다.
- 다음과 같이 마이크로 어레이 슬라이드 세척을 진행합니다.
- 첫째, 세 개의 슬라이드 염색 요리를 준비하고 적절한 세척 버퍼로 채우기 : RT에서 세척 버퍼 1 2 요리와 사전 예열 (37 ° C) 세척 버퍼 2 1 접시.
- 하이브리드 챔버를 분해하고 샌드위치를 제거합니다. 이 위를 향하도록 마이크로 어레이 슬라이드 숫자 바코드로, 실온에서 세척 버퍼 1로 채워진 첫 번째 슬라이드 염색 접시에 샌드위치 잠수함과 깨끗한 집게의 도움으로, 마이크로 어레이 슬라이드에서 가스켓을 분리합니다. 빠르게 슬라이드 랙에 마이크로 어레이 슬라이드를 전송하고 세척 버퍼 1로 채워진 두 번째 슬라이드 염색 접시에 배치RT에서.
- 자석 교반기에 슬라이드 염색 접시를 넣고 적당한 교반하면서 1 분을 씻는다. 신속하게 사전 예열 (37 ° C) 세척 버퍼 2로 가득 세 번째 슬라이드 염색 접시로 슬라이드 랙을 전송하고 적당한 교반하면서 1 분을 씻는다.
- 천천히 슬라이드 염색 접시에서 랙을 제거하고 조심스럽게 방울을 피하고, 랙에서 슬라이드를 제거합니다.
참고 : 세척 버퍼 트리톤 X-102을 추가하고 아세토 니트릴 세척 단계를 생략하지 마십시오.
- 실온에서 어둠 속에서 세척 된 칩을 저장합니다.
- 마이크로 어레이를 스캔하여 현저한 특징을 추출합니다.
- 적합한 스캐너에 마이크로 어레이 슬라이드를 놓고 마이크로 어레이 제조업체의 지침 설명서에서 권장하는 매개 변수 설정을 사용하여 각 배열을 검사합니다. 여기에 설명 된 결과, 주사 절차를 용이하게하기 위해 슬라이드 홀더에 각각 마이크로 어레이 슬라이드를 배치했다.
- 출력을 가져 .SHP숫자 형광 값으로 디지털 신호를 변환 할 수있는 적절한 소프트웨어 파일. 혼성화 과정이 성공했는지 확인하기 위해 품질 관리 리포트를 확인한다.
- 여기에서 설명한 결과, 피쳐 추출 소프트웨어의 사용 설명서에 권장 파라미터 설정을 사용하여 표 4에 열거 된 파라미터는 제조자에 의해 주어진 정상 범위 내에 있다는 것을 확인하기 위해 품질 관리 리포트를 검사한다.
| 메트릭 이름 | 상한 | 하한 | 기술 |
| AnyColorPrcntFeatNonUnif | 1.00 | NA | 기능의 백분율 어느 채널 기능 비 균일 특이하다 그 |
| DetectionLimit | 2.00 | 0.10 | 평균 1을 더한 선형 농도 범위 아래의 스파이크 기능의 표준 편차 |
| absGE1E1aSlope | 1.20 | 0.90 | E1A 프로브의 농도 대 신호에 적합의 기울기의 절대 |
| MedCVProcSignal | 8.00 | NA | 처리 된 신호에 대한 중간 %의 CV |
| gNegCtrlAveBGSubSig | 5.00 | -10.00 | 배경의 평균 (BGSubSignal 신호를 의미하는 기능에서 BGUsed라는 값을 substracting에 의해 계산된다) 모든 적합 점 음성 대조군의 신호를 감산 |
| gNegCtrlAveNetSig | 40.00 | NA | 모든 적합 점 음성 대조군의 순 신호의 평균 |
| gNegCtrlSDevBGSubSig | 10.00 | NA | 의 표준 편차배경은 모두 적합 점 음성 대조군의 신호를 감산 |
| gNonCntrlMedCVProcSignal | 8.00 | NA | 비 제어 프로브 처리 된 신호에 대한 중간 값의 % CV |
| gSpatialDetrendRMSFilter | 15.00 | NA | 배경 디트 랜딩 맞는 잔류 |
표 4 : 주요 파라미터는 마이크로 어레이 혼성화의 품질을 확인하기 위해 체크한다.
- 마이크로 어레이 데이터를 처리합니다.
- 모든 마이크로 어레이 슬라이드 스캔하고 품질 관리 긍정적 평가되면, 각 프로브의 원료 형광 강도를 나타내는 각각의 단일 서브 어레이 결과 파일에서 gProcessedSignal 값을 선택하여 탭 구분 매트릭스 데이터를 준비한다.
- 스프레드 시트에서 각 배열 (P 값)과 calcul 내 75 번째 백분위 수의 데이터를 정상화는 R 비율을 계산하기 위해 모든 다른 서브 - 어레이들 사이의 모든 P 값의 평균을 먹었다.
- 이어서, 동일한 스프레드 시트 자체 서브 어레이의 R 비가 각각 gProcessedSignal 값을 정규화.
대사 체학 및 Transcriptomics 데이터의 자세한 통계 분석을 수행하십시오
- 통계 분석 소프트웨어를 준비합니다.
참고 :이 사례 연구, PCA을 수행 할 수 소프트웨어에서, PLS-DA와 O2PLS-DA를 사용 하였다.- 대사 체학 및 transcriptomics 데이터를 가져옵니다. MZmine 소프트웨어를 사용하여 얻은 데이터 행렬을 가져 오는 새로운 정규 프로젝트 → → 새 정규 프로젝트 파일로 이동합니다. 편집 → 전체 행렬 → 홈 트랜스하고 적절한 기본 및 보조 ID의 → 마침을 할당에서 다음을 클릭합니다.
- 평균 데이터 센터와 파레토 스케일을 사용하여 확장. 홈 창에서 편집 → M1에 가서 적절한 parame 변경데이터 의존 TERS. 여기에 설명 된 결과를 파에 단위 차이에서 스케일을 변경합니다.
- 단위 분산 설정을 사용하여 transcriptomics 데이터를 확장 할 수 있습니다.
- 다변량 통계 분석을 실시하고 있습니다.
- F의 igure 2에 도시 한 바와 같이, PCA를 구현한다. 이런 사례에서, PCA는 샘플들 사이의 주요 차이점은 상이한 숙성 단계를 반영하는 계절 성장을 보여준다.
- 작업 세트 창에서 모델 유형으로 PCA-X를 선택합니다. 를 눌러 자동 맞춤. 그들이 모델의 품질에 대한 아이디어를 제공으로 R2X (정액)와 2 분기 (정액)에주의는 값.
주 : 일반적으로, 매우 높은 R2X (정액)을 더 높은 값 모델이지만 모델 데이터를 오버 맞는 것이다. 2 분기 (정액) 값이 감소하기 시작하면 경험적 규칙으로, 우리는 주요 구성 요소를 추가 중지. - 그런 다음, 샘플의 수 그룹화를 보여줍니다 플롯을 볼 수 점수 → 분산을 선택합니다.
- PCA에 검사플롯 점수. 좋은 모델이 얻어지면 (Q2cum> 0.5)와, O2PLS-DA 모델 (단계 4.2.2)를 구축하는 샘플의 같은 클래스를 사용합니다.
- 작업 세트 창에서 모델 유형으로 PCA-X를 선택합니다. 를 눌러 자동 맞춤. 그들이 모델의 품질에 대한 아이디어를 제공으로 R2X (정액)와 2 분기 (정액)에주의는 값.
- 매크로 영역별로 분류 된 샘플들을 사용하여 두 O2PLS-DA 모델을 구축 200 순열 순열 시험을 사용하여 모델을 확인.
- 새로 → M1 → 관측 → 홈 화면으로 이동하여 클래스를 지정합니다. 원하는 클래스를 설정합니다. 그런 다음, O2PLS-DA를하는 PCA-X에서 모델 유형을 변경합니다. 를 눌러 자동 맞춤. PLS-DA 모델의 구성 요소의 개수가 O2PLS-DA와 동일한 것을 보장한다.
- O2PLS-DA 모델을 확인하려면, CV-ANOVA 분석으로 이동하고 오른쪽 p- 값을 참조하십시오. 또한, M2 → 홈 창에서와 같이 새를 클릭 PLS-DA에 O2PLS-DA의 모델 유형을 변경합니다. 를 눌러 자동 맞춤.
- 순열 → 분석 및 (200) 순열 (선택 해제 새로 고침 순열 옵션)을 수행하기 위해 이동합니다.
주 : 최종 출력 창이 표시되는 R2 값의 수오2 분기는 Y 축의 음의 부분에 충돌한다 동안 LD는 일반적으로 0.4 아래 값에서 Y 축했다. R2 및 / 또는 Q2 값이 잘못된 경우, 부품의 PLS-DA와 O2PLS-DA 모델 수를 줄이십시오. - 프로젝트 창에서 M2를 선택하고 플롯 및 샘플 클래스의 위치를 볼 수 분산 → 점수를 클릭하십시오로 이동합니다. 하나 이상의 특정 클래스의 특성을 대사 무엇을 관찰하려면 / 목록 → 분산 형 플롯로 이동합니다.
- 관찰에 선택 데이터 형식을 변경하고 존재하는 경우 부하는 → 각각 1, 2 또는 추가하거나 구성 요소로 PQ (CORR)와 Pred를 가산 컴포지션으로 X 축과 시리즈의 항목 시리즈를 추가하고 수정합니다.
- 플롯을 누르면 마우스의 오른쪽 버튼으로 분자와 클래스 사이 플롯 기호를 구분하는 조건으로 부동산 → 색상으로 이동하여 선택합니다. 원하는 특성을 가진 플롯을 수정 축 및 / 또는 스타일 → 레이아웃 → 형식 플롯으로 이동합니다.
<대사 체학 O2PLS-DA 분석, 히스토그램 등의 대사 또는 대사 특정 클래스의 상대적인 수준으로 존재 차이의 결과에 기초하여 리>. - transcriptomics의 O2PLS-DA 분석의 결과를 바탕으로, 검색 및 유전자 온톨로지 (GO)이 28 분류에 차등 변조 유전자를 할당합니다.
- 수동으로 대사 수준과 유전자 발현 사이의 관계를 확인합니다.
- F의 igure 2에 도시 한 바와 같이, PCA를 구현한다. 이런 사례에서, PCA는 샘플들 사이의 주요 차이점은 상이한 숙성 단계를 반영하는 계절 성장을 보여준다.
결과
이 문서에서 설명하는 사례 연구는 상대적으로 189 샘플 중 정량 분자 이온 플러스 자신의 동위 원소, 부가 물 및 일부 조각을 포함하여 552 신호 (m / Z 기능), (7 포도원 × 3 숙성 단계 × 3 성장 계절 X를 포함하는 최종 데이터 매트릭스를 산출 3 생물 복제). 데이터 포인트의 총 수는 따라서 104,328이었다. 분할 트리 분석은 대사 플러스 부가 물, 동위 원소 및 조각에 해당?...
토론
이 문서에서는 포도 베리 떼루아 개념을 해석하는 데 사용되는 대사 체학, transcriptomics 및 통계 분석 프로토콜을 설명합니다. HPLC-ESI-MS에 의해 대사 체학 분석 동시에 대사 산물을 대량을 검출하기에 충분히 민감하지만, 상대적 정량은 매트릭스 효과 이온 억제 / 강화에 의해 영향을 받는다. 그러나 유사한 접근 방식은 이미 조기는 열매의 숙성 및 수확 후 고사를 설명하는 데 사용되었으며, 매트릭...
공개
The authors have nothing to disclose.
감사의 말
This work benefited from the networking activities coordinated within the EU-funded COST ACTION FA1106 "An integrated systems approach to determine the developmental mechanisms controlling fleshy fruit quality in tomato and grapevine". This work was supported by the 'Completamento del Centro di Genomica Funzionale Vegetale' project funded by the CARIVERONA Bank Foundation and by the 'Valorizzazione dei Principali Vitigni Autoctoni Italiani e dei loro Terroir (Vigneto)' project funded by the Italian Ministry of Agricultural and Forestry Policies. SDS was financed by the Italian Ministry of University and Research FIRB RBFR13GHC5 project "The Epigenomic Plasticity of Grapevine in Genotype per Environment Interactions".
자료
| Name | Company | Catalog Number | Comments |
| Mill Grinder | IKA | IKA A11 basic | |
| HPLC Autosampler | Beckman Coulter | - | System Gold 508 Autosampler |
| HPLC System | Beckman Coulter | - | System Gold 127 Solvent Module HPLC |
| C18 Guard Column | Grace | - | Alltima HP C18 (7.5 mm x 2.1 mm; 5 μm) Guard Column |
| C18 Column | Grace | - | Alltima HP C18 (150 mm x 2.1 mm; 3 μm) Column |
| Mass Spectometer | Bruker Daltonics | - | Bruker Esquire 6000; The mass spectometer was equipped with an ESI source and the analyzer was an ion trap. |
| Extraction solvents and HPLC buffers | Sigma | 34966 | Methanol LC-MS grade |
| Sigma | 94318 | Formic acid LC-MS grade | |
| Sigma | 34967 | Acetonitrile LC-MS grade | |
| Sigma | 39253 | Water LC-MS grade | |
| Minisart RC 4 Syringe filters (0.2 μm) | Sartorius | 17764 | |
| Softwares for data collection (a) and processing (b) | Bruker Daltonics | - | Bruker Daltonics Esquire 5.2 Control (a); Esquire 3.2 Data Analysis and MzMine 2.2 softwares (b) |
| Spectrum Plant Total RNA kit | Sigma-Aldrich | STRN250-1KT | For total RNA extractino from grape pericarps |
| Nanodrop 1000 | Thermo Scientific | 1000 | |
| BioAnalyzer 2100 | Agilent Technologies | G2939A | |
| RNA 6000 Nano Reagents | Agilent Technologies | 5067-1511 | |
| RNA Chips | Agilent Technologies | 5067-1511 | |
| Agilent Gene Expression Wash Buffer 1 | Agilent Technologies | 5188-5325 | |
| Agilent Gene Expression Wash Buffer 2 | Agilent Technologies | 5188-5326 | |
| LowInput QuickAmp Labeling kit One-Color | Agilent Technologies | 5190-2305 | |
| Kit RNA Spike In - One-Color | Agilent Technologies | 5188-5282 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-5242 | |
| RNeasy Mini Kit (50) | Qiagen | 74104 | For cRNA Purification |
| Agilent SurePrint HD 4X44K 60-mer Microarray | Agilent Technologies | G2514F-048771 | |
| eArray | Agilent Technologies | - | https://earray.chem.agilent.com/earray/ |
| Gasket slides | Agilent Technologies | G2534-60012 | Enable Agilent SurePrint Microarray 4-array Hybridization |
| Thermostatic bath | Julabo | - | |
| Hybridization Chamber | Agilent Technologies | G2534-60001 | |
| Microarray Hybridization Oven | Agilent Technologies | G2545A | |
| Hybridization Oven Rotator Rack | Agilent Technologies | G2530-60029 | |
| Rotator Rack Conversion Rod | Agilent Technologies | G2530-60030 | |
| Staining kit | Bio-Optica | 10-2000 | Slide-staining dish and Slide rack |
| Magnetic stirrer device | AREX Heating Magnetic Stirrer | F20540163 | |
| Thermostatic Oven | Thermo Scientific | Heraeus - 6030 | |
| Agilent Microarray Scanner | Agilent Technologies | G2565CA | |
| Scanner Carousel, 48-position | Agilent Technologies | G2505-60502 | |
| Slide Holders | Agilent Technologies | G2505-60525 | |
| Feature extraction software v11.5 | Agilent Technologies | - | inside the Agilent Microarray Scanner G2565CA |
| SIMCA + V13 Software | Umetrics |
참고문헌
- Jessome, L. L., Volmer, D. A. Ion suppression: A major concern in mass spectrometry. Lc Gc N Am. 24 (5), 498-510 (2006).
- Kim, H. K., Choi, Y. H., Verpoorte, R. NMR-based plant metabolomics: where do we stand, where do we go?. Trends Biotech. 29 (6), 267-275 (2011).
- Sumner, L. W., Mendes, P., Dixon, R. A. Plant metabolomics: large-scale phytochemistry in the functional genomics era. Phytochem. 62 (6), 817-836 (2003).
- Bottcher, C., von Roepenack-Lahaye, E., Willscher, E., Scheel, D., Clemens, S. Evaluation of matrix effects in metabolite profiling based on capillary liquid chromatography electrospray ionization quadrupole time-of-flight mass spectrometry. Anal Chem. 79 (4), 1507-1513 (2007).
- Toffali, K., et al. Novel aspects of grape berry ripening and post-harvest withering revealed by untargeted LC-ESI-MS metabolomics analysis. Metabolomics. 7 (3), 424-436 (2011).
- Martin, J. C., et al. Can we trust untargeted metabolomics? Results of the metabo-ring initiative, a large-scale, multi-instrument inter-laboratory study. Metabolomics. 11 (4), 807-821 (2015).
- Jaillon, O., et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 449 (7161), 463-467 (2007).
- Velasco, R., et al. A high quality draft consensus sequence of the genome of a heterozygous grapevine variety. Plos One. 2 (12), (2007).
- Tornielli, G. B., Zamboni, A., Zenoni, S., Delledonne, M., Pezzotti, M., Gerós, H., Chaves, M., Delrot, S. Ch. 11. The Biochemestry of the Grape Berry. 11, (2012).
- Anesi, A., et al. Towards a scientific interpretation of the terroir concept: plasticity of the grape berry metabolome. BMC Plant Biol. 15, 1-17 (2015).
- Berdeja, M., et al. Water limitation and rootstock genotype interact to alter grape berry metabolism through transcriptome reprogramming. Hort Res. 2, 1-13 (2015).
- Carbonell-Bejerano, P., et al. Solar ultraviolet radiation is necessary to enhance grapevine fruit ripening transcriptional and phenolic responses. BMC Plant Biol. 14, 1-16 (2014).
- Carbonell-Bejerano, P., et al. Reducing sampling bias in molecular studies of grapevine fruit ripening: transcriptomic assessment of the density sorting method. Theor Exp Plant Phys. 28 (1), 109-129 (2016).
- Carbonell-Bejerano, P., et al. Circadian oscillatory transcriptional programs in grapevine ripening fruits. BMC Plant Biol. 14, 1-15 (2014).
- Cavallini, E., et al. Functional diversification of grapevine MYB5a and MYB5b in the control of flavonoid biosynthesis in a petunia anthocyanin regulatory mutant. Plant & Cell Physiol. 55 (3), 517-534 (2014).
- Cramer, G. R., et al. Transcriptomic analysis of the late stages of grapevine (Vitis vinifera cv. Cabernet Sauvignon) berry ripening reveals significant induction of ethylene signaling and flavor pathways in the skin. BMC Plant Biol. 14, 1-21 (2014).
- Dal Santo, S., et al. The plasticity of the grapevine berry transcriptome. Genome Biol. 14 (6), 1-17 (2013).
- Fasoli, M., et al. The Grapevine Expression Atlas Reveals a Deep Transcriptome Shift Driving the Entire Plant into a Maturation Program. Plant Cell. 24 (9), 3489-3505 (2012).
- Gambino, G., et al. Co-evolution between Grapevine rupestris stem pitting-associated virus and Vitis vinifera L. leads to decreased defence responses and increased transcription of genes related to photosynthesis. J Exp Bot. 63 (16), 5919-5933 (2012).
- Ghan, R., et al. Five omic technologies are concordant in differentiating the biochemical characteristics of the berries of five grapevine (Vitis vinifera L.) cultivars. BMC Genomics. 16 (1), 1-26 (2015).
- Pastore, C., et al. Selective defoliation affects plant growth, fruit transcriptional ripening program and flavonoid metabolism in grapevine. BMC Plant Biol. 13, 1-13 (2013).
- Pastore, C., et al. Increasing the source/sink ratio in Vitis vinifera (cv Sangiovese) induces extensive transcriptome reprogramming and modifies berry ripening. BMC Genomics. 12, 1-23 (2011).
- Rinaldo, A. R., et al. A Grapevine Anthocyanin Acyltransferase, Transcriptionally Regulated by VvMYBA, Can Produce Most Acylated Anthocyanins Present in Grape Skins. Plant Physiol. 169 (3), 1897-1916 (2015).
- Royo, C., et al. Developmental, transcriptome, and genetic alterations associated with parthenocarpy in the grapevine seedless somatic variant Corinto bianco. J Exp Bot. , 259-273 (2015).
- Venturini, L., et al. De novo transcriptome characterization of Vitis vinifera cv. Corvina unveils varietal diversity. BMC Genomics. 14, 1-13 (2013).
- Commisso, M., Strazzer, P., Toffali, K., Stocchero, M., Guzzo, F. Untargeted metabolomics: an emerging approach to determine the composition of herbal products. Comput Struct Biotechnol J. 4, 1-7 (2013).
- Pluskal, T., Castillo, S., Villar-Briones, A., Oresic, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 1-11 (2010).
- Ashburner, M., et al. Gene Ontology: tool for the unification of biology. Nat Genet. 25 (1), 25-29 (2000).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유