È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Il concetto Terroir interpretato attraverso uva a bacca Metabolomica e Trascrittomica
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo articolo descrive l'applicazione di metabolomica non mirati, trascrittomica e l'analisi statistica multivariata per le trascrizioni di bacche di uva e metaboliti al fine di ottenere una visione nel concetto terroir, vale a dire, l'impatto dell'ambiente sulla caratteristiche qualitative di bacche.
Abstract
Terroir si riferisce alla combinazione di fattori ambientali che influenzano le caratteristiche delle colture quali vite (Vitis vinifera) in base a particolari habitat e pratiche di gestione. Questo articolo mostra come certe firme terroir possono essere rilevati nel metaboloma bacche e trascrittoma della Corvina vite cultivar utilizzando l'analisi statistica multivariata. Il primo metodo richiede un piano di campionamento adeguato. In questo caso di studio, un clone specifico della cultivar Corvina è stato scelto per ridurre al minimo le differenze genetiche, ed i campioni sono stati raccolti da sette vigneti che rappresentano tre diverse macro-zone nel corso di tre diverse stagioni di crescita. Un approccio non mirati LC-MS metabolomica è raccomandato a causa della sua alta sensibilità, accompagnato da trattamento efficiente dei dati utilizzando il software MZmine e una strategia di identificazione metabolita sulla base dell'analisi albero frammentazione. analisi del trascrittoma completa può essere effettuata mediante microarraycontenenti sonde copre ~ 99% di tutti i geni di vite previsti, consentendo l'analisi simultanea di tutti i geni differenzialmente espressi nel contesto di diversi terroir. Infine, l'analisi dei dati multivariati basato su metodi di proiezione può essere utilizzato per superare il forte effetto specifico epoca, permettendo ai metabolomica e dati trascrittomica da integrare e analizzati in dettaglio per identificare correlazioni informativi.
Introduzione
l'analisi dei dati su larga scala basata sui genomi, la trascrittomica, proteomi e metabolomes di piante fornisce una visione senza precedenti sul comportamento di sistemi complessi, come ad esempio le caratteristiche terroir di vino che riflettono le interazioni tra piante di vite e il loro ambiente. Poiché il terroir di un vino può essere distinto anche quando cloni di vite identiche sono coltivate in vigneti diversi, l'analisi genomica è di scarsa utilità, perché i genomi clonali sono identici. Invece è necessario esaminare le correlazioni tra l'espressione genica e le proprietà metaboliche dei frutti di bosco, che determinano le caratteristiche qualitative del vino. L'analisi dell'espressione genica a livello delle prestazioni trascrittoma dalle proprietà chimiche simili di tutte le trascrizioni, che facilita l'analisi quantitativa sfruttando caratteristiche universali come ibridazione di sonde immobilizzate su microarray. Al contrario, i metodi analitici universali proteomica unND metabolomica sono più difficili a causa della enorme diversità fisica e chimica delle singole proteine e metaboliti. Nel caso di metabolomica questa diversità è ancora più estremo, perché i singoli metaboliti differiscono notevolmente in termini di dimensioni, la polarità, l'abbondanza e la volatilità, in modo che nessun singolo processo di estrazione o metodo analitico offre un approccio olistico.
Tra le piattaforme analitici adatti per i metaboliti non volatili, quelli basati sulla cromatografia liquida ad alta prestazione accoppiata alla spettrometria di massa (HPLC-MS) sono molto più sensibili rispetto alle alternative quali HPLC con raggi ultravioletti o diodi rivelatori a serie (HPLC-UV, HPLC-DAD ) o spettroscopia di risonanza magnetica nucleare (NMR), ma l'analisi quantitativa mediante HPLC-MS può essere influenzata da fenomeni quali l'effetto matrice e soppressione ionica / enhancement 1-3. L'indagine di tali effetti durante l'analisi di acini d'uva Corvina mediante HPLC-MS utilizzando una sorgente di ionizzazione elettrospray (HPLC-ESI-MS), ha mostrato che gli zuccheri e le altre molecole con i tempi di ritenzione più bassi sono stati fortemente underreported, probabilmente riflette il gran numero di molecole in questa zona, e che l'abbondanza di altre molecole potrebbe essere sottostimato, sopravvalutati o influenzato dalla effetto matrice , ma la normalizzazione dei dati per l'effetto della matrice sembrava avere impatto limitato sul risultato 4,5 complessiva. Il metodo descritto nel presente documento è ottimizzato per l'analisi dei metaboliti medio di polarità che si accumulano ad alti livelli nel acini d'uva durante la maturazione, e che sono significativamente influenzato dal terroir. Essi comprendono antociani, flavonoli, flavan-3-oli, procianidine, altri flavonoidi, resveratrolo, stilbeni, acidi idrossicinnamici e acidi idrossibenzoici, che insieme determinano il colore, il gusto e le proprietà relative alla salute dei vini. Altri metaboliti, come gli zuccheri e acidi organici alifatici, vengono ignorati perché quantificazione mediante HPLC-MS non è affidabile a causa della matrice di effect e soppressione ionica fenomeni 5. All'interno della gamma polarità selezionata con questo metodo, l'approccio è non mirati in quanto mira a rilevare come molti metaboliti differenti possibili 6.
Metodi trascrittomica che permettono di migliaia di trascrizioni di vite da monitorare simultaneamente sono facilitati dalla disponibilità della completa sequenza del genoma della vite 7,8. metodi trascrittomica I primi a base di high-throughput sequencing cDNA si sono evoluti con l'avvento di sequenziamento di prossima generazione in un insieme di procedure note come tecnologia di RNA-Seq. Questo approccio sta rapidamente diventando il metodo di scelta per gli studi di trascrittomica. Tuttavia, un grande corpo di letteratura basata su microarray, che consentono di migliaia di trascrizioni da quantificare in parallelo per ibridazione, ha accumulato per vite. Infatti, prima di RNA-Seq è diventata una tecnologia mainstream, molte piattaforme di microarray commerciali dedicati erano statisviluppato permettendo vite trascrittoma da ispezionare in grande dettaglio. Tra la vasta gamma di piattaforme, solo due ammessi genome-wide analisi del trascrittoma 9. La matrice più evoluti consentito l'ibridazione di un massimo di 12 campioni indipendenti su un unico dispositivo, riducendo così i costi di ogni esperimento. I 12 sub-array ciascuno composti 135.000 sonde 60-mer rappresentano 29,549 trascrizioni di vite. Questo dispositivo è stato utilizzato in un gran numero di studi 10-24. Queste due piattaforme sono state ormai fuori produzione, ma un nuovo microarray personalizzato è stato recentemente progettato e rappresenta uno sviluppo più recente in quanto contiene un numero ancora maggiore di sonde che rappresentano ulteriori geni di recente scoperta di vite 25.
I set di dati di grandi dimensioni-vendita prodotti da trascrittomica e metabolomica analisi richiedono metodi statistici per l'analisi dei dati, comprese le tecniche multivariate per determinare le correlazioni tra forma diversas di dati. Le tecniche multivariate più utilizzati sono quelli a base di proiezione, e questi possono essere sorvegliati, come ad esempio analisi delle componenti principali (PCA), o sotto la supervisione, come ad esempio bidirezionale proiezione ortogonale alle strutture latenti analisi discriminante (O2PLS-DA) 26. Il protocollo presentato in questo articolo utilizza PCA per l'analisi dei dati esplorativi e O2PLS-DA per identificare le differenze tra gruppi di campioni.
Protocollo
1. Selezionare materiali appropriati e costruire un piano di campionamento
- Inizia l'esperimento attraverso lo sviluppo di un piano di campionamento adeguato. Non esiste un approccio generico e universale in modo valutare ogni piano, caso per caso. Assicurarsi che il piano di campionamento afferma i luoghi di campionamento, i tempi e le modalità di campionamento preciso. Vedere la Figura 1 per il piano di campionamento utilizzato in questo caso di studio.
NOTA: In questo caso di studio, acini da un singolo clone (. Vitis vinifera cv Corvina, clone 48) sono stati raccolti da sette vigneti commerciali in tre diverse macro-zone in provincia di Verona (Lago di Garda, Valpolicella e Soave). Le caratteristiche principali di ciascun vigneto sono riassunti nella tabella 1. Bacche sono stati raccolti nel corso di tre stagioni di crescita (2006, 2007, e 2008) a tre punti di tempo, corrispondente alla invaiatura (l'insorgenza di maturazione), a metà maturazione e bacche mature.- Per ciascuna delle adesioni (vineyard / anno / fase di maturazione), raccolto 30 gruppi provenienti da diverse posizioni lungo due filari, con altezze e le posizioni sulla pianta randomizzati.
- Selezionare tre bacche a caso da ogni cluster, evitando quelli con danni visibili e / o segni di infezione.
- Ripetere i punti 1.1.1 e 1.1.2 per ottenere tre piscine indipendenti.
- Togliamo i semi le bacche e congelare il pericarpo immediatamente in azoto liquido.
- Crush 10 frutti di bosco surgelati da ogni pool con una smerigliatrice automatica mulino, e dividere ogni campione in polvere in due parti uguali, una per l'analisi trascrittomica e uno per l'analisi metabolomica.
- Conservare le polveri a -80 ° C.
| AM | BA | BM | CS | FA | MN | PM | |
| Macrozona | Soave | Lago di Garda | Valpolicella | Lago di Garda | Valpolicella | Valpolicella | Soave |
| Altezza (m) | 250 | 120 | 450 | 100 | 130 | 250 | 130 |
| portainnesto | 41B | S04 | K5BB | 420A | 420A | K5BB | 41B |
| direzione Row | EW | NS | EW | EW | EW | NS | NS |
| sistema di allevamento | Sistema Overhead (Pergola) | Sistema Overhead (Pergola) | Spara verticale di posizionamento (Guyot) | Sistema Overhead (Pergola) | Overhead System (Pergola) | Spara verticale di posizionamento (Guyot) | Spara verticale di posizionamento (Guyot) |
| tipo di terreno | argilla limosa | terra grassa | Argilla | terra grassa | argilloso | Silt terriccio | argilloso |
| Sesto d'impianto (m) | 3.20 x 1.00 | 4,50 x 0,80 | 4.00 x 1.25 | 3,50 x 1,20 | 3,50 x 0,75 | 2.80 x 1.00 | 1,80 x 0,80 |
| Calce Totale% | 3.9 | 19.3 | 18,3 | 14.4 | 31 | 5.9 | 27.9 |
| Attivo calce% | 0.5 | 2.6 | 9.4 | 6.3 | 11.3 | 3.1 | 8.3 |
| sabbia% | 15 | 47 | 66 | 42 | 29 | 13 | 36 |
| Loam% | 43 | 36 | 21 | 37 | 39 | 67 | 36 |
| argilla% | 42 | 17 | 13 | 21 | 32 | 20 | 28 |
| pH del terreno | 8.3 | 7.9 | 7.8 | 8.2 | 8.2 | 7.8 | 7.9 |
| Sostanza organica (%) | 2.9 | 2.5 | 2.2 | 1.2 | 2.9 | 1.6 | 2.5 |
| Fosforo intercambiabile (mg / kg) | 26 | 73 | 73 | 68 | 48 | 47 | 64 |
| Potassio scambiabile (mg / kg) | 190 | 376 | 620 | 230 | 168 | 154 | 126 |
| Magnesio intercambiabile (mg / kg) | 272 | 468 | 848 | 623 | 294 | 293 | 183 |
| Calcio scambiabile (mg / kg) | 6500 | 5380 | 7358 | 6346 | 4652 | 10055 | 2878 |
| Berry zuccheri riduttori 2006 | 211.25 ± 1.20 | 176.20 ± 0.42 | 187,40 ± 0.00 | 203.70 ± 1.13 | 212,55 ± 0,64 | 195.20 ± 0.00 | 211,65 ± 0,64 |
| Berry zuccheri riduttori 2007 | 190.00 ± 1.27 | 165.25 ± 0.49 | 153.00 ± 0.42 | 203,60 ± 0,71 | 210.90 ± 0.71 | 192,25 ± 0,64 | 188.70 ± 1.84 |
| Berry zuccheri riduttori 2008 | 191,35 ± 0,64 | 178.90 ± 0.57 | 170,05 ± 0,49 | 205.15 ± 1.48 | 188.70 ± 0.57 | 169.35 ± 0.49 | 108.05 ± 1.06 |
| Berry pH 2006 | 3.01 ± 0.01 | 2.96 ± 0.01 | 2.84 ± 0.00 | 2.9 ± 0.00 | 2.98 ± 0.00 | 3.02 ± 0.00 | 3.06 ± 0.01 |
| Berry pH 2007 | 2.97 ± 0.00 | 3.00 ± 0.00 | 2.74 ± 0.00 | 3.07 ± 0.01 | 2.98 ± 0.00 | 2.87 ± 0.01 | 3.09 ± 0.00 |
| Berry pH 2008 | 2.83 ± 0.00 | 3.04 ± 0.01 | 2.71 ± 0.00 | 2.98 ± 0.01 | 2.98 ± 0.00 | 2.82 ± 0.00 | 3.11 ± 0.00 |
| prelievo Data | 2006 | 2007 | 2008 | ||||
| Veraison | 8-ago | 18-lug | 12-ago | ||||
| Mid Maturazione | 4-Set | 8-ago | 2-Set | ||||
| Maturo | 18-Sep | 29-ago | 23-set |
Tabella 1: Principali caratteristiche di ogni vigneto ecampione di raccolta date. m = metri, EW = Eat-Ovest, NS = Nord-Sud. 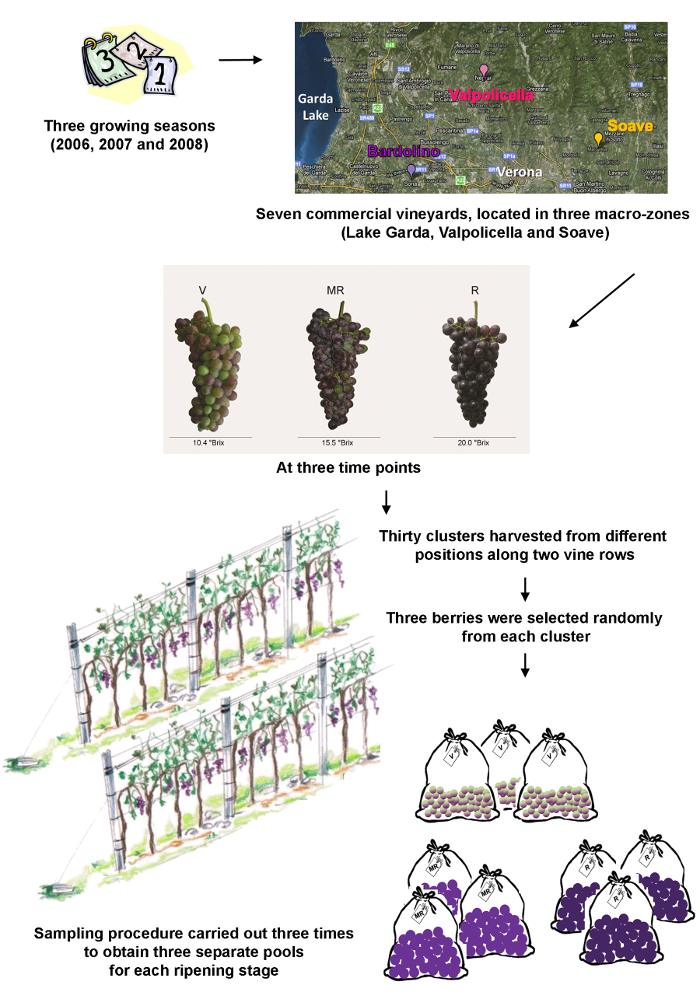
Figura 1:. Rappresentazione schematica della procedura di campionamento I macro-zone di produzione di vino e tre si trovano nei dintorni della città di Verona, regione Veneto, Italia. I tre punti di tempo sono invaiatura (V) che rappresenta l'inizio della maturazione in viticoltura, a metà maturazione (MR) e di frutti di bosco maturi (R). Cliccate qui per vedere una versione più grande di questa figura.
2. Preparare Berry in polvere Estratti, Analizzare i metaboliti ed elaborare i dati
- Preparare i campioni Berry in polvere per l'analisi.
- Preparare gli estratti metaboliche dei campioni bacca a temperatura ambiente in tre volumi (w / v) di metanolo acidificato con 0,1% (v / v) perL'acido microfono in un bagno a ultrasuoni a 40 kHz per 15 min. Utilizzare acqua LC-MS-grade e acido formico, e HPLC-grade metanolo.
- Centrifugare gli estratti a 16.000 xg per 10 minuti a 4 ° C.
- Diluire il surnatante dal punto 2.1.2 in due volumi (v / v) di acqua deionizzata e passare attraverso un filtro da 0,2 micron.
- Analizzare i estratti di bacche di HPLC-ESI-MS.
- Impostare il sistema HPLC-ESI-MS secondo le raccomandazioni del fornitore.
NOTA: In questo caso di studio, l'installazione comprende un sistema HPLC dotato di un campionatore automatico, collegato in linea con uno spettrometro di massa a trappola ionica e la sorgente di ionizzazione elettrospray. - Collegare il sistema HPLC con una precolonna C18 (7,5 x 2,1 mm 2) ed una colonna a fase inversa C18 (150 x 2,1 mm 2, granulometria 3 micron).
- Preparare i solventi usati come fase mobile. Utilizzare 5% (v / v) di acetonitrile, 0,5% (v / v) di acido formico in acqua come solvente A e 100% acetonitrile come solvent B. Usare acetonitrile LC-MS-grade, acqua e acido formico.
- Eseguire la separazione HPLC con un gradiente lineare di solventi A e B con un flusso costante di 0,2 ml min -1 utilizzando un volume di iniezione campione di 30 microlitri.
- Inizialmente equilibrare la colonna con 100% A. solvente Dopo l'iniezione del campione, stabilire un gradiente da 0% al 10% di solvente B in 5 minuti, dal 10% al 20% di solvente B in 20 min, dal 20% al 25% di solvente B in 5 min, e dal 25% al 70% solvente B in 15 min.
- Analizzare ogni campione in duplicato. Randomizza analisi del campione per evitare gli effetti dello strumento-driven. Lasciare 20 minuti per riequilibrio con 100% Un solvente tra ogni analisi.
- Acquisire spettri di massa in modalità di ionizzazione negativi e positivi alternativi. Per i risultati descritti in questo caso di studio, impostare i parametri come nella tabella 2. La regolazione precisa della macchina dipende dalla piattaforma specifica.
NOTA: In alternativa, utilizzare altro sememetodi in grado secondo la piattaforma specifica.- In tutti i casi, come con qualsiasi piattaforma di separazione, assicurarsi di utilizzare un adeguato tempo di ri-equilibrio, in modo da avere il tempo di ritenzione riproducibilità. Quando il numero di campioni è elevato (superiore dieci campioni), analizzarli in lotti di 9-10 campioni, con programmi di pulizia colonna cromatografica (pendenze lenti tra i due solventi di eluizione) tra ciascun lotto. Per avere il tempo di ritenzione riproducibilità, assicurarsi che la prima analisi cromatografica di ogni lotto è un'analisi vuota (cioè, l'analisi del solvente).
- Acquisire anche spettri di massa in modalità di ioni negativi e positivi frammentati impostando le opzioni di frammentazione (due precursori di ioni, MS 3). Annotate i metaboliti mediante analisi albero di frammentazione (MS / MS e MS 3) secondo le istruzioni del produttore.
NOTA: Questo è particolarmente adatta per i metaboliti vegetali perché sono spesso glicosilate, therrima il primo frammentazione (MS / MS) tende a rimuovere le frazioni di zucchero lasciando lo ione aglicone libera, e la conseguente frammentazione (MS 3) aiuta a identificare l'aglicone contro una libreria standard autentico. - Acquisire MS / MS e MS 3 spettri nel range m / z 50-1,500 con un'ampiezza frammentazione di 1 V. In alternativa, utilizzare altri metodi idonei a seconda della piattaforma specifica.
- Per ogni segnale m / z, confrontare la MS / MS e MS 3 modelli di frammentazione e tempi di ritenzione per l'autentico libreria standard in base alle istruzioni del produttore.
NOTA: molti tipi di piattaforme comprendono servizi software per costruire questo tipo di biblioteca attraverso l'analisi di standard autentici. Questo dovrebbe identificare molti dei segnali, ma non tutti i segnali verranno annotati. - Per i segnali anonimi rimanenti, confrontare i MS / MS e MS modelli 3 frammentazione con quelli pubblicati in letteratura, SearcHing per i valori / Z M (vale a dire, cifre "m / z 353" per cercare le pubblicazioni in cui le molecole con tanta m / z sono menzionati) con qualsiasi motore di ricerca comuni liberamente disponibili, oppure utilizzare i database online come MassBank (www .massbank.jp / it / database.html) e il metaboloma umano Database (www.hmdb.ca/search/spectra?type=ms_search).
- Impostare il sistema HPLC-ESI-MS secondo le raccomandazioni del fornitore.
| Componenti spettrometro di massa | Funzione | parametri |
| Elettrospray ionizzazione Fonte | nebulizzazione Gas | 50 psi, 350 ° C |
| gas di essiccazione | 10 L min -1 | |
| Ion trappola e rivelatore | Scansione | Modalità di scansione completa, 13.000 m / z per secondo, gamma 50-1,500 m / z |
| Di massa di destinazione | 400 m / z | |
| Collision Gas | Elio | |
| pressione di vuoto | 1.4 x 10 -5 mbar | |
| fonte capillare | 4000 V | |
| Offset Piastra terminale | -500 V | |
| scrematrice | -40 V | |
| uscita Cap | -121 V | |
| 1 ottobre DC | -12 V | |
| 2 ottobre DC | -1.7 V | |
| Obiettivo 1 | +5 V | |
| Obiettivo 2 | +60 V | |
| ICC per la modalità di ionizzazione positiva | 20.000 | |
| ICC per la modalità ionizzazione negativa | 7.000 |
Tabella 2: set di parametri principale per l'acquisizione di spettri di massa.
| operazione | Selezione | Funzione | parametri | Valori |
| rilevamento di picco | rilevazione di masse | centroid | Livello di rumore | 3.500 |
| cromatogramma builder | Punto più alto dei dati | min lasso di tempo | 0.15 | |
| altezza minima | 4.000 | |||
| m / z tolleranza | 0.3 | |||
| rilevamento di picco | deconvoluzione picco | Locale di ricerca minima | soglia cromatografica | 70 |
| Cerca minimo nella gamma RT (min) | 00:50 | |||
| altezza relativa minima | 15% | |||
| Altezza minima assoluta | 4.000 | |||
| rapporto minimo del picco superiore / bordo | 2 | |||
| gamma Durata (min) | 0-10 | |||
| isotopi | picchi isotopici cernia | - | m / z tolleranza | 1.2 |
| tolleranza RT | 00:50 | |||
| forma monotona | No | |||
| carica massima | 3 | |||
| isotopo rappresentante | No | |||
| Allineamento | Partecipa allineatore | - | m / z tolleranza | 1.2 |
| Peso per m / z | 10 | |||
| Tolleranza Tempo di ritenzione | 00:50 | |||
| Di peso per RT | 5 | |||
| Richiede stesso stato di carica | No | |||
| Richiede stesso ID | No | |||
| Confronta modello isotopo | No | |||
| riempimento Gap | finder Peak | - | tolleranza intensità | 20% |
| m / z tolleranza | 0.9 | |||
| Tolleranza Tempo di ritenzione | 00:40 | |||
| correzione RT | No | |||
| Filtraggio | Duplicate filtro Peak | - | m / z tolleranza | 1.2 |
| tolleranza RT | 00:30 | |||
| Richiede stessa identificazione | No |
Tabella 3: flusso di lavoro Mzmine con valori specifici per elaborare i file di dati di uva a bacca negativi LC-MS.
- Elaborare il LC-MS dati.
- Access oscaricare un pacchetto software di elaborazione dati in grado di estrarre informazioni rilevanti da molti cromatogrammi prime e costruire una matrice di dati in cui ogni metabolita rilevato viene quantificato in ogni campione.
NOTA: passi il protocollo di seguito sono su misura per il software open-source MZmine v2.14 (http://mzmine.sourceforge.net). Si tratta di un software open-source per l'elaborazione dei dati spettrometria di massa, con l'obiettivo principale su dati LC-MS. 27 - Trasformare i dati cromatogramma LC-MS in formato netCDF utilizzando il software fornito dal produttore dell'apparecchiatura. Per i risultati qui descritti, utilizzare il v5.2 Bruker Daltonics Esquire e software v3.2 analisi dei dati e eseguire le operazioni secondo le istruzioni del produttore.
NOTA: Altri convertitori possono essere utilizzati (vari convertitori libero-ware sono disponibili). - Importare i file cdf nel software.
- Attuare le procedure di filtro di rilevamento di picco, di allineamento, riempimento divario e di picco con la PARAMETRIERS riportati nella Tabella 3.
- Come primo passo, selezionare un file cdf importato, quindi passare alla visualizzazione → TIC / XIC Visualizer. Posizionare il cursore sulla base del più piccolo picco nel cromatogramma e prendere nota sul segnale minima intensità dello ione base. Poi vai a prime metodi di data → rilevamento di picco.
- Selezionare massa di rilevamento e riempire il livello del segnale per rilevare ioni individuali per ogni scansione e creare un elenco di ioni.
NOTA: Controllare l'algoritmo prima di eseguire la rilevazione di masse - dipende spettrometro di massa. Nel nostro caso, selezioniamo Centroid. - Selezionare Cromatogramma Builder e riempire con il livello di segnale per collegare i punti dati dalla lista di ioni e costruire un cromatogramma per ogni valore di massa. Vedere i parametri specificati nel manuale del spettrometro di massa per regolare il minimo del periodo di tempo e m / z tolleranza.
- Poi, vai a Peak List Metodi → Peak Detection → Cromatogramma Deconvoluzione. Selezionare l'algoritmo appropriato (in questo caso di ricerca locale minima) per consentire la separazione di ciascun cromatogramma in singoli picchi.
- controllare manualmente i cromatogrammi per scoprire i valori appropriati per i seguenti parametri. Impostare la Soglia cromatografica al 30% per rimuovere il rumore e Search minima in RT Gamma 2 (min) per identificare la presenza di locali minime di discriminare due picchi.
- Impostare minimo relativo Altezza al 2.0. ispezionare manualmente il cromatogramma per identificare l'altezza minimo assoluto di segnale che corrisponde a un picco e non allo sfondo; impostare questo valore come altezza assoluto (ad esempio, 10.000 nell'esperimento presentato).
- Impostare il rapporto minimo di Peak Top / Edge 1.1 per indicare il rapporto minimo tra la parte superiore e il più basso intensità dei punti dati di un picco di essere riconosciuto come un vero e proprio picco.
- ispezionare manualmente i cromatogrammi per vedere quale è la durata minima dei diversi picchi in Chromat usatocondizioni nienza geografica (per esempio, tra 0,2 e 2 minuti, a seconda del composto, negli esperimenti presentati), e di conseguenza utilizzano questi valori come Durata Peak Range (min) per impostare l'intervallo di lunghezza picco accettabile durata.
- Poi vai a picco metodi delle liste → → Isotopi isotopica Peaks Cernia di gruppo isotopi un picco, di solito nella più intensa. Nota: I parametri dipendono dalla risoluzione dello spettrometro di massa e la riproducibilità dei tempi di ritenzione.
- Infine, andare a picco Lista Metodi → → Allineamento Iscriviti Aligner per allineare i picchi in funzione del loro m / z e ritenzione tempo utilizzando un punteggio partita.
- Dopo aver allineato le cime, colmare eventuali lacune di dati cliccando sul Peak metodi delle liste → Gap Filling → Peak Finder. Infine, il filtro duplicare i punti dati cliccando sul Peak List Metodi → Filtrazione → Duplica Filtro Peak.
- Esportare il set di dati risultante come csv file.
- Modificare manualmente il. Estensione csv ad una. Estensione txt. Se non ci sono le estensioni dei file sono visibili, modificare le impostazioni del computer per rivelare le estensioni dei file. Vai a File Explorer Opzioni → Visualizza → Impostazioni avanzate e deselezionare le "Nascondi le estensioni per i tipi di file conosciuti 'box.
- Importare i file txt in un foglio di calcolo. Questo produce una matrice di dati in cui tutti i metaboliti rilevati, riconosciuti da un tempo numero di identificazione, m / z valore e ritenzione, vengono quantificati in tutti i campioni in termini di valori di area di picco.
- Access oscaricare un pacchetto software di elaborazione dati in grado di estrarre informazioni rilevanti da molti cromatogrammi prime e costruire una matrice di dati in cui ogni metabolita rilevato viene quantificato in ogni campione.
3. Preparare Berry in polvere Estratti per analisi del trascrittoma ed elaborare i dati
- Estratto RNA totale dai campioni Berry e determinano la qualità di RNA.
- Estratto RNA totale dai campioni di bacche.
NOTA: In questo caso di studio, un kit proprietario e una procedura modificata che assicura la completa rimozione di molecole che trafere con RNA analisi, quali polisaccaridi e polifenoli, sono stati utilizzati. Le istruzioni che seguono sono specifici per questo kit 18.- Pesare 400 mg di polvere di bacche per ogni campione, dividerlo in due parti e posizionare 200 mg ciascuna in due tubi microcentrifuga.
- Aggiungere 900 microlitri della soluzione di lisi contenente β-mercaptoetanolo (fornito nel kit) per ogni 200 mg di polvere e miscelare immediatamente e vigorosamente per almeno 30 secondi. Riscaldare il campione a 56 ° C per 5 min agitando a 800 rpm.
- Centrifugare i campioni a massima velocità in una microcentrifuga da banco per 10 minuti per far sedimentare i detriti cellulari.
- Dispensare 700 ml di surnatante nella colonna di filtrazione fornito nel kit (anello blu fermo) seduto in un tubo di raccolta da 2 ml. Chiudere il tappo e centrifugare a massima velocità in una microcentrifuga da banco per 1 minuto per rimuovere i detriti residui. Ripetere questa operazione due volte utilizzando la stessa colonna di filtrazione, ma un tubo collezione fresca, con conseguente tre tUBE ciascuna contenente ~ 700 ml di lisato chiarificato.
- Dispensare 750 ml di soluzione di rilegatura (fornito nel kit) in ciascun tubo di lisato chiarificato e mescolare immediatamente pipettando su e giù almeno cinque volte. Trasferire 700 ml di questa miscela alla colonna vincolante fornito nel kit (anello di fermo rosso) seduto in un tubo di raccolta da 2 ml. Chiudere il tappo e centrifugare a massima velocità in una microcentrifuga da banco per 1 minuto per legare l'RNA.
- Decantare la frazione flow-through, invertire il tubo di raccolta e toccare brevemente su un rilievo pulito di carta assorbente per drenare il liquido residuo.
- Ritorna la colonna al tubo di raccolta e seminare la miscela restante nella stessa colonna e ripetere la centrifugazione e decantazione passi. Ripetere fino l'intera miscela è stata filtrata nella stessa colonna rossa vincolante.
- Ora seguire le istruzioni rimanenti nel kit ed eluire l'RNA nel tampone di eluizione 50 microlitri (fornito nel kit) e memorizzare it a -80 ° C pronti per le fasi di controllo di qualità.
- Determinare la quantità e la purezza del RNA utilizzando uno spettrofotometro. Registrare i rapporti di assorbanza che rivelano il grado di contaminazione con proteine (A 260/280) e polifenoli / polisaccaridi (A 260/230).
NOTA: RNA adatto per microarray ibridazione dovrebbe segnare almeno 1.8 per entrambi i rapporti. - Determinare l'integrità dell'RNA.
NOTA: Vari sistemi possono essere utilizzati. Un acquirente digitale che esegue una corsa elettroforetica capillare in combinazione con un colorante fluorescente è stato utilizzato in questo caso di studio. RNA adatto per microarray ibridazione dovrebbe avere un RNA Integrity Number (RIN) di almeno 8.
- Estratto RNA totale dai campioni di bacche.
- Preparare Campioni e ibridazione l'RNA di un microarray personalizzato.
- Impostare l'importo di partenza di RNA totale per l'analisi di microarray a 200 ng diluendo la soluzione di RNA ottenuto nel passaggio 3.1.1.8 con acqua priva di RNasi deionizzata. Aggiungi iladeguata quantità di RNA in una provetta da 1,5 ml microcentrifuga in un volume finale di 1,5 ml.
- Aggiungere 2 ml di mix picco diluito a ciascun campione di RNA e seguire le istruzioni del produttore per sintetizzare cDNA primo filone, trascrivere questo in cRNA ed etichettare il cRNA con cianina 3CTP.
- Purificare il cRNA etichettata secondo le istruzioni del produttore ed eluire in 30 acqua priva di RNasi microlitri.
- Determinare l'attività resa e specifico di ogni cRNA registrando tre valori su uno spettrofotometro: cianina 3 concentrazione di colorante (pmol ml -1), RNA di purezza (A 260/280) e concentrazione cRNA (ng ml -1). Utilizzare le formule si trovano in manuale di istruzioni del produttore per calcolare il rendimento cRNA (mg) e l'attività specifica (pmol Cy3 per mcg cRNA).
NOTA: positiva rese e attività specifiche differiscono sulla base del formato microarray specifico. In questo caso di studio un formato 44K 4-Pack è statoprescelto, la resa consigliato era 1,65 e l'attività specifica era 9. - Progettare il microarray personalizzato utilizzando il software adatto per la progettazione della sonda.
- Per i risultati descritti in questo caso di studio, preparare un nuovo microarray personalizzato, progettato sul formato 44K 4-pack utilizzando un'applicazione web-based per i disegni di microarray personalizzati e biblioteche oligonucleotidi. Sonde di design da abbinare 34,651 trascrizioni di destinazione, tra cui 29.971 predetto trascrizioni dal Pinot nero serie V1, 4.500 nuovi loci identificati nella Pinot cultivar da ricostruzione trascrittoma Corvina e 180 geni Corvina privato 25.
NOTA: Ciò ha comportato la produzione di 34,651 specifiche sonde 60-mer, che comprende 29.798 Pinot nero previsto trascrizioni, 4.392 nuovi Pinot loci e 179 geni Corvina privati.
- Per i risultati descritti in questo caso di studio, preparare un nuovo microarray personalizzato, progettato sul formato 44K 4-pack utilizzando un'applicazione web-based per i disegni di microarray personalizzati e biblioteche oligonucleotidi. Sonde di design da abbinare 34,651 trascrizioni di destinazione, tra cui 29.971 predetto trascrizioni dal Pinot nero serie V1, 4.500 nuovi loci identificati nella Pinot cultivar da ricostruzione trascrittoma Corvina e 180 geni Corvina privato 25.
- Preparare l'assemblaggio ibridazione sulla base di specifiche di formato 4-Pack come segue.
- Posizionare 1,65 mg di Cy3-marcato cRNA in un volume finale di 41,8ml di acqua priva di RNasi deionizzata. Aggiungere 11 ml di 10x agente di blocco e 2.2 ml 25x frammentazione tampone. Incubare a 60 ° C per 30 minuti in un bagno termostatico a frammentare RNA. Raffreddare immediatamente in ghiaccio per 1 minuto.
- Infine, aggiungere 55 ml di tampone di ibridazione 2x, mescolare bene pipettando e centrifugare per 1 min a 15.500 xg a temperatura ambiente. Prontamente posizionare la provetta in ghiaccio. Utilizzare immediatamente, non conservarlo.
- Caricare il microarray personalizzato come segue.
- Caricare un vetrino guarnizione con l'etichetta rivolta verso l'alto nella base di una camera di ibridazione. Lentamente caricare 100 microlitri di campione ibridazione ottenuti nel passaggio 3.2.6.2 su ogni guarnizione bene, erogazione del liquido con la punta della pipetta, evitando bolle.
- Lentamente collocare il microarray personalizzato rivolto verso il basso, in modo che il codice a barre numerico è rivolto verso l'alto. Assicurarsi che la coppia panino è allineato correttamente. Infine posizionare il coperchio della camera di ibridazione sul sandwichscivoli e serrare a mano il morsetto sulla camera. Ruotare la camera assemblato per valutare la mobilità di bolle.
- Posizionare camera di scorrimento assemblato in una rosticceria in un forno di ibridazione impostato a 65 ° C. Impostare il rotatore ruotare a 10 rpm. Lasciare ibridazione di procedere per 17 ore.
- Procedere con il lavaggio microarray diapositiva come segue.
- In primo luogo, preparare tre piatti di scorrimento-colorazione e riempirli con le opportune buffer di lavaggio: 2 piatti con tampone di lavaggio 1 a RT e 1 piatto con pre-riscaldato (37 ° C) il tampone di lavaggio 2.
- Smontare la camera di ibridazione e rimuovere il panino. Con il microarray diapositive codice a barre numerico rivolto verso l'alto, immergere il panino nel primo piatto slide-colorazione riempita con tampone di lavaggio 1 a temperatura ambiente e, con l'aiuto di pinze pulite, separare la guarnizione dalla diapositiva microarray. trasferire rapidamente il vetrino microarray in un rack scorrevole e posizionarlo nel secondo piatto slide-colorazione piena di Wash Buffer 1a RT.
- Mettere il piatto scorrevole colorazione su un agitatore magnetico e lavare 1 min con agitazione moderata. trasferire rapidamente il rack scorrevole nel terzo piatto slide-colorazione piena di pre-riscaldato (37 ° C) il tampone di lavaggio 2 e lavare 1 min con agitazione moderata.
- Rimuovere lentamente il rack dal piatto slide-colorazione e rimuovere con attenzione il vetrino dal rack, evitando goccioline.
NOTA: Non aggiungere Triton X-102 per i buffer di lavaggio e omettere la fase di lavaggio acetonitrile.
- Conservare il chip lavato al buio a temperatura ambiente.
- Eseguire la scansione del microarray ed estrarre le caratteristiche salienti.
- Posizionare il vetrino microarray in uno scanner adeguato e la scansione di ogni array utilizzando le impostazioni dei parametri consigliati nel manuale di istruzioni del produttore microarray. Per i risultati qui descritti, posizionare ogni diapositiva microarray in un portavetrini per facilitare la procedura di scansione.
- Importare l'uscita .shpdepositare in un apposito software che è in grado di convertire un segnale digitale in valori numerici fluorescenti. Verificare la relazione di controllo di qualità per garantire che la procedura di ibridazione ha avuto successo.
- Per i risultati qui descritti, utilizzare le impostazioni dei parametri raccomandati nel manuale di istruzioni del software di estrazione delle caratteristiche e controllare la relazione di controllo di qualità per garantire che i parametri elencati nella tabella 4 sono all'interno del normale intervallo indicato dal costruttore.
| Nome Metric | Limite superiore | Limite inferiore | Descrizione |
| AnyColorPrcntFeatNonUnif | 1.00 | N / A | Percentuale di caratteristiche che sono dotate di valori anomali non uniformità in uno dei due canali |
| DetectionLimit | 2.00 | 0.10 | più media 1 deviazione standard dei ins picco al di sotto del range di concentrazione lineare |
| absGE1E1aSlope | 1.20 | 0.90 | Assoluto della pendenza della forma per Signal vs. concentrazione di sonde E1A |
| MedCVProcSignal | 8.00 | N / A | % CV mediana per il segnale processato |
| gNegCtrlAveBGSubSig | 5.00 | -10.00 | Media di sfondo segnale di tutti i controlli negativi inlier sottratto (BGSubSignal è calcolato sottraendo un valore chiamato BGUsed dalla funzione significare segnale) |
| gNegCtrlAveNetSig | 40.00 | N / A | Media di segnale netto di tutti i controlli negativi inlier |
| gNegCtrlSDevBGSubSig | 10.00 | N / A | Deviazione standardsfondo sottratto i segnali di tutti i controlli negativi inlier |
| gNonCntrlMedCVProcSignal | 8.00 | N / A | % CV mediana per il segnale elaborato delle sonde non-controllo |
| gSpatialDetrendRMSFilter | 15.00 | N / A | Residuo di fondo l'eliminazione del trend in forma |
Tabella 4: principali parametri da controllare per verificare la qualità dei microarray ibridazione.
- Elaborare i dati microarray.
- Una volta che tutte le diapositive microarray sono stati analizzati ed il controllo di qualità è stato valutato positivamente, preparare un data-matrix delimitato da tabulazioni selezionando i valori gProcessedSignal da ogni singolo file-sottoarray risultato, che rappresenta le intensità di fluorescenza prime di ogni sonda.
- In un foglio di calcolo, normalizzare i dati sui 75 ° percentile all'interno di ciascun array (valori di P) e calculmangiato la media di tutti i valori di P tra tutti i diversi sub-array per calcolare il rapporto R.
- Poi, sullo stesso foglio, normalizzare ogni valore gProcessedSignal al rapporto R del proprio sub-array.
4. effettuare l'analisi statistica dettagliata delle Metabolomica e Trascrittomica dati
- Preparare il software di analisi statistica.
NOTA: In questo caso di studio, un software in grado di eseguire PCA, PLS-DA e O2PLS-DA è stato utilizzato.- Importare i metabolomica e dati trascrittomica. Vai a File → Nuovo progetto Regolare → Nuovo progetto regolare per importare la matrice di dati ottenuti con il software MZmine. Quindi fare clic su Modifica → Trasposizione l'intera matrice → Casa e assegnare l'appropriato ID → Fine primario e secondario.
- Significa centrare i dati e scalare utilizzando la scala di Pareto. Nella finestra di casa, andare in Modifica → M1 e cambiare il para appropriataTERS a seconda dei dati. Per i risultati qui descritti, cambiare la scala da unità di varianza a Par.
- Scalare i dati trascrittomica utilizzando l'impostazione Unità di varianza.
- Effettuare l'analisi statistica multivariata.
- Implementare il PCA come mostrato in F IGURA 2. In questo caso di studio, PCA rivela le principali differenze tra i campioni, che riflettono le diverse fasi di maturazione e di crescita stagioni.
- Nella finestra workset, selezionare PCA-X come tipo di modello. Press Autofit. Prestare attenzione alla R2X (cum) e Q2 (cum) valori che danno un'idea circa la qualità del modello.
NOTA: In generale, più alto è il valore migliore è il modello, ma i modelli con altissima R2X (cum) potrebbero over-adattarsi ai dati. Come regola empirica, smettiamo di aggiungere componenti principali quando il valore Q2 (cum) inizia a diminuire. - Quindi, selezionare punteggi → Scatter per vedere il grafico che mostra l'eventuale raggruppamento dei campioni.
- Controllare l'APCPunteggio Plot. Se si ottiene un buon modello (Q2cum> 0.5), utilizzare le stesse classi di campioni di costruire un modello O2PLS-DA (fase 4.2.2).
- Nella finestra workset, selezionare PCA-X come tipo di modello. Press Autofit. Prestare attenzione alla R2X (cum) e Q2 (cum) valori che danno un'idea circa la qualità del modello.
- La costruzione di due modelli O2PLS-DA che utilizzano i campioni suddivisi per macro-zone e validare i modelli utilizzando un test di permutazione con 200 permutazioni.
- Assegnare le classi andando a finestra Portale → Nuova Come → → M1 Osservazioni. Impostare le classi desiderati. Quindi, cambiare il tipo di modello da APC-X per O2PLS-DA. Press Autofit. Assicurarsi che il numero dei componenti del modello PLS-DA è uguale a quella del O2PLS-DA.
- Per validare il modello O2PLS-DA, va ad analizzare CV-ANOVA e vedere il diritto p-value. Inoltre, fare clic su Nuovo Come nella finestra Home → M2 e cambiare il tipo di modello da O2PLS-DA per PLS-DA. Press Autofit.
- Vai a Analizzare → permutazioni e di eseguire 200 permutazioni (de-selezionare opzione Ricalcola permutazioni).
Nota: L'output finale mostra una finestra in cui il valore R2 should generalmente colpito l'asse Y in valori sotto 0,4, mentre il Q2 dovrebbe colpire la parte negativa dell'asse Y. Se i valori R2 e / o Q2 sono corretti, ridurre il numero di componenti sia in modelli PLS-DA e O2PLS-DA. - Procedere per selezionare M2 nella finestra del progetto e cliccare su punteggi → Scatter a vedere la trama e la posizione delle classi di esempio. Per osservare ciò metaboliti caratterizzano uno o più specifici classi, andare per tracciare / Elenco → Scatter.
- Modificare il Seleziona tipo di dati di osservazioni e Carichi → Aggiungi Series e modificare la voce in asse X e serie in pq (corr) e Pred Comp in 1 e 2 o ulteriori componenti quando presente, rispettivamente.
- Con il tasto destro del mouse premuto sulla trama, andare a Proprietà → Colore e selezionare da termini di distinguere i simboli trama tra le molecole e le classi. Vai al formato → Format Plot → Asse e / o stili di modificare la trama con le caratteristiche desiderate.
- Sulla base dei risultati delle analisi metabolomica O2PLS-DA, le differenze presenti nei relativi livelli di metaboliti specifici o classi di metaboliti come istogrammi.
- Sulla base dei risultati delle analisi trascrittomica O2PLS-DA, recuperare e assegnare geni differenzialmente modulati a Gene Ontology (GO) le classificazioni 28.
- Identificare le relazioni tra i livelli di metaboliti e l'espressione genica manualmente.
- Implementare il PCA come mostrato in F IGURA 2. In questo caso di studio, PCA rivela le principali differenze tra i campioni, che riflettono le diverse fasi di maturazione e di crescita stagioni.
Risultati
Il caso di studio descritto in questo articolo ha prodotto una matrice di dati finale comprendente 552 segnali (m / z caratteristiche) tra ioni molecolari più i loro isotopi, addotti e alcuni frammenti, relativamente quantificati tra i 189 campioni (7 vigneti x 3 stadi di maturazione x 3 stagioni di crescita x 3 repliche biologiche). Il numero totale di punti di dati è stato quindi 104.328. Analisi dell'albero La frammentazione ha portato l'annotazione di 282 funzioni ...
Discussione
Questo articolo descrive le metabolomica, trascrittomica e protocolli di analisi statistica utilizzate per interpretare il concetto di terroir uva a bacca. Analisi Metabolomica mediante HPLC-ESI-MS è abbastanza sensibile da rilevare un gran numero di metaboliti simultaneamente, ma relativa quantificazione è influenzata dall'effetto matrice e ioni soppressione / miglioramento. Tuttavia, un approccio simile è già stato utilizzato per descrivere la maturazione e appassimento post-raccolta di frutti di bosco Corvina...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This work benefited from the networking activities coordinated within the EU-funded COST ACTION FA1106 "An integrated systems approach to determine the developmental mechanisms controlling fleshy fruit quality in tomato and grapevine". This work was supported by the 'Completamento del Centro di Genomica Funzionale Vegetale' project funded by the CARIVERONA Bank Foundation and by the 'Valorizzazione dei Principali Vitigni Autoctoni Italiani e dei loro Terroir (Vigneto)' project funded by the Italian Ministry of Agricultural and Forestry Policies. SDS was financed by the Italian Ministry of University and Research FIRB RBFR13GHC5 project "The Epigenomic Plasticity of Grapevine in Genotype per Environment Interactions".
Materiali
| Name | Company | Catalog Number | Comments |
| Mill Grinder | IKA | IKA A11 basic | |
| HPLC Autosampler | Beckman Coulter | - | System Gold 508 Autosampler |
| HPLC System | Beckman Coulter | - | System Gold 127 Solvent Module HPLC |
| C18 Guard Column | Grace | - | Alltima HP C18 (7.5 mm x 2.1 mm; 5 μm) Guard Column |
| C18 Column | Grace | - | Alltima HP C18 (150 mm x 2.1 mm; 3 μm) Column |
| Mass Spectometer | Bruker Daltonics | - | Bruker Esquire 6000; The mass spectometer was equipped with an ESI source and the analyzer was an ion trap. |
| Extraction solvents and HPLC buffers | Sigma | 34966 | Methanol LC-MS grade |
| Sigma | 94318 | Formic acid LC-MS grade | |
| Sigma | 34967 | Acetonitrile LC-MS grade | |
| Sigma | 39253 | Water LC-MS grade | |
| Minisart RC 4 Syringe filters (0.2 μm) | Sartorius | 17764 | |
| Softwares for data collection (a) and processing (b) | Bruker Daltonics | - | Bruker Daltonics Esquire 5.2 Control (a); Esquire 3.2 Data Analysis and MzMine 2.2 softwares (b) |
| Spectrum Plant Total RNA kit | Sigma-Aldrich | STRN250-1KT | For total RNA extractino from grape pericarps |
| Nanodrop 1000 | Thermo Scientific | 1000 | |
| BioAnalyzer 2100 | Agilent Technologies | G2939A | |
| RNA 6000 Nano Reagents | Agilent Technologies | 5067-1511 | |
| RNA Chips | Agilent Technologies | 5067-1511 | |
| Agilent Gene Expression Wash Buffer 1 | Agilent Technologies | 5188-5325 | |
| Agilent Gene Expression Wash Buffer 2 | Agilent Technologies | 5188-5326 | |
| LowInput QuickAmp Labeling kit One-Color | Agilent Technologies | 5190-2305 | |
| Kit RNA Spike In - One-Color | Agilent Technologies | 5188-5282 | |
| Gene Expression Hybridization Kit | Agilent Technologies | 5188-5242 | |
| RNeasy Mini Kit (50) | Qiagen | 74104 | For cRNA Purification |
| Agilent SurePrint HD 4X44K 60-mer Microarray | Agilent Technologies | G2514F-048771 | |
| eArray | Agilent Technologies | - | https://earray.chem.agilent.com/earray/ |
| Gasket slides | Agilent Technologies | G2534-60012 | Enable Agilent SurePrint Microarray 4-array Hybridization |
| Thermostatic bath | Julabo | - | |
| Hybridization Chamber | Agilent Technologies | G2534-60001 | |
| Microarray Hybridization Oven | Agilent Technologies | G2545A | |
| Hybridization Oven Rotator Rack | Agilent Technologies | G2530-60029 | |
| Rotator Rack Conversion Rod | Agilent Technologies | G2530-60030 | |
| Staining kit | Bio-Optica | 10-2000 | Slide-staining dish and Slide rack |
| Magnetic stirrer device | AREX Heating Magnetic Stirrer | F20540163 | |
| Thermostatic Oven | Thermo Scientific | Heraeus - 6030 | |
| Agilent Microarray Scanner | Agilent Technologies | G2565CA | |
| Scanner Carousel, 48-position | Agilent Technologies | G2505-60502 | |
| Slide Holders | Agilent Technologies | G2505-60525 | |
| Feature extraction software v11.5 | Agilent Technologies | - | inside the Agilent Microarray Scanner G2565CA |
| SIMCA + V13 Software | Umetrics |
Riferimenti
- Jessome, L. L., Volmer, D. A. Ion suppression: A major concern in mass spectrometry. Lc Gc N Am. 24 (5), 498-510 (2006).
- Kim, H. K., Choi, Y. H., Verpoorte, R. NMR-based plant metabolomics: where do we stand, where do we go?. Trends Biotech. 29 (6), 267-275 (2011).
- Sumner, L. W., Mendes, P., Dixon, R. A. Plant metabolomics: large-scale phytochemistry in the functional genomics era. Phytochem. 62 (6), 817-836 (2003).
- Bottcher, C., von Roepenack-Lahaye, E., Willscher, E., Scheel, D., Clemens, S. Evaluation of matrix effects in metabolite profiling based on capillary liquid chromatography electrospray ionization quadrupole time-of-flight mass spectrometry. Anal Chem. 79 (4), 1507-1513 (2007).
- Toffali, K., et al. Novel aspects of grape berry ripening and post-harvest withering revealed by untargeted LC-ESI-MS metabolomics analysis. Metabolomics. 7 (3), 424-436 (2011).
- Martin, J. C., et al. Can we trust untargeted metabolomics? Results of the metabo-ring initiative, a large-scale, multi-instrument inter-laboratory study. Metabolomics. 11 (4), 807-821 (2015).
- Jaillon, O., et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 449 (7161), 463-467 (2007).
- Velasco, R., et al. A high quality draft consensus sequence of the genome of a heterozygous grapevine variety. Plos One. 2 (12), (2007).
- Tornielli, G. B., Zamboni, A., Zenoni, S., Delledonne, M., Pezzotti, M., Gerós, H., Chaves, M., Delrot, S. Ch. 11. The Biochemestry of the Grape Berry. 11, (2012).
- Anesi, A., et al. Towards a scientific interpretation of the terroir concept: plasticity of the grape berry metabolome. BMC Plant Biol. 15, 1-17 (2015).
- Berdeja, M., et al. Water limitation and rootstock genotype interact to alter grape berry metabolism through transcriptome reprogramming. Hort Res. 2, 1-13 (2015).
- Carbonell-Bejerano, P., et al. Solar ultraviolet radiation is necessary to enhance grapevine fruit ripening transcriptional and phenolic responses. BMC Plant Biol. 14, 1-16 (2014).
- Carbonell-Bejerano, P., et al. Reducing sampling bias in molecular studies of grapevine fruit ripening: transcriptomic assessment of the density sorting method. Theor Exp Plant Phys. 28 (1), 109-129 (2016).
- Carbonell-Bejerano, P., et al. Circadian oscillatory transcriptional programs in grapevine ripening fruits. BMC Plant Biol. 14, 1-15 (2014).
- Cavallini, E., et al. Functional diversification of grapevine MYB5a and MYB5b in the control of flavonoid biosynthesis in a petunia anthocyanin regulatory mutant. Plant & Cell Physiol. 55 (3), 517-534 (2014).
- Cramer, G. R., et al. Transcriptomic analysis of the late stages of grapevine (Vitis vinifera cv. Cabernet Sauvignon) berry ripening reveals significant induction of ethylene signaling and flavor pathways in the skin. BMC Plant Biol. 14, 1-21 (2014).
- Dal Santo, S., et al. The plasticity of the grapevine berry transcriptome. Genome Biol. 14 (6), 1-17 (2013).
- Fasoli, M., et al. The Grapevine Expression Atlas Reveals a Deep Transcriptome Shift Driving the Entire Plant into a Maturation Program. Plant Cell. 24 (9), 3489-3505 (2012).
- Gambino, G., et al. Co-evolution between Grapevine rupestris stem pitting-associated virus and Vitis vinifera L. leads to decreased defence responses and increased transcription of genes related to photosynthesis. J Exp Bot. 63 (16), 5919-5933 (2012).
- Ghan, R., et al. Five omic technologies are concordant in differentiating the biochemical characteristics of the berries of five grapevine (Vitis vinifera L.) cultivars. BMC Genomics. 16 (1), 1-26 (2015).
- Pastore, C., et al. Selective defoliation affects plant growth, fruit transcriptional ripening program and flavonoid metabolism in grapevine. BMC Plant Biol. 13, 1-13 (2013).
- Pastore, C., et al. Increasing the source/sink ratio in Vitis vinifera (cv Sangiovese) induces extensive transcriptome reprogramming and modifies berry ripening. BMC Genomics. 12, 1-23 (2011).
- Rinaldo, A. R., et al. A Grapevine Anthocyanin Acyltransferase, Transcriptionally Regulated by VvMYBA, Can Produce Most Acylated Anthocyanins Present in Grape Skins. Plant Physiol. 169 (3), 1897-1916 (2015).
- Royo, C., et al. Developmental, transcriptome, and genetic alterations associated with parthenocarpy in the grapevine seedless somatic variant Corinto bianco. J Exp Bot. , 259-273 (2015).
- Venturini, L., et al. De novo transcriptome characterization of Vitis vinifera cv. Corvina unveils varietal diversity. BMC Genomics. 14, 1-13 (2013).
- Commisso, M., Strazzer, P., Toffali, K., Stocchero, M., Guzzo, F. Untargeted metabolomics: an emerging approach to determine the composition of herbal products. Comput Struct Biotechnol J. 4, 1-7 (2013).
- Pluskal, T., Castillo, S., Villar-Briones, A., Oresic, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 1-11 (2010).
- Ashburner, M., et al. Gene Ontology: tool for the unification of biology. Nat Genet. 25 (1), 25-29 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon