Method Article
Визуализация ДНК уплотнения в цианобактерий, томография высокого напряжения крио электрон
В этой статье
Резюме
Этот протокол описывает, как визуализировать переходных уплотнения ДНК в синезеленых водорослей. Синхронные культивирования, мониторинг микроскопии флуоресцирования, быстрое замораживание и высокого напряжения крио электронная томография используются. Представил протокол для этих методик, и будущие приложения и события обсуждаются.
Аннотация
Этот протокол описывает, как визуализировать переходных уплотнения ДНК в синезеленых водорослей. ДНК уплотнения является событием драматического цитоплазмы, недавно обнаружили в некоторых цианобактерий до деления клеток. Однако из-за большой размер и переходный характер, трудно исследовать структуру в деталях. Чтобы преодолеть трудности, во-первых, ДНК уплотнения можно воспроизвести производится в цианобактерии Synechococcus удлинённый PCC 7942 синхронных культуры с использованием 12 h каждого цикла свет/темно. Во-вторых ДНК Сжатие контролируется микроскопии флуоресцирования и захвачен быстрое замораживание. В-третьих, подробная структура ДНК уплотненных клетки визуализируется в трех измерениях (3D), крио электронная томография высокого напряжения. Этот набор методов широко применяется для изучения временных структур в бактерии, например деление клеток, хромосома сегрегации, фаг инфекции и т.д., которые контролируются микроскопии флуоресцирования и непосредственно визуализированное крио электронная томография в соответствующее время точках.
Введение
ДНК уплотнения является драматического цитоплазматических событие, которое определило в некоторых цианобактерий. Когда Synechococcus удлинённый был культивированный под 12 h каждого цикла свет/темно, ДНК, конденсированной появилась в конце периода света, который был явно отличается от его внешний вид, в то время очка1. Было высказано предположение о том, что этот процесс контролируется суточного часов, основанный на Кай белки2. Секи et al. сообщили что ДНК, окрашенных с Hoechst 33342 уплотняется в S. удлинённый клетки к концу периода света и показал волнистые стержня форму под флуоресцентным микроскопом. Уплотненный ДНК затем разделены на две в центре стержня как ячейки разделены и наконец вернулся в нормальное равномерное распределение в каждой ячейке дочь3. Однако его временный характер и большого размера для электронной микроскопии препятствия структурного анализа. Murata et al. в сочетании нескольких методов, включая синхронных культуры, микроскопии флуоресцирования, быстрое замораживание и высокого напряжения электрон крио томография (крио HVET) и удалось в определении структуры переходных ДНК уплотнения, включая кинетику полифосфат органов (ППБ)4. Рукопись обеспечивает визуальное объяснение такой сложный материал в деталях, объединяя экспериментальных процедур.
S. удлинённый имеет форму капсул, длиной 2-5 мкм, шириной о 0,5 мкм и идеальный ДНК уплотнения появляется в живых клетках только на очень короткое время. Таким образом структурные изменения, происходящие в цианобактерий уплотнения ДНК были неизвестны в деталях. Для того, чтобы расследовать эти структуры электронной микроскопии, необходимо преодолеть две основные технические проблемы. Одно наблюдение за такой толстый образца всего бактерии в вблизи собственных условий, и другой быстрой фиксации динамической структурой. Что касается первой проблемы неупругого означает свободный путь (iMFP) электронов зависит от ускоряющего напряжения микроскопа5. В просвечивающий электронный микроскоп (ТЕА) 300 кв, это менее чем 350 Нм. Например, когда лед встроенный цианобактерии (толщина образца ≈ 600 Нм) наблюдается в 200kV ТЕА (iMFP ≈ 250 Нм), структуры внутри ячейки трудно соблюдать. С другой стороны, 1 MV ТЕА (iMFP ≈ 500 Нм) может дать и изображения цитоплазматических структуры всей клетки (рис. 1). В этом протоколе, как часть решения, электронный микроскоп высокого напряжения (HVEM) на ускоряющего напряжения 1 MV было занято. Однако услуги, реализующих HVEM ограничены во всем мире. Возможные альтернативные решения, также обсуждаются в разделе обсуждения. Вторая проблема была решена путем крио электронная микроскопия (крио EM). Это мощный инструмент для визуализации динамических структур на вблизи родной условий, где быстро образец замораживается в жидкий Этан, используя устройство быстрого замораживания, и замороженные момент наблюдается непосредственно с минимальными изменениями6. Сочетание с томография, снимок трехмерной (3D) структуры могут быть реконструированы из серии наклона7. В этом эксперименте уплотнения ДНК был воспроизведен в S. удлинённый с использованием синхронных культуры под 12 h каждый свет/темно цикла и времени замораживания образца определяется мониторинга под флуоресцентным микроскопом.
Подходы, описанные здесь широко применяются для изучения динамических структур клетки бактерий, например деление клеток, хромосома сегрегации и ФАГ инфекции и имеют потенциал, чтобы открыть новые возможности в микробиологических исследованиях.
протокол
1. синхронные культура цианобактерий
- Культуры S. удлинённый PCC 7942 табличке стерилизованные BG 11 (в 9 см стерильных пластиковых Петри) содержащих агар 1,5% (w/v) и 0,3% (w/v) натрия тиосульфат8.
- Поместите пластины в камере роста при 23 ° C с интенсивностью освещения 50 µE/m2/s и с учетом 12 h свет/12 h темные циклов.
- Передача клетки на свежие плиты агара BG11 один раз в неделю.
Примечание: Культур на агаре будет отображаться как зеленые полосы активно пролиферирующих клеток после одной недели при условии этой культуры. - Взять зеленые Купы клеток с пламенем стерилизовать утопленными дужками и полоска клеток на свежий BG11 агар пластину. Это можно сделать на лавочке чистой.
2. Мониторинг по микроскопии флуоресцирования
- Используйте клетки культивировали на пластину агар для 6 дней наблюдать ДНК уплотнения. Соберите клетки от пластины в конце периода света, поливая 1 мл раствора сахарозы 0,2 М над клетки. Повторите, опрокинув решение на клетки так, что большинство клеток собираются. Передача приостановлена клеточной решения в капиллярной трубки для окрашивания ДНК.
- Добавьте ДНК, окрашивание раствор красителя (например Hoechst 33342) до 500 мкл раствора подвесной клетки в капиллярной трубки до конечной концентрации 1 мкг/мл. Затем держите трубку в темноте за 10 мин.
- Центрифуга для 1 мин на 2000 x g отложениях клетки. Отменить супернатант и 10 мкл раствора 0,2 М сахарозы для получения густой ячейки подвеска.
- Передача 1 мкл раствора, содержащего окрашенных клеток на слайд стакан, положите крышку выскальзования и наблюдать с флуоресцентным микроскопом с УФ фильтром, с помощью объектива с увеличением 100 X и Масло иммерсионное.
- Убедитесь, что уплотнение ДНК наблюдается в этот момент в большинстве клеток, а затем подготовить образец для следующего шага замораживания.
3. образец замораживания для крио HVET
- Настройка устройства Плунге замораживания. Заполнить бак с жидким азотом и начать охлаждения крио палата после подключения бака и камеры с тефлоновой трубкой.
- Заполнения жидким этаном в горшочке небольшой медные внутри камеры после охлаждения камере до температуры жидкого азота. Носите очки во время операции, так как жидкий Этан является взрывоопасной.
- Тлеющим разрядом стороне углерода EM Холи углерода покрытием сетки (Холи ГРИД) для 30 s 50 мА, с помощью bombarder ионов плазмы.
- Применять 1 мкл BSA золото трассировщик (15 Нм) для Холи сетки как маркер фидуциальный.
- Применить 2,5 мкл Алиготе клеток на стадии уплотнения ДНК к сетке Холи. Пятно от излишки раствора с фильтровальной бумаги. Сетки в пучину жидкий Этан с использованием плунжера в Плунге замораживание устройства немедленно.
- Храните замороженные сетки в жидком азоте хранения до тех пор, пока они изучаются.
4. крио HVET
- Настройка HVEM высоким напряжением 1 МВ.
- Смонтировать замороженных сетки в крио образец держатель для HVEM, охладить до-150 ° C с жидким азотом внутри крио станции и загрузить его в HVEM. Будьте осторожны избежать загрязнения по льду.
- Выберите изображения область в нижней увеличение 1, 000 X. Отрегулируйте высоту eucentric по оси z.
- Наклона этап образца до-60 ° и удалите зазор наклона вращения.
- Отрегулируйте фокус вблизи целевое расположение в масштабе 10, 000 X. Установить в фокусе 6-10 мкм, отклонение от целенаправленного изображения. Для изображений, установите доза 2 e–/Å-2 или меньше на образец заранее.
- Измерения дозы электронов по плотности тока на экране изображение в HVEM. Возьмите изображение на пленке электрона или цифровой камерой в том же масштабе как процесс фокусировки.
- Соберите изображения наклона вручную в том же порядке, как и в (4.5) от-60 ° до + 60 ° с шагом угол наклона 2 °-4 °.
Примечание: В многих современных электронных микроскопов, приобретение наклона серии автоматизированных сочетание цифровой камеры. В этом случае следуйте инструкции по эксплуатации. Для негативных пленок, развивать фильмы для 12 мин при 20° C в баке разработчика с помощью разработчик полную силу и исправить в закрепитель для 10 мин оцифровка фильмов с разрешением 4000 dpi (0,635 нм/пиксела на изображении) с помощью планшетного сканера. В случае цифровой камеры при условии собраны изображения напрямую к следующему шагу обработки изображений. При необходимости, уменьшите размер изображения, медианный фильтр, с помощью двух-четырех биннинга (размер снижения фактора) и соответствующего программного обеспечения (например ImageJ).
5. томографических реконструкция
- Сделайте файл образа стека от индивидуальных наклона изображения с помощью команды «tif2mrc» или «newstack» в IMOD программного обеспечения9.
- Запустите программное обеспечение eTomo GUI в IMOD и задать параметры изображения: размер пикселя, фидуциальный диаметр, вращение изображения, и т.д. Затем создайте скрипты.
- Выполнение индивидуальных программ по программное обеспечение, перечисленное в таблицах материалов, где наклон серии выравнивается с помощью координатных маркеров (с средняя остаточная ошибка меньше, чем 0,5). Наконец реконструировать 3D томограммы, с использованием алгоритма Сырт в IMOD.
- Область интересов (ROI) из томограммы и denoise, используя фильтр denoise: Фильтр анизотропного диффузии IMOD, двусторонних фильтра в Эман10или математическая морфология фильтровать11и т.д., с соответствующими параметрами для повышения контрастности.
6. Сегментация функцию интереса
Примечание: Описанной ниже процедуры зависит от используемого программного обеспечения (см. Таблицу материалов), но вместо него может использоваться другие пакеты программного обеспечения. Обратитесь к их руководство пользователя.
- В окне 3D просмотра откройте файл томограммы Amira программного обеспечения и создания OrthoSlice.
- В окне редактора сегментации создайте файл сегментации, выбрав новое поле «Метка».
- Вручную отслеживать границы функцию интерес (FOI). Следуйте FOI через все срезы томограмм. Для второй фол создайте новый «материал» и повторить ту же операцию.
- Создание поверхности отрисовки, выбрав в меню «SurfaceGen». Чтобы визуализировать сегментирована тома, выберите в меню «SurfaceView». Для перемещения, поворота и масштабирования в 3D-объем, используйте инструменты в окне просмотра 3D.
- Для автоматической сегментации используйте «Волшебная палочка». Нажмите на объект и отрегулируйте ползунки в отображения и маскировки, чтобы покрыть диапазон значений таким образом, чтобы полностью выбран объект, его особенности.
Результаты
В точного синхронного культуры под 12 h каждый свет/темно цикла ДНК, помечены Hoechst показывает нормальное равномерное распределение в темной состоянии (Рисунок 2a). Однако он постепенно сжимает внутри клетки в легких период и отображается в виде волнистой стержня как структура (рис. 2b) в конце периода света. Наконец стержень делит в центре (стрелки на рис. 2 c), и две его части распространяются в дочь клетки (Рисунок 2d). После деления клеток уплотненных ДНК исчезает сразу же, и ДНК возвращается к нормальной равномерное распределение.
Когда Алиготе клеток, содержащих клеток на заключительной стадии ДНК уплотнения сразу же перевели на Холи сетку и быстрое заморожены в жидкий Этан, и замороженные сетки наблюдалось на 1 MV крио HVEM, внутренние структуры цианобактерий включая ДНК, тилакоидной мембраны слои, клеточной стенки и ППБ, появился как моментальный снимок на данный момент замораживания (рис. 3a). Многие клетки показали различные уплотнения ДНК в клетках (белые стрелки на рисунке 3a) и может быть легко отличить от нормальных клеток (белые стрелки на рисунке 3b). Некоторые выставлены сужение в центре клетки, как ожидалось до деления клеток (желтая стрелка на рисунке 3a).
В 3D томограмм может дробиться основные Органеллы клетки; клеточной стенки, тилакоидной мембраны слои, ДНК и ППБ можно выделить (рис. 4). В частности уплотненных ДНК была отделена собственный разрыв в цитоплазму, где ДНК была окружена низкой плотности материала и тилакоидной мембраны слои были искажены вдоль волнистые штанги уплотненного ДНК. Динамическое поведение ППБ было вновь отмечено: в ДНК клетки, сжатой многие малые ППБ были замечены присоединиться к ДНК, в то время как они являются большими и меньше в нормальных клетках. Кроме того большая часть ППБ появилась как пары, и некоторые ДНК, по-видимому, в процессе отделения от ППБ. Это предполагает, что ППБ, сами разделяются два дублирования ДНК и функции как поставщиков фосфата для синтеза ДНК.
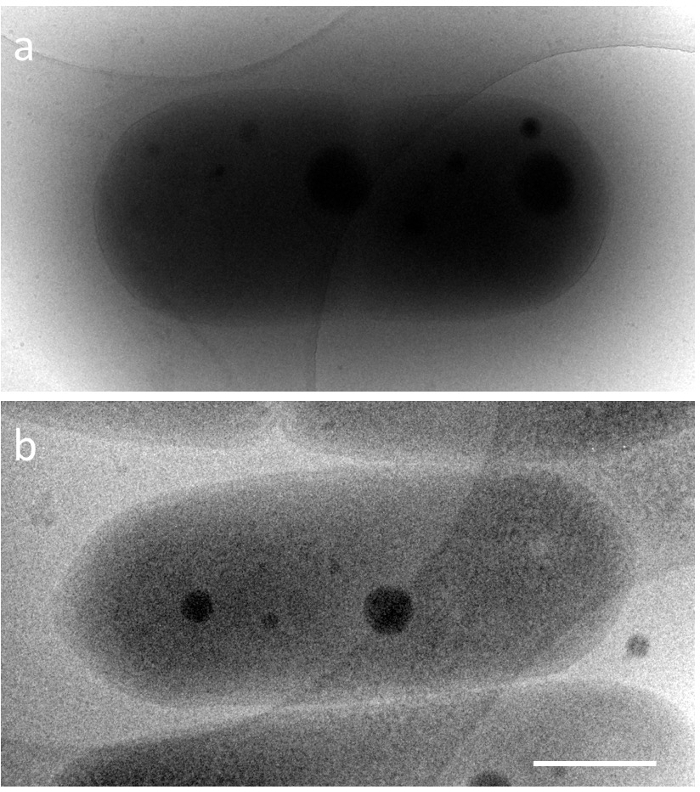
Рисунок 1. Крио-EM образы льда встроенный нормальной цианобактерий, S. удлинённый PCC 7942 различных ускоряющего напряжения в сети. ) 200kV и b) 1000кв. Шкалы бар = 500 Нм. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
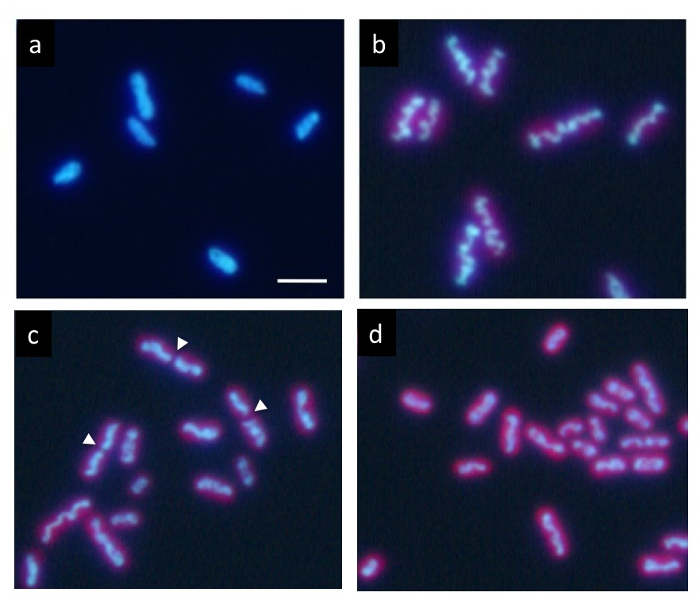
Рисунок 2: Микроскопии флуоресцирования цианобактерии клеток S. удлинённый . После синхронной культуры младше 12 час каждый свет/темно циклов клетки окрашивали Hoechst 33342. () клетки после 2 h наступлением темной государства демонстрируют единообразной маркировки ДНК. ДНК в клетках многих постепенно уплотняются в светлое время. В конечном счете он сформирован густой волнистой стержня (b) , типичный ДНК уплотнения. После этого этапа, уплотненной структуры ДНК, быстро разделены в их центрах (стрелок) во время деления клеток (c) и два фрагмента разделены на клетки дочи (d). Дочь клетки вернулся в единообразных ДНК маркировки по снова в темное время суток. Шкалы бар = 2 мкм. пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
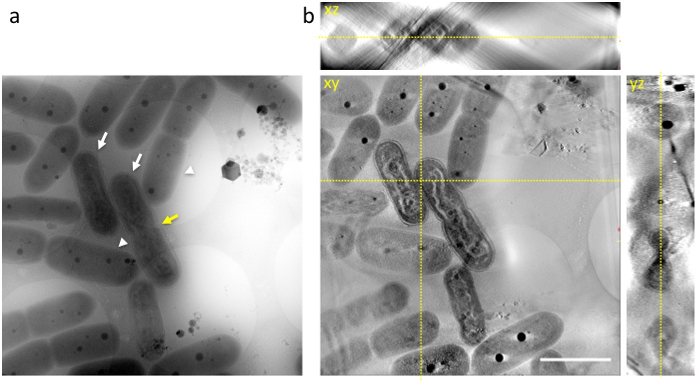
Рисунок 3: крио-HVEM изображение цианобактерий, встроенные в аморфных ice. (a) показывает изображения raw в наклона 0 °. Изображения были взяты в конце периода света. Некоторые клетки показывают волнистые палочковидные ДНК тела похож на хромосомы эукариот сгущенное (белые стрелки). В нормальных клетках цианобактерий экспонат заметной структуры ДНК в цитоплазме (белые стрелки). Некоторые демонстрируют сужение в центре клетки, как ожидалось до деления клеток (желтая стрелка). (b) xy, xz, yz ломтики 3D томограмм. Желтые пунктирные линии показывают пересечения фрагментов. Шкалы бар = 2 мкм. пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
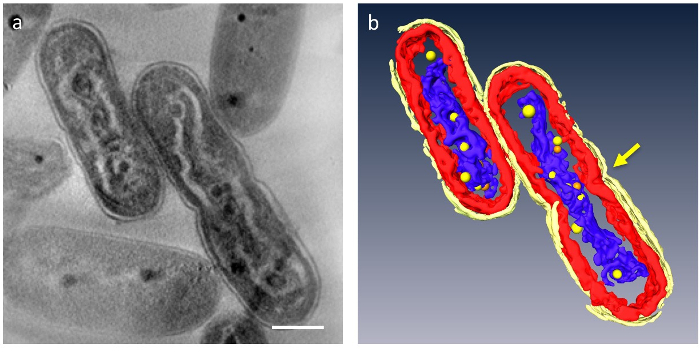
Рисунок 4: ультраструктуру клетки, содержащие уплотненных ДНК. Основными компонентами являются сегментированные: клеточной стенки (ярко-желтый), тилакоидной мембраны (красный) и ДНК (синий). (a) показывает z ломтик 3D томограмм. (b) все сегменты. Сужение, указанием разделения клеток появляется в центре ячейки (стрелка). ППБ моделируются как желтый или оранжевый сферах; Каждый оранжевый шар представляет двойники ближайшего желтые сферы. Шкалы бар = 500 Нм. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Обсуждение
Мы представили последовательность протоколов для визуализации переходных ДНК уплотнения в синезеленых водорослей. Основная концепция аналогична коррелятивных света и электронной микроскопии (Клем)12. Кроме того в этом методе, живой цианобактерий были под наблюдением микроскопии флуоресцирования, быстро заморожены на ет сетки и непосредственно визуализированы с крио электронная томография высокого напряжения. Как первое приложение, подробная структура ДНК уплотненных бактериальной клетки успешно был визуализирован в 3D. В настоящее время эта процедура относится только к этому вопросу, но он будет применяться более широко, с изменение методологии в некоторых случаях. Здесь обсуждаются преимущества, недостатки и будущие возможности этого метода.
Одним из преимуществ данного метода является 3D визуализация всей ячейки. 1 MV HVEM успешно визуализирована динамического сжатия структура внутриклеточных органелл в ДНК клетки. Однако тонкой структуры внутри нормальные клетки не могут различаться из-за низкой контрастности. Увеличение неупругого и многократного рассеяния в толстых образцах размывает изображение13. Нулевых потерь и большинство вероятная потери изображений, фильтрация фильтром энергии может улучшить контрастность изображения путем сокращения глубоконеупругого рассеяния14,15, но он не будет работать для образцов толще, чем iMFP. Вершины нулевых потерь и большинство вероятная потеря резко уменьшается с толщины образца. Это особенно трудно получить достаточно соотношение сигнал шум для чувствительных льда встроенный образцов электрона. Murata et al. показали, что 1 МВ сканирования передачи микроскопии (STEM) дает высокий контраст изображения чем светлые области изображения в пластик встроенный дрожжевых клеток толщиной 5 мкм, где контраст изображения определяется главным образом амплитуда контраст13 . Однако как ожидается, что эффект домино ущерба на более ускоренных электронов создает еще одно ограничение для дозы облучения для повреждения чувствительных крио образцы16. Приложение Вольта и Цернике фазы пластины17,18 для HVEM может иметь возможность уменьшить повреждения домино, уменьшая общая доза в будущем. Еще одно ограничение использования HVEM для толстых образцов происходит из того факта, что пользователь услуги, которые предоставляют HVEM ограничены во всем мире.
Использование альтернативной методологии для наблюдения за толщиной образцов, крио стволовых томография на 300 кв продемонстрировал высокий контраст изображения на замороженные гидратированных образцов с толщиной свыше нескольких сотен нанометров19. Чтобы получить фазового контраста в крио СТЕБЕЛЬ, pthychographic микроскопии также введена, в котором фазы пластины в объектив конденсатора транспонирует фаза модулированных дифракции чтобы неровной 2D детектор20. Изображения получаются путем расчета от нескольких дифракций. Для прямой и быстрый 3D крио изображений больших родной замороженных образцов, крио БМУ-SEM может также быть используется21, где серийный секционирование с целенаправленной ионного пучка и блокировать изображений лица применяется для визуализации полностью гидратированных замороженных образцов. Хотя эти технологии расширения спектра просмотра биологических образцов, трудно найти в целевое расположение бактерий, например помечены бактерий, потому что цель полностью под лед и не могут быть идентифицированы перед отделкой.
ДНК сжатие производит четкую структуру в синезеленых водорослей. Уплотненный ДНК клетки легко выделяются даже без витражи из-за большой плотности смещения внутри клетки, которая не присутствует в нормальных клетках. Однако для того, чтобы визуализировать более местные события в ячейке, необходимо перевести в электронный микроскоп дневно обозначенные ROI. Для корреляционного света и электронной микроскопии (Клем) световой микроскоп изображения и изображения микроскопа обычно связаны как с использованием флуоресцентных латексные шарики или квантовых точек на EM finder сетки12. Маркировки частицы должны быть высокой электронной плотности помимо флуоресценции. Они могут точно и достоверно коррелируют позиции между двумя изображениями. Кроме того подтверждая помечены зона с крио свет микроскопии, полное перекрытие ROI может быть достигнуто между двумя микроскопы. При характеристике более подробные структурные события в ДНК уплотнения, эти частицы и Микроскоп крио свет будет незаменимым инструментом для более надежной и точной корреляции в будущем.
Эта статья показывает, как характеризовать переходные структуры ДНК уплотнения в цианобактерий сочетанием синхронных культуры, флуоресцентной микроскопии и крио электронная томография высокого напряжения. Этот протокол сосредоточена на наблюдении за уплотненной ДНК. Объединив этот метод с других новых технологий, упомянутых выше, будет возможность исследовать процесс уплотнения ДНК более подробно, и измененном методы широко применяются для других динамических структурных мероприятий в бактерии.
Раскрытие информации
Авторы заявляют не конкурирующих финансовых интересов.
Благодарности
Авторы благодарят Tammo Reisewitz для критических чтении рукопись и как Мако Хаяси и Саюри Hagiwara для тщательного выращивания и наблюдения цианобактерий, Yoshitaka Kimori для обработки изображения, Chihong песни, Наоюки Миядзаки и Миёко Нагайоши за помощь с сегментация структур. Эта работа была поддержана совместной программы исследований Национального института для физиологических наук (НПВ) ю.к.
Материалы
| Name | Company | Catalog Number | Comments |
| Hoechst 33342 solution | Dojindo | 346-07951 | 1mg/mL in H2O |
| Agar Powder (for plant culture) | Wako | 016-11875 | |
| Boric acid | Wako | 027-02192 | 99.5% |
| Manganese chloride tetrahydrate | Wako | 139-00722 | 99% |
| Zinc sulfate heptahydrate | Wako | 264-00405 | 99.5% |
| Sodium molybdate | Wako | 196-02472 | 99% |
| Copper sulfate pentahydrate | Wako | 039-04412 | 99.5% |
| Cobalt nitrate hexahydrate | Wako | 031-03752 | 99.5% |
| Sodium nitrate | Wako | 191-02542 | 99% |
| Magnesium sulfate heptahydrate | Wako | 131-00405 | 99.5% |
| Calcium chloride dehydrate | Wako | 031-00435 | 99.5% |
| Citric acid | Wako | 036-05522 | 98% |
| EDTA-2Na | Dojindo | 343-01861 | 99.5% |
| Sodium carbonate | Wako | 197-01581 | 99.8% |
| Potassium phosphate dibasic | Wako | 164-04295 | 99% |
| TES (Good’s buffer) | Dojindo | 344-02653 | 99% |
| Ferric ammonium citrate | Wako | 092-00802 | 1st Grade |
| Sodium thiosulfate pentahydrate | Wako | 197-03585 | 99% |
| BSA gold tracer 15nm | Aurion | 215.133 | |
| Quantifoil EM grid | Quantifoil MicroTools | R3.5/1 Copper grid | |
| Electron films | Kodak | SO-163 | |
| Developer | Kodak | D19 | |
| Fixer | Kodak | Rapid fixer | Solution |
| Filter paper | Whatman | Grade 1 | |
| Growth chamber | NKsystem | LH-100SP | |
| Fluorescent microscope | Nikon | ECLIPSE 50i | |
| High voltage TEM | HItachi | H1250M | |
| Cryo-specimen holder for HVEM | Gatan | ||
| plunge-freezing device | Leica | EM CPC | |
| Plasma Ion bombarder | Vacuum device | PIB-10 | |
| Liquid nitrogen storage | Taylor-Wharton | 25LDB | |
| Developing tank | Dosaka EM | TB-3-75 | |
| flatbed scanner | Nikon | Coolscan 9000ED | |
| Segmentation software | FEI | Amira | https://www.fei.com/software/amira |
| Tomographic Reconstruction software | eTOMO | http://bio3d.colorado.edu/imod |
Ссылки
- Smith, R. M., Williams, S. B. Circadian rhythms in gene transcription imparted by chromosome compaction in the cyanobacterium Synechococcus elongatus. Proc. Nat. Acad. Sci. USA. 103 (22), 8564-8569 (2006).
- Kondo, T. A cyanobacterial circadian clock based on the kai oscillator. CSHS Quant. Biol. 72, 47-55 (2007).
- Seki, Y., Nitta, K., Kaneko, Y. Observation of polyphosphate bodies and DNA during the cell division cycle of Synechococcus elongatus PCC 7942. Plant biol. 16 (1), 258-263 (2014).
- Murata, K., Hagiwara, S., Kimori, Y., Kaneko, Y. Ultrastructure of compacted DNA in cyanobacteria by high-voltage cryo-electron tomography. Sci. Rep. 6, 34934 (2016).
- Koster, A. J., et al. Perspectives of molecular and cellular electron tomography. J. struct. boil. 120 (3), 276-308 (1997).
- Lučić, V., Rigort, A., Baumeister, W. Cryo-electron tomography: The challenge of doing structural biology in situ. J. Cell Biol. 202 (3), 407-419 (2013).
- Murata, K., et al. Visualizing Adsorption of Cyanophage P-SSP7 onto Marine Prochlorococcus. Sci. Rep. 7, 44176 (2017).
- Rippka, R., Deruelles, J., Waterbury, J. B., Herdman, M., Stanier, R. Y. Generic assignments, strain histories and properties of pure cultures of Cyanobacteria. J. Gen. Microbiol. 111, 1-61 (1979).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J. struct. boil. 116, 71-76 (1996).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. J. Struct. Biol. 144, 114-122 (2003).
- Kimori, Y. Morphological image processing for quantitative shape analysis of biomedical structures: effective contrast enhancement. J. Sync. Rad. 20, 848-853 (2013).
- Jun, S., Zhao, G., Ning, J., Ga Gibson, ., Watkins, S. C., Zhang, P. Correlative microscopy for 3D structural analysis of dynamic interactions. J. Visual. Exper. 76, e50386 (2013).
- Murata, K., Esaki, M., Ogura, T., Arai, S., Yamamoto, Y., Tanaka, N. Whole-cell imaging of the budding yeast Saccharomyces cerevisiae by high-voltage scanning transmission electron tomography. Ultramicros. 146, 39-45 (2014).
- Kortje, K. H., Paulus, U., Ibsch, M., Rahmann, H. Imaging of thick sections of nervous tissue with energy-filtering transmission electron microscopy. J. Microsc. 183, 89-101 (1996).
- Bouwer, J. C., et al. Automated most-probable loss tomography of thick selectively stained biological specimens with quantitative measurement of resolution improvement. J. Struct. Biol. 148 (3), 297-306 (2004).
- Egerton, R. F., Li, P., Malac, M. Radiation damage in the TEM and SEM. Micron. 35 (6), 399-409 (2004).
- Danev, R., Buijsse, B., Khoshouei, M., Plitzko, J. M., Baumeister, W. Volta potential phase plate for in-focus phase contrast transmission electron microscopy. Proc. Nat. Acad. Sci. USA. 111 (44), 15635-15640 (2014).
- Murata, K., et al. Zernike phase contrast cryo-electron microscopy and tomography for structure determination at nanometer and subnanometer resolutions. Structure. 18 (8), 903-912 (2010).
- Wolf, S. G., Houben, L., Elbaum, M. Cryo-scanning transmission electron tomography of biological cells. Nat. Methods. 11 (4), 423-428 (2014).
- Ophus, C., et al. Efficient linear phase contrast in scanning transmission electron microscopy with matched illumination and detector interferometry. Nat. Comm. 7, 1-7 (2016).
- Schertel, A., et al. Cryo FIB-SEM: Volume imaging of cellular ultrastructure in native frozen specimens. J. Struct. Biol. 184 (2), 355-360 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены