JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
'Peroxidative'와 '산화'히드록실 급진적 Footprinting에 의해 RNA 구조의 모니터링 평형 변경
요약
이 프로토콜은 MG (II) - 종속 히드록실 급진 footprinting 두 가지 방법으로 RNA 차 구조 형성을 계량하는 방법을 설명합니다.
초록
RNA의 분자 생물학에서 필수적인 역할을한다. 유전 정보를 전달하는 이외에, RNA는 레귤레이터, 바인더 또는 촉매와 같은 특정 생물 학적 역할을 수행 고유한 삼차 구조로 접을 수 있습니다. 차 접촉 형성에 관한 정보는 RNA 분자의 기능을 이해하는 것이 필수적입니다. 히드록실 래디 칼 (• OH)가 높은 반응과 작은 크기로 인해 핵산의 구조 독특한 프로브입니다. 1 footprinting 프로브로 사용하면, 히드록실 래디 칼과 함께 DNA 1과 RNA 2의 phosphodiester 백본의 용매 접근 표면을지도 단일 뉴클레오 티드 해상도만큼 괜찮아요. 히드록실 라디칼 footprinting는 DNA - 단백질 1과 RNA - 단백질 단지의 예 분자간 접촉면 내에 세포핵을 식별하는 데 사용할 수 있습니다. 평형 3 번과 4 번 전환 운동은 soluti의 함수로 수산기 라디칼 footprinting을 실시하여 확인할 수 있습니다에서 변수 또는 시간을 각각. footprinting의 주요 기능은 프로브에게 노출 제한 (예 : '싱글 히트 속도론') 폴리머의 각 염기의 유니폼을 샘플링 결과입니다 5.
이 비디오 문서에서, 우리는 RNA 샘플의 준비와 MG (II) - 중재 접는 등온선의 결정을 설명하기 Tetrahymena ribozyme의 P4 - P6 도메인을 사용합니다. 우리는 H 2 O 2 (우리가이 'peroxidative'프로토콜 호출)와 자연스럽게 녹아 O 2를 사용하는 귀중한하지만, 널리 알려진 아니라, 대안을 (우리가 부르는 '가 필요합니다 잘 알려진 수산기 라디칼 footprinting 프로토콜의 사용을 설명 산화 '프로토콜). 데이터 감소, 변환 및 분석 절차의 개요가 제공됩니다.
프로토콜
1. Footprinting의 시약의 준비
- 100 MM 나트륨 cacodylate, 1 ㎜ EDTA (에틸렌 다이아 민 테트라 초산), 1 M KCl을 포함 10X 반응 버퍼를 준비합니다. 7.4로 산도를 조정합니다. 0.2 μm의 아세테이트 필터 장치 (Nalgene)를 사용하여 버퍼를 필터링합니다. 비고가 : 10X 버퍼에 직접 pipet의 RNA를하지 않습니다.
- 로 표 1에 표시된 각 반응에 대한 적정 반응 믹스를 준비합니다. 적정 믹스의 볼륨 (원하는 농도에 1X 버퍼와 MG (II)는) 1X 버퍼에서 RNA의 10μl를 추가하기 전에, 90 μl해야합니다.
- 6.63M 요소, 20mM 나트륨 구연산, EDTA (에틸렌 다이아 민 테트라 초산) 1mM, 0.25 μg / μl tRNA, 0.025 %의 크실렌의 cyanol, 그리고 0.025 %의 bromphenol 블루를 포함하는 RNAse T1의 소화 버퍼를 준비합니다. 이 버퍼는 최대 6 개월까지 4 ° C에 저장할 수 있습니다.
- 펜톤 반응은 250 MM 철 (NH 4) 2 (SO 4) 2, 250 MM EDTA (에틸렌 다이아 민 테트라 초산), 500 MM 나트륨 L - 아스코 브 산의 신선한 수성 솔루션을 필요로합니다. 농도는 제 1에 명시된0.5은 래디 칼의 다양한 이가의 이온 농도를 청소하지 않는 등 나트륨 cacodylate로 버퍼에 평형 실험에 최적입니다.
- peroxidative (1.5.1)과 산화 (1.5.2) footprinting 반응에 대한 시약.
- peroxidative 펜톤 반응은 100 MM 철 - EDTA (에틸렌 다이아 민 테트라 초산) 솔루션, 50 MM 나트륨 L - 아스코 브 산 및 1.5 % H 2 O 2의 신선한 수성 솔루션을 필요로합니다. 철 - EDTA (에틸렌 다이아 민 테트라 초산) 솔루션은 철분 이상의 EDTA (에틸렌 다이아 민 테트라 초산) 1.1 배 초과를 가질 EDTA (에틸렌 다이아 민 테트라 초산)과 혼합 철 (NH 4) 2 (SO 4) 2 준비가되어 있습니다.
- 그냥 산화 절단 반응 (3.4.2)를 시작하기 전에 μl 철 2 μl (NH 4) 2 (SO 4) 2 (250 ㎜), EDTA (에틸렌 다이아 민 테트라 초산) 2.2 μl (250 ㎜), 62.5를 결합하여 펜톤 반응 믹스를 만들 나트륨 L - 아스코 브 산 (500 ㎜), 22.5 μl 반응 10X 버퍼와 0 footprinting 반응 혼합물 (3.4.2)에 최종 농도에 도달하는 H 2 O의 135.5 μl.10 MM, 0.11 MM, 그리고 6.61 MM, 각각.
2. Footprinting 실험을위한 RNA 준비
- 짧은 50 세포핵 (DNA 통합 기술, www.idtdna.com에서 예) 구입할 수있다면 RNA는 DNA 템플릿 6 시험 관내 전사의 표준에 의해 생산 또는 수 있습니다. footprinting 실험 중에 RNA의 샘플 무결성을 유지하는 것이 좋습니다 실험실 청결 절차를 따르십시오. 7
- 체외 베꼈는데 RNA에의 Dephosphorylation 8 이전 RNA의 5 '끝에 32 P 라벨이 필요합니다. RNA 10 pmol는 kinasing에 의해 최종 분류됩니다. 9 솔루션은 인산 가수 분해 효소의 불활 성화 성공 후 뿌연한다.
- denaturing 겔 전기 영동을 사용하여 radioactively 레이블 RNA 정화. RNA는 0.3 M의 아세트산 나트륨 (산도 5.5) 사용하여 두 후속 젤 extractions하여 복구할 수 있습니다. RNA는 1 ML의 에탄올과 이후로 0.5 ML 수용액을 혼합하여 시켰던 것입니다드라이 아이스 1 분간 인큐베이션. 4 ° C, 0.5 시간을 위해 12,500 RPM에서 샘플을 돌린다. 뜨는을 취소하고 70 % 에탄올로 RNA 펠렛을 씻는다. 추가 원심 분리 후 뜨는 제거하고 진공에서 펠렛을 건조.
- 반응 관의 RNA 솔루션, 에탄올과 진공 건조 RNA와 침전물의 1X Buffer.Pipet 30 μl의 330 UL에서 32 P - 표시 RNA 샘플을 분해, 참조 사다리 (단계 3.5) 생성이 튜브를 유지. 95에서 가열하여 버퍼 RNA 솔루션 ° 2 분 C를 변성. 15 분 상온에서 샘플을 냉정하고 다시 솔루션에 튜브의 뚜껑에 condensates을 가지고 빠른 스핀을 제공합니다.
3. Footprinting 실험
- 전기 장식은 분석 젤 당 데이터 요소의 최대 개수를 결정합니다. 우리는 30 잘 빗을 사용합니다. 참조 사다리 2 우물과 uncleaved 컨트롤 하나를 가득 메우고 있습니다. 27 반응 튜브를 준비합니다. 최종 MG (II) concentrati기능은 고르게 적정의 midpoint 주위 규모의 주문을 걸쳐 로그 규모 간격해야합니다.
- 별도로, 5 분 50 RNA와 MG (II) 솔루션 ° C를 품어. 100 μl의 최종 볼륨을 도달 1X 버퍼에 MG의 해당 금액 (II) 90 μl와 RNA 용액 10 μl를 섞는다. 50 30 분 솔루션을 품어 ° C.
- 접는가 발생하는 동안 솔루션은 25 ° C 1 시간에 대한 평형하자.
- Peroxidative (3.4.1) 또는 산화 (3.4.2) 수산기 라디칼 footprinting 반응.
- 2 μl 철 - EDTA (에틸렌 다이아 민 테트라 초산) (100 ㎜)의 방울을 배치하여 peroxidative 반응을 시작, 2 μl 나트륨 L - 아스코 브 산 (50 ㎜) 2 μl H 2 O 2 (1.5 %)는 반응의 맨 안쪽에 서로 구분 튜브는 RNA 솔루션을 포함하고 활발한 혼합하여 footprinting 반응을 시작합니다. 300 μl 감기 절대 에탄올을 추가로 약 15 초 후에 반응을 중지합니다. 튜브에게 3-5 번 돌립니다. 침전, 세척 및2.4 단계로 펠렛을 건조.
- 신선한 펜톤 반응 믹스 (1.6) 5 μl을 추가하여 산화 히드록실 급진 footprintingreaction를 시작합니다. 25 30 분 품어 ° C. 튜브에게 3-5 번 회전하여 반응과 혼합을 담금질하기 위해 추운 절대 에탄올 300 μl를 추가합니다. 침전, 세척 및 펠렛대로 단계 2.4 표시된 건조.
- 단계 1.3 준비 솔루션을 사용하여 표준 절차에 따라 10 RNase T1 다이제스트 참조 예제를 생성합니다.
- 8 μl 젤로드 염료 II (AMBION)와 확인 RNA에서 RNA의 알약을 분해은 가이거 계수기를 사용하여 resuspended 있습니다.
- 11.로드 샘플이 참조 및 제어를 포함하여 표준 프로토콜에 따라 denaturing, 8 % polyacrylamide의 순서 젤을 준비합니다. 60 W에서 별도의 RNA 조각 - 2.5 시간에 대해 75 W. 하룻밤 저장 형광체 화면으로 건조 겔을 쉽게받을 수 있습니다. filmless autoradiography 예위한 이미징 시스템 스캔 형광체 화면. 폭풍 865 (GE 헬스케어) 또는 태풍 (GE 헬스케어). 컴퓨터에 겔 이미지 파일을 전송합니다.
4. 데이터 분석
- 각 염기의 수산기 라디칼 반응은 분석 젤의 각 밴드의 강도를 quantifying에 의해 결정됩니다. 다운로드, 설치 및 오픈 사파, 단일 밴드 피팅 및 부량을위한 오픈 소스 소프트웨어를 사용하기 편리합니다. 12, 13 사용 설명서의 지침을 따르십시오. 간단히 같은 RNA 시퀀스를로드합니다. txt 파일은. 겔 파일로 겔 사진으로 따라갔다. 밴드 농도를 조절하고, 차선을 정의 앵커 차선을 선택하고 젤 정렬을 수행합니다. 세포핵에 밴드의 임무는 RNase T1의 소화 사다리를 참조에서 발생합니다. 밴드 integrals를 정할 가능성이 불변량 잔류물을 정상화하고 할당 정상화 / colorplot 기능을 사용합니다. . txt 파일과 같은 출력을 저장합니다.
- 사파가에서 대표 겔에 골목길과 행을 대표하는 열을 포함하는 스프레드 시트를 출력RNA의 조각에 해당하는 각각의 밴드의 밴드 밀도를 tegrated. 첫째, 끝점 MG (II) 농도 (예제 50 ㎜)의 프로필과 함께 MG (II) 샘플 노에서 파생 보호 프로파일을 비교하여 용매 접근에 눈에 띄는 변화를 표시 보호 사이트를 식별합니다. 낮은 값을, 더 뉴클레오 티드가 히드록실 래디 칼과 반대의 공격으로부터 보호합니다.
- 개인 또는 MG (II) 농도 간의 세포핵의 그룹의 표준 대역 integrals에서 접는 등온선을 만듭니다. •을 - 등온선은 개별적 - Fi 인터넷 접속 = L + (L U)에 의해 부분 채도로 결정됩니다
 F는 밴드의 통합 밀도 (들)을 나타냅니다 어디에 분석하고, L과 U는 전환 아래쪽과 위쪽 한계를 나타내며
F는 밴드의 통합 밀도 (들)을 나타냅니다 어디에 분석하고, L과 U는 전환 아래쪽과 위쪽 한계를 나타내며 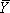 분수 채도입니다. 14 데이터가 적합한 비선형 리터를 사용하는힐 방정식 (1) 동쪽 평방 분석 프로그램 (우리도 마찬가지 유래 (OriginLab) 또는 GraphPad (GraphPad 소프트웨어, 주식 회사)를 사용합니다.)
분수 채도입니다. 14 데이터가 적합한 비선형 리터를 사용하는힐 방정식 (1) 동쪽 평방 분석 프로그램 (우리도 마찬가지 유래 (OriginLab) 또는 GraphPad (GraphPad 소프트웨어, 주식 회사)를 사용합니다.) 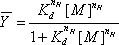
K D의 평형이 어디 해리 상수, [M] 접는 반응을 mediates 변수,이 예제에서 MG (II)의 농도에 해당하고, N H는 힐 계수이다. 이 절차는 비늘 부분 포화로의 전환은 그 midpoint을 결정하고 전환이 sigmoidal 여부의 현상 학적 테스트를 제공합니다. 사파 (그림 3B)에 의해 분석하고 위에 설명된대로 맞는 순서 젤 (그림 3A)에서 파생된 등온선은 그림 3C에 표시됩니다. 그림 3에 표시된 부분 포화 등온선은 스프레드 시트에서 스케일링에 의해 생성되었다 방정식 분율을 사용하여 최고 맞게 위턱과 아래턱에 한계 (U와 L)에 대한 사파에 의해 생성된 값을 = 값 / (U - L) - L / (U - L).
5. 대표 결과 :
그림 3은 P4 - P6 RNA • OH의 footprinting 실험에서 담당자 결과를 보여줍니다. 겔 이미지 (왼쪽) 단일 염기 과제로 인해 T1 차선 (각 G의 RNase T1의 앞을)에서 잘 정의된 밴드 가능합니다)) 배경 절단은 (오른쪽 차선) B 최소한의 것을 나타냅니다하고, C) 수산기 라디칼 RNA의 유도 조각은 잘 배경 위에있다. 높은 MG (II)로 낮은에서 전환은 개인 및 RNA 차 구조의 형성을 나타내는 밴드 그룹의 통합 밴드 밀도를 감소와 연관되어있다. 단일 또는 그의 절단 변경 concomitantly '보호'라고 인접한 뉴클레오 티드 그룹. 밀접한의 결정 구조에서 관찰 분자의 용매 액세스할 지역에 해당하는 MG (II)의 특징 • OH 보호는 P4 - P6 RNA의 접는을 중재. 15
접힌 RNA 분자 C 분명 구조적 또는 동적 현상과 상관되지 않은 차 연락 보호 매우 다른 extents (즉, 어떻게 배경 밴드 밀도 감소를 닫습니다)가. 따라서 일부 RNA 분자는 그림 3과 같이, 잘 정의된 구조 전환을 표시되며, 몇몇은하지 않습니다. 밴드 농도는 사파 12 분석 (그림 3B)에 의해 계량 및 표준입니다. 출력 • OH에 대한 보호의 상대 정도를 시각화 '열'플롯이다. 컬러 전환은 MG (II) 또한시 접근의 변화에 대해 설명합니다. 각각 빨간색 또는 파란색 보여줍 더 접근 이상의 세포핵 보호에 화이트. 음영의 각 학위는 보호 곡선 (그림 3C)로 꾸몄다과 힐 방정식 (1)과 같은 바인딩 모델을 분석할 수 수치와 연관되어있다. 지속적인 분리가 제한 153-155 부속 평형 보호 163-164의 해당 가치의 약 두배입니다.
ontent ">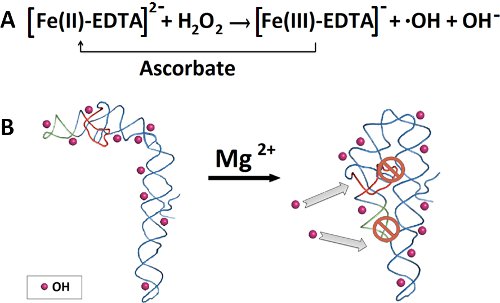
그림 1. 수산기 라디칼 생산과 P4 - P6 RNA를 접는. A) 철 (II)는 과산화수소의 히드록실 래디 칼의 생성을 catalyzes. 아스코 브 산이 함유 철에 철 (III) 다시 줄어 듭니다. B) MG (II)의 부재에서 더 P4 - P6 차 구조는 히드록실 래디 칼은 모든 접근 백본 위치를 침투하고 다니엘 있도록 형성되지 않습니다. MG의 추가 (II)는 오직 용매 접근 백본은 히드록실 래디 칼에 의해 죽습 수 있도록, P4 - P6의 폴딩 시작합니다.
그림 2. 히드록실 급진적인 footprinting 실험에 대한 개요. A) Dephophorylation와 RNA의 32 P 5' 엔드 라벨. B) 32 정제는 denaturing polyacrylamide 젤에서 RNA를 P - 레이블. C) RNA 밴드의 절단, 후속 RNA 추출, 에탄올 침전. D) Prefolding과RNA의 접는. E) 갓 준비 펜톤 반응 혼합물의 추가는 히드록실 래디 칼을 생성합니다. polyacrylamide 겔 전기 영동을 denaturing하여 F) RNA 조각 분리. 사파 소프트웨어에 의해 RNA 조각의 G) Quantitation.
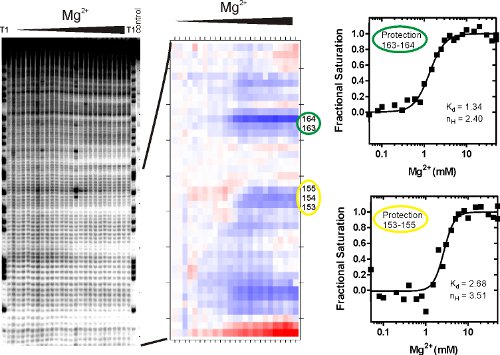
그림 3. peroxidative 펜톤의 footprinting 반응 후 RNA 조각의 분석. (A) RNA는 히드록실 래디 칼에 노출되었고 절단 제품은 denaturing polyacrylamide 겔 전기 영동 (PAGE)를 사용하여 해결이되었습니다. 사진은 MG의 농도 증가 (II)와 peroxidative 절단 제품을 보여줍니다. 참조 및 제어 골목길이 표시됩니다. 이 이미지 파일은 각각의 밴드의 볼륨을 quantitates 사파 프로그램에 입력됩니다. (B) '열'줄거리는 사파에 의해 생성된. (C) 사파에서 염기 번호를 출력 통합 밴드 밀도와 관련된 가치의 스프레드 시트는 같은 힐 equati으로 바인딩 모델로 분석됩니다에 (1). 보호 지역은 MG (II) 농도의 함수로 밴드 통합 밀도의 증가에 따라 선정되었습니다. K D는 RNA의 절반이 모니터링되고있는 사이트에 접혀있는의 MG (II) 농도입니다. 힐 계수, N H은 구속력 cooperativity에 대한 정보를 제공하고이 특정 사이트의 접이식에 관련된 마그네슘 이온의 개수에 대한 낮은 견적을 제공 곡선의 기울기의 측정이다.
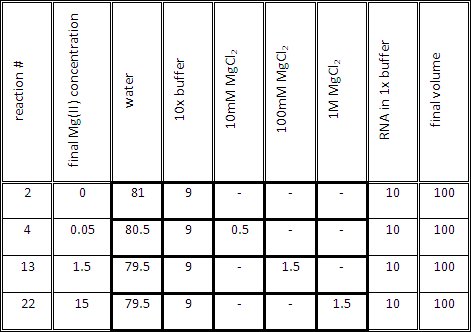
표 1. 다른 MG (II) 농도를 포함하는 RNA 샘플을 생성하는 대표적인 볼륨.
토론
히드록실 급진 footprinting는 핵산의 용매 접근 표면적을 평가하는 유용한 도구입니다. 차 구조 14 질적 및 양적 형성은 이러한 이온 종류와 농도, 산도, 온도, 바인딩 단백질이나 접는 공동 요인으로 매개 변수의 함수로 다음 수 있습니다. 정직하고 저렴한 프로토콜과 그 결과 용매 접근성 및 단일 뉴클레오 티드 수준에서 접는 정보의 강력한 조합이 방법은 매우 매력적합니다. 전통적 • OH?...
공개
관심 없음 충돌 선언하지 않습니다.
감사의 말
이 작품은 건강 RO1 - GM085130 국립 연구소와 국립 과학 재단 (National Science Foundation) MCB0929394에서 보조금에 의해 지원되었다. 우리는 그녀의 환대와 그녀의 실험실에서 영화 우리에게 허용에 대한 박사 메리언 슈미트 감사합니다.
자료
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
참고문헌
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유