È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Singolo ovociti Mutagenesi bisolfito
In questo articolo
Riepilogo
Mutagenesi bisolfito è il gold standard per l'analisi della metilazione del DNA. Il nostro protocollo modificato per l'analisi consente di metilazione del DNA al livello di singola cellula ed è stato specificamente progettato per i singoli ovociti. Può anche essere utilizzato per il clivaggio stadio embrioni.
Abstract
L'epigenetica comprende tutte ereditarie e le modifiche reversibili alla cromatina che l'accessibilità alter gene, e quindi sono i principali meccanismi per la regolazione della trascrizione genica 1. Metilazione del DNA è una modificazione epigenetica che agisce prevalentemente come un segno repressivo. Attraverso l'aggiunta covalente di un gruppo metilico su citosine in dinucleotidi CpG, può assumere altre proteine repressivi e modifiche istone avviare processi di condensazione della cromatina e il silenziamento genico 2. Metilazione del DNA è essenziale per il normale sviluppo in quanto svolge un ruolo fondamentale nella programmazione dello sviluppo, differenziamento cellulare, la repressione degli elementi retrovirali, inattivazione del cromosoma X e imprinting genomico.
Uno dei metodi più potenti per l'analisi della metilazione del DNA è mutagenesi bisolfito. Bisolfito di sodio è un mutageno DNA che deamina cytosines in uracils. Dopo l'amplificazione PCR e seque ncing, questi eventi di conversione vengono rilevati come thymines. Citosine metilate sono protetti da deaminazione e quindi rimangono come citosine, che consente l'identificazione di metilazione del DNA a livello di singolo nucleotide 3. Sviluppo del saggio mutagenesi bisolfito è avanzato da quelli originariamente riportati 4-6 verso quelli che sono più sensibili e riproducibile 7. Un progresso fondamentale è stato l'incorporamento piccole quantità di DNA in un cordone di agarosio, proteggendo in tal modo il DNA dal trattamento bisolfito duro 8. Questa analisi di metilazione in grado di essere eseguita su pool di ovociti e di embrioni allo stadio di blastocisti 9. Il più sofisticato protocollo di mutagenesi bisolfito ad oggi è per i singoli embrioni allo stadio di blastocisti 10. Tuttavia, poiché blastocisti hanno in media 64 celle (contenente 120-720 pg di DNA genomico), questo metodo non è efficace per gli studi sulla metilazione ovociti individuali o clivaggio stadi embrioni. tenda "> Assunzione di indizi da incorporare agarosio di somme di DNA minuto tra cui 11 ovociti, qui vi presentiamo un metodo per cui gli ovociti vengono direttamente incorporati in una soluzione di agarosio e lisi cordone immediatamente dopo il recupero e la rimozione della zona pellucida dell'ovocita dal. Questo ci permette di bypassare le due sfide principali di mutagenesi singolo ovocita: bisolfito. proteggere una piccola quantità di DNA dalla degradazione, e conseguente perdita durante le fasi di protocollo numerosi importante, poiché i dati sono ottenuti da ovociti singoli, il problema del bias PCR in pool Inoltre viene eliminato. , contaminazione accidentale cella cumulo rilevabili mediante questo metodo poiché ogni campione con più di un pattern di metilazione può essere esclusi dall'analisi 12. Questo protocollo fornisce un metodo migliorato per analisi di successo e riproducibile di metilazione del DNA a livello di singola cellula ed è ideale ovociti per i singoli così come scissione stadio embrioni.
Protocollo
1 ° GIORNO
Preparare le seguenti soluzioni fresche del giorno di raccolta degli ovociti con sterile, acqua distillata come l'acqua GIBCO. Per ridurre la possibilità di contaminazione del DNA, cambiare i guanti spesso e usare puntali con filtro. Tenere tubi angolato lontano quando aperto, e richiuderli tutte le provette quando non in uso. Si raccomanda che le soluzioni sono state rilasciate n +1.
3% agarosio LMP
Punto di Fusione 30 mg Low (LMP) Agarose
fino a 1 ml GIBCO H 2 O
sciogliere a 70 ° C
Soluzione di Lisi
8 pl di tampone di lisi
1 ml proteinasi K
1 pl Igepal 10%
posto su ghiaccio fino al momento dell'uso
02:01 agarosio: Lysis Solution (10 microlitri per ogni singolo ovocita, l'importo è di 3 ovociti)
20 pl 3% agarosio LMP
10 Soluzione Lysis pl
mescolare @ 70 ° C
SDS Lysis Buffer (501 pl per singolo ovocita)
| 1x TE pH 7,5 | 450 microlitri |
| 10% SDS | 50 microlitri |
| Proteinasi K | 1 pl |
| 501 microlitri |
1. Ovociti Collection
- Posizionare i ovidotti del mouse sezionati nei media M2, e strappare le ampolle per estrarre il complesso di celle cumulus.
- Separare gli ovociti dal complesso cella cumulo utilizzando 0,3 mg / ml soluzione ialuronidasi una goccia in 30 pl di supporto M2. Conservare gli ovociti in unica soluzione il tempo necessario per rimuovere le cellule del cumulo, in quanto l'esposizione prolungata può danneggiarli. Lavare gli ovociti 3x goccia in 30 microlitri di mezzi di M2, la rimozione di cellule del cumulo periodicamente.
- Rimuovere la zona pellucida con soluzione acida Tyrode. Posizionare gli ovociti in una goccia 30 ml di soluzione, e poi trasferisca ad un altro 30goccia pl, come i supporti realizzati lungo diluire l'acido e ridurre la sua efficacia. Conservare gli ovociti in unica soluzione il tempo necessario per rimuovere la zona, come l'esposizione prolungata può danneggiarli. Nota: una maggiore concentrazione di soluzione acida di Tyrode o pronasi possono essere utilizzati per campioni umani, come la zona pellucida umano è più resistente al trattamento con la soluzione acida Tyrode che il mouse.
- Lavare gli ovociti ancora una volta in un calo del 30 microlitri di supporto M2.
2. Agarose Embedding e Lisi
- Per eseguire l'incorporamento di agarosio, posizionare la soluzione di lisi su un 70 ° C heatblock. Aggiungere l'agarosio LMP preriscaldato alla soluzione di lisi, producendo un 2:1 agarosio: soluzione di lisi.
- Posizionare un singolo ovocita su un vetrino pulito in minima dei media M2. Raccogliere 10 pl di agarosio: soluzione di lisi in una punta di pipetta, e (sotto un microscopio) delicatamente espellere una piccola quantità (~ 1 pl o meno) sul vetrino, permettendogli di miscelare con la minimaal media. Delicatamente sollevare l'ovocita nella punta della pipetta e mettere tutte le 10 pl in una provetta Eppendorf olio minerale con 300 microlitri in modo che il tallone forma una sfera.
Nota: questo processo deve essere fatto abbastanza velocemente, come l'agarosio si indurisce se la temperatura scende un minimo di 5 ° C inferiore a 70 ° C. - Incubare la provetta in ghiaccio per 10 minuti. Per procedere alla lisi, rimuovere l'olio minerale 300 microlitri e aggiungere 500 microlitri di tampone di lisi SDS. Incubare per una notte in un bagnomaria a 50 ° C.
Nota: soluzione di lisi (Tabella 1) possono anche essere utilizzati per questo scopo.
2 ° GIORNO
Preparare le seguenti soluzioni nuove il giorno di mutagenesi bisolfito. Per ridurre le possibilità di contaminazione del DNA, cambiare i guanti spesso e usare puntali con filtro. Tenere tubi angolato lontano quando aperto, e richiuderli tutte le provette quando non in uso. Si raccomanda che le soluzioni sono state rilasciate n +1.
| 3 M NaOH | 2,4 g di NaOH in 20 ml autoclavato ddh 2 O |
| 0,1 M NaOH | 0,5 ml di 3M in 14,5 ml autoclavato ddh 2 O |
| 0,3 M NaOH | 1,5 ml di 3M in 13,5 ml autoclavato ddh 2 O |
2,5 M soluzione di bisolfito
- 3,8 g di sodio bisolfito
5,5 ml GIBCO distillata H 2 O
1 ml 3 M NaOH
sciogliere @ temperatura ambiente - 110 mg Idrochinone
1 ml GIBCO distillata H 2 O
sciogliere @ 90 ° C (solo per il tempo necessario per sciogliere, mescolare regolarmente)
Quando è completamente dissolto, miscelare la soluzione (a) e (b)
* Tenere lontano dalla luce *
3. Bisolfito di Mutagenesi
- Completamente rimuovere il 500 pl tampone di lisi SDS e aggiungere 300 microlitri di olio minerale (~ 20 ore). Qualsiasi tampone di lisi rimanente diluire thagarosio e quando viene riscaldato e il tallone sarà più suscettibile a dissolvere nei passaggi successivi. Procedere con la mutagenesi bisolfito immediatamente, o conservare a -20 ° C per un massimo di 5 giorni.
- Se necessario, rimuovere gli ovociti dal freezer e lasciare scongelare (solo fino al tallone agarosio è relativamente trasparente). Incubare per 2,5 minuti a 90 ° C con blocco termico, dopo che incubare in ghiaccio per 10 minuti.
Nota: Non mescolare o agitare, estendere più di 2,5 minuti, o fluttuare temperatura. - Per eseguire la denaturazione, rimuovere l'olio minerale e aggiungere 1 ml di NaOH 0,1 M ad ogni flick tubo, e capovolgere 5-6 volte.
- Incubare per 15 minuti a 37 ° C a bagno-maria, invertendo ogni 3-4 minuti. Il tallone dovrebbe galleggiare in NaOH.
- Per eseguire il trattamento bisolfito, girare delicatamente il tubo, quindi rimuovere il NaOH e aggiungere 300 microlitri di olio minerale e 500 microlitri soluzione di bisolfito. Incubare la provetta per 3,5 ore a 50 ° C in bagno maria. * Tenere lontano dalla luce *
Nota: Lunghezza di incubazione può essere necessario essere determinata empiricamente per gene di interesse. - Per eseguire desulfonation, incubare in ghiaccio per 3 minuti, quindi rimuovere l'olio minerale e la soluzione di bisolfito, girare delicatamente e aggiungere 1 ml 0.3 M NaOH. Flick e capovolgere 5-6 volte.
- Incubare per 15 minuti a 37 ° C a bagno-maria, invertendo ogni 3-4 minuti. Il tallone dovrebbe galleggiare in NaOH.
- Lavare i campioni, in primo luogo girare delicatamente, quindi rimuovere il NaOH e aggiungere 1 ml di TE 1x pH 7.5. Agitare per 5-10 minuti a temperatura ambiente (su un agitatore). Spin dolcemente di nuovo, quindi rimuovere il TE 1x. Ripetere questo processo di lavaggio due volte.
- Aggiungere 1 ml autoclavato ddh 2 O. Agitare per 5-10 minuti a temperatura ambiente (su un agitatore). Spin delicatamente, quindi rimuovere la H 2 O. Ripetere ddh 2 O lavaggio due volte.
- Controllare il pH del surnatante, che dovrebbe avere un pH 5,0. Se ancora troppo semplice, sciacquare con H 2 O. Rimuovere tutti i surnatante, lasciando solo l'agarosio siaannuncio.
4. 1 ° e 2 ° Turno di amplificazione
- Preparare 1 turno PCR ** mix mentre ci si lava **
| Primer 10 pM Forward esterno | 0,5 pl |
| 10 Primer Reverse Outer pM | 0,5 pl |
| 240 ng / ml tRNA | 1 pl |
| H 2 O | 13 microlitri |
Aggiungi ai Illustra Ready-to-Go Hot Start PCR perline
Far scorrere delicatamente il solido agarosio tallone nel tubo PCR (~ 10 pl)
Riscaldare a 70 ° C e mescolare
Aggiungere 25 microlitri di olio minerale
Totale: 50 pl
- Amplificare
Nota: Un esempio di condizioni di ciclo per mouse SNRPN è denaturazione per 2 minuti a 94 ° C, seguiti da 40 cicli di 30 secondi a 94 ° C, 1 minuto a 50 ° C, e 1minuto a 68 ° C, e una fase di allungamento finale 10 minuti a 68 ° C. Ricottura temperatura per 1 turno H19 PCR per il mouse e Peg3 è di 50 ° C. - Preparare 2 ° turno PCR mix
| Primer 10 pM Forward interno | 0,5 pl |
| 10 Primer Reverse pM interna | 0,5 pl |
| H 2 O | 19 microlitri |
Aggiungi ai Illustra Ready-to-Go Hot Start PCR perline
Aggiungere 5 ul 1 prodotto turno come modello. Riscaldare il prodotto 1 turno a 70 ° C per 1 minuto per ammorbidire l'agarosio. Assicurati di pipettare di sotto dello strato di olio minerale.
Aggiungere 25 microlitri di olio minerale
Totale: 50 pl
Nota: sequenze di innesco nidificate per SNRPN, H19, e Peg3 può essere trovato in Market-sentenza Velker et al 10,12.
- Amplificare
Nota: condizioni di ciclo per mouse SNRPN è denaturazione per 2 minuti a 94 ° C, seguiti da 40 cicli di 30 secondi a 94 ° C, 1 minuto a 50 ° C, e 1 minuto a 68 ° C, e un allungamento finale 10 minuti passaggio a 68 ° C 10. Mouse H19 e Peg3 richiedono a 50 ° C temperatura di ricottura per 2 ° turno PCR. - Come test diagnostico, secondo i campioni circolari può essere tagliato con un enzima di restrizione che è metilazione-specifica o ceppo.
| 2 ° Turno del prodotto | 4 microlitri |
| Enzima di restrizione | 1 pl |
| Buffer | 1 pl |
| H 2 O | 4 microlitri |
- I prodotti della digestione elettroforesi su un gel di acrilammide 8%. Tutte le fasce eterogenee rappresentano più di un sequeNCE.
5. TA clonazione e PCR Colony
- Per clonare 2 ° prodotto tondo, primo calore a 70 ° C per 1 minuto per ammorbidire l'agarosio, poi legare nel vettore utilizzando il pGEM-T Vector Promega Sistema (Fisher Scientific Cat # A1360).
| 2 ° Turno PCR | 1 pl |
| pGEMT-EASY vettore | 1 pl |
| Ligasi | 1 pl |
| H 2 O | 2 microlitri |
| Legatura Buffer 2x | 5 pl |
Incubare @ 4 ° C in macchina PCR.
- Scongelare le cellule competenti di E.coli in ghiaccio per 15 minuti (Zymo Research Corp Cat # T3009). Aggiungere 3 reazione ul legatura a 8 pl E.coli e ligazione incubare in ghiaccio per 15 minuti.
- Shock termico per 40 secondi in un bagno d'acqua 42 ° C,e incubare su ghiaccio per 2 minuti. Aggiungere 60 microlitri mezzo SOC e incubare a 37 ° C per 1 ora (in shaker).
- Mettere tutta la miscela di reazione su un LB / Agar / IPTG / Xgal / Amp piastra e piastra incubare a 37 ° C per una notte.
- Preparare colonia mix PCR
| 20 pM M13 Forward Primer | 0,7 pl |
| 20 pM M13 Reverse Primer | 0,7 pl |
| 5X Green Go Taq Buffer | 7,0 pl |
| 10 mM dNTP | 0,7 pl |
| Taq DNA polimerasi | 0,28 pl |
| H 2 O | 25,62 pl |
| 35 Totale pl |
Aggiungere 35 microlitri mix PCR Colony maestro in una provetta PCR. Scegli una colonia bianca batterica dalla piastra con una pipetta, e turbinare esso nella reazione PCR.
- Amplificazione con denaturazione per 10 minuti a 94 ° C, seguita da 30 cicli di 45 secondi a 94 ° C, 30 secondi a 57 ° C, e 1 minuto a 72 ° C, ed una fase finale di allungamento 10 minuti a 72 ° C. 4 microlitri elettroforesi su un gel di agarosio 1,5%. Invia ~ 30 pl del prodotto PCR per il sequenziamento.
Nota: Per ovociti, 5 colonie prodotti di PCR vengono sequenziati. - Una volta che i risultati si ottengono sequencing, pattern di metilazione può essere letto. Qualsiasi CG originale che è rimasto come un CG è stato metilato, e qualsiasi CG originale che ora è un TG è stato non metilato.
6. Risultati rappresentativi
Nel nostro lavoro, abbiamo impresso test di metilazione in singoli ovociti ed embrioni (Figura 1). Dopo amplificazione nested PCR usando primer bisolfitici convertiti, è possibile confermare una conversione da visualizzare una dimensione corretta frammento su gel di agarosio (Figura 2). Un oocita individualerappresenta un allele parentale, e in teoria, ha un modello impressa metilazione. Come tale, secondo turno prodotti di PCR può essere testato per la contaminazione accidentale. Un enzima di restrizione sensibili alla metilazione del DNA (ad esempio HinfI o DpnII) può essere utilizzato per digerire il secondo prodotto tondo PCR per valutare se esso contiene un allele metilata o non metilato (Figura 3). Un C metilato nella sequenza di riconoscimento dell'enzima viene clivato mentre un C non metilato che viene convertito a T non è più riconosciuto dall'enzima ed è tagliato. Ogni campione degli ovociti MII contenente sia metilato e non metilato alleli deve essere scartato, come è indicativo di contaminazione cumulo cellulare (Figura 3). Dopo ligazione e trasformazione, successo colonia amplificazione PCR può essere visualizzato su un gel di agarosio per garantire campioni con la dimensione prodotto corretto vengono inviati per sequenziamento (Figura 4). Infine, la sequenza di cinque cl individualequelli di un ovocita MII dovrebbe produrre cinque schemi di metilazione uguali e identici tassi di conversione nonCpG (Figura 5a). Tutti i campioni che contengono più di un modello deve essere scartato (figura 5b). Poiché ovulati ovociti MII hanno due copie cromosomiche o un corpo attaccato polare, vi è la possibilità di ottenere due schemi analoghi di sequenza (Figura 5c). Si consiglia eliminando i dati da ovociti che hanno schemi di metilazione molto dissimili in quanto la contaminazione delle cellule cumulo non si può escludere.

Figura 1. Schematica del singolo test Mutagenesi bisolfito ovociti.
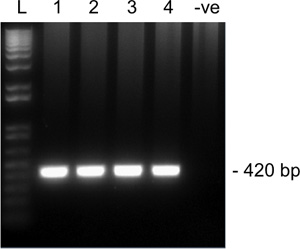
I risultati rappresentativi Figura 2. Dal 2 ° turno di amplificazione per SNRPN da cantareLe ovocita MII su un gel di agarosio 1,5%. Lanes 1-4 sono quattro singoli ovociti MII e corsia 5 è un controllo negativo (nessun ovocita). Dimensioni amplicone previsto per SNRPN è di 420 bp. L, scala.
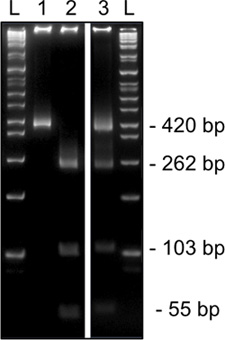
Risultati rappresentativi Figura 3. Da 2 ° turno metilazione-specifica digestione di restrizione per SNRPN da un oocita singolo MII su un gel di acrilammide 8%. Digestione di restrizione HinfI diagnostica mostra DNA non metilato che ospita una T che abolisce il sito di restrizione (420bp, corsia 1) o DNA metilato che contiene un sito di riconoscimento all'interno C (taglio, 262, 103, e 54 bp, corsia 2). La digestione mostrando sia metilati e non metilato siti di enzimi di restrizione (bande di taglio e non tagliato, corsia 3) sono indicativi di contaminazione cumuli cella. L, scala.
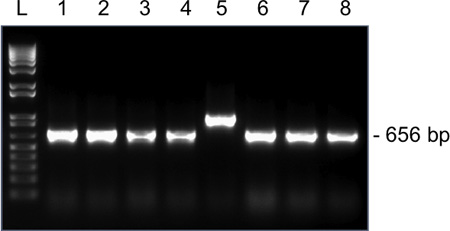
Figura 4. I risultati rappresentativi per l'amplificazione colonia PCR per SNRPN da un singolo ovocita MII su un gel di agarosio 1,5%. Dimensioni amplicone previsto dopo la legatura del SNRPN nel pGEM-T vector facile e l'utilizzo di M13 in avanti e primers inversa è 656 bp. Lane, 1-8, amplificati da cloni 1-8. Clone 5 ha una dimensione errata amplicone e non dovrebbe essere trasmessa per il sequenziamento.

Figura 5. Rappresentante sequenziamento risultati per SNRPN da un singolo ovocita MII. SNRPN è metilato in ovociti. Cerchi neri indicano CpG metilati. Cerchi bianchi indicano CpG non metilato. Il numero e il posizionamento CpG è rappresentativo di un mouse B6 ceppo femminile. a) i risultati attesi per il sequenziamento SNRPN da un singolo ovocita MII. Solo un singolo filamento di DNA dovrebbe amplificare in tutte le cinque cloni. Ovociti con un modello di metilazione sola e identica non-CpG patter di conversionen dovrebbero essere inclusi nelle analisi (conversione percentuale di non-CpG indicato a destra è stata calcolata come il numero di non-CpG citosine convertiti timina come percentuale del totale non-CpG citosine). b) i risultati di sequenziamento per SNRPN da un singolo ovocita MII alla contaminazione cumulus cell. Notare la diversità tra gli stati di metilazione ed i modelli di conversione che indicano l'amplificazione multipla Strand. c) Sequencing risultati per SNRPN da un singolo ovocita MII con entrambe le copie di cromosomi o di inclusione corpo polare.
Discussione
Questo saggio ovocita singolo contiene molti passi con un numero che sono critiche e richiedono particolare attenzione. Il primo è lavaggio ovocita. È particolarmente importante lavare ciascun ovocita più volte in mezzo fresco scende dopo il trattamento ialuronidasi per rimuovere come cellule del cumulo numero possibile. Inoltre, durante il trasferimento di ovociti alla soluzione acida Tyrode per la rimozione della zona pellucida assicurarsi che circonda mezzo è chiaro cellule del cumulo. L'ovocita è molto appi...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalla University of Western Ontario, il Dipartimento di Ostetricia e Ginecologia, e una borsa di ER06-02-188 da Ministryof di ricerca e innovazione, Premio ricercatore alle prime armi. MMD è stata sostenuta da un programma di formazione CIHR in riproduzione, lo sviluppo precoce e l'impatto sulla salute (REDIH) Graduate Scholarship.
Materiali
Tabella di reagenti e attrezzature specifiche.
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| Ovociti Collection | |||
| Ialuronidasi | Sigma | H4272 | |
| Acida Tyrode | Sigma | T1788 | |
| Proteinasi K | Sigma | P5568 | |
| 10% Igepal | Bioshop | NON999.500 | |
| Soluzione di Lisi | |||
| Tris pH 7,5 | Bioshop | TRS001.5 | |
| LiCl | Sigma | L9650 | & Nbsp; |
| EDTA pH 8,0 | Sigma | E5134 | |
| COPERCHI | Bioshop | LDS701.10 | |
| DTT | Invitrogen | P2325 | |
| SDS Lysis Buffer | |||
| TE pH 7,5 | Bioshop (Tris) Sigma (EDTA) | TRS001.5 E5134 | |
| 10% SDS | Bioshop | SDS001.500 | |
| Bisolfito di conversione | |||
| Di sodio idrossido | Sigma | S8045 | |
| Di sodio bisolfito (bisolfito di sodio) | Sigma | 243973 | ; |
| Idrochinone | Sigma | H9003 | |
| Basso punto di fusione (LMP) Agarose | Sigma | A9414 | |
| Olio minerale | Sigma | M8410 | |
| Media M2 | Sigma | M7167 | |
| GIBCO Acqua distillata | Invitrogen | 15230-196 | |
| Autoclavato bidistillata (dd) l'acqua | |||
| PCR | |||
| Illustra Hot Start Mix RTG | GE Healthcare | 28-9006-54 | |
| 240 ng / ml lievito tRNA | Invitrogen | 15401-011 | |
| 5x Verde GoTaq Tampone di reazione | Professionistamega | M7911 | |
| Primers annidati interni ed esterni | Sigma | ||
| Ligation | |||
| Promega pGEM-T Vector Facile | Fisher Scientific | A1360 | |
| TA Cloning | |||
| Competenti E.coli cellule | Zymo Research Corp. | T3009 | |
| Attrezzatura | |||
| Dissezione Microscopio | |||
| 70 ° C e 90 ° C di calore Blocks | |||
| 37 ° C e 50 ° C bagni d'acqua (42 ° C per le trasformazioni) | |||
| Sedia a dondolo | |||
| PCR macchina | |||
Riferimenti
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33, 245-254 (2003).
- Rodenhiser, D., Mann, M. Epigenetics and human disease: translating basic biology into clinical applications. CMAJ. 174, 341-348 (2006).
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827-1831 (1992).
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Feil, R., Charlton, J., Bird, A. P., Walter, J., Reik, W. Methylation analysis on individual chromosomes: improved protocol for bisulphite genomic sequencing. Nucleic Acids Res. 22, 695-696 (1994).
- Raizis, A. M., Schmitt, F., Jost, J. P. A bisulfite method of 5-methylcytosine mapping that minimizes template degradation. Anal. Biochem. 226, 161-166 (1995).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA Methylation: Bisulphite Modification and Analysis. J. Vis. Exp. (56), e3170 (2011).
- Olek, A., Oswald, J., Walter, J. A modified and improved method for bisulphite based cytosine methylation analysis. Nucleic Acids Res. 24, 5064-5066 (1996).
- Mann, M. R. Selective loss of imprinting in the placenta following preimplantation development in culture. Development. 131, 3727-3735 (2004).
- Market-Velker, B. A., Zhang, L., Magri, L. S., Bonvissuto, A. C., Mann, M. R. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose-dependent manner. Hum. Mol. Genet. 19, 36-51 (2010).
- Meng, L. H., Xiao, S. Q., Huang, X. F., Zhou, Y., Xu, B. S. A study on bisulfite sequencing method for methylation status of imprinted genes in single human oocytes. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 25, 289-292 (2008).
- Denomme, M. M., Zhang, L., Mann, M. R. Embryonic imprinting perturbations do not originate from superovulation-induced defects in DNA methylation acquisition. Fertil. Steril. 96, 734-738 (2011).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Hajj, N. E. l. Limiting dilution bisulfite (pyro)sequencing reveals parent-specific methylation patterns in single early mouse embryos and bovine oocytes. Epigenetics. 6, 1176-1188 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon