È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevamento dei microRNA nella microglia da Real-time PCR in CNS normale e durante neuroinfiammazione
In questo articolo
Riepilogo
Microglia risiedono macrofagi che forniscono la prima linea di difesa e sorveglianza immunitaria del sistema nervoso centrale. I microRNA sono molecole regolatrici che svolgono un ruolo importante in molti processi fisiologici tra cui l'attivazione e la differenziazione dei macrofagi. In questo articolo, si descrive il metodo per la misurazione dei microRNA in cellule microgliali.
Abstract
Microglia sono cellule della linea mieloide che risiedono nel sistema nervoso centrale (CNS) 1. Queste cellule hanno un ruolo importante in patologie di molte malattie associate neuroinfiammatoria come la sclerosi multipla (MS) 2. Microglia nel sistema nervoso centrale di un normale esprimere macrofagi CD11b marcatore e mostrano un fenotipo di riposo da esprimere bassi livelli di marker di attivazione quali CD45. Durante gli eventi patologici del sistema nervoso centrale, microglia si attivano come determinato dal upregulation di CD45 e di altri marcatori 3. I fattori che influenzano il fenotipo e le funzioni di microglia nel sistema nervoso centrale non sono ben studiati. I microRNA (miRNA) sono una famiglia crescente di molecole conservate (circa 22 nucleotidi) che sono coinvolte in molti processi fisiologici come la crescita e la differenziazione cellulare 4 e patologie come l'infiammazione 5. MicroRNA downregulate l'espressione di geni bersaglio determinati legame sequenze complementari dimRNA ir e svolgono un ruolo importante nella attivazione di cellule immunitarie innate tra macrofagi e microglia 6 7. Al fine di indagare miRNA-mediate percorsi che definiscono la microglia fenotipo, funzione biologica, e per distinguere microglia da altri tipi di macrofagi, è importante per valutare quantitativamente l'espressione dei microRNA in particolari sottogruppi distinti del SNC residente microglia. Metodi comuni per misurare l'espressione dei miRNA nel SNC includono quantitativa PCR da intero tessuto neuronale e ibridazione in situ. Tuttavia, quantitativa PCR da omogenato tissutale intero non consente la valutazione dell'espressione di miRNA in microglia, che rappresentano solo 5-15% delle cellule del tessuto neuronale. Ibridazione in situ permette di valutare l'espressione di microRNA in specifici tipi cellulari nelle sezioni di tessuto, ma questo metodo non è del tutto quantitativa. In questo rapporto descriviamo una quantitativa e sensitive metodo per la rilevazione di miRNA dal real-time PCR in microglia isolato dal sistema nervoso centrale normali o durante neuroinfiammazione con encefalomielite autoimmune sperimentale (EAE), un modello murino di sclerosi multipla. Il metodo descritto sarà utile per misurare il livello di espressione dei microRNA in cellule microgliali in CNS normali o durante neuroinfiammazione associato a varie patologie tra cui sclerosi multipla, ictus, lesioni traumatiche, morbo di Alzheimer e tumori cerebrali.
Protocollo
1. Isolamento della microglia dal CNS normali
Isolamento di microglia viene eseguita come descritto in precedenza con modifiche 3.
- Preparare strumenti per la dissezione e la perfusione: forbici, pinze, siringa da 10 ml con ago 27g, 15 ml Dounce omogeneizzatore (Teflon / vetro), nylon 40 micron filtro cellulare, il 40% e 70% soluzioni di Percoll, 1X PBS.
- Euthanize topi per asfissia rapido (privazione di ossigeno) in CO 2 fotocamera e immediatamente trasferire banco di laboratorio per le manipolazioni chirurgiche e perfusione.
- Eseguire le manipolazioni chirurgiche (forbici e pinze di utilizzo e il 70% di etanolo come antisettico): aperto cavità peritoneale, tagliare il, cavità toracica e diaframma aperto snip l'atrio destro con le forbici.
- Eseguire corpo intero perfusione intracardial iniettando siringa da 10 ml in ventricolo sinistro e pressione. Passare 8-10 ml di PBS 1X attraverso la circolazione del sangue pa lento e costantece.
- Ripetere i passaggi da 1,2-1,4 per tutti i topi del gruppo di topi (tipicamente 5-10). Dopo la perfusione tenere il mouse su ghiaccio fino a quando tutti i topi sono perfusi.
- Dopo la perfusione di tutti i topi, sezionare il cervello e midolli spinali. (Opzionale: studiare l'espressione dei miRNA nelle diverse aree del cervello, sezionare queste aree specifiche, quali il cervelletto, ecc ippocampo per il gruppo di topi 5-10 e piscina insieme per omogeneizzazione).
- Omogeneizzare cervelli e midolli spinali con 15 ml Dounce omogeneizzatore. Posizionare un cervello e del midollo spinale in uno omogeneizzatore e aggiungere 5 ml di PBS 1X. Eseguire 10-20 scioperi dolci; omogenato passare attraverso il filtro delle cellule in 50 ml-conica tubo e certificato a 2000 giri minuto su una centrifuga grande 5-7.
- Gettare il surnatante, risospendere cervello / omogenato di midollo spinale di topi in 2-3 ml 5-7 del 70% Percoll e overlay 7 ml di Percoll al 40%. Spin a 2000 rpm (no freno!) Per 30 minuti.
- Eliminare le cellule danneggiate / detriti mielina in cima ad una del 70% e raccogliere monocellule nucleari a 40% / 70% di interfaccia. Diluire le cellule raccolte almeno a 1:1 con 1X PBS e centrifugare a 2000 rpm per 5-7 minuti. Risospendere le cellule in 0,5 ml di PBS e trasferirli in provetta 1,7 ml ependorf. Resa tipica è ~ 500 x 10 3 cellule mononucleari di per topo, il che darebbe il numero di cellule microgliali ~ 1x10 6 a 0,5 ml per la preparazione delle cellule dal cervello e midolli spinali di 2-3 topi.
2. Controllo della Purezza della microglia mediante citometria a flusso
Colorazione con anticorpi viene eseguita come descritto in precedenza con modifiche 7,8.
- Preparare tampone FACS (1X PBS con 2% FBS), anticorpi FCR bloccanti, anti-CD11b - PE, anti-CD45-APC-Cy7 anticorpi, 1% paraformaldeide in PBS.
- Prendere 50 microlitri di cellule a partire dal punto 1.9 e trasferirli al nuovo 1.7 ml tubo Eppendorf ed aggiungere 350 microlitri di tampone FACS.
- Aggiungi ul 5-10 di anticorpi anti-FCR e incubare 10-15minuti su ghiaccio.
- Dividere le celle in due provette Eppendorf. Aggiungi ad un tubo di 1 ml di anti-CD11b - PE e 2 pl anti-CD45-APC-Cy7 anticorpi e incubare 10-15 minuti su ghiaccio. Secondo tubo sarà utilizzato come controllo negativo per la colorazione anticorpale. Dopo l'incubazione con anticorpi aggiungere 1 ml di tampone FACS per entrambi i tubi e spin in centrifuga a 5400 rpm piccola per 5 min. Dopo la filatura fissare le cellule risospendendo in 400 pl di 1% paraformaldeide in PBS e vortex.
- Effettuare analisi di citometria di flusso al citofluorimetro (LSR II, BD Biosciences) utilizzando compubeads BD (BD Biosciences) per i controlli di compensazione come è stato descritto 8. Esempi di preparati positivi e negativi sono mostrati nella fig. 1a, b.
3. Isolamento della microglia e macrofagi periferici dal CNS infiammate in modello murino di neuroinfiammazione
Encefalite autoimmune sperimentale (EAE) viene indotta come è stato descritto in precedenzanel nostro documento 7; ordinamento FACS viene eseguita in modo simile al protocollo pubblicato 8.
- Indurre EAE mediante immunizzazione sottocutanea con 150 mg MOG 35-55 peptide in 4 mg / ml di adiuvante completo di Freund (CFA) del gruppo di 5-10 di 8-12-settimane di età topi C57BL / 6. La tossina della pertosse deve essere somministrato ip (150 ng / topo) nei giorni giorno zero e due post-immunizzazione. I topi sono stati osservati segni di malattia a partire dal giorno 10 post-immunizzazione, e la gravità della malattia è stato segnato in una scala numerica 0-5 come segue: 0) nessuna malattia: 1) la coda debole o piedi traballante, 2) paresi degli arti posteriori; 3) la paralisi degli arti posteriori; 4) la paralisi degli arti anteriori e posteriori, e 5) la morte o l'eutanasia per motivi umanitari. I topi ai vertici della malattia (d21 post-immunizzazione) sono utilizzati per sperimentare punteggio tipica malattia da 1,5 a 2,5.
- Isolare le cellule mononucleate del CNS di gruppo di topi 5-10 come nei passi 1.1-1.4 e colorare le cellule con anticorpi anti-CD11b e anti-CD45 come nei passi 2.1-2.4.Nel passo 2,4 non risolvono le cellule, invece risospendere in 400 microlitri di PBS 1X invece di 1% paraformaldeide.
- Selezione delle cellule viene eseguita da FACSAria per le popolazioni di CD11b + CD45 CD11b bassa microglia e cellule CD45 + hi (~ 80% di loro sono macrofagi periferici e ~ 20% di loro vengono attivati microglia 3, in futuro si farà riferimento come queste cellule " macrofagi "). Usare 1,7 ml ependorf tubi per la raccolta dei campioni. 300 pl di 1X PBS con 10% FBS dovrebbe essere aggiunto ai tubi di raccolta per evitare danni delle cellule selezionate. Esempi di tre popolazioni di microglia, macrofagi e linfociti sono mostrati nella fig. 2a con impostazioni appropriate di gate di ordinamento di fig. 2b. Purezze ordinamento tipici erano al 98-100% come determinato da rianalizzati ordinati popolazioni di microglia (Fig. 2c) e macrofagi (Fig. 2d). Tipico è resa 20-50 x10 3 per microglia e50-100 x10 3 per macrofagi isolati dal gruppo di cinque topi.
4. Isolamento di RNA
RNA viene isolato usando il kit Mirvana secondo le istruzioni del fabbricante con le modifiche.
- Spin microglia dal CNS normali a partire dal punto 1.9 o sottoinsiemi ordinati di microglia e macrofagi a partire dal punto 3.3 in 1,7 ml provette Eppendorf a 5400 rpm per 5-7 minuti in centrifuga piccola, con attenzione scartare il surnatante e congelare i pellet cellulari in ghiaccio secco. Passare al punto 4.2 o trasferire i pellet cellulari congelati a -70 C, dove può essere conservato per 2-3 mesi.
- Trasferimento pellet cellulari congelati in ghiaccio regolare per lo scongelamento. Aggiungere 300 ul di tampone di lisi dal kit Mirvana e mescolare delicatamente dal Pipette 5-7 volte.
- Aggiungere 30 microlitri di Additive omogeneizzato da Mirvana kit, vortex 30 secondi per mescolare, tenere in ghiaccio per 10 minuti
- Aggiungere 300 ml di acido-Pehnol: Cloroformio da Mirvana kit, vortex 30 secondi, spin per 5 ma 10.000 giri in centrifuga sulla piccola.
- Estrarre con cura superiore fase acquosa (300 pl) e trasferire in un'altra provetta 1,7 ml Eppendorf, mescolare con 375 microlitri di etanolo al 100%, trasferimento a cartuccia filtrante da Mirvana kit e centrifugare per 1 min a 10.000 rpm.
- Eliminare flow-through tampone, aggiungere 700 ml di soluzione di lavaggio da Mirvana I kit, e centrifugare per 1 min a 10.000 rpm.
- Lavare due volte con 500 microlitri di soluzione di lavaggio II da Mirvana kit e infine eluire l'RNA dalla cartuccia con 60 microlitri di pre-riscaldata di acqua priva di nucleasi.
- La quantità e la qualità di RNA è valutata mediante spettrofotometro NanoDrop. La concentrazione di RNA dovrebbe essere da 5 a 20 ng / ml e OD λ260 / λ280 rapporto nell'intervallo 1,7-2,0. Se la concentrazione di RNA è inferiore a 3 ng / ml e OD 260λ/280λ è inferiore a 1,6, i campioni vengono scartati. I campioni di RNA possono essere conservati a -70 ° C per 6-12 mesi.
- Per ulteriore stima della qualità dell'RNA, AR campionie analizzati mediante elettroforesi su gel 15% in TBE Urea-Gel (Life Technologies). Tipici esempi di buona qualità e di cattiva preparati RNA sono mostrati nella fig. 3a e Fig.3b, rispettivamente.
5. Rilevazione di miRNA in microglia e macrofagi
Per l'analisi dell'espressione miRNA, quantitativa in tempo reale reverse transcriptase PCR (qRT-PCR) analisi è effettuata utilizzando saggi TaqMan con primer e sonde fluorescenti per microRNA particolare interesse e uno degli RNA ubiquitariamente espresse brevi (ad esempio snoRNA-55, snoRNA -135 o U6) per la normalizzazione. Primers e sonde fluorescenti per specifici gruppi di miRNA umano o del mouse può essere acquistato da Life Technologies Inc. Qui useremo miR-124 come un esempio per miRNA di interesse e snoRNA-55 come esempio per la normalizzazione.
- Eseguire la reazione della trascrittasi inversa per fare cDNA da campioni di RNA con kit trascrizione inversa TaqMan:
| Voce | Volume (pl) per campione |
| 5x RT Primer | 1,00 |
| RNA del campione | 1,67 |
| RT Enzyme Mix | |
| 25 mm ciascuno (totale 100 mM) dNTPs | 0,050 |
| MultiScribe trascrittasi inversa) | 0,333 |
| RT Buffer 10X | 0,500 |
| AB inibitore della RNasi | 0,063 |
| Acqua priva di nucleasi | 1,388 |
| Totale | 5,00 |
Diluire i campioni di RNA concentrazione di 3 ng / ul. Preparare Mix enzimi RT e mettere 3,33 ul ai tubi PCR e aggiungere 1,67 ml di campione RNA. Incubare in PCR Instrument (BioRad) a 16 ° Cper 30 min, 42 ° C per 30 min, 85 ° C per 5 minuti, e fissato a 4 ° C in attesa.
- Eseguire real-time PCR con TaqMan PCR mix:
| Voce | Volume (pl) per campione |
| RT prodotto | 1,0 |
| 2X TaqMan Master Mix | 5.0 |
| Sonda 5X | 2,0 |
| Acqua priva di nucleasi | 2,0 |
| Totale | 10,0 |
Utilizzare una piastra da 384 pozzetti chiara reazione ottico (Life Technologies) per ogni reazione e real-time PCR strumento AB 7900 HT. Condizioni bici: 95 ° C per 10 min, [95 ° C per 5 sec, 60 ° C per 60 sec x 40 cicli].
- Curve tipiche per l'importo di fluorescently etichettati specifici prodotti di PCR (y-assi) per snoRNA-55 e miR-124 campioni di RNA (per ogni campione è in duplicati) isolati da macrofagi e microglia derivate dal midollo osseo (BMDMs) rispetto al numero di cicli (x assi) sono mostrato nella fig. 4a, b.
6. Normalizzazione e Analisi dei dati
Livelli di espressione relativi miRNA di interesse (miR-124) sono calcolate utilizzando il metodo ΔΔCT come descritto in precedenza 7,9 utilizzando snoRNA-55 per la normalizzazione della quantità iniziale di RNA.
- L'esempio di calcolo del relativo livello di espressione di miR-124 per la microglia e BMDMs è mostrato nella Tabella I.
- Come primo passo, i valori Ct per snoRNA-55 e miR-124 sono determinati per la microglia e BMDMs da qRT-PCR curve (Fig.4, tabella I, colonne: snoRNA-55 e miR-124).
- Come seconda fase, i valori ACTsono calcolati per ogni duplicato di ogni campione per sottrazione di Ct per snoRNA-55 (normalizzazione) dal Ct per il miR-124 (esperimento) (tabella I, colonne: Ct (Norm), CT (Exp) e ACT).
- Come terzo passo. ΔΔCt valori sono calcolati sottraendo ACT del campione di riferimento (ACT (Ref)) da valori ACT di altri campioni (Tabella I, colonna: ΔΔCt). Un campione viene definito come riferimento, il livello relativo di espressione miRNA per questo esempio sarà definito come 1,0. Tutti gli altri campioni saranno confrontati a questo campione di riferimento. Nel nostro caso, si definisce il livello di miR-124 nel primo campione per la microglia, come 1.0, quindi ACT (Ref) = 0,49 (Tabella I, prima fila, ha segnato in rosso).
- Infine, il livello relativo di espressione è calcolato come 2-ΔΔCt (Tabella I, colonna: miR-124 expression).
- Tutti RT-PCR vengono eseguiti in triplicato o in duplicati, e il relativo livello di espressione è presentato come media ± deviazione standard (SD) per triplica (Fig. 5a, b) oppure come entrambi i lati duplicati lato, come mostrato in fig. 5c.
7. Risultati rappresentativi
Curve tipiche di real-time PCR e valori di CT per miR-124 (microRNA di interesse) e snoRNA-55 (normalizzazione) e relativi livelli di espressione di miR-124 sono mostrati in figura. 4 e tabella I. Nei nostri esperimenti abbiamo usato snoRNA-55 come controllo per la normalizzazione. Come abbiamo accennato in precedenza, altri RNA onnipresenti può essere anche utilizzata per la normalizzazione, come snoRNA-135 e U6.
Gli esempi di espressione di miR-124 in adulti microglia vs midollo osseo derivati da macrofagi (BMDMs) sono mostrate in fig. 5 bis (BMDMs sono stati coltivati nel presence di M-CSF come descritto 7). In Fig. 5b, abbiamo confrontato l'espressione di miR-124 ordinati in popolazioni di CD45 bassa microglia vs hi macrofagi CD45. In entrambi questi esperimenti abbiamo dimostrato che cellule microgliali sono diversi da altri macrofagi esprimere elevati livelli di miR-124. Esperimenti simili può essere eseguita in futuro per la cinetica di attivazione microglia in vivo o in vitro.
Espressione di miR-124 nella microglia isolato dal CNS di topi C57BL / 6 a diversi stadi di sviluppo è mostrato in fig. 5c. Questi dati dimostrano che microglia nelle prime fasi di sviluppo esprimono bassi livelli di miR-124, che correlato con la loro fenotipo attivato 7. Un'analisi simile può essere effettuata per studiare le funzioni di microglia nel sistema nervoso centrale normali quali il confronto di espressione dei miRNA particolari microglia isolate da diverse aree del sistema nervoso centrale: il cervelletto, scavo di Pinal, ecc ippocampo
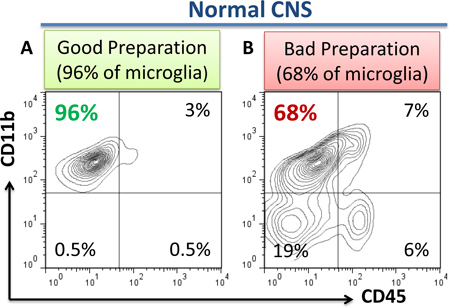
Figura 1. Gli esempi di buoni e cattivi preparazioni di microglia come determinato mediante citometria a flusso. Nel caso di isolamento "buono" della microglia dal CNS normali, 95-98% delle cellule CD45 + rappresentano CD11b microgla basso con 2-5% di CD11b + CD45 hi macrofagi perivascolari. Meno dell'1% di CD11b - hi linfociti CD45 o CD45 CD11b - cellule - astrogliali o oligodendrogliale o frammenti di cellule dovrebbero essere presenti (a). Il caso della preparazione "cattivo" viene qui mostrato (b) dove il 18% di contaminazione CD11b - CD45 - cellule è presente (b, quadrante in basso a sinistra) suggerendo insufficiente separazione su gradiente Percoll. Nel caso di una buona preparazione, 95-98% delle cellule dovrebbe rappresentare CD11b + CD45bassa microglia (a, quadrante in alto a sinistra), 2-5% di tutte le cellule dovrebbero rappresentare CD11b + CD45 macrofagi perivascolari hi (a, quadrante in alto a destra), e meno dell'1% di tutte le cellule dovrebbero rappresentare CD11b cellule negative contaminanti (a, quadrante inferiore a sinistra). In caso di "cattiva preparazione", la contaminazione significativa di CD11b - CD45 - cellule o frammenti di cellule (astroglia, particelle di mielina, ecc) sono presenti (b, quadrante inferiore a sinistra), che rende la preparazione delle cellule non idonei per l'isolamento di RNA e ulteriormente analisi. Inoltre, CD11b - cellule CD45 hi potrebbe essere presente anche in caso di "cattiva preparazione" (b, quadrante in basso a destra) che indica la contaminazione da linfociti del sangue a causa di perfusione insufficiente.
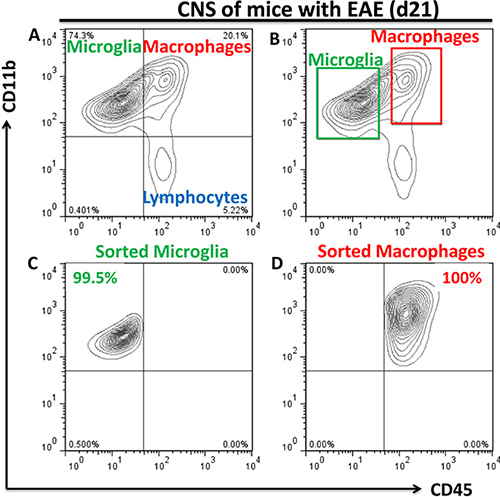
Figura 2. Citometria a flusso l'analisi dells e smistamento delle popolazioni di microglia e macrofagi periferici dal CNS malate (a) Durante neuroinfiammatoria tre popolazioni di cellule sono presenti nel SNC: CD11b CD45 + bassa (microglia), CD11b + CD45 hi (macrofagi), e CD11b - CD45. hi (linfociti), come mostrato in alto a sinistra, destra quadranti superiore ed inferiore destro, rispettivamente. Gates per l'ordinamento delle popolazioni di CD11b + CD45 CD11b bassa microglia e macrofagi CD45 + hi sono indicati in (b) e le purezze delle popolazioni rianalizzati ordinati sono indicate in (c, d). Gating preliminare sulle cellule vitali sulla base di FSC / SSC parametri è stato attuato.
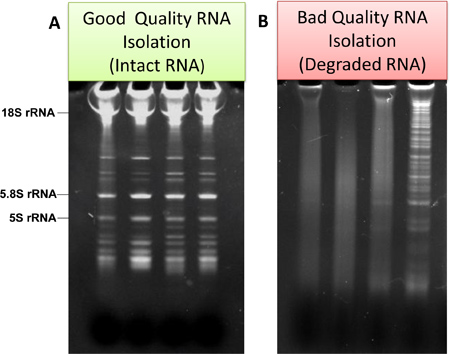
Figura 3. Esempi di qualit "buono" e "cattivi"zioni del RNA isolati da microglia e analizzati mediante elettroforesi su gel. Nel caso di buona qualità, scaletta RNA e RNA ribosomiale è evidente (a). In caso di cattiva qualità, striscio, e basso peso molecolare prodotti di degradazione sono evidenti (b).
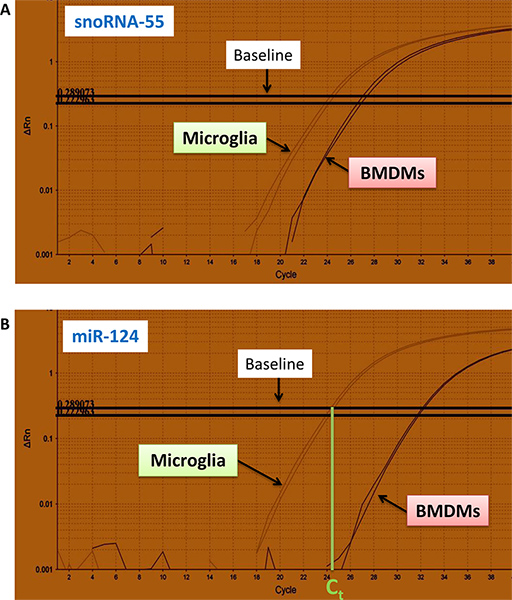
Figura 4. Real-time RT-PCR curve per fluorescenza etichettato miR-124 e snoRNA-55 prodotti di PCR. Curve per snoRNA-55 (a) e miR-124 (b) sono indicati per microglia e macrofagi di midollo osseo derivate (BMDMs). L'esperimento è stato eseguito in duplicato. Valori Ct sono stati determinato dall'intersezione della linea di base con la parte inferiore del range lineare del qRT-PCR curva come mostrato in (b). per vedere figura maggiore .
| snoRNA-55 | miR-124 | DCT = Ct (Esp)-Ct (Norm) | DDCT = DCT-DCT (Ref) | Livello relativo di espressione = 2-DDCT | |
| Ct (Norm) | Ct (Exp) | DCT | DDCT | miR-124 Espressione | |
| Microglia dal CNS normali | 24,082 | 24,572 | 0,49 (Ref) | 0 | 1,0 |
| 23,766 | 24,291 | 0,525 | 0,035 | 1,02 | |
| Midollo osseo macrofagi derivati (BMDMs) | 26,879 | 320,231 | 5,352 | 5,317 | 0,025 |
| 26,526 | 32,078 | 5,552 | 5,517 | 0,022 |
Tabella I. Calcolo dei livelli relativi di espressione di miR-124 in microglia vs macrofagi derivate dal midollo osseo a base di C valori di t per il miR-124 (microRNA di interesse) e snoRNA-55 (controllo per la normalizzazione).
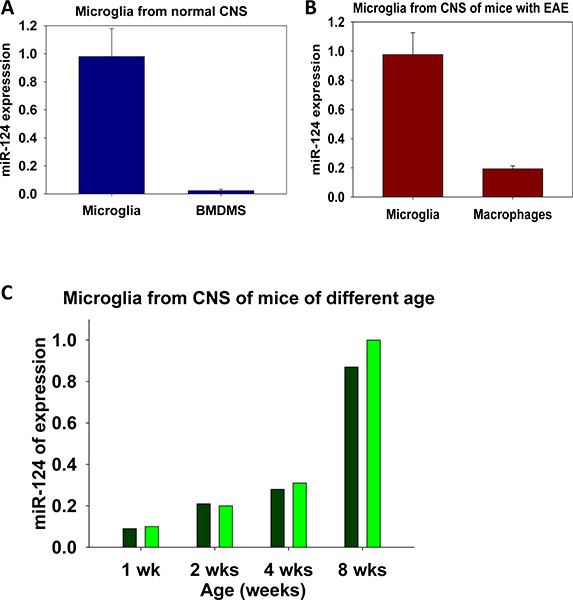
Figura 5. Analisi di miR-124 espressione microglia e macrofagi. Confronto di miR-124 espressione in cellule microgliali dal CNS normali vs macrofagi derivate dal midollo osseo (BMDMs) è mostrata in (a). Confronto di miR-124 in microglia vs macrofagi isolati da periferici di CNS in topi con neuroinfiammatoria è mostrata in (b). Variazionilivello di espressione di miR-124 in microglia durante lo sviluppo in topi di 1, 2 e 4 settimane, rispetto ai topi adulti (a 8 settimane di età) è mostrato in (c). In (a, b) l'esperimento è stato eseguito in triplicato con media ± SD di tre reazioni separate PCR mostrato. In (c) esperimento è stato eseguito in duplicato come indicato.
Discussione
Recentemente notevole interesse è stato dato a microglia e il coinvolgimento di queste cellule in molte patologie associate neuroinfiammazione e neurodegenerazione. Inoltre, il ruolo dei microRNA nella differenziazione delle cellule immunitarie e cancro è drammaticamente cresciuta negli ultimi anni diversi 5,10. Abbiamo precedentemente riportato il ruolo di miR-124 nel mantenimento del fenotipo non attivata o di riposo della microglia nel CNS normali 7, ma il ruolo degli altri tipi di microRNA ne...
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Il lavoro è stato supportato dal NIH R01 NS071039-01A1 assegno di ricerca.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| PBS 1X | GIBCO | 14190 | Può essere preparati da componenti secchi nel pH laboratorio,: 7,2-7,4 |
| 10X PBS | Thermo Scientific | SH30258.02 | Può essere preparati da componenti secchi nel pH laboratorio,: 7,2-7,4 |
| DMEM; FBS | GBCO | 11965, 10438 | |
| Mirvana kit | Invitrogen | AM1561 | |
| Percoll | Sigma | P4937-500 ml | 100 ml di Percoll 100% viene preparato miscelando 10 ml di PBS 10X e 90 ml di Percoll da Sigma. 50 ml di Percoll 70% èpreparata miscelando di 35 ml di Percoll al 100% e 15 ml di DMEM 50 ml di Percoll 40% viene preparata miscelando 20 ml di Percoll al 100% e 30 ml di 1X PBS |
| MOG 35-55 peptide | AnaSpec Inc | 60130-5 | |
| CFA | DIFCO | 263810 | |
| Tossina della pertosse | Sigma | P7208 | |
| FcR bloccando gli anticorpi | BD Biosciences | 553142 | Clone 2.4G2 |
| Anti-CD11b - PE mAb | BD Biosciences | 553311 | Può essere utilizzato con fluorofori differenti (es. AF488) |
| Anti-CD45-APC-Cy7 mAbs | Biolegend | 103116 | Può essere utilizzato con fluorofori differenti (ad es APC) |
| Paraformalformaldeide (16%) | EMS Inc | 15710 | Diluire 1:15 in PBS 1X |
| 15% Urea TBE-Gel | Life Technologies Inc. | EC68855BOX | |
| TaqMan MicroRNA Reverse Transcription Kit | Life Technologies Inc | 4366596 | |
| TaqMan Universal PCR Master Mix | Life Technologies Inc | 4304437 | |
| Saggi TaqMan MicroRNA mmu-miR-124a | Life Technologies Inc | 4427975 ID 001182 | |
| Saggi TaqMan MicroRNA snoRNA-55 | Life Technologies Inc | 4427975 ID 001228 | |
| Acqua priva di nucleasi | Life Technologies Inc | AM9937 | Acqua priva di nucleasi |
| 384 pozzetti di reazione chiara ottica | Life Technologies Inc. | 4309849 | Possono essere sostituiti con altri (ad esempio piastre da 96 pozzetti) in tempo reale diversi strumenti PCR |
| Omogeneizzatore Dounce (15 ml) | Wheaton Science Products. | 358044 | Teflon / vetro |
| 40 μ nylon filtro cella | Falco | 352340 | |
| Centrifuga Big | Sorval | RT 6000B | Diametro rotore 18,5 centimetri |
| Centrifuga Piccolo | Eppendorf | 5415 R | Diametro rotore 6,5 centimetri |
| Real-time PCR Instrument | Life Technologies Inc. | AB 7900 HT | Possono essere sostituiti con altri strumenti |
| Citometro a flusso | BD Biosciencess | LSR II | Possono essere sostituiti con altri strumenti |
| FACS | BD Biosciences | FACSAria | Possono essere sostituiti con altri strumenti |
| Spettrofotometro NanoDrop | Thermo Scientific | NanoDrop 1000 |
Riferimenti
- Tremblay, M. E. The Role of Microglia in the Healthy Brain. J. Neurosci. 31, 16064-16069 (2011).
- Graeber, M. B., Li, W., Rodriguez, M. L. Role of microglia in CNS inflammation. FEBS Lett. , (2011).
- Ponomarev, E. D., Shriver, L. P., Maresz, K., Dittel, B. N. Microglial cell activation and proliferation precedes the onset of CNS autoimmunity. J. Neurosci. Res. 81, 374-389 (2005).
- Perruisseau-Carrier, C., Jurga, M., Forraz, N., McGuckin, C. P. miRNAs stem cell reprogramming for neuronal induction and differentiation. Mol. Neurobiol. 43, 215-227 (2011).
- McCoy, C. E. The role of miRNAs in cytokine signaling. Front Biosci. 17, 2161-2171 (2010).
- He, M., Xu, Z., Ding, T., Kuang, D. M., Zheng, L. MicroRNA-155 regulates inflammatory cytokine production in tumor-associated macrophages via targeting C/EBPbeta. Cell Mol. Immunol. 6, 343-352 (2009).
- Ponomarev, E. D., Veremeyko, T., Barteneva, N., Krichevsky, A. M., Weiner, H. L. MicroRNA-124 promotes microglia quiescence and suppresses EAE by deactivating macrophages via the C/EBP-alpha-PU.1 pathway. Nat. Med. 17, 64-70 (2011).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of Specific Cell Population by Fluorescence Activated Cell Sorting (FACS). J. Vis. Exp. (41), e1546-3791 (2010).
- Gabriely, G. MicroRNA 21 promotes glioma invasion by targeting matrix metalloproteinase regulators. Mol. Cell Biol. 28, 5369-5380 (2008).
- Caffarelli, E., Filetici, P. Epigenetic regulation in cancer development. Front Biosci. 17, 2682-2694 (2011).
- Cardona, A. E., Huang, D., Sasse, M. E., Ransohoff, R. M. Isolation of murine microglial cells for RNA analysis or flow cytometry. Nat. Protoc. 1, 1947-1951 (2006).
- Gordon, R. A simple magnetic separation method for high-yield isolation of pure primary microglia. J. Neurosci. Methods. 194, 287-296 (2011).
- Moltzahn, F., Hunkapiller, N., Mir, A. A., Imbar, T., Blelloch, R. High Throughput MicroRNA Profiling: Optimized Multiplex qRT-PCR at Nanoliter Scale on the Fluidigm Dynamic ArrayTM IFCs. J. Vis. Exp. (54), e2552 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon