Method Article
Visualizzazione di compattazione del DNA nei cianobatteri tramite tomografia ad alta tensione Cryo-elettrone
In questo articolo
Riepilogo
Questo protocollo viene descritto come visualizzare la compattazione DNA transitoria nei cianobatteri. Coltivazione sincrono, monitoraggio di microscopia di fluorescenza, congelamento rapido e alta tomografia di cryo-elettrone di tensione sono usati. Un protocollo per queste metodologie è presentato, e sono discussi gli sviluppi e le applicazioni future.
Abstract
Questo protocollo viene descritto come visualizzare la compattazione DNA transitoria nei cianobatteri. Compattazione del DNA è un evento drammatico citoplasmico recentemente risultato in alcuni cianobatteri prima divisione cellulare. Tuttavia, a causa delle dimensioni di grandi cellule e il carattere transitorio, è difficile indagare la struttura in dettaglio. Per superare le difficoltà, in primo luogo, compattazione del DNA è riproducibile prodotto in cyanobacterium Synechococcus elongatus PCC 7942 da cultura sincrono utilizzando 12 h ogni ciclo luce/buio. In secondo luogo, compattazione del DNA è monitorata mediante microscopia a fluorescenza e catturato mediante congelamento rapido. In terzo luogo, la struttura dettagliata del DNA compattato celle viene visualizzata in tre dimensioni (3D) tramite tomografia del cryo-elettrone ad alta tensione. Questo insieme di metodi è ampiamente applicabile per indagare strutture transitori in batteri, ad esempio la divisione cellulare, segregazione del cromosoma, infezione dei fagi ecc., che sono monitorati mediante microscopia a fluorescenza e visualizzate direttamente da tomografia di Cryo-elettrone a intervalli di tempo appropriati.
Introduzione
Compattazione del DNA è un evento drammatico citoplasmatico che è stato individuato in alcuni cianobatteri. Quando Synechococcus elongatus è stato coltivato sotto 12 h ogni ciclo luce/buio, DNA è apparso condensata alla fine del periodo di luce, che era chiaramente diverso dalla sua apparizione nel momento punti1. È stato suggerito che questo processo è controllato da un orologio circadiano basato sul Kai proteine2. Seki et al hanno riferito che il DNA colorato con Hoechst 33342 è stato compattato in cellule di S. elongatus verso la fine del periodo di luce e ha mostrato una rod-forma ondulata sotto un microscopio a fluorescenza. Il DNA compattato, poi separato in due al centro dell'asta come la cellula diviso e infine tornò a una normale distribuzione uniforme in ogni cellula figlia3. Tuttavia, la natura transitoria e grandi dimensioni per microscopia elettronica ostacolato analisi strutturale. Murata et al combinato diversi metodi, tra cui cultura sincrono, microscopia a fluorescenza, congelamento rapido e ad alta tensione cryo-tomografia elettronica (cryo-HVET) ed è riuscito a individuare la struttura di compattazione DNA transitoria, compresa la cinetica di polifosfato corpi (PPBs)4. Il manoscritto fornisce spiegazione visiva di un materiale così difficile in dettaglio combinando le procedure sperimentali.
S. elongatus ha una forma di capsula, una lunghezza di 2 a 5 µm, una larghezza di circa 0,5 µm e la perfetta compattazione DNA appare in cellule viventi solo per un tempo molto breve. Di conseguenza, i cambiamenti strutturali che si verificano la compattazione DNA cianobatterica erano sconosciuti in dettaglio. Per studiare queste strutture da microscopia elettronica, è necessario superare due principali problemi tecnici. Uno è l'osservazione di un esemplare di spesso del batterio intero al vicino circostanze natali, e l'altro è il fissaggio rapido di una struttura dinamica. Per quanto riguarda il primo problema, il percorso libero medio (iMFP) anelastico di elettroni dipende la tensione accelerare del microscopio elettronico5. In un microscopio elettronico a trasmissione (TEM) di 300 kV, è inferiore a 350 nm. Ad esempio, quando un cianobatterio ghiaccio incorporato (esemplare spessore ≈ 600 nm) è osservato a 200kV TEM (≈ iMFP 250 nm), le strutture all'interno delle cellule sono difficili da osservare. Per contro, 1 MV TEM (iMFP ≈ 500 nm) può dare e immagine della struttura citoplasmatica in tutta la cellula (Figura 1). In questo protocollo, come una parte della soluzione, un microscopio elettronico ad alta tensione (HVEM) a una tensione di accelerazione di 1 MV è stato impiegato. Tuttavia, i servizi che implementano HVEM sono limitati in tutto il mondo. Possibili soluzioni alternative inoltre sono discussi nella sezione discussione. Il secondo problema è stato risolto da cryo-microscopia elettronica (cryo-EM). Si tratta di un potente strumento per la visualizzazione dinamica delle strutture a vicino a circostanze natali, dove il campione è congelare rapidamente in etano liquido utilizzando un dispositivo di congelamento rapido, e il momento congelato è osservato direttamente con un minimo di modifiche6. Combinando con tomografia, uno snapshot di strutture tridimensionali (3D) può essere ricostruito dalla inclinazione serie7. In questo esperimento, compattazione del DNA è stata riprodotta in s. elongatus utilizzando sincrono cultura ciclo di meno di 12 h ogni luce/buio, e la tempistica del congelamento del campione è stata determinata mediante monitoraggio sotto un microscopio a fluorescenza.
Gli approcci descritti qui sono ampiamente applicabili allo studio delle strutture dinamiche in cellule batteriche, ad esempio la divisione cellulare, segregazione del cromosoma ed infezione dei fagi e hanno un potenziale per aprire nuove vie nella ricerca microbiologica.
Protocollo
1. sincrono cultura di cianobatteri
- S. elongatus PCC 7942 sterilizzato BG 11 piastra (in un piatto di petri in plastica sterile cm 9) contenente 1,5% (p/v) di agar e 0,3% (p/v) di tiosolfato di sodio8della coltura.
- Posizionare le piastre in una camera di crescita a 23 ° C con un'intensità luminosa di 50 µE/m2/s e soggette a cicli di scuro h luce/12 h 12.
- Trasferire le cellule su piastre di agar BG11 freschi una volta a settimana.
Nota: Le culture sull'agar verranno visualizzato come bande verdi di cellule attivamente proliferanti dopo una settimana in questa condizione di cultura. - Prendere verde ciuffi di cellule con un cappio di filo sterilizzato di fiamma e striscia le cellule su una piastra di agar BG11 fresca. Eseguire questa operazione su un banco pulito.
2. monitoraggio mediante microscopia a fluorescenza
- Utilizzare cellule coltivate sulla piastra di agar per 6 giorni per osservare la compattazione del DNA. Raccogliere le cellule dal piatto alla fine del periodo di luce da versare 1 mL di soluzione di saccarosio di 0,2 M sopra le cellule. Ripetere l'operazione versando la soluzione sulle cellule in modo che la maggior parte delle cellule sono raccolti. Trasferire la soluzione sospesa-cellula in una provetta micro per la colorazione del DNA.
- Aggiungere DNA macchiando soluzione colorante (ad es. Hoechst 33342) a 500 µ l della soluzione di sospensione cellulare in un micro tubo ad una concentrazione finale di 1 µ g/mL. Quindi, mantenere la provetta al buio per 10 min.
- Centrifugare per 1 min a 2.000 x g di sedimentare le cellule. Scartare il surnatante e aggiungere 10 µ l di soluzione di saccarosio di 0,2 M per ottenere una sospensione cellulare denso.
- Trasferire 1 µ l di soluzione contengono cellule marcate per un bicchiere di diapositiva, mettere un vetrino coprioggetto e osservare con un microscopio a fluorescenza equipaggiato con un filtro UV utilizzando un obiettivo con un ingrandimento di 100x e olio da immersione.
- Confermare che la compattazione del DNA è osservata a questo punto nella maggior parte delle cellule, quindi preparare il campione per il prossimo passo di congelamento.
3. il campione di congelamento per Cryo-HVET
- Configurare un dispositivo di tuffo-congelamento. Riempire il serbatoio con azoto liquido e avviare il cryo-camera di raffreddamento dopo il collegamento del serbatoio e la camera con un tubo in Teflon.
- Riempire etano liquido in una pentola di rame piccolo all'interno della camera dopo il raffreddamento della camera alla temperatura dell'azoto liquido. Indossare occhiali durante il funzionamento, perché etano liquido è esplosivo.
- Bagliore di scarico carbonio-lato di un holey rivestita di carbonio EM griglia (griglia holey) per 30 s a 50 mA utilizzando un bombarder di ioni al plasma.
- Applicare 1 µ l di BSA oro marcato (15 nm) alla griglia holey come un marcatore fiduciale.
- Applica un'aliquota di 2,5 µ l di cellule in fase di compattazione del DNA alla griglia holey. Della macchia fuori la soluzione in eccesso con una carta da filtro. Tuffatevi nella griglia etano liquido utilizzando un pistone nel dispositivo congelamento tuffo immediatamente.
- Memorizzare le griglie congelate in azoto liquido archiviazione fino a quando sono esaminati.
4. Cryo-HVET
- Impostare il HVEM ad un'alta tensione di 1 MV.
- Montare la griglia congelata in un supporto di cryo-campione per HVEM preraffreddata a-150 ° C con azoto liquido all'interno della cryo-workstation e caricarlo il HVEM. Fare attenzione a evitare la contaminazione da ghiaccio.
- Selezionare un'area di imaging a un ingrandimento minore di 1, 000 X. Regolare l'altezza dell'asse z eucentric.
- Il tavolino a-60 ° di inclinazione e rimuovere il contraccolpo della rotazione inclinazione.
- Regolare la messa a fuoco nei pressi della località di destinazione ad un ingrandimento di 10, 000 X. Impostare un sotto fuoco di 6 a 10 µm di deviazione dall'immagine messa a fuoco. Per l'imaging, impostare la dose a 2 e–å-2 o meno sul campione in anticipo.
- Misurare la dose di elettroni per la densità di corrente sullo schermo di immagine in HVEM. Prendere un'immagine su una pellicola di elettroni o dalla fotocamera digitale allo stesso ingrandimento come nel processo di messa a fuoco.
- Raccogliere immagini di inclinazione manualmente con la stessa procedura come in (4,5) da-60 ° a + 60 ° a un incremento di angolo di inclinazione di 2 ° e 4 °.
Nota: In molti moderni microscopi elettronici, l'acquisizione di tilt series è automatizzato tramite la combinazione di una fotocamera digitale. In tal caso, seguire il manuale di istruzioni. Per le pellicole negative, sviluppare le pellicole per 12 min a 20° C in un serbatoio di sviluppatore utilizzando uno sviluppatore piena forza e difficoltà in un fissatore per 10 min. digitalizzare i film con una risoluzione di 4.000 dpi (0,635 nm/pixel dell'immagine) utilizzando uno scanner piano. Nel caso di una fotocamera digitale, è necessario sottoporre le immagini raccolte direttamente per la successiva fase di elaborazione di immagine. Se necessario, è possibile ridurre le dimensioni dell'immagine di un filtro mediano utilizzando binning (fattore riduzione dimensionale) ed il software adatto (ad es. ImageJ) da due a quattro.
5. tomografica ricostruzione
- Rendere un file di immagine dello stack dall'individuo inclinare immagini utilizzando il comando "tif2mrc" o "newstack" in IMOD software9.
- Avviare il software eTomo GUI in IMOD e impostare i parametri di immagine: dimensione dei pixel, diametro fiducial, rotazione immagine, ecc. Quindi creare script.
- Eseguire programmi individuali secondo il software elencato nelle tabelle dei materiali, dove la serie di inclinazione è allineata con marker fiduciali (con un medio errore residuo inferiore a 0,5). Infine, ricostruire un tomogramma 3D utilizzando l'algoritmo SIRT in IMOD.
- Estrarre una regione di interesse (ROI) dal tomogramma e denoise utilizzando un filtro di denoise: un filtro anisotropico diffusione in IMOD, un filtro bilaterale in EMAN10o una morfologia matematica filtro11, ecc., con i parametri appropriati per migliorare il contrasto.
6. segmentazione della caratteristica di interesse
Nota: La procedura descritta di seguito è specifica per il software usato (Vedi Materiali tavolo) ma altri pacchetti software possono essere utilizzati invece. Consultare la Guida utente.
- Nella finestra del visualizzatore 3D, aprire il file tomogramma Amira software e generare un OrthoSlice.
- Nella finestra dell'Editor di segmentazione, creare un file di segmentazione selezionando un nuovo "campo di etichetta".
- Manualmente seguite il bordo della caratteristica di interesse (FOI). Seguire la FOI attraverso tutte le sezioni di tomogramma. Per il secondo FOI, creare un nuovo "materiale" e ripetere la stessa operazione.
- Generare un rendering superficie selezionando il menu "SurfaceGen". Per visualizzare il volume segmentato, selezionare il menu "SurfaceView". Per spostare, ruotare e ingrandire il volume 3D, è possibile utilizzare gli strumenti nella finestra del visualizzatore 3D.
- Per la segmentazione automatica, utilizzare lo strumento bacchetta magica. Fare clic su un oggetto e regolate i cursori nel Display e mascheratura per coprire l'intervallo di valori in modo che l'oggetto è completamente selezionato per le sue caratteristiche.
Risultati
In una cultura sincrona precisa ciclo di meno di 12 h ogni luce/buio, DNA etichettato con Hoechst Mostra una distribuzione uniforme normale in condizioni di buio (Figura 2a). Tuttavia, progressivamente compatta all'interno della cellula durante il periodo di luce e appare come una struttura ondulata di rod-like (Figura 2b) alla fine del periodo di luce. Infine, l'asta si divide al centro (frecce in Figura 2C) e sue due parti sono distribuiti in cellule della figlia (figura 2d). Dopo la divisione cellulare, il DNA compattato scomparirà immediatamente, e il DNA restituisce ad una normale distribuzione uniforme.
Quando un'aliquota di cellule che contengono le cellule nella fase finale di compattazione del DNA sono stati immediatamente trasferiti su una griglia holey e rapida congelati in etano liquido, e la griglia congelata è stata osservata da 1 MV cryo-HVEM, le strutture interne dei cianobatteri compreso DNA, strati di membrana tilacoide, pareti cellulari e PPBs, apparve come un'istantanea al momento del congelamento (Figura 3a). Molte cellule hanno mostrato distinti compattazione del DNA nelle cellule (frecce bianche in Figura 3a) e potrebbero essere facilmente distinguibili dalle cellule normali (bianchi punte di freccia in Figura 3b). Alcuni hanno esibito una costrizione al centro delle cellule come previsto prima divisione cellulare (freccia gialla in Figura 3a).
In 3D tomogrammi, principali organelli della cellula possono essere segmentati; parete cellulare, strati di membrana tilacoide, DNA e PPBs potrebbe essere distinto (Figura 4). In particolare, il DNA compattato era separato da uno spazio distinto nel citoplasma dove il DNA era circondato da materiale di bassa densità e gli strati di membrana tilacoide erano distorti lungo l'asta ondulata del DNA compattato. Recentemente è stato osservato un comportamento dinamico del PPBs: nel DNA compattato cellule, molti piccoli PPBs sono stati veduti ad aderire al DNA, mentre essi sono grandi e meno in cellule normali. Inoltre, la maggior parte del PPBs apparve come coppie, e alcuni DNA è sembrato essere in un processo di separazione dal PPBs. Ciò ha suggerito che PPBs essi stessi sono divisi in due dalla duplicazione del DNA e la funzione come i fornitori di fosfato per la sintesi del DNA.
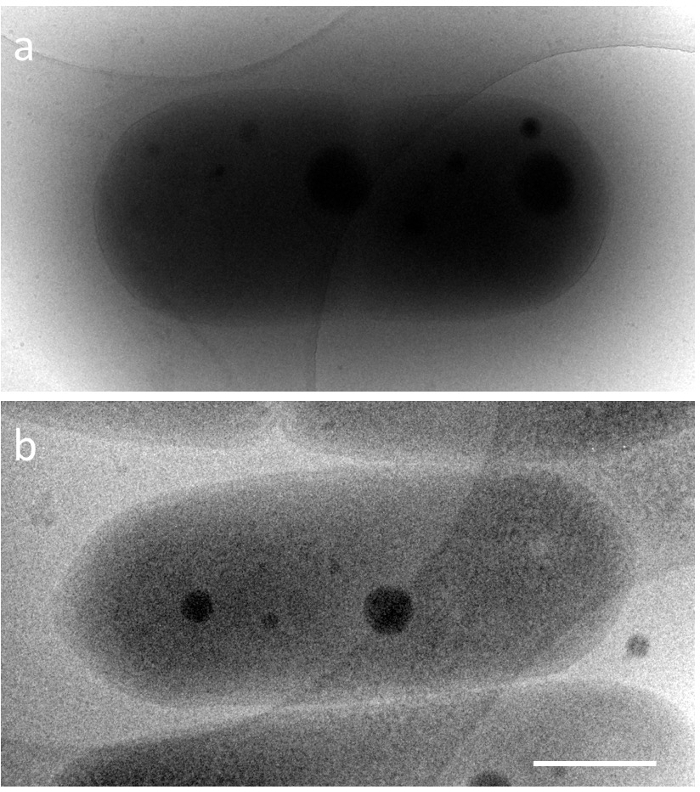
Figura 1. Immagini di cryo-EM di cianobatteri incorporato ghiaccio normale, S. elongatus PCC 7942 alle diverse tensioni di accelerazione. a) 200kV e b) 1000kV. Barra della scala = 500 nm. Clicca qui per visualizzare una versione più grande di questa figura.
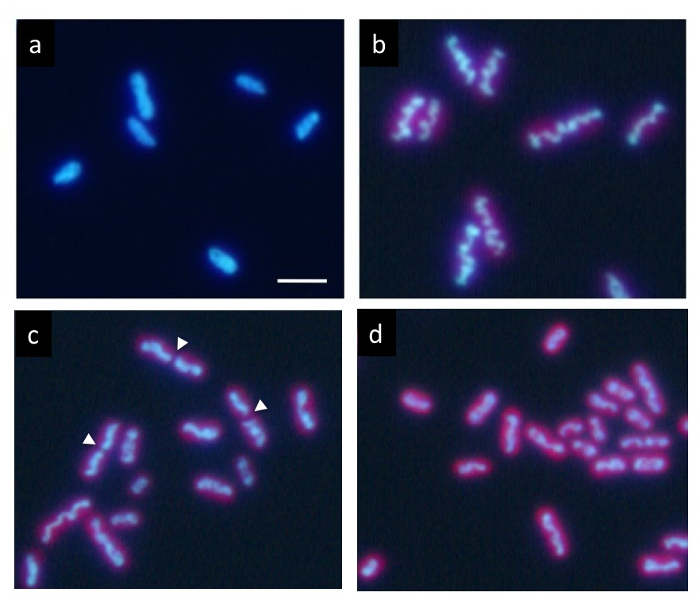
Figura 2: Microscopia di fluorescenza delle cellule di S. elongatus cianobatterio. Dopo coltura sincrona cicli di meno di 12 ore ogni luce/buio, le cellule sono state macchiate con Hoechst 33342. (a) cellule dopo 2 h di insorgenza dello stato scuro mostrano etichettatura uniforme di DNA. Il DNA in molte cellule condensato gradualmente durante il periodo di luce. In ultima analisi, formava un asta di ondulato spessore (b) tipico di compattazione del DNA. Dopo questa fase, le strutture di DNA compattate rapidamente divise loro centri (frecce) durante la divisione cellulare (c) e i due frammenti separati in cellule della figlia (d). Le cellule della figlia restituite al contrassegno nuovamente nel periodo scuro uniforme del DNA. Barra della scala = 2 µm. Clicca qui per visualizzare una versione più grande di questa figura.
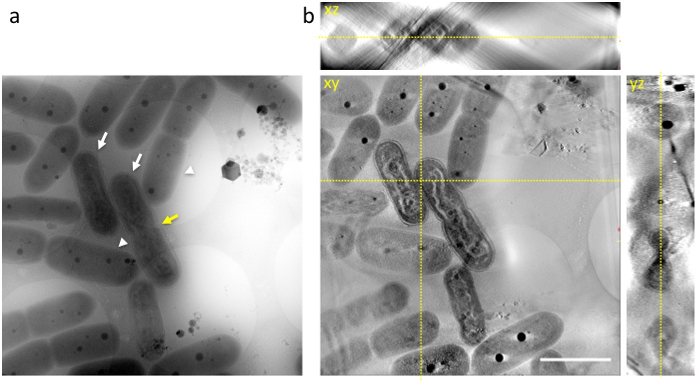
Figura 3: immagine di Cryo-HVEM di cianobatteri incorporato nel ghiaccio amorfo (a) Mostra un'immagine raw a tilt 0 °. Immagini sono state scattate alla fine del periodo di luce. Alcune cellule mostrano ondulato asta-a forma di DNA corpi simili a cromosomi eucariotici condensati (frecce bianche). In cellule normali, i cianobatteri esibiscono una struttura di DNA percepibile all'interno del citoplasma (frecce bianche). Alcuni presentano una costrizione al centro delle cellule come previsto prima divisione cellulare (freccia gialla). (b) xy, xz, yz-fette di un tomogramma 3D. Linee gialle tratteggiate mostrano intersezioni delle fette. Barra della scala = 2 µm. Clicca qui per visualizzare una versione più grande di questa figura.
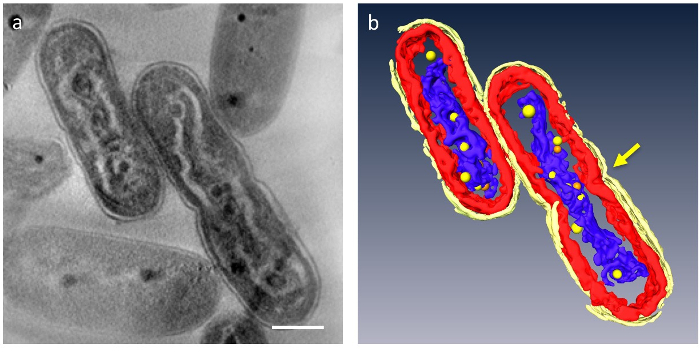
Figura 4: compattato ultrastruttura delle cellule contenenti DNA. I componenti principali sono segmentati: parete cellulare (giallo brillante), membrane tilacoidali (rosse) ed il DNA (blu). (a) Mostra una z-fetta di un tomogramma 3D. (b) tutti i segmenti. La costrizione che indica separazione cellulare appare al centro della cella (freccia). PPBs sono modellate come sfere gialle o arancioni; ogni sfera arancione rappresenta la controparte della sfera gialla più vicina. Barra della scala = 500 nm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Abbiamo presentato una sequenza di protocolli per la visualizzazione transitoria compattazione del DNA nei cianobatteri. Il concetto di base è simile a quella del correlativo luce e microscopia elettronica (CLEM)12. Inoltre, in questo metodo, live cianobatteri sono stati monitorati mediante microscopia a fluorescenza, congelati rapidamente sulle griglie di EM e visualizzati direttamente con tomografia cryo-elettrone ad alta tensione. Come una prima applicazione, la struttura dettagliata del DNA compattato cellule batteriche è stato correttamente visualizzato in 3D. Attualmente, questa procedura è specifica per questo argomento, ma essa si applicherà più estesamente, con metodologia modificata in alcuni casi. Qui, i vantaggi, i limiti e le possibilità future di questo metodo sono discussi.
Uno dei vantaggi di questo metodo è la visualizzazione 3D di tutta la cella. 1 MV HVEM visualizzati correttamente la dinamica struttura degli organelli sottocellulari nel DNA compattato le cellule. Tuttavia, la struttura fine all'interno di cellule normali non si distinguevano a causa del contrasto dell'immagine in basso. Aumentando anelastica e quantistico negli esemplari di spessi sfoca l'immagine13. Perdita zero e la maggior parte-probabile-perdita immagine filtro da un filtro di energia può migliorare il contrasto dell'immagine riducendo la dispersione anelastica14,15, ma non funzionerà per gli esemplari più spessi di iMFP. I picchi di perdita zero e la maggior parte-probabile-perdita drasticamente diminuiscono con spessore provini. È particolarmente difficile ottenere un rapporto segnale-rumore sufficiente per i campioni di ghiaccio incorporato sensibile dell'elettrone. Murata et al hanno dimostrato che la trasmissione scansione 1MV dà di microscopia (STEM) maggiore contrasto di immagine rispetto a un'immagine di campo luminoso in plastica incorporato di cellule di lievito con 5 µm di spessore, dove il contrasto dell'immagine è dato principalmente da contrasto ampiezza13 . Tuttavia, si prevede che l'effetto di trascinamento danno il maggiori elettroni accelerati crea un'altra limitazione alla dose di irradiazione per danni sensibili cryo-campioni16. L'applicazione di Volta e Zernike fase piastre17,18 per HVEM può essere in grado di ridurre i danni secondari riducendo la dose totale in futuro. Un'altra limitazione all'utilizzo HVEM per spessi esemplari deriva dal fatto che le strutture di utente che forniscono HVEM sono scarse in tutto il mondo.
Utilizzando una metodologia alternativa di osservare esemplari di spessi, tomografia di cryo-staminali a 300 kV ha dimostrato immagini ad alto contrasto su campioni congelati-idrato con spessore superiore a diverse centinaia di nanometri19. Per recuperare il contrasto di fase in cryo-staminali, microscopia elettronica pthychographic è stato introdotto anche, in cui la piastra di fase nella lente condensatore traspone una diffrazione modulato in fase a un pixelated detector 2D20. Le immagini vengono recuperate mediante calcolo da più le diffrazioni. Per cryo-imaging 3D veloce e diretto di grande nativo congelati campioni, cryo-FIB-SEM può anche essere usato21, dove seriale sezionamento con un fascio ionico focalizzato e bloccare viso imaging viene applicati per imaging completamente idratata campioni congelati. Anche se queste tecnologie ampliare la gamma di osservazione di campioni biologici, è difficile trovare il percorso di destinazione dei batteri, ad esempio etichettato come batteri, perché l'obiettivo è completamente sotto il ghiaccio e non può essere identificato prima di tagliare.
Compattazione del DNA produce una struttura distinta nei cianobatteri. DNA compattato cellule sono prontamente distinto anche senza essere macchiato a causa di una distorsione di grande densità all'interno delle cellule che non è presente nelle cellule normali. Tuttavia, al fine di visualizzare altri eventi locali all'interno della cellula, è necessario trasferire il ROI fluorescente contrassegnato nel microscopio elettronico. Per correlativo luce e microscopia elettronica (CLEM), immagini del microscopio ottico e microscopio elettronico immagini sono generalmente correlati utilizzando granuli di lattice fluorescente o punti quantici su EM finder griglie12. Le particelle di etichettatura devono essere di densità dell'elettrone alta oltre a fluorescenza. Essi possono correlare con precisione e in modo affidabile le posizioni tra le due immagini. Inoltre, confermando l'area con etichetta con microscopia crio-luce, completa sovrapposizione del ROI può essere raggiunto tra i due microscopi. Quando che caratterizza eventi più dettagliati strutturali nella compattazione del DNA, queste particelle e cryo-luce microscopio sarà uno strumento indispensabile per una correlazione più robusto e preciso in futuro.
In questo articolo viene illustrato come caratterizzare la struttura transitoria di compattazione del DNA nei cianobatteri da una combinazione di cultura sincrono, microscopia a fluorescenza e tomografia cryo-elettrone ad alta tensione. Questo protocollo si concentra sull'osservazione del DNA compattato. Combinando questo metodo con altre nuove tecnologie di cui sopra, sarà possibile studiare il processo di compattazione del DNA in dettaglio e metodi opportunamente modificate sono ampiamente applicabili agli altri eventi dinamici strutturali nei batteri.
Divulgazioni
Gli autori non dichiarano concorrenti interessi finanziari.
Riconoscimenti
Gli autori ringraziano Tammo Reisewitz per lettura critica del manoscritto e sia Mako Hayashi e Sayuri Hagiwara per coltivazione attenta e l'osservazione di cianobatteri, Yoshitaka Kimori per elaborazione di immagini, Chihong Song, Naoyuki Miyazaki e Miyoko Nagayoshi per aiutare con la segmentazione delle strutture. Questo lavoro è stato supportato dal programma di studio collaborativo dell'Istituto Nazionale Scienze fisiologiche (NIPS) per Y.K.
Materiali
| Name | Company | Catalog Number | Comments |
| Hoechst 33342 solution | Dojindo | 346-07951 | 1mg/mL in H2O |
| Agar Powder (for plant culture) | Wako | 016-11875 | |
| Boric acid | Wako | 027-02192 | 99.5% |
| Manganese chloride tetrahydrate | Wako | 139-00722 | 99% |
| Zinc sulfate heptahydrate | Wako | 264-00405 | 99.5% |
| Sodium molybdate | Wako | 196-02472 | 99% |
| Copper sulfate pentahydrate | Wako | 039-04412 | 99.5% |
| Cobalt nitrate hexahydrate | Wako | 031-03752 | 99.5% |
| Sodium nitrate | Wako | 191-02542 | 99% |
| Magnesium sulfate heptahydrate | Wako | 131-00405 | 99.5% |
| Calcium chloride dehydrate | Wako | 031-00435 | 99.5% |
| Citric acid | Wako | 036-05522 | 98% |
| EDTA-2Na | Dojindo | 343-01861 | 99.5% |
| Sodium carbonate | Wako | 197-01581 | 99.8% |
| Potassium phosphate dibasic | Wako | 164-04295 | 99% |
| TES (Good’s buffer) | Dojindo | 344-02653 | 99% |
| Ferric ammonium citrate | Wako | 092-00802 | 1st Grade |
| Sodium thiosulfate pentahydrate | Wako | 197-03585 | 99% |
| BSA gold tracer 15nm | Aurion | 215.133 | |
| Quantifoil EM grid | Quantifoil MicroTools | R3.5/1 Copper grid | |
| Electron films | Kodak | SO-163 | |
| Developer | Kodak | D19 | |
| Fixer | Kodak | Rapid fixer | Solution |
| Filter paper | Whatman | Grade 1 | |
| Growth chamber | NKsystem | LH-100SP | |
| Fluorescent microscope | Nikon | ECLIPSE 50i | |
| High voltage TEM | HItachi | H1250M | |
| Cryo-specimen holder for HVEM | Gatan | ||
| plunge-freezing device | Leica | EM CPC | |
| Plasma Ion bombarder | Vacuum device | PIB-10 | |
| Liquid nitrogen storage | Taylor-Wharton | 25LDB | |
| Developing tank | Dosaka EM | TB-3-75 | |
| flatbed scanner | Nikon | Coolscan 9000ED | |
| Segmentation software | FEI | Amira | https://www.fei.com/software/amira |
| Tomographic Reconstruction software | eTOMO | http://bio3d.colorado.edu/imod |
Riferimenti
- Smith, R. M., Williams, S. B. Circadian rhythms in gene transcription imparted by chromosome compaction in the cyanobacterium Synechococcus elongatus. Proc. Nat. Acad. Sci. USA. 103 (22), 8564-8569 (2006).
- Kondo, T. A cyanobacterial circadian clock based on the kai oscillator. CSHS Quant. Biol. 72, 47-55 (2007).
- Seki, Y., Nitta, K., Kaneko, Y. Observation of polyphosphate bodies and DNA during the cell division cycle of Synechococcus elongatus PCC 7942. Plant biol. 16 (1), 258-263 (2014).
- Murata, K., Hagiwara, S., Kimori, Y., Kaneko, Y. Ultrastructure of compacted DNA in cyanobacteria by high-voltage cryo-electron tomography. Sci. Rep. 6, 34934 (2016).
- Koster, A. J., et al. Perspectives of molecular and cellular electron tomography. J. struct. boil. 120 (3), 276-308 (1997).
- Lučić, V., Rigort, A., Baumeister, W. Cryo-electron tomography: The challenge of doing structural biology in situ. J. Cell Biol. 202 (3), 407-419 (2013).
- Murata, K., et al. Visualizing Adsorption of Cyanophage P-SSP7 onto Marine Prochlorococcus. Sci. Rep. 7, 44176 (2017).
- Rippka, R., Deruelles, J., Waterbury, J. B., Herdman, M., Stanier, R. Y. Generic assignments, strain histories and properties of pure cultures of Cyanobacteria. J. Gen. Microbiol. 111, 1-61 (1979).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J. struct. boil. 116, 71-76 (1996).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. J. Struct. Biol. 144, 114-122 (2003).
- Kimori, Y. Morphological image processing for quantitative shape analysis of biomedical structures: effective contrast enhancement. J. Sync. Rad. 20, 848-853 (2013).
- Jun, S., Zhao, G., Ning, J., Ga Gibson, ., Watkins, S. C., Zhang, P. Correlative microscopy for 3D structural analysis of dynamic interactions. J. Visual. Exper. 76, e50386 (2013).
- Murata, K., Esaki, M., Ogura, T., Arai, S., Yamamoto, Y., Tanaka, N. Whole-cell imaging of the budding yeast Saccharomyces cerevisiae by high-voltage scanning transmission electron tomography. Ultramicros. 146, 39-45 (2014).
- Kortje, K. H., Paulus, U., Ibsch, M., Rahmann, H. Imaging of thick sections of nervous tissue with energy-filtering transmission electron microscopy. J. Microsc. 183, 89-101 (1996).
- Bouwer, J. C., et al. Automated most-probable loss tomography of thick selectively stained biological specimens with quantitative measurement of resolution improvement. J. Struct. Biol. 148 (3), 297-306 (2004).
- Egerton, R. F., Li, P., Malac, M. Radiation damage in the TEM and SEM. Micron. 35 (6), 399-409 (2004).
- Danev, R., Buijsse, B., Khoshouei, M., Plitzko, J. M., Baumeister, W. Volta potential phase plate for in-focus phase contrast transmission electron microscopy. Proc. Nat. Acad. Sci. USA. 111 (44), 15635-15640 (2014).
- Murata, K., et al. Zernike phase contrast cryo-electron microscopy and tomography for structure determination at nanometer and subnanometer resolutions. Structure. 18 (8), 903-912 (2010).
- Wolf, S. G., Houben, L., Elbaum, M. Cryo-scanning transmission electron tomography of biological cells. Nat. Methods. 11 (4), 423-428 (2014).
- Ophus, C., et al. Efficient linear phase contrast in scanning transmission electron microscopy with matched illumination and detector interferometry. Nat. Comm. 7, 1-7 (2016).
- Schertel, A., et al. Cryo FIB-SEM: Volume imaging of cellular ultrastructure in native frozen specimens. J. Struct. Biol. 184 (2), 355-360 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon