Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement et purification évolutifs de vésicules extracellulaires à partir d’Escherichia coli et d’autres bactéries
Dans cet article
Résumé
Les bactéries sécrètent des vésicules extracellulaires (VE) de taille nanométrique transportant des molécules biologiques bioactives. La recherche sur les véhicules électriques se concentre sur la compréhension de leur biogenèse, de leur rôle dans les interactions microbes-microbes et hôte-microbe et des maladies, ainsi que de leurs applications thérapeutiques potentielles. Un flux de travail pour l’isolation évolutive des véhicules électriques de diverses bactéries est présenté pour faciliter la normalisation de la recherche sur les véhicules électriques.
Résumé
Diverses espèces bactériennes sécrètent ~20-300 nm de vésicules extracellulaires (VE), composées de lipides, de protéines, d’acides nucléiques, de glycanes et d’autres molécules dérivées des cellules parentales. Les VE fonctionnent comme des vecteurs de communication intra et inter-espèces tout en contribuant à l’interaction entre les bactéries et les organismes hôtes dans le contexte de l’infection et de la colonisation. Compte tenu de la multitude de fonctions attribuées aux VE dans la santé et la maladie, il y a un intérêt croissant pour l’isolement des VE pour les études in vitro et in vivo . On a émis l’hypothèse que la séparation des VE en fonction des propriétés physiques, à savoir la taille, faciliterait l’isolement des vésicules provenant de diverses cultures bactériennes.
Le flux de travail d’isolement comprend la centrifugation, la filtration, l’ultrafiltration et la chromatographie d’exclusion de taille (SEC) pour l’isolement des VE à partir de cultures bactériennes. Une étape de filtration à flux tangentiel (TFF) entraînée par pompe a été incorporée pour améliorer l’évolutivité, permettant l’isolation du matériau des litres de culture cellulaire de départ. Escherichia coli a été utilisé comme système modèle exprimant la nanoluciférase associée à EV et la mCherry non associée à EV en tant que protéines rapporteures. La nanoluciférase a été ciblée sur les VE en fusionnant son extrémité N avec la cytolysine A. Les premières fractions chromatographiques contenant 20-100 nm EVs avec cytolysine A - nanoLuc associée étaient distinctes des fractions ultérieures contenant les protéines libres. La présence de nanoluciférase associée à l’EV a été confirmée par marquage immuno-or et microscopie électronique à transmission. Ce flux de travail d’isolation EV est applicable à d’autres espèces bactériennes à Gram négatif et à Gram positif associées à l’intestin humain. En conclusion, la combinaison de la centrifugation, de la filtration, de l’ultrafiltration/TFF et de la SEC permet une isolation évolutive des VE de diverses espèces bactériennes. L’utilisation d’un flux de travail d’isolement normalisé facilitera les études comparatives des VE microbiennes entre les espèces.
Introduction
Les vésicules extracellulaires (VE) sont des structures de type liposome, de taille nanométrique, composées de lipides, de protéines, de glycanes et d’acides nucléiques, sécrétées par les cellules procaryotes et eucaryotes1. Depuis les premières études visualisant la libération de VE par les bactéries à Gram négatif2, le nombre de fonctions biologiques attribuées aux VE bactériens (20-300 nm de diamètre) n’a cessé de croître au cours des dernières décennies. Leurs fonctions comprennent le transfert de la résistance aux antibiotiques3, la formation de biofilm4, la détection du quorum5 et l’administration de toxines6. L’utilisation des VE bactériens comme thérapeutique suscite également un intérêt croissant, en particulier en vaccinologie7 et en traitement du cancer8.
Malgré l’intérêt croissant pour la recherche sur les véhicules électriques, il existe encore des défis techniques concernant les méthodes d’isolement. Plus précisément, il existe un besoin de méthodes d’isolement reproductibles, évolutives et compatibles avec divers organismes producteurs de VE. Afin de créer un ensemble unifié de principes pour la planification et le rapport sur l’isolement des VE et les méthodes de recherche, la Société internationale pour les vésicules extracellulaires publie et met à jour le document de positionMISEV 9. En outre, le consortium EV-TRACK fournit une plate-forme ouverte pour rendre compte des méthodologies détaillées pour l’isolation des VE utilisées dans les manuscrits publiés afin d’améliorer la transparence10.
Dans ce protocole, les méthodologies précédentes utilisées pour l’isolement des VE à partir de cultures de cellules de mammifères ont été adaptées11,12 pour permettre l’isolement des VE à partir de la culture de cellules bactériennes. Nous avons cherché à utiliser des méthodes qui permettent d’isoler les VE d’une variété de microbes, ce qui peut être évolutif, et d’équilibrer la pureté et le rendement des VE (comme discuté dans le document de positionMISEV 9). Après élimination des cellules bactériennes et des débris par centrifugation et filtration, le milieu de culture est concentré soit par ultrafiltration par dispositif centrifuge (pour un volume allant jusqu’à ~100 mL), soit par TFF entraîné par pompe (pour les plus grands volumes). Les véhicules électriques sont ensuite isolés par SEC à l’aide de colonnes optimisées pour la purification des petits véhicules électriques.
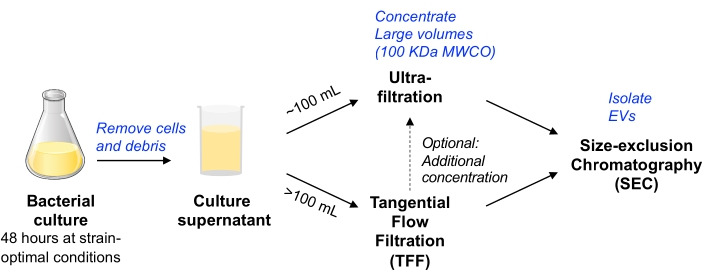
Figure 1 : Vue d’ensemble schématique du flux de travail d’isolation bactérienne des véhicules électriques. Abréviations : EV = vésicule extracellulaire; TFF = filtration à flux tangentiel; SEC = chromatographie d’exclusion de taille; MWCO = seuil de poids moléculaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Une souche commenssale de souris d’Escherichia coli (c.-à-d. E. coli MP1 13) a été utilisée comme organisme modèle et modifiée pour exprimer la nanoluciférase associée à l’EV par fusion avec la cytolysine A, comme indiqué précédemment14. Les méthodes utilisées ici peuvent traiter au moins jusqu’à plusieurs litres de cultures bactériennes et séparer efficacement les protéines associées à EV des protéines non associées à EV. Enfin, cette méthode peut également être utilisée pour d’autres espèces bactériennes à Gram positif et à Gram négatif. Toutes les données pertinentes des expériences rapportées ont été soumises à la base de connaissances EV-TRACK (EV-TRACK ID: EV210211)10.
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE : Veiller à ce que tous les travaux impliquant des bactéries et de l’ADN recombinant suivent les pratiques exemplaires en matière de confinement de la biosécurité appropriées au niveau de risque pour la biosécurité de chaque souche. Le travail doit être effectué conformément aux réglementations locales, nationales et internationales en matière de biosécurité.
1. Souches bactériennes et conditions de culture
REMARQUE : Les souches bactériennes utilisées dans cette étude étaient Escherichia coli MP113, Akkermansia mucinophila, Bacteroides thetaiotaomicron, Bifidobacterium breve et Bifidobacterium dentium.
- Pour E. coli, utiliser une boucle stérile pour inoculer des colonies individuelles dans 250 à 1 000 ml de bouillon Luria-Bertani (LB) et incuber en aérobie dans un incubateur à agitation à 300 tr/min et 37 °C pendant 48 h avant de traiter la culture. Pour la souche MP1 d’E. coli recombinante contenant p114-mCherry-Clyluc (méthode supplémentaire et figure supplémentaire S1), ajouter du chloramphénicol à la gélose LB et au bouillon à une concentration finale de 17 μg/mL.
- Pour A. mucinophila, B. thetaiotaomicron, B. breve et B . dentium, strester sur des plaques de gélose pour perfusion cœur de cerveau (BHI) et incuber en anaérobiose à l’intérieur d’une chambre anaérobie vinylique. Inoculer les colonies individuelles dans 100 ml de bouillon BHI préréduit et incuber pendant 48 h en anaérobiose.
2. Isolation des véhicules électriques
- Clarification du milieu de culture bactérien par centrifugation et filtration
- Transférer les cultures de cellules bactériennes inoculées à l’étape 1 pour nettoyer les flacons centrifugeuses en polypropylène de 250 ml ou 500 ml en versant. Centrifuger les bouteilles dans un rotor à angle fixe de grande capacité à 4 °C et 5 000 × g pendant 15 min. Transférer le surnageant dans les flacons de centrifugation propres en versant soigneusement et centrifuger à nouveau à 10 000 × g pendant 15 min.
REMARQUE : Réutilisez les bouteilles après un nettoyage et une décontamination adaptés à la biosécurité.- Si de grosses pastilles de cellules bactériennes sont présentes après la deuxième centrifugation, répétez la centrifugation dans un flacon propre pour éliminer davantage les cellules.
- Transférer le surnageant dans un dispositif filtrant à vide en polyéthersulfone de 0,22 μm de taille appropriée en versant. Filtrer en connectant le dispositif de filtration à une alimentation en paroi sous vide. Si le taux de filtration diminue de manière significative, déplacez simplement tout matériau non filtré vers un nouvel appareil. Conserver le milieu filtré à 4 °C pendant la nuit et poursuivre le protocole le lendemain si vous le souhaitez.
REMARQUE: Les centrifugations ci-dessus permettent généralement le traitement de ~2x le volume indiqué de culture cellulaire à travers chaque dispositif. Par exemple, un seul dispositif filtrant de 500 mL pourrait filtrer ~1 000 mL de culture précentrifugée. Ces appareils ne sont généralement pas réutilisés. L’utilisation de filtres à seringues à cette étape n’est pas recommandée sans optimisation, car des pertes importantes ont été constatées avec les modèles testés. C’est un point d’arrêt potentiel. - Vérifier l’élimination complète des cellules viables à ce stade en étalant une partie aliquote du surnageant filtré sur des plaques de gélose appropriées et s’assurer de l’absence de colonies après l’incubation dans des conditions optimales pour la souche bactérienne. Si des bactéries sont détectées, optimisez davantage la procédure ci-dessus en effectuant des centrifugations et / ou des filtrations supplémentaires.
- Transférer les cultures de cellules bactériennes inoculées à l’étape 1 pour nettoyer les flacons centrifugeuses en polypropylène de 250 ml ou 500 ml en versant. Centrifuger les bouteilles dans un rotor à angle fixe de grande capacité à 4 °C et 5 000 × g pendant 15 min. Transférer le surnageant dans les flacons de centrifugation propres en versant soigneusement et centrifuger à nouveau à 10 000 × g pendant 15 min.
- Concentration du milieu filtré
- Si vous travaillez avec des volumes importants >100 mL, passez à l’étape 2.2.2. Si vous travaillez avec des volumes de ~100 mL, chargez 90 mL de milieu de culture filtré sur le réservoir d’un dispositif d’ultrafiltration centrifuge de seuil de poids moléculaire (MWCO) de capacité respective de 100 kDa à l’aide de pipettes sérologiques. Toujours équilibrer avec un dispositif d’ultrafiltration correspondant et centrifuger dans un rotor de godet oscillant à 4 °C et 2 000 × g pendant des intervalles de 15 à 30 minutes, jusqu’à ce que le volume du fluide dans le réservoir supérieur ait été concentré à <0,5 mL.
- Remplissez le réservoir avec tout milieu de culture filtré restant. Si vous « rechargez », retirez le flux continu situé au bas de l’appareil et rééquilibrez tous les appareils.
REMARQUE : Il a été observé que le volume maximal de milieu de culture filtré pouvant être concentré à l’aide de ces dispositifs est <2 fois supérieur au volume recommandé. - Si la viscosité du milieu concentré dans le réservoir est visiblement augmentée (matière sombre et visqueuse), diluer avec une solution saline tamponnée au phosphate (PBS) et reconcentrer par centrifugation pour diluer toutes les protéines non EV inférieures au MWCO de 100 kDa.
REMARQUE : Il s’agit d’un point d’arrêt potentiel. - Transférer le milieu concentré dans un tube à faible liaison aux protéines, conserver à 4 °C pendant la nuit et poursuivre le protocole le lendemain si désiré.
- Remplissez le réservoir avec tout milieu de culture filtré restant. Si vous « rechargez », retirez le flux continu situé au bas de l’appareil et rééquilibrez tous les appareils.
- Si vous travaillez avec des volumes d’une taille significative > 100 ml, sélectionnez un périphérique TFF de taille appropriée (MWCO de 100 kDa) pour s’adapter au volume à traiter.
REMARQUE : Des dispositifs de filtration pour le traitement de 100 ml à >1 000 ml sont disponibles dans le commerce. La disponibilité locale, le coût et la compatibilité avec la pompe et les tuyaux/connexions dicteront quels modèles particuliers seront les plus utiles. Jusqu’à 2 L de milieu de culture ont été traités avec le dispositif indiqué dans le tableau des matériaux avant de devoir nettoyer le filtre (voir l’étape 2.3 ci-dessous pour le protocole de nettoyage).- Assembler un circuit de filtration avec un tube #16 à faible liaison/lixiviation, des adaptateurs de tuyau de 1/8 pouce vers Luer, le dispositif TFF et une pompe péristaltique, comme indiqué à la figure supplémentaire S2.
REMARQUE : Effectuer une TFF dans une enceinte de biosécurité afin de minimiser le risque de contamination de la préparation du véhicule électrique par des bactéries environnementales. - À température ambiante, commencer à faire circuler le milieu filtré et conditionné à environ 200 mL/min (minimum 100 mL/min). Déterminer le régime approprié correspondant au débit désiré en pompant 200 mL de PBS dans une cuve graduée. Lors de la circulation d’un milieu filtré et conditionné, recueillir les molécules < 100 kDa traversant la membrane d’ultrafiltration en tant que déchets dans un récipient séparé.
REMARQUE: L’exemple ci-dessous supposera un volume de départ de 2 L de culture. - Continuer à faire circuler le milieu conditionné jusqu’à ce que son volume ait été réduit à ~ 100-200 mL. Passez à des navires plus petits au besoin. Diluer 2 fois avec du PBS et continuer à circuler avec la pompe, en se concentrant jusqu’à 75-100 mL. Diluer 2 fois avec du PBS et continuer à circuler jusqu’à un volume final de 25 mL. Diluer 2 fois avec du PBS et continuer à circuler jusqu’à <10 mL.
- Soulevez le tube d’alimentation hors du réservoir d’échantillon et continuez à pomper pour purger le filtre et récupérer la quantité maximale d’échantillon.
REMARQUE : Il s’agit d’un point d’arrêt potentiel. - Transférer l’échantillon concentré dans un tube conique et conserver toute la nuit à 4 °C si désiré. Vous pouvez également poursuivre le protocole.
- Déplacer l’échantillon concentré vers un dispositif d’ultrafiltration centrifuge MWCO d’une capacité de 15 mL et 100 kDa. Centrifuger dans un rotor à godet oscillant à 4 °C et 2 000 × g pendant des intervalles de 15 à 30 minutes jusqu’à ce que le volume du milieu dans le réservoir supérieur ait été concentré à <2 mL.
REMARQUE : Il s’agit d’un point d’arrêt potentiel. - Transférer le milieu concentré dans un tube à faible teneur en protéines et conserver à 4 °C pendant la nuit, en poursuivant le protocole le lendemain si désiré.
- Assembler un circuit de filtration avec un tube #16 à faible liaison/lixiviation, des adaptateurs de tuyau de 1/8 pouce vers Luer, le dispositif TFF et une pompe péristaltique, comme indiqué à la figure supplémentaire S2.
- Si vous travaillez avec des volumes importants >100 mL, passez à l’étape 2.2.2. Si vous travaillez avec des volumes de ~100 mL, chargez 90 mL de milieu de culture filtré sur le réservoir d’un dispositif d’ultrafiltration centrifuge de seuil de poids moléculaire (MWCO) de capacité respective de 100 kDa à l’aide de pipettes sérologiques. Toujours équilibrer avec un dispositif d’ultrafiltration correspondant et centrifuger dans un rotor de godet oscillant à 4 °C et 2 000 × g pendant des intervalles de 15 à 30 minutes, jusqu’à ce que le volume du fluide dans le réservoir supérieur ait été concentré à <0,5 mL.
- Nettoyage du périphérique TFF (facultatif)
REMARQUE: Le taux de filtration diminue lorsque le dispositif TFF commence à « se boucher » pendant le processus (encrassement). Si nécessaire, le dispositif de filtration peut être nettoyé pour faciliter la filtration d’échantillons supplémentaires dans le même cycle de purification. Bien que théoriquement possible, un filtre TFF nettoyé n’a pas été utilisé pour un cycle de purification différent afin d’éviter la contamination croisée.- Pour nettoyer, retirez tous les tubes et bouchons du dispositif TFF et vidangez tout liquide résiduel.
- Utiliser la pompe péristaltique et le tube pour inonder les compartiments intérieur et extérieur du dispositif TFF (c.-à-d. via les orifices parallèles et perpendiculaires du modèle indiqué dans le tableau des matériaux) avec ~100 mL d’eau distillée. Retirez tous les tubes/bouchons et vidangez le dispositif TFF.
- Boucher les orifices externes (perpendiculaires, filtrat) et faire circuler 250 mL d’éthanol à 20 % dans de l’eau distillée à >200 mL/min pendant 10 min dans le compartiment intérieur. Égoutter, inonder d’eau distillée et égoutter à nouveau comme ci-dessus.
- Faire circuler 250 mL de solution de NaOH frais 0,5 N pendant 30 min dans le compartiment intérieur et égoutter à nouveau.
- Rebrancher tous les tubes et les bouchons aux orifices d’entrée, de sortie et de filtrat, comme dans la figure supplémentaire S2, et faire circuler à nouveau la solution de NaOH 0,5 N jusqu’à ce qu’un volume de NaOH > surface filtrante de 1 mL/cm2 pénètre à travers la membrane filtrante et soit recueilli sous forme de filtrat ou de déchets.
- Rincez l’appareil TFF avec de l’eau distillée comme ci-dessus. Utilisez immédiatement l’appareil TFF ou inondez-le de ~100 mL d’éthanol à 20 % et conservez-le toute la nuit à 4 °C.
REMARQUE : S’il est conservé dans de l’éthanol, assurez-vous de égoutter, de rincer à l’eau, de vider et de faire circuler 250 ml de PBS dans l’appareil jusqu’à ce qu’un volume de surface filtrante de >1 mL/cm2 pénètre à travers la membrane filtrante et soit recueilli sous forme de filtrat ou de déchets pour éliminer l’éthanol résiduel avant le traitement de l’échantillon.
- Chromatographie d’exclusion de taille (SEC)
REMARQUE: SEC est utilisé pour augmenter la pureté des VE et éliminer la protéine non vésiculeuse.- Utiliser une petite colonne SEC (volume de lit de 10 mL) pour isoler les VE de <100 mL de matière de départ et une colonne plus grande (47 mL de volume de lit) pour isoler les VE de >100 mL de matière de départ.
Remarque : L’exemple ci-dessous répertorie les volumes pour la plus grande colonne, avec les volumes pour la plus petite colonne entre parenthèses. - Amenez la colonne SEC et le PBS à température ambiante pendant plusieurs heures. Stabiliser la colonne SEC en position verticale à l’aide d’un support de laboratoire et d’un support standard. Vous pouvez également utiliser des supports de colonne de chromatographie commerciale.
- Avant de vous connecter à la colonne SEC, hydratez le réservoir d’échantillon en laissant 5 mL de PBS s’écouler à travers la fritte et dans un conteneur à déchets. Dévissez le bouchon d’entrée de la colonne SEC, ajoutez 2 mL de PBS au réservoir d’échantillon et connectez délicatement le réservoir à la colonne pendant que le PBS s’égoutte à travers la fritte (ne s’applique pas aux petites colonnes SEC).
REMARQUE: Cette étape précédente empêche les bulles d’air de se coincer en haut de la colonne SEC. Si de l’air est emprisonné, retirez le réservoir, appuyez sur la colonne pour extraire la bulle d’air et répétez la procédure de connexion. Pour la plus petite colonne, il suffit de décapsuler le haut de la colonne SEC et de fixer la trémie d’échantillon. - Ajouter 47 mL (10 mL) de PBS dans le réservoir d’échantillon et déboucher le bas de la colonne SEC. Laissez tout le tampon d’échantillon chargé circuler à travers la colonne pour l’équilibre. Jetez le flux continu.
- Charger un maximum de 2 mL (0,5 mL) d’échantillon sur le réservoir d’échantillon, jeter l’écoulement continu et laisser l’échantillon pénétrer complètement dans la colonne.
- Ajouter immédiatement du PBS au réservoir ou à la trémie à un volume de 14,25 mL moins le volume de l’échantillon (3 mL moins le volume de l’échantillon, pour la petite colonne). Laissez la solution s’écouler à travers la colonne et ignorez cette quantité égale au volume vide de la colonne.
REMARQUE : Pour un échantillon typique de 2 mL, la quantité de PBS à ajouter au réservoir ou à la trémie d’échantillon sera de 12,25 mL. - Placez un microtube de 2 mL à faible liaison directement sous la colonne SEC. Ajouter immédiatement 2 mL (0,5 mL) de PBS dans le réservoir de l’échantillon et le laisser entrer dans la colonne. Étiquetez ces 2 premiers mL (0,5 mL) d’écoulement continu comme fraction 1. Continuer d’ajouter 2 mL (0,5 mL) à la fois dans le réservoir d’échantillon pour prélever chaque fraction subséquente.
REMARQUE: La plupart des VE bactériens éluent dans les 5 premières fractions. Lors de l’optimisation, les 12 premières fractions ont été collectées. - Conserver les fractions à 4 °C pour un stockage à court terme (jours) ou à -80 °C pour un stockage à long terme.
- Nettoyage et stockage des colonnes SEC réutilisables
REMARQUE: Les colonnes SEC décrites dans ce protocole peuvent être réutilisées jusqu’à 5 fois selon le fabricant. Si le débit des colonnes SEC diminue après <5 utilisations, le fabricant recommande de centrifuger les échantillons concentrés à 10 000 x g pendant 10 minutes pour éliminer les agrégats avant la SEC. Chargez ensuite le surnageant de cette centrifugation sur la colonne SEC pour l’isolation EV.- Pour nettoyer et stocker la colonne SEC après chaque utilisation, ajouter 2 mL (0,5 mL) de 0,5 M de NaOH et lui permettre de pénétrer complètement dans la colonne. Faites passer 100 ml (20 ml) d’éthanol à 20 % dans la colonne et conservez-le à 4 °C jusqu’à la prochaine utilisation. Avant la prochaine utilisation, équilibrer l’éthanol à température ambiante comme ci-dessus et l’échanger avec un tampon PBS en faisant passer 150 mL (30 mL) de PBS dans la colonne.
- Pour nettoyer et réutiliser immédiatement la colonne SEC après chaque utilisation, ajouter 2 mL (0,5 mL) de 0,5 M de NaOH et laisser entrer complètement dans la colonne. Appliquez environ 150 ml (30 ml) de tampon PBS pour éliminer le NaOH. Lorsque le pH de l’éluat est égal au PBS (~7), un nouvel échantillon peut être chargé.
- Utiliser une petite colonne SEC (volume de lit de 10 mL) pour isoler les VE de <100 mL de matière de départ et une colonne plus grande (47 mL de volume de lit) pour isoler les VE de >100 mL de matière de départ.
3. Contrôle de la qualité de la préparation des véhicules électriques
- Test de stérilité
REMARQUE: Comme ces VE proviennent de cultures bactériennes, il est essentiel d’assurer la stérilité avant l’utilisation en aval.- Obtenir 100 μL (20 μL) des fractions à utiliser dans les essais et inoculer 3 mL du milieu utilisé pour cultiver la bactérie source. Culture dans les conditions optimales respectives pendant au moins 3 jours et observation de la turbidité. Alternativement, appliquez les échantillons de fraction sur des plaques de gélose contenant le milieu utilisé pour cultiver les bactéries productrices et recherchez la formation de colonies.
REMARQUE: Si une contamination bactérienne est détectée, il n’est pas recommandé d’utiliser la préparation EV pour l’expérimentation. Répétez plutôt l’isolement, en prenant soin de minimiser le risque de contamination bactérienne en (a) effectuant une centrifugation/filtration suffisante du milieu de culture cellulaire bactérien conditionné, (b) en utilisant des bouteilles, des tubes, des filtres et des colonnes de chromatographie propres, et (c) en utilisant des techniques aseptiques appropriées.
- Obtenir 100 μL (20 μL) des fractions à utiliser dans les essais et inoculer 3 mL du milieu utilisé pour cultiver la bactérie source. Culture dans les conditions optimales respectives pendant au moins 3 jours et observation de la turbidité. Alternativement, appliquez les échantillons de fraction sur des plaques de gélose contenant le milieu utilisé pour cultiver les bactéries productrices et recherchez la formation de colonies.
- Quantification des protéines
REMARQUE : Une trousse de quantification des protéines à haute sensibilité basée sur la fluorescence a été utilisée (voir le tableau des matériaux). Le kit fonctionne avec un fluorimètre exclusif correspondant à des longueurs d’onde d’excitation / émission de 485/590 nm.- Apportez tous les réactifs, étalons et échantillons à température ambiante.
- Préparer un mélange principal de réactif protéique et de tampon en ajoutant 1 μL du réactif à 199 μL de tampon pour chaque échantillon et étalon à doser. À l’aide de tubes PCR à paroi mince de 0,5 mL, ajouter 10 μL standard + 190 μL de mélange maître à chaque tube standard.
NOTE: Pour être dans la plage de l’essai, la quantité de chaque fraction à ajouter à chaque tube d’échantillon dépend du rendement en protéines attendu de la purification. En règle générale, 5 μL de chaque fraction + 195 μL de mélange maître ont été utilisés. Le volume final de l’échantillon + mélange maître doit être de 200 μL. - Vortex des tubes d’essai et incuber pendant au moins 15 minutes à température ambiante dans l’obscurité.
- Mesurez les étalons sur le fluorimètre exclusif approprié (voir le tableau des matériaux) en sélectionnant l’option Dosage des protéines à l’aide des boutons fléchés et en appuyant sur le bouton GO pour confirmer. Suivez les instructions à l’écran, insérez chaque tube standard et appuyez sur GO.
- Insérer le tube d’échantillon expérimental; appuyez sur GO pour lire; et notez le résultat affiché, qui est la concentration réelle de protéines dans le mélange tampon/échantillon de l’essai. Pour obtenir la concentration en protéines dans l’échantillon, utilisez les touches fléchées pour sélectionner l’option Calculer la concentration de l’échantillon, appuyez sur GO et utilisez les touches fléchées pour sélectionner le volume d’échantillon ajouté au tampon de dosage pour l’échantillon donné. Appuyez sur GO et enregistrez la concentration de protéines de l’échantillon. Répétez cette étape pour chaque échantillon à analyser.
- Comptage des particules et distribution granulométrique
REMARQUE : La détection microfluidique des impulsions résistives (MRPS) a été utilisée pour quantifier la concentration et la distribution de la taille des VE.- Diluer les échantillons dans du PBS supplémenté avec du Tween-20 à 1 % qui a été filtré à travers un filtre à seringue de 0,02 μm à une concentration de protéines d’environ 0,1 μg/mL.
NOTE: L’objectif de la dilution est d’atteindre une concentration de particules prévue de l’ordre de 10à 10 particules/mL dans les fractions contenant des VE. Il peut être nécessaire de déterminer empiriquement la dilution optimale. Peu de VE sont attendus pour les fractions ultérieures (au-delà de la fraction 6). Ainsi, la concentration de particules sera probablement de <10à 10 particules/mL malgré l’analyse à de faibles dilutions. - Chargez 3 μL de chaque échantillon dans la cartouche microfluidique jetable à l’aide d’une micropipette, insérez la cartouche dans l’instrument MRPS et appuyez sur le bouton métallique avec un bord éclairé bleu.
- Cliquez sur Go! sur le logiciel d’acquisition et attendez que l’échantillon soit analysé par l’instrument. Acquérir 1 000 à 10 000 événements de particules pour minimiser l’erreur statistique technique d’analyse. À ce stade, cliquez sur Arrêter et Terminer l’exécution pour terminer l’acquisition de l’exemple.
REMARQUE: Avec les fichiers de données brutes, l’instrument produit une feuille de calcul récapitulative répertoriant la concentration de particules dans l’échantillon. Corriger cette valeur en fonction de la dilution de l’échantillon effectuée. - À l’aide d’un logiciel d’analyse, chargez les données brutes et générez des graphiques personnalisés de la distribution par taille.
- Diluer les échantillons dans du PBS supplémenté avec du Tween-20 à 1 % qui a été filtré à travers un filtre à seringue de 0,02 μm à une concentration de protéines d’environ 0,1 μg/mL.
4. Stockage des véhicules électriques
- Fractions individuelles ou groupées aliquotes à 25-50% de la taille de la fraction individuelle (selon la taille de la colonne utilisée) dans des tubes à faible liaison aux protéines et conserver à -80 ° C pour éviter les cycles de congélation-décongélation.
REMARQUE : Différentes applications peuvent nécessiter des aliquotes plus petites ou plus grandes selon la quantité attendue utilisée dans chaque expérience. Cela devra être déterminé empiriquement. Les fractions ne contenant pas de VE peuvent être éliminées si elles ne sont pas applicables aux objectifs de la recherche.
5. Microscopie électronique à transmission
- Coloration négative
- Ajouter 5 μL de l’échantillon EV à la grille de 400 mailles en cuivre revêtu de carbone et incuber à température ambiante pendant 10 min. Laver le côté de l’échantillon avec 5 gouttes de tampon Tris de 5 mM (pH 7,1) puis avec 5 gouttes d’eau distillée.
- Colorer le côté de l’échantillon avec 5 gouttes d’acétate d’uranyle à 2%. Effacez toute quantité supplémentaire de tache avec du papier filtre et laissez la grille sécher complètement pendant plusieurs heures ou toute la nuit. Visualisez les échantillons avec un microscope électronique fonctionnant à 80 kV.
- Marquage Immunogold
- Appliquer 10 μL de la suspension EV sur une grille formvar/carbone 400 mailles et incuber à température ambiante pendant 1 h. Laver la grille dans PBS trois fois, puis appliquer 4% de paraformaldéhyde pendant 10 minutes pour fixer l’échantillon. Lavez les grilles cinq fois avec PBS.
- Bloquer la grille avec trois lavages de PBS contenant 0,1% d’albumine sérique bovine (BSA). Ensuite, appliquez 10 μL d’un anticorps primaire pendant 40 minutes à température ambiante (ici, 1 μg/mL d’anticorps nluc). Laver trois fois de plus avec du PBS contenant 0,1% de BSA.
- Ajouter 10 μL d’anticorps secondaire marqué à l’or dans la grille et incuber pendant 40 minutes à température ambiante. Lavez les grilles trois fois avec PBS.
REMARQUE: Ici, un anticorps anti-souris de chèvre conjugué avec des nanoparticules d’or de 10 nm a été utilisé après dilution 1:10 dans le tampon de blocage. Si le marquage de l’or obscurcit la visualisation de l’EV, des anticorps secondaires avec des nanoparticules d’or plus petites (par exemple 5 nm) peuvent être utilisés à la place. - Post-fixer la grille avec 10 μL de glutaraldéhyde à 2,5% pendant 10 minutes à température ambiante. Lavez trois fois dans PBS. Effectuer une coloration négative avec de l’acétate d’uranyle à 2 % (10 μL) pendant 15 min. Incorporer les échantillons dans 10 μL d’acétate d’uranyle à 0,5 % et de solution de méthylcellulose à 0,13 % pendant 10 min.
- Laisser sécher les grilles d’échantillons pendant la nuit à température ambiante avant d’imager au microscope électronique.
- Sur le logiciel d’acquisition de microscope, déterminez empiriquement l’exposition pour obtenir la qualité optimale de l’image (par exemple, 0,80851 s dans cette configuration particulière) et ajustez-la en tapant cette valeur dans la case d’option du temps d’exposition . Sélectionnez l’option 80 kV , puis cliquez sur Démarrer l’acquisition pour capturer l’image.
Access restricted. Please log in or start a trial to view this content.
Résultats
Pour évaluer quelles fractions chromatographiques SEC ont été enrichies pour les VE, la colonne SEC a été chargée de 2 mL de milieu de culture conditionné à E. coli MP1 qui avait été concentré 1 000 fois par TFF, et des fractions séquentielles ont été recueillies. À l’aide de MRPS, il a été constaté que les fractions 1 à 6 contenaient le plus de VE (figure 2A). Les fractions suivantes contenaient très peu de VE, comprenant au lieu de protéines sans VE (
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans le protocole ci-dessus, une méthode est décrite qui est évolutive et isole de manière fiable les VE de diverses bactéries gram-négatives/positives et aérobies/anaérobies. Il a plusieurs points d’arrêt potentiels tout au long de la procédure, bien qu’il soit préférable d’éviter de prendre plus de 48 heures pour isoler les VE des milieux de culture bactériens conditionnés.
Tout d’abord, il consiste à cultiver des bactéries pour générer un milieu de culture bactér...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
La recherche décrite ci-dessus a été soutenue par la subvention de formation NIH TL1 TR002549-03. Nous remercions les Drs John C. Tilton et Zachary Troyer (Case Western Reserve University) d’avoir facilité l’accès à l’instrument d’analyse granulométrique; Lew Brown (Spectradyne) pour l’assistance technique à l’analyse des données de distribution granulométrique; Dr David Putnam de l’Université Cornell pour avoir fourni le plasmide14 pClyA-GFP; et le Dr Mark Goulian de l’Université de Pennsylvanie pour nous avoir fourni le MP113 de E. coli.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.5 mL flat cap, thin-walled PCR tubes | Thermo Scientific | 3430 | it is important to use thin-walled PCR tubes to obtain accurate readings with Qubit |
| 16% Paraformaldehyde (formaldehyde) aqueous solution | Electron microscopy sciences | 15700 | |
| 250 mL Fiberlite polypropylene centrifuge bottles | ThermoFisher | 010-1495 | |
| 500 mL Fiberlite polypropylene centrifuge bottles | ThermoFisher | 010-1493 | |
| 65 mm Polypropylene Round-Bottom/Conical Bottle Adapter | Beckman Coulter | 392077 | Allows Vivacell to fit in rotor |
| Akkermansia mucinophila | ATCC | BAA-835 | |
| Amicon-15 (100 kDa MWCO) | MilliporeSigma | UFC910024 | |
| Avanti J-20 XPI centrifuge | Beckman Coulter | No longer sold by Beckman. Avanti J-26XP is closest contemporary model. | |
| Bacteroides thetaiotaomicron VPI 5482 | ATCC | 29148 | |
| Bifidobacterium breve | NCIMB | B8807 | |
| Bifidobacterium dentium | ATCC | 27678 | |
| Brain Heart infusion (BHI) broth | Himedia | M2101 | After autoclaving, Both BHI broth and agar were introduced into the anaerobic chamber, supplemented with Menadione (1 µg/L), hematin (1.2 µg/L), and L-Cysteine Hydrochloride (0.05%). They were then incubated for at least 24 h under anaerobic conditions before inoculation with the anaerobic bacterial strains. |
| C-300 microfluidics cartridge | Spectradyne | ||
| Chloramphenicol | MP Biomedicals | ICN19032105 | |
| Electron microscope | FEI company | Tecnai G2 SpiritBT | |
| Escherichia coli HST08 (Steller competent cells) | Takara | 636763 | |
| Escherichia coli MP1 | Dr. Mark Goulian (gift) | commensal bacteria derived from mouse gut | |
| Fiberlite 500 mL to 250 mL adapter | ThermoFisher | 010-0151-05 | used with Fiberlite rotor to enable 250 mL bottles to be used for smaller size of starting bacterial culture |
| Fiberlite fixed-angle centrifuge rotor | ThermoFisher | F12-6x500-LEX | fits 6 x 500 mL bottles |
| Formvar Carbon Film 400 Mesh, Copper | Electron microscopy sciences | FCF-400-CU | |
| Glutaraldehyde (EM-grade, 10% aqeous solution) | Electron microscopy sciences | 16100 | |
| Hematin | ChemCruz | 207729B | Stock solution was made in 0.2 M L-histidine solution as 1.2 mg/mL |
| Infinite M Nano+ Microplate reader | Tecan | This equibment was used to measure the mCherry fluorescence | |
| In-Fusion HD Cloning Plus | Takara | 638909 | For cloning of the PCR fragements into the PCR-lineraized vectors |
| JS-5.3 AllSpin Swinging-Bucket Rotor | Beckman Coulter | 368690 | |
| Lauria Bertani (LB) broth, Miller | Difco | 244620 | |
| L-Cysteine Hydrochloride | J.T. Baker | 2071-05 | It should be weighed and added directly to the autoclaved BHI media inside the anaerobic chamber |
| Masterflex Fitting, Polypropylene, Straight, Female Luer to Hose Barb Adapter, 1/8" ID; 25/PK | cole-parmer - special | HV-30800-08 | connection adapters for filtration tubing circuit |
| Masterflex Fitting, Polypropylene, Straight, Male Luer to Hose Barb Adapter, 1/8" ID; 25/PK | cole-parmer - special | HV-30800-24 | connection adapters for filtration tubing circuit |
| Masterflex L/S Analog Variable-Speed Console Drive, 20 to 600 rpm | Masterflex | HV-07555-00 | |
| Masterflex L/S Easy-Load Head for Precision Tubing, 4-Roller, PARA Housing, SS Rotor | Masterflex | EW-07514-10 | |
| Masterflex L/S Precision Pump Tubing, PharmaPure, L/S 16; 25 ft | Cole Palmer | EW-06435-16 | low-binding/low-leaching tubing |
| Menadione (Vitamin K3) | MP | 102259 | Stock solution was made in ethanol as 1 mg/mL |
| MIDIKROS 41.5CM 100K MPES 0.5MM FLL X FLL 1/PK | Repligen | D04-E100-05-N | TFF device we have used to filter up to 2 L of E. coli culture supernatant |
| Nano-Glo Luciferase Assay System | Promega | N1110 | This assay kit was used to measure the luminescence of the nluc reporter protein |
| NanoLuc (Nluc) Luciferase Antibody, clone 965808 | R&D Systems | MAB10026 | |
| nCS1 microfluidics resistive pulse sensing instrument | Spectradyne | ||
| nCS1 Viewer | Spectradyne | Analysis software for particle size distribution | |
| OneTaq 2x Master Mix with Standard Buffer | NEB | M0482 | DNA polymerase master mix used to perform the routine PCR reactions for colony checking |
| Protein LoBind, 2.0 mL, PCR clean tubes | Eppendorf | 30108450 | |
| Q5 High-Fidelity 2x Master Mix | NEB | M0492 | DNA polymerase master mix used to perform the PCR reactions needed for cloning |
| qEV original, 35 nm | Izon | maximal loading volume of 0.5 mL | |
| qEV rack | Izon | for use with the qEV-original SEC columns | |
| qEV-2, 35 nm | Izon | maximal loading volume of 2 mL | |
| Qubit fluorometer | ThermoFisher | Item no longer available. Closest available product is Qubit 4.0 Fluorometer (cat. No. Q33238) | |
| Qubit protein assay kit | ThermoFisher | Q33211 | Store kit at room temperature. Standards are stored at 4 °C. |
| Sorvall Lynx 4000 centrifuge | ThermoFisher | 75006580 | |
| SpectraMax i3x Microplate reader | Molecular Devices | This equipment was used to measure the nanoluciferase bioluminescence | |
| Stericup Quick-release-GP Sterile Vacuum Filtration system (150, 250, or 500 mL) | MilliporeSigma | S2GPU01RE S2GPU02RE S2GPU05RE | One or multiple filters can be used to accommodate working volumes. In our experience, you can filter twice the volume listed on the product size. |
| Uranyl acetate | Electron microscopy sciences | 22400 | |
| Vinyl anaerobic chamber | Coy Lab | ||
| Vivacell 100, 100,000 MWCO PES | Sartorius | VC1042 | |
| Whatman Anotop 10 Plus syringe filters (0.02 micron) | MilliporeSigma | WHA68093002 | to filter MRPS diluent |
Références
- Yanez-Mo, M., et al. Biological properties of extracellular vesicles and their physiological functions. Journal of Extracellular Vesicles. 4, 27066(2015).
- Chatterjee, S. N., Das, J. Electron microscopic observations on the excretion of cell-wall material by Vibrio cholerae. Journal of General Microbiology. 49 (1), 1-11 (1967).
- Ciofu, O., Beveridge, T. J., Kadurugamuwa, J., Walther-Rasmussen, J., Hoiby, N. Chromosomal beta-lactamase is packaged into membrane vesicles and secreted from Pseudomonas aeruginosa. Journal of Antimicrobial Chemotherapy. 45 (1), 9-13 (2000).
- Yonezawa, H., et al. Outer membrane vesicles of Helicobacter pylori TK1402 are involved in biofilm formation. BMC Microbiology. 9, 197(2009).
- Mashburn, L. M., Whiteley, M. Membrane vesicles traffic signals and facilitate group activities in a prokaryote. Nature. 437 (7057), 422-425 (2005).
- Kato, S., Kowashi, Y., Demuth, D. R. Outer membrane-like vesicles secreted by Actinobacillus actinomycetemcomitans are enriched in leukotoxin. Microbial Pathogenesis. 32 (1), 1-13 (2002).
- Petousis-Harris, H., et al. Effectiveness of a group B outer membrane vesicle meningococcal vaccine against gonorrhoea in New Zealand: a retrospective case-control study. Lancet. 390 (10102), 1603-1610 (2017).
- Kim, O. Y., et al. Bacterial outer membrane vesicles suppress tumor by interferon-gamma-mediated antitumor response. Nature Communications. 8 (1), 626(2017).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750(2018).
- Consortium, E. -T., et al. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14 (3), 228-232 (2017).
- Watson, D. C., et al. Efficient production and enhanced tumor delivery of engineered extracellular vesicles. Biomaterials. 105, 195-205 (2016).
- Watson, D. C., et al. Scalable, cGMP-compatible purification of extracellular vesicles carrying bioactive human heterodimeric IL-15/lactadherin complexes. Journal of Extracellular Vesicles. 7 (1), 1442088(2018).
- Lasaro, M., et al. Escherichia coli isolate for studying colonization of the mouse intestine and its application to two-component signaling knockouts. Journal of Bacteriology. 196 (9), 1723-1732 (2014).
- Kim, J. Y., et al. Engineered bacterial outer membrane vesicles with enhanced functionality. Journal of Molecular Biology. 380 (1), 51-66 (2008).
- Beveridge, T. J. Structures of gram-negative cell walls and their derived membrane vesicles. Journal of Bacteriology. 181 (16), 4725-4733 (1999).
- Reimer, S. L., et al. Comparative analysis of outer membrane vesicle isolation methods with an Escherichia coli tolA mutant reveals a hypervesiculating phenotype with outer-inner membrane vesicle content. Frontiers in Microbiology. 12, 628801(2021).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon