Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement de vecteurs de thérapie génique de nouvelle génération par l’ingénierie, le codage à barres et le criblage de variantes de capside du virus adéno-associé (AAV)
Dans cet article
Résumé
Génération de bibliothèque d’affichage de peptides AAV et validation ultérieure par le codage à barres de candidats ayant de nouvelles propriétés pour la création d’AAV de nouvelle génération.
Résumé
Les vecteurs de livraison de gènes dérivés du virus adéno-associé (AAV) sont l’un des outils les plus prometteurs pour le traitement des maladies génétiques, comme en témoignent les données cliniques encourageantes et l’approbation de plusieurs thérapies géniques AAV. Deux raisons majeures du succès des vecteurs AAV sont (i) l’isolement préalable de divers sérotypes viraux naturels ayant des propriétés distinctes, et (ii) la mise en place ultérieure de technologies puissantes pour leur ingénierie moléculaire et leur réutilisation à haut débit. Le potentiel de ces techniques est récemment mis en œuvre pour coder à barres des capsides AAV sélectionnées au niveau de l’ADN et de l’ARN, permettant leur stratification in vivo complète et parallèle dans tous les principaux organes et types de cellules d’un seul animal. Ici, nous présentons un pipeline de base englobant cet ensemble de voies complémentaires, en utilisant l’affichage peptidique AAV pour représenter l’arsenal diversifié de technologies d’ingénierie de capside disponibles. En conséquence, nous décrivons d’abord les étapes cruciales de la génération d’une bibliothèque d’affichage de peptides AAV pour la sélection in vivo de candidats ayant les propriétés souhaitées, suivie d’une démonstration de la façon de coder à barres les variantes de capside les plus intéressantes pour le criblage secondaire in vivo. Ensuite, nous illustrons la méthodologie de création de bibliothèques pour le séquençage de nouvelle génération (NGS), y compris l’amplification de codes-barres et la ligature d’adaptateurs, avant de conclure par un aperçu des étapes les plus critiques de l’analyse des données NGS. Comme les protocoles rapportés ici sont polyvalents et adaptables, les chercheurs peuvent facilement les exploiter pour enrichir les variantes optimales de capside AAV dans leur modèle de maladie préféré et pour des applications de thérapie génique.
Introduction
La thérapie par transfert de gènes est l’introduction de matériel génétique dans les cellules pour réparer, remplacer ou modifier le matériel génétique cellulaire afin de prévenir, traiter, guérir ou améliorer la maladie. Le transfert de gènes, à la fois in vivo et ex vivo, repose sur différents systèmes d’administration, non viraux et viraux. Les virus ont évolué naturellement pour transduire efficacement leurs cellules cibles et peuvent être utilisés comme vecteurs de livraison. Parmi les différents types de vecteurs viraux utilisés en thérapie génique, les virus adéno-associés ont été de plus en plus utilisés, en raison de leur absence de pathogénicité, de leur innocuité, de leur faible immunogénicité et, surtout, de leur capacité à maintenir une expression non intégratrice à long terme 1,2,3. La thérapie génique AAV a donné lieu à des réalisations considérables au cours de la dernière décennie; trois thérapies ont été approuvées par l’Agence européenne des médicaments et la Food and Drug Administration des États-Unis pour une utilisation chez l’homme 3,4. Plusieurs essais cliniques sont également en cours pour traiter diverses maladies, comme l’hémophilie, les maladies musculaires, cardiaques et neurologiques, comme on l’a vu ailleurs3. Malgré des décennies de progrès, le domaine de la thérapie génique a connu une série de revers ces dernières années4, surtout des décès dans les essais cliniques5 qui ont été suspendus en raison de toxicités limitant la dose, en particulier pour les tissus massifs, tels que les muscles, ou difficiles à atteindre, tels que le cerveau6.
Les vecteurs AAV actuellement utilisés dans les essais cliniques appartiennent aux sérotypes naturels à quelques exceptionsprès 1. L’ingénierie AAV offre la possibilité de développer des vecteurs avec une spécificité et une efficacité supérieures pour les organes ou les cellules. Au cours des deux dernières décennies, plusieurs approches ont été appliquées avec succès, telles que l’affichage peptidique, l’échange de boucles, le brassage de l’ADN de la capside, la PCR sujette aux erreurs et la conception ciblée, pour générer des variantes AAV individuelles ou des bibliothèques de celles-ci avec diverses propriétés7. Ceux-ci sont ensuite soumis à de multiples cycles d’évolution dirigée pour sélectionner les variantes en leur sein avec les propriétés souhaitées, comme examiné ailleurs 1,3. De toutes les stratégies d’évolution de la capside, les bibliothèques AAV d’affichage peptidique ont été les plus largement utilisées, en raison de certaines propriétés uniques: elles sont relativement faciles à générer et peuvent atteindre une grande diversité et un séquençage à haut débit, ce qui permet de suivre leur évolution.
Les premières bibliothèques AAV d’insertion de peptides réussies ont été décrites il y a près de 20 ans. Dans l’un des premiers, Perabo et al.8 ont construit une bibliothèque de capsides AAV2 modifiées, dans laquelle un pool d’oligonucléotides générés aléatoirement a été inséré dans un plasmide à une position qui correspond à l’acide aminé 587 de la protéine de capside VP1, dans le triple axe dépassant de la capside. En utilisant la co-infection par l’adénovirus, la bibliothèque AAV a été développée à travers plusieurs cycles de sélection, et les variantes finales reciblées se sont révélées capables de transduire des lignées cellulaires réfractaires à l’AAV2parental 8. Peu de temps après, Müller et al.9 ont introduit le système en deux étapes pour la production en bibliothèque, une amélioration significative du protocole. Initialement, la bibliothèque plasmidique, ainsi qu’un plasmide auxiliaire adénoviral, sont utilisés pour produire une bibliothèque AAV qui contient des capsides chimériques. Cette bibliothèque de navettes AAV est utilisée pour infecter des cellules à faible multiplicité d’infection (MOI), dans le but d’introduire un génome viral par cellule. La co-infection par l’adénovirus assure la production d’AAV avec un génome et une capside9 correspondants. Environ une décennie plus tard, Dalkara10 a utilisé l’évolution dirigée in vivo pour créer la variante 7m8. Cette variante a une insertion de 10 acides aminés (LALGETTRPA), dont trois agissent comme des agents de liaison, et cible efficacement la rétine externe après injection intravitréenne10. Cette capside artificielle est une réussite exceptionnelle, car c’est l’une des rares capsides modifiées à se rendre à la clinique jusqu’àprésent11.
Le domaine a connu un deuxième coup de pouce avec l’introduction de techniques de séquençage de nouvelle génération (NGS). Deux publications d’Adachi et al.12 en 2014 et de Marsic et al.13 en 2015, ont montré la puissance du NGS pour suivre la distribution des bibliothèques de capsides AAV à code-barres avec une grande précision. Quelques années plus tard, le NGS des régions à code-barres a été adapté à la région d’insertion peptidique pour suivre l’évolution de la capside. Körbelin et coll.14 ont effectué un dépistage guidé par NGS pour identifier une capside pulmonaire ciblée basée sur AAV2. L’analyse NGS a permis de calculer trois scores de notation : le score d’enrichissement entre les tours de sélection, le score de spécificité générale pour déterminer la spécificité tissulaire, et enfin le score combiné14. Le laboratoire Gradinaru15 a publié le système d’évolution ciblée AAV basé sur la recombinaison Cre (CREATE) la même année, qui facilite une sélection spécifique au type cellulaire. Dans ce système, la bibliothèque de capsides porte un commutateur Cre-inversible, car le signal polyA est flanqué de deux sites loxP. La bibliothèque AAV est ensuite injectée chez les souris Cre, où le signal polyA est inversé uniquement dans les cellules Cre+, fournissant le modèle pour la liaison d’une amorce de PCR inverse avec l’amorce directe dans le gène de la capside. Ce sauvetage PCR très spécifique a permis l’identification de l’AAV-PHP. Variante B qui peut traverser la barrière hémato-encéphalique15. Ce système a ensuite évolué pour devenir M-CREATE (Multiplexed-CREATE), dans lequel NGS et la génération de bibliothèques synthétiques ont été intégrées dans le pipeline16.
Une version améliorée à base d’ARN de ce système du laboratoire Maguire17, iTransduce, permet de sélectionner au niveau de l’ADN des capsides qui transduisent fonctionnellement les cellules et expriment leurs génomes. Le génome viral de la bibliothèque d’affichage peptidique comprend un gène Cre sous le contrôle d’un promoteur ubiquitaire et le gène de la capside sous le contrôle du promoteur p41. La bibliothèque est injectée chez des souris qui ont une cassette loxP-STOP-loxP en amont de tdTomato. Les cellules transduites avec des variantes AAV qui expriment le génome viral et donc Cre expriment tdTomato et, en combinaison avec des marqueurs cellulaires, peuvent être triées et sélectionnées17. De même, Nonnenmacher et al.18 et Tabebordbar et al.19 ont placé la bibliothèque de gènes de la capside sous le contrôle de promoteurs spécifiques aux tissus. Après injection dans différents modèles animaux, l’ARN viral a été utilisé pour isoler les variantes de capside.
Une autre approche consiste à utiliser le code-barres pour baliser les bibliothèques de capsides. Le laboratoire Björklund20 a utilisé cette approche pour les bibliothèques de capsides d’insertion de peptides à code-barres et a développé l’évolution rationnelle du vecteur AAV à code-barres (BRAVE). Dans un plasmide, la cassette Rep2Cap est clonée à côté d’un transgène à code-barres à répétitions terminales inversées (ITR) exprimant une protéine fluorescente jaune (YFP). En utilisant des sites loxP entre la fin du bouchon et le début du code-barres, une recombinaison Cre in vitro génère un fragment suffisamment petit pour le NGS, permettant ainsi l’association de l’insertion peptidique avec le code-barres unique (table de consultation, LUT). La production d’AAV est réalisée à l’aide de la bibliothèque plasmidique et les codes-barres exprimés dans l’ARNm sont criblés après application in vivo , toujours avec NGS20. Lorsque les bibliothèques de capsides comprennent des variantes de l’ensemble du gène de la capside (c.-à-d. bibliothèques mélangées), le séquençage à lecture longue doit être utilisé. Plusieurs groupes ont utilisé des codes-barres pour étiqueter ces diverses bibliothèques, ce qui permet à NGS d’avoir une profondeur de lecture plus élevée. Le laboratoire Kay21 a étiqueté des bibliothèques mélangées de capsides très diverses avec des codes-barres en aval du signal polyA du capuchon . Dans un premier temps, une bibliothèque de plasmides à code-barres a été générée et la bibliothèque de gènes de capside mélangée y a été clonée. Ensuite, une combinaison de MiSeq (lecture courte, profondeur de lecture plus élevée) et PacBio (lecture longue, profondeur de lecture inférieure) NGS ainsi que le séquençage Sanger a été utilisée pour générer leur LUT21. En 2019, Ogden et ses collègues du laboratoireChurch 22 ont délimité l’aptitude de la capside AAV2 à plusieurs fonctions à l’aide de bibliothèques comportant des mutations, des insertions et des suppressions ponctuelles dans chaque position, ce qui a finalement permis une conception guidée par machine. Pour la génération de la bibliothèque, des fragments plus petits du gène de la capside ont été synthétisés, marqués avec un code-barres, séquencés de nouvelle génération, puis clonés dans le gène complet de la capside. Les données NGS ont été utilisées pour générer une LUT. La bibliothèque a ensuite été examinée à l’aide uniquement des codes-barres et du séquençage de lecture courte, ce qui permet une profondeur de lecture plus élevée22.
Les bibliothèques à code-barres ont été principalement utilisées pour examiner un pool de variantes connues, naturelles et artificielles après plusieurs cycles de sélection de bibliothèques de capsides ou indépendamment d’une étude de l’évolution de la capside. L’avantage de ces bibliothèques est la possibilité de cribler plusieurs capsides, tout en réduisant le nombre d’animaux et en minimisant les variations entre les animaux. Les premières études qui ont introduit cette technologie dans le domaine de l’AAV ont été publiées il y a près de dix ans. Le laboratoire Nakai 12 a marqué 191 mutants doubles alanine couvrant les acides aminés 356 à 736 sur le VP1 de AAV9 avec une paire de codes-barres12 nucléotides. À l’aide de NGS, la bibliothèque a été examinée in vivo pour la liaison au galactose et d’autres propriétés12. Marsic et ses collègues ont délimité la biodistribution des variantes AAV en utilisant également une analyse à double barcordée 1 an plus tard13. Une étude plus récente chez des primates non humains a comparé la biodistribution dans le système nerveux central de 29 capsides en utilisant différentes voies d’administration23. Notre laboratoire a récemment publié des criblages de bibliothèque AAV à code-barres de 183 variantes comprenant des AAV naturels et artificiels. Ces criblages au niveau de l’ADN et de l’ARN ont conduit à l’identification d’une variante AAV24 hautement myotrope chez la souris ainsi que d’autres présentant une spécificité de type cellulaire élevée dans le cerveau de souris25.
Ici, nous décrivons la méthodologie utilisée dans ce travail et l’élargissons pour inclure le criblage des bibliothèques d’affichage de peptides AAV. Cela comprend la génération de bibliothèques d’affichage de peptides AAV2, une méthode de PCR numérique par gouttelettes (dd-PCR) pour la quantification, et enfin un pipeline NGS pour analyser les variantes AAV, basé en partie sur les travaux de Weinmann et ses collègues24. Enfin, une description de la génération de bibliothèques AAV à code-barres et du pipeline NGS utilisé dans la même publication est fournie.
Protocole
1. Préparation de la bibliothèque d’affichage de peptides 7-mer aléatoires AAV2
REMARQUE: Pour la préparation d’une bibliothèque d’affichage peptidique aléatoire AAV2, synthétisez les oligonucléotides dégénérés sous forme d’ADN simple brin, convertissez-le en ADN double brin, digérez, ligaturez le plasmide accepteur et électroporate.
- Conception d’oligonucléotides dégénérés
- Ordonner les oligonucléotides dégénérés et éviter le biais de codon. Dans l’oligonucléotide 5' CAGTCGGCCAG AG W GGC (X01)7 GCCCAGGCGGCTGACGAG 3', X01 correspond à 20 codons, chacun codant pour l’un des 20 acides aminés. Le W peut être A ou T, produisant les codons AGA ou AGT, qui codent pour les acides aminés arginine (R) ou sérine (S).
- Commandez l’amorce d’amplification : 5' CTCGTCAGCCGCCTGG 3' (voir la figure 1 pour plus de détails). Cela produit l’insert protéique suivant: R / S G X7. La diversité théorique est calculée comme suit : 1 x 2 x 207 = 2,56 x 109 variantes uniques.
REMARQUE : Il convient de noter que cette diversité peut être limitée par l’efficacité de la transformation.
- Synthèse du second brin
- Resuspendre les deux oligonucléotides (oligonucléotides dégénérés et amorce d’amplification) à une concentration finale de 100 μM avec un tampon TE.
- Pour la réaction de PCR, mettre en place une réaction de 50 μL avec 1 μL de chaque amorce, 10 μL du tampon, 1,5 μL de DMSO, 0,5 μL de dNTP (10 mM), 0,5 μL de polymérase II à démarrage à chaud haute fidélité et 35,5 μL d’eau sans nucléase.
- Transférer la réaction dans un thermocycleur et exécuter une étape de pré-incubation pendant 10 s à 98 °C, suivie de trois cycles de 10 s à 98 °C, 30 s à 59 °C et 10 s à 72 °C, puis 5 min à 72 °C et une dernière étape de refroidissement.
- Purifier la réaction à l’aide d’un kit d’élimination des nucléotides et éluer dans 100 μL d’eau sans nucléase.
- Confirmer l’efficacité de la synthèse du second brin par analyse sur un bioanalyseur (voir Figure 2). Analyser la taille et la pureté de l’insert double brin en chargeant 1 μL de la réaction sur une puce microfluidique à partir d’un kit de réactifs DNA 1000 selon les instructions du fabricant. Ce kit est optimisé pour mesurer la taille et la concentration de fragments d’ADN double brin de 25 à 1 000 bps.
- Resuspendre les deux oligonucléotides (oligonucléotides dégénérés et amorce d’amplification) à une concentration finale de 100 μM avec un tampon TE.
- Digestion de l’insert et du vecteur plasmidique
- Digérer 85 μL de l’insert purifié avec 10 μL de tampon 10x et 5 μL d’enzyme BglI dans un volume de réaction final de 100 μL (voir la figure 1 pour plus de détails). Incuber à 37 °C pendant une nuit. Purifier à l’aide d’un kit d’élimination des nucléotides, éluer dans 50 μL d’eau sans nucléase et quantifier en utilisant le type « Oligo DNA » dans un spectrophotomètre.
- Digérer 10 μg d’un plasmide AAV (pRep2Cap2_PIS)26 (génome viral flanqué ITR) avec 20 μL de tampon 10x et 10 μL d’enzyme SfiI dans un volume de réaction final de 200 μL (voir la figure 1 pour plus de détails). Incuber à 50 °C pendant la nuit. Purifier le vecteur sur un gel d’agarose à 1% à l’aide du kit d’extraction de gel suivi d’une étape de purification supplémentaire à l’aide d’un kit de purification de l’ADN. Quantifier la concentration dans un spectrophotomètre.
- Ligature de l’insert au vecteur
- Ligate 955 ng de vecteur plasmidique avec 45 ng d’insert avec 2 μL de tampon et 2 μL de ligase dans une réaction de ligature de 20 μL. Incuber à 16 °C pendant la nuit, suivi de 10 min à 70 °C pour inactiver la ligase à la chaleur.
- Transformation, calcul de complexité et préparation de la bibliothèque plasmidique
- Purifier la réaction avec un kit de purification de l’ADN en suivant les instructions du fabricant. Éluer la réaction dans environ 80% du volume de départ d’eau exempte de nucléases et stocker sur de la glace pour une transformation ultérieure.
- Transformer les cellules électrocompétentes : décongeler un flacon de cellules électrocompétentes sur de la glace pendant 10 min. Ajoutez ensuite 1 à 2 μL de la réaction de ligature purifiée à 30 μL (un flacon) de cellules électrocompétentes et mélangez en tapotant doucement. Ensuite, pipeter soigneusement le mélange cellule/ADN dans une cuvette d’électroporation à intervalle pré-refroidi de 1 mm sans introduire de bulles d’air.
- Électroporate en utilisant les réglages suivants : 1800 V, 600 Ω et 10 μF. Dans les 10 s suivant l’impulsion d’électroporation, ajouter 970 μL de fluide de récupération préchauffé (muni des cellules électrocompétentes) à la cuvette et mélanger par pipetage. Enfin, transférer les cellules dans un tube microcentrifuge et incuber pendant 1 h à 37 °C à 250 tr/min. Pour obtenir la diversité souhaitée, effectuez 10 à 100 réactions et, après l’incubation, regroupez toutes les réactions dans un seul tube.
- Calculer la diversité en diluant 10 μL des transformations groupées 10, 100 ou 1 000 fois dans le PBS et étaler 100 μL sur des plaques de gélose nutritive contenant l’antibiotique approprié (75 mg/mL d’ampicilline). Incuber les plaques de gélose pendant une nuit à 37 °C, puis compter les colonies sur les plaques de gélose.
- Calculez la diversité théorique comme suit :
Diversité maximale théorique = 10 x facteur de dilution x nombre de colonies x nombre de réactions d’électroporation.
Remarque : Pour confirmer la qualité de la bibliothèque, séquencez au moins 20 colonies par séquençage de Sanger. La plupart des clones doivent contenir un insert, et tous doivent être uniques. - Inoculer 400 à 1 000 mL de milieu LB contenant l’antibiotique approprié avec le reste des transformations regroupées et incuber pendant une nuit à 37 °C, 180 tr/min.
- Préparation de la bibliothèque plasmidique
- À partir de la culture de nuit, préparer un bouillon de glycérol (mélanger des volumes égaux de culture bactérienne et une solution de glycérol à 50% dans de l’eau sans nucléase et congeler à -80 °C) et purifier la bibliothèque plasmidique à l’aide d’un kit maxi plasmidique.
- Production de la bibliothèque virale AAV
- Préparez la bibliothèque virale comme décrit précédemment27. Transfecter la bibliothèque plasmidique (pRep2Cap2_PI, insert peptidique) avec un plasmide adéno-auxiliaire aux cellules HEK293T à l’aide d’un réactif de transfection tel que la polyéthylène (PEI).
- Recueillir les cellules après 3 jours et les soumettre à trois cycles de gel-décongélation. Purifier le lysat viral par ultracentrifugation à gradient de chlorure de césium, suivie d’un échange tampon au PBS, et enfin concentrer les particules virales.
- Titrage vectoriel AAV par dd-PCR
- Diluer en série 2 μL du stock de vecteurs AAV dans 198 μL d’eau exempte de nucléases pour obtenir une dilution finale de 1:106 . Bien mélanger à chaque fois à l’aide d’une pipette de 200 μL. Ajoutez un contrôle sans modèle (NTC) en tant que contrôle négatif.
NOTA: Des dilutions supplémentaires plus faibles ou supérieures peuvent être dosées (1:105-1:107). - Préparez un mélange amorce-sonde 20x. Ajouter 3,6 μL de chacune des amorces de 100 μM (avant et arrière, Rep2 et ITR), 1 μL chacune des sondes dd-PCR de 100 μM (Rep2 et ITR) et 3,6 μL d’eau sans nucléase dans un tube centrifuge de 1,5 mL.
REMARQUE: La bibliothèque AAV est mesurée à l’aide d’un ensemble d’amorces-sondes ciblant les transgènes (Rep2) détectées avec une sonde marquée FAM, et d’un ensemble d’amorces-sondes ciblant ITR détecté avec une sonde marquée HEX. - Préparer une réaction PCR de 22 μL en ajoutant 5,5 μL d’échantillon, 1,1 μL de mélange amorce-sonde 20x, 11 μL de supermix dd-PCR pour sondes (pas de dUTP) et 4,4 μL d’eau sans nucléase. Cela donne des concentrations de 900 nM et 250 nM pour les amorces et la sonde, respectivement.
- Générer les gouttelettes à l’aide d’un générateur de gouttelettes, transférer la réaction sur une plaque de 96 puits, placer la plaque dans un thermocycleur et exécuter une étape de dénaturation pendant 10 min à 94 °C, suivie de 40 cycles de 30 s à 94 °C et de 1 min à 58 °C. Ensuite, inactivez la polymérase à la chaleur pendant 10 min à 98 °C et ajoutez une dernière étape de refroidissement. Lire les réactions dans un lecteur de gouttelettes et procéder à l’analyse28.
- Ouvrez le fichier de plaque dd-PCR enregistré à l’aide du logiciel d’analyse. Utilisez l’outil de seuil dans l’onglet Amplitude 1D (amplitude de fluorescence par rapport au numéro d’événement) pour séparer les gouttelettes négatives et positives pour chaque canal, en utilisant le NTC comme guide, et exportez les données dans un fichier csv.
- Pour calculer la concentration du vecteur, calculez d’abord le facteur de correction CF à l’aide de la formule:
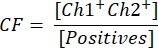
La mucoviscidose détermine la proportion de gouttelettes positives pour le transgène [positifs] qui sont positives pour le transgène et l’ITR [Ch1+ Ch2+], afin d’assurer la détection des particules vectorielles fonctionnelles. La concentration finale du vecteur c peut maintenant être calculée à l’aide de l’équation suivante: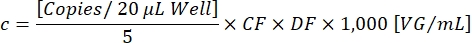
DF est le facteur de dilution (1:105-1:107 tel que déterminé précédemment). Les copies par réaction de 20 μL/puits correspondent à 5 μL de l’échantillon dilué. Le facteur 1 000 corrige l’échelle à VG/mL (génome viral/mL). Un exemple de résultat de titrage est présenté dans le tableau 1 et la figure 3.
- Diluer en série 2 μL du stock de vecteurs AAV dans 198 μL d’eau exempte de nucléases pour obtenir une dilution finale de 1:106 . Bien mélanger à chaque fois à l’aide d’une pipette de 200 μL. Ajoutez un contrôle sans modèle (NTC) en tant que contrôle négatif.
- Analyse de la banque virale AAV par NGS
- Amplifier le fragment d’insertion du peptide 96 nucléotidiques en mettant en place une réaction PCR de 20 μL à l’aide d’un kit de relecture de la polymérase (2x; voir Figure 4). Ajouter 1 μL de stock AAV contenant 1 x 108 vg, 0,5 μL de chacun des 100 μM d’amorce (NGS_forward et NGS_reverse) et 10 μL du mélange d’enzymes à la réaction. Régler le volume final à 20 μL avec de l’eau sans nucléase.
- Transférer la réaction dans un thermocycleur et exécuter une étape de dénaturation pendant 3 min à 98 °C, suivie de 30 à 35 cycles de 10 s à 98 °C, 10 s à 59 °C et 20 s à 72 °C, suivis de 5 min à 72 °C et d’une dernière étape de refroidissement.
- Purifier les échantillons à l’aide d’un kit de purification PCR. Quantifier la concentration dans un spectrophotomètre et utiliser un gel d’agarose à 3% pour vérifier la pureté et la taille du fragment.
- Traiter les fragments PCR à l’aide du système de bibliothèque pour le kit d’échantillons de faible complexité conformément aux instructions du fabricant pour la préparation d’une bibliothèque NGS. Effectuer la réaction de réparation finale avec 30 ng de fragment de PCR, suivie de la ligature de l’adaptateur et de l’amplification de la PCR pendant 10 cycles. Utilisez le kit de purification PCR pour la purification des réactions.
- Traiter les produits finaux sur un bioanalyseur pour vérifier la taille et la pureté, en utilisant un kit de réactifs ADN selon les instructions du fabricant.
- Quantifier les amplicons à l’aide d’un fluoromètre et les mettre en commun. Quantifier à nouveau la bibliothèque NGS finale regroupée sur un fluoromètre (selon les instructions du fabricant) et vérifier la qualité sur un bioanalyseur.
- Séquencez les bibliothèques NGS en mode mono-extrémité (SE), à l’aide d’un kit de sortie élevée de 75 cycles, avec une longueur de lecture de 84 et un index 1 sur 8.
REMARQUE : Le séquençage des exemples de cet article a été effectué dans les installations GeneCore de l’EMBL Heidelberg (http://www.genecore.embl.de/). - Analysez les données de séquençage NGS avec Python 3 et biopython. Les fichiers peuvent être trouvés à https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (alternativement à https://doi.org/10.5281/zenodo.7032215). L’analyse NGS est composée de deux étapes.
- Dans un premier temps, recherchez dans les fichiers de séquences les séquences qui répondent à certains critères (présence de séquences de reconnaissance flanquant le site d’insertion) (voir Figure 4, étape 1.9.8.5.). Cela se fait à l’aide d’un script (Script#1) et d’un fichier de configuration qui fournit les informations nécessaires. Une fois la séquence correcte identifiée, le programme extrait et stocke la séquence dans le fichier de sortie, qui est un fichier txt portant le même nom que le fichier de séquençage.
- La deuxième étape est l’analyse des fichiers de sortie. Les séquences de la bibliothèque commencent par l’un des six nucléotides (AGWggc, W = A / T) dans l’insert de neuf acides aminés. Sur la base de cette séquence de départ, le peptide est traduit. Cela génère les fichiers de sortie qui contiennent les variantes peptidiques (PV).
- Préparez deux dossiers : Script et Data. Dans le dossier Données, copiez les fichiers compressés au format gzip résultant du séquencement. Dans le dossier Script, copiez les fichiers Python suivants : Script#1_DetectionExtraction_JoVE_Py3.py; Fichier Python : Script#2_PV_extraction_and_ranking_Py3.py; Fichier de configuration : Barcode_Script_JoVE.conf ; et fichier de table de correspondance (LUT) : Zuordnung.txt.
- Avant d’exécuter les scripts, modifiez les fichiers suivants dans le dossier Script. Ouvrez le fichier « Zuordnung.txt » et ajoutez deux colonnes séparées par des tabulations, les noms des fichiers gzip (colonne 1) et le nom final souhaité (colonne 2; valeurs séparées par des tabulations).
Remarque : exemples de fichiers txt se trouvent dans le dossier GitHub « PV_analysis_script ». Les fichiers fournis dans le dossier GitHub sont préparés pour l’analyse de trois exemples de données de la bibliothèque ci-dessus : xaa.txt.gz, xab.txt.gz et xac.txt.gz. Les fichiers de sortie sont également fournis. - Modifiez les variables suivantes dans le fichier de configuration « Barcode_Script_JoVE.conf » :
my_dir = « ~/données/ »
filename_sample_file = « ~/Script/Zuordnung.txt »
Les variables spécifiques à la séquence : BCV_size = 27, BCVgauche = TCCAGGGCCAG, BCVdroite = GCCCAGG, BCVloc = 30,marge BCV = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC et BCVloc_revcomp = 41 (voir Figure 4 pour plus de détails). - Utilisez la commande suivante pour appeler la détection et l’extraction de séquence de variantes :
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
REMARQUE: Les résultats sont des fichiers txt avec les séquences d’ADN extraites et leur nombre de lectures. L’en-tête de ce fichier contient des données statistiques (c’est-à-dire le nombre total de lectures et les lectures extraites). Ces données sont transférées vers les fichiers suivants. Ces données txt sont les fichiers d’entrée pour Script # 2, dans lequel les séquences d’ADN sont traduites, classées et analysées. - Effectuez l’extraction et l’analyse PV à l’aide de la commande suivante:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - Analysez les fichiers de sortie texte de Script#2. Les fichiers de sortie de Script#2 sont nommés en utilisant la deuxième colonne de la LUT dans « Zuordnung.txt » avec des extensions basées sur le type d’analyse.
REMARQUE: Assurez-vous que les trois fichiers de sortie contiennent des données statistiques dans les premières lignes (« # de lectures PV valides », « # de lectures PV non valides » et « # de lectures PV uniques »), une première colonne avec l’index de chaque séquence d’ADN des fichiers txt d’entrée (sortie du script # 1) et les colonnes suivantes: (1) « ... analyzed_all.csv »: « Échantillon: » (séquence d’ADN), « # » (nombre de lectures), « Frw ou Rev » (lecture directe ou inverse) et « PVs » (séquence peptidique traduite). Les séquences non valides ont « NA » et « non valide » dans les deux dernières colonnes. (2) "... analyzed_validSeq.csv »: identique au fichier précédent, filtré pour les séquences valides. (3) "... analyzed_PV.csv »: « PVs » (séquence peptidique traduite), « # » (nombre de lectures) et « count » (les comptes frw et rev dans les fichiers précédents sont fusionnés et le nombre est donné 1 ou 2). - Visualisez les fichiers de sortie à l’aide des logiciels disponibles en fonction des besoins de l’utilisateur.
2. Sélection de la bibliothèque d’affichage aléatoire de peptides 7-mer AAV2
- Utiliser la bibliothèque AAV après quantification et contrôle de la qualité (section 1) pour l’évolution dirigée dans un modèle de choix afin de sélectionner de manière itérative les candidats ayant les propriétés souhaitées (voir Figure 5)16,18,21.
REMARQUE : Ces candidats sont ensuite utilisés pour la génération d’une bibliothèque de codes à barres comme décrit ci-dessous dans la section 3.
3. Préparation et analyse de la bibliothèque de capsides AAV à code-barres
REMARQUE: Après l’identification d’un ensemble de capsides AAV potentiellement spécifiques et efficaces dans l’écran d’affichage peptidique, vérifiez la fonctionnalité des séquences peptidiques identifiées et comparez-les avec un ensemble de variantes de capside AAV de référence couramment utilisées ou bien décrites. Pour ce faire, la séquence de capside est insérée dans une construction d’assistance Rep/Cap sans RTI.
- Production d’une bibliothèque AAV à code-barres
- Effectuer la production d’AAV recombinants pour chaque variante de capside en utilisant le système à trois plasmides, comme décrit précédemment24.
REMARQUE: Pour distinguer les différentes variantes de capside, le plasmide transgène rapporteur flanqué d’ITR abrite un code-barres unique de 15 nucléotides de longueur. Le code-barres est situé à 3' UTR (région non traduite) entre la protéine fluorescente jaune améliorée (EYFP) et le signal polyA (voir Figure 6A). L’expression de l’EYFP est déterminée par un puissant promoteur du cytomégalovirus ubiquitaire (CMV) qui fournit des niveaux suffisants de transcrits d’ARN. - Concevoir des codes-barres de 15 nucléotides de longueur avec des homopolymères de moins de trois nucléotides, une teneur en GC de <65%29 et une distance de Hamming supérieure à quatre nucléotides24.
- Produire chaque capside séparément en combinaison avec un plasmide transgénique portant un code-barres unique. De cette façon, chaque variante de capside est étiquetée avec un code-barres distinct qui permet son suivi spécifique (voir Figure 6B).
- Effectuer la production d’AAV recombinants pour chaque variante de capside en utilisant le système à trois plasmides, comme décrit précédemment24.
- Titrage vectoriel AAV par dd-PCR
- Effectuez le titrage AAV comme décrit précédemment dans la section 1.8, en remplaçant la paire d’amorces Rep2 par la paire d’amorces YFP.
- Quantifier les productions AAV individuelles et mettre en commun des quantités égales de chaque production pour générer la bibliothèque finale à code-barres.
- Quantifier à nouveau la bibliothèque finale pour vérifier la concentration finale et la qualité (voir la figure 7).
- Bibliothèque AAV à code-barres application in vivo
- Appliquer systématiquement la bibliothèque AAV à code-barres au système modèle de votre choix (par exemple, systématiquement chez la souris24).
- Prélever des tissus ON et OFF cibles (foie, poumon, cœur, diaphragme, muscle lisse, duodénum, pancréas, côlon, biceps, ovaires, estomac, oreille interne, rein, aorte abdominale, aorte thoracique, cerveau, graisse brune et blanche et rate) ou des types de cellules en fonction de l’expérience. Congelez-les à -80 °C, extrayez l’ADN/ARN et appliquez l’analyse de quantification NGS, comme décrit dans la section suivante.
- Extraction d’ADN/ARN
- Extrayez l’ADN et l’ARN des tissus d’intérêt à l’aide du mini kit ADN/ARN.
- Placer un petit morceau du tissu d’intérêt (1 mm3, environ 5 mg) dans un tube de réaction de 2 mL.
- Ajouter 350 μL de tampon de lyse mélangé avec du β-mercaptoéthanol (1 %) et des billes d’acier de 5 mm dans le tissu (manipuler les échantillons avec du β-mercaptoéthanol sous une hotte).
- Homogénéiser le tissu dans un tissuLyser pendant 45 s à 40 Hz.
- Ajouter 10 μL de protéinase K (10 mg/mL) et incuber pendant 15 min à 55 °C tout en agitant à 400 rpm.
- Centrifuger à 20 000 x g pendant 3 min à température ambiante, recueillir le surnageant et procéder au protocole du fabricant du kit ADN/ARN.
- Divisez l’étape de lavage en deux étapes avec 350 μL de tampon de lavage à chaque étape. Entre ces étapes de lavage, digérer l’ADN restant sur la colonne avec la DNase I sans RNase. Ajouter 80 μL de la solution de DNase I, préparée selon les instructions du fabricant, sur la colonne et incuber à température ambiante pendant 15 min.
- Élute ARN/ADN de la colonne avec de l’eau sans nucléase. Stocker l’ARN isolé à -80 °C et l’ADNg à -20 °C.
- Synthèse de l’ADNc
- Soumettre les échantillons d’ARN à une autre série de traitement par la DNase I de 15 à 30 minutes (pour l’élimination complète de l’ADN contaminant des échantillons d’ARN) avant la réaction de transcription inverse. Ajouter 1 μL de la solution de DNase I, 4 μL de tampon (fourni avec le kit) et de l’eau sans nucléase à un volume final de 40 μL à 212 ng d’ARN. Incuber pendant 30 min à température ambiante et inactiver la chaleur à 70 °C pendant 10 min.
- Synthétiser l’ADNc, en utilisant 150 ng d’ARN à l’aide d’un kit selon les instructions du fabricant. Inclure des contrôles sans transcriptase inverse, pour s’assurer de l’absence d’ADN viral contaminant de l’échantillon. L’ADNc est stocké à -20 °C.
REMARQUE: La quantité d’ARN d’entrée pour une transcription inverse optimale peut varier en fonction du type de tissu et de l’efficacité de transduction attendue dans le tissu respectif.
- Analyse de la bibliothèque virale AAV (in-vivo) par NGS
- Pour obtenir une profondeur de séquençage élevée à faible coût, effectuez le NGS via le séquençage Illumina comme décrit précédemment (section 1.9). Amplifiez la séquence de codes-barres, puis ligaturez les adaptateurs de séquençage sur l’amplicon.
- En raison de la courte longueur de lecture et de la ligature des adaptateurs de séquençage des deux côtés de l’amplicon, lors de la conception, vérifiez que l’amplicon est suffisamment petit pour assurer la présence de la séquence de codes-barres dans la lecture NGS. Pour le séquençage des codes-barres dans les génomes viraux et les transcrits viraux, l’amplicon PCR est conçu pour avoir une longueur de 113 pb (voir Figure 8).
- Amplifiez la région à code-barres avec les amorces BC-seq avant et BC-seq inverse. Préparer la réaction PCR suivante : 0,5 μL d’ADN polymérase haute fidélité, 10 μL de tampon 5x, 0,25 μL de chaque amorce 100 μM (BC-seq fw/BC-seq rv) et 1 μl de 10 mM dNTP. Utilisez 25 ng d’ADNc ou d’ADN/réaction comme matrice et ajustez le volume final à 50 μL avec de l’eau sans nucléase.
- Préparez le mélange-maître PCR sous une hotte PCR propre pour éviter toute contamination. Utilisez les conditions de cycle suivantes: 30 s à 98 °C, suivi de 40 cycles à 98 °C pendant 10 s et 72 °C pendant 20 s, et un pas final de 5 minutes à 72 °C.
- Inclure des contrôles PCR pour confirmer l’absence d’ADN contaminant dans le mélange maître de PCR. Pour les échantillons d’ADNc, inclure les témoins sans transcriptase inverse. Enfin, incluez un exemple avec la bibliothèque d’entrées AAV. Ces informations seront utilisées pour générer le fichier Normalization_Variant.txt utilisé dans l’analyse.
- Vérifier la taille du fragment PCR de chaque échantillon par électrophorèse sur gel avant purification par PCR. Ce dernier est réalisé en utilisant soit des billes magnétiques disponibles dans le commerce, soit des systèmes de purification de l’ADN à base de colonnes (voir le tableau des matériaux).
- Préparer la bibliothèque NGS à l’aide du système de bibliothèque pour les échantillons de faible complexité conformément aux instructions du fabricant, comme décrit précédemment à la section 1.9.
- Déterminer la concentration d’ADN via le kit dsDNA HS et analyser la qualité de la bibliothèque comme décrit précédemment (section 1.9.6), suivi d’une mise en commun. Quantifier la bibliothèque regroupée sur un fluoromètre et évaluer la qualité sur un bioanalyseur.
- Effectuer le séquençage NGS comme indiqué à la section 1.9.7.
- Quantifier par qPCR le nombre de copies du transgène (génomes viraux) et du gène d’entretien pour évaluer la distribution de la bibliothèque mise en commun entre les tissus ou les organes de l’ADN.
- Mettre en place une réaction qPCR de 30 μL comme suit, pour déterminer le nombre de copies de EYFP (transgène) et GAPDH (glycéraldéhyde 3-phosphate déshydrogénase, gène d’entretien ménager):
- Préparer un mélange amorce/sonde 60x pour le FEP (1,5 μM YFP_fw, 1,5 μM YFP_rv et 0,6 μM YFP_probe; voir le tableau des matériaux). Utiliser le mélange amorce/sonde GAPDH (voir le tableau des matières) pour déterminer le numéro de copie du gène de la gouvernante. Mettez en place la réaction sur la glace.
- Préparez un mélange principal PCR (15 μL, voir le tableau des matériaux) et ajoutez 60x mélange amorce/sonde (0,5 μL) pour tous les échantillons et étalons (pour calculer le nombre d’exemplaires pour les étalons, utilisez le lien suivant : http://cels.uri.edu/gsc/cndna.html). Mettez en place la réaction sur la glace.
- Transférer 15,5 μL du mélange maître dans une plaque de 96 puits et ajouter 14,5 μL d’échantillon (75 ng de concentration d’ADN total) ou d’étalon au puits respectif. Scellez la plaque de 96 puits avec du papier d’aluminium, du vortex et faites tourner brièvement.
- Transférer 10 μL de chaque échantillon dans une plaque de 384 puits en doubles. Sceller la plaque avec du papier d’aluminium et faire tourner à 800 x g pendant 5 min à 4 °C.
- Incuber le mélange réactionnel dans un thermocycleur à une température initiale de 50 °C pendant 2 min, suivie d’une étape d’activation initiale de 10 min à 95 °C. Effectuer 40 cycles de dénaturation à 95 °C pendant 15 s et de recuit/extension à 60 °C pendant 1 min24.
- Pour obtenir le nombre de génomes diploïdes (dg), utilisez le nombre de copies GAPDH et divisez par deux. Ensuite, prenez la valeur du nombre de copies du PEJ et divisez-la par le nombre de dg, ce qui donne des génomes vectoriels par génome diploïde (vg/dg). Utilisez cette valeur pour générer le fichier Normalization_Organ.txt pour l’analyse bioinformatique.
- Effectuer l’analyse des données de séquençage NGS comme Weinmann et al.24, en utilisant du code personnalisé dans Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Le flux de travail comprend la détection de séquences de codes-barres guidées par des séquences flanquantes, leur longueur et leur emplacement (Script#1_BarcodeDetection.py), ainsi que l’analyse de l’enrichissement et de la distribution des codes-barres sur l’ensemble des tissus (Script#2_BarcodeAnalysis.py).
- Détectez les codes-barres et attribuez-les aux variantes AAV. Placez les données de séquençage sous forme de fichiers fastq archivés dans un répertoire (par exemple, « Data_to_analyze »). Le fichier de données de séquençage de la bibliothèque d’entrée est inclus dans ce répertoire et utilisé uniquement pour calculer les proportions de capside dans la bibliothèque d’entrée.
- Avant d’exécuter le script, créez deux fichiers texte délimités par des tabulations : le fichier de variantes de capside (voir le fichier d’exemple « Variantes.txt ») avec les séquences de codes-barres attribuées aux noms de variantes de capside AAV, et le fichier de contamination (voir « Contaminations.txt ») avec des séquences de codes-barres provenant d’une contamination possible (autres codes à barres disponibles en laboratoire, contribuant à la contamination).
- Enfin, modifiez le fichier de configuration « Barcode_Script.conf » pour inclure les informations suivantes : chemin d’accès au dossier avec les données de séquençage (par exemple, « Data_to_analyze »), séquence des régions flanquantes des codes-barres, leur position et la taille de la fenêtre pour la détection des codes-barres (similaire à 1.9.8.5, voir la figure 8).
- Utilisez la commande suivante pour appeler la détection de code-barres avec les chemins d’accès fournis à Script#1_BarcodeDetection.py et aux fichiers de configuration :
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
REMARQUE: La sortie de l’exécution du script # 1_BarcodeDetection.py est constituée de fichiers texte avec le nombre de lectures par variante de capside ainsi que le nombre total de lectures récupérées à partir des données brutes. - Évaluez la distribution des capsides AAV à code-barres parmi les tissus ou les organes, en exécutant Script#2_BarcodeAnalysis.py avec les fichiers txt suivants :
- Dans le fichier « Zuordnung.txt », attribuez le nom à chaque fichier txt obtenu à partir de l’exécution de détection de code-barres à un nom de tissu/organe : noms des fichiers txt dans la première colonne et noms de tissus/organes correspondants dans l’affectation délimitée par des tabulations.
REMARQUE: Pour un exemple, vérifiez dans le dossier « Exemple » (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Il convient de noter que le nom du tissu ou de l’organe peut inclure des caractères définissant la mesure de l’ADNc ou de l’ADNg et le nombre de réplications biologiques (M1, M2, etc.). - Créez un fichier texte « organes.txt » avec la liste des noms des organes cibles ON et OFF, qui correspondent aux noms donnés dans le fichier d’affectation « Zuordnung.txt » (voir dossier « Exemple »: https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- Créez des fichiers texte délimités par des tabulations « Normalization_Organ.txt » et « Normalization_Variant.txt » avec des valeurs normalisées pour toutes les variantes de capside et tous les organes/tissus. Dans la première colonne du fichier « Normalization_Organ.txt », écrivez les noms donnés pour chaque organe (comme dans le fichier d’affectation « Zuordnung.txt ») et dans la deuxième colonne les valeurs de normalisation pour les tissus correspondants, générées dans la section 3.6.11.
- Remplissez la première colonne du fichier « Normalization_Variant.txt » avec la liste des noms de capside et la deuxième colonne avec les valeurs normalisées du nombre de lectures pour chaque capside dans la bibliothèque regroupée (la normalisation peut être calculée sur la base du fichier de sortie txt pour la bibliothèque d’entrée résultant du premier script).
- Modifiez le fichier de configuration en spécifiant les chemins d’accès complets à tous les fichiers supplémentaires mentionnés ci-dessus. Exécuter le script#2_BarcodeAnalysis.py en tant que :
>python3 /Script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
REMARQUE: Le script d’analyse de code-barres génère plusieurs fichiers: des fichiers texte avec des valeurs de concentration relative (RC) de la distribution de la capside dans différents tissus en fonction de plusieurs étapes de normalisation décrites précédemment, et le fichier de feuille de calcul qui combine les données de fichier texte dans les données de matrice fusionnées. Ce dernier peut être utilisé pour l’analyse et la visualisation de clusters. - Visualisez les données et effectuez une analyse par grappes des données matricielles afin de distinguer les propriétés de la capside et d’évaluer leurs similitudes en fonction des profils RC entre les tissus. Utilisez le script supplémentaire PCA_heatmap_plot. R placé dans le référentiel :
>Rscript --vanille ~/PCA. R ~/concentration relative.xls
Remarque : Le script prend la concentration relative.xls fichiers comme entrée et génère deux tracés de carte thermique de cluster hiérarchique et d’analyse en composantes principales (ACP). - Pour modifier des tracés (axes de carte thermique, composantes principales de PCA) ou des paramètres png (couleur, taille, étiquetage), ouvrez le script R et suivez les instructions fournies dans les sections commentées.
- Dans le fichier « Zuordnung.txt », attribuez le nom à chaque fichier txt obtenu à partir de l’exécution de détection de code-barres à un nom de tissu/organe : noms des fichiers txt dans la première colonne et noms de tissus/organes correspondants dans l’affectation délimitée par des tabulations.
Résultats
Génération d’une bibliothèque d’affichage de peptides AAV2. Comme première étape vers la sélection d’AAV techniques, la génération d’une bibliothèque de plasmides est décrite. L’insert peptidique est produit à l’aide d’amorces dégénérées. Réduire la combinaison de codons chez ceux de 64 à 20 a l’avantage d’éliminer les codons stop et de faciliter l’analyse NGS, en réduisant la diversité des bibliothèques sur l’ADN mais pas au niveau de la protéine. L’insert ...
Discussion
Dans ce protocole, les étapes nécessaires à l’ingénierie de la capside AAV d’affichage peptidique et au criblage de bibliothèques AAV à code-barres, ainsi qu’à l’analyse bioinformatique de la composition de la bibliothèque et des performances de la capside, sont décrites. Ce protocole se concentre sur les étapes qui facilitent l’analyse bioinformatique de ces types de bibliothèques, car la plupart des laboratoires de virologie accusent un retard dans les compétences en programmation correspondant à...
Déclarations de divulgation
D.G. est co-fondateur d’AaviGen GmbH. D.G. et K.R. sont des inventeurs sur une demande de brevet en instance liée à la génération de variantes de capside AAV échappant au système immunitaire. Les autres auteurs n’ont rien à divulguer.
Remerciements
D.G. apprécie grandement le soutien de la Fondation allemande pour la recherche (DFG) par l’intermédiaire des centres de recherche collaborative DFG SFB1129 (Projektnummer 240245660) et TRR179 (Projektnummer 272983813), ainsi que du Centre allemand de recherche sur les infections (DZIF, BMBF; TTU-VIH 04.819).
matériels
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
Références
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621 (2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756 (2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075 (2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041 (2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432 (2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819 (2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899 (2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272 (2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200 (2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon