Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
AMEBaS: Extracción automática de la línea media y sustracción de fondo de time-lapses de fluorescencia radiométrica de células individuales polarizadas
En este artículo
Resumen
Los métodos actuales para analizar la dinámica intracelular de células individuales polarizadas son a menudo manuales y carecen de estandarización. Este manuscrito presenta una novedosa línea de análisis de imágenes para automatizar la extracción de la línea media de células polarizadas individuales y cuantificar el comportamiento espacio-temporal de los lapsos de tiempo en una interfaz en línea fácil de usar.
Resumen
La polaridad celular es un fenómeno macroscópico establecido por un conjunto de moléculas y estructuras espacialmente concentradas que culminan en la aparición de dominios especializados a nivel subcelular. Se asocia con el desarrollo de estructuras morfológicas asimétricas que subyacen a funciones biológicas clave como la división celular, el crecimiento y la migración. Además, la alteración de la polaridad celular se ha relacionado con trastornos relacionados con los tejidos, como el cáncer y la displasia gástrica.
Los métodos actuales para evaluar la dinámica espacio-temporal de los reporteros fluorescentes en células polarizadas individuales a menudo implican pasos manuales para trazar una línea media a lo largo del eje principal de las células, lo que lleva mucho tiempo y es propenso a fuertes sesgos. Además, aunque el análisis radiométrico puede corregir la distribución desigual de las moléculas reporteras utilizando dos canales de fluorescencia, las técnicas de sustracción de fondo suelen ser arbitrarias y carecen de apoyo estadístico.
Este manuscrito presenta una nueva línea computacional para automatizar y cuantificar el comportamiento espacio-temporal de células individuales utilizando un modelo de polaridad celular: crecimiento del tubo polínico/pelo radicular y dinámica de iones citosólicos. Se desarrolló un algoritmo de tres pasos para procesar imágenes ratiométricas y extraer una representación cuantitativa de la dinámica y el crecimiento intracelular. El primer paso segmenta la celda del fondo, produciendo una máscara binaria a través de una técnica de umbral en el espacio de intensidad de píxeles. El segundo paso traza un camino a través de la línea media de la célula a través de una operación de esqueletización. Finalmente, el tercer paso proporciona los datos procesados como un lapso de tiempo radiométrico y produce un quimógrafo radiométrico (es decir, un perfil espacial 1D a través del tiempo). Para comparar el método se utilizaron datos de imágenes radiométricas adquiridas con reporteros fluorescentes codificados genéticamente de tubos polínicos en crecimiento. Esta canalización permite una representación más rápida, menos sesgada y más precisa de la dinámica espacio-temporal a lo largo de la línea media de las células polarizadas, avanzando así en el conjunto de herramientas cuantitativas disponibles para investigar la polaridad celular. El código fuente de Python de AMEBaS está disponible en: https://github.com/badain/amebas.git
Introducción
La polaridad celular es un proceso biológico fundamental en el que la acción concertada de una colección de moléculas y estructuras espacialmente concentradas culmina en el establecimiento de dominios morfológicos subcelulares especializados. La división, el crecimiento y la migración celular dependen de dichos sitios de polaridad, mientras que su pérdida se ha asociado con el cáncer en trastornos relacionados con el tejido epitelial2.
Las células que crecen apicalmente son un ejemplo dramático de polaridad, donde el sitio de polaridad en la punta generalmente se reorienta a las señales extracelulares3. Estos incluyen neuritas en desarrollo, hifas fúngicas, pelos radiculares y tubos polínicos, donde múltiples procesos celulares muestran diferencias pronunciadas desde la punta de la célula hacia el vástago. En los tubos polínicos, en particular, la polimerización de actina, el tráfico de vesículas y las concentraciones iónicas están marcadamente polarizadas, mostrando gradientes centrados en la punta4. Los tubos polínicos son los gametofitos masculinos de las plantas con flores y son responsables de llevar los espermatozoides al óvulo al crecer exclusivamente en el ápice de la célula a una de las tasas de crecimiento más rápidas conocidas para una sola célula. Los gradientes centrados en la punta de iones como el calcio 5 (Ca2+) y los protones 6 (H+) juegan un papel importante en el mantenimiento del crecimiento del tubo polínico, que es esencial para cumplir su función biológica principal que culmina en una doble fertilización 5,6. Por lo tanto, los métodos cuantitativos para analizar la dinámica espacio-temporal a lo largo de la línea media de las células en crecimiento apical son esenciales para investigar los mecanismos celulares y moleculares que subyacen al crecimiento polarizado 7,8,9. Los investigadores a menudo usan quimógrafos, es decir, una matriz que representa las intensidades de píxeles de la línea media de la célula (por ejemplo, columnas) a través del tiempo (por ejemplo, filas), lo que permite visualizar el crecimiento y la migración celular en diagonal (Figura 1). A pesar de su utilidad, los quimógrafos se extraen con frecuencia trazando manualmente la línea media, siendo propensos a sesgos y errores humanos, además de ser bastante laboriosos. Esto requiere un método automatizado de extracción de la línea media que es la primera característica de la tubería introducida en este documento llamada AMEBaS: una extracción Midline Eutomática y unaextracción S de Mckground de lapsos de tiempo defluorescencia ratiométricos de celdas individuales polarizadas.
En cuanto a los procedimientos experimentales, se pueden obtener imágenes cuantitativas de iones/moléculas/especies de interés en células individuales con sondas fluorescentes codificadas genéticamente10. Entre las opciones cada vez más amplias, las sondas radiométricas son una de las más precisas, ya que emiten diferentes longitudes de onda de fluorescencia cuando se unen/desunen a las moléculas de interés11. Esto permite corregir la heterogeneidad espacial en la concentración intracelular de la sonda utilizando la relación de dos canales con su fondo específico del canal restado. Sin embargo, estimar el umbral de fondo para cada canal y punto de tiempo puede ser una tarea compleja, ya que a menudo varía en el espacio debido a efectos como el sombreado, donde las esquinas de la imagen tienen variación de luminosidad en relación con el centro, y en el tiempo debido al desvanecimiento del fluoróforo (fotoblanqueo)12. Aunque existen múltiples métodos posibles, este manuscrito propone determinar la intensidad de fondo de forma automática utilizando el umbral de segmentación obtenido con el algoritmo Isodata13, que luego se suaviza a través de fotogramas mediante regresión polinómica como estándar. Sin embargo, los componentes espaciales derivados de la heterogeneidad de la fluorescencia no relacionada con la célula diana eliminada en12 fueron ignorados por este método. El umbral automático se puede realizar mediante varios métodos, pero el algoritmo Isodata produjo los mejores resultados empíricamente. Por lo tanto, la sustracción automática del valor de fondo y el cálculo radiométrico son la segunda característica principal de AMEBaS (Figura 1), que, en conjunto, recibe como entrada una pila de imágenes de microscopía de fluorescencia de doble canal, estima la línea media de la célula y el fondo específico del canal, y genera cimogramas de ambos canales y su relación (salida principal # 1) después de la sustracción de fondo, el suavizado y la eliminación de valores atípicos. junto con una pila de imágenes radiométricas (salida principal #2).
AMEBaS se probó con lapsos de tiempo de fluorescencia de tubos polínicos de Arabidopsis en crecimiento obtenidos bajo un microscopio, ya sea con sensores radiométricos de Ca2+ (CaMeleon)8 o pH (pHluorin)6 expresados bajo el promotor LAT52 específico del polen. Las imágenes de cada canal se tomaron cada 4 s acopladas a un microscopio invertido, una cámara con iluminación frontal (2560 píxeles × 2160 píxeles, tamaño de píxel 6,45 μm), un iluminador de fluorescencia y una lente objetivo de inmersión en agua 63x, 1.2NA. Los ajustes de los filtros utilizados para CaMeleon fueron: excitación 426-450 nm (CFP) y 505-515 nm (YFP), emisión 458-487 nm (CFP) y 520-550 nm (YFP), mientras que para pHluorin, excitación 318-390 nm (DAPI) y 428-475 nm (FITC), emisión 435-448 nm (DAPI) y 523-536 nm (FITC). Se agregó un conjunto completo de datos para las pruebas en Zenodo (DOI: 10.5281/zenodo.7975350)14.
Además, la tubería se probó con datos de pelos radiculares, donde se realizaron imágenes con un microscopio de lámina de luz (SPIM) como se describió previamente 15,16 con pelos radiculares de Arabidopsis que expresan el reportero Ca2+ codificado genéticamente NES-YC3.6 bajo el control del promotor UBQ1017. El software casero LabView, que controlaba la adquisición de la cámara, la traslación de la muestra y el obturador del microscopio de lámina de luz, permitió la observación de los dos canales cpVenus y CFP, pero también la visualización de su relación en tiempo real. Cada imagen de relación del lapso de tiempo representó una proyección de máxima intensidad (MIP) entre las imágenes de los canales fluorescentes cpVenus y CFP obtenidas a partir de 15 cortes de la muestra espaciados a 3 μm de distancia. Se guardó la relación cpVenus/CFP de lapso de tiempo de los MIP y se utilizó directamente para el análisis AMEBaS.
Aunque esta tubería puede trabajar con múltiples tipos de células en crecimiento y migración, fue diseñada específicamente para analizar células en crecimiento que crecen exclusivamente en la punta, como los tubos polínicos, los pelos radiculares y las hifas fúngicas, donde existe una correspondencia de las regiones citoplasmáticas no en crecimiento entre los marcos. Cuando dicha correspondencia no está presente, el usuario debe elegir la opción complete_skeletonization en el paso 1.3.1.1 (consulte la sección Discusión para obtener más detalles).
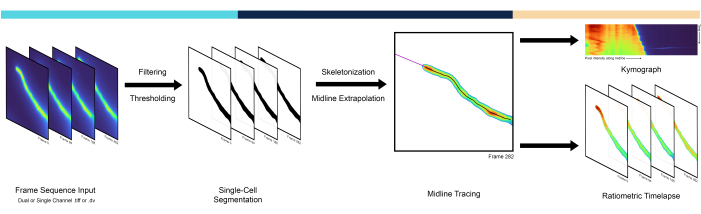
Figura 1: Descripción general del flujo de trabajo de la canalización. La canalización AMEBaS analiza y procesa lapsos de tiempo microscópicos en tres pasos principales: segmentación de una sola célula, trazado de la línea media y generación de quimógrafos. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Protocolo de cuaderno interactivo
El cuaderno de Jupyter Notebook se puede usar directamente en la web mediante Google Colab en https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb, donde se basaron las instrucciones siguientes. Como alternativa, Jupyter Notebook está disponible en https://github.com/badain/amebas, donde se puede descargar y configurar para que se ejecute localmente en Jupyter (Anaconda puede proporcionar un proceso de instalación fácil y multiplataforma). Se pueden encontrar datos completos de la prueba en Zenodo (https://doi.org/10.5281/zenodo.7975350), que contiene datos de canal simple y doble de los tubos polínicos de Arabidopsis que expresan indicadores de pH o Ca2+ 14. La canalización se ha dividido en partes, donde cada paso se puede ejecutar haciendo clic en el botón de reproducción después de configurar las opciones específicas del usuario. Los archivos necesarios para este estudio están disponibles en la carpeta zip principal de AMEBaS (Archivo de codificación suplementaria 1).
- Abra Jupyter Notebook y lea los archivos de lapso de tiempo.
- Navegue a la página de inicio del bloc de notas interactivo en Google Colab mencionado anteriormente o descargue y abra el bloc de notas AMEBaS_Local.ipynb desde GitHub.
- Prepare la configuración del directorio para los datos de entrada y salida:
- Si utiliza la versión local, coloque el time-lapse de fluorescencia como un archivo TIFF o un archivo DV dentro de una carpeta llamada data que debe estar en la carpeta raíz del programa. Se debe crear una carpeta con nombre out para recibir los datos generados. A continuación, ejecute el bloque de código de instalación.
- Si usa el bloc de notas en Google Colab, ejecute el bloque de código de instalación para generar automáticamente las carpetas de datos y salida .
- Ejecute el bloque de código de entrada de archivo para leer los datos de lapso de tiempo haciendo clic en el botón de reproducción. Si utiliza la versión de Google Colab del bloc de notas, haga clic en el botón Elegir archivo para cargar directamente el archivo de lapso de tiempo en la carpeta de datos.
NOTA: El número de canales se detectará automáticamente en función de la dimensión de la imagen. - Elija si se generarán salidas adicionales de cada paso estableciendo el parámetro 'verbose' en Verdadero o Falso.
- Detecte la celda principal y el segmento del fondo (Figura 2).
- Ejecute el bloque de código Segmentación de celda única para separar automáticamente la celda de interés del fondo haciendo clic en el botón de reproducción .
NOTA: Los filtros mediano y gaussiano se aplicarán como paso de preprocesamiento para eliminar el ruido no deseado antes de segmentar el primer plano del fondo mediante el umbral de isodatos y el aislamiento de la región del área más grande para eliminar los artefactos no deseados.- Ajuste el valor sigma utilizado por el gaussiano en la variable 'sigma' para ajustar la suavidad de la máscara de segmentación. El valor predeterminado es 2.0.
- Establezca la estimación de la variable en Falso para almacenar el umbral estimado a partir de Isodata directamente o en True para suavizarlo a través de fotogramas vecinos mediante la regresión polinómica local (LOESS). Ajuste su función cambiando la variable n_points . El valor predeterminado es 40.
- Ejecute el bloque de código Segmentación de celda única para separar automáticamente la celda de interés del fondo haciendo clic en el botón de reproducción .
- Traza la línea media a lo largo de la extensión de la celda (Figura 3).
- Ejecute el bloque de código Cell Midline Tracing haciendo clic en el botón de reproducción para esqueletizar automáticamente la celda utilizando el método18 de Lee y extender la punta del último esqueleto a través de la extrapolación lineal.
- Elija trazar la línea media solo en el último fotograma o una vez por fotograma ajustando el argumento complete_skeletonization .
NOTA: Cuando todos los fotogramas están esqueletizados, se omite la extrapolación. - Establezca la fracción de puntos del esqueleto que se va a interpolar durante la extrapolación ajustando la variable interpolation_fraction . El valor predeterminado es 0,25.
- Elija la longitud de la extrapolación de la línea media cambiando la variable extrapolation_length. El valor predeterminado es -1, que extiende el esqueleto hasta el borde más cercano.
- Elija trazar la línea media solo en el último fotograma o una vez por fotograma ajustando el argumento complete_skeletonization .
- Ejecute el bloque de código Cell Midline Tracing haciendo clic en el botón de reproducción para esqueletizar automáticamente la celda utilizando el método18 de Lee y extender la punta del último esqueleto a través de la extrapolación lineal.
- Genere kymographs para cada canal ( Figura 4).
- Ejecute el primer bloque de código de visualización de datos haciendo clic en el botón de reproducción para generar automáticamente kymographs para ambos canales.
- Elija el tamaño del kernel gaussiano utilizado para el suavizado ajustando la variable kymograph_kernel.
NOTA: Corresponde al tamaño de la vecindad (en píxeles) sobre la que se promedian las intensidades de píxel. El valor predeterminado es 3 píxeles x 3 píxeles. - Los esqueletos no extendidos generan kymogramas limitados que deben usar un mapa de colores personalizado para mostrar correctamente sus intensidades. Elige el porcentaje fraccionario de las intensidades que se asignará al color de fondo, negro, ajustando la variable shift_fraction . El valor predeterminado es 0,7.
- Elija el tamaño del kernel gaussiano utilizado para el suavizado ajustando la variable kymograph_kernel.
- Ejecute el primer bloque de código de visualización de datos haciendo clic en el botón de reproducción para generar automáticamente kymographs para ambos canales.
- Calcule la relación entre canales (Figura 5).
- Ejecute el segundo bloque de código de visualización de datos haciendo clic en el botón de reproducción para generar automáticamente un quimógrafo radiométrico y un timelapse radiométrico (Figura 6).
NOTA: Este paso solo está disponible cuando se utilizan lapsos de tiempo de doble canal. El umbral de intensidad de fondo almacenado en el paso 1.2.1.2 se resta de cada canal.- Ajuste la variable switch_ratio para cambiar el orden de los canales utilizados como numerador y denominador durante los cálculos de proporción. El valor predeterminado es False.
- Elija si el lapso de tiempo de relación debe suavizarse aún más con una pasada de filtro mediana ajustando la variable smooth_ratio . El valor predeterminado es False.
- Elija si desea eliminar los valores atípicos producidos por la señal baja del canal del denominador manipulando la variable reject_outliers . El valor predeterminado es True y define los valores atípicos como valores 1,5 veces el rango intercuartílico por encima del tercer cuartil (donde se encuentra el 75% de los valores).
- Elija si el fondo de la salida radiométrica debe exportarse ajustando la variable background_ratio. El valor predeterminado es False, que lo reemplaza con ceros.
- Ejecute el segundo bloque de código de visualización de datos haciendo clic en el botón de reproducción para generar automáticamente un quimógrafo radiométrico y un timelapse radiométrico (Figura 6).

Figura 2: Paso de segmentación de una sola célula. Se utilizan técnicas de procesamiento de imágenes como el filtrado, el umbral y el etiquetado de áreas para aislar la señal de interés (paso 1.2). Estos datos en particular tenían los siguientes valores para la intensidad más baja: 2556, la mediana: 3441 y la intensidad más alta: 32125. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Descripción general del trazado de la línea media: la línea media de la celda individual se obtiene calculando su esqueleto (blanco). La punta (magenta) se extrapola linealmente desde los últimos puntos al final del esqueleto (paso 1.3). En esta composición, tanto la línea media como su punta se superponen sobre la celda original. Haga clic aquí para ver una versión más grande de esta figura.
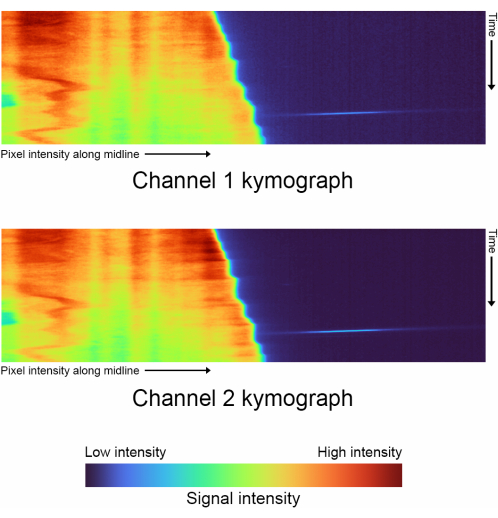
Figura 4: Quimógrafos de Timelapse: comparación de los quimógrafos de cada canal generado con 'complete_skeletonization' desactivado (paso 1.4). El eje vertical describe la progresión del tiempo, y el eje horizontal representa la intensidad media de la trayectoria extrapolada de la línea media seguida de una sola celda. Para estos datos en particular, el mapa de colores representa los siguientes valores para la intensidad más baja del canal 1: 2886, Mediana: 3167, Intensidad más alta: 21021. Canal 2 Intensidad más baja: 3030, Mediana: 3400, Intensidad más alta: 29688. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Suavizado del umbral de fondo: el umbral de segmentación de fondo se estima a través del algoritmo de isodatos y luego se suaviza mediante regresión polinómica local (paso 1.5). Haga clic aquí para ver una versión más grande de esta figura.
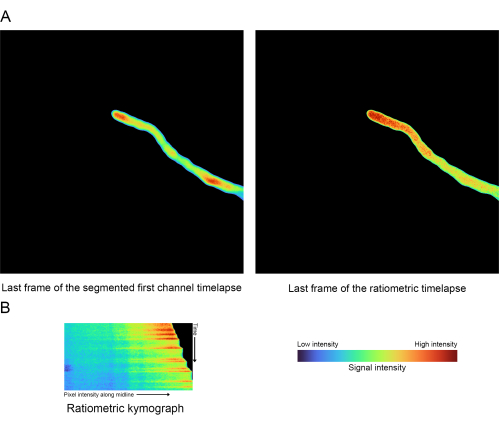
Figura 6: Resultados ratiométricos- (A) Comparación entre el último fotograma del timelapse radiométrico y el primer canal original segmentado. (B) Quimógrafo generado a partir del timelapse radiométrico (paso 1.5). Haga clic aquí para ver una versión más grande de esta figura.
2. Protocolo de modo por lotes
- Descargue y coloque el archivo pipeline.py del repositorio de GitHub de AMEBaS en el mismo directorio que los datos.
- Escriba la ubicación del archivo en la línea de comandos después del archivo de programa.
- Incluya - -v como argumento posicional para mostrar los pasos internos de la canalización, si lo desea.
- Incluya - -s para elegir en el valor sigma utilizado en el paso de preprocesamiento del filtro gaussiano en preparación para la segmentación celular. El valor predeterminado es 2.
- Incluya - -a para trazar la línea media de cada fotograma del lapso de tiempo. De forma predeterminada, la canalización usa solo el último fotograma.
- Incluya - -f para elegir la fracción [0,1] del esqueleto que se utilizará en la interpolación. El valor predeterminado es 0,25.
- Incluya -e para elegir la longitud en píxeles del esqueleto extrapolado. El valor predeterminado es -1, que extiende el esqueleto hasta el borde más cercano.
- Incluya - -sf para elegir la fracción de la gama de colores que se desplazará al fondo en los kymographs no extrapolados. El valor predeterminado es 0,7.
- Incluya - -k para determinar el tamaño del kernel utilizado en el filtrado gaussiano del cimógrafo. El valor predeterminado es 3.
- Incluya - -eb para estimar la intensidad del umbral de fondo global a través de la regresión polinómica LOESS de las intensidades del umbral de fondo específico de la trama.
- Personalice el número de puntos utilizados en el suavizado LOESS de los valores de umbral de fondo modificando el parámetro - -n. El valor predeterminado es 40.
- Cambie los canales utilizados como numerador y denominador durante los cálculos de proporción, incluidos - r o - -switch_ratio, si el lapso de tiempo tiene dos canales. Por defecto, el segundo canal es el numerador y el primero es el denominador.
- Elija si el lapso de tiempo de proporción debe suavizarse aún más con una pasada de filtro mediano con el argumento - -sm . El valor predeterminado es False.
- Incluya -o para rechazar píxeles con intensidades anormales durante la generación de lapso de tiempo ratiométrico.
- Elija si el fondo de la salida radiométrica debe exportarse mediante el argumento - -b. El valor predeterminado es False, que lo reemplaza con ceros.
- Pulse Intro para ejecutar. La salida se generará en el mismo directorio que el archivo de programa.
Resultados
La canalización AMEBaS automatiza la extracción de la dinámica de la línea media de células individuales polarizadas de pilas de imágenes de microscopía de fluorescencia, lo que hace que consuma menos tiempo y sea menos propenso a errores humanos. El método cuantifica estos lapsos de tiempo mediante la generación de quimógrafos y pilas de imágenes radiométricas (Figura 1) en células individuales en crecimiento. Se puede ajustar para trabajar en la migración de células individu...
Discusión
El novedoso método que se presenta aquí es una potente herramienta para agilizar y automatizar el análisis de pilas de imágenes de microscopía de fluorescencia de células polarizadas. Los métodos actuales descritos en la literatura, como los complementos ImageJ Kymograph, requieren el trazado manual de la línea media de la celda polarizada de interés, una tarea que no solo requiere mucho tiempo, sino que también es propensa a errores humanos. Dado que la definición de la línea media en este conducto se apoya ...
Divulgaciones
Los autores de este manuscrito declaran no tener intereses financieros en competencia ni otros conflictos de intereses.
Agradecimientos
Los autores agradecen las subvenciones de la FAPESP 2015/22308-2, 2019/23343-7, 2019/26129-6, 2020/06744-5, 2021/05363-0, CNPq, NIH R01 GM131043 y las subvenciones de la NSF MCB1714993, MCB1930165 por el apoyo financiero. Los datos del pelo radicular se produjeron con la infraestructura y bajo la supervisión del Prof. Andrea Bassi y el Prof. Alex Costa.
Materiales
| Name | Company | Catalog Number | Comments |
| Github | Github | https://github.com/badain/amebas | |
| Google Colab | https://colab.research.google.com/github/badain/amebas/blob/main/AMEBAS_Colab.ipynb |
Referencias
- Drubin, D. G., Nelson, W. J. Origins of cell polarity. Cell. 84 (3), 335-344 (1996).
- Wodarz, A., Näthke, I. Cell polarity in development and cancer. Nature Cell Biology. 9 (9), 1016-1024 (2007).
- Palanivelu, R., Preuss, D. Pollen tube targeting and axon guidance: parallels in tip growth mechanisms. Trends in Cell Biology. 10 (12), 517-524 (2000).
- Portes, M. T., et al. . The Pollen Tube Oscillator: Integrating Biophysics and Biochemistry into Cellular Growth and Morphogenesis. Rhythms in Plants: Dynamic Responses in a Dynamic Environment. , (2015).
- Wudick, M. M., et al. CORNICHON sorting and regulation of GLR channels underlie pollen tube Ca2+ homeostasis. Science. 360 (6388), 533-536 (2018).
- Hoffmann, R. D., et al. Plasma membrane H+-ATPases sustain pollen tube growth and fertilization. Nature Communications. 11 (1), 1-15 (2020).
- Michard, E., et al. Glutamate receptor-like genes form Ca2+ channels in pollen tubes and are regulated by pistil D-serine. Science. 332 (6028), 434-437 (2011).
- Damineli, D. S., Portes, M. T., Feijó, J. A. Oscillatory signatures underlie growth regimes in Arabidopsis pollen tubes: computational methods to estimate tip location, periodicity, and synchronization in growing cells. Journal of Experimental Botany. 68 (12), 3267-3281 (2017).
- Li, K., et al. An optimized genetically encoded dual reporter for simultaneous ratio imaging of Ca2+ and H+ reveals new insights into ion signaling in plants. New Phytologist. 230 (6), 2292-2310 (2021).
- Sadoine, M., et al. Designs, applications, and limitations of genetically encoded fluorescent sensors to explore plant biology. Plant Physiology. 187 (2), 485-503 (2021).
- Grenzi, M., et al. Illuminating the hidden world of calcium ions in plants with a universe of indicators. Plant Physiology. 187 (2), 550-571 (2021).
- Munglani, G., Vogler, H., Grossniklaus, U. Fast and flexible processing of large FRET image stacks using the FRET-IBRA toolkit. PLoS Computational Biology. 18 (4), 1009242 (2022).
- Ridler, T., Calvard, S. Picture thresholding using an iterative selection method. IEEE Transactions on Systems, Man, and Cybernetics. 8 (8), 630-632 (1978).
- Portes, M. T., Feijó, J. . Growing Arabidopsis pollen tubes expressing genetically encoded reporters for calcium and pH. , (2023).
- Candeo, A., Doccula, F. G., Valentini, G., Bassi, A., Costa, A. Light sheet fluorescence microscopy quantifies calcium oscillations in root hairs of Arabidopsis thaliana. Plant & Cell Physiology. 58 (7), 1161-1172 (2017).
- Romano Armada, N., et al. In vivo light sheet fluorescence microscopy of calcium oscillations in Arabidopsis thaliana. Methods in Molecular Biology. 1925, 87-101 (2019).
- Krebs, M., et al. FRET-based genetically encoded sensors allow high-resolution live cell imaging of Ca2+ dynamics. The Plant Journal. 69 (1), 181-192 (2012).
- Lee, T. -. C., Kashyap, R. L., Chu, C. -. N. Building skeleton models via 3-D medial surface axis thinning algorithms. CVGIP: Graphical Models and Image Processing. 56 (6), 462-478 (1994).
- Nunez-Iglesias, J., Blanch, A. J., Looker, O., Dixon, M. W., Tilley, L. A new Python library to analyse skeleton images confirms malaria parasite remodelling of the red blood cell membrane skeleton. PeerJ. 6, 4312 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados