Method Article
Yaşayan Hücrelerde Protein-fragmanı tamamlama Assay (PCA) ile genom Protein-protein etkileşimi Eleme
Bu Makalede
Özet
Proteins interact with each other and these interactions determine in a large part their functions. Protein interaction partners can be identified at high-throughput in vivo using a yeast fitness assay based on the dihydrofolate reductase protein-fragment complementation assay (DHFR-PCA).
Özet
Proteins are the building blocks, effectors and signal mediators of cellular processes. A protein’s function, regulation and localization often depend on its interactions with other proteins. Here, we describe a protocol for the yeast protein-fragment complementation assay (PCA), a powerful method to detect direct and proximal associations between proteins in living cells. The interaction between two proteins, each fused to a dihydrofolate reductase (DHFR) protein fragment, translates into growth of yeast strains in presence of the drug methotrexate (MTX). Differential fitness, resulting from different amounts of reconstituted DHFR enzyme, can be quantified on high-density colony arrays, allowing to differentiate interacting from non-interacting bait-prey pairs. The high-throughput protocol presented here is performed using a robotic platform that parallelizes mating of bait and prey strains carrying complementary DHFR-fragment fusion proteins and the survival assay on MTX. This protocol allows to systematically test for thousands of protein-protein interactions (PPIs) involving bait proteins of interest and offers several advantages over other PPI detection assays, including the study of proteins expressed from their endogenous promoters without the need for modifying protein localization and for the assembly of complex reporter constructs.
Giriş
Protein etkileşim ağları (PIN) proteinleri fonksiyonel hücrede 1 organize nasıl bir düşük çözünürlüklü haritası sunuyoruz. Her fiziksel iki protein arasında bağlantı veya protein-protein etkileşimi (PPI) örneğin protein kompleksleri içinde bulunanlar gibi zaman içinde istikrarlı bir ilişki, temsil edebilir ve o hücrenin yapısal organizasyonu katkıda bulunur. Bu bağlantıları da etkinlik, istikrar, yerelleştirme ve iki ortağı etkileşimlerini düzenleyen geçici dernekleri temsil edebilir. Belirli bir proteinin fiziksel etkileşim ortakları belirlemek nedenle bu proteinin 2,3 fonksiyonu ve yönetmelik zengin bilgi sağlar. Bu nedenlerden dolayı, büyük çabalar Escherichia coli 4-6, Arabidopsis thaliana 7, Saccharomyces cerevisiae 8-12, Drosophila melanogaster model organizmaların içine PIN haritalanması doğru konulmuştur/ Em> 13, Caenorhabditis elegans 14 ve Homo sapiens 15. Bu çalışmalar proteinler daha önce bilinmeyen fonksiyonları ile proteinler üzerinde hücreye ve böylece önemli bilgilere organize nasıl önemli bilgiler vermiştir.
Birkaç stratejiler PIN incelemek için yılda geliştirilmiştir. Bu teknolojileri genel (16-18 gözden) onlar PPI sağladığınız bilgilerin türüne göre üç kategoride toplanabilir. Birincisi, maya iki-hibrid ve türevleri 19 dayanır. Bu teknolojiler ikili ağları inşa sağlar proteinlerin çiftleri arasında doğrudan ilişki, hakkında bilgi vermek. ikinci bir familyası, yem proteinlerinin eğilim saflaştırılmasına ve bu kütle spektrometresi 20 ardından afinite temizliği gibi ilgili ortakları belirlenmesine dayanmaktadır. Bu yaklaşımlar doğrudan proteinlerin gruplarını belirlemekya da dolaylı olarak, genel olarak, bir kararlı bir şekilde, eşlik eden ve protein kompleksleri tespit etmek çok güçlüdür. Üçüncü yaklaşım protein fragmanı tamamlama deneyleri (PCA) 11,21 dayanmaktadır. Bu proteinler arasında doğrudan ve yakın dernekler tespit veriyor gibi bu yaklaşım, iki eski yaklaşımlar arasındaki çözünürlük orta düzeyde sağlar. Son zamanlarda 18 gözden gibi her tekniği, kendi güçlü ve zayıf yönleri vardır.
onun proteomu PPİ önce tahlil edilmiştir tespit etmek için diğer model ökaryotlarda ve yüksek verimli deneyler için olduğundan daha az karmaşık ve daha verimli olduğu için en iyi tarif edilen ökaryotik bir PIN kısmen çiçek mayası Saccharomyces cerevisiae'nin kadar bir kişi tarafından bir Bu model organizma 9-12 uygulanan. Maya sistemi için özellikle güçlü bir yöntem dihidrofolat redüktaz protein fragmanı tamamlama analizi (DHFR PCA) olmuştur bir deneydirStandart ve tedirgin koşullarda 11,22-26 maya PIN incelemek için farklı bağlamlarda kullanılan. Bu yöntem endojen ifade seviyeleri ve kantitatif bir şekilde 27 etkileşim ortakları 11,21 yerli subselüler lokalizasyonu hem de belirli bir yem proteini doğrudan ve yakın doğrudan PPİ algılama sağlayan bir hayatta kalma tahlil dayanmaktadır. Bu deneyi (yüksek-yoğunluklu koloni diziler örneğin koloni büyüklüğü) kullanılarak elde edilen sinyal, vahşi tür hücrelerinde birine hemen hemen eşit bir hücresel ortamda yem ve av arasında oluşan protein kompleksleri miktarını yansıtır. Deney folat metabolizmasında rol oynayan bir raportör enzim olarak yeniden oluşturulmasına dayanır, dihidrofolat redüktaz (DHFR), burada ilgilenilen iki proteinlere kaynaşmış DHFR iki tamamlayıcı parçalan, iki proteinin, etkileşime girdiğinde yakın ettirildiği bu da enzim aktivitesinin 1 tersine çevrilebilir yeniden yol açarOrta içeren metotreksat gerginlik 1 ve büyüme (MTX; Şekil 1). Bu bileşik, endojen DHFR enziminin ama tahlilinde 28 kullanılmaz, mutasyona uğramış bir inhibe eder. PCA suşları iki koleksiyonları içeren bir Mata ile suşları ~ 4.300 ORF DHFr F kaynaşmış bir [1,2] fragmanı ve ORF DHFr [3] fragmanına erimiş bir, için satın alınabilir ile suşları α ~ 4.800 MAT içeren bir Herhangi bir laboratuvarda küçük veya büyük ölçekte DHFr-PCA uygulamak. Burada, biz bir yem protein ve bu testi kullanılarak ~ 4,800 av proteinleri arasındaki PPI için ekrana genel ancak ayrıntılı protokol açıklar.
Protokol
1. İnşaat / Yem Suşlarının doğrulama
- Ilgi yem suşu Mata DHFr F [1,2] koleksiyonunda mevcut ise adım 1.1.1 açıklandığı gibi, adım 1.1.2 anlatıldığı gibi aksi gerginlik inşa, toplama onu almak.
NOT: Bu koleksiyon DHFr F [1,2] koleksiyon daha fazla suşları içeren burada açıklanan protokol, bir DHFr F avlayan bir yem ve DHFr F [3] toplama gibi [1,2] suşu kullanır. Bununla birlikte, tek bir interactome yüksek kapsama gerektiriyorsa yem soyu, DHFR K [3] toplama bölgesi veya her iki yönde de kullanılabilir ise yaklaşık ekranı başka bir şekilde yapılması mümkündür.- Bir saat boyunca buz üzerinde yem suşu içeren gliserol stok plaka çözülme. % 95 etanol kullanılarak plaka kapsayan alüminyum folyo sterilize. Pierce steril ucu ile folyo, seçici maya özü pepton üzerindeki gliserol stokunun hücreleri ve çizgi 2-3 ul tekrar süspansiyon aşağı yukarı pipet vedekstroz (YPD) + 100 ug tek koloniler izole etmek için / ml Nourseothricin (Nat). 30 ° C'de iki gün için inkübe edilir.
- Bir yem PCA soyunun İnşaatı (Mata DHFr F [1,2]).
- Plazmid pAG25-bağlayıcı-DHFr F DHFr F yükseltmek, yüksek sadakat polimeraz ve bir standart PCR protokolü kullanılarak [1,2] kaset [1,2] -ADHterm taşkın oligonükleotidler kullanılarak ORF en son 40 bp ile homolog biter durdurma kodonu (ileri doğru primer) ve genin 3 'UTR (Ters primer) (Şekil 1A) ilk 40 bp hariç 3'-ucu.
- 29 (Şekil 1A) standart olarak LiOAc / PEG maya dönüşüm protokolü kullanılarak (genellikle BY4741 suşu olarak) yetkili maya hücrelerine PCR ürünü Transform.
- Seçici YPD'de + Uyruk ortamında plaka pozitif transformantların izole etmek.
- Uygun DHFr F [1,2] füzyon onaylamak için izole koloniler üzerinde bir tanı koloni PCR gerçekleştirin. Pr kullanınimers gen kodlama dizisinin (İleri oligo), DHFR füzyon üst akışında yaklaşık 100 bp ve 2) kaset (oligo Ters) (Şekil 1B) ADH sonlandırıcısı olarak) 1 tavlanması.
- Uygun gen füzyonu onaylamak için Sanger dizileme ile PCR ürünü sırası.
- -80 ° C'de% 25 gliserol içinde teyit yem suşu Arşiv.
NOT: protokolü bu aşamada durdurulmuş olabilir.
2. Pin-aracı Sterilizasyon ve Baskı İşlemleri
NOT: Aşağıda açıklanan sterilizasyon işlemi BM3-BC (S & P Robotik) robotik platformu tarafından manipüle pin-araçlar için optimize edilmiş, ancak aynı zamanda diğer platformlara adapte edilebilir. Bu bölüm, protokol geri kalanı için başka bir ortama hücreler aktarmak için kullanılır Pin aracı sterilizasyon ve baskı işlemlerini açıklamaktadır. Bu rutinleri gerçekleştirmek için kullanılan içi komut istek üzerine temin edilebilir. Not that tüm adımları manuel pim aracı 30 ile bir robot platformun gerek kalmadan gerçekleştirilebilir.
- Robotik platformda uygun pin-aleti monte edin.
- Aşağıdaki gibi temizlik ve ıslak istasyonları hazırlayın:
- Su banyosu istasyonunda steril su, 500 ml ilave edilir.
- Fırça istasyonunda steril su 320 ml ilave edilir.
- Bir agar plakasından sıvı kültürleri ihtiva eden bir mikro-titre plaka çoğaltma zaman agar plaka, ya da 400 ml (katılaşmış ortam 35 ml içeren, 86 x 128 mm omnitray) agar plakasından çoğaltma zaman sonikatörde,% 70 etanol içinde 380 ml ilave edilir.
- Islak istasyonu (steril boş omnitray oluşan) steril su, 35 ml ilave edilir.
- Robotik platform gerektirir her gün başında, sonikatör banyosunda bir dakika pin-araçlar beş kez sterilize. Beş dakika robot muhafaza sterilize etmek için arada, UV lambasını açmak.
NOT: Bu soymak eğer gerekli değildirot steril kaputun altında yer almaktadır. - Aşağıdaki gibi her pin-alet çoğaltma turunda arasında, pin-aracı sterilize:
- Hücre kümeleri kaldırmak için su banyosu istasyonunda pin-aracını 10 sn için beş kez bekletin.
- Iki kez ileri ve geri fırça istasyonu pin-aracı ıslatın.
NOT: Döner fırça artık hücreleri çıkarmak olacaktır. - Sonikatör istasyonunda 20 saniye boyunca iki kez pin aracı ıslatın.
NOT: iğneler üzerinde hücreleri Kalan sonication tarafından kaldırıldı veya etanol tarafından öldürülen edilecektir. - Pim daldırma derinliği doğru sterilizasyon sağlamak için her ardışık banyo artar emin olun.
- 25 sn için hava kurutucu istasyonunda pin-aracı kurulayın.
- Kaynak plakaları üzerinde hücrelerin almadan önce, bir omnitray steril su 35 ml içeren ıslak istasyonunda işaretçilerini ıslak.
- Kaynak plaka koloniler iki kez işaretçilerine batırın.
- Ag dokunarak taze döktü hedef plakası Yazdırar yüzeyi iki kez (bundan sonra "baskı" bir dizi eylem olarak da adlandırılır).
Otomatik Robotik Platformu Kullanma 1.536 Diziler DHFR F [3] Collection 3. Yoğunlaşma
- Buz üzerinde çözülme, DHFR F [3] bir koleksiyonu olan (60 ve 96-delikli plakalar), ve L-DHFR F [3] Kontrol soyu, DHFR F içerir (Şekil 1C), [3] fragmanı ile dolu bir ilave 96 oyuklu plaka ve üst baş bağlayıcı bir negatif kontrol olarak tek başına ifade edilmiştir.
Not: ilke olarak, bu fragman bir DHFR fragmanı füzyon proteini (ayrıntılar için tartışmaya bakınız) ile etkileşime girmemelidir. - Kuyular arasında çapraz-bulaşma riskleri önlemek için alüminyum folyo çıkarmadan önce santrifüj plakaları (hızlı sıkma).
- 384 suşları (Şekil 2A) ve 16 dizileri üzerine toplama yoğunlaşır. Bunu yapmak için, her 384 dizisi için, bir seçici YPD'de + 250 ug / ml Higromisin B (HygB) dört kadran üzerindeki baskı, dört gliserol plakaları th kullanarak omnitraye 96 pin-aracı (burada, 384 dizisi her 2 x 2 matris düzeninde 96 pozisyonları oluşan, dört eşit interspaced kadranda ayrılabilir). Tam dört 1536 dizileri dolgu 64 plakalarının son kümesi elde etmek için 60 diğer plakalar arasında L-DHFR F [3] negatif kontrol ihtiva eden dört adet 96 oyuklu plakalar yerleştirin. Aşama 2'de tarif edildiği gibi, her çoğaltma çevrimi ile pim aracı sterilize.
Not: Dört son 1,536 dizilerin her birinde böyle bir plaka elde etmek için L-DHFR F [3] plakalar yerleştirin. - 30 ° C'de iki gün süre ile inkübe edin. NOT: Bu aşamada, DHFR toplama agar plakaları üzerinde en fazla bir ay boyunca 4 ° C'de 384 biçimde depolanabilir.
- 1536 soylarının (Şekil 2A) dört dizilerine toplama yoğunlaşır. Bunu yapmak için, her bir 1536 dizisi için, burada (384 pim aracı kullanarak aşama 3.2'de olduğu gibi, aynı ortamın bir plakanın dört çeyrek ile 384 soylarının dört dizi baskı, bir 1,536 dizisi dört e bölünebilirqually) 2 x 2 matris düzeninde her 384 pozisyonların oluşan kadranlar interspaced.
- 30 ° C'de iki gün süre ile inkübe edin. NOT: Bu aşamada, DHFR toplama agar plakaları üzerinde en fazla bir ay için 4 ° C'de bir 1536 biçimde depolanabilir.
- Bir 1.536 pin-aracını kullanarak koloni boyutu standardize etmek, aynı ortamda dört dizileri çoğaltın.
- 30 ° C'de iki gün süre ile inkübe edin.
4. Yüksek verim DHFr-PCA Prosedürü
- 50 ml'lik bir tüp (Şekil 2B) 'de, sıvı YPD + Nat 20 ml aşama 1.1.1 ya da 1.1.2 elde edilen yem suşu (DHFR K [1,2] füzyon) içindeki bir kültürü inoküle.
- Kültür doygunluk ulaşmasına izin verecek biçimde, 250 rpm'de çalkalanarak 30 ° C'de iki gün için inkübe edilir.
- Inkübasyon iki gün, plaka sonra YPD + Nat kültürün 5 ml omnitray. Hücreler, 5-10 dakika için yüzeye adsorbe olsun ve fazla sıvı (Şekil 2B) çıkarın. Tekrar tAkil üç suret için.
- 30 ° C'de iki gün için inkübe edilir.
- Dört kat daha fazla her bir hücre çim kullanılarak 1.536 pin-aracı ile (DHFr F [3] toplama × üç suret dört tabak çiftleşme için yeterli) adımlar 4.3 ve 4.4 12 YPD plakaları yem gerginlik yazdırın.
- 1.536 pin-aracı (Şekil 2C) kullanarak yem hücreleri üstünde DHFr F [3] koleksiyonu uygun bir dizi yazdırın.
- Suşu 30 ° C'de iki gün süre ile inkübe edilmesi ile çiftleşme olsun.
- YPD + HygB + Nat (Şekil 2C) içeren omnitrays koloniler yazdırarak diploid hücreleri seçin.
- 30 ° C'de iki gün için inkübe edilir.
- Adım 4.8 ve 4.9 (Şekil 2B) 'de tarif edildiği gibi tekrar diploit seçim.
- . Bir toksik bileşik olarak MTX ile çalışırken her zaman eldiven giyin dikkatli olun, prote: MTX (MTX ortamı) bir gün içeren medya kullanmadan önce bu adımları 21 (DİKKAT aşağıdaki tabak hazırlayınction gözlük ve işleme bir laboratuvar önlüğü) (Şekil 2C):
- 10x Lys hazırlayın / deiyonize su içinde / ade amino asit damla-out bir araya geldi. Büyük miktarlarda gerekirse, 0.2 mikron şişe üst huni (Tablo 1) 0.2 mikron steril şırınga filtre kullanarak steril bir şişede açılan Çıkış Filtre-sterilize veya.
- Dimetil sülfoksit (DMSO) içinde 10 mg / mL metotreksat stok çözelti hazırlayın. Çözeltisinin hazırlanması hemen sonra kullanarak ve -20 ° C 'de kalan dondurma. O ışığa olduğu gibi ışıktan koruyun. Bunu çözündükten sonra MTX tekrar dondurulmamalıdır.
- Aşağıdaki gibi (. Ortamı maddeleri ve miktarları Tarassov diğ 11 kullanılanlar ile aynıdır) ortam hazırlanması:
- Ortamın bir litre için, iki ayrı şişelerde karışımı: 1) amino asit içermeyen ve amonyum sülfat olmayan maya azot baz 6.69 g deiyonize su, 330 ml; Iyonu giderilmiş su ağarla ve 500 ml 2), 25 gr.
- 20 dakika boyunca 121 ° C'de otoklav şişe hazırlanmıştır.
- En az bir saat süreyle 55 ° C'de bir su banyosu içinde sıcaklığı dengelenmesi.
- İki tüp birlikte karıştırın ve steril% 40 glikoz, 50 mi, 100 10x steril damla Çıkış ml ve metotreksat 10 mg / ml, 20 ml ekleyin.
- 35 ml dökün omnitrays ortamın (aşağıda Not bakınız). En az bir saat bir buçuk katılaşmaya edelim. Işıktan Kalkan plakaları.
Not: Burada omnitray başına ortam 35 ml dökme tüm alt aşamada önemli olan eşit plaka kalınlığını sağlamak için çok önemlidir.
- Robot platformu ile veya üniforma plaka aydınlatma ile normal bir dijital kamera ile diploid seçimi ikinci tura görüntü plakaları. Mansap analizini yaparken diziler boş pozisyonları belirlemek için bu resimleri kullanın. Kameranın parametreler her zaman aynı ve robot ışık açık olduğundan emin olun.
- MTX medya usi diploid hücreleri yazdırmak1.536 pin-aracı ng.
- Kurumasını önlemek için plastik torbalar içinde, 30 ° C'de dört gün boyunca inkübe edilir.
- Adım 4.11 tarif edildiği gibi MTX ortamı içeren omnitrays bir ikinci parti hazırlayın.
- Inkübasyon dört gün sonra, görüntü plakalar robot platformu veya düzenli dijital kamerayı kullanarak. Kameranın parametreler her zaman aynı ve robot ışık açık olduğundan emin olun.
- MTX medyanın ikinci toplu üzerindeki hücrelerin çoğaltarak MTX seçimi ikinci tura gerçekleştirin.
NOT: Bu PCA suşları arka plan büyümesini azaltmak ve kantitatif çözünürlüğü artacaktır. - Kurumasını önlemek için plastik torbalar içinde, 30 ° C'de dört gün boyunca inkübe edilir.
- Gibi görüntü plakaları adım 4.16 tarif.
5. Görüntü Analizi
- Özel ImageJ ile 31 komut koloni dizileri görüntüleri analiz ya da sömürge, Ht koloni ızgara analizörü, Hücre profiler, Koloni Görüntüsü olarak yayınlanan yazılımlar kullanarak(32 derlenmiş) r, ScreenMill, YeastXtract ve gitter. Görüntü analizi çıkışı bir ya da her dizinin her pozisyon için koloni boyutları içeren çok sayıda elektronik tablolar, tüm alt analizler için bu koloni boyutları kullanmalısınız.
NOT: Bu çalışmada, biz Leducq diğ özel ImageJ komut dosyası kullanılan 33 (Daha fazla bilgi için tartışma bölümüne bakınız)..
6. Veri Analizi
NOT: Görüntü analiz sonuçları gibi excel veya R 34 gibi bir betik dili kullanarak bir tabulator işlenebilir. Aşağıdaki adımlar, özel bir ImageJ 31 komut dosyası kullanarak prosedürü tarif.
- Özel bir komut dosyası kullanarak, görüntü analizi çıktı dosyalarını birleştirmek ve plaka ile her satır açıklama ve Ek Tablo 1 gibi bilgileri süzün.
- Koloni boyutu değerlerini (Entegre yoğunluğu veya koloni alanı 2 log dönüşümü, burada, sütun "IntDenBackSub & #8221; Ek, Tablo 1) kullanılmıştır.
NOT: Değer dağılımı Şekil 3A olarak bakacağız. - Her plaka medyan değerinin çıkarılmasıyla bu değerleri Normale.
NOT: Otomatik görüntü elde etme eşitsiz medya miktar veya değişiklik sonucu, ve inter-suret varyans (Şekil 3B) azaltır olabilir plaka önyargı Bu adım, kontrol. - Çoğaltır deneylerin tekrarlanabilirliğini değerlendirmek için birbirlerine (Şekil 3C) ile ilişkili olduğunu doğrulayın.
- Olmayan etkileşim yem-av çiftleri etkileşim ayırt etmek için, L-DHFr F [3] kontrolleri dağılımı 95 persentil karşılık gelen yüksek güven eşiğini ayarlayın.
NOT: Bu deneyde, bu durum 3.39 (Şekil 3D) karşılık gelmektedir. Seçenek olarak ise, bu tür BioGRID 35 bildirilenler gibi bilinen fiziksel uygulayıcı ile üst üste binme göre bir eşik kullanılabilir.Daha fazla bilgi için tartışma bakın. - DHFr-PCA ekranlarında yalancı pozitif etkileşimler dahil (Daha fazla bilgi için tartışma bakınız) ve (sütununda "1" olarak tanımlanan "süzülmüş") Ek Tablo 2'de listelenen herhangi bir yem için, filtre tespit avlıyor.
- (Ek Tablo 2 sütun "cevaplarını bulacaksınız"), her etkileşim üç tekrardan log2 normalize koloni boyutları ortalama.
Fiziksel interactors 7. Doğrulama Küçük ölçekli deneyler kullanma
Not: Bir Yukarıda puan veya katı veya sıvı MTX ortamında büyüme tahlili kullanarak küçük ölçekli deneysel tasarım DHFr-PCA analizi kullanılarak valide edilebilir uygulanan eşiğe yakın olan özel ilgi herhangi ÜFE. Aşağıdaki adımlar elle diploid PCA suşları inşa ve MTX orta nokta analizleri gerçekleştirmek prosedürünü göstermektedir. deneyci, tüm Mutlaka bu adımları uygulamanız gerekiry kontroller (Bait-DHFr F [1,2] x U-DHFr F [3], Fermuar-bağlayıcı-DHFr diploid suşları ve bağlayıcı-DHFr diploid soy).
- 1.1.2.6 üretilen yem suşu gliserol stokunun Levha 2-3 ul), L-DHFr F [3] Kontrol suşu, YPD + Nat, YPD Fermuar-bağlayıcı-DHFR'nin ve bağlayıcı-DHFR'nin diploit suşları + HygB iki kez YPD + NAT + HygB ortam, sırasıyla.
- Yerine YPD + Uyruk orta YPD + HygB ortamı üzerinde zorlanma DHFr F [3] koleksiyonunda ilgi av Al ve adım 1.1.1 yönergeleri izleyin, ama çizgi.
- Av lokus ve sırayla ürün, [3] füzyon DHFr F onaylamak için 1.1.2.4 olarak bir teşhis PCR gerçekleştirin.
- Çiftleşmeye haploid suşlar ile Sıvı YPD besiyeri içinde 1 ml ile inoküle (yem yem x U-DHFR F [3], kontrol, yırtıcı x) ve diploitler oluşmasının sağlanması için 30 ° C'de en az iki gün büyür.
- Katı YPD + HygB + Uyruk ortamında 7.4 kültürün 4-5 ul çizgiler ile diploidlerin seçin. 30 ° C'de iki gün büyütün.
- SelecT, bir izole edilmiş koloni ve sıvı kültür (1 mi) içinde gece boyunca büyümeye büyüme deneyi yapmak için.
- Kullanmadan önce bir gün MTX ve DMSO (MTX ortamı aynı maddeler, ancak MTX olmadan) tabak hazırlayın. Daha fazla bilgi için adım 4.11 bakın.
- Kontrolün (DMSO) ve seçme plakaları (MTX), farklı kültürlerde seri dilüsyonları tatbik edilmesi suretiyle büyüme deneyi yapın.
- OD = 1 ön kültür sulandırmak.
- Bir steril 96 oyuklu bir plaka içerisinde (625 seyreltme faktörü kadar), beş misli dilüsyonları yapın.
- Nokta PCA medya (DMSO ve MTX) her seyreltme 4 ul.
- Kurumasını önlemek için plastik torbalar içinde 30 ° C'de inkübe edilir.
- Robot platformu veya normal bir dijital kamera kullanarak 1 inkübasyon 7 gün arasında görüntü plakaları.
Sonuçlar
Ek Tablo 2 Nup82, DHFR F yem olarak [1,2] fragmanına kaynaşık maya proteini kullanılarak elde edilen temsili sonuçlar bir örnektir. L-DHFR F tanımlanmış eşik [3] kontrol yüksek güven isabet (Şekil 3D ve 3E) belirlemek için deneysel bir eşik değeri olarak kullanılabilir. Alternatif olarak, puan sıralamasında Gen Ontoloji zenginleşme gerçekleştirmek için kullanılan veya fonksiyonel diğer altın standartlarına dayalı 37 36 analizler yapılabilir. yem bilinen fiziksel interaktörler BioGRID 35 gibi veritabanlarından alınan ve veri (Şekil 3E & 3F) üstüne eklenebilir. Bu örnekte, beş, sekiz dışarı yüksek güven hit önce Nup82 interactors olarak bildirilmiştir ve iki Nup82 subcomplex, Nup116 ve Nup159 (Şekil 3F ve 3G) bir parçasıdır. kompleks, Nsp1 diğer üyesi, bir interac göstermezBizim deneyde yon. İki avlayan, Ade17 ve (değil Şekil 3F gösterilmiştir) Tef2, uygulanan sabit eşiğin üstünde puan vardı, ama bunlar onlar (yayınlanmamış sonuçlar) gerçekleştirdik PCA ekranlarında hemen hemen her yem proteini ile etkileşim gibi yanlış pozitif olması muhtemeldir. Öte yandan, Pex30 Nup82 yeni bir fiziksel interaktör temsil edebilir ve biz düşük-throughput (Şekil 3G) ile DHFr-PCA kullanarak bu etkileşimi teyit başardık. Pex30 bir peroksisomal zar proteinidir ve birkaç doğrudan etkileşimler çekirdek gözenek kompleks (NPC) ile bu organel arasındaki bildirilmiştir. Iki melez eleme Pex30 38 fiziksel uygulayıcı olarak, diğer iki NPC proteinini Nup53 ve Asm4 (Nup59) tarafından tanımlanan, ve Pex30 ve Nup170 arasındaki genetik etkileşimin 39 bildirilmiştir. Diğer iki etkileşim eşleri, tespit, Nup120 ve Nup85 (Şekil 3F ve 3G), yeteneğini gösteren Nup82 alt kompleksin bir parçası değildirDHFr-PCA içinde ve daha geniş kompleksleri 11 subcomplexes arasındaki etkileşimleri tespit etmek.
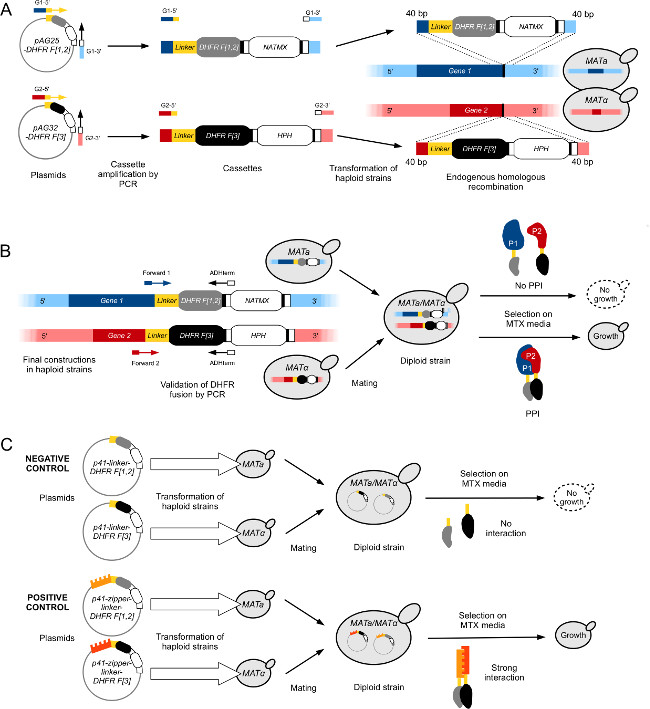
Şekil 1:. Haploid Mata ve MAT α soylarının yüksek verimli, DHFR-PCA için teknikler maya cinsleri (. Leducq ve diğerlerinden adapte Şekil 2012 33), (A) Yapı DHFR ile Gene1 (G1) Gene2 (G2) birleşmesi F sırasıyla [1,2] -NatMX ve DHFr F [3] -HPH kasetleri,. Kasetler, G1-3 pAG25-DHFR F [1,2] ve pAG32-DHFR F [3] ileri primerler G1-5 've G2-5' ile geri primerler plasmidlerden büyütülmüş 've G2-3', ve daha sonra homolog rekombinasyon ile hedef genin 3 'ucunda genomuna eklenir. Elde edilen proteinler, P1 ve P2, ayrı ayrı D kaynaşmışHFR F [1,2] fragmanı (Mata) ve esnek bir bağlayıcı vasıtasıyla, DHFR F [3] fragmanı (MATα). (B), inşaat doğrulanması (A) Gene1 en ve Gene2 en ORF'ler arasında bağlantılarını sıralamak için gerçekleştirilir DHFR kasetleri. çiftleşme tipleri karşı inşa PCA suşları daha sonra bir diploid oluşturmak evlendirilen vardır. Iki tamamlayıcı DHFR fragmanları, DHFR enziminin aktivitesini yeniden oluşturulmasını P1 ve P2 arasındaki bir etkileşim ile yakınlık içine getirildiğinde, eğer diploid suşlar MTX ortamında büyür. (C), Yapı kontrol diploid PCA DHFR PCA ekranlar için zorlamaktadır. Negatif kontroller (L-DHFr) plazmidler p41-bağlayıcı-DHFr F [1,2] ve p41-bağlayıcı-DHFr F [3] 11, sırasıyla ayrı ayrı haploit Mata ve MATα suşları dönüştürerek inşa edilir. İki soy, DHFR fragmanları mümkün olduğu bir negatif kontrol diploid soy elde edilir jantbirbirlerine (üst) tamamlayacak. Pozitif kontroller negatif kontroller için aynı yaklaşımı kullanılarak inşa edilir, ancak plazmidler haploit suşları (p41-fermuar-bağlayıcı-DHFr F [1,2] (p41-ZL-DHFr F [1,2]) ve p41 dönüştürülmüş -zipper-bağlayıcı-DHFR F [3] (p41-ZL-DHFR F [3])) DHFR aktivitesi tekrar oluşturur, güçlü ve konstitütif etkileşimine yol açar, tamamlayıcı, DHFR fragmanlara bağlanmış iki GCN4 paralel lösin zipper fragmanları içerir (altta ). Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
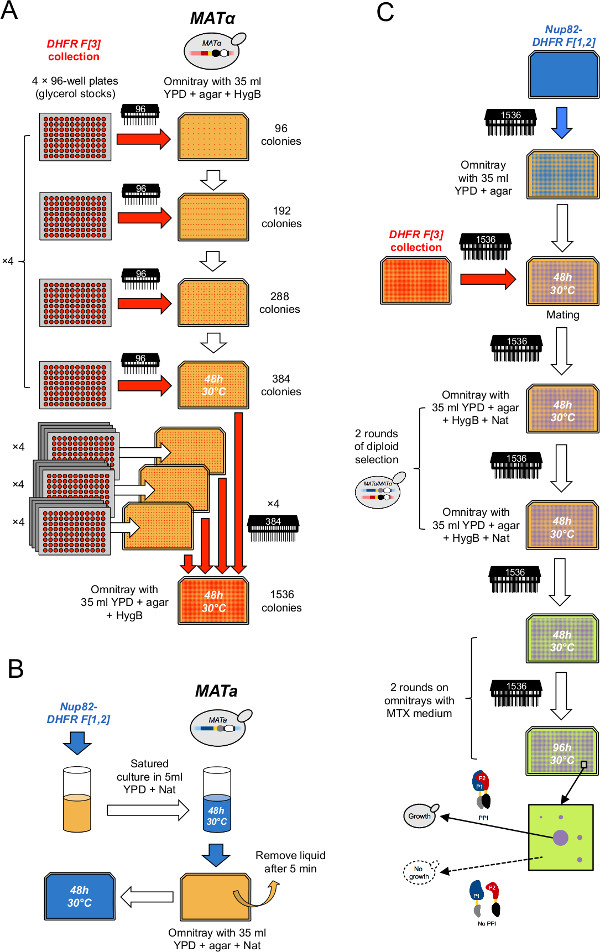
Şekil 2: Yüksek verim, DHFR-PCA prosedürü (Leducq diğ 33 uyarlanmıştır Şekil.) (A), DHFR F [3] toplama α MAT. yoğunlaştırma iki ardışık mermi ile 1536 biçimde yoğunlaştırılır. İlk olarak, gliserol stok plakaları 96 pin-aracını kullanarak 384 formatında seçici YPD + HygB ortamında dörtlü gruplar tarafından birleştirilir. İkincisi, 384 dizileri 384 pin-aracını kullanarak bir 1536 formatında seçici YPD + HygB ortamında dörtlü gruplar tarafından birleştirilir. MATa PCA yem suşu (B) Hücre çimler içinde yem gerginlik doymuş kültürünü büyüyen hazırlanır Bir YPD + Nat omnitray. (C) Bu çimler üzerinde kültürünü kaplama seçici YPD + Uyruk orta ve YPD ortamında DHFr F [3] koleksiyonu ile yem gerginlik çiftleşmek için kullanılır. Hücreler arda diploidlerin ve iki kez PCA gerçekleştirmek için MTX ortamında seçmek için YPD + HygB + Uyruk ortamına kez aktarılır. DHFr fragmanları yem ve av proteinleri arasındaki etkileşim, aşağıdaki birbirini tamamlayacak eğer Büyüme sadece MTX ortamında görülecektir.e.jpg "target =" _ blank "> bu rakamın daha büyük bir versiyonunu görmek için burayı tıklayınız.
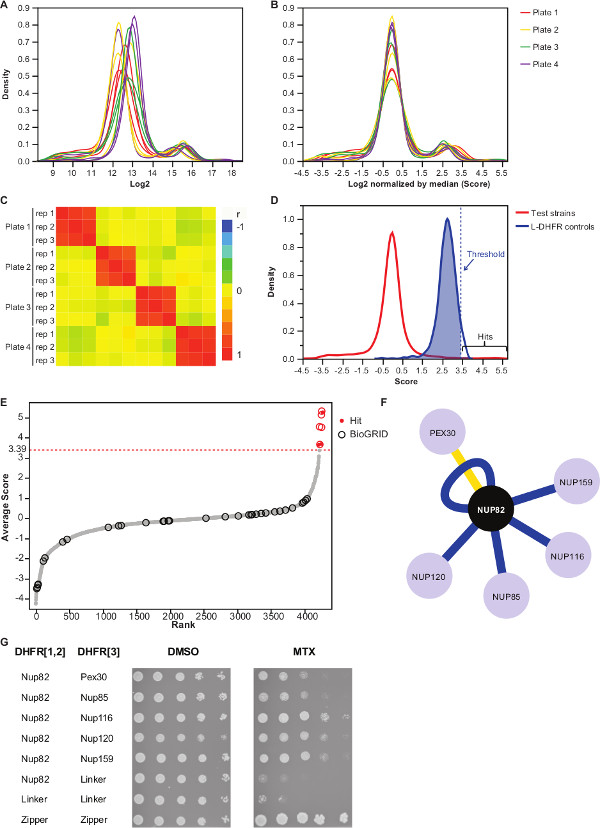
Şekil 3,.:., Her görüntünün medyan, Veri normalleşme bir anlam eşik değerine, ve pozitif etkileşimlerinin belirlenmesi belirlenmesi adımları analiz koloni boyutu (A) yoğunluk dağılımı, her plaka (2 log) (B) Normalleştirme önyargılara düzeltir Plaka-plaka etkileri ile ilişkili. (C) Heatmap prosedürü röprodüsibilitesini teyit plakalar arasında Spearman korelasyon katsayısı gösteren. test PPİ için puanları (D) Dağıtım ve L-DHFr F [3] kontrolleri. Sert eşik 95 inci persentil ayarlanabilir L-DHFr F [3] noktalı dikey tarafından temsil edilen yüksek güven PPIs tanımlamak için dağıtım (Her yırtıcı ortalama puanı hattı). (E) Derece sipariş dağılımı. BioGRID 35 Nup82p önceden bildirilen fiziksel etkileşim ortakları çevreler tarafından tespit edilir ve bu çalışmada bildirilen bu kırmızı nokta ile tanımlanır. (D) 'de tanımlanan eşik noktalı bir çizgi olarak gösterilir. (F) Ağ Bu çalışmada belirlenen yüksek güven PPIs gösteren. Mavi kenarları daha önce bildirilen göstermek fiziksel interaktörler (BioGRID 35) ve sarı kenarları Pex30 ile daha önce bildirilmemiş etkileşim gösterir. (G) Nup82-DHFr F [1,2] ve av-DHFr F [3 içeren PCA Diploid suşları Spot-seyreltme deneyi ] çiftleri Bu çalışmada Nup82p fiziksel interactors olarak belirledi. Büyüme deney DMSO ortamında (MTX çözücü, sol panel) ve MTX ortam (sağ panel) ile gerçekleştirilmiştir. F Nup82-DHFR oluşan Negatif kontroller, [1,2] - Bağlayıcı-DHFR K [3] ve Bağlayıcı-DHFR F [1,2] - Bağlayıcı-DHFR K [3] ve bir pozitif kontrol t oluşanİki lösin fermuar kısımları arasındaki o kuvvetli etkileşim (fermuar-DHFr F [1,2] - fermuar-DHFr F [3]) dahil edildi. MTX ortamında negatif kontroller üstün hücre büyümesi fiziksel etkileşim olarak yorumlanmalıdır. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
| Amino asit | Miktar (g) |
| Adenin sülfat * | 0.2 |
| L-Triptofan | 0,4 |
| L-Tirozin | 0.3 |
| L-Fenilalanin metilik ester | 0.5 |
| L-Glutamik asit (monosodyum tuzu) | 1.0 |
| L-Asparagin | 1.0 |
| L-Valin | 1.5 |
| L-treonin | 2.0 |
| L-Serin | 3.75 |
| Urasil | 0.2 |
| L-histidin HCI | 0.2 |
| L-Arginin HCI | 0.2 |
| L-metionin * | 0.2 |
| L-Lizin * | 0.2 |
| L-Lösin | 0.6 |
| * (Bu protokol gibi) standart PCA performans ancak diğer amaçlar için eklenebilir Çekilmiş. | |
Tablo 1:. MTX ortamında 10X lys / tanıştı / ade açılan-out Kompozisyon Miktarları 10x damla-out bir litre içindir. Yıldız standart MTX orta geri çekilmelidir bileşikleri belirtmektedir, ama başka amaçlar için ilave edilebilir.
Ek Tablo 1:. 12 Test plakalarından Kombine verileri Tablo 1 sıralı dekar içeriyorImageJ analiz ta her satır, her 1536 dizisinde tek bir konumuna karşılık gelen bir özel ImageJ komut kullanılarak gerçekleştirilmiştir. Ayrıca, her satır, ORF ve protein adını çoğaltmak, görüntü dosyası, DHFr toplama plakası hakkında bilgi açıklamalı olmuştur.
Ek Tablo 2:. Suş başına ortalama normalize büyüme Tablo 2 standart sapma ile birlikte koleksiyonun her bir suş için ortalama Giriş 2 normalize büyüme içerir. Filtreli avlayan, hit ve bilinen fiziksel interaktörler ayrı sütunlarda tanımlanır.
Tartışmalar
Biz yüksek verimlilik de herhangi bir yem proteini fiziksel interactors sistematik kimlik sağlayan DHFr-PCA tahlil dayalı bir protokol açıklar. Bu protokol, daha fazla yemler için tarama tarafından uyarlanan ve bu çoğalma istenilen düzeyde olabilir. Biz Nükleer Gözenek Kompleksi'nde yer alan bir yem proteini için fiziksel etkileşim ortakları belirlenmesi ile bu protokolün güvenilirliğini göstermektedir: Nup82. Yaptığımız analizler maya proteini interactome çalışma yöntemi yeteneğini vurgulayarak, beş önceden bildirilen inter ve bir daha önceden bildirilmemiş interaktör (Şekiller 3F ve 3G) bulmak için etkin.
Burada açıklanan protokol deneyci dikkat etmelidir hangi birkaç kritik adımlar içermektedir. Biz yem DHFr F [1,2] füzyon (Şekil 1B) doğru olduğundan emin olun) 1 tavsiye; Bu yapı ve kuv dizilenmesi ile elde edilebilirbir anti-DHFR F [1,2] ya da anti-DHFR F [3] bir antikor kullanarak, uygun protein ekspresyonu ing; 2) ekranını başlamadan önce, bu ilgi herhangi bir yem PCA ekranlarında karışık etkileşimleri sergiler doğrulamak için tavsiye edilir. Bu, uygun, L-DHFR kumanda veya manuel olarak uygun L-DHFR kontrol yem çiftleşme ve MTX ortamında büyüme tahlili yapılarak çapraz yemler ile kontrol ekranlarında gerçekleştirerek yapılabilir. Onlar nem baskı işlemi sırasında agar yüzeyinde hücre yapışması için en uygun olacak şekilde kullanılmadan önce 3) Tabaklar gün dökülür olmalıdır; 4) Kaynak plakaları hedef plaka üzerinde yeterli hücre aktarmak için en az dört kez kullanılmamalıdır. Hedef plaka kopya sayısını arttırmak genişleme ardışık adımlarla (-> 16 kopya -> 64 kopya örneğin 4 kopya) tarafından yapılabilir. Alternatif olarak, hücreler çimler üzerinde veya çoğaltma farklı mermi arasında koloni farklı pozisyonlarda alınabilir; 5) Eğer birkaç positions diploid seçimi (ler), kaynak plakaları çiftleşme aşamasında pek çok kez kullanılmış değil emin olun sonra eksik (4,5-4,7 adımları); 6) MTX orta sağ konsantrasyonlarda gerekli tüm malzemeyi içerdiğinden emin olun. Hiç büyüme MTX ortamında gözlenmesi durumunda Nitekim, bu olabilir ya hiç etkileşim ilgi proteinlerin veya MTX ortamı düzgün hazır değildi çünkü PCA tarafından algılanamaz çünkü. Ortam, DHFR tamamlanmasını fragmanları gösteren türlerinin büyümesini sağlar emin olmak için, bir kurucu etkileşim toplama boş pozisyonlarında ilave edildi ve bu lösin zipper kısımların 33 (Şekil 1C) kaynaşmış, DHFR-fragmanları gibi, bir pozitif kontrol olarak kullanılabilir. Bağlayıcı-DHFr fragmanları veya fermuar-bağlayıcı-DHFr parçaları kullanarak Paralel testler descr'in gibi, yanlış pozitif etkileşim yapmak eğilimindedir tüm hücrelerin büyümesine izin koşulları (düşük MTX konsantrasyonu veya yem protein arasında ayrım sağlayacakAşağıdaki IBED) ve tüm türlerinin büyümesini (MTX konsantrasyonu çok yüksek veya gerekli madde ortamda eksik) önlemek koşullar; Örneğin, pin-alet çoğaltma mermi ve / veya son su arasındaki düzgün sterilize değil, eğer 7) PCA başka bir ortamdan tekerrür ardışık tur üzerinden gerçekleştirildiğini göz önüne alındığında, farklı plakalar arasındaki suşları arasında çapraz-bulaşma oluşabilir sterilizasyon işlemi banyo (yani ıslak istasyonu) Önceki çoğaltma mermi koloniler tarafından kirletilmektedir. Diziler çeşitli pozisyonları boş olduğu ve bu nedenle herhangi bir büyüme çapraz kirletmesini tespit etmek için dikkat edilmelidir kontrol pozisyonları olarak kullanılabilir.
Görüntü analizi birkaç yayınlanan yazılımları (protokol bölümüne 5) veya herhangi bir özel komut dosyası kullanılarak yapılabilir. 1) komut dosyası co için her plaka değerlerini piksel için boş bir plaka piksel değerlerini çıkarır: Bu çalışmada, özel komut dosyası aşağıdaki adımları yürütüraydınlatma önyargıları için rrect. 2) komut dosyası kenar koloniler üzerinde bir dikdörtgen kaplayan tarafından belirlenen her plakanın her 1.536 pozisyonları için) 10. 3 bir piksel değeri eşiğini kullanarak ikili her arka plan-düzeltilmiş resmi dönüştürür, komut ImageJ "parçacıklar Analiz çalışır ... dairesel bir seçimde "fonksiyonu. dairesel seçim iki pozisyon eksi 10 piksel arasında aralığına eşit bir yarıçap ile ayarlanır. 4) komut seçim merkezine en yakın parçacık seçer ve konumu, iki koloniler arasında değil yarısından fazlasını aralığı uzakta seçim merkezine ise bir koloni olarak onaylar. 5) komut arka-düzeltilmiş resimde seçilen parçacığın piksel değerlerini ölçer. 6) ayrıca kalan arka plan aydınlatma önyargılarını düzeltmek için, komut koloninin piksel değerlerine bir parçacığın parçası değildi dairesel seçimden tüm piksellerin ortalama değeri çıkarır. Bu düzeltilmiş piksel değerinin toplamıEk, Tablo 1 sütun "IntDenBackSub" depolanan s, koloni büyüklüğü bir ölçüsü olarak kullanılır.
Analiz parçası içinde kritik bir adım önemi eşiği seçimdir. Burada, negatif L-DHFr F [3] kontrolleri dağılımına dayalı bir eşiği seçtim, ancak ekranın hedefi bağlı, böyle eşiği çok sıkı olabilir. Tamamlayıcı parçaları kendiliğinden birbirini tamamlayıcı olabilir ve bu nedenle çoğu proteinlerin ifade temsilcisi olmadığını Nitekim, L-DHFr F [3] kontrolleri aşırı salgılandığı (güçlü TEF promotör) gibi. Bu L-DHFr F dağılımı [3] kontrolleri arka plan büyüme (Şekil 3D) ortalamasından yüksek olduğu gerçeği ile vurgulanır. Böylece, bazı bu sıkı eşiğin altında puan olan etkileşimleri ama bu arka plan büyüme dağılımı dışında açıkça örneği, geçici ya da zayıf Inter temsil edebilir varsayılan hit olarak kabul edilebilireylemler. Bunlar daha ele alınabilir ve örneğin, iki protein L-DHFr denetimleri gibi DHFr parçalarının kendiliğinden tamamlanmasını izin verebilir seviyelerde ifade edilmez, eğer çapraz valide. Alternatif olarak, bir yanlış pozitif üzerinde gerçek pozitif oranını maksimize etmek için BioGRID 35 gibi veritabanlarında bildirilen fiziksel interactors ile örtüşme oranı dayalı bir önemi eşiğini ayarlayabilirsiniz. Örneğin, bilinen bir fiziksel uygulayıcı sayısı yeterince yüksek değilse Ancak L-DHFR dağılımının kullanımı farklı olarak, bu alternatif her zaman mümkün olmayabilir. Ayrıca, önemi eşik seçimi son veri setindeki yanlış pozitif ve yanlış negatif oranı üzerinde bir etkisi vardır. Daha önce de belirtildiği gibi, örneğin, proteinin yüksek oranda bol, Gerçekten de, diğer herhangi bir PPI tarama deneyinde olduğu gibi, yanlış pozitif DHFR füzyon proteini ile bir proteinin spesifik etkileşimi sonucu olabilir. BuBazı pençesine sistematik böylece PCA ekranlar tüm yem proteinleri ile etkileşime girecek ve bu gerçeği ile tarif edilmektedir, analizi 11 (örneğin, Tef2 ve Ade17 ve Katkı Tablo 2) çıkarılabilir olması gerekir. Bu sorunu, uygun L-DHFr kontrolüne karşı iki koleksiyon bir kontrol PCA ekranını aşmak için (F [1,2] veya F [3]) spontan DHFr tamamlama belirli koşullarda yapılabilir fragmanları sergileyen yemler ve kurbanlarına tanımlamak için Her ekranın. Belirli bir yem işlevi bilinen Ayrıca, eğer bir Gen Ontoloji zenginleştirme analizi yapmak veri güven artırabilir. Öte yandan, DHFR PCA birçok nedenden dolayı yanlış negatif ortaya çıkmasına neden olabilir: 1) Bütün proteinler, bu proteinlerin kararsız hale ya da, eğer, örneğin, kendi lokalizasyonu değiştirebilir olarak, DHFR fragmanlarına füzyonlu olabilir, hiç DHFR füzyon C-terminali bir lokalizasyon sinyali ile girişim; Bazı hücresel bölmeleri m 2) DHFr sulandırıldıktanÖrneğin, folat sentezi için gerekli bir öncül mevcut değildir, eğer ay folat üretmek değil; DHFr tamamlama 11 gerçekleşmesi için 3) C-termini ihtiyaç 8 nm mesafede olması. C-terminalleri alanda yeterince yakın değildir Böylece, iyi bilinen bir etkileşim tespit edilemez. Bu dolaylı olarak çoğu veri tabanları, bildirilen Nup82 fiziksel etkileşim büyük bir bölümü, tahlilimizde saptanmamış gerçeği ile örneklenmektedir. DHFr tamamlanmasını fragmanları ve 11 tespit olmayacaktır benzer, C-Termini zara trans akrabası olan hangi zar proteinleri arasındaki etkileşimler neden olmayacak. Sınırlamalar 1) ve 3) proteinin N-termini, DHFR fragmanı füzyon tarafından nispeten kolaylıkla yapılabilir. Yani C-terminalleri yakınında bir lokalizasyon sinyali ile müdahale engelleyebilir ve zar proteinleri, N ve C-ucu arasındaki bir etkileşimi saptamak için izin verebilir sis nisbetle olan Amaçlızara.
Çeşitli zorluklar (2,3 gözden) PIN'lerin çalışmada kalır. Şimdiye kadar üretilen PIN'lerden haritalar büyük ölçüde her tür için tek bir deneysel durumda tarif edilmiş ve dolayısıyla protein ağları organize olabilir nasıl tek bir anlık teklif var. PIN geliştirme veya aşağıdaki mutasyonlar arasında çevresel değişiklikler, belirli uyaranlara yanıt olarak yeniden olabilir nasıl görmek için diğer deneysel koşulların araştırılması için bir ihtiyaç vardır. Bu zorluklar canlı hücreler içinde, gerçek zamanlı PPIs sorguya için yeni teknolojilerin geliştirilmesi ile ve laboratuarların daha büyük bir topluluk tarafından kullanılabilir, böylece mevcut teknikleri uyarlayarak üstesinden edilecektir. DHFr tamamlama miktarındaki değişiklikleri algılayabilen bir nicel tekniği 27 kompleksleri gibi, DHFr-PCA bu zorlukların üstesinden gelmek için adapte edilebilir ve PPI'lar ajan 22 zarar bir DNA tarafından nasıl etkilendiğini incelemek için kullanılır olmuştur , Kimyasal maddeler, 25, gen silme 23,26 ya da maya türlerinde ve bunların melezleri 33. Bu yeni boyutlar keşfetmek PIN dinamiğini ortaya çıkarmak için daha önemli hale gelecektir.
Açıklamalar
Bu yazı için açık erişim yayın ücretleri Bölüm S & P Robotics tarafından ödendi.
Teşekkürler
Bu çalışma Kanada Sağlık Araştırma Enstitüsü (CIHR) 191.597, 299.432 ve 324.265, Grants tarafından desteklenen bir Doğa Bilimleri ve Kanada Keşif hibe Mühendislik Araştırma Konseyi ve CRL İnsan Frontier Bilim Programı hibe. CRL CIHR Yeni Araştırmacı olduğunu. Guillaume Diss bir PROTEO bursu ile desteklenmektedir. Samuel Rochette NSERC ve FRQNT arkadaş tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| BioMatrix Robot, Bench-top Configuration | S&P Robotics Inc. | BM5-BC | |
| 96-format Pin-tool | S&P Robotics Inc. | PH-96-10 | Standard 96-format Pin-tool with 96 high-precision floating pins |
| 384-format Pin-tool | S&P Robotics Inc. | PH-384-10 | Standard 384-format Pin-tool with 384 high-precision floating pins |
| 1536-format Pin-tool | S&P Robotics Inc. | PH-1536-05 | Custom 1536-format Pin-tool with 0.5mm high-precision floating pins |
| Automated imaging module | S&P Robotics Inc. | IMG-02 | |
| Methotrexate | Bioshop Canada Inc. | MTX440 | CAUTION: toxic compound |
| Hygromycin B | Bioshop Canada Inc. | HYG003 | |
| Nourseothricin dihydrogen sulfate | Werner BioAgents | 5010000 | |
| Yeast-Interactome Collection | Thermo Scientific | YSC5849 | |
| Omni Tray w/lid sterile | Thermo Scientific | 242811 | |
| Anti-DHFR F[1,2] antibody | Sigma-Aldrich | D1067 | |
| Anti-DHFR F[3] antibody | Sigma-Aldrich | D0942 |
Referanslar
- Alberts, B. The cell as a collection of protein machines: preparing the next generation of molecular biologists. Cell. 92, 291-294 (1998).
- Diss, G., et al. Integrative avenues for exploring the dynamics and evolution of protein interaction networks. Curr Opin Biotechnol. 24, 775-783 (2013).
- Vidal, M., Cusick, M. E., Barabasi, A. L. Interactome networks and human disease. Cell. 144, 986-998 (2011).
- Hu, P., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS biology. 7, e96 (2009).
- Arifuzzaman, M., et al. Large-scale identification of protein-protein interaction of Escherichia coli K-12. Genome Res. 16, 686-691 (2006).
- Rajagopala, S. V., et al. The binary protein-protein interaction landscape of Escherichia coli. Nature biotechnology. 32, 285-290 (2014).
- Arabidopsis-Interactome-Mapping-Consortium. Evidence for network evolution in an Arabidopsis interactome map. Science. 333, 601-607 (2011).
- Babu, M., et al. Interaction landscape of membrane-protein complexes in Saccharomyces cerevisiae. Nature. 489, 585-589 (2012).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Gavin, A. C., et al. Functional organization of the yeast proteome by systematic analysis of protein complexes. Nature. 415, 141-147 (2002).
- Tarassov, K., et al. An in vivo map of the yeast protein interactome. Science. 320, 1465-1470 (2008).
- Uetz, P., et al. A comprehensive analysis of protein-protein interactions in Saccharomyces cerevisiae. Nature. 403, 623-627 (2000).
- Guruharsha, K. G., et al. A protein complex network of Drosophila melanogaster. Cell. 147, 690-703 (2011).
- Li, S., et al. A map of the interactome network of the metazoan C. elegans. Science. 303, 540-543 (2004).
- Rual, J. F., et al. Towards a proteome-scale map of the human protein-protein interaction network. Nature. 437, 1173-1178 (2005).
- Landry, C. R., Levy, E. D., Abd Rabbo, D., Tarassov, K., Michnick, S. W. Extracting insight from noisy cellular networks. Cell. 155, 983-989 (2013).
- Berggard, T., Linse, S., James, P. Methods for the detection and analysis of protein-protein interactions. Proteomics. 7, 2833-2842 (2007).
- Wodak, S. J., Vlasblom, J., Turinsky, A. L., Pu, S. Protein-protein interaction networks: the puzzling riches. Current opinion in structural biology. 23, 941-953 (2013).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Dunham, W. H., Mullin, M., Gingras, A. C. Affinity-purification coupled to mass spectrometry: basic principles and strategies. Proteomics. 12, 1576-1590 (2012).
- Michnick, S. W., Ear, P. H., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods Enzymol. 470, 335-368 (2010).
- Rochette, S., Gagnon-Arsenault, I., Diss, G., Landry, C. R. Modulation of the yeast protein interactome in response to DNA damage. Journal of proteomics. 100, 25-36 (2014).
- Diss, G., Dube, A. K., Boutin, J., Gagnon-Arsenault, I., Landry, C. R. A systematic approach for the genetic dissection of protein complexes in living cells. Cell Rep. 3, 2155-2167 (2013).
- Gagnon-Arsenault, I., et al. Transcriptional divergence plays a role in the rewiring of protein interaction networks after gene duplication. Journal of proteomics. 81, 112-125 (2013).
- Schlecht, U., Miranda, M., Suresh, S., Davis, R. W., St Onge, R. P. Multiplex assay for condition-dependent changes in protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 109, 9213-9218 (2012).
- Lev, I., et al. Reverse PCA, a systematic approach for identifying genes important for the physical interaction between protein pairs. PLoS Genet. 9, e1003838 (2013).
- Freschi, L., Torres-Quiroz, F., Dube, A. K., Landry, C. R. qPCA: a scalable assay to measure the perturbation of protein-protein interactions in living cells. Mol Biosyst. 9, 36-43 (2013).
- Pelletier, J. N., Campbell-Valois, F. X., Michnick, S. W. Oligomerization domain-directed reassembly of active dihydrofolate reductase from rationally designed fragments. Proceedings of the National Academy of Sciences of the United States of America. 95, 12141-12146 (1998).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods Enzymol. 350, 87-96 (2002).
- Schuldiner, M., Collins, S. R., Weissman, J. S., Krogan, N. J. Quantitative genetic analysis in Saccharomyces cerevisiae using epistatic miniarray profiles (E-MAPs) and its application to chromatin functions. Methods. 40, 344-352 (2006).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature methods. 9, 671-675 (2012).
- Wagih, O., Parts, L. gitter: A Robust and Accurate Method for Quantification of Colony Sizes From Plate Images. G3 (Bethesda). 4 (3), 547-552 (2014).
- Leducq, J. B., et al. Evidence for the robustness of protein complexes to inter-species hybridization. PLoS Genet. 8, e1003161 (2012).
- . . Development Core Team: A language and environment for statistical computing. , (2008).
- Stark, C., et al. BioGRID: a general repository for interaction datasets. Nucleic Acids Res. 34, D535-D539 (2006).
- Vinayagam, A., et al. Protein complex-based analysis framework for high-throughput data sets. Science signaling. 6, rs5 (2013).
- Jansen, R., Gerstein, M. Analyzing protein function on a genomic scale: the importance of gold-standard positives and negatives for network prediction. Current opinion in microbiology. 7, 535-545 (2004).
- Ito, T., et al. A comprehensive two-hybrid analysis to explore the yeast protein interactome. Proceedings of the National Academy of Sciences of the United States of America. 98, 4569-4574 (2001).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-431 (2010).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır