Method Article
RNA'nın 1 H R1φ Gevşeme Dispersiyon Deneylerinin Numune Hazırlama ve Kurulumunun Pratik Yönleri
Bu Makalede
Özet
13 C/ 15 Netiketli ve etiketsiz RNA'da mikro-milisaniye dinamikleri1H R1φ gevşeme dağılımı nükleer manyetik rezonans (NMR) spektroskopisi ile ölçmek için bir protokol sunuyoruz. Bu protokolün odak noktası, NMR deneylerinin yüksek saflıkta numune hazırlanması ve kurulumunda yatmaktadır.
Özet
RNA, yapılardaki değişikliklerin RNA moleküllerinin hücresel haberciler ve modülatörler olarak yürüttüğü işlevlerde önemli roller oynadığı son derece esnek bir biyomoleküldür. Bu dinamik durumlar çoğu yapısal yönteme gizli kalırken, R1φ gevşeme dağılımı (RD) spektroskopisi, mikro-milisaniye rejimindeki konformasyonsal dinamiklerin atomik çözünürlükte incelenmesini sağlar. Gözlemlenen çekirdek olarak 1H'nin kullanılması, kapsanan zaman rejimini daha da genişletir ve hidrojen bağlarına ve baz eşleştirmesine doğrudan erişim sağlar.
Böyle bir çalışmadaki zorlu adımlar, potansiyel olarak 13 C ve 15 Netiketli yüksek saflık ve yüksek verimli numune hazırlamanın yanı sıra, daha önce görünmeyen devletin popülasyonunu, döviz kurunu ve ikincil yapısını çıkarmak için deneylerin kurulumu ve verilerin takılmasıdır. Bu protokol, uygun bir RNA örneğinin hazırlanmasını ve hem izotopik olarak etiketlenmiş hem de etiketlenmemiş RNA örnekleriyle 1H R1φ deneyinin kurulumunu sağlamak için numune hazırlamada çok önemli uygulamalı adımlar sağlar.
Giriş
RNA'lar hücrede çok sayıda düzenleyici1,katalitik2ve yapısal 3 işlevi gerçekleştirir,bunların çoğu esnek bir moleküler yapı ve bu yapıların karmaşık değişiklikleri ile ilişkilidir4,5,6,7. Düşük nüfuslu durumlar, yapı belirleme yöntemlerinin çoğunda görünmez kalır veya bu gizli durumların yüksek atomik çözünürlükte incelenmesine izin vermez. Çözüm durumu nükleer manyetik rezonans (NMR) spektroskopisi, bireysel atom çekirdeklerine erişim sağlayarak ve tüm zaman rejimleri boyunca dinamikleri hedefleyen büyük bir deney araç kutusu sunarak her iki yönü birleştirir8. RD NMR denemeleri, temel eşleştirme desenlerinde ve yerel yapısal yeniden düzenlemelerde değişikliklerin beklenebilir olduğu ara zaman ölçeğinde konformasyonsal değişime erişim sağlar5,9,10,11,12,13,14. RD deneyleri, Carr-Purcell-Meiboom-Gill darbe treni 15 şeklinde uzun R2 ölçümleri veya R1φ RD deneyleri16 olarak adlandırılan dönen çerçevede gevşeme ölçümleri olarakgerçekleştirilir.
Her ikisi de nüfus ve döviz kuru ve kimyasal kayma farkını küçük duruma çıkarmak için kullanılabilse de, R1φ RD deneyleri aynı zamanda heyecanlı devletin kimyasal kayma farkının işaretini verir. Bu, RNA yapılarındaki kimyasal kayma ile güçlü bir şekilde ilişkili olan ikincil yapı üzerinde bir çıkarım sağlar17. Kimyasal kayma, nükleobazlardaki aromatik protonlar ve karbonlar, imino protonlar için baz eşleştirme ortakları ve C4' ve C1' atomları üzerindeki şeker büzücüleri durumunda helicity'nin iyi bir göstergesidir18,19. Son zamanlarda daha yüksek spin kilidi (SL) gücü kullanan bir kimyasal değişim doygunluk transferi (CEST) deneyinin, böylece CEST deneyinin uygulanabilirliğini daha hızlı değişim zaman ölçeklerine kaydırdığı, heyecanlı bir duruma sahip sistemler için R1φ RD deneyine alternatif olarak yayınlandığı belirtilmelidir.
Yapısal değişime erişmek için sıklıkla 13C ve 15N izotopları kullanılmış olsa da, bu laboratuvardan yapılan son çalışmalarda aromatik ve imino protonlar konformasyonsal değişim için prob olarakkullanılmıştır 9,10. Gözlemlenen çekirdek olarak 1H'nin kullanılması, örneğin daha hızlı ve daha yavaş zaman ölçeklerinde değişime erişim, daha yüksek hassasiyet ve daha kısa ölçüm süreleri gibi çeşitli avantajlar getirir. Bu, heteronükleer manyetizasyon transferi yerine homonükleer skaler kavramalar kullanarak tek boyutlu (1D) spektrumun kafeinsizleştirilmesi yoluyla aromatik protonlara erişim sağlayan ve izotop etiketlerine olan ihtiyacı ortadan kaldırarak SELective Optimize Edilmiş Proton Deneyi (SELOPE) yaklaşımı ile daha da kolaylaştırılmıştır20. Bu protokol, eşit 13C/15 N etiketli ve etiketsiz numunelerin 1H R1φRD deneylerindeki ölçümü ele alıyor. Bu nedenle, bu makale farklı numune hazırlama ihtiyaçları için en çok yönlü olduğu tespit edilen bir örnek hazırlama yöntemi sunun21 ve bu makalenin son bölümünde alternatifleri ele almaktadır (Şekil 1).
Bu noktada okuyucu, 1 H R 1φRD deneyleri için diğer numune hazırlama tekniklerinin kabul edilebilir olduğunu, sunulan teknikle sentezlenen örneklerle yapısal ve fonksiyonel analizin diğer yöntemlerinin de yapılabileceğini belirtmelidir. 1 H R1φ RD deneyleri, moleküler dinamiklerin güvenilir bir şekilde karakterizeini sağlamak için hem RNA uzunluğunda hem de yapısal konformasyonda yüksek RNA konsantrasyonları (ideal olarak >1 mM) ve yüksek homojenlik gerektirir. In vitro transkripsiyon (IVT), etiketli nükleozid trifosfatların (NTP' ler) ve facile indüksiyonunun enzymatic reaksiyon22'debulunması nedeniyle birçok araştırmacının 13C/15N etiketli RNA örnekleri üretmesi için tercih edilen yöntemdir. Bununla birlikte, yaygın olarak kullanılan T7 RNA polimeraz (T7RNAP)23,24,25, belirli başlatma dizileri durumunda düşük 5' homojenliktenmuzdariptir 26 , 27 ve genellikle transkripsiyon ikinci tur28sırasında 3' homojenlik . Hedef RNA türlerinin saflaştırılması, büyük miktarlarda ~ 200 nmol ihtiyacı nedeniyle daha pahalı ve zahmetli hale gelir. Burada kullanılan yöntem daha önce avantajların tartışıldığı21. Kısacası, açıklanan sorunları, daha sonra Escherichia coli RNase H tarafından özellikle bölünen, kimerik oligonükleotid29,30 tarafından yönlendirilen daha büyük bir tandem transkript yazarak çözer (ayrıntılar için Bkz. Şekil 2).
Tandem transkriptin 5' ve 3' uçlarında bir aralayıcı dizisinin dahil edilmesi, yüksek verimli bir başlatma dizisinin kullanılmasına ve plazmid şablonunun doğrusallaştırma bölgesine yakın terminal çıkıntilarının kaldırılmasına izin verir (Şekil 2B). Yöntemin, daha karmaşık bir şablon sentezi ve ek bir enzim ve oligonükleotid ihtiyacı ile maliyet ve işçiliği azaltırken, verimi önemli ölçüde artırdığı gösterilmiştir. RNase H bölünmesinin yüksek özgüllüğü, benzer boyut aralığında RNA türlerinin eksikliği nedeniyle arınma sağlar. Mevcut protokol, bu laboratuvar tarafından yakın zamanda yayınlanan bir iyon değişimi yüksek performanslı sıvı kromatografisi (HPLC) adımı kullanır31Diğer yöntemler olası alternatifler olmasına rağmen. 1 H R1φ RD, genel olarak, iki ilgili darbe dizisine sahip etiketli veya etiketsiz örneklerde elde edilebilir, "etiketli" 1H R1φ heteronuclear tek kuantum korelasyonu (HSQC) tabanlı deney ile 13C dolaylı boyut10 ve "etiketsiz" 1H R1φ SELOPE tabanlı deney ile 1H indirekt boyut20.
Bu iki boyutlu (2D) deneyler, R1φ zaman ölçeğindeki dinamiklerin örnekte bulunup bulunmadığına bakılmaksızın ilk denetim görevi görebilirsiniz. Spektrumdaki tüm çözümlenmiş tepeler için RD'ye genel bir bakış elde edilebilir ve daha kapsamlı bir RD analizi için ilgi çekici zirveler belirlenebilir. Bu, etiketlenmemiş numunelerin bile daha pahalı, etiketli bir örnek üretme kararı almadan önce kontrol edilebileceği anlamına gelir. Konformasyonsal değişim katkısına sahip bir zirve daha ayrıntılı bir şekilde çalışılmak üzere seçildikten sonra, yukarıdaki deneylerin 1D sürümlerine geçmek en iyisidir (zirve hala çözülebilirse) sözde rezonans dışı deneyler yapmak için. Etiketli versiyon için, 13C'ye HSQC aktarımı, SELOPE deneyi durumunda,13 C R 1φdeneylerinde 32 , 33 ,34 ,35 'te kullanıldığı gibi seçici bir heteronükleer çapraz polarizasyon(HCP)adımı ile değiştirilir, deney sadece bir 1D olarak çalıştırılır, bu da özellikle 2D'de diyagonal üzerinde yatan H8 ve H2 sinyalleri için yararlıdır. Her ikisinin de etiketli ve etiketsiz bir örneğin mevcut olması koşuluyla, hangi sıranın kullanılacağına ilişkin bir kriter, iki deneyde ilgi zirvesinin ne kadar iyi yalıtılmış olduğudur.
Genel olarak, SELOPE deneyi 50 nükleotide kadar RNA örnekleri için önerilir. Daha büyük RNA'lar için çakışma daha büyük olacaktır; bununla birlikte, yapısal olarak ilginç nükleotitler genellikle daha az üst üste binen ve daha büyük RNA'larda hala erişilebilir olabilen kimyasal kayma bölgelerinde görülür. Başka bir argüman, etiketlenmemiş örneklerde, 1H ile 12C arasında J bağlantısı oluşmadığıdır. Bununla birlikte, minimum döndürme kilidi gücü, etiketli deneyde bu iki dönüşü (~1 kHz) ayırmak için kullanılan minimum güçle tanımlandığı için, etiketlenmemiş deneme, daha geniş bir spin kilidi (SL) güçlü yönlerinin kullanılmasına ve bu nedenle daha geniş bir değişim zaman ölçeğine erişime izin verir. Bu rezonans dışı deneyler, heyecanlı devletin popülasyonu (alternatif konformör), p ES gibi kex'eek bilgiler veΔω (zemin durumunun kimyasal kayma farkı ve heyecanlı durum) şeklinde çok değerli kimyasal kayma bilgileri sağlar.
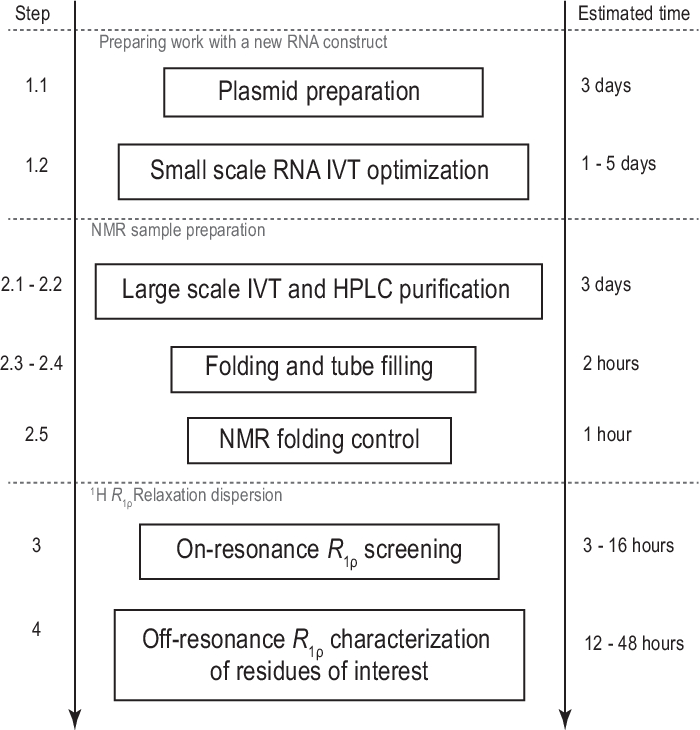
Şekil 1: Sunulan protokolün iş akışı. Şablon hazırlama ve başarılı in vitro transkripsiyon ve RNase H dekolte teyidsinden oluşan gerçek büyük ölçekli numune üretiminden önce hazırlık. HPLC saflaştırma, NMR tüpünün doldurulması ve RNA katlama onayı dahil olmak üzere büyük ölçekli üretim. Izotop etiketli sentez durumunda, aynı gün gradyan optimizasyonu için etiketsiz bir saflaştırma yapılmalıdır. R1φ deneyleri ile konformasyonsal dinamiklerin NMR karakterizasyonu. Her adım bağımsız olarak gerçekleştirilebilir, örneğin 1H R1φ RD analizi başka bir yöntemle üretilen herhangi bir uygun RNA örneğine uygulanabilir. Kısaltmalar: IVT = in vitro transkripsiyon; HPLC = yüksek performanslı sıvı kromatografisi; NMR = nükleer manyetik rezonans; RD = gevşeme dağılımı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Bu protokolün amacı, RNA saç tokası moleküllerinde 1 H R 1φ gevşeme dağılımı ile konformasyonsal dinamiklerin incelenmesi için pratik ayrıntılar ve kritik parametreler sağlamaktır. 13C/15N etiketli sürümler olarak tüm, bazıları veya hiçbiri NTP'leri kullanılarak gerçekleştirilebilen bir hedef RNA'nın tasarımı, sentezi ve iyon değişimi HPLC saflaştırmasının ayrıntılı bir protokolünü sağladıktan sonra, NMR örneğini sonlandırma ve NMR spektroskopisi ile konformasyonel değişimi onaylama iş akışı açıklanmıştır. Son olarak, Bruker NMR spektrometresi üzerinde 1H R1φ RD deneylerinin kurulumu için ayrıntılar açıklanmıştır (Şekil 1). Protokol, etiketli örnekler ve ek yorumlar için 1D sürümü ayarlamak için her adımı ve SELOPE sürümünün(Tablo 2)kurulumu için ayarlamak üzere bir tablo verir. Protokolden sonra, kritik adımlar ve örnek hazırlama ve 1 H R 1φRD kurulumu için alternatif yollar tartışılır.
Protokol
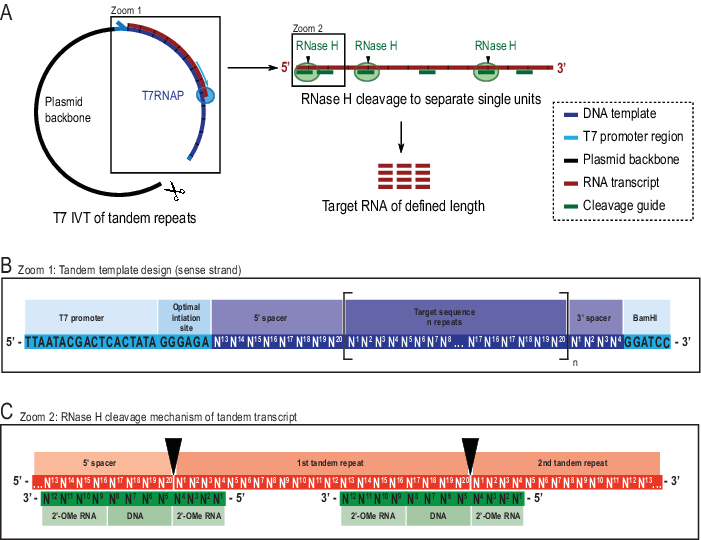
Şekil 2: Bildirilen tandem IVT protokolünün şematik gösterimi. (A) T7RNAP (solda) ile doğrusallaştırılmış bir plazmid şablonundan tandem transkripsiyon ve bir kimerik DNA kılavuzu (sağda) tarafından yönlendirilen hedef uzunluk RNA'sına ulaşmak için transkriptin RNA'sı tarafından ardışık bölünme. (B) Viral T7RNAP promotöründen başlayarak tandem şablonunun ayrıntılı şeması, bir başlatma dizisi. Hedef sıra (koyu mavi, örnek burada 20 nt uzunluğunda) "n" kez yinelenir. Tekrarlar, ilk ve son tekrar ünitesinden başlatma ve kısıtlama sıralarının kaldırılmasına izin vermek için sırasıyla son sekiz ve ilk dört nükleotitten oluşan 5′ ve 3′ ara dizileri tarafından kuşatılmıştır. (C) Tandem transkript (kırmızı) ve kimerik bölünme kılavuzlarının (yeşil) hibridizasyonu. RNase H, DNA 5′ ucunun karşısındaki RNA'yı ayırır. 2′-OMe RNA kanatları, göğüs dekolte kılavuzunun hedef RNA'ya bağlayıcı benzeşimini artırarak özgüllüğü arttırır. Bu rakam 21'den değiştirilmiştir. Kısaltmalar: T7RNAP = T7 RNA polimeraz. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
1. Yeni bir RNA yapısı için çalışma hazırlama
- Plazmid tasarımı ve hazırlanması
- Şablon dizisini bir klonlama aracına yazın, örneğinSeri Klonlayıcı.
- T7 promotör sırasını alın ve yüksek verimli bir başlatma dizisi ekleyin (T7: 5'-TAATACGACTCACTATA ^GGGAGA-3').
NOT: Transkripsiyon, bir şapka (^) ile belirtilen nükleotide başlayacaktır. Başlatma sırası GGGAGA değişkendir, ancak kuvvetle sıraya bağlıdır; bu nedenle, bu sıranın kullanılması önerilir. - Hedef sıranın son 8 nükleotidini (nt) 5' aralayıcı (5'ler) olarak ekleyin.
- Hedef sıranın (TS) yinelemelerini ekleyin.
- Tekrarlardan sonra ilk dört nükleotitleri 3' aralayıcı olarak ekleyin (5'ler).
- BamHI kısıtlama sitesi (RS) veya benzer benzersiz kısıtlama sitesi ekleyin.
NOT: Gösterilen toplam sıra klonlanacak veya bakteriyel bir yüksek kopya plazmidinde(örneğin,pUC19): 5'-T7-5'S-(TS)n -3'S-RS-3'(Şekil 2B)olarak sıralanacaktır. Tekrar sayısı gen sentezinin izin verdiği kadar yüksek olmalıdır (bu protokolde en fazla 600 nt). - Ticari bir kit kullanarak E. coli'deki plazmidi güçlendirin.
- Saflaştırılmış plazmidi uygun kısıtlama bölgesini kullanarak 20 ng/μL'de doğrusallaştırın. Ölçek kısıtlaması BamHI ile 1 mL'ye kadar sindirilir.
- Sindirilen plazmidi arındırın ve% 1 agarose jel üzerinde başarılı doğrusallaşmayı onaylayın. Doğrusal plazmidi -20 °C'de birkaç ay saklayın.
- Dekolte kılavuzu tasarımı (Şekil 2C)
- Hedef RNA dizisinin son sekiz nükleotidini 5'-3' yönde yazın ve hedef RNA dizisinin ilk dört nükleotidini 3' uçta da 5'-3' yönde ekleyin.
- Bu dizinin ters DNA tamamlayıcısını oluştur
- Nükleotid harfinden önce bir 'm' ekleyerek ilk ve son dört nükleotitleri 2'-OMe modifikasyonlarına değiştirin.
NOT: Sentez için mT yerine mU kullanılır. - Standart tuzdan arındırma ile oligo sipariş edin.
NOT: Oluşturulan oligonun iki RNA dizisinin bağlantısı dışında başka bir yere bağlanıp bağlanamayacağını kontrol edin. Orta dört DNA nükleotidinde tam tamamlayıcılık gerekirken, yan bölgeler uyumsuzluklara izin verebilir. Gerekirse, benzersiz bir bağlama dizisi oluşturmak için kanatları 18 nt'ye kadar uzatın36.
- Küçük ölçekli IVT
NOT: RNase içermeyen çalışmalar için tüm reaktifleri steril ve RNase içermeyen koşullar altında hazırlayın. Çalışma yüzeylerini ve pipetleri kullanmadan önce temizlemek için RNase dekontaminasyon reaktifi (Bkz. Malzeme Tablosu)ve % 95 v/v etanol kullanın. Eldivenleri % 95 etanol ile yıkayın ve tüy bırakmayan uzun kollu kıyafetler giyin. RNase kontaminasyonünü en aza indirmek için açık tüpler üzerinde nefes almayın.- Tris-Cl (pH 8.0), dithiothreitol, MgCl2, spermidin ve NTP/GMP (unbuffered) stok çözeltilerini hazırlayın. Reaktifleri Tablo 1'degösterildiği gibi karıştırın. Enzimlerin veya nükleik asitlerin eklenmesinden önce, bu reaktiflerin ana karışımını önceden hazırlayın.
NOT: Donmuş reaktifler kullanıyorsanız, çözdükten sonra iyice karıştırın. Reaktifler çok yüksek konsantrasyonlarda karıştırılırsa çökelebilir, bu nedenle Tablo 1'dekisırayı izlemeniz şiddetle önerilir. - Aşağıdaki sırayla ekleyin: plazmid, dekolte kılavuzu, inorganik fosfataz (IPPase), RNase H, T7RNAP. Enzim aktivitesi şirket içinde üretilen enzimler için değişebileceğinden, en iyisini seçmeden önce birkaç konsantrasyonu test edin.
NOT: Eksik bir hedef bandı kusurlu RNase H dekoltesine bağlamak ve başarısız transkripsiyona değil, örneğin RNase H olmadan bölünme reaksiyonu için negatif bir kontrol ekleyin. - Reaksiyonu 37 °C'de 1 saat boyunca kuluçkaya yatırın ve denatüre poliakrilamid jel elektroforez (PAGE)(Şekil 3A)üzerindeki reaksiyonu onaylayın. Numuneyi yükleme çözeltisinde 10 kat seyreltin ve jel üzerine 1 μL yükleyin.
NOT: Jel karışımı: 1x TBE'de 8 M üre, %20 akrilamid (19:1 akrilamid:bisacrylamide). Yükleme çözeltisi: 5 mM etilenediamin tetraasetik asit (EDTA), formamidde 300 μM bromofenol mavisi. RNase H dekolte reaksiyonlarının 1 saat sonra tamamlanması beklenemez, çünkü sürekli yeni RNA üretilir. Bu noktada, net bir hedef bandına ve benzer moleküler ağırlıkta bir türün yokluğuna dikkat edin(örneğin,±3 nükleotid (nt) ürünleri).
- Tris-Cl (pH 8.0), dithiothreitol, MgCl2, spermidin ve NTP/GMP (unbuffered) stok çözeltilerini hazırlayın. Reaktifleri Tablo 1'degösterildiği gibi karıştırın. Enzimlerin veya nükleik asitlerin eklenmesinden önce, bu reaktiflerin ana karışımını önceden hazırlayın.
| Reaktif | Stok konsantrasyonu | Miktar küçük ölçekli (μL) |
| H2O | - | 24 |
| Tris | 1 M | 5 |
| MgCl2 | 1 M | 0.5 |
| DTT | 1 M | 0.5 |
| Spermidin | 250 mM | 5 |
| GMP | 100 mM | 2.5 |
| ATP | 100 mM | 1.5 |
| GTP | 100 mM | 1.5 |
| UTP | 100 mM | 1.5 |
| CTP | 100 mM | 1.5 |
| Plasmid | 20 ng/μL | 5 |
| Dekolte kılavuzu | 100 μM | 10 |
| iPPase | 10 mg/mL | 0.5 |
| RNase H | 10 μg/mL | 2 |
| T7 RNA polimeraz | 5 mg/mL | 2 |
Tablo 1: Tandem IVT ve eşzamanlı RNase H dekolte için reaktif tablo. Stok konsantrasyonlarıkullanıcınınrahatlığına uyarlanabilir. T7 IVT'den sonra RNase H dekoltesi yapılması gerekiyorsa, T7RNAP ısı inaktivasyonundan sonra dekolte kılavuzu ve RNase H ekleyin. Reaksiyon ölçeği ile doğrusal olarak ölçeklenmiş olarak kullanılan tutarlar. Kısaltmalar: T7RNAP = T7 RNA polimeraz; IVT = in vitro transkripsiyon.
2. NMR numune hazırlama
- Reaksiyonun istediğiniz birime (genellikle 10 mL) ölçeğini yükseltin ve reaksiyonun gece boyunca çalıştırın. Ertesi gün bir denatüre PAGE jeli ile reaksiyon tamamlama testi (Şekil 3A).
NOT: Eksik bölünme reaksiyonı, hedef bandın üzerindeki daha yüksek moleküler ağırlık türleri tarafından gösterilir.- Dekolte başarılı veya eksiksiz değilse, çözeltiyi 15 s için 450 W'ta geleneksel bir mikrodalgada ısıtarak reanneal RNA ve reaksiyon kabındaki bölünme kılavuzu.
- Çözeltiyi 40 dakika boyunca yavaşça 37 °C'ye soğutun. 1 mL'nin altındaki hacimler için bir ısıtma bloğu kullanın. Yeni çökeltici oluşumuna dikkat edin.
- Daha fazla IPPase ve RNase H ekleyin ve 37 °C'de 1-3 saat daha kuluçkaya yaslanın. Dekolte reaksiyonunun tamamlandığını denatüre PAGE ile onaylayın.
- RNase H bölünme reaksiyonu tamamlandığında, 50 mM son konsantrasyona ve girdaplara EDTA ekleyerek reaksiyonu iyice söndürün.
NOT: Potansiyel pirofosfat çökeltme çözünür ve yeni protein çökeltir. - Çözeltiyi 0,2 μm şırındıcı filtreden filtreleyin ve enjeksiyon döngüsü boyutuna bağlı olarak HPLC sistemine enjekte edilebilen bir hacme konsantre olun.
NOT: Protokol burada -20 °C'de numune dondurularak duraklatılabilir.
- Büyük ölçekli HPLC saflaştırma
- İyon değişimi arabelleği A ve B'yi kullanımdan sonraki bir hafta içinde hazırlayın. Tamponları filtreleyin ve gazdan arındırın.
NOT: Tampon A: 20 mM sodyum asetat; 20 mM sodyum perklorat, pH 6.5. Tampon B: 20 mM sodyum asetat; 600 mM sodyum perklorat, pH 6.5. - Sütunu %100 B tamponu ve ardından 75 °C'de en az 2 sütun birimi için %100 arabellek A ile dengele.
- HPLC dizisini (Şekil 3B) 5,5 mL / dak'lık bir akış hızında hazırlayın. 20 ila 30 nt arasında büyüklükte bir RNA'nın saflaştırılması için aşağıdaki sırayı kullanın: 0-7 dk: %0 B; 7-16 dk: gradyan %0-20 B; 16-46 dk: tipik olarak% 20-30 B gradyan ile elution (iztiyaca göre optimize edin); 46-62 dk: %100 B; 62-73 dk: %0 B.
NOT: Akış hızında 5,5 mL/dk'ya yapılan bir değişiklik bu protokoldeki ayrımı etkilemedi. - Bir seferde 1 mL transkripsiyon reaksiyonuna (etiketsiz) eşdeğer bir enjeksiyonla elution gradyanını optimize edin.
NOT: Daha fazla ayrıntı ve tartışma için Karlsson ve ark.31 ve Feyrer ve ark.21'e bakın. - Denatüre page üzerinde toplanan kesirleri test edin. Ana elution zirvesi iyi yalıtılmışsa ve saf hedef RNA'yı içeriyorsa, saflaştırmayı 10 mL transkripsiyon reaksiyonuna eşdeğer bir şekilde ölçeklendirin.
- İlgi kesirlerini toplayın, yoğunlaştırın ve arabelleği NMR arabelleğiyle değiştirin. 50 mL'nin üzerindeki hacimler için ultrasantrifüj filtre ünitesi kullanın (Malzeme Tablosunabakın).
NOT: NMR tamponu: 15 mM sodyum fosfat; 25 mM sodyum klorür; 0.1 mM EDTA, pH 6.5. Plastik boru duvarlarına bağlı kalan RNA'dan kaynaklanan kaybı en aza indirmek için, tüm sıvıları toplamak için tüm toplama tüplerini 1 mL su, girdap ve santrifüjle yıkayın. - Ultraviyole spektroskopisi ile konsantrasyonu belirleyin. Reaksiyon verimini Feyrer veark.
NOT: RD deneyleri için bir NMR örneğinin konsantrasyonu, NMR tüpleri(Malzeme Masası)kullanılarak 250 μL'lik bir numune hacminde 500 μM'ye karşılık gelen 130 nmol'ün altında olmamalıdır.
- İyon değişimi arabelleği A ve B'yi kullanımdan sonraki bir hafta içinde hazırlayın. Tamponları filtreleyin ve gazdan arındırın.
- RNA örneğinin katlanır
- ~10 mL'lik bir hacmin örneğini tüp başına 1 mL'ye seyreltin ve aliquot edin.
- RNA aliquots'ı 5 dakika boyunca 95 °C'ye ısıtın.
- Numuneleri buza veya su-buz-tuz karışımına yerleştirerek soğutun ve 30 dakika kuluçkaya yatırın.
- Havuz örnekleri ve konsantre 2 mL santrifüj filtre ünitesinde ~250 μL.
- NMR tüpünün doldurulması
- NMR tüp temizleyicislerindeki NMR tüpünü bol su, RNaz dekontaminasyon reaktifi, su, %95 etanol (EtOH) ve tekrar su ile temizleyerek temizleyin. Kurumaya bırakın.
- Pistonu suyla durulayarak ve RNase dekontaminasyon reaktifi ve% 95 EtOH ile tiftiksiz bir mendil kullanarak silerek temizleyin. Kurumaya bırakın.
- NMR örneğine%10(v/v) D 2 O ekleyin.
- RNA örneğini büyük bir pipet ucu kullanarak NMR tüpüne doldurun. Sıvının tüp duvarının yan tarafı boyunca akmasına izin verin.
- Pistonu takın ve pistonu hızlı bir büküm hareketiyle birlikte aşağı iterek hava kabarcıklarını çıkarın.
- Pistonu yeni hava kabarcıkları oluşturmadan yavaşça yukarı çekin ve parafin balmumu filmi ile sabitleyin.
- NMR tarafından katlamayı onaylayın.
NOT: Bu noktada, RNA örneğinin ikincil yapısını doğrulamak ve konformasyonsal dinamiklerin incelenmesi için ilgi çekici bölgeleri belirlemek için en azından kısmi rezonans ataması yapmak gerekir. RNA rezonans ataması ile ilgili kapsamlı bir açıklama bu protokolü aşalamıştır, bu nedenle bu noktada köklü literatüre atıfta bulunuyoruz19,37,38. Elektroforetik hareket kabiliyeti değişimi tahlilleri (EMSA), RNA katlamanın yararlı bir göstergesi olabilir ve NMR deneyleri için tamamlayıcı veri görevi görebilirsiniz.- 1 H R 1φRD deneylerinin yapıldığı numunenin aşağıdaki spektrumunu düzgün katlanmış referans örneğiyle karşılaştırın (Şekil 4): 1H 1D, özellikle imino bölgesi 10–15 ppm; Aromatik 1H,13C-HSQC; 1 H,1H-SELOPE (isteğe bağlı).
NOT: Aromatik bir parmak izi de gereklidir, imino sinyalleri arasında anlaşma durumunda bile, çünkü dimer oluşumu genellikle bir RNA saç tokası ile aynı veya benzer imino sinyallerini gösterir. SELOPE deneyi aromatik parmak izi için 1H,13C-HSQC'nin yerini alabilir, çünkü etiketlenmemiş örnekler üzerinde heteronükleer deneyler çok zaman alıcıdır. - UUCG döngüsünü parmak izi referansı olarak kullanın (varsa).
- 1 H R 1φRD denemeleri kaydedilmeden önce her seferinde bu karşılaştırmayı gerçekleştirin.
- 1 H R 1φRD deneylerinin yapıldığı numunenin aşağıdaki spektrumunu düzgün katlanmış referans örneğiyle karşılaştırın (Şekil 4): 1H 1D, özellikle imino bölgesi 10–15 ppm; Aromatik 1H,13C-HSQC; 1 H,1H-SELOPE (isteğe bağlı).
3. 1H R1φ Gevşeme dağılımı—on-rezonans (etiketli 1D versiyon)
NOT: Aşağıdaki adımlarda, HSQC tabanlı RD darbe sırasının 1B sürümünü kullanarak etiketli bir örnek için RD denemelerinin kurulumu açıklanmaktadır. Etiketlenmemiş numuneler için SELOPE tabanlı 1D dizi için aynı adımları izleyin. Her iki durum için parametre adlarına ve ayarlarına genel bakış Tablo 2'de gösterilmiştir. 1D sürümlere odaklanılması, rezonans dışı ölçümler için daha pratik olmalarıdır ve SELOPE ve HSQC tabanlı deneylerin 2D sürümlerinin kurulumu sırasıyla Schlagnitweit ve ark.20 ve Steiner ve ark.10tarafından ayrıntılı olarak tartışılmıştır.
- Sert 90° darbe (P1) için 1 H güç belirleyin.
- A Seçeneği: Bruker pulsecal komutunu kullanın.
- B Seçeneği: Bir zg deneyinde, su zirvesindeki proton sert darbe güç seviyesinde bir fındıklama eğrisi ölçerek 360° darbeyi belirleyin.
NOT: 90° darbe uzunluğu, sıfır sinyalin gözlemlendiği sürenin dörtte biridir (tam bir fındıklama eğrisi ölçülürse, ikinci sıfırdır; ancak pratikte, sadece 360° için beklenen değerin etrafındaki bölge örneklenir).
- Her R 1φölçümünden önce RNA katlamayı onaylamak için adım 3.1'de belirlenen darbe uzunluğunu kullanarak 1 H 1D spektrum zgesgp.f2f3dec çalıştırın.
NOT: 1H SL deneyleri ilk kez çalıştırılırsa, hesaplanan SL gücünün istenen her bant genişliği için SL gücünü kalibre ederek örneğe teslim edilen güce karşılık gelip gelip olmadığını kontrol edin. Ayrıntılı kalibrasyon adımları Steiner ve ark.10'da açıklanmıştır. - Etiketli veri kümesi için 1H R1φ oluşturun ve anahtar parametreleri ayarlayın.
- Yeni bir veri kümesi oluşturun; ideal olarak RNAataması için tam etiketli RNA örneklerinde kullanılan 1 H-13C aromatik HSQC veri kümesine dayanmaktadır.
NOT: Bu, 13C'nin yanı sıra 15N güç ve ayırma gücünün zaten kurulmuş olmasını sağlayacaktır. - Genel parametreleri Tablo 2'ninilk bölümüne göre ayarlayın.
- RD'ye özgü parametreleri Tablo 2'ninikinci bölümüne göre ayarlayın.
- Test için 1H SL gücü en düşük değere (1,2 kHz) ayarlayın.
- Yalnızca bir girişle bir test vd listesi oluşturun, 0 ms(adım 3.4'te açıklandığı gibi vd listesini en iyi duruma getirmek için), TDF1'i 1 olarak ayarlayın ve D30'ugüncelleştirin.
- Bu ayarlarla bir test spektrumu çalıştırın.
- Yeni bir veri kümesi oluşturun; ideal olarak RNAataması için tam etiketli RNA örneklerinde kullanılan 1 H-13C aromatik HSQC veri kümesine dayanmaktadır.
- VD listesini en iyi duruma getirme (kullanılacak SL uzunluklarının listesi).
- Deneyi bir test vd listesiyle çalıştırın(örneğin,altı giriş: 0 m, 5 m, 10 m, 20 m, 30 m, 40 m; ısıtma nedeniyle sistematik hataları önlemek için bu değerleri çırpın).
- D30 ve TDF1'i buna göre güncelleştirin (bu örnekte, D30 = 42m ve TDF1 = 6).
- Tepenin çizim yoğunluğu ile SL uzunluğu. Orijinal tepenin yoğunluğunun 1/3'e düştüğü SL uzunluğunu tanımlayın.
- Aşağıdakileri göz önünde bulundurarak denemede kullanılacak son vd listesini oluşturun: önceki adımda açıklandığı gibi en uzun SL uzunluğunu belirleyin; azalan veya artan düzene sahip bir liste kullanmaktan kaçının; ve istatistiksel çalışmalar için bazı kopyalar ekleyin. VD listesinde her değişiklik olduğunda D30 ve TDF1'i güncellemeyi unutmayın.
NOT: Deneme, vd listesinde verilen farklı SL uzunluklarıyla sahte bir şekilde çalıştırılır. - Listenin en zayıf zirvesinin sinyal-gürültü oranına (SINO) en az 10 olması için tarama sayısını seçin.
NOT: VD listesi düşük bir SL gücü (1,2 kHz) için optimize edilmiş olsa da, bu vd listesi kullanılacak en yüksek SL gücünde(örneğin,15 kHz) test edilmelidir. Bunun nedeni, çürümenin önemli kEX katkısına sahip tepeler için yüksek SL gücünde çok daha yavaş olmasıdır. Bu nedenle, yeterli bir çürüme de yüksek SL gücünde doğrulanmalıdır.
| Parametre Açıklaması | Darbe sırasında parametre adı | |
| 1D etiketli | 1D SELOPE | |
| on-rezonans 1D'ler için darbe programı | 1HR1rho_HCP_onres1D.es | 1HR1r_HH_onres1D.js |
| 1 H taşıyıcı frekansları (ppm) | O1P = ppm'de su rezonansı | O1P = ilgi zirvesinin kimyasal kayması (ppm) |
| CNST28 = ilgi zirvesinin kimyasal kayması (ppm) | CNST29 = ppm'de su rezonansı | |
| 1 H sert 90º darbe | P1 @ PL1 (3.1.1'de kalibre edilmiş olarak) | P1 @ PL1 (3.1.1'de kalibre edilmiş olarak) |
| Su bastırma için şekilli bakliyatlar ve güçler | P25 = 1000 us @ sp3 | P12 = 2000 abd @ sp1 |
| (Watergate) | (heyecanlandırma heykel) | |
| 13 C taşıyıcı frekansı, ilgi zirvesinin 13C kimyasal kayması ile on-rezonans | O2P | – |
| 15 N taşıyıcı frekansı, ayrıştırılme için ortalama 15N kimyasal kayma (aromatik HSQC'de kullanıldığı gibi) | O3P | – |
| 13 C/15N ayırma (HSQC'de olduğu gibi ayarlanır) | pcpd2, cpd2 | – |
| pcpd3, cpd3 | ||
| HCP transferi (örneğin, p=1/J @ 100 Hz) | – | |
| darbe ve pulsef2 komutları sert darbelerden gelen güçleri belirlemek için kullanılabilir | ||
| İlginin zirvesinin süresi (1/J(1H-13C) olarak ayarlanır) | P11 | |
| Güç 1H ve Güç açık 13C | SP1, SP12 | |
| SELOPE transferi (d = 1/4J(H5-H6)) | – | D5 |
| SELOPE için seçici nabız (örneğin aromatik bölge) (4000 us, Eburp) | – | P13 & amp4 |
| SL / RD'ye özgü parametreler: | ||
| 1 Kalibre edilmiş sert darbeden elde edilen H SL gücü (örneğin, darbe komutu kullanılarak). | Pl25 ve CNST12 (1,2 – 15 kHz) | Pl24 (50 Hz – 15 kHz) |
| SL süresi için değişken gecikme listesi (başlangıçta 1 giriş, 0, optimizasyon 3.1.3 altında açıklanmıştır) | vdlist (~ 0 – 40 ms) | vdlist (~ 0 – 150 ms, etiketlenmemiş örneklerdeki düşük R2 nedeniyle) |
| VD listesindeki TDF1 girdi sayısı (başlangıçta 1) | TDF1 | TDF1 |
| Isı telafisi: | ||
| D30 = vd listesindeki en büyük değer + 2ms | D30 | D30 |
| Çok geniş SL'ler için ek ısı telafisi | PL25 | |
| Rezonans dışı özel parametreler: | ||
| rezonans dışı 1D'ler için darbe programı | 1HR1rho_HCP_offres1D.es | 1HR1r_HH_offres1D.js |
| Rezonans dışı deneyler için ofset | CNST30 | CNST30 |
Tablo 2: 1D HCP tabanlı ve 1D SELOPE tabanlı 1 H R1 φdeneyleri ayarlamak için parametrelere genel bakış. Kısaltmalar: 1D = tek boyutlu; HCP = heteronuclear çapraz polarizasyon; SELOPE = SELective Optimize Proton Deneyi; ppm = milyon başına parça; HSQC= heteronuclear spin kuantum korelasyonu; SL = döndürme kilidi; RD = gevşeme dağılımı
- On-rezonans 1 H R 1φdeneylerinin kurulumu ve edinimi
- Denemeyi bölüm 3.4'ten Topspin'deki yeni bir klasöre kopyalayın.
- Bu klasörde, pl25 ve CNST12'yi her seferinde değiştirerek farklı SL güçlerinde denemeler ayarlayın. Darbe komutunu kullanarak her SL gücü için doğru güç seviyesini belirleyin. Daha düşük SL mukavemetleri için daha yoğun örnekleme ile 1,2 ila 15 kHz arasında değişen SL mukavemetlerini kullanın (seçilen SL mukavemetleri için Şekil 5G'ye bakın). Bazı SL güçlü yönleri için yinelenenlere sahip olmak için bazı denemelerin kopyalarını ekleyin.
- Bu deneyleri yapın.
- On-rezonans 1H R1φ deneylerinin analizi
- TopSpin'de, xf2 komutunu kullanarak aynı işleme parametrelerini(örneğin, hat genişletme, faz) kullanarak her sahte 2Dveri kümesinin her dilimini işleyin ve Bruker AU programı split2D'yi kullanarak veri kümesini 1D'ye bölün.
- Her 1D dilim için sinyal yoğunlukları ve hacimleri elde edin.
NOT: Pratikte, potansiyel olarak çakışan zirvelerden katkılardan kurtulmak için spektrumu deconvolve etmek daha iyidir ve bruker AU programının kullanımına izin verir multidcon, tüm dilimlerin zirvelerinin yoğunluklarını veya alanlarını tek bir deneyde kolayca özetler decall.txt, daha sonra diğer programlarla kolayca okunabilir (Steiner ve ark.10tarafından açıklandığı gibi burada şirket içinde yazılmış Python komut dosyaları kullanılmıştır) 3.6.3 ve 3.6.4 adımlarında. - R1φ (veya on-rezonans, R 2+REX)değerini elde etmek için her SL mukavemeti için tek üstel bir bozunma takın.
- Bu R2+REX değerlerini (y) ve .'ye göreçizin. SL mukavemeti (x) (Şekil 5F, G).
NOT: Düşük SL mukavemetleri için değerler önemli ölçüde daha yüksekse ve daha yüksek SL gücüyle azalırsa (Şekil 5G'degösterildiği gibi), araştırılan tepe dağılım gösterir ve heyecanlı devletinpopülasyonu ve kimyasal kayma farkı hakkında bilgi edinmek için ek (rezonans dışı) deneyler yapmak ilginç olabilir. zemin durumu.
4. 1H R1φ Gevşeme dağılımı — rezonans dışı (etiketli 1D versiyon)
- Rezonans dışı 1 H R 1φdeneylerinin kurulumu ve edinimi
- Yeni bir topspin klasöründe, belirli bir SL gücünde denemeler ayarlayın(genellikle REX katkısı orada en yüksek olduğu için en düşük SL gücünde, bkz.
- Şekil 5H,I'degörülebileceği gibi, 0 ofset etrafında daha yoğun bir örnekleme ile ± (3 veya 4)* SL mukavemetine kadar ofsetler kullanın.
- Bu deneyleri yapın.
- Rezonans dışı 1H R1φ deneylerinin analizi
- Her uzaklık için bir R1φ değeri belirlemek için 3.6.1–3.6.3'te olduğu gibi aynı işleme stratejisini kullanın.
- Bu değerleri ofsete karşı çizin (Şekil 5G).
NOT: Bu eğrideki bir asimetri, heyecanlı durum için kimyasal kaydırma bilgilerinin zaten elde edilebileceğini gösterebilir. Bloch-McConnell veya Laguerre denklemleri kullanılarak kapsamlı montaj ve analiz, kEX, pES ve Δω10 ,20 (Şekil 5G)hakkında bilgi edinmek için yapılmalıdır. Her iki 1D deneme için örnek veri kümeleri, darbe programları ve makrolar Petzold Lab Github deposunda (https://github.com/PetzoldLab) bulunabilir. Parametrelere genel bir bakış Tablo 2'de verilmiştir.
Sonuçlar
RNA üretimi protokolü, yüksek saflıkta transkriptlerin üretilmesi yoluyla saflaştırmayı kolaylaştırır. Şekil 3A, tandem transkriptlerin birkaç bölünme reaksiyonunun sonuçlarını gösterir ve hem başarılı hem de başarısız reaksiyonlar sağlar. Lane 1, yan ürünlerin sadece silik izlerine sahip tamamen bölünmüş bir transkriptin en uygun kasasını gösterir. Lane 2a, yeniden tavlama ve daha fazla RNase H eklenmesiyle çözülebilen eksik bölünme gösterir (Lane 2b, adım 2.1.2). 1, 2a ve 2b şeritlerinin RNA yapıları aynıdır. Şerit 3'teki örnek başarısız dekolte gösteriyor. Bu reaksiyonun giderilmesi, bölünme kılavuzu dizisinin, DNA şablonunun saflığının ve tavlama sıcaklıklarının kontrolunu içerir. Potansiyel olarak, RNase H dekoltesi, örnek 2 için gösterildiği gibi T7 IVT'den sonra yapılmalıdır.
Şerit 4'teki örnek, iyon değişimi HPLC ile çıkarılması zor olan önemli miktarda bölünme yan ürünü gösterir. Böyle bir numunenin sorunlarının giderilmesi, (a) sıcaklığın, RNase H miktarının veya reaksiyon süresinin azaltılmasını, (b) elution gradyan ve enjeksiyon hacminin azaltılmasını ve hedef kesirlerin yan ürünlerden ayrılmasına çalışılmasını içerebilir. İyon değişiminde çözünürlüğün nasıl artırılacağı hakkında daha fazla bilgi HPLC saflaştırma Karlsson ve ark.31. HPLC, hedef RNA'yı daha uzun veya daha kısa nükleik asitlerden ve protein veya küçük moleküllü kirleticilerden ayırır. Şekil 3B, iyon değişimi HPLC saflaştırması için en uygun sonucu göstermektedir. Elution gradyanı, hedef RNA türünün bir sonraki küçük türden sonra en az bir sütun hacmini (bu örnekte: 35 mL) ve bir sonraki büyük türden önce bir sütun hacmini atlatacak şekilde seçilmelidir.
Bu yöntemdeki daha küçük türler arasında tek nükleotitler, iptal edici ürünler (8-12 nt), 3' ve 5' ara sıraları (5-14 nt) ve bölünme kılavuzu (12 nt kimerik nükleik asit) bulunurken, daha uzun diziler potansiyel olarak temizlanmamış tandem tekrarları ve plazmiddir. İyi ayrılmış bir elution zirvesi elde edildiğinde, saflaştırma enjeksiyon başına ~20 mL IVT reaksiyonuna eşdeğer olacak şekilde ölçeklendirilebilir. Bir RNA örneğinin doğru katlanmış olması RD deneyleri için çok önemlidir ve her ölçümden önce onaylanması gerekir. Şekil 4, 2.4 (mavi) adımdaki katlama protokolü uygulanmadan önce A etiketli 22 mer RNA'yı ve doğru katlama elde edildikten sonra aynı örneği göstermektedir (kırmızı). Bir Mc-Fold ikincil yapı tahmini (Şekil 4C) sunulan saç tokası yapısını 4 baz çifti ile 5 imino sinyalle sonuçlanır.
Şekil 4A'daki her iki spektrum da, biraz farklı göreceli yoğunluklarda da olsa, bu tahmin edilen sinyalleri doğrular, bu da bazı yanlış katlanmış yapının (burada, bir dimer) sadece 1 H 1Dspektrumla değerlendirilmesinde sorunlu olabileceğini gösterir. Aromatik 1H,13C-HSQC spektrumu (Şekil 4B), ancak katlama protokolünden (mavi) önce numune için aromatik sinyallerin sadece 3'ünü gösterir, ancak 2.4 adımına (kırmızı) göre katlanmış numune için 4 sinyalin tümünü gösterir. Mavi renkte gösterilen örnek muhtemelen saç tokası ile aynı imino sinyallerle sonuç olacak bir homodimer (Şekil 4D'deönerilen yapı) oluşturmuştur. A13H2 sinyali değişim açısından genişlemiş görünüyor. Bu sonuçlar, her RD deneyinden önce hem imino hem de aromatik parmak izi deneyleri ile katlama onayının önemini vurgulamaya yardımcı olur. Bu protokolde açıklanan 1H R1φ darbe dizileri, ara değişim rejimindeki dinamiklerin algılanmasını sağlar. Başlangıçta bir on-rezonans eğrisi kaydedilir ve belirli bir kalıntı için dinamikler varsa, elde edilen R2+REX değerleri içinde bir dağılım görünürken, bu eğri değişimsiz kalıntılar için düzdür.
Şekil 5, sentetik bir RNA saç tokasında iki farklı H8 atomunda elde edilen temsili rezonans eğrilerini gösterir (Şekil 5A), burada G6H8 değişim yaşar (Şekil 5C), A4H8 ise (Şekil 5B). Değişim bu örnekte nispeten yavaş olduğundan(kEX = 292 ± 40 Hz), SELOPE deneyinin düşük SL güçlü yanlarına ulaşma avantajından yararlanıldı ve iki rezonans eğrisi darbe dizisinin 1D sürümü kullanılarak kaydedildi. Aynı darbe dizisi daha sonra on-rezonans profilinde dağılım gösteren kalıntı için rezonans dışı veri elde etmek için kullanıldı. Şekil 5D, elde edilen R1φ değerlerini ve eğrinin hafif bir asimetrisinin zaten Δωişaretini gösterdiği ofseti gösterir.
Bu, R1 katkısının kaldırıldığı R2+REX arsasında daha da belirgin hale gelir ( Şekil5E). Aynı şeklin sağ sütunu, iki farklı H8 atom için elde edilen temsili on-rezonans eğrilerini daha hızlı değişime sahip biraz farklı sentetik RNA saç tokasında gösterir, burada G6H8 değişim yaşar (Şekil 5G), A4H8 ise (Şekil 5F). Daha hızlı döviz kuru(kEX = 43.502 ± 38.478 Hz), hem açık hem de rezonans dışı verileri (Şekil 5H,I'degörüntülenen G6H8 verileri) elde etmek için SELOPE 2D sürümünü kullanarak tüm aromatik protonların rd kaydına izin verdi.
Olumlu ve olumsuz sonuçlar için genel tanımlayıcılar
Tandem IVT ve RNase H dekoltesinde olumlu sonuçlar şu şekilde tanımlanabilir: 1) Hedef bant denatüre PAGE jeldeki en güçlü banttır. 2) Ana bandın etrafında zayıf bantlar yoktur veya sadece zayıf bantlar yoktur. 3) Daha yüksek moleküler ağırlıklı türler yoktur veya sadece zayıftır. 4) HPLC kromatogramı, hedef RNA'nın iyi ayrılmış bir zirvesini gösterir. 5) Ana tepe örneklendiğinde, denatüre page jel üzerinde sadece bir bant görünür.
Tandem IVT ve RNase H dekoltesinde negatif sonuçlar şu şekilde mevcuttur: 1) Denatüre page jel üzerinde hayır veya sadece zayıf bir ana bant görülebilir. 2) RNA tandem tekrarlarından yüksek moleküler ağırlıklı türlerin bir deseni görülebilir. 3) Ana bant mevcut olmasına rağmen, benzer yoğunlukta bantlar ± 3 nt içindeki ana bandın üstünde veya altındadır.
İyi katlanmış bir örnek aşağıdaki gibi tanımlanabilir: 1) Gözlemlenen imino protonların sayısı, ikincil bir yapı simülasyonundan beklenen imino proton sayısıyla eşleşir (örneğin,Mc-Fold39, Şekil 4A). 2) Bir UUCG döngüsündeki (varsa) syn G-U sallantı taban çifti ~ 9,5 ppm'de görülebilir, bazen sadece daha düşük sıcaklıkta görülebilir. UUCG döngüsünün daha fazla parmak izi Fürtig ve meslektaşları tarafından tanımlanmıştır40. 3) Aromatik parmak izi, doğru katlandığında onaylanmış daha önce atanmış bir örnekle aynı fikirdedir (Şekil 4C).
Yanlış katlanmış veya bozulmuş bir örnek aşağıdaki gibi tanımlanabilir: 1) İkincil bir yapı simülasyonunun öngördüğünden daha fazla imino sinyali vardır (NOT: daha az imino sinyali, kapanış taban çiftleri genellikle görünmediği ve konformasyonsal değişim çizgileri genişletdiği için mutlaka yanlış katlanma anlamına gelmez). 2) İmino sinyallerinin yokluğu. 3) Aromatik bölgede tek nükleotid bozunma ürünlerini gösteren yüksek yoğunluklu dar sinyaller. 4) Onaylanmış katlama referans örneğine imino veya aromatik sinyaller arasında ayrışma (Şekil 4C).
Algılanabilir zaman ölçeğinde değişim göstermeyen bir atom aşağıdaki gibi tanımlanabilir: 1) düz bir RD profilinden (uygulanan SL gücüne göre değişen eksik REX katkısı nedeniyle) (Şekil 5B ve Şekil 5F). 2) kEX ve Δω aynı büyüklükteki olduğunda yavaş ara değişim durumunda dikkatli olunmalıdır. Bu durumda, on-rezonans katkısı Şekil 5C'de görülebileceği gibi çok küçük olabilir (bu durumda takılan parametreler kEX = 292 ± 40 Hz ve Δω = 112 ± 4 Hz'dir). Şüpheniz varsa, doğrulama için düşük bir SL rezonans eğrisi kaydedilebilir.
Ara zaman ölçeğinde değişimi gösteren bir atom, 1) on-rezonans RD deneyinde düz olmayan bir gevşeme dağılımı profilinden tanımlanabilir (Şekil 5B ve Şekil 5F); 2) HSQC veya SELOPE deneyinde daha geniş bir çizgi genişliği de değişim için bir gösterge olabilir.
Rezonans dışı eğriler için iyi seçilmiş SL güç değerleri (Şekil 5E, F): 1) on-rezonans eğrisinde önemli bir k EX katkısına sahiptir (seçilen SL güç değerleri Şekil 5C ve Şekil 5G'debelirtilmiştir). 2) En az 3 SL güç değeri için rezonans dışı eğriler ölçüldüğünden, seçilen SL güç değerleri kEX katkı ile on-rezonans eğrisinin bölgesine yayılmalıdır. 3) Laguerre sığdıktan sonra düz olmayan R2+REX eğrilerine yol açar (örneğin, Şekil 5D: SL mukavemetleri 25, 50 ve 75 Hz; Şekil 5E).
Rezonans dışı eğriler için kötü seçilmiş SL güç değerleri (Şekil 5E, F) Laguerre sığdıktan sonra düz R2+REX eğrilerine yol açar. Bir örnek gösterilmiştir Şekil 5E, burada 100 Hz rezonans eğrisi çok düzdür ve bu nedenle Δωhakkında önemli bilgi sağlamaz.
Dönen çerçeveli nükleer Overhauser etkisi (ROE) eserleri için endikasyonlar: 1) Rezonans eğrilerinden elde edilen Δω, nükleer Overhauser etkisi spektroskopisi (NOESY) spektrumundaki ilginin zirvesi ile çapraz bir tepe gösteren uzaysal çevre / protonlardaki protonların kimyasal kaymalarıyla eşleşir. (örneğin, Şekil 5I, hızlı ara değişim için beklendiği gibi geniş rezonans dışı eğriler gösterir, ancak eğriler -3000 Hz ve +1500 Hz'de daha keskin özelliklere de sahiptir. Bunlar büyük olasılıkla farklı bir konformörde bu H8 için kimyasal bir kayma yerine bir ROE eserinden kaynaklanmaktadır). 2) Laguerre fit işe yarar, ancak yüksek SINO (>20)(örneğin, kEX = 43.502 ± 38.478 Hz) ile yapılan deneylerden üsteller elde edilmiş olsa da, bir on-rezonans ve en az 3 rezonans eğrisi için iyi çalışmaz (yüksek hatalar veya fiziksel olarak imkansız değerler verir). Genellikle her SL ayrı ayrı iyi uyar, ancak bunları birbirine uydurmak çok daha yüksek bir hata verir; gerçek bir heyecanlı durum için tam tersi bir davranış beklenir.
"Gerçek" değişim endikasyonları Δω: 1) Rezonans dışı eğrilerden elde edilen Δω, NOESY spektrumunda ilginin zirvesi ile çapraz bir zirve gösteren uzaysal çevre / protonlardaki protonların kimyasal kaymalarıyla eşleşmez (örneğin, Şekil 5E). 2) Laguerre fit, on-rezonans için düşük hatalar ve en az 3 rezonans dışı eğri verir(örneğin, Şekil 5E vs. Şekil 5I, uygun sonuçlar için resim yazısına bakın).
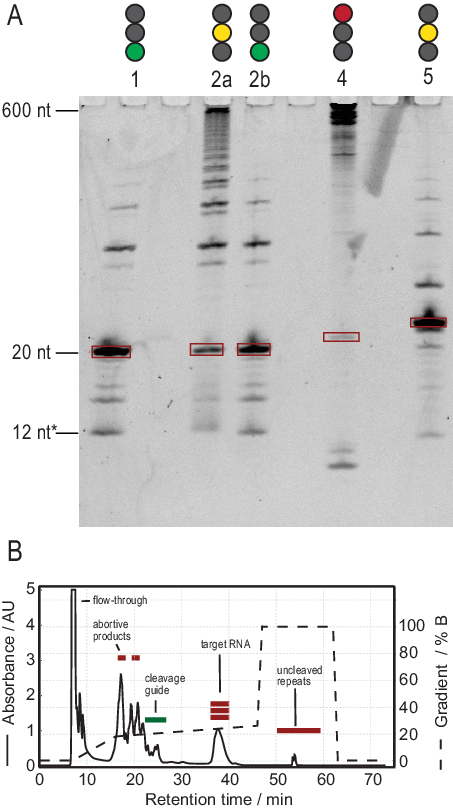
Şekil 3: T7 tandem IVT ve RNase H dekolte reaksiyonunun örnek üretimi. (A) Tandem IVT ve RNase H dekoltesinin olumlu ve olumsuz sonuçlarının denatüre SAYFASI. Merdiven yüksekliği RNA referanslarını, 12* ise kimerik dekolte kılavuzunu ifade eder. Şerit 1: 20 nt hedef RNA'nın başarılı üretimi. Birkaç kısa ve uzun ürün mevcuttur. Şerit 2a: Tandem transkriptinin eksik bölünmesi. HPLC saflaştırması mümkün olsa da, çok fazla malzeme boşa harcanır. Lane 2b: Lane 2'nin devam eden RNase H bölünmesi, HPLC enjeksiyonuna hazır temiz bir örnek üretir (Lane 1 ile aynıdır). Lane 4: RNase H dekoltesi büyük ölçüde başarısız oldu ve hedef bant üretilmedi. Tam uzunlukta tandem transkript hala 600 nt'de görülebilir. Lane 5: Bir hedef bant üretildi, ancak güçlü bir -1 bandı mevcut. HPLC yapılabilmesine rağmen, yan ürünün dikkatli bir şekilde çıkarılması gerekir. (B) Başarılı bir HPLC enjeksiyonu örneği. 38 dakikadaki zirve, hedef uzunlukta saf RNA içerirken, daha uzun ve daha kısa ürünler hedef RNA'dan iyi ayrılır. Panel B 21'den değiştirildi. Kısaltmalar: IVT = in vitro transkripsiyon; HPLC = yüksek performanslı sıvı kromatografisi; nt = nükleotitler; AU = rasgele birimler. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
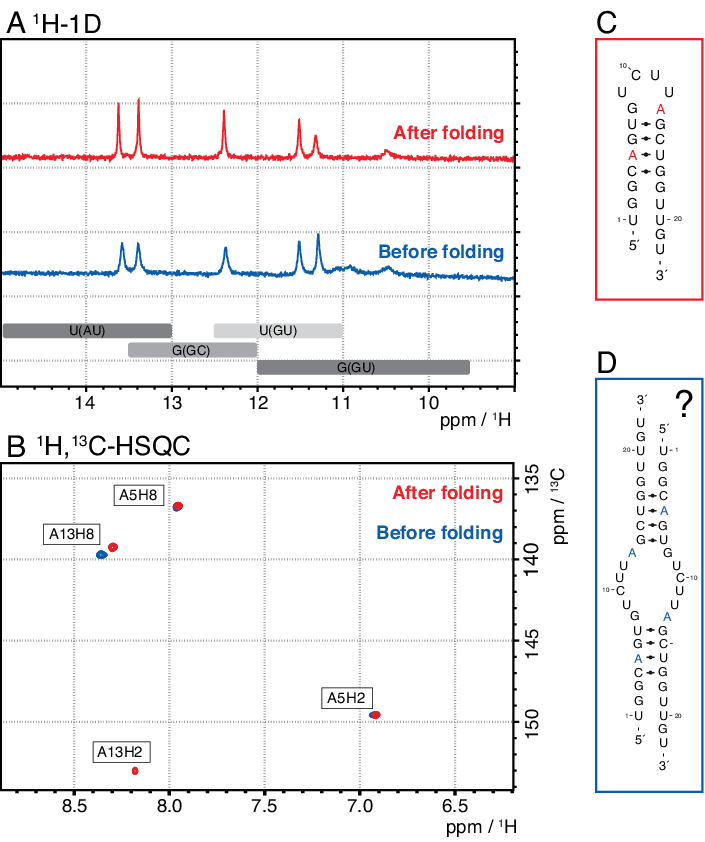
Şekil 4: A etiketli22-mer RNA'nın 1 H-1D spektrumunun NMR. (A ) Imino bölgesindeki katlama adımı 2.4 (bkz. protokol) öncesi (mavi) ve sonra (kırmızı) RNA saç tokası örneği. imino sinyallerinin baz çifti kimliği için beklenen bölgeler aşağıda gri ile belirtilmiştir. (B) 1H,13C-HSQC spektrumu RNA'nın aromatik rezonanslarının panel A'dan. Katlamadan sonraki örnek (kırmızı) beklendiği gibi 4 sinyal gösterirken, katlamadan önceki örnek (mavi) yalnızca 3 sinyal gösterir. (C) 22-mer RNA'nın saç tokası olarak Mc-Fold tahmini. Panel A. (D) 22 mer RNA tarafından oluşturulan bir homodimerin önerilen yapısında her iki örnekte de bulunabilen bu ikincil yapıdan beş imino sinyali beklenebilir ve bu da saç tokası yapısıyla aynı 5 baz çifti ile sonuçlanır. Kısaltmalar: NMR = nükleer manyetik rezonans; 1D = tek boyutlu; HSQC = heteronükleer tek kuantum korelasyonu; ppm = milyon başına parça. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
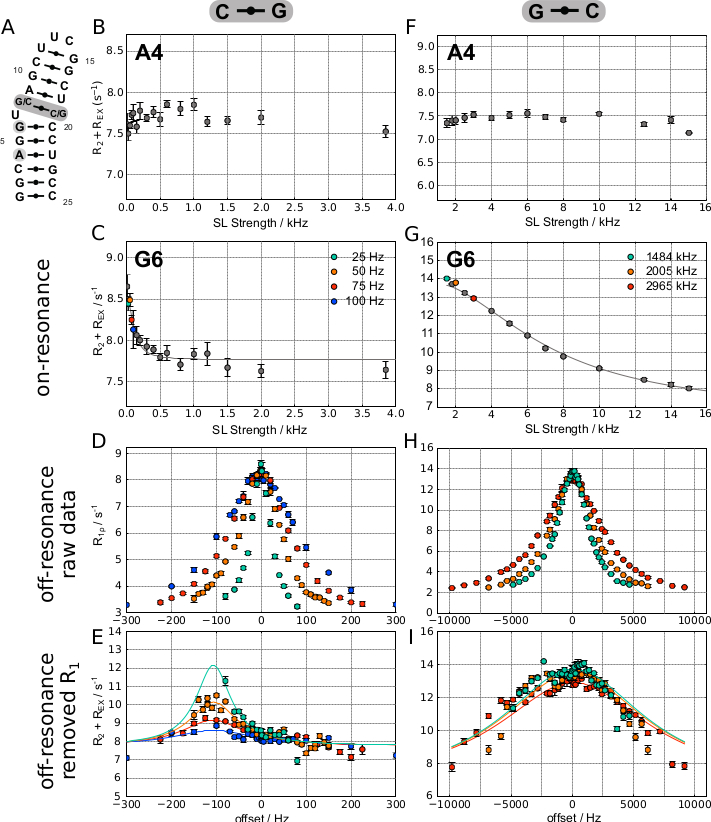
Şekil 5: RNA saç tokası bazlı iki farklı yapı için 1H R 1φ RD temsili sonuçları. (A) Sol sütun, şişkin U'nun üzerinde bir C-G taban çifti ile RNA'da elde edilen sonuçları gösterirken, sağ sütun, taban çiftinin yerine G-C'ye geçirildiği bir örnekte elde edilen sonuçları gösterir. (B) ve (F) iki yapı için A4H8 için elde edilen düz dispersiyon profillerini gösterir ve konformasyonsal değişim olmadığını gösterir. (C–E) (G-C) yapısında G6 için elde edilen on-rezonans, off-rezonans ve takılı verileri gösterir. Laguerre uyumu aşağıdaki sonuca yol açar: R1 = 2.87 ± 0.01 Hz, R2 = 7.76 ± 0.03 Hz, kEX =292 ± 40 Hz, pES = 0,31 ± %0,03, Δω = 112 ± 4 Hz. (G–I) (G-C) yapısında G6 için elde edilen on-rezonans, off-rezonans ve takılı verileri gösterir. Laguerre uyumu aşağıdaki sonuca yol açar: R1 = 1.93 ± 0.02 Hz, R2 = 6,71 ± 0,86 Hz, k EX = 43,502 ± 38,478 Hz, p ES = 27 ± %16, Δω = 203 ± 166 Hz. Bu rakam 20'den değiştirildi. Kısaltma: SL = spin lock. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Burada sunulan protokol, daha önce 10,20 ,21,31araştırma makaleleri şeklinde yayınlanan çeşitli protokollerin bir sentezidir. Bu nedenle, protokolün bölümleri uygulanabilirken, diğerleri okuyucunun tercihine değiştirilebilir. Örneğin, R1φ ölçümleri, katlama ve homojenlik uzunluğu varsayıldığı göz önüne alındığında, herhangi bir yöntemle üretilen bir RNA numunesi üzerinde gerçekleştirilebilir. Ayrıca, protokol RNA dizisinin rezonans ataması hakkında bilgi içermez-RD deneyleri için gerekli bir adım- bu önceki literatür19,37,38'dekapsamlı bir şekilde ele alınmıştır. Kısmi, segmental veya siteye özgü etiketleme şemaları36,41,42,43,44, rezonans atamasını kolaylaştırmak veya RD deneylerinde ilgi çekici olan ve literatürde uzun uzadıya açıklanan rezonansların çakışmasını azaltmak için yaklaşımlardır. Bu yöntem, rezonans atamasını önemli ölçüde basitleştirebilen herhangi bir nükleotid kimliğinin tekdüze etiketlemesinin kullanılmasına izin verir.
Burada sunulan IVT yöntemi, diziler ve etiketleme ile bilinen sorunların üstesinden gelir, verimi artırır ve maliyet ve çalışma süresini diğer yöntemlere göre azaltır. Viral başlatma dizisinin kullanımı reaksiyon optimizasyonu ihtiyacını azaltır, bu da alanda yapılması zaman alıcı olabilen bilinen bir sorundur ve G olmayan başlatma durumunda transkriptin sadece birkaç kopyasını verir. Tandem transkriptin T7 IVT ve RNase H dekoltesi aynı kapta aynı anda yapılabilir. Reaksiyon sırasında, RNase H reaksiyonunun tamamlanmasıyla hedef RNA'da tek bir bantta birleşen denatüre page jel üzerinde multimerik tandem tekrarlarının bir deseni görülebilir(Şekil 3A, şeritler 1 ve 2b). Bu yöntemi kullanan tipik verimler 1 mL IVT başına 30 ila 70 nmol RNA arasında değişmektedir. Yine de, tandem tekrarlarının RNase H bölünmesine dayanan yöntem, kendi başına belirli sorunlar olmadan gelmez. RNase H bölünme reaksiyoni genellikle T7 transkripsiyon ile aynı anda çalıştırıldığında tamamlanmaz(Şekil 3A, şerit 2a).
Tandem ünitelerin ayrılması, transkript için dekolte kılavuzu tavlanarak ve daha fazla RNase H eklenerek sonlandırılabilir(Şekil 3A, şerit 2b, adım 2.1.2). Büyük hacimlerin ısıtılması yavaş olduğundan ve RNA'nın Mg2+katalizli hidrolizine yol açtığından, numuneyi 10-15 s'de >95 ° C'ye ısıtan geleneksel bir mikrodalga fırın kullanılmıştır. Üretilen numuneler üzerinde şu ana kadar olumsuz etkiler gözlenmedi. Bazı yapılar reaksiyon koşullarının optimizasyonu ile ortadan kaldırılamayan küçük bir ikinci bant göstermektedir(Şekil 3A, şerit 4). Genellikle bunlar, iyi optimize edilmiş bir elution gradyanı kullanılırsa ve çıkarılabilirse HPLC kromatogramında bir omuz olarak oldukça net bir şekilde görülebilir (adım 2.2.5). Aşağıdaki tartışma, özellikle konformasyonsal dinamiklerin yorumlanmasına izin veren yüksek kaliteli verilerin elde edilmesiyle ilgili olarak protokoldeki kritik adımları vurgulamayı amaçlamaktadır.
RNase kontaminasyonu
Hücre dışı RNas'lar her yerde bulunur, son derece kararlıdır ve NMR örneklerinin uzun vadeli stabilitesi için en büyük tehdidi oluşturur. Bu nedenle, RNase içermeyen bir ortamda çalışmak ve tüm reaktifleri ve plastik yazılım RNase'leri içermeden tutmak çok önemlidir. Filtre uçlarının ve hatta yüz maskelerinin kullanılması önerilir. Bu, HPLC saflaştırmadan sonra özellikle önemlidir. RNases ile kontamine olmuş NMR örnekleri tipik olarak tek nükleotid bozulma ürünleri nedeniyle günler veya haftalar sonra 1H-1D spektrumda görülebilen dar tepeler sergiler. Böyle bir örnek R1φ ölçümleri için uygun değildir.
NMR örneği
Yüksek yüklü doğası nedeniyle, RNA çoğu proteine kıyasla yağış olmadan yüksek konsantrasyonlarda kullanılabilir. Shigemi NMR tüplerinin kullanımı (Malzeme Tablosunabakın), yüksek konsantre numunenin bobinin ortalamasına izin verirken, duyarlılıkla eşleşen cam alt ve piston nedeniyle ideal shimming ve kilitleme koşulları sağlarken avantajlıdır. Bu şekilde, B1-inhomogeneity azalır ve daha dar çizgilere yol açtı. Bir NMR tüpündeki tipik numune hacmi 250 μL'dir ve tipik konsantrasyon 1-2 mM'dir. 500 μM'nin altındaki örnekler RD deneyleri için önerilmez, çünkü deney çok uzun sürer ve iyi bir dolgu. Benzer şekilde, 200 μL'nin altındaki numune hacmi önerilmez, çünkü iyi bir dolgu ve alan stabilitesi (kilit) gereklidir. Pistonu takarken, numunede kabarcık oluşumunu önlemek çok önemlidir (adım 2.4.5). Düzgün sabitlenmezse, piston numuneye doğru kayarak algılanabilir birimi azaltabilir. Ayrıca, sıcaklıktaki hızlı değişiklikler numunede yeni kabarcıkların oluşmasına neden olabilir. Bu nedenle, numune taşınırken ve NMR spektrometresunda prob sıcaklığını değiştirirken dikkatli olunmalıdır. Daha uzun bir süre sonra tekrar ölçüm yaparken numunede kabarcık olup olmadığını kontrol edin.
RNA katlama
Dinamik RNA molekülleri düzgün katlandığında birden fazla konformasyonda bulunabilir. İkincil yapıların erime sıcaklıkları oda sıcaklığının sadece biraz üzerinde olsa da, ölçümden önce kapsamlı bir ısıtma ve snap soğutma prosedürü önerilir. Kinetik kontrol (ısıtma ve snap-soğutma) altında katlanan yüksek konsantre saç tokası örnekleri zamanla homodimer oluşturabilir ve bu da her NMR ölçümünden önce RNA katlamanın sıkı kontrolünü gerektirir. Ölçülen RNA bir saç tokası yapısı değil, RNA dubleks ise termodinamik kontrol altında yavaş katlama uygulanmalıdır.
Bu durumda, ısıtmadan sonra soğutma işlemi saat aralığında olmalıdır, RNA ise NMR numunesinde son hacminde ve konsantrasyonunda kullanılır. Beklenen imino ve aromatik rezonansların ilk sayısı, numunenin homojenliği hakkında fikir verebilir. Örnek beklendiği gibi görünmüyorsa, yeniden katlanmalıdır. Mg2+ (klorür tuzu olarak eklenir) katlanır RNA yapıları ile yardımcı olabilir45. Pratikte, katlama kontrolü, en azından NMR rezonanslarını kısmen atamak ve ikincil yapıyı deneysel olarak çözmek için kullanılan bir örnekle karşılaştırma görevi görür.
Dönüş kilidi gücü ve ısıtma konuları
1 H R 1φRD deneylerinin2B genel bakış deneyleri olarak çalıştırılması durumunda, SL gücü 1,2 kHz'den düşük olmamalıdır. Radyofrekans verici frekansı, ilgi çekici zirvelerin ppm bölgesinin ortasına yerleştirilmelidir(örneğin,aromatik protonlar için 7,5 ppm). 1.2 kHz bant genişliği daha sonra bu protonları herhangi bir büyük rezonans etkisi olmadan spin-lock yapacak kadar büyük olacaktır. Bu tür efektler RD profilinde tanımlanabilir. Bunlar oluşursa, özellikledüşük SL gücü için artan SL güç değerleriyle R2+REX değerleri azalmak yerine artar. Hesaplanan SL güç değerlerinin örneğe teslim edilen güce karşılık gelip olmadığını kontrol edin. Pratikte, 1H 90 ° sert darbe daha yeni spektrometrelerde dikkatlice kalibre edilmişse, hesaplanan SL gücü kullanılabilir; ancak, istenen her bant genişliği için SL gücü kalibre edilerek bu kontrol edilebilir.
1 H R 1φRD deneylerinde kullanılabilen SL güç aralığı çok geniştir ve değişen numune ısıtmasına yol açmaktadır (HCP tabanlı diziler için HSQC için 1,2 kHz ila 15 kHz ve SELOPE deneyleri için 50 Hz ila 15 kHz). Eşit olmayan numune ısıtma, düşük güçlü SL'ler için elde edilen 1D'ler karşılaştırıldığındakimyasal kaymada hafif bir değişiklik olarak tespit edilebilir. yüksek güçlü SL'ler. Bu etki genellikle heteronuclei üzerindeki R1φ deneylerinde ısı telafilerinde dikkate alınmaz. Bu deneylerdeki ısı telafisi genellikle her spin kilidi güç serisinin vd listesinde belirtilen farklı dönüş kilidi süreleri nedeniyle farklı ısıtma için düzeltecek şekilde ayarlanır. Özellikle SELOPE deneyi için,20'de açıklandığı gibi tüm uygulanan SL güçlerinde ikinci bir ısı telafisi kullanılmalıdır.
vd listesiyle ilgili önemli noktalar
Daha önce de belirtildiği gibi, vd listesi önemli bir yoğunluk bozunması elde etmek için yeterince uzun bir zaman noktası içermelidir (ideal olarak ilk sinyalin% 30'una kadar veya probun özellikleri içinde% 70'lik bir çürümeye ulaşmak mümkün değilse mümkün olduğunca düşük). VD listesi düşük bir SL gücü (1,2 kHz) için optimize edilmiş olsa da, bu vd listesi kullanılacak en yüksek SL gücünde(örneğin,15 kHz) test edilmelidir. Bunun nedeni, önemli REX katkısına sahip zirveler için, çürümenin yüksek SL gücünde çok daha yavaş olacağı gerçeğidir. Bu nedenle, yüksek SL gücünde yeterli bir çürüme de doğrulanmalıdır. Aynı durum, rezonans dışı deneylerde yüksek ofsetlerdeki çürümeler için de düşünülmelidir. VD listesinin ideal maksimum zaman noktası, dağılım deneyinin farklı bölgeleri için önemli ölçüde farklı olabilir. Bu durumda, vd listesine daha fazla nokta eklenebilir ve analiz sırasında daha yüksek SL gücü veya daha yüksek uzaklıklar için daha uzun vD liste noktaları, yol açacakları düşük SINO'ya bağlı olarak atılabilir. Genel olarak, 5-8 vd liste noktalarının J-kaplin gibi üstel olmayan çürümelere yol açan potansiyel eserleri tespit edebileceği düşünülmelidir (aşağıya bakın).
1D-HCP seçicilik konuları
HCP tabanlı 1D sürümü çalıştırırken, 2D HSQC tabanlı deneyin 1H boyutunda ilginin zirvesiyle çakışan başka bir tepe varsa, özel dikkat gösterilmelidir. HCP tabanlı transferler çok, ancak asla% 100 seçicidir ve bu nedenle başka bir tepenin 1D'deki ilgi zirvesinin yoğunluğuna ve çürüme davranışına katkıda bulunduğu gerçekleşebilir. Bunun için bir gösterge, etiketli deneyin 1D ve 2D sürümleri kullanılarak elde edilen on-rezonans R 1φ değerlerinde bir fark olacaktır.
ROE ile ilgili önemli noktalar:
Yavaş ara değişime sahip atomların rezonans dışı eğrileri için, elde edilen Δω'nin NOESY veya ROESY spektrumu ile karşılaştırılması temel alınarak ROE eserleri tanımlanabilir. Bir çapraz tepe Δω'yekarşılık gelen kimyasal bir kayma farkıyla tanımlanabilirse, gözlemlenen heyecanlı durum aslında bir ROE eseri olabilir(örneğin,ROE'ler aromatik protonlar arasında bulundu, hepsi aynı kimyasal kayma aralığındadır ve bu nedenle rezonans dışı eğriler tarafından kaplanmıştır20). Deneyime göre, bu her zaman büyük hatalarla zayıf uyumlara yol açtı, muhtemelen ROE'nun artan SL gücüyle REX ile aynı deseni takip etmemesi nedeniyle. Orta-hızlı değişim için durum daha da zorlaşır. On-rezonans eğrisi (komşu çekirdek üzerinde elde edilen 13C verisi ile karşılaştırıldığında) hala GS ve ES arasındaki değişim sürecini temsil ederken, rezonans dışı eğri birden fazla ROE eserinden etkilenir.
Bu durumda, değişim işlemini algılamak için SL gücü daha büyüktür (>1,5 kHz) ve bu nedenle, rezonans dışı eğriler çeşitli ROE adaylarının kimyasal kayma farklılıklarına yayıldığı için daha fazla sayıda protona yayılır (H8 için bunlar: ca'daki amino protonlar. ±1000 Hz, H5/H1'ler ca. -1200 Hz, imino protonlar yaklaşık 3500 Hz). Noe/ROE katkısı NOESY spektrası ile hariç tutulamazsa, bu yöntemle gerçek Δω hakkında güvenilir bir bilgi çıkarılamadığından, şimdiye kadar bu ROE eserlerini bastıracak bir yöntem bulunamadı (kısmen kısırlaştırılmış nükleotitler46kullanmak dışında) ve off-rezonans verileri hızlı-ara değişim için kaydedilmemelidir.
J-Kaplin (Hartmann-Hahn) dikkat edilmesi gereken noktalar
H6 gibi homonükleer J-bağlantılı protonlar için on-rezonans eğrileri başarıyla kaydedilmiş olsada, 10,20, rezonans dışı ölçümler için özel dikkat gösterilmelidir, özellikle Hartmann-Hahn eşleştirme koşulları araştırılan ofsetlerin geniş bir yelpazesini kapsayabileceğinden düşük SL gücü için. Hartmann-Hahn eserleri, üstel çürüme üzerinde salınımlar veya on-rezonans RD arazilerinde artan SL güçleri ile artanR2+REX değerleri olarak tanımlanabilir20.
Açıklamalar
K.P., RNA'yı hedefleyen küçük molekülleri keşfeden Arrakis Therapeutics şirketinin danışmanıdır.
Teşekkürler
Karolinska Institutet'teki protein bilimi tesisine (PSF) T7 RNA polimerazının ve E. coli RNase H'nin ifade ve saflaştırılması için, Martin Hällberg'e inorganik fosfatazın cömert hediyesi için ve tüm Petzoldlab'a değerli tartışmalar için teşekkür ederiz. U-bulge yapılarının hazırlanması için Luca Retattino'ya ve makrolara ve uygun komut dosyalarına katkılarından dolayı Emilie Steiner ve Carolina Fontana'ya teşekkür ederiz. Karolinska Enstitüsü ve Tıbbi Biyokimya ve Biyofizik Bölümü'nü 600 MHz spektrometre ve pozisyon finansmanı (KI FoAss ve KID 2-3707/2013) satın alımını desteklemek için kabul ediyoruz. Vetenskapsrådet (#2014-4303), Stiftelsen för strategisk Forskning (ICA14-0023 ve FFL15-0178) ve Ragnar Söderberg Stiftelse (M91-14), Harald och Greta Jeansson Stiftelse'nin (J) finansal katkıları için minnettarız.S20140009), Carl Tryggers stiftelse (CTS14-383 ve 15-383), Eva och Oscar Ahréns Stiftelse, Åke Wiberg Stiftelse (467080968 ve M14-0109), Cancerfonden (CAN 2015/388), J.S. Marie Skłodowska-Curie IF (AB H2020, MSCA-IF proje no. 747446).
Malzemeler
| Name | Company | Catalog Number | Comments |
| 40% Acrylamide/Bis Solution | Bio-Rad | 161-0144 | |
| 5-alpha Competent E. coli | NEB | C2987I | |
| Acetic Acid | Sigma-Aldrich | 49199 | |
| Acetonitrile | Sigma-Aldrich | 34851 | |
| AFC-3000, HPLC Fraction collector | Thermo Scientific | 5702.1 | |
| Agarose | Sigma-Aldrich | A9414 | |
| Amersham ImageQuant 800 UV | GE Healthcare | 29399482 | Replacing LAS-4000 or equivalent |
| Amicon ultra centrifugal filter unit | Sigma-Aldrich | UFC900324 | |
| Ammonium persulfate | Sigma-Aldrich | A3678 | |
| Ampicillin | Sigma-Aldrich | A9518 | |
| ATP | Sigma-Aldrich | A2383 | |
| ATP-13C10/15N5 | Sigma-Aldrich | 645702 | |
| BamHI restriction enzyme | NEB | R0136L | |
| Bottle top filter | VWR | 514-1019 | |
| Bromophenol Blue | Sigma-Aldrich | 1081220005 | |
| Cleavage guide | IDT | N/A | or equivalent |
| CTP | Sigma-Aldrich | C1506 | |
| CTP-13C10/15N5 | Sigma-Aldrich | 645699 | |
| D2O | Sigma-Aldrich | 151882 | |
| Dionex Ultimate 3000 UHPLC system | Thermo Scientific | N/A | |
| DL-Dithiotreitol | Sigma-Aldrich | 43815 | |
| DMSO | Sigma-Aldrich | D8418 | |
| DNAPac PA200 22x250 Semi-Prep column | Thermo Scientific | SP6734 | |
| DNAPac PA200 22x50 guard column | Thermo Scientific | SP6731 | |
| E.coli RNase H | NEB | M0297L | or made in-house uniprot ref. P0A7Y4 |
| EDTA | Sigma-Aldrich | E6758 | |
| Eppendorf centrifuge, rotor: A-4-44 | Eppendorf | 5804R | |
| Ethanol 95% | Fisher scientific | 11574139 | |
| Ethanol 95% denatured | VWR | 85829.29 | |
| Formamide | Sigma-Aldrich | 47671 | |
| GelRed | VWR | 41003 | |
| GeneRuler 1kbp Plus | Fisher Scientific | SM1333 | Optional |
| GMP | Sigma-Aldrich | G8377 | |
| GMP-13C10/15N5 | Sigma-Aldrich | 650684 | |
| GTP | Sigma-Aldrich | G8877 | |
| GTP-13C10/15N5 | Sigma-Aldrich | 645680 | |
| Hydrochloric Acid | Sigma-Aldrich | H1758 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-100UN | or made in-house uniprot ref. P0A7A9 |
| Invitrogen UltraPure 10X TBE-buffer | Sigma-Aldrich | T4415 | |
| Julabo TW8 Water bath | VWR | 461-3117 | |
| kuroGEL Midi 13 Horizontal gel electrophoresis | VWR | 700-0056 | or comparable |
| LB broth (Lennox) | Sigma-Aldrich | L3022 | |
| LB broth with agar (Lennox) | Sigma-Aldrich | L2897 | |
| Low Range ssRNA Ladder | NEB | N0364S | Optional |
| LPG-3400RS Pump | Thermo Scientific | 5040.0036 | |
| Magnesium chloride hexahydrate | Sigma-Aldrich | 63068 | |
| microRNA Marker | NEB | N2102S | |
| Microwave oven | Samsung | MS23F301EAW | |
| Mini-PROTEAN electrophoresis equipment | Bio-Rad | 1658004 | |
| NucleoBond Xtra Maxi | Machinery-Nagel | 740414.10M | |
| pUC19 plasmid containing tandem insert | Genscript | N/A | or equivalent |
| RNaseZAP | Sigma-Aldrich | R2020 | |
| Shigemi tube 5mm | Sigma-Aldrich | Z529427 | |
| Single-use syringe, Luer lock tip | VWR | 613-2008 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium chloride | Sigma-Aldrich | 730-1470 | |
| Sodium perchlorate | Sigma-Aldrich | 71853 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S3264 | |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Spermidine trihydrochloride | Sigma-Aldrich | 85578 | |
| SYBR Gold | ThermoFisher | S11494 | |
| Syringe filters | VWR | 514-0061 | |
| T7 RNA polymerase | Sigma-Aldrich | 10881767001 | or made in-house uniprot ref. P00573 |
| TCC-3000RS Column thermostat | Thermo Scientific | 5730 | |
| Tetramethylethylenediamine | Sigma-Aldrich | T9281 | |
| Tris Base | Fisher Scientific | 10103203 | |
| UMP | Sigma-Aldrich | U6375 | |
| UMP-13C9/15N2 | Sigma-Aldrich | 651370 | |
| Urea | Sigma-Aldrich | U5378 | |
| UTP | Sigma-Aldrich | U6625 | |
| UTP-13C10/15N5 | Sigma-Aldrich | 645672 | |
| VWD-3100 Detector | Thermo Scientific | 5074.0005 |
Referanslar
- Djebali, S., et al. Landscape of transcription in human cells. Nature. 489 (7414), 101-108 (2012).
- Doudna, J. A., Cech, T. R. The chemical repertoire of natural ribozymes. Nature. 418 (6894), 222-228 (2002).
- Sehgal, P. B., Westley, J., Lerea, K. M., DiSenso-Browne, S., Etlinger, J. D. Biomolecular condensates in cell biology and virology: phase-separated membraneless organelles (MLOs). Analytical Biochemistry. , 597(2020).
- Herschlag, D., Allred, B. E., Gowrishankar, S. From static to dynamic: the need for structural ensembles and a predictive model of RNA folding and function. Current Opinion Structural Biology. 30, 125-133 (2015).
- Kimsey, I. J., Petzold, K., Sathyamoorthy, B., Stein, Z. W., Al-Hashimi, H. M. Visualizing transient Watson-Crick-like mispairs in DNA and RNA duplexes. Nature. 519 (7543), 315-320 (2015).
- Dethoff, E. A., Petzold, K., Chugh, J., Casiano-Negroni, A., Al-Hashimi, H. M. Visualizing transient low-populated structures of RNA. Nature. 491 (7426), 724-728 (2012).
- Baisden, J. T., Boyer, J. A., Zhao, B., Hammond, S. M., Zhang, Q. Visualizing a protonated RNA state that modulates microRNA-21 maturation. Nature Chemical Biology. 17 (1), 80-88 (2021).
- Marušič, M., Schlagnitweit, J., Petzold, K. RNA dynamics by NMR spectroscopy. Chembiochem. 20 (21), 2685-2710 (2019).
- Baronti, L., et al. Base-pair conformational switch modulates miR-34a targeting of Sirt1 mRNA. Nature. 583 (7814), 139-144 (2020).
- Steiner, E., Schlagnitweit, J., Lundström, P., Petzold, K. Capturing excited states in the fast-intermediate exchange limit in biological systems using 1H spectroscopy. Angewandte Chemie International Edition. 55 (51), 15869-15872 (2016).
- Moschen, T., et al. Ligand-detected relaxation dispersion NMR spectroscopy: dynamics of preQ1-RNA binding. Angewandte Chemie International Edition. 54 (2), 560-563 (2015).
- LeBlanc, R. M., Longhini, A. P., Tugarinov, V., Dayie, T. K. NMR probing of invisible excited states using selectively labeled RNAs. Journal of Biomolecular NMR. 71 (3), 165-172 (2018).
- Strebitzer, E., Nußbaumer, F., Kremser, J., Tollinger, M., Kreutz, C. Studying sparsely populated conformational states in RNA combining chemical synthesis and solution NMR spectroscopy. Methods. 1148, 39-47 (2018).
- Rangadurai, A., Shi, H., Al-Hashimi, H. M. Extending the sensitivity of CEST NMR spectroscopy to micro-to-millisecond dynamics in nucleic acids using high-power radio-frequency fields. Angewandte Chemie International Edition. 59 (28), 11262-11266 (2020).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. Using relaxation dispersion NMR spectroscopy to determine structures of excited, invisible protein states. Journal of Biomolecular NMR. 41 (3), 113-120 (2008).
- Lundström, P., Akke, M. Off-resonance rotating-frame amide proton spin relaxation experiments measuring microsecond chemical exchange in proteins. Journal of Biomolecular NMR. 32 (2), 163-173 (2005).
- Lee, J., Dethoff, E. A., Al-Hashimi, H. M. Invisible RNA state dynamically couples distant motifs. Proceedings of the National Academy of Sciences of the United States of America. 111 (26), 9485-9490 (2014).
- Schnieders, R., Keyhani, S., Schwalbe, H., Fürtig, B. More than proton detection- new avenues for NMR spectroscopy of RNA. Chemistry. 26 (1), 102-113 (2020).
- Fürtig, B., Richter, C., Wöhnert, J., Schwalbe, H. NMR spectroscopy of RNA. Chembiochem. 4 (10), 936-962 (2003).
- Schlagnitweit, J., Steiner, E., Karlsson, H., Petzold, K. Efficient detection of structure and dynamics in unlabeled RNAs: The SELOPE approach. Chemistry. 24 (23), 6067-6070 (2018).
- Feyrer, H., Munteanu, R., Baronti, L., Petzold, K. One-pot production of RNA in high yield and purity through cleaving tandem transcripts. Molecules. 25 (5), 1142(2020).
- Baronti, L., Karlsson, H., Marušič, M., Petzold, K. A guide to large-scale RNA sample preparation. Analytical and Bioanalytical Chemistry. 410 (14), 3239-3252 (2018).
- Brunelle, J. L., Green, R. In vitro transcription from plasmid or PCR-amplified DNA. Methods in Enzymology. 530, 101-114 (2013).
- Borkotoky, S., Murali, A. The highly efficient T7 RNA polymerase: A wonder macromolecule in biological realm. International Journal of Biological Macromolecules. 118, Pt A 49-56 (2018).
- Arnaud-Barbe, N., Cheynet-Sauvion, V., Oriol, G., Mandrand, B., Mallet, F. Transcription of RNA templates by T7 RNA polymerase. Nucleic Acids Research. 26 (15), 3550-3554 (1998).
- Guillerez, J., Lopez, P. J., Proux, F., Launay, H., Dreyfus, M. A mutation in T7 RNA polymerase that facilitates promoter clearance. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5958-5963 (2005).
- Kuzmine, I., Gottlieb, P. A., Martin, C. T. Binding of the priming nucleotide in the initiation of transcription by T7 RNA polymerase. Journal of Biological Chemistry. 278 (5), 2819-2823 (2003).
- Gholamalipour, Y., Karunanayake Mudiyanselage, A., Martin, C. T. 3' end additions by T7 RNA polymerase are RNA self-templated, distributive and diverse in character - RNA-Seq analyses. Nucleic Acids Research. 46 (18), 9253-9263 (2018).
- Inoue, H., Hayase, Y., Iwai, S., Ohtsuka, E. Sequence-dependent hydrolysis of RNA using modified oligonucleotide splints and RNase H. FEBS Letters. 215 (2), 327-330 (1987).
- Wang, X., Li, C., Gao, X., Wang, J., Liang, X. Preparation of small RNAs using rolling circle transcription and site-specific RNA disconnection. Molecular Therapy - Nucleic Acids. 4, 215(2015).
- Karlsson, H., Baronti, L., Petzold, K. A robust and versatile method for production and purification of large-scale RNA samples for structural biology. RNA. 26 (8), 1023-1037 (2020).
- Hartmann, S. R., Hahn, E. L. Nuclear double resonance in the rotating frame. Physical Review. 128 (5), 2042-2053 (1962).
- Chiarparin, E., Pelupessy, I., Bodenhausen, G. Selective cross-polarization in solution state NMR. Molecular Physics. 95 (5), 759-767 (1998).
- Korzhnev, D. M., Orekhov, V. Y., Kay, L. E. Off-resonance R 1ρ NMR studies of exchange dynamics in proteins with low spin-lock fields: an application to a Fyn SH3 domain. Journal of the American Chemical Society. 127 (2), 713-721 (2005).
- Hansen, A. L., Nikolova, E. N., Casiano-Negroni, A., Al-Hashimi, H. M. Extending the range of microsecond-to-millisecond chemical exchange detected in labeled and unlabeled nucleic acids by selective carbon R 1ρ NMR spectroscopy. Journal of the American Chemical Society. 131 (11), 3818-3819 (2009).
- Duss, O., Maris, C., von Schroetter, C., Allain, F. H. -T. A fast, efficient and sequence-independent method for flexible multiple segmental isotope labeling of RNA using ribozyme and RNase H cleavage. Nucleic Acids Research. 38 (20), 188(2010).
- Krähenbühl, B., Lukavsky, P., Wider, G. Strategy for automated NMR resonance assignment of RNA: application to 48-nucleotide K10. Journal of Biomolecular NMR. 59 (4), 231-240 (2014).
- LeBlanc, R. M., Longhini, A. P., Le Grice, S. F. J., Johnson, B. A., Dayie, T. K. Combining asymmetric 13C-labeling and isotopic filter/edit NOESY: a novel strategy for rapid and logical RNA resonance assignment. Nucleic Acids Research. 45 (16), 146(2017).
- Parisien, M., Major, F. The MC-Fold and MC-Sym pipeline infers RNA structure from sequence data. Nature. 452 (7183), 51-55 (2008).
- Fürtig, B., Richter, C., Bermel, W., Schwalbe, H. New NMR experiments for RNA nucleobase resonance assignment and chemical shift analysis of an RNA UUCG tetraloop. Journal of Biomolecular NMR. 28 (1), 69-79 (2004).
- Keyhani, S., Goldau, T., Blümler, A., Heckel, A., Schwalbe, H. Chemo-enzymatic synthesis of position-specifically modified RNA for biophysical studies including light control and NMR spectroscopy. Angewandte Chemie International Edition. 57 (37), 12017-12021 (2018).
- Marchanka, A., Kreutz, C., Carlomagno, T. Isotope labeling for studying RNA by solid-state NMR spectroscopy. Journal of Biomolecular NMR. 71, 151-164 (2018).
- Becette, O., Olenginski, L. T., Dayie, T. K. Solid-phase chemical synthesis of stable isotope-labeled RNA to aid structure and dynamics studies by NMR spectroscopy. Molecules. 24 (19), 3476(2019).
- Zhang, X., Li, M., Liu, Y. Optimization and characterization of position-selective labelling of RNA (PLOR) for diverse RNA and DNA sequences. RNA Biology. 17 (7), 1009-1017 (2020).
- Roh, J. H., et al. Effects of preferential counterion interactions on the specificity of RNA folding. The Journal of Physical Chemistry Letters. 9 (19), 5726-5732 (2018).
- Juen, M. A., et al. Excited states of nucleic acids probed by proton relaxation dispersion NMR spectroscopy. Angewandte Chemie German Edition. 55 (39), 12008-12012 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır