Method Article
Aspectos práticos da preparação e configuração da amostra de 1H R1ρ Experimentos de dispersão de relaxamento do RNA
Neste Artigo
Resumo
Apresentamos um protocolo para medir a dinâmica micro a milissegundo em 13C/15N rotulado e sem rótulo RNA com 1H R1ρ de dispersão nuclear de ressonância magnética (NMR). O foco deste protocolo está na preparação e configuração de amostras de alta pureza de experimentos de RMN.
Resumo
O RNA é uma biomolécula altamente flexível, onde as mudanças nas estruturas desempenham papéis cruciais nas funções que as moléculas de RNA executam como mensageiros e moduladores celulares. Embora esses estados dinâmicos permaneçam ocultos à maioria dos métodos estruturais, a espectroscopia de dispersão de relaxamento R1ρ (RD) permite o estudo da dinâmica conformacional no regime micro-a milissegundo na resolução atômica. O uso de 1H como núcleo observado expande ainda mais o regime de tempo coberto e dá acesso direto a ligações de hidrogênio e emparelhamento de base.
Os passos desafiadores em tal estudo são a preparação de amostras de alta pureza e de alto rendimento, potencialmente com 13C e 15N rotulados, bem como a configuração de experimentos e a montagem de dados para extrair população, taxa de câmbio e estrutura secundária do estado anteriormente invisível. Este protocolo fornece etapas práticas cruciais na preparação da amostra para garantir a preparação de uma amostra de RNA adequada e a configuração de experimentos de 1H R1ρ com amostras de RNA isototicamente rotuladas e sem rótulo.
Introdução
As RNAs realizam uma infinidade de funções regulatórias1,catalítica2e estrutural3 na célula, muitas das quais estão correlacionadas a uma estrutura molecular flexível e mudanças intrincadas dessas estruturas4,5,6,7. Estados pouco populosos permanecem invisíveis para a maioria dos métodos de determinação da estrutura ou não permitem o estudo desses estados ocultos em alta resolução atômica. A espectroscopia de ressonância magnética nuclear (NMR) combina ambos os aspectos, fornecendo acesso a núcleos atômicos individuais, bem como oferecendo uma grande caixa de ferramentas de experimentos visando dinâmicas através de todos os regimes de tempo8. Os experimentos de RD NMR fornecem acesso à troca conformacional na escala de tempo intermediária, onde podem ser esperadas alterações nos padrões de emparelhamento de base e rearranjos estruturais locais5,9,10,11,12,13,14. Os experimentos RD são realizados como medidas longas R2 na forma de um trem de pulso Carr-Purcell-Meiboom-Gill15 ou como medidas de relaxamento no quadro rotativo, chamado R1ρ RD experimentos16.
Embora ambos possam ser usados para extrair diferença populacional e cambial e de mudança química para o estado menor, os experimentos R1ρ RD também dão o sinal da diferença de mudança química do estado animado. Isso permite uma inferência sobre a estrutura secundária, que se correlaciona fortemente com a mudança química nas estruturas de RNA17. A mudança química é um bom indicador de helicidade no caso de prótons aromáticos e carbonos nas nucleobases, de parceiros de emparelhamento de base para prótons imino, e de discos de açúcar nos átomos C4' eC1' 18,19. Deve-se notar que recentemente um experimento de transferência de saturação de câmbio químico (CEST) usando maior potência de bloqueio de giro (SL), mudando assim a aplicabilidade do experimento CEST para escalas de tempo de câmbio mais rápidas, foi publicado como uma alternativa ao experimento R1ρ RD para sistemas com um estado animado.
Embora os isótopos de 13C e 15N tenham sido frequentemente usados para acessar a troca estrutural, trabalhos recentes deste laboratório usaram prótons aromáticos e iminos como sondas para troca conformacional9,10. O uso de 1H como núcleo observado traz várias vantagens, por exemplo, acesso à troca em escalas de tempo mais rápidas e lentas, maior sensibilidade e tempos de medição mais curtos. Isso é facilitado ainda pela abordagem SELective Optimized Proton Experiment (SELOPE), proporcionando acesso a prótons aromáticos através da despovoamento do espectro unidimensional (1D) utilizando acoplamentos escalares homonucleares, em vez de uma transferência de magnetização heteronuclear, e eliminando a necessidade de rótulos de isótopos20. Este protocolo aborda a medição em experimentos RD de 1H R1ρ de amostras uniformemente rotuladase sem rótulosN. Portanto, este artigo apresenta um método de preparação amostral que foi considerado o mais versátil para diferentes necessidades de preparação amostral21 e discute alternativas na última seção deste artigo (Figura 1).
Neste ponto, o leitor deve observar que outras técnicas de preparação da amostra são aceitáveis para experimentos rd de 1H R1ρ, e que outros métodos de análise estrutural e funcional podem ser realizados com as amostras sintetizadas com a técnica apresentada. 1 Os experimentos H R1ρ RD requerem altas concentrações de RNA (idealmente >1 mM) bem como alta homogeneidade, tanto no comprimento do RNA quanto na conformação estrutural para garantir uma caracterização confiável da dinâmica molecular. A transcrição in vitro (IVT) é o método de escolha para muitos pesquisadores produzirem amostras de RNA com 13C/15N rotulados devido à disponibilidade de triptosfatos nucleosídeos rotulados (NTPs) e incorporação fácil na reação enzimática22. No entanto, a polimerase T7 RNA amplamente utilizada (T7RNAP)23,24,25 sofre de baixa homogeneidade de 5' no caso de certas sequências de iniciação26,27 e muitas vezes também 3' homogeneidade durante o escoamento de transcrição28. A purificação das espécies de RNA alvo torna-se mais cara e trabalhosa devido à necessidade de grandes quantidades de ~200 nmol. O método aqui utilizado foi apresentado anteriormente onde as vantagens foram discutidas em geral21. Resumindo, ele resolve os problemas descritos transcrevendo uma transcrição em tandem maior que é então especificamente cortada por Escherichia coli RNase H, guiada por um oligonucleotídeoquimérico 29,30 (ver Figura 2 para detalhes).
A incorporação de uma sequência espaçadora nas extremidades de 5' e 3' da transcrição tandem permite o uso de uma sequência de iniciação de alto rendimento e remoção de saliências terminais próximas ao local de linearização do modelo plasmídeo, respectivamente (Figura 2B). O método mostrou-se para melhorar significativamente os rendimentos, ao mesmo tempo em que reduz o custo e a mão-de-obra, com a ressalva de uma síntese de modelo mais complexa e a necessidade de uma enzima adicional e oligonucleotídeo. A alta especificidade do decote RNase H facilita a purificação devido à falta de espécies de RNA em uma faixa de tamanho semelhante. O presente protocolo utiliza uma etapa de cromatografia líquida de alto desempenho (HPLC) de intercâmbio de íons que foi publicada por este laboratório recentemente31,embora outros métodos sejam alternativas possíveis. 1 H R1ρ RD pode, em geral, ser adquirido em amostras rotuladas ou não com duas respectivas sequências de pulso, o experimento "rotulado" 1H R1ρ heteronuclear single correlação (HSQC)-based experimento com uma dimensão indireta de 13C10 e o "unlabeled" 1H R1ρPE-based experimento com uma dimensão indireta 20H .
Esses experimentos bidimensionais (2D) podem servir como uma primeira verificação, independentemente de a dinâmica na escala de tempo R1ρ estar presente na amostra. Uma visão geral do RD para todos os picos resolvidos nos espectros pode ser obtida, e picos de interesse para uma análise rd mais completa podem ser identificados. Isso significa que mesmo amostras não rotuladas podem ser verificadas antes da decisão de produzir uma amostra mais cara e rotulada. Uma vez que um pico com contribuição de troca conformacional é selecionado para ser estudado mais detalhadamente, é melhor mudar para as versões 1D dos experimentos acima (se o pico ainda pode ser resolvido) para realizar os chamados experimentos fora de ressonância. Para a versão rotulada, a transferência do HSQC para 13C é substituída por um passo seletivo de polarização cruzada heteronuclear (HCP) como usado em experimentos 13C R1ρ 32,33,34,35, enquanto no caso do experimento SELOPE, o experimento é simplesmente executado como um 1D, o que é especialmente útil para sinais H8 e H2 que estão deitados na diagonal no 2D de qualquer maneira. Um critério sobre qual sequência de uso, desde que tanto, uma amostra rotulada e não rotulada estejam disponíveis, é o quão bem isolado o pico de interesse está nos dois experimentos.
Em geral, recomenda-se o experimento SELOPE para amostras de RNA de até 50 nucleotídeos. Para rnas maiores, a sobreposição será maior; no entanto, nucleotídeos estruturalmente interessantes frequentemente aparecem em regiões de mudança química que são menos sobrepostas e ainda podem ser acessíveis em RNAs ainda maiores. Outro argumento seria que, em amostras sem rótulo, nenhum acoplamento J ocorre entre 1H e 12C. No entanto, como o poder mínimo de bloqueio de rotação é definido pela potência mínima usada para desacoplar esses dois giros (~1 kHz) no experimento rotulado, o experimento sem rótulo permite o uso de uma gama mais ampla de pontos fortes de bloqueio de giro (SL) e, portanto, acesso a uma escala de tempo mais ampla de troca. Esses experimentos de ressonância extra fornecem informações adicionais para kex, como população do estado animado (conformr alternativo), pES, bem como informações de mudança química muito valiosas na forma de Δω (a diferença de mudança química do estado terrestre e do estado animado).
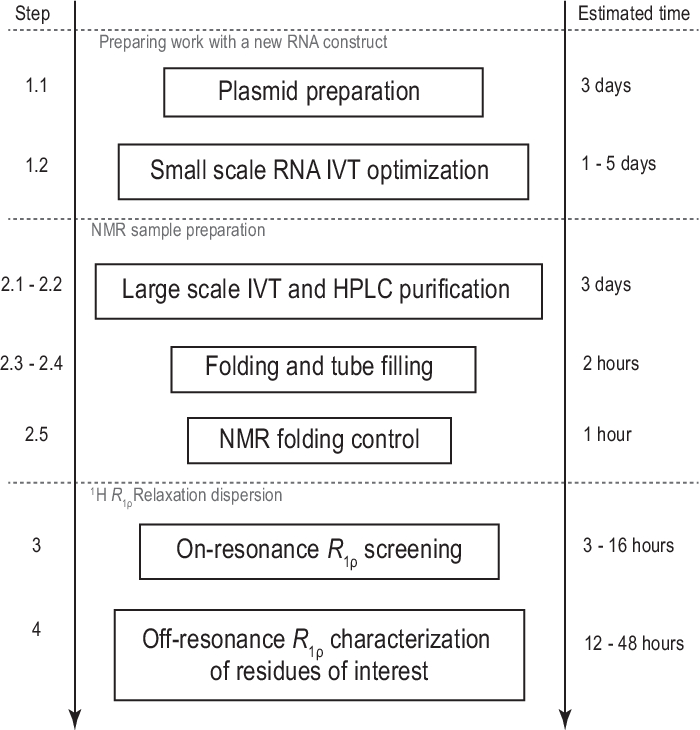
Figura 1: Fluxo de trabalho do protocolo apresentado. Preparação antes da produção real de amostras em larga escala, consistindo na preparação do modelo e confirmação da transcrição in vitro bem sucedida e do decote RNase H. Produção em larga escala, incluindo purificação hplc, enchimento do tubo NMR e confirmação de dobramento de RNA. Em caso de síntese rotulada por isótopos, deve ser realizada uma purificação sem rótulo para otimização de gradiente no mesmo dia. Caracterização nMR da dinâmica conformacional com experimentos R1ρ. Cada etapa pode ser realizada de forma independente, por exemplo,a análise RD de 1H R1ρ pode ser aplicada a qualquer amostra de RNA adequada produzida com outro método. Abreviaturas: IVT = transcrição in vitro; HPLC = cromatografia líquida de alto desempenho; NMR = ressonância magnética nuclear; RD = dispersão de relaxamento. Clique aqui para ver uma versão maior desta figura.
O objetivo deste protocolo é fornecer detalhes práticos e parâmetros críticos para o estudo da dinâmica conformacional com dispersão de relaxamento de 1H R1ρ em moléculas de grampos de RNA. Depois de fornecer um protocolo detalhado do design, síntese e purificação hplc de troca de íons de um RNA de destino que pode ser realizado usando todos, alguns ou nenhum NTPs como versões com 13C/15N, foi descrito o fluxo de trabalho de finalizar a amostra NMR e confirmar a troca conformacional com espectroscopia NMR. Finalmente, são descritos os detalhes para a configuração de experimentos 1H R1ρ RD em um espectrômetro Bruker NMR(Figura 1). O protocolo dá cada passo para configurar a versão 1D para amostras rotuladas e comentários adicionais e uma tabela para ajustar para a configuração da versão SELOPE (Tabela 2). Após o protocolo, são discutidas etapas críticas e rotas alternativas para a preparação da amostra e a configuração RD de1 H R1ρ.
Protocolo
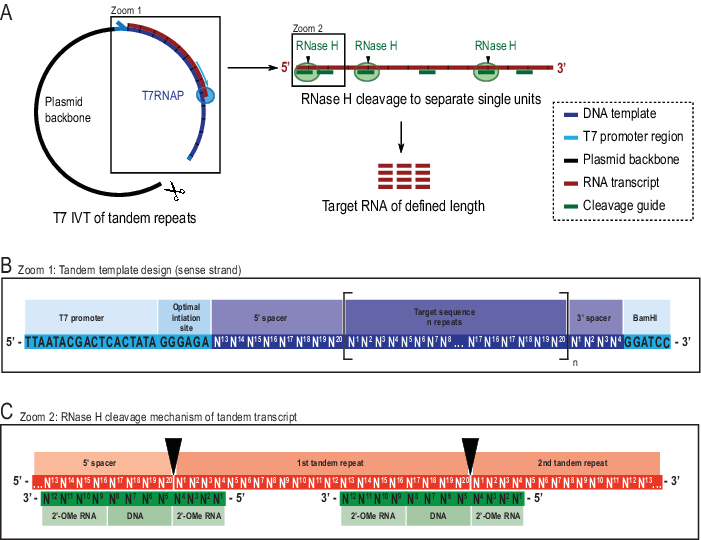
Figura 2: Representação esquemática do protocolo ivt tandem relatado. (A) Transcrição tandem de um modelo plasmídeo linearizado com T7RNAP (esquerda) e sucessivas decotes por RNAse H da transcrição para alcançar o comprimento-alvo RNA, dirigido por um guia de DNA quimérico (à direita). (B) Esquema detalhado do modelo tandem começando com o promotor T7RNAP viral, uma sequência de iniciação. A sequência de destino (azul escuro, exemplo aqui tem 20 nt de comprimento) é repetida "n" vezes. As repetições ladeadas por uma sequência espaçadora de 5′ e 3′ constituídas pelos últimos oito e quatro primeiros nucleotídeos, respectivamente, para permitir a remoção das sequências de iniciação e restrição da primeira e última unidade de repetição. (C) Hibridização da transcrição tandem (vermelho) e das guias de decote quimérico (verde). RNase H aperta o RNA oposto ao DNA 5′ final. Os flancos RNA 2'-OMe aumentam a especificidade aumentando a afinidade vinculante do guia de decote ao RNA alvo. Este número foi modificado a partir de 21. Abreviaturas: T7RNAP = T7 RNA polymerase. Clique aqui para ver uma versão maior desta figura.
1. Preparando o trabalho para uma nova construção de RNA
- Projeto e preparação plasmid
- Escreva a sequência de modelos em uma ferramenta de clonagem, por exemplo,Serial Cloner.
- Pegue a sequência de promotor T7 e adicione uma sequência de iniciação de alto rendimento (T7: 5'-TAATACGACTACTATA ^GGGAGA-3').
NOTA: A transcrição começará no nucleotídeo indicado com um caret (^). A sequência de iniciação GGGAGA é variável, mas fortemente dependente de sequência; portanto, recomenda-se o uso desta sequência. - Adicione os últimos 8 nucleotídeos (nt) da sequência de destino como um espaçador de 5''(5'S).
- Adicionar repetições da sequência de destino (TS).
- Adicione os quatro primeiros nucleotídeos como espaçador de 3' após as repetições (5'S).
- Adicione um site de restrição BamHI (RS) ou um site de restrição exclusivo semelhante.
NOTA: A sequência total mostrada será clonada ou prontamente ordenada em um plasmídeo bacteriano de alta cópia(por exemplo,pUC19): 5'-T7-5'S-(TS)n-3'S-RS-3'(Figura 2B). O número de repetições deve ser tão alto quanto o permitido pela síntese genética ( um máximo de 600 nt neste protocolo). - Amplie o plasmídeo em E. coli usando um kit comercial.
- Linearize o plasmídeo purificado a 20 ng/μL usando o local de restrição apropriado. A restrição de escala digere com BamHI em até 1 mL.
- Purifique o plasmídeo digerido, e confirme a linearização bem sucedida em um gel de 1% de agarose. Armazene o plasmídeo linearizado a -20 °C por vários meses.
- Design do guia de decote(Figura 2C)
- Escreva os últimos oito nucleotídeos da sequência de RNA alvo na direção de 5'-3', e adicione os quatro primeiros nucleotídeos da sequência de RNA alvo no final de 3' também na direção de 5'-3'.
- Gerar o complemento de DNA reverso dessa sequência
- Altere o primeiro e último quatro nucleotídeos para suas modificações de 2'-OMe adicionando um 'm' antes da letra nucleotídea.
NOTA: Para síntese, o mU é usado em vez de mT. - Ordene o oligo com purificação de desalização padrão.
NOTA: Verifique se o oligo gerado pode se ligar em outro lugar que não seja a conexão de duas sequências de RNA. A complementaridade total nos quatro núcleos centrais de DNA é necessária, enquanto as regiões de flanqueamento podem permitir uma incompatibilidade. Se necessário, estenda os flancos até 18 nt para gerar uma sequência de ligação única36.
- IVT de pequena escala
NOTA: Para o trabalho sem RNase, prepare todos os reagentes em condições estéreis e livres de RNase. Use reagente de descontaminação RNase (ver tabela de materiais) e 95% v/v de etanol para limpar superfícies de trabalho e pipetas antes de usar. Lave luvas com 95% de etanol e use roupas de manga comprida sem fiapos. Para minimizar a contaminação rnase, não respire sobre tubos abertos.- Preparar soluções de estoque de Tris-Cl (pH 8.0), dithiothreitol, MgCl2, espermidina e NTPs/GMP (não oferecido). Misture reagentes como mostrado na Tabela 1. Prepare uma mistura mestre desses reagentes com antecedência, antes da adição de enzimas ou ácidos nucleicos.
NOTA: Se usar reagentes congelados, misture-os completamente após o descongelamento. Os reagentes podem precipitar-se se misturados em concentrações muito altas, por isso é fortemente recomendado seguir a ordem na Tabela 1. - Adicione na seguinte ordem: plasmid, guia de decote, fosfatismo inorgânico (IPPase), RNase H, T7RNAP. Como a atividade enzimática pode variar para enzimas produzidas internamente, teste várias concentrações antes de selecionar a melhor.
NOTA: Inclua um controle negativo para a reação do decote, por exemplo, sem RNase H, para atribuir uma faixa de alvo ausente ao decote RNase H falho e não à transcrição mal sucedida. - Incubar a reação a 37 °C por 1h e confirmar a reação em uma eletroforese de gel de poliacrilamida de desnaturação (PAGE) (Figura 3A). Diluir a amostra 10 vezes na solução de carregamento e carregar 1 μL no gel.
NOTA: Mistura de gel: 8 M de ureia, 20% acrilamida (19:1 acrilamida:bisacrilamida) em 1x TBE. Solução de carregamento: 5 mM ácido tetraáctico de etilenodiamina (EDTA), 300 μM azul bromofenol em formamida. As reações de decote RNase H não podem ser completas após 1h, já que o novo RNA é produzido constantemente. Neste ponto, procure uma faixa-alvo clara e a ausência de uma espécie de peso molecular semelhante (por exemplo,±3 produtos nucleotídeos (nt).
- Preparar soluções de estoque de Tris-Cl (pH 8.0), dithiothreitol, MgCl2, espermidina e NTPs/GMP (não oferecido). Misture reagentes como mostrado na Tabela 1. Prepare uma mistura mestre desses reagentes com antecedência, antes da adição de enzimas ou ácidos nucleicos.
| Reagente | Concentração de estoque | Quantidade de pequena escala (μL) |
| H2O | - | 24 |
| Tris | 1 M | 5 |
| MgCl2 | 1 M | 0.5 |
| DTT | 1 M | 0.5 |
| Esperdina | 250 mM | 5 |
| GMP | 100 mM | 2.5 |
| ATP | 100 mM | 1.5 |
| GTP | 100 mM | 1.5 |
| UTP | 100 mM | 1.5 |
| CTP | 100 mM | 1.5 |
| Plasmídeo | 20 ng/μL | 5 |
| Guia de decote | 100 μM | 10 |
| iPPase | 10 mg/mL | 0.5 |
| RNase H | 10 μg/mL | 2 |
| Polimerase T7 RNA | 5 mg/mL | 2 |
Tabela 1: Tabela reagente para TANDEM IVT e decote RNase H simultâneo. As concentrações de estoque podem ser adaptadas à conveniênciadousuário. Se o decote RNase H deve ser realizado após t7 IVT, adicione guia de decote e RNase H após a inativação de calor de T7RNAP. As quantidades utilizadas escalam linearmente com escala de reação. Abreviaturas: T7RNAP = T7 RNA polymerase; IVT = transcrição in vitro.
2. Preparação da amostra de NMR
- Dimensione a reação ao volume desejado (tipicamente 10 mL) e execute a reação durante a noite. Teste para conclusão de reação no dia seguinte com um gel PAGE desnaturado(Figura 3A).
NOTA: A reação incompleta do decote é mostrada por espécies de peso molecular mais altas acima da faixa-alvo.- Se o decote não foi bem sucedido ou completo, o RNA reanneal e o guia de decote no vaso de reação aquecendo a solução em um micro-ondas convencional a 450 W para 15 s.
- Esfrie a solução lentamente a 37 °C por 40 min. Use um bloco de aquecimento para volumes abaixo de 1 mL. Note a formação de novo precipitado.
- Adicione mais IPPase e RNase H, e incubar por mais 1-3 h a 37 °C. Confirme a conclusão da reação do decote com a DEsnaturação PAGE.
- Quando a reação de decote RNase H estiver concluída, sacie a reação adicionando EDTA a 50 mM de concentração final e vórtice completamente.
NOTA: A precipitação potencial do pirofosfato se dissolverá, e novas formas de precipitação de proteínas. - Filtre a solução através de um filtro de seringa de 0,2 μm e concentre-se em um volume injetável em um sistema HPLC, dependendo do tamanho do loop de injeção.
NOTA: O protocolo pode ser pausado aqui congelando a amostra a -20 °C.
- Purificação hplc em larga escala
- Prepare os buffers de troca de íons A e B dentro de uma semana de uso. Filtrar e desgas os buffers.
NOTA: Tampão A: acetato de sódio de 20 mM; Perclorato de sódio de 20 mM, pH 6,5. Tampão B: acetato de sódio de 20 mM; Perclorato de sódio de 600 mM, pH 6,5. - Equilibre a coluna com 100% de buffer B seguido de 100% tampão A para pelo menos 2 volumes de coluna a 75 °C.
- Prepare a sequência HPLC(Figura 3B) a uma vazão de 5,5 mL/min. Use a seguinte sequência para purificação de um RNA de tamanho entre 20 e 30 nt: 0-7 min: 0% B; 7-16 min: gradiente 0-20% B; 16-46 min: elução, tipicamente com gradiente de 20-30% B (otimizar de acordo com as necessidades); 46-62 min: 100% B; 62-73 min: 0% B.
NOTA: Uma mudança na vazão de 5,5 para 8 mL/min não influenciou a separação neste protocolo. - Otimize o gradiente de elução pela injeção de um equivalente a 1 mL de reação de transcrição (sem rótulo) de cada vez.
NOTA: Para maiores detalhes e discussão, consulte Karlsson et al.31 e Feyrer et al.21. - Teste as frações coletadas em uma PÁGINA de desnaturação. Se o pico de elução principal estiver bem isolado e contiver o RNA de destino puro, aumente a purificação para um equivalente a 10 mL de reação de transcrição.
- Colete as frações de juros, concentre-se e troque o buffer com o buffer NMR. Use uma unidade de filtro ultracentrifugal (ver a Tabela de Materiais) para volumes acima de 50 mL.
NOTA: Tampão de RMN: fosfato de sódio de 15 mM; Cloreto de sódio de 25 mM; 0,1 mM EDTA, pH 6.5. Para minimizar a perda da adesão do RNA às paredes dos tubos de plástico, lave todos os tubos de coleta com 1 mL de água, vórtice e centrífuga para coletar todo o líquido. - Determine a concentração através de espectroscopia ultravioleta. Calcule o rendimento da reação de acordo com Feyrer et al21.
NOTA: A concentração de uma amostra de RMN para experimentos rd não deve ser inferior a 130 nmol, o que corresponde a 500 μM em um volume amostral de 250 μL usando tubos de RN(Tabela de Materiais).
- Prepare os buffers de troca de íons A e B dentro de uma semana de uso. Filtrar e desgas os buffers.
- Dobramento de uma amostra de RNA
- Diluir e alíquotar a amostra de um volume de ~10 mL em 1 mL por tubo.
- Aqueça as alíquotas de RNA a 95 °C por 5 minutos.
- Esfrie as amostras colocando-as no gelo ou em uma mistura de água-gelo-sal e incubar por 30 minutos.
- Possua amostras e concentre-se em ~250 μL em uma unidade de filtro centrífugo de 2 mL.
- Enchimento de um tubo NMR
- Limpe o tubo NMR no limpador de tubos NMR lavando com água abundante, reagente de descontaminação RNase, água, 95% de etanol (EtOH) e água novamente. Deixe secar.
- Limpe o êmbolo enxaguando com água e limpando com reagente de descontaminação RNase e 95% EtOH usando um lenço sem fiapos. Deixe secar.
- Adicione 10% (v/v) de D2O à amostra NMR.
- Encha a amostra de RNA no tubo NMR usando uma grande ponta de pipeta. Deixe o líquido fluir ao longo da lateral da parede do tubo.
- Insira o êmbolo e remova as bolhas de ar empurrando o êmbolo para baixo junto com um movimento de torção rápida.
- Puxe o êmbolo lentamente sem criar novas bolhas de ar e fixá-lo com filme de cera de parafina.
- Confirme a dobra por NMR.
NOTA: Neste ponto, é necessário realizar pelo menos uma atribuição de ressonância parcial para confirmar a estrutura secundária da amostra de RNA e identificar regiões de interesse para o estudo da dinâmica conformacional. Uma descrição exaustiva sobre a atribuição de ressonância RNA excederia este protocolo, portanto nos referimos à literatura bem estabelecida neste ponto19,37,38. Um ensaio de mudança de mobilidade eletroforética (EMSA) pode ser um indicador útil de dobramento de RNA e servir como dados complementares para experimentos de RN.- Compare os seguintes espectros da amostra para os quais são realizados experimentos de 1H R1ρ RD com a amostra de referência devidamente dobrada (Figura 4): 1H 1D, especialmente a região imino 10–15 ppm; Aromático 1H,13C-HSQC; 1 H,1H-SELOPE (opcional).
NOTA: Uma impressão digital aromática também é necessária, mesmo em caso de concordância entre sinais iminos, pois a formação de dimer muitas vezes mostra os mesmos sinais iminos ou similares como um grampo de cabelo de RNA. O experimento SELOPE pode substituir um 1H,13C-HSQC por impressão digital aromática, já que experimentos heteronucleares em amostras não rotuladas são muito demorados. - Use o loop UUCG como referência de impressão digital (se estiver presente).
- Realize esta comparação todas as vezesantes que os experimentos 1 H R1ρ RD sejam registrados.
- Compare os seguintes espectros da amostra para os quais são realizados experimentos de 1H R1ρ RD com a amostra de referência devidamente dobrada (Figura 4): 1H 1D, especialmente a região imino 10–15 ppm; Aromático 1H,13C-HSQC; 1 H,1H-SELOPE (opcional).
3. 1H R1ρ Dispersão de relaxamento—naressonância (versão 1D rotulada)
NOTA: As etapas abaixo descrevem a configuração de experimentos RD para uma amostra rotulada usando a versão 1D da sequência de pulso RD baseada em HSQC. Siga os mesmos passos para a sequência 1D baseada em SELOPE para amostras sem rótulo. Uma visão geral dos nomes dos parâmetros e configurações para ambos os casos são mostradas na Tabela 2. O foco nas versões 1D é porque elas são mais práticas para medições fora da ressonância, e a configuração das versões 2D dos experimentos baseados em SELOPE e HSQC foram discutidas detalhadamente por Schlagnitweit et al.20 e Steiner et al.10, respectivamente.
- Determine a potência de 1H para um pulso rígido de 90° (P1).
- Opção A: Use o comando pulsecal Bruker.
- Opção B: Em um experimento zg, determine o pulso de 360° medindo uma curva de noz no nível de energia de pulso rígido de prótons no pico da água.
NOTA: O comprimento do pulso de 90° é um quarto da duração onde o sinal zero é observado (se uma curva de noz completa é medida, então é o segundo zero; porém, na prática, apenas a região ao redor do valor esperado para o 360° é amostrada).
- Execute um espectro 1H 1D zgesgp.f2f3dec usando o comprimento do pulso determinado na etapa 3.1 para confirmar o RNA dobrando antes de cada medição R1ρ.
NOTA: Se os experimentos de 1H SL forem executados pela primeira vez, verifique se a potência SL calculada corresponde à potência fornecida à amostra calibrando a potência SL para cada largura de banda desejada. As etapas detalhadas de calibração são descritas em Steiner et al.10. - Crie um conjunto de dados de 1 H R1ρ para o conjunto de dados rotulado e defina parâmetros-chave.
- Criar um novo conjunto de dados; idealmente baseado em um conjunto de dados H-13 C aromáticos HSQC de 1H-13C, usado em amostras de RNA totalmente rotuladas para atribuição de RNA.
NOTA: Isso garantirá que 13C, bem como 15N de potência e energia de dissociação já estejam configurados. - Defina os parâmetros gerais de acordo com a primeira parte da Tabela 2.
- Defina parâmetros específicos do RD de acordo com a segunda parte da Tabela 2.
- Defina 1H de potência SL ao menor valor (1,2 kHz) para testes.
- Gere uma lista vd de teste com apenas uma entrada, 0 ms, (para otimizar a lista vd, conforme descrito na etapa 3.4), definir TDF1 para 1 e atualizar D30.
- Execute um espectro de teste com essas configurações.
- Criar um novo conjunto de dados; idealmente baseado em um conjunto de dados H-13 C aromáticos HSQC de 1H-13C, usado em amostras de RNA totalmente rotuladas para atribuição de RNA.
- Otimize a lista vd (lista de comprimentos SL a serem usados).
- Execute o experimento com uma lista de vds de teste (por exemplo,seis entradas: 0 m, 5 m, 10 m, 20 m, 30 m, 40 m; embaralhar esses valores para evitar erros sistemáticos devido ao aquecimento).
- Atualização D30 e TDF1 em conformidade (neste exemplo, D30 = 42m e TDF1 = 6).
- Intensidade do gráfico do pico vs. comprimento SL. Identifique o comprimento de SL no qual a intensidade do pico original diminui para 1/3.
- Criar a lista vd final a ser usada no experimento, levando em consideração o seguinte: determinar o comprimento SL mais longo descrito na etapa anterior; evitar o uso de uma lista com ordem decrescente ou ascendente; e adicionar algumas duplicatas para estudos estatísticos. Lembre-se de atualizar D30 e TDF1 toda vez que houver alterações na lista vd.
NOTA: O experimento é executado com diferentes comprimentos de SL, conforme dado na lista vd de forma pseudo-2D. - Selecione o número de varreduras para que o pico mais fraco da lista tenha uma relação sinal-ruído (SINO) de pelo menos 10.
NOTA: Embora a lista vd tenha sido otimizada para uma potência SL baixa (1,2 kHz), esta lista vd também deve ser testada na maior potência SL a ser usada (por exemplo,15 kHz). Isso porque a decadência será muito mais lenta na alta potência de SL para picos com contribuição significativa kEX. Portanto, uma decadência suficiente também deve ser verificada com alta potência de SL.
| Descrição do parâmetro | Nome do parâmetro na sequência de pulso | |
| 1D rotulado | 1D SELOPE | |
| programa de pulso para 1Ds de ressonância on-resonance | 1HR1rho_HCP_onres1D.es | 1HR1r_HH_onres1D.js |
| 1 Frequências portadoras de H (ppm) | O1P = ressonância hídrica em ppm | O1P = mudança química de pico de juros (ppm) |
| CNST28 = mudança química de pico de juros (ppm) | CNST29 = ressonância hídrica em ppm | |
| 1 Pulso rígido de 90º | P1 @ PL1 (conforme calibrado em 3.1.1) | P1 @ PL1 (conforme calibrado em 3.1.1) |
| Pulsos e poderes moldados para supressão de água | P25 = 1000 us @ sp3 | P12 = 2000 us @ sp1 |
| (Watergate) | (escultura de excitação) | |
| 13 C frequência portadora, em ressonância com mudança química de 13C de pico de interesse | O2P | – |
| 15 N frequência portadora, mudança química média de 15N para desacoplamento (como usado no HSQC aromático) | O3P | – |
| 13 Desacoplamento C/15N (configurado como no HSQC) | pcpd2, cpd2 | – |
| pcpd3, cpd3 | ||
| Transferência de HCP (por exemplo, p=1/J @ 100 Hz) | – | |
| pulso e comandos pulsef2 podem ser usados para determinar poderes de pulsos rígidos | ||
| Duração (definida para 1/J(1H-13C) de pico de juros) | P11 | |
| Potência 1H e Potência em 13C | SP1, SP12 | |
| Transferência SELOPE (d = 1/4J(H5-H6)) | – | D5 |
| Pulso seletivo (por exemplo, região aromática) para SELOPE (4000 us, Eburp) | – | P13 & SP4 |
| Parâmetros específicos de SL / RD: | ||
| 1 H SL power, obtido a partir de pulso duro calibrado (por exemplo, usando o comando de pulso). | Pl25 & CNST12 (1,2 – 15 kHz) | Pl24 (50 Hz – 15 kHz) |
| Lista de atraso variável para duração de SL (inicialmente 1 entrada, 0, otimização descrita abaixo de 3.1.3) | vdlist (~ 0 – 40 ms) | vdlist (~ 0 – 150 ms devido ao baixo R2 em amostras não rotuladas) |
| Número TDF1 de entradas na lista vd (inicialmente 1) | TDF1 | TDF1 |
| Compensação térmica: | ||
| D30 = maior valor na lista vd + 2ms | D30 | D30 |
| Compensação adicional de calor para uma gama muito ampla de SLs | PL25 | |
| Parâmetros específicos de ressonância: | ||
| programa de pulso para 1Ds fora de ressonância | 1HR1rho_HCP_offres1D.es | 1HR1r_HH_offres1D.js |
| Deslocamento para experimentos fora de ressonância | CNST30 | CNST30 |
Tabela 2: Visão geral dos parâmetros para configurar experimentos baseados em HCP 1D e 1D-SELOPE 1H R1ρ. Abreviaturas: 1D = unidimensional; HCP = polarização cruzada heteronuclear; SELOPE = Experimento de próton otimizado selectivo; ppm = partes por milhão; HSQC= correlação quântica de spin heteronuclear; SL = trava de giro; RD = dispersão de relaxamento
- Configuração e aquisição de experimentos on-resonance 1H R1ρ
- Copie o experimento da seção 3.4 em uma nova pasta em Topspin.
- Nesta pasta, configure experimentos em diferentes pontos fortes de SL, sempre alterando PL25 e CNST12. Determine o nível de alimentação correto para cada força SL usando o comando de pulso. Use pontos fortes SL variando de 1,2 a 15 kHz, com uma amostragem mais densa para forças SL mais baixas (ver Figura 5G para pontos fortes SL selecionados). Adicione cópias de alguns dos experimentos para ter duplicatas para alguns dos pontos fortes da SL.
- Executar esses experimentos.
- Análise de experimentos on-resonance 1H R1ρ
- No TopSpin, processe cada fatia de cada conjunto de dados pseudo-2D usando os mesmos parâmetros de processamento (por exemplo, ampliação da linha, fase) usando o comando xf2e divida o conjunto de dados em 1Ds usando o programa Bruker AU split2D.
- Obtenha intensidades de sinal e volumes para cada fatia 1D.
NOTA: Na prática, é melhor desconvolver o espectro para se livrar de contribuições de picos potencialmente sobrepostos e permite o uso do programa Bruker AU multidcon, que convenientemente resume as intensidades ou áreas dos picos de todas as fatias em um experimento no arquivo de texto decall.txt, que podem ser lidos facilmente com outros programas (scripts Python escritos internamente foram usados aqui, como descrito por Steiner et al.10) nas etapas 3.6.3 e 3.6.4. - Encaixe uma decadência mono exponencial para cada força SL para obter o valor R1ρ (ou on-resonance, R2+REX).
- Plote esses valores R2+REX (y) vs. Força SL (x) (Figura 5F,G).
NOTA: Se os valores forem significativamente mais elevados para baixos pontos fortes da SL e diminuírem com maior potência SL (como mostrado na Figura 5G),então o pico investigado mostra dispersão, e pode ser interessante realizar experimentos adicionais (fora da ressonância) para obter informações sobre a diferença populacional e de mudança química do estado animado vs. o estado terrestre.
4. 1H R1ρ Dispersão de relaxamento — fora da ressonância (versão 1D rotulada)
- Configuração e aquisição de experimentos off-resonance 1H R1ρ
- Em uma nova pasta topspin, configure experimentos com uma certa força SL (geralmente primeiro na menor resistência sl como a contribuição REX é mais alta lá, ver Figura 5G para uma seleção representativa de SLs fora de ressonância), mas com diferentes deslocamentos, cada vez mudando CNST30.
- Use compensações até ± (3 ou 4)*SL de força, com uma amostragem mais densa em torno de 0 offset, como pode ser visto na Figura 5H,I.
- Executar esses experimentos.
- Análise de experimentos fora de ressonância 1H R1ρ
- Use a mesma estratégia de processamento, como em 3.6.1-3.6.3, para determinar um valor de R1ρ para cada deslocamento.
- Plote esses valores vs. offset(Figura 5G).
NOTA: Uma assimetria nesta curva já pode indicar que informações de mudança química para o estado animado podem ser obtidas. A montagem e análise minuciosas usando equações bloch-McConnell ou Laguerre devem ser realizadas para obter informações sobre kEX, pES, bem como Δω10,20 (Figura 5G). Exemplos de conjuntos de dados, programas de pulso e macros para ambos os experimentos 1D podem ser encontrados no repositório Petzold Lab Github (https://github.com/PetzoldLab). Uma visão geral dos parâmetros é dada na Tabela 2.
Resultados
O protocolo para produção de RNA facilita a purificação através da geração de transcrições de alta pureza. A Figura 3A mostra os resultados de várias reações de decote de transcrições tandem, fornecendo reações bem sucedidas e mal sucedidas. A faixa 1 mostra o caso ideal de uma transcrição totalmente cortada com apenas traços fracos de produtos laterais. A pista 2a mostra decote incompleto, que pode ser resolvido re-annealing e a adição de mais RNase H (Lane 2b, passo 2.1.2). As construções RNA das pistas 1, 2a e 2b são as mesmas. A amostra na faixa 3 mostra decote mal sucedido. A solução de problemas nesta reação envolveria uma verificação da sequência do guia de decote, pureza do modelo de DNA e temperaturas de ressarcial. Potencialmente, o decote RNase H terá que ser realizado após T7 IVT, como mostrado para a amostra 2.
A amostra na faixa 4 mostra uma quantidade significativa de produtos laterais de decote, que são difíceis de remover via HPLC de troca de íons. A solução de problemas como essa amostra pode envolver (a) redução da temperatura, quantidade de RNase H ou tempo de reação, (b) reduzir o gradiente de elução e o volume de injeção e tentar separar as frações-alvo dos produtos laterais. Mais informações sobre como aumentar a resolução na purificação do HPLC de intercâmbio de íons foram discutidas por Karlsson et al.31. O HPLC separa o RNA alvo de ácidos nucleicos mais longos ou mais curtos e contaminantes de proteínas ou pequenas moléculas. A Figura 3B mostra o resultado ideal para a purificação HPLC de troca de íons. O gradiente de elução deve ser escolhido de tal forma que a espécie RNA alvo elutes pelo menos um volume de coluna (neste exemplo: 35 mL) após as próximas espécies menores e um volume de coluna antes da próxima espécie maior.
Espécies menores neste método incluem nucleotídeos únicos, produtos abortivos (8-12 nt), sequências espaçadoras de 3' e 5' (5-14 nt) e guia de decote (ácido nucleico quimérico de 12 nt), enquanto sequências mais longas são potencialmente impuros e o plasmídeo. Quando um pico de elução bem separado é alcançado, a purificação pode ser dimensionada até o equivalente a ~20 mL de reação IVT por injeção. A dobra correta de uma amostra de RNA é crucial para experimentos rd e deve ser confirmada antes de cada medição. A Figura 4 mostra um RNA de 22-mer com rótulo A antes que o protocolo de dobramento na etapa 2.4 (azul) seja aplicado, e a mesma amostra após a dobra correta tenha sido alcançada (vermelho). Uma previsão de estrutura secundária mc-fold(Figura 4C) propõe a estrutura de grampos apresentado com 4 pares de base resultando em 5 sinais imino.
Ambos os espectros na Figura 4A confirmam esses sinais previstos, embora com intensidades relativas ligeiramente diferentes, o que indica que alguma estrutura desdobrada (aqui, um dimer) pode ser problemática para avaliar com apenas 1H 1D espectros. Um espectro aromático de 1H,13C-HSQC (Figura 4B),no entanto, mostra apenas 3 dos sinais aromáticos para a amostra antes do protocolo de dobramento (azul), mas todos os 4 sinais para a amostra que foi dobrado de acordo com a etapa 2.4 (vermelho). A amostra mostrada em azul provavelmente formou um homodimer (estrutura proposta na Figura 4D) que resultaria nos mesmos sinais iminos que o grampo de cabelo. O sinal do A13H2 parece ampliado. Esses resultados ajudam a destacar a importância da confirmação dobrável com experimentos de impressão digital imino e aromática antes de cada experimento rd. As sequências de pulso 1H R1ρ descritas neste protocolo permitem a detecção de dinâmicas no regime de troca intermediária. Inicialmente é registrada uma curva de ressonância e, se houver dinâmica para um resíduo específico, uma dispersão é visível dentro dos valores R 2+REX obtidos, enquanto esta curva é plana para resíduos sem troca.
A Figura 5 mostra curvas representativas de ressonância obtidas para dois átomos H8 diferentes em um grampo de RNA sintético(Figura 5A),em que as experiências G6H8 trocam(Figura 5C),enquanto A4H8 não(Figura 5B). Como a troca é relativamente lenta nesta amostra (kEX = 292 ± 40 Hz), a vantagem do experimento SELOPE para alcançar baixos pontos fortes de SL foi explorada, e as duas curvas de ressonância foram registradas usando a versão 1D da sequência de pulso. A mesma sequência de pulso foi então usada para obter dados de ressonância para o resíduo mostrando dispersão no perfil de ressonância. A Figura 5D mostra os valores R1ρ obtidos versus deslocamento em que uma ligeira assimetria da curva já indica o sinal de Δω.
Isso se torna ainda mais evidente no lote R2+REX onde a contribuição R1 é removida(Figura 5E). A coluna direita da mesma figura mostra curvas representativas de ressonância obtidas para dois átomos H8 diferentes em um grampo de RNA sintético ligeiramente diferente com troca mais rápida, em que as experiências G6H8 trocam(Figura 5G),enquanto A4H8 não(Figura 5F). A taxa de câmbio mais rápida (kEX = 43.502 ± 38.478 Hz) permitiu o registro rd de todos os prótons aromáticos de uma só vez usando a versão SELOPE 2D para obter dados de ressonância contínua e externa (dados G6H8 exibidos na Figura 5H,I).
Identificadores gerais para resultados positivos e negativos
Resultados positivos no tandem IVT e RNase H decote podem ser identificados da seguinte forma: 1) A banda alvo é a banda mais forte no gel PAGE desnaturação. 2) Não há bandas fracas ou apenas fracas ao redor da banda principal. 3) Não existem ou apenas espécies de peso molecular mais fracas. 4) O cromatógrafo HPLC mostra um pico bem separado do RNA alvo. 5) Quando o pico principal é amostrado, apenas uma banda aparece em um gel PAGE desnaturado.
Resultados negativos no tandem IVT e RNase H decote presente da seguinte forma: 1) Não ou apenas uma faixa principal fraca é visível em um gel PAGE desnaturando. 2) Um padrão de espécies de alto peso molecular de RNA tandem repete é visível. 3) Embora a banda principal esteja presente, bandas de intensidade semelhante estão acima ou abaixo da faixa principal dentro de ± 3 nt.
Uma amostra bem dobrada pode ser identificada da seguinte forma: 1) O número de prótons imino observados corresponde ao número de prótons imino esperados de uma simulação de estrutura secundária (por exemplo,Mc-Fold39, Figura 4A). 2) O par de bases de oscilação syn G-U em um loop UUCG (se presente) é visível a ~9,5 ppm, às vezes apenas visível em temperatura mais baixa. Outras impressões digitais do loop UUCG foram descritas por Fürtig e colegas40. 3) A impressão digital aromática concorda com uma amostra previamente atribuída que foi confirmada para dobrar corretamente(Figura 4C).
Uma amostra descasada ou degradada pode ser identificada da seguinte forma: 1) Há mais sinais de imino do que uma simulação de estrutura secundária prevê (NOTA: menos sinais imino não implicam necessariamente dobras erradas, pois os pares de base de fechamento muitas vezes não são visíveis, e a troca conformacional amplia as linhas). 2) Ausência de sinais imino. 3) Sinais estreitos de alta intensidade na região aromática, indicando produtos de degradação de nucleotídeos únicos. 4) Divergência entre sinais iminos ou aromáticos a uma amostra de referência de dobramento confirmado(Figura 4C).
Um átomo que não mostra nenhuma troca na escala de tempo detectável pode ser identificado da seguinte forma: 1) a partir de um perfil RD plano (devido à contribuição REX ausente variando com a potência SL aplicada) (Figura 5B e Figura 5F). 2) É preciso ter cuidado para o caso de troca lenta-intermediária quando kEX e Δω são da mesma magnitude. Nesse caso, a contribuição sobre ressonância pode ser muito pequena como pode ser visto na Figura 5C (neste caso os parâmetros montados são kEX = 292 ± 40 Hz e Δω = 112 ± 4 Hz). Em caso de dúvida, uma curva de baixa ressonância SL pode ser registrada para verificação.
Um átomo mostrando troca na escala de tempo intermediário pode ser identificado 1) a partir de um perfil de dispersão de relaxamento não plano em um experimento RD de ressonância(Figura 5B e Figura 5F); 2) uma largura de linha mais ampla no experimento HSQC ou SELOPE também pode ser um indicador de troca.
Os valores de potência SL bem selecionados para curvas fora da ressonância(Figura 5E,F): 1) têm uma contribuição considerável kEX na curva de ressonância (os valores de potência SL selecionados são indicados na Figura 5C e Figura 5G). 2) À medida que as curvas de ressonância são medidas para pelo menos 3 valores de potência SL, os valores de energia SL selecionados devem ser espalhados sobre a região da curva de ressonância com contribuição kEX. 3) Leve a curvas não planas R2+REX após o ajuste de Laguerre (por exemplo, Figura 5D: Pontos fortes SL 25, 50 e 75 Hz; Figura 5E).
Valores de potência SL mal selecionados para curvas fora de ressonância(Figura 5E,F) levam a curvas planas R2+REX após o ajuste de Laguerre. Um exemplo é mostrado na Figura 5E, onde a curva de 100 Hz fora de ressonância é muito plana e, portanto, não fornece informações significativas sobre Δω.
Indicações para artefatos nucleares de overhauser de quadro rotativo (ROE): 1) Δω obtidos a partir de curvas de ressonância fora correspondem a mudanças químicas de prótons nas proximidades espaciais / prótons, que mostram um pico cruzado com o pico de interesse no espectro nuclear de espectro de efeito Overhauser (NOESY). (por exemplo,a Figura 5I mostra curvas largas fora de ressonância como esperado para troca rápida-intermediária, mas as curvas também têm características mais nítidas, por exemplo,a -3000 Hz e +1500 Hz. Estes são muito prováveis devido a um artefato ROE em vez de uma mudança química para este H8 em um conformr diferente). 2) Laguerre fit funciona, mas não funciona bem (dá altos erros ou valores fisicamente impossíveis) para uma ressonância on-resonance e pelo menos 3 curvas fora de ressonância, embora os exponenciais tenham sido obtidos a partir de experimentos com alto SINO (>20) (por exemplo, kEX = 43.502 ± 38.478 Hz). Muitas vezes cada SL se encaixa individualmente bem, mas encaixá-los em conjunto dá um erro muito maior; o comportamento oposto é esperado para um verdadeiro estado animado.
Indicações para troca "verdadeira" Δω: 1) Δω obtidas a partir de curvas fora de ressonância não correspondem a mudanças químicas de prótons em proximidade espacial/prótons, que mostram um pico cruzado com o pico de interesse no espectro NOESY (por exemplo, Figura 5E). 2) O ajuste de Laguerre dá baixos erros para uma ressonância on-resonance e pelo menos 3 curvas fora de ressonância (por exemplo, Figura 5E vs. Figura 5I, veja legenda para resultados adequados).
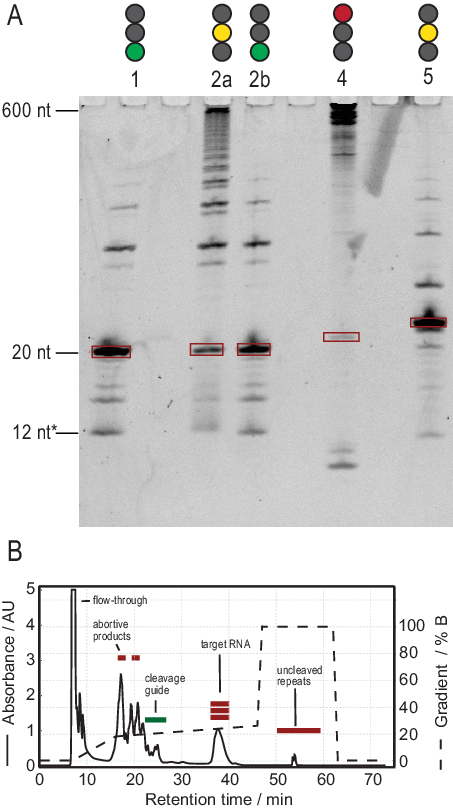
Figura 3: Produção amostral por T7 tandem IVT e RNase H reação de decote. (A) Desnaturação PAGE de resultados positivos e negativos de tandem IVT e RNase H decote. A altura da escada refere-se às referências de RNA, 12* refere-se ao guia quimérico de decote. Raia 1: Geração bem sucedida de um RNA alvo de 20 nt. Poucos produtos mais curtos e longos estão presentes. Faixa 2a: Decote incompleto da transcrição tandem. Embora a purificação do HPLC seja possível, muito material seria desperdiçado. Pista 2b: O decote RNase H continuado da Faixa 2 produz uma amostra limpa pronta para injeção HPLC (idêntica à Faixa 1). Lane 4: O decote RNase H não teve sucesso, e nenhuma banda alvo foi produzida. A transcrição em conjunto ainda é visível a 600 nt. Lane 5: Uma banda alvo foi produzida, mas uma banda forte -1 está presente. Embora o HPLC possa ser realizado, é necessária uma remoção cuidadosa do produto lateral. (B) Exemplo de uma injeção hplc bem sucedida. O pico em 38 min contém RNA puro do comprimento alvo, enquanto produtos mais longos e mais curtos são bem separados do RNA alvo. O painel B foi modificado a partir de 21. Abreviaturas: IVT = transcrição in vitro; HPLC = cromatografia líquida de alto desempenho; nt = nucleotídeos; UA = unidades arbitrárias. Clique aqui para ver uma versão maior desta figura.
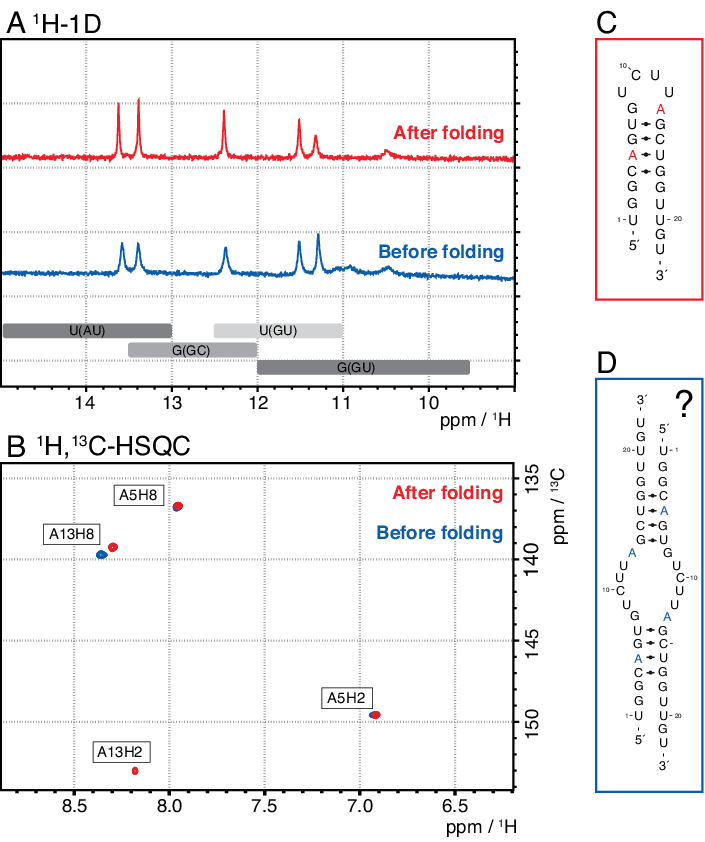
Figura 4: Exemplo de um grampo de RNA antes (azul) e depois (vermelho) da etapa dobrável 2.4 (ver protocolo) em NMR. (A) região Imino de um espectro 1H-1D de um RNA de 22-mer rotulado A. As regiões esperadas para a identidade do par de bases de sinais imino são indicadas em cinza abaixo. (B) 1H,13C-HSQC espectro das ressonâncias aromáticas do RNA do painel A. A amostra após a dobra (vermelho) mostra 4 sinais como esperado, enquanto a amostra antes de dobrar (azul) mostra apenas 3 sinais. (C) Previsão mc-fold do RNA 22-mer como um grampo de cabelo. Cinco sinais imino são esperados a partir desta estrutura secundária, que pode ser encontrada em ambas as amostras no painel A. (D) Estrutura proposta de um homodimer formado pelo RNA de 22-mer, resultando nos mesmos 5 pares de base que a estrutura do grampo de cabelo. Abreviaturas: NMR = ressonância magnética nuclear; 1D = unidimensional; HSQC = correlação quântica única heteronuclear; ppm = partes por milhão. Clique aqui para ver uma versão maior desta figura.
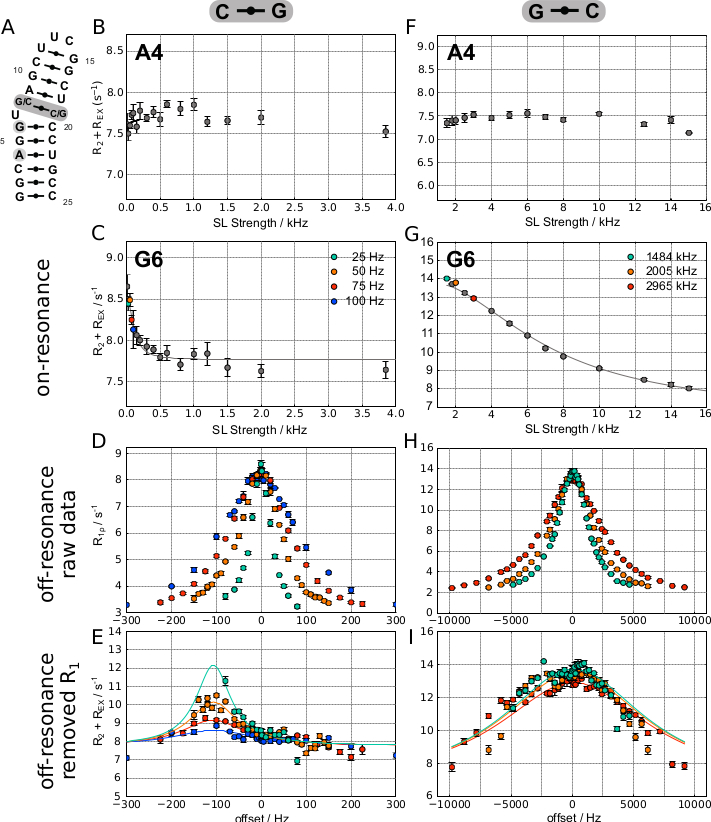
Figura 5: 1H R1ρ RD resultados representativos para duas construções diferentes baseadas em um grampo de cabelo RNA. (A) A coluna esquerda mostra os resultados obtidos no RNA com um par de base C-G acima do U abaulado, enquanto a coluna direita mostra os resultados obtidos em uma amostra onde o par base foi trocado para G-C em vez disso. (B) e (F) apresentam perfis de dispersão plana obtidos para A4H8 para as duas construções, indicando nenhuma troca conformacional. (C–E) mostrar sobre ressonância, off-resonance e dados instalados obtidos para G6 na construção (G-C). O fit laguerre leva ao seguinte resultado: R1 = 2,87 ± 0,01 Hz, R2 = 7,76 ± 0,03 Hz, kEX =292 ± 40 Hz, pES = 0,31 ± 0,03 %, Δω = 112 ± 4 Hz. (G–I) mostram sobre ressonância, off-resonance, e dados instalados obtidos para G6 na construção (G-C). O fit de Laguerre leva ao seguinte resultado: R1 = 1,93 ± 0,02 Hz, R2 = 6,71 ± 0,86 Hz, k EX = 43.502 ± 38.478 Hz, p ES = 27 ± 16 %, Δω = 203 ± 166 Hz. Este número foi modificado a partir de 20. Abreviação: SL = trava de giro. Clique aqui para ver uma versão maior desta figura.
Discussão
O protocolo aqui apresentado é uma síntese de diversos protocolos publicados anteriormente na forma dos artigos de pesquisa10,20,21,31. Assim, segmentos do protocolo podem ser aplicados, enquanto outros podem ser trocados à preferência do leitor. Por exemplo, as medidas R1ρ podem ser realizadas em uma amostra de RNA produzida com qualquer método, dado que a dobra e a homogeneidade do comprimento são assumidas. Além disso, o protocolo não contém informações sobre a atribuição de ressonância da sequência de RNA - uma etapa necessária para experimentos RD - uma vez que isso foi extensivamente coberto na literatura anterior19,37,38. Esquemas de rotulagem parciais, segmentais ou específicos do local36,41,42,43,44 são abordagens para facilitar a atribuição de ressonância ou reduzir a sobreposição de ressonâncias que são de interesse em experimentos de RD e foram descritas longamente na literatura. Este método permite o uso de rotulagem uniforme de qualquer identidade nucleotídea, que já pode simplificar significativamente a atribuição de ressonância.
O método IVT aqui apresentado supera questões conhecidas com sequências e rotulagem, aumenta o rendimento e diminui o custo e o tempo de trabalho em comparação com outros métodos. O uso da sequência de iniciação viral reduz a necessidade de otimização da reação, que é um problema conhecido no campo que pode ser demorado para realizar e rende apenas algumas cópias da transcrição no caso de iniciação não-G. O decote T7 IVT e RNase H da transcrição tandem pode ser realizado simultaneamente no mesmo vaso. Um padrão de repetições multiméricas de tandem pode ser visto em um gel PAGE desnaturing durante a reação, que se une a uma única banda no RNA alvo após a conclusão da reação RNase H(Figura 3A,faixas 1 e 2b). Os rendimentos típicos usando este método variam entre 30 e 70 nmol RNA por 1 mL IVT. No entanto, o método baseado no decote RNase H de repetições tandem não vem sem certos problemas próprios. A reação de decote RNase H muitas vezes não vai para a conclusão quando executada simultaneamente com transcrição T7(Figura 3A, faixa 2a).
A separação das unidades tandem pode ser finalizada com a ressarção do guia de decotes à transcrição e adicionando mais RNase H (Figura 3A, faixa 2b, passo 2.1.2). Como o aquecimento de grandes volumes é lento e leva à hidrólise catalisada mg2+de RNA, foi utilizado um forno de micro-ondas convencional, que aquece a amostra para >95 °C em 10-15 s. Os efeitos adversos nas amostras produzidas não foram observados até agora. Algumas construções mostram uma pequena segunda banda que não poderia ser eliminada pela otimização das condições de reação (Figura 3A, faixa 4). Geralmente estes são claramente visíveis como um ombro no cromatógrafo HPLC, se um gradiente de elução bem otimizado for usado, e pode ser removido (passo 2.2.5). A discussão a seguir tem como objetivo destacar etapas críticas no protocolo, especificamente no que diz respeito à obtenção de dados de alta qualidade que permitam uma interpretação da dinâmica conformacional.
Contaminação rnase
As RNases extracelulares são onipresentes, altamente estáveis e representam a maior ameaça para a estabilidade a longo prazo das amostras de RMR. Portanto, é crucial trabalhar em um ambiente livre de RNase e manter todos os reagentes e plásticos livres de RNase. Recomenda-se o uso de dicas de filtro e talvez até máscaras faciais. Isso é especificamente importante após a purificação do HPLC. Amostras de RMR contaminadas com RNases normalmente exibem picos estreitos visíveis em espectros de 1H-1D após dias ou semanas devido a produtos de degradação de nucleotídeos únicos. Tal amostra não é adequada para medições R1ρ.
Amostra de NMR
Devido à sua natureza altamente carregada, o RNA pode ser usado em altas concentrações sem precipitação quando comparado com a maioria das proteínas. O uso de tubos De Ressonância Magnética Shigemi (ver a Tabela de Materiais) é vantajoso, pois permitem o centro da amostra altamente concentrada no centro da bobina, ao mesmo tempo em que fornecem condições ideais de shimming e locking devido ao fundo de vidro e êmbolo compatível com suscetibilidade. Dessa forma, a B1-inhomogeneidade é reduzida, dando origem a linhas mais estreitas. O volume típico da amostra em um tubo NMR é de 250 μL, e a concentração típica é de 1-2 mM. Amostras abaixo de 500 μM não são recomendadas para experimentos rd, pois o experimento levaria muito tempo e um bom calço. Da mesma forma, o volume amostral abaixo de 200 μL não é recomendado porque é necessário um bom calço e estabilidade de campo (bloqueio). Ao inserir o êmbolo, é crucial evitar a formação de bolhas na amostra (etapa 2.4.5). Se não for fixado corretamente, o êmbolo pode deslizar para baixo na amostra, reduzindo o volume detectável. Além disso, mudanças rápidas na temperatura podem levar à formação de novas bolhas na amostra. Portanto, deve-se tomar cuidado ao transportar a amostra e ao alterar a temperatura da sonda no espectrômetro NMR. Verifique se há bolhas na amostra ao medir novamente após um período mais longo.
Dobramento de RNA
Moléculas dinâmicas de RNA podem existir em múltiplas conformações quando não dobradas corretamente. Embora as temperaturas de fusão das estruturas secundárias possam ser apenas ligeiramente acima da temperatura ambiente, um procedimento de aquecimento e resfriamento completo é recomendado antes da medição. Amostras de grampos altamente concentradas dobrando sob controle cinético (aquecimento e resfriamento) podem formar homodimers ao longo do tempo, o que requer um controle rigoroso da dobra de RNA antes de cada medição de RMR. Se o RNA medido não for uma estrutura de grampos, mas um duplex RNA, deve ser aplicado dobramento lento sob controle termodinâmico.
Neste caso, o processo de resfriamento após o aquecimento deve estar na faixa de horas, enquanto o RNA é usado em seu volume final e concentração na amostra de RMN. Uma contagem inicial de ressonâncias iminos e aromáticas esperadas pode fornecer insights sobre a homogeneidade da amostra. Se a amostra não parecer esperada, deve ser re-dobrada. Mg2+ (adicionado como sal de cloreto) pode ajudar com estruturas de RNA dobráveis45. Na prática, o controle dobrável serve como comparação com uma amostra que tem sido usada para pelo menos atribuir parcialmente as ressonâncias NMR e resolver a estrutura secundária experimentalmente.
Considerações de potência de bloqueio de giro e aquecimento
No caso de executar os experimentos 1H R1ρ RD como experimentos de visão geral 2D, a energia SL não deve ser inferior a 1,2 kHz. A frequência do transmissor de radiofrequência deve ser colocada no meio da região ppm dos picos de interesse (por exemplo,7,5 ppm para prótons aromáticos). A largura de banda de 1,2 kHz será então grande o suficiente para travar esses prótons sem grandes efeitos de ressonância. Tais efeitos podem ser identificados no perfil RD. Se ocorrerem, os valores R2+REX aumentam em vez de diminuir com o aumento dos valores de potência SL, especialmente para baixo poder SL. Verifique se os valores de alimentação SL calculados correspondem à potência fornecida à amostra. Na prática, a potência SL calculada pode ser usada se o pulso rígido de 1H 90° for calibrado cuidadosamente em espectrômetros mais novos; no entanto, isso pode ser verificado calibrando a potência SL para cada largura de banda desejada.
A gama de energia SL, que pode ser usada em experimentos rd de 1H R1ρ é muito ampla, levando a um aquecimento amostral variado (1,2 kHz a 15 kHz para HSQC para sequências baseadas em HCP e 50 Hz a 15 kHz para experimentos SELOPE). O aquecimento desigual da amostra pode ser detectado como uma pequena mudança na mudança química ao comparar 1Ds obtidos para SLs de baixa potência vs. SLs de alta potência. Esse efeito geralmente não é considerado em compensações térmicas em experimentos R1ρ em heteronucleos. A compensação de calor nesses experimentos é geralmente configurada para corrigir para diferentes aquecimentos devido às diferentes durações de bloqueio de giro especificadas na lista vd de cada série de potência de bloqueio de spin. Especialmente para o experimento SELOPE, uma compensação de segundo calor deve ser usada em todos os pontos fortes SL aplicados, conforme descrito em20.
considerações da lista vd
Como mencionado anteriormente, a lista vd deve conter um ponto de tempo tempo suficiente para obter uma decadência significativa de intensidade (idealmente até 30% do sinal inicial, ou o mais baixo possível se não for possível alcançar uma decadência de 70% dentro das especificações da sonda). Embora a lista vd tenha sido otimizada para uma baixa potência SL (1,2 kHz), esta lista vd também deve ser testada na maior potência SL a ser usada (por exemplo,15 kHz). Isso se deve ao fato de que, para picos com contribuição significativa de REX, a decadência será muito mais lenta na alta potência de SL. Assim, uma decadência suficiente também deve ser verificada em alta potência SL. O mesmo deve ser considerado para decaimentos em altas compensações em experimentos fora da ressonância. O ponto de tempo máximo ideal da lista vd poderia ser significativamente diferente para as diferentes regiões do experimento de dispersão. Nesse caso, mais pontos poderiam ser incluídos na lista vd, e os pontos de lista vd mais longos para maior potência SL ou compensações mais altas durante a análise, com base no baixo SINO que eles levarão, poderiam ser descartados. Em geral, os pontos de lista de 5-8 vd devem ser considerados capazes de detectar artefatos potenciais que levam a decaimentos não exponenciais, como o acoplamento J (veja abaixo).
1D- Consideraçõesde seletividade do HCP
Deve-se tomar cuidado especial ao executar a versão 1D baseada em HCP se houver outro pico sobreposto com o pico de interesse na dimensão 1H do experimento baseado em HSQC 2D. As transferências baseadas em HCP são muito, mas nunca 100% seletivas, e, portanto, pode acontecer que outro pico contribua para a intensidade e o comportamento de decadência do pico de interesse no 1D. Uma indicação para isso seria uma diferença nos valores de ressonância R1ρ obtidos utilizando as versões 1D e 2D do experimento rotulado.
Considerações do ROE:
Para curvas fora de ressonância de átomos com câmbio lento-intermediário, os artefatos ROE podem ser identificados com base em uma comparação do Δω obtido com um espectro NOESY ou ROESY. Se um pico cruzado pode ser identificado em uma diferença de mudança química correspondente a Δω, então o estado animado observado pode de fato ser um artefato ROE (por exemplo,ROEs foram encontrados entre prótons aromáticos, que estão todos na mesma faixa de mudança química e, portanto, cobertos por aquelas curvas fora de ressonância20). Pela experiência, isso sempre levou a ataques ruins com grandes erros, possivelmente devido ao ROE não seguir o mesmo padrão que o REX com o aumento do poder SL. A situação se torna mais difícil para a troca intermediária-rápida. Enquanto a curva de ressonância (a partir da comparação com os dados de 13C obtidos no núcleo vizinho) ainda representa o processo de troca entre o GS e o ES, a curva de ressonância fora é influenciada por múltiplos artefatos roe.
Nesse caso, a potência SL para detectar o processo de troca é maior (>1,5 kHz) e, portanto, abrange um número maior de prótons à medida que as curvas de ressonância off-resonance se estendem sobre as diferenças de mudança química de vários candidatos ao ROE (para H8 estes seriam: amino protons na ca. ±1000 Hz, H5/H1's em ca. -1200 Hz, imino prótons em cerca de 3500 Hz). Até agora, nenhum método foi encontrado para suprimir esses artefatos roe (além do uso de nucleotídeos parcialmente deutados46), e dados fora da ressonância não devem ser registrados para troca rápida-intermediária, pois nenhuma informação confiável sobre o Δω real pode ser extraída com este método, se a contribuição NOE/ROE não puder ser excluída através de espectros NOESY.
J-Acoplamento (Hartmann-Hahn) considerações
Embora as curvas de ressonância para prótons homonucleares acoplados a J, como H6, tenham sido registradas com sucesso10,20, cuidados especiais devem ser tomados para medições fora da ressonância, especialmente para baixo poder SL, pois as condições de correspondência Hartmann-Hahn podem abranger uma ampla gama de deslocamentos investigados. Os artefatos hartmann-hahn podem ser identificados como oscilações na decadência exponencial ou aumento dos valores R2+REX com o aumento dos pontos fortes SL nas parcelas RD de ressonância20.
Divulgações
K.P. é consultor da Arrakis Therapeutics, uma empresa que descobre pequenas moléculas voltadas para o RNA.
Agradecimentos
Agradecemos à instalação de ciência de proteínas (PSF) do Instituto Karolinska pela expressão e purificação da polimerase T7 RNA e E. coli RNase H, Martin Hällberg pelo generoso dom da fosfatase inorgânica, e de todo o Petzoldlab para discussões valiosas. Agradecemos a Luca Retattino pela preparação das construções u-bulge e Emilie Steiner e Carolina Fontana por sua contribuição às macros e roteiros adequados. Reconhecemos o Instituto Karolinska e o Departamento de Bioquímica Médica e Biofísica pelo apoio à compra de um espectrômetro de 600 MHz e financiamento de posições (KI FoAss e KID 2-3707/2013). Somos gratos pela contribuição financeira de Vetenskapsrådet (#2014-4303), Stiftelsen för strategisk Forskning (ICA14-0023 e FFL15-0178) e The Ragnar Söderberg Stiftelse (M91-14), Harald och Gretason Stiftelse (M91-14), Harald och Gretas JS20140009), Carl Tryggers stiftelse (CTS14-383 e 15-383), Eva och Oscar Ahréns Stiftelse, Åke Wiberg Stiftelse (467080968 e M14-0109), Cancerfonden (CAN 2015/388), J.S. reconhece financiamento através de uma Marie Skłodowska-Curie IF (EU H2020, Projeto MSCA-IF nº 747446).
Materiais
| Name | Company | Catalog Number | Comments |
| 40% Acrylamide/Bis Solution | Bio-Rad | 161-0144 | |
| 5-alpha Competent E. coli | NEB | C2987I | |
| Acetic Acid | Sigma-Aldrich | 49199 | |
| Acetonitrile | Sigma-Aldrich | 34851 | |
| AFC-3000, HPLC Fraction collector | Thermo Scientific | 5702.1 | |
| Agarose | Sigma-Aldrich | A9414 | |
| Amersham ImageQuant 800 UV | GE Healthcare | 29399482 | Replacing LAS-4000 or equivalent |
| Amicon ultra centrifugal filter unit | Sigma-Aldrich | UFC900324 | |
| Ammonium persulfate | Sigma-Aldrich | A3678 | |
| Ampicillin | Sigma-Aldrich | A9518 | |
| ATP | Sigma-Aldrich | A2383 | |
| ATP-13C10/15N5 | Sigma-Aldrich | 645702 | |
| BamHI restriction enzyme | NEB | R0136L | |
| Bottle top filter | VWR | 514-1019 | |
| Bromophenol Blue | Sigma-Aldrich | 1081220005 | |
| Cleavage guide | IDT | N/A | or equivalent |
| CTP | Sigma-Aldrich | C1506 | |
| CTP-13C10/15N5 | Sigma-Aldrich | 645699 | |
| D2O | Sigma-Aldrich | 151882 | |
| Dionex Ultimate 3000 UHPLC system | Thermo Scientific | N/A | |
| DL-Dithiotreitol | Sigma-Aldrich | 43815 | |
| DMSO | Sigma-Aldrich | D8418 | |
| DNAPac PA200 22x250 Semi-Prep column | Thermo Scientific | SP6734 | |
| DNAPac PA200 22x50 guard column | Thermo Scientific | SP6731 | |
| E.coli RNase H | NEB | M0297L | or made in-house uniprot ref. P0A7Y4 |
| EDTA | Sigma-Aldrich | E6758 | |
| Eppendorf centrifuge, rotor: A-4-44 | Eppendorf | 5804R | |
| Ethanol 95% | Fisher scientific | 11574139 | |
| Ethanol 95% denatured | VWR | 85829.29 | |
| Formamide | Sigma-Aldrich | 47671 | |
| GelRed | VWR | 41003 | |
| GeneRuler 1kbp Plus | Fisher Scientific | SM1333 | Optional |
| GMP | Sigma-Aldrich | G8377 | |
| GMP-13C10/15N5 | Sigma-Aldrich | 650684 | |
| GTP | Sigma-Aldrich | G8877 | |
| GTP-13C10/15N5 | Sigma-Aldrich | 645680 | |
| Hydrochloric Acid | Sigma-Aldrich | H1758 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-100UN | or made in-house uniprot ref. P0A7A9 |
| Invitrogen UltraPure 10X TBE-buffer | Sigma-Aldrich | T4415 | |
| Julabo TW8 Water bath | VWR | 461-3117 | |
| kuroGEL Midi 13 Horizontal gel electrophoresis | VWR | 700-0056 | or comparable |
| LB broth (Lennox) | Sigma-Aldrich | L3022 | |
| LB broth with agar (Lennox) | Sigma-Aldrich | L2897 | |
| Low Range ssRNA Ladder | NEB | N0364S | Optional |
| LPG-3400RS Pump | Thermo Scientific | 5040.0036 | |
| Magnesium chloride hexahydrate | Sigma-Aldrich | 63068 | |
| microRNA Marker | NEB | N2102S | |
| Microwave oven | Samsung | MS23F301EAW | |
| Mini-PROTEAN electrophoresis equipment | Bio-Rad | 1658004 | |
| NucleoBond Xtra Maxi | Machinery-Nagel | 740414.10M | |
| pUC19 plasmid containing tandem insert | Genscript | N/A | or equivalent |
| RNaseZAP | Sigma-Aldrich | R2020 | |
| Shigemi tube 5mm | Sigma-Aldrich | Z529427 | |
| Single-use syringe, Luer lock tip | VWR | 613-2008 | |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium chloride | Sigma-Aldrich | 730-1470 | |
| Sodium perchlorate | Sigma-Aldrich | 71853 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S3264 | |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Spermidine trihydrochloride | Sigma-Aldrich | 85578 | |
| SYBR Gold | ThermoFisher | S11494 | |
| Syringe filters | VWR | 514-0061 | |
| T7 RNA polymerase | Sigma-Aldrich | 10881767001 | or made in-house uniprot ref. P00573 |
| TCC-3000RS Column thermostat | Thermo Scientific | 5730 | |
| Tetramethylethylenediamine | Sigma-Aldrich | T9281 | |
| Tris Base | Fisher Scientific | 10103203 | |
| UMP | Sigma-Aldrich | U6375 | |
| UMP-13C9/15N2 | Sigma-Aldrich | 651370 | |
| Urea | Sigma-Aldrich | U5378 | |
| UTP | Sigma-Aldrich | U6625 | |
| UTP-13C10/15N5 | Sigma-Aldrich | 645672 | |
| VWD-3100 Detector | Thermo Scientific | 5074.0005 |
Referências
- Djebali, S., et al. Landscape of transcription in human cells. Nature. 489 (7414), 101-108 (2012).
- Doudna, J. A., Cech, T. R. The chemical repertoire of natural ribozymes. Nature. 418 (6894), 222-228 (2002).
- Sehgal, P. B., Westley, J., Lerea, K. M., DiSenso-Browne, S., Etlinger, J. D. Biomolecular condensates in cell biology and virology: phase-separated membraneless organelles (MLOs). Analytical Biochemistry. , 597 (2020).
- Herschlag, D., Allred, B. E., Gowrishankar, S. From static to dynamic: the need for structural ensembles and a predictive model of RNA folding and function. Current Opinion Structural Biology. 30, 125-133 (2015).
- Kimsey, I. J., Petzold, K., Sathyamoorthy, B., Stein, Z. W., Al-Hashimi, H. M. Visualizing transient Watson-Crick-like mispairs in DNA and RNA duplexes. Nature. 519 (7543), 315-320 (2015).
- Dethoff, E. A., Petzold, K., Chugh, J., Casiano-Negroni, A., Al-Hashimi, H. M. Visualizing transient low-populated structures of RNA. Nature. 491 (7426), 724-728 (2012).
- Baisden, J. T., Boyer, J. A., Zhao, B., Hammond, S. M., Zhang, Q. Visualizing a protonated RNA state that modulates microRNA-21 maturation. Nature Chemical Biology. 17 (1), 80-88 (2021).
- Marušič, M., Schlagnitweit, J., Petzold, K. RNA dynamics by NMR spectroscopy. Chembiochem. 20 (21), 2685-2710 (2019).
- Baronti, L., et al. Base-pair conformational switch modulates miR-34a targeting of Sirt1 mRNA. Nature. 583 (7814), 139-144 (2020).
- Steiner, E., Schlagnitweit, J., Lundström, P., Petzold, K. Capturing excited states in the fast-intermediate exchange limit in biological systems using 1H spectroscopy. Angewandte Chemie International Edition. 55 (51), 15869-15872 (2016).
- Moschen, T., et al. Ligand-detected relaxation dispersion NMR spectroscopy: dynamics of preQ1-RNA binding. Angewandte Chemie International Edition. 54 (2), 560-563 (2015).
- LeBlanc, R. M., Longhini, A. P., Tugarinov, V., Dayie, T. K. NMR probing of invisible excited states using selectively labeled RNAs. Journal of Biomolecular NMR. 71 (3), 165-172 (2018).
- Strebitzer, E., Nußbaumer, F., Kremser, J., Tollinger, M., Kreutz, C. Studying sparsely populated conformational states in RNA combining chemical synthesis and solution NMR spectroscopy. Methods. 1148, 39-47 (2018).
- Rangadurai, A., Shi, H., Al-Hashimi, H. M. Extending the sensitivity of CEST NMR spectroscopy to micro-to-millisecond dynamics in nucleic acids using high-power radio-frequency fields. Angewandte Chemie International Edition. 59 (28), 11262-11266 (2020).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. Using relaxation dispersion NMR spectroscopy to determine structures of excited, invisible protein states. Journal of Biomolecular NMR. 41 (3), 113-120 (2008).
- Lundström, P., Akke, M. Off-resonance rotating-frame amide proton spin relaxation experiments measuring microsecond chemical exchange in proteins. Journal of Biomolecular NMR. 32 (2), 163-173 (2005).
- Lee, J., Dethoff, E. A., Al-Hashimi, H. M. Invisible RNA state dynamically couples distant motifs. Proceedings of the National Academy of Sciences of the United States of America. 111 (26), 9485-9490 (2014).
- Schnieders, R., Keyhani, S., Schwalbe, H., Fürtig, B. More than proton detection- new avenues for NMR spectroscopy of RNA. Chemistry. 26 (1), 102-113 (2020).
- Fürtig, B., Richter, C., Wöhnert, J., Schwalbe, H. NMR spectroscopy of RNA. Chembiochem. 4 (10), 936-962 (2003).
- Schlagnitweit, J., Steiner, E., Karlsson, H., Petzold, K. Efficient detection of structure and dynamics in unlabeled RNAs: The SELOPE approach. Chemistry. 24 (23), 6067-6070 (2018).
- Feyrer, H., Munteanu, R., Baronti, L., Petzold, K. One-pot production of RNA in high yield and purity through cleaving tandem transcripts. Molecules. 25 (5), 1142 (2020).
- Baronti, L., Karlsson, H., Marušič, M., Petzold, K. A guide to large-scale RNA sample preparation. Analytical and Bioanalytical Chemistry. 410 (14), 3239-3252 (2018).
- Brunelle, J. L., Green, R. In vitro transcription from plasmid or PCR-amplified DNA. Methods in Enzymology. 530, 101-114 (2013).
- Borkotoky, S., Murali, A. The highly efficient T7 RNA polymerase: A wonder macromolecule in biological realm. International Journal of Biological Macromolecules. 118, 49-56 (2018).
- Arnaud-Barbe, N., Cheynet-Sauvion, V., Oriol, G., Mandrand, B., Mallet, F. Transcription of RNA templates by T7 RNA polymerase. Nucleic Acids Research. 26 (15), 3550-3554 (1998).
- Guillerez, J., Lopez, P. J., Proux, F., Launay, H., Dreyfus, M. A mutation in T7 RNA polymerase that facilitates promoter clearance. Proceedings of the National Academy of Sciences of the United States of America. 102 (17), 5958-5963 (2005).
- Kuzmine, I., Gottlieb, P. A., Martin, C. T. Binding of the priming nucleotide in the initiation of transcription by T7 RNA polymerase. Journal of Biological Chemistry. 278 (5), 2819-2823 (2003).
- Gholamalipour, Y., Karunanayake Mudiyanselage, A., Martin, C. T. 3' end additions by T7 RNA polymerase are RNA self-templated, distributive and diverse in character - RNA-Seq analyses. Nucleic Acids Research. 46 (18), 9253-9263 (2018).
- Inoue, H., Hayase, Y., Iwai, S., Ohtsuka, E. Sequence-dependent hydrolysis of RNA using modified oligonucleotide splints and RNase H. FEBS Letters. 215 (2), 327-330 (1987).
- Wang, X., Li, C., Gao, X., Wang, J., Liang, X. Preparation of small RNAs using rolling circle transcription and site-specific RNA disconnection. Molecular Therapy - Nucleic Acids. 4, 215 (2015).
- Karlsson, H., Baronti, L., Petzold, K. A robust and versatile method for production and purification of large-scale RNA samples for structural biology. RNA. 26 (8), 1023-1037 (2020).
- Hartmann, S. R., Hahn, E. L. Nuclear double resonance in the rotating frame. Physical Review. 128 (5), 2042-2053 (1962).
- Chiarparin, E., Pelupessy, I., Bodenhausen, G. Selective cross-polarization in solution state NMR. Molecular Physics. 95 (5), 759-767 (1998).
- Korzhnev, D. M., Orekhov, V. Y., Kay, L. E. Off-resonance R 1ρ NMR studies of exchange dynamics in proteins with low spin-lock fields: an application to a Fyn SH3 domain. Journal of the American Chemical Society. 127 (2), 713-721 (2005).
- Hansen, A. L., Nikolova, E. N., Casiano-Negroni, A., Al-Hashimi, H. M. Extending the range of microsecond-to-millisecond chemical exchange detected in labeled and unlabeled nucleic acids by selective carbon R 1ρ NMR spectroscopy. Journal of the American Chemical Society. 131 (11), 3818-3819 (2009).
- Duss, O., Maris, C., von Schroetter, C., Allain, F. H. -. T. A fast, efficient and sequence-independent method for flexible multiple segmental isotope labeling of RNA using ribozyme and RNase H cleavage. Nucleic Acids Research. 38 (20), 188 (2010).
- Krähenbühl, B., Lukavsky, P., Wider, G. Strategy for automated NMR resonance assignment of RNA: application to 48-nucleotide K10. Journal of Biomolecular NMR. 59 (4), 231-240 (2014).
- LeBlanc, R. M., Longhini, A. P., Le Grice, S. F. J., Johnson, B. A., Dayie, T. K. Combining asymmetric 13C-labeling and isotopic filter/edit NOESY: a novel strategy for rapid and logical RNA resonance assignment. Nucleic Acids Research. 45 (16), 146 (2017).
- Parisien, M., Major, F. The MC-Fold and MC-Sym pipeline infers RNA structure from sequence data. Nature. 452 (7183), 51-55 (2008).
- Fürtig, B., Richter, C., Bermel, W., Schwalbe, H. New NMR experiments for RNA nucleobase resonance assignment and chemical shift analysis of an RNA UUCG tetraloop. Journal of Biomolecular NMR. 28 (1), 69-79 (2004).
- Keyhani, S., Goldau, T., Blümler, A., Heckel, A., Schwalbe, H. Chemo-enzymatic synthesis of position-specifically modified RNA for biophysical studies including light control and NMR spectroscopy. Angewandte Chemie International Edition. 57 (37), 12017-12021 (2018).
- Marchanka, A., Kreutz, C., Carlomagno, T. Isotope labeling for studying RNA by solid-state NMR spectroscopy. Journal of Biomolecular NMR. 71, 151-164 (2018).
- Becette, O., Olenginski, L. T., Dayie, T. K. Solid-phase chemical synthesis of stable isotope-labeled RNA to aid structure and dynamics studies by NMR spectroscopy. Molecules. 24 (19), 3476 (2019).
- Zhang, X., Li, M., Liu, Y. Optimization and characterization of position-selective labelling of RNA (PLOR) for diverse RNA and DNA sequences. RNA Biology. 17 (7), 1009-1017 (2020).
- Roh, J. H., et al. Effects of preferential counterion interactions on the specificity of RNA folding. The Journal of Physical Chemistry Letters. 9 (19), 5726-5732 (2018).
- Juen, M. A., et al. Excited states of nucleic acids probed by proton relaxation dispersion NMR spectroscopy. Angewandte Chemie German Edition. 55 (39), 12008-12012 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados