Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Çeviri Yönetmeliğinde RNA-Protein Etkileşimlerini İncelemek için Optik Cımbız
Bu Makalede
Özet
Bu protokol, optik cımbız kullanarak RNA-protein etkileşimlerini incelemek için eksiksiz bir deneysel iş akışı sunar. Optik cımbızın konfokal mikroskopi ile kombinasyonu da dahil olmak üzere çeşitli olası deneysel kurulumlar özetlenmiştir.
Özet
RNA, işlevleri için gerekli olan ve böylece hücredeki çeşitli süreçleri etkileyebilen çeşitli yapısal kıvrımları benimsemiştir. Ek olarak, bir RNA'nın yapısı ve işlevi proteinler, metabolitler veya diğer RNA'lar gibi çeşitli trans-etkili faktörler tarafından modüle edilebilir. Örneğin, karek değiştirme RNA molekülleri, ribozomları alternatif bir açık okuma çerçevesine yönlendiren ve böylece gen anahtarları olarak hareket eden kodlama bölgelerinde bulunan düzenleyici RNA'lardır. Ayrıca proteinlere veya diğer trans faktörlere bağlanmasından sonra farklı kıvrımları benimseyebilirler. RNA bağlayıcı proteinlerin çevirideki rolünü ve RNA yapısını ve stabilitesini nasıl modüle ettiklerini incelemek için, bu RNA-protein komplekslerinin etkileşim ve mekanik özelliklerini aynı anda incelemek çok önemlidir. Bu çalışma, RNA-protein komplekslerinin konformasyonsal ve termodinamik manzarasını yüksek çözünürlükte keşfetmek için tek moleküllü floresan bağlantılı optik cımbızın nasıl kullanılacağını göstermektedir. Örnek olarak, SARS-CoV-2 programlı ribozomal çerçeve değiştirme elemanının çinko-parmak antiviral proteininin trans-etkili faktör kısa izoformu ile etkileşimi ayrıntılı olarak açıklanmıştır. Ek olarak, floresan etiketli ribozomlar konfokal ünite kullanılarak izlendi ve bu da sonuçta çeviri uzamasının incelenmesini sağlayacaktı. Floresan bağlantılı OT tahlili, çeviriyi düzenleyen çeşitli RNA-protein komplekslerini veya trans-etkili faktörleri araştırmak için yaygın olarak uygulanabilir ve RNA tabanlı gen düzenleme çalışmalarını kolaylaştırabilir.
Giriş
Genetik bilgilerin DNA'dan proteinlere mRNA'lar yoluyla aktarılması, hücrelerin içindeki makromoleküler etkileşimler yoluyla tüm seviyelerde hassas bir şekilde düzenlenen karmaşık bir biyokimyasal süreçtir. Çevirisel düzenleme için, RNA-protein etkileşimleri çeşitli uyaranlara ve sinyallere hızlı bir şekilde tepki vermek için kritik bir rol oynar1,2. Bazı RNA-protein etkileşimleri mRNA stabilitesini etkiler ve böylece bir RNA'nın çevirisel olarak aktif olduğu zamanı değiştirir. Diğer RNA-protein etkileşimleri stop-codon readthrough, bypass veya programlanmış ribozomal frameshifting (PRF)3,4,5,6,7 gibi yeniden kodlama mekanizmaları ile ilişkilidir. Son zamanlarda, hücrede ne zaman ve ne kadar yeniden kodlama olacağını dikte etmek için uyarıcı mRNA elemanları ve çeviri makineleri ile etkileşime giren bir dizi RNA bağlayıcı protein (RBP) gösterilmiştir7,8,9,10,11. Bu nedenle, RNA bağlayıcı proteinlerin çevirideki rolünü ve RNA yapısını ve stabilitesini nasıl modüle ettiklerini incelemek için, bu RNA-protein komplekslerinin etkileşim ilkelerini ve mekanik özelliklerini ayrıntılı olarak incelemek çok önemlidir.
Onlarca yıllık çalışma, hız ve doğruluk elde etmek için çeviri makinelerinin RNA ve protein bileşenleri arasındaki karmaşık iletişime dayanan çok adımlı ve çok bileşenli çeviri sürecini incelemek için temel atmıştır12,13,14. Karmaşık düzenleyici olayları anlamanın önemli bir sonraki adımı, çeviri sırasındaki kuvvetleri, zaman ölçeklerini ve yapısal belirleyicileri yüksek hassasiyetle belirlemektir12,15,16,17. RNA konformasyonel dinamiklerinin incelenmesi ve özellikle trans-etkili yardımcı faktörlerin çeviri sırasında RNA yapısı üzerinde nasıl hareket ettiği, optik cımbız veya sıfır mod dalga kılavuzları dahil olmak üzere tek moleküllü araçların ortaya çıkmasıyla daha da aydınlatılmıştır16,17,18,19,20,21,22,23,24 ,25,26.
Optik cımbızlar (OT), transkripsiyon ve çeviri dahil olmak üzere birçok RNA'ya bağımlı dinamik süreci incelemek için uygulanan son derece hassas bir tek molekül tekniğini temsil eder26,27,28,29,30,31,32. Optik cımbız kullanımı moleküler etkileşimlerin, nükleik asit yapılarının ve termodinamik özelliklerin, kinetiğin ve enerjiklerinin ayrıntılı olarak araştırılmasına izin almıştır16,17,22,33,34,35,36,37,38,39 . Optik cımbız tahlilleri, mikroskobik nesnelerin odaklanmış bir lazer ışını ile tuzağa getirilmesine dayanır. Tipik bir OT deneyinde, ilgi molekülü iki şeffaf (genellikle polistiren) boncuk (Şekil 1A)27 arasında birbirine bağlıdır. Bu boncuklar daha sonra yaylar gibi davranan optik tuzaklar tarafından yakalanır. Böylece moleküle uygulanan kuvvet, boncukun odaklanmış lazer ışınının (tuzak merkezi) merkezinden yer değiştirmesi esas alınarak hesaplanabilir. Son zamanlarda optik cımbız konfokal mikroskopi (Şekil 1B) ile birleştirilerek floresan veya Förster rezonans enerji transferi (FRET) ölçümleri40,41,42 olarak mümkün olmuştur. Bu, eşzamanlı ölçüme ve dolayısıyla kuvvet spektroskopisi ve floresan verilerinin kesin korelasyonuna izin vererek yepyeni bir olası deney alanı açar.
Burada, çevirisel çerçevelemeyi düzenleyen protein-RNA etkileşimlerini incelemek için konfokal mikroskopi ile birlikte optik cımbız kullanarak deneyler gösteriyoruz. Amaç ve kondenser arasında, beş kanallı bir akış hücresi laminer akış ile sürekli numune uygulaması sağlar. Mikroakışkan kanallar aracılığıyla, çeşitli bileşenler doğrudan enjekte edilebilir, bu da uygulamalı zamanı azaltır ve deney boyunca çok az numune tüketimine izin verir.
İlk olarak, OT deneylerinin tasarımına yardımcı olacak temel bir kılavuz önerilmektedir ve avantajların yanı sıra çeşitli kurulumların tuzakları tartışılmaktadır. Daha sonra, örneklerin ve deneysel iş akışlarının hazırlanması açıklanır ve veri analizi için bir protokol sağlanır. Bir örneği temsil etmek için, sars-CoV-2 çerçeve değiştirme RNA elemanını (Şekil 2A) trans-etkili faktörle incelemek için RNA germe deneylerinden elde edilen sonuçları özetliyoruz, çinko-parmak antiviral proteininin (ZAP) kısa izoformunu, viral RNA'nın alternatif bir okuma çerçevesinden çevirisini değiştiren43. Ek olarak, çeviri makinelerinin prosesliğini ve hızını izlemek için yararlı olacak bu OT konfokal testinde floresan etiketli ribozomların kullanılabileceği gösterilmiştir. Burada sunulan yöntem, çevirinin çeşitli yönlerini incelemek için farklı tamponların, ligandların veya diğer hücresel bileşenlerin etkisini hızlı bir şekilde test etmek için kullanılabilir. Son olarak, yaygın deneysel tuzaklar ve bunların nasıl giderileceği tartışılır. Aşağıda, deneysel tasarımda bazı önemli noktalar özetlenmiştir.
Tasarım oluşturma
Prensip olarak, OT uyumlu bir RNA yapısı oluşturmak için iki ortak yaklaşım vardır. İlk yaklaşım, tamamlayıcı DNA tutamaçlarıyla melezlenen uzun bir RNA molekülü kullanmaktadır, böylece ortada tek iplikli bir RNA dizisini kuşatan iki RNA / DNA melez bölgesinden oluşan bir yapı elde edilir (Şekil 2B). Bu yaklaşım çoğu OT RNA deneyinde 33,44,45'te kullanılır.
İkinci yaklaşım, kısa (yaklaşık 20 nt) çıkıntilı dsDNA tutamaçlarından yararlanır15,17. Bu çıkıntılar daha sonra RNA molekülü ile melezlenir. Tasarımda daha karmaşık olmasına rağmen, dsDNA tutamaklarının kullanımı DNA / RNA-hibrid sisteminin bazı sınırlamalarının üstesinden geliyor. Prensip olarak, konfokal ölçümler için daha uygun olan çok uzun tutamaklar (>10kb) bile uygulanabilir. Ek olarak, RNA molekülü, bağ stabilitesini artırmak için DNA tutamaklarına bağlanabilir.
Son etiketleme stratejisi
Yapı, güçlü bir moleküler etkileşim yoluyla boncuklara bağlanmalıdır. Sapların boncuklara doğru kurcalanabilir bağlanması için yaklaşımlar mevcut olsa da, streptavidin-biotin ve digoksigenin-antikor gibi güçlü ancak yaygın olmayan etkileşimler OT deneylerinde yaygın olarak kullanılmaktadır15,33,35,45. Açıklanan protokolde, yapı biotin veya digoksigenin ile etiketlenmiştir ve boncuklar sırasıyla streptavidin veya digoksigenin'e karşı antikorlarla kaplanmıştır (Şekil 1A). Bu yaklaşım, yaklaşık 60 pN'a (bağlama başına)47'ye kadar kuvvet uygulamak için uygundur. Ayrıca, farklı 5' ve 3' etiketleme stratejilerinin kullanılması, boncuklar arasında oluşan bağın yönünün belirlenmesine izin verir17.
Floresan ölçümleri için protein etiketleme
Konfokal görüntüleme için floresan etiketleme için yaygın olarak kullanılan birkaç yaklaşım vardır. Örneğin, floroforlar proteinlerde doğal olarak bulunan veya reaktif organik bir grup aracılığıyla bölgeye yönlendirilmiş mutajensis tarafından tanıtılan amino asit kalıntılarına aktif olarak bağlanabilir. Sistein ve lizin kalıntılarının etiketlenebilmesi için sırasıyla tiyol veya amin reaktif boyalar kullanılabilir. Etiketlemenin özgüllüğünü artırmak için birkaç ters çevrilebilir koruma yöntemi vardır48,49, ancak yerel proteinler genellikle birden fazla kalıntıda etiketlenir. Floroforun küçük boyutu bir avantaj sağlasa da, spesifik olmayan etiketleme protein aktivitesini etkileyebilir ve bu nedenle sinyal yoğunluğu değişebilir49. Ayrıca, etiketleme verimliliğine bağlı olarak sinyal yoğunluğu farklı deneyler arasında farklılık gösterebilir. Bu nedenle, denemeden önce bir etkinlik denetimi yapılmalıdır.
İlgi proteini, His-tag veya strep-tag gibi bir N veya C-terminal etiketi içeriyorsa, bu etiketlerin özel etiketlemesi başka bir popüler yaklaşımı temsil eder. Ayrıca, etiket hedefli etiketleme, floroforun protein aktivitesini engelleme şansını azaltır ve çözünürlüğü artırabilir49. Bununla birlikte, etikete özgü etiketleme genellikle mono-florofor etiketli proteinler verir ve bu da tespit edilmesi zor olabilir. Spesifik etiketlemenin başka bir yolu antikorlar kullanılarak gerçekleştirilebilir.
Mikroakışkanlar kurulumu
OT'nin mikroakışkanlar sistemi ile kombinasyonu, farklı deneysel koşullar arasında hızlı bir geçiş sağlar. Ayrıca, akım sistemleri akış hücresinin içindeki laminar akışı korumaktan yararlanır, bu da diğer kanallardan gelen sıvıların akış yönüne göre dik yönde karıştırılmasını önler. Bu nedenle, laminar akış özellikle deneysel tasarım için avantajlıdır. Şu anda, 5 kanala kadar akış hücreleri yaygın olarak dirilmektedir (Şekil 3).
Protokol
1. Numune hazırlama
- İlgi sırasını, sap dizileri olarak hizmet eden Lambda DNA parçalarını içeren vektöre klonlayın (Şekil 2)43,50.
- İlk önce PCR ile sonraki in vitro transkripsiyon için bir DNA şablonu oluşturun (Şekil 2B; reaksiyon 1). Bu PCR adımında, T7 promotörü dna molekülünün 5' ucuna eklenir32,33,43,50. PCR reaksiyonlarını Tablo 1'e göre ayarlayın. PCR'ı termosikler içinde uygun döngülerle 50 μL aliquots'ta çalıştırın.
- Tutamaçları iki ayrı PCR reaksiyona göre hazırlayın (Tablo 1, Şekil 2B; reaksiyon 2 ve 3). İlk olarak, PCR ile 5' tutamacını oluşturun. Ardından, 3' tutamağı oluşturun ve aynı anda 5' digoksigenin etiketli bir astar32,33,43,50 kullanarak digoxigenin ile etiketlendir.
- PCR'den sonra, silika spin sütunlarını kullanarak DNA'yı arındırın.
- T7 RNA polimeraz kullanarak in vitro transkripsiyon reaksiyonunu gerçekleştirin (Tablo 2)32,33,43,50. Reaksiyonun 37 °C'de 2-4 saat boyunca RNA'nın uzunluğuna bağlı olarak kuluçkaya yatırın. Ardından, reaksiyona I. Dayı'yı ekleyin ve DNA şablonını sindirmek için 37 °C'de 30 dakika kuluçkaya yatırın. Silika döndürme sütunlarını kullanarak RNA'yı arındırın.
- 5' sapının etiketleme reaksiyonu sırasında (Tablo 3), kolun 3' ucuna T4 DNA polimeraz38,50 ile biotin-16-dUTP ekleyin. Reaksiyonun oda sıcaklığında 1-2 saat boyunca gerçekleştirilmesi. Daha sonra, silika spin sütunlarını kullanarak DNA'yı arındırın.
NOT: 5' tutamacın 3' ucunda (Şekil 2B) etiketli olması gerektiğinden, etiketleme PCR sırasında gerçekleştirilemez. - Yukarıda belirtilen bileşenleri karıştırın - 5' tutamak (3' biotin ile etiketlenmiş), 3' tutamak (5' digoksinin ile etiketlenmiş) ve RNA - tavlama tamponunda 1:1:1 molar oranında (%80 formamid, 400 mM NaCl, 40 mM HEPES, pH 7.5, 0.5 mM EDTA, pH 8), istenen RNA/DNA melezini elde etmek için (Tablo 4). Tavlama karışımını 10 dakika boyunca 85 °C'ye kadar ısıtın ve ardından yavaşça 4 °C'ye kadar soğutun.
- Tavlanmış numuneyi 1/10 hacimde 3 M sodyum asetat (pH 5), 3 hacim buz gibi etanol ile karıştırın ve -80 °C'de en az 1 saat veya -20 °C'de bir gecede kuluçkaya yatırın.
- Numuneleri 15.000 × g'da 4 °C'de 30 dakika santrifüj edin. Süpernatant atın ve peletin (genellikle görünmez) vakum altında kurutun.
- Son olarak, 50 μL RNase içermeyen suda pelet resuspend ve aliquots yapmak. Aliquotları kullanılana kadar -80 °C'de saklayın. Kısa süreli depolama için, numuneler -20 ° C'de de saklanabilir.
2. Enstrüman kurulumu
NOT: Aşağıdaki protokol LUMICKS şirketinin ticari optik cımbız cihazı C-Trap için optimize edilmiştir. Bu nedenle, diğer optik cımbız cihazları kullanırken sunulan adımlarda ayarlamalar gerekebilir. Kullanılmadığı takdirde, makinenin mikroakışkan sistemi çamaşır suyunda (sodyum hipoklorit çözeltisi) tutulur ve kullanılmadan önce yıkanmalıdır.
- Çamaşır suyunu atın ve şırınnaları 1 mL RNase içermeyen su ile doldurun.
- RNase içermeyen suyun en az 1 mL'sine 50 μL 0,5 M sodyum tiyosülfat ekleyin ve sistemde kalan çamaşır suyunu ortadan kaldırmak için sistemi (1 bar, en az 0,5 mL) iyice yıkayın.
- Sodyum tiyosülfat çözeltisini şırıngamlardan atın. Şırınnaları tazeleriyle değiştirin ve sistemi en az 0,5 mL RNase içermeyen suyla yıkayın.
NOT: Mikroakışkanlar sisteminin, sistemdeki hava kabarcıklarını önlemek için asla kuru çalışmasına dikkat edin. - Hedefin üzerine 2 damla daldırma yağı (1,33 kırma indisi) veya yaklaşık 70 μL su koyun.
- Akış hücresini tutma çerçevesinin içine kendi konumuna getirin.
- Akış hücresinin üzerine 2 damla daldırma yağı (1,51 kırma indeksi) koyun.
- Cımbız makinesindeki lazer cihazını açın. Çalıştırıldıktan sonra, yazılım arayüzündeki bindirme lazerini % 100 açın.
- Tanılama kameralarını (Z bulucu) kullanarak, mikro vidayı çevirerek kırılma halkalarının en büyük olduğu ikinci ve üçüncü yansımalar (arayüzler) arasındaki odanın ortasına Z eksenini ayarlayın.
NOT: Amaç ölçüm odasına her taşındında ve hedefin odak düzlemi arayüzü iki aşama arasında geçtiğinde, Z bulucu modunda bir yansıma tanınabilir. 4 arayüz mümkündür: (i) su/daldırma yağı ve alt cam (ii) odanın içindeki alt cam ve tampon (iii) odanın içindeki tampon ve kondenser için üst cam (iv) üst cam ve daldırma yağı. - Kondenser konumunu ayarlayın (bindirme lazerini yaklaşık% 50'ye ayarlayın), böylece kondenser ölçüm odasının üstündeki daldırma yağına dokunur.
- Kondenserle yavaşça aşağı/yukarı hareket derek odağı ayarlayın, böylece ay modunda yaklaşık 10 ışık bandı gösterilir (tanılama kameraları).
3. Numune ölçümü
- Örnek yapılarla (3 μL% 0.1 (w/v) AD boncuk süspansiyonu + 4 μL numune) ve 1 μL RNase inhibitörleri ve 8 μL tahlil tamponu (300 mM KCl, 5 mM MgCl2, 20 mM HEPES, pH 7.6, 0.05% Ara 20, 5 mM DTT) 10-20 dk için RT'de. Kuluçkadan sonra, numuneyi 500 μL test tamponunda seyreltin.
NOT: Oksidatif hasarı önlemek için özellikle floresan ölçümleri sırasında tampona oksijen leş yiyiciler eklenmesi önerilir. Burada glikoz (8.3 mg/mL), glikoz oksidaz (40 U/mL) ve katalaz (185 U/mL) içeren oksijen çöpçü sistemi kullanıldı. - 0,8 μL%1 (w/v) streptavidin kaplı (SA) boncukları 1 mL tahlil tamponu ile karıştırın.
- Şırınnalardan su atın ve şırınnaları ilgili süspansiyonlar / çözeltilerle doldurun. Yaklaşık 1 barda en az 2 dakika yıkayın ve ardından boncuk yakalamaya başlayın.
NOT: Deneysel kuruluma bağlı olarak farklı kanal düzenlemeleri kullanılabilir (Şekil 3). Tipik olarak, bir akış kanalı RNA molekülini taşıyan anti-digoksigenin boncukları ile doldurulur. İkinci bir kanal streptavidin kaplı boncuklarla doludur. Tampon kanalı, bağlamaları oluşturmak için kullanılır. RNA bağlama proteinini yüklemek için dördüncü bir kanal kullanılabilir (Şekil 3C) veya alternatif olarak RBP doğrudan tampon kanalına eklenebilir (Şekil 3B). - Boncukları yakalamak için optik tuzakları birbirinden ayırın. İlk olarak AD kanalına geçin ve tuzak 1'de bir AD-boncuk yakalayın. Ardından, sahneyi SA kanalına taşıyın ve tuzak 2 ile tek bir SA boncuk yakalayın.
NOT: Zaten yakalanmış olan boncukları kaybetmemek veya aynı tuzakla birden fazla boncuk yakalamayı önlemek için tampon ve boncuk kanallarının arayüzünde kalmaya çalışın. - Doğru boyuttaki boncuklar yakalandıktan sonra tampon kanalına geçin ve laminar akışı durdurun. Ardından, tuzak sertliğini kontrol etmek için kuvvet kalibrasyonu gerçekleştirin. İlgili sertlik değerleri x/y ekseninde% 10-15'ten fazla farklı olmamalıdır.
NOT: Lazer gücünü veya lazeri boncuk boyutuna göre tuzaklar arasında bölün. Boncuk şablonları eşleştiği sürece her boncuk çifti için kuvvet kalibrasyonu yapılması gerekmez (benzerlik puanı > 0,9). Ancak düzenli olarak veya en azından tahlil koşulları her değiştiğinde yapılmalıdır. - Boncukları birbirine yaklayarak, birkaç saniye bekleyerek ve sonra tekrar birbirinden ayırarak bir bağ için balık tutmaya başlayın, bir bağ oluşana kadar tekrarlayın. Bir bağ oluşumu, iki boncuk birbirinden uzaklaştıktan sonra ölçülen kuvvetin artmasına neden olur.
NOT: Birden fazla bağın oluşmasını önlemek için boncuklar çok yakına taşınmamalıdır. İki boncuk arasında bir bağ yakaladıktan sonra, aşırı uzanma platosu bulunarak bağlama kalitesi kontrol edilebilir. Plato tek bir bağ için 50 ila 60 pN arasında olmalıdır. - Bir bağ elde ettikten sonra ölçüme başlayın. Çalışılan olguya bağlı olarak farklı ölçüm kurulumları seçilmelidir (Şekil 1B-D).
NOT: Genellikle deneyin başında, bağlama kalitesini kontrol etmek ve davranışı araştırmak için bir kuvvet rampası deneyi yapılır. Daha sonra, durum geçişlerini daha fazla incelemek için sabit kuvvet veya sabit konum deneylerine de başlanabilir. Bir RNA örneğinde davranışını belirlemek için yeterli sayıda ölçüm yapıldıktan sonra, konfokal ölçümleri gerçekleştirmek için sisteme etiketli faktörler eklenebilir. - Floresan ölçümleri yapmak için optik cımbız cihazındaki konfokal lazerleri ve foton sayacı ünitesini açın.
- Yazılım arayüzünde istenen dalga boyunun uyarıcı lazerini açın ve florofora bağlı olarak lazerin gücünü% 5 veya daha yüksek olarak ayarlayın.
NOT: Ölçüm yapmamakla birlikte, numuneye aşırı fotodamajdan kaçınmak için heyecan lazerinin güç ayarını % 0'a düşürür. - Yazılımın görüntü işlevlerini kullanarak örneği görüntülemeye başlayın.
NOT: İyi odaklanmış görüntüler elde etmek için, konfokal mikroskop ve optik tuzakların odak düzleminin hizalanmış olması gerekir. Bu amaçla mavi lazer kanalındaki polistiren boncukların otoflüoresansı kullanılabilir. Optik tuzakların odak düzlemi, boncukların görüntüsü en yüksek çapa ulaşana kadar z ekseninde yukarı veya aşağı taşınır. Bu pozisyonda boncuklar arasında bağlı molekülden gelen floresan sinyali ölçülebilir. - Kimograf işlevini kullanmak için, kimograf ekseninin x-y konumunu belirtin, böylece boncuklar arasındaki bağın algılanmasını sağlar.
- Ölçüm boyunca, boncukların farklı kanallara taşınması veya mikroakışkanlar sisteminde sağlanan tamponun değiştirilmesiyle tampon bileşimi kolayca değiştirilebilir.
4. Veri analizi
- Ham veri ön işleme
- Basit bir komut dosyası kullanarak, ham verileri (i) daha hızlı sonraki veri işlemeye izin vermek, ancak (ii) hala tüm kritik bilgileri içerecek kadar altörnekleyin (Şekil 4A). Genellikle 100-5000 Hz bu amaç için uygundur.
NOT: Optik cımbız deneylerinde veri toplama sıklığı genellikle analiz için gerekli olandan daha yüksektir - sunulan deneylerde, veri toplama sıklığı varsayılan olarak 78 125 Hz olarak ayarlanır. Depolama alanı sınırlı olduğundan, verilerin örnekleme hızını azaltmak için kullanışlı ve zaman kazandırandır. Burada ham veriler 30 kat daha fazla altörneklendi. - Ardından, sinyalden gelen yüksek frekanslı ölçüm gürültüsünü azaltmak için bir sinyal filtresi kullanın (Şekil 4A). Farklı deneylerin veri çıkışını optimize etmek için filtre derecesini ve kesme frekans parametrelerini buna göre ayarlayın (Şekil 5).
NOT: Sinyal filtreleri arasında Butterworth filter51 en yaygın kullanılanlardan biridir. Ek verilerde ham verilerin önceden işlenmesine izin veren özel yazılmış bir python komut dosyası sağlanır. Altörnekleme ve sinyal filtreleme parametrelerinin (kesme frekansı, filtre derecesi) farklı deneyler için optimize edilmesi gerekir.
- Basit bir komut dosyası kullanarak, ham verileri (i) daha hızlı sonraki veri işlemeye izin vermek, ancak (ii) hala tüm kritik bilgileri içerecek kadar altörnekleyin (Şekil 4A). Genellikle 100-5000 Hz bu amaç için uygundur.
- Kuvvet rampası veri çözümlemesi için aşağıdaki adımları kullanın.
- Kuvvet yörüngesi çiziminde karşılık gelen noktaları bularak veya özel yazılmış komut dosyaları kullanarak adımları el ile işaretleyin. Açılma adımları, kuvvet mesafesi (FD) eğrisinde mesafe artışı ile birlikte ani bir kuvvet düşüşü ile karakterize edilir.
- Açılma olayları işaretlendikten sonra, uygun modelleri kullanarak FD eğrisinin farklı bölgelerine sığar (Şekil 4D).
NOT: İlk açılma adımından önceki bölge için, bağ "çift telli" olarak kabul edilebilir ve genişletilebilir bir Solucan benzeri zincir modeli (WLC)47,52,53 kullanılarak yaygın olarak uygundur. İlk açılma olayından sonraki parçalar, çift telli nükleotitler (tutamaklar) ve tek iplikli nükleotidlerin (açılmamış RNA molekülü) bir kombinasyonu olarak kabul edilir. Bu nedenle, veri uydurma daha karmaşıktır - genellikle 2 WLC modeli veya WLC ve Serbest eklemli zincir (FJC) modellerinin bir kombinasyonu 36,39,52 kullanılır. Genişletilebilir WLC modeli, kontur uzunluğu (LC) ve kalıcılık uzunluğu (LP) olmak için iki ana sığdırma parametresi vardır. Kontur uzunluğu tamamen gerilmiş molekülün uzunluğuna karşılık gelir ve kalıcılık uzunluğu, ilgi molekülünün bükülme özelliklerini tanımlar. Model aşağıdaki denklemle açıklanabilir (1). WLC, hem katlanmış hem de açılmış bölgelerin davranışını modellemek için kullanılabilir, ancak bunların her biri için farklı parametrelere sahip ayrı bir model kullanılması gerekir.
(1)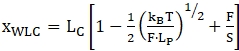
burada x uzatma, LC kontur uzunluğu, F kuvvet, LP kalıcılık uzunluğu, kB Boltzmann sabit, T Termodinamik sıcaklık ve S streç modülüdür.
Serbest eklemli zincir (FJC) adı verilen ikinci model, açılmamış tek iplikli bölgelerin davranışını tanımlamak için yaygın olarak kullanılır. Polimerlerin benzer parametrelerini kullanır, ancak "zincirin" her bir birimini, burada açılmış tek iplikli bölgenin nükleotitlerine karşılık gelen sert bir çubuk olarak değerlendirir. Aşağıdaki denklem (2) bu modeli açıklar:
(2)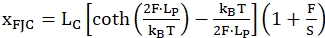
NOT: Laboratuvarımız son zamanlarda "Pratik Optik Cımbız Analizi TOol (PATATES)54" adı verilen ham kuvvet rampası verilerinin toplu olarak işlenmesine izin veren bir algoritma geliştirmiştir. Algoritma verileri altörnekler ve filtreler, daha sonra olası açılma adımlarını tanımlar ve son olarak veri sığdırma gerçekleştirir. PATATES, kullanıcı dostu bir grafik kullanıcı arabiriminde (GUI) (https://github.com/REMI-HIRI/POTATO) oluşturulmuştır.
- Sabit kuvvet verilerini aşağıdaki gibi işleyin:
NOT: Aşağıdaki talimatlar sabit konum verilerine analojik olarak uygulanabilir.- Sabit kuvvet verileri için, zaman içinde mesafeyi çizin (Şekil 5). Pozisyondaki göreceli değişime göre farklı konformasyonların sıklığını (sayılarını) gösteren bir histogram, çeşitli baskın ve küçük durumları karakterize etmenin yararlı bir yoludur (Şekil 7).
- Belirli bir kuvvetteki bireysel konformatörlerin genel yüzdesini tahmin etmek için (birden fazla) Gauss fonksiyonunu kullanarak histogramı takın (Şekil 7C). Gauss uyum, ortalama konum ve standart sapma farklı popülasyonlar arasındaki kuvvetle ilgili ilişkiyi özetler.
NOT: Ek verilerde, sabit kuvvet verilerinin ön işleme ve temel bimodal Gauss montajına izin veren özel olarak yazılmış bir python komut dosyası sağlanır. Parametrelerin (kesme frekansı, filtre derecesi, beklenen araçlar, standart sapma değerleri ve genlikler) farklı deneyler için optimize edilmesi gerekir. - Daha sonra, ek katlanır ara maddeleri (konformatörler)55'i ortaya çıkarabilecek durumları daha fazla analiz etmek için Gizli Markov modelini kullanın. Sabit kuvvet ve Gizli Markov modeli hakkında daha fazla bilgi için 55,56,57,58'e atıfta bulunulabilir.
Sonuçlar
Bu bölümde ağırlıklı olarak floresan optik cımbız ile RNA-protein/ligand etkileşimlerinin ölçümlerine odaklanılmalıdır. Genel RNA optik cımbız deneylerinin ve buna karşılık gelen temsili sonuçların açıklaması için bkz. RNA/DNA-protein etkileşimlerinin daha ayrıntılı tartışılması için ayrıca bkz.
...Tartışmalar
Burada, çeşitli ligandlarla RNA moleküllerinin etkileşimlerini ve dinamik davranışlarını incelemek için floresan bağlantılı optik cımbızların kullanımını gösteriyoruz. Aşağıda, mevcut tekniğin kritik adımları ve sınırlamaları tartışılmıştır.
Protokolde kritik adımlar
Diğer birçok yöntemde olduğu gibi, numunenin kalitesi güvenilir veri elde etmek için çok önemlidir. Bu nedenle, mümkün olan en yüksek kalitede numuneler elde etmek ...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Anuja Kibe ve Jun. Prof. Redmond Smyth'e makaleyi eleştirel bir şekilde inceledikleri için teşekkür ederiz. Uzman teknik yardım için Tatyana Koch'a teşekkür ederiz. Kristyna Pekarkova'ya deneysel videoların kaydediltiren yardımı için teşekkür ederiz. Laboratuvarımızdaki çalışmalar Helmholtz Derneği tarafından desteklenmekte ve Avrupa Araştırma Konseyi (ERC) Grant Nr. 948636 (NC'ye) fon sağlamıştır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| E. coli HB101 | lab collection | N/A | cloning of the vectors |
| Chemicals and enzymes | |||
| Sodium chloride | Sigma-Aldrich | 31424 | Buffers |
| Biotin-16-dUTP | Roche | 11093070910 | Biotinylation |
| BSA | Sigma-Aldrich | A4737 | Buffers |
| Catalase | Lumicks | N/A | Oxygen scavanger system |
| Dithiothreitol (DTT) | Melford Labs | D11000 | Buffers |
| DNAse I from bovine pancreas | Sigma-Aldrich | D4527 | in vitro transcription |
| dNTPs | Th.Geyer | 11786181 | PCR |
| EDTA | Sigma-Aldrich | E9884 | Buffers |
| Formamide | Sigma-Aldrich | 11814320001 | Buffers |
| Glucose | Sigma-Aldrich | G8270-1KG | Oxygen scavanger system |
| Glucose-oxidase | Lumicks | N/A | Oxygen scavanger system |
| HEPES | Carl Roth | HN78.3 | Buffers |
| Magnesium chloride | Carl Roth | 2189.1 | Buffers |
| Phusion DNA polymerase | NEB | M0530L | Gibson assembly, cloning |
| Potassium chloride | Merck | 529552-1KG | Buffers |
| PrimeSTAR GXL DNA Polymerase | Takara Bio Clontech | R050A | PCR |
| Pyrophosphotase, thermostabile, inorganic | NEB | M0296L | in vitro transcription |
| RNase Inhibitor | Molox | 1000379515 | Buffers |
| rNTPS | life technologies | R0481 | in vitro transcription |
| Sodium thiosulophate | Sigma-Aldrich | S6672-500G | Bleach deactivation |
| Sytox Green | Lumicks | N/A | confocal measurements |
| T4 DNA Polymerase | NEB | M0203S | Biotinylation |
| T5 exonuclease | NEB | M0363S | Gibson assembly, cloning |
| T7 RNA polymerase | Produced in-house | N/A | in vitro transcription |
| Taq DNA polymerase | NEB | M0267S | PCR |
| Taq ligase | Biozym | L6060L | Gibson assembly, cloning |
| TWEEN 20 BioXtra | Sigma-Aldrich | P7949 | Buffers |
| Kits | |||
| Monolith Protein Labeling Kit RED-NHS 2nd Generation (Amine Reactive) | Nanotemper | MO-L011 | Used for ribosome labeling |
| Purefrex 2.0 | GeneFrontier | PF201-0.25-EX | Ribosomes used for the labeling |
| Oligonucleotides | |||
| 5' handle T7 forward | Microsynth | custom order | 5’ - CTTAATACGACTCACTATAGGTC CTTTCTGTGGACGCC - 3’, used to generate OT in vitro transcription template in PCR 1 |
| 3’ handle reverse | Microsynth | custom order | 5' - GTCAAAGTGCGCCCCGTTATCC - 3', used to generate OT in vitro transcription template in PCR 1 |
| 5' handle forward | Microsynth | custom order | 5' - TCCTTTCTGTGGACGCCGC - 3' , used to generate 5' handle in PCR 2 |
| 5’ handle reverse | Microsynth | custom order | 5’ - CATAAATACCTCTTTACTAATATA TATACCTTCGTAAGCTAGCGT - 3’, used to generate 5' handle in PCR 2 |
| 3’ handle forward | Microsynth | custom order | 5' - ATCCTGCAACCTGCTCTTCGCC AG - 3', used to generate 3' handle in PCR 3 |
| 3’ handle reverse 5’labeled with digoxigenin | Microsynth | custom order | 5' -[Dig]-GTCAAAGTGCGCCCCGTTATCC - 3', used to generate 3' handle in PCR 3 |
| DNA vectors | |||
| pMZ_OT | produced in-house | N/A | further description in "Structural studies of Cardiovirus 2A protein reveal the molecular basis for RNA recognition and translational control" Chris H. Hill, Sawsan Napthine, Lukas Pekarek, Anuja Kibe, Andrew E. Firth, Stephen C. Graham, Neva Caliskan, Ian Brierley bioRxiv 2020.08.11.245035; doi: https://doi.org/10.1101/2020.08.11.245035 |
| Software and Algorithms | |||
| Atom | https://atom.io/packages/ide-python | N/A | |
| Bluelake | Lumicks | N/A | |
| Graphpad | https://www.graphpad.com/ | N/A | |
| InkScape 0.92.3 | https://inkscape.org/ | N/A | |
| Matlab | https://www.mathworks.com/products/matlab.html | N/A | |
| POTATO | https://github.com/lpekarek/POTATO.git | N/A | |
| RNAstructure | https://rna.urmc.rochester.edu/RNAstructure.html | N/A | |
| Spyder | https://www.spyder-ide.org/ | N/A | |
| Other | |||
| Streptavidin Coated Polystyrene Particles, 1.5-1.9 µm, 5 ml, 1.0% w/v | Spherotech | SVP-15-5 | |
| Anti-digoxigenin Coated Polystyrene Particles, 2.0-2.4 µm, 2 ml, 0.1% w/v | Spherotech | DIGP-20-2 | |
| Syringes | VWR | TERUMO SS+03L1 | |
| Devices | |||
| C-trap | Lumicks | N/A | optical tweezers coupled with confocal microscopy |
Referanslar
- Balcerak, A., Trebinska-Stryjewska, A., Konopinski, R., Wakula, M., Grzybowska, E. A. RNA-protein interactions: disorder, moonlighting and junk contribute to eukaryotic complexity. Open Biology. 9 (6), 190096 (2019).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Firth, A. E., Brierley, I. Non-canonical translation in RNA viruses. Journal of General Virology. 93, 1385-1409 (2012).
- Caliskan, N., Peske, F., Rodnina, M. V. Changed in translation: mRNA recoding by −1 programmed ribosomal frameshifting. Trends in Biochemical Sciences. 40 (5), 265-274 (2015).
- Jaafar, Z. A., Kieft, J. S. Viral RNA structure-based strategies to manipulate translation. Nature Reviews Microbiology. 17 (2), 110-123 (2019).
- Eswarappa, S. M., et al. Programmed translational readthrough generates antiangiogenic VEGF-Ax. Cell. 157 (7), 1605-1618 (2014).
- Rodnina, M. V., et al. Translational recoding: canonical translation mechanisms reinterpreted. Nucleic Acids Research. 48 (3), 1056-1067 (2020).
- Li, Y., et al. Transactivation of programmed ribosomal frameshifting by a viral protein. Proceedings of the National Academy of Sciences. 111 (21), 2172 (2014).
- Napthine, S., et al. Protein-directed ribosomal frameshifting temporally regulates gene expression. Nature Communications. 8 (1), 15582 (2017).
- Patel, A., et al. Molecular characterization of the RNA-protein complex directing -2/-1 programmed ribosomal frameshifting during arterivirus replicase expression. Journal of Biological Chemistry. 295 (52), 17904-17921 (2020).
- Napthine, S., Bell, S., Hill, C. H., Brierley, I., Firth, A. E. Characterization of the stimulators of protein-directed ribosomal frameshifting in Theiler's murine encephalomyelitis virus. Nucleic Acids Research. 47 (15), 8207-8223 (2019).
- Marshall, R. A., Aitken, C. E., Dorywalska, M., Puglisi, J. D. Translation at the Single-Molecule Level. Annual Review of Biochemistry. 77 (1), 177-203 (2008).
- Rodnina, M. V. The ribosome in action: Tuning of translational efficiency and protein folding. Protein science : A publication of the Protein Society. 25 (8), 1390-1406 (2016).
- Rodnina, M. V., Fischer, N., Maracci, C., Stark, H. Ribosome dynamics during decoding. Philosophical Transactions of Royal Society of London B Biological Sciences. 372 (1716), (2017).
- Yan, S., Wen, J. D., Bustamante, C., Tinoco, I. Ribosome excursions during mRNA translocation mediate broad branching of frameshift pathways. Cell. 160 (5), 870-881 (2015).
- Liu, T., et al. Direct measurement of the mechanical work during translocation by the ribosome. eLife. 3, 03406 (2014).
- Desai, V. P., et al. Co-temporal force and fluorescence measurements reveal a ribosomal gear shift mechanism of translation regulation by structured mRNAs. Molecular Cell. 75 (5), 1007-1019 (2019).
- Choi, J., O'Loughlin, S., Atkins, J. F., Puglisi, J. D. The energy landscape of -1 ribosomal frameshifting. Science Advances. 6 (1), (2020).
- Prabhakar, A., Puglisi, E. V., Puglisi, J. D. Single-molecule fluorescence applied to translation. Cold Spring Harbor Perspectives in Biology. 11 (1), 032714 (2019).
- Bao, C., et al. mRNA stem-loops can pause the ribosome by hindering A-site tRNA binding. Elife. 9, 55799 (2020).
- Chen, J., Tsai, A., O'Leary, S. E., Petrov, A., Puglisi, J. D. Unraveling the dynamics of ribosome translocation. Current Opinion in Structural Biology. 22 (6), 804-814 (2012).
- Qu, X., et al. The ribosome uses two active mechanisms to unwind messenger RNA during translation. Nature. 475 (7354), 118-121 (2011).
- Zheng, Q., et al. Ultra-stable organic fluorophores for single-molecule research. Chemical Society Reviews. 43 (4), 1044-1056 (2014).
- Blanchard, S. C. Single-molecule observations of ribosome function. Current Opinion in Structural Biology. 19 (1), 103-109 (2009).
- Juette, M. F., et al. The bright future of single-molecule fluorescence imaging. Current Opinion in Chemical Biology. 20, 103-111 (2014).
- McCauley, M. J., Williams, M. C. Mechanisms of DNA binding determined in optical tweezers experiments. Biopolymers. 85 (2), 154-168 (2007).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters. 11 (5), 288-290 (1986).
- Bustamante, C., Smith, S. B., Liphardt, J., Smith, D. Single-molecule studies of DNA mechanics. Current Opinion in Structural Biology. 10 (3), 279-285 (2000).
- Choudhary, D., Mossa, A., Jadhav, M., Cecconi, C. Bio-molecular applications of recent developments in optical tweezers. Biomolecules. 9 (1), 23 (2019).
- Moffitt, J. R., Chemla, Y. R., Smith, S. B., Bustamante, C. Recent advances in optical tweezers. Annual Reviews of Biochemistry. 77, 205-228 (2008).
- Li, P. T. X., Vieregg, J., Tinoco, I. How RNA Unfolds and Refolds. Annual Review of Biochemistry. 77 (1), 77-100 (2008).
- Stephenson, W., Wan, G., Tenenbaum, S. A., Li, P. T. Nanomanipulation of single RNA molecules by optical tweezers. Journal of Visualized Experiments. (90), e51542 (2014).
- Halma, M. T. J., Ritchie, D. B., Cappellano, T. R., Neupane, K., Woodside, M. T. Complex dynamics under tension in a high-efficiency frameshift stimulatory structure. Proceedings of the National Academy of Sciences. 116 (39), 19500 (2019).
- Hansen, T. M., Reihani, S. N. S., Oddershede, L. B., Sørensen, M. A. Correlation between mechanical strength of messenger RNA pseudoknots and ribosomal frameshifting. Proceedings of the National Academy of Sciences of the United States of America. 104 (14), 5830-5835 (2007).
- Zhong, Z., et al. Mechanical unfolding kinetics of the SRV-1 gag-pro mRNA pseudoknot: possible implications for -1 ribosomal frameshifting stimulation. Science Reports. 6, 39549 (2016).
- McCauley, M. J., Rouzina, I., Li, J., Núñez, M. E., Williams, M. C. Significant differences in RNA structure destabilization by HIV-1 GagDp6 and NCp7 proteins. Viruses. 12 (5), 484 (2020).
- de Messieres, M., et al. Single-molecule measurements of the CCR5 mRNA unfolding pathways. Biophysics Journal. 106 (1), 244-252 (2014).
- Yang, L., et al. Single-molecule mechanical folding and unfolding of RNA hairpins: Effects of single A-U to A·C pair substitutions and single proton binding and implications for mRNA structure-induced -1 ribosomal frameshifting. Journal of American Chemical Society. 140 (26), 8172-8184 (2018).
- McCauley, M. J., et al. Targeted binding of nucleocapsid protein transforms the folding landscape of HIV-1 TAR RNA. Proceedings of the National Academy of Sciences of the United States of America. 112 (44), 13555-13560 (2015).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. High-resolution "Fleezers": Dual-trap optical tweezers combined with single-molecule fluorescence detection. Methods in Molecular Biology. 1486, 183-256 (2017).
- Yerramilli, V. S., Kim, K. H. Labeling RNAs in live cells using malachite green aptamer scaffolds as fluorescent probes. ACS Synthetic Biology. 7 (3), 758-766 (2018).
- Gross, P., Farge, G., Peterman, E. J., Wuite, G. J. Combining optical tweezers, single-molecule fluorescence microscopy, and microfluidics for studies of DNA-protein interactions. Methods in Enzymology. 475, 427-453 (2010).
- Zimmer, M. M., et al. The short isoform of the host antiviral protein ZAP acts as an inhibitor of SARS-CoV-2 programmed ribosomal frameshifting. Nature Communications. 12 (1), 7193 (2021).
- Neupane, K., Yu, H., Foster, D. A. N., Wang, F., Woodside, M. T. Single-molecule force spectroscopy of the add adenine riboswitch relates folding to regulatory mechanism. Nucleic acids research. 39 (17), 7677-7687 (2011).
- Ritchie, D. B., Soong, J., Sikkema, W. K., Woodside, M. T. Anti-frameshifting ligand reduces the conformational plasticity of the SARS virus pseudoknot. Journal of the American Chemical Society. 136 (6), 2196-2199 (2014).
- Janissen, R., et al. Invincible DNA tethers: covalent DNA anchoring for enhanced temporal and force stability in magnetic tweezers experiments. Nucleic Acids Research. 42 (18), 137 (2014).
- Smith, S. B., Cui, Y., Bustamante, C. Overstretching B-DNA: The elastic response of individual double-stranded and single-stranded DNA molecules. Science. 271 (5250), 795 (1996).
- Puljung, M. C., Zagotta, W. N. Labeling of specific cysteines in proteins using reversible metal protection. Biophysical Journal. 100 (10), 2513-2521 (2011).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Hill, C. H., et al. Structural and molecular basis for Cardiovirus 2A protein as a viral gene expression switch. Nature Communications. 12 (1), 7166 (2021).
- Butterworth, S. On the theory of filter amplifiers. Experimental Wireless and the Wireless Engineer. 7, 536-541 (1930).
- Wang, M. D., Yin, H., Landick, R., Gelles, J., Block, S. M. Stretching DNA with optical tweezers. Biophysics Journal. 72 (3), 1335-1346 (1997).
- Mukhortava, A., et al. Structural heterogeneity of attC integron recombination sites revealed by optical tweezers. Nucleic Acids Research. 47 (4), 1861-1870 (2019).
- Buck, S., Pekarek, L., Caliskan, N. POTATO: An automated pipeline for batch analysis of optical tweezers data. bioRxiv. , (2021).
- Zhang, Y., Jiao, J., Rebane, A. A. Hidden Markov modeling with detailed balance and its application to single protein folding. Biophysical Journal. 111 (10), 2110-2124 (2016).
- Sgouralis, I., Pressé, S. An introduction to infinite HMMs for single-molecule data analysis. Biophysics Journal. 112 (10), 2021-2029 (2017).
- Müllner, F. E., Syed, S., Selvin, P. R., Sigworth, F. J. Improved hidden Markov models for molecular motors, part 1: basic theory. Biophysical Journal. 99 (11), 3684-3695 (2010).
- Elms, P. J., Chodera, J. D., Bustamante, C. J., Marqusee, S. Limitations of constant-force-feedback experiments. Biophysical Journal. 103 (7), 1490-1499 (2012).
- Re, A., Joshi, T., Kulberkyte, E., Morris, Q., Workman, C. T. RNA-protein interactions: an overview. Methods Molecular Biology. 1097, 491-521 (2014).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature reviews. Molecular Cell Biology. 16 (9), 533-544 (2015).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Sunbul, M., Jäschke, A. SRB-2: a promiscuous rainbow aptamer for live-cell RNA imaging. Nucleic Acids Research. 46 (18), 110 (2018).
- Sanchez de Groot, N., et al. RNA structure drives interaction with proteins. Nature Communications. 10 (1), 3246 (2019).
- Zeffman, A., Hassard, S., Varani, G., Lever, A. The major HIV-1 packaging signal is an extended bulged stem loop whose structure is altered on interaction with the Gag polyprotein. Journal of Molecular Biology. 297 (4), 877-893 (2000).
- Mangeol, P., et al. Probing ribosomal protein-RNA interactions with an external force. Proceedings of the National Academy of Sciences. 108 (45), 18272 (2011).
- Luo, X., et al. Molecular mechanism of RNA recognition by Zinc-Finger antiviral protein. Cell Reports. 30 (1), 46-52 (2020).
- Qu, X., Lancaster, L., Noller, H. F., Bustamante, C., Tinoco, I. Ribosomal protein S1 unwinds double-stranded RNA in multiple steps. Proceedings of the National Academy of Science U. S. A. 109 (36), 14458-14463 (2012).
- Chandra, V., Hannan, Z., Xu, H., Mandal, M. Single-molecule analysis reveals multi-state folding of a guanine riboswitch. Nature Chemical Biology. 13 (2), 194-201 (2017).
- Savinov, A., Perez, C. F., Block, S. M. Single-molecule studies of riboswitch folding. Biochimica et Biophysica Acta. 1839 (10), 1030-1045 (2014).
- Kelly, J. A., et al. Structural and functional conservation of the programmed ribosomal frameshift signal of SARS coronavirus 2 (SARS-CoV-2). Journal of Biological Chemistry. 295 (31), 10741-10748 (2020).
- Neupane, K., et al. Structural dynamics of single SARS-CoV-2 pseudoknot molecules reveal topologically distinct conformers. Nature Communications. 12 (1), 4749 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zheng, Q., Jockusch, S., Zhou, Z., Blanchard, S. C. The contribution of reactive oxygen species to the photobleaching of organic fluorophores. Photochemistry and Photobiology. 90 (2), 448-454 (2014).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Rill, N., Mukhortava, A., Lorenz, S., Tessmer, I. Alkyltransferase-like protein clusters scan DNA rapidly over long distances and recruit NER to alkyl-DNA lesions. Proceedings of the National Academy of Science U. S. A. 117 (17), 9318-9328 (2020).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Wen, J. -. D., et al. Force unfolding kinetics of RNA using optical tweezers. I. Effects of experimental variables on measured results. Biophysical journal. 92 (9), 2996-3009 (2007).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır