É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Pinças ópticas para estudar interações RNA-proteína no regulamento de tradução
Neste Artigo
Resumo
Este protocolo apresenta um fluxo de trabalho experimental completo para estudar interações RNA-proteína usando pinças ópticas. Várias possíveis configurações experimentais são delineadas, incluindo a combinação de pinças ópticas com microscopia confocal.
Resumo
O RNA adota diversas dobras estruturais, essenciais para suas funções e, assim, podem impactar diversos processos na célula. Além disso, a estrutura e a função de um RNA podem ser moduladas por vários fatores trans-acionados, como proteínas, metabólitos ou outras RNAs. As moléculas de RNA de mudança de quadro, por exemplo, são RNAs regulatórias localizadas em regiões de codificação, que direcionam ribossomos de tradução em um quadro de leitura aberto alternativo e, assim, agem como interruptores genéticos. Eles também podem adotar diferentes dobras após a ligação a proteínas ou outros fatores trans. Para dissecar o papel das proteínas vinculantes ao RNA na tradução e como elas modulam a estrutura e a estabilidade do RNA, é crucial estudar as características interpulsão e mecânica desses complexos de proteína RNA simultaneamente. Este trabalho ilustra como empregar pinças ópticas acopladas a uma molécula única para explorar a paisagem conformacional e termodinâmica dos complexos de proteína RNA em alta resolução. Como exemplo, elabora-se a interação do elemento ribossômico programado SARS-CoV-2 com o fator trans-ação de isoforme curto da proteína antiviral de dedo de zinco. Além disso, ribossomos rotulados por fluorescência foram monitorados utilizando-se a unidade confocal, o que, em última análise, permitiria o estudo do alongamento da tradução. O ensaio de fluorescência acoplado de OT pode ser amplamente aplicado para explorar diversos complexos de proteína RNA ou fatores trans-atuantes que regulam a tradução e poderia facilitar estudos de regulação genética baseada em RNA.
Introdução
A transferência de informações genéticas do DNA para as proteínas através dos mRNAs é um processo bioquímico complexo, que é precisamente regulado em todos os níveis através de interações macromoleculares dentro das células. Para a regulação translacional, as interações RNA-proteína conferem um papel crítico para reagir rapidamente a vários estímulos e sinais1,2. Algumas interações RNA-proteína afetam a estabilidade do mRNA e, assim, alteram o tempo em que um RNA é tratilicamente ativo. Outras interações RNA-proteína estão associadas a mecanismos de recodificação, como leitura stop-codon, bypass ou mudança de quadro ribossômico programada (PRF)3,4,5,6,7. Recentemente, uma série de proteínas de ligação de RNA (RBPs) foram demonstradas para interagir com elementos estimulantes de mRNA e a máquina de tradução para ditar quando e quanta reco codificação ocorrerá nas células7,8,9,10,11. Assim, dissecar o papel das proteínas vinculantes ao RNA na tradução e como elas modulam a estrutura e a estabilidade do RNA, é fundamental estudar os princípios de interação e propriedades mecânicas desses complexos de proteína RNA em detalhes.
Décadas de trabalho estabeleceram as bases para estudar o processo de tradução multi-passo e multicomponente, que conta com a comunicação intrincada entre o RNA e componentes proteicos da máquina de tradução para alcançar velocidade e precisão12,13,14. Um próximo passo crucial na compreensão de eventos regulatórios complexos é determinar as forças, escalas de tempo e determinantes estruturais durante a tradução em alta precisão12,15,16,17. O estudo da dinâmica conformacional do RNA e especialmente como fatores auxiliares trans-acionam na estrutura do RNA durante a tradução foram ainda mais iluminados pelo surgimento de ferramentas de molécula única, incluindo pinças ópticas ou guias de ondas de modo zero16,17,18,19,20,21,22,23,24 25,26.
A pinça óptica (OT) representa uma técnica de molécula única altamente precisa, que tem sido aplicada para estudar muitos tipos de processos dinâmicos dependentes de RNA, incluindo transcrição, e tradução26,27,28,29,30,31,32. O uso de pinças ópticas permitiu a sondagem de interações moleculares, estruturas de ácido nucleico e propriedades termodinâmicas, cinéticas e energéticas desses processos em detalhes16,17,22,33,34,35,36,37,38,39 . O ensaio óptico da pinça é baseado na armadilha de objetos microscópicos com um raio laser focado. Em um experimento típico de OT, a molécula de interesse é amarrada entre duas contas transparentes (geralmente poliestireno) (Figura 1A)27. Essas contas são então capturadas por armadilhas ópticas, que se comportam como molas. Assim, a força aplicada na molécula pode ser calculada com base no deslocamento da conta do centro do raio laser focal focado (centro da armadilha). Recentemente, pinças ópticas foram combinadas com microscopia confocal (Figura 1B), permitindo medidas de fluorescência ou transferência de energia de ressonância Förster (FRET)40,41,42. Isso abre um novo campo de possíveis experimentos permitindo medição simultânea e, portanto, correlação precisa de dados de espectroscopia de força e fluorescência.
Aqui, demonstramos experimentos usando as pinças ópticas combinadas com microscopia confocal para estudar interações proteína-RNA regulando a mudança de quadro translacional. Entre o objetivo e o condensador, uma célula de fluxo com cinco canais permite a aplicação contínua da amostra com fluxo laminar. Através dos canais microfluidos, vários componentes podem ser injetados diretamente, o que diminui o tempo prático, além de permitir muito pouco consumo amostral ao longo do experimento.
Em primeiro lugar, uma diretriz básica para auxiliar o desenho de experimentos OT é proposta e vantagens, bem como armadilhas de várias configurações são discutidas. Em seguida, são descritas amostras e fluxos de trabalho experimentais e um protocolo para a análise dos dados. Para representar um exemplo, descrevemos os resultados obtidos a partir de experimentos de alongamento de RNA para estudar o elemento RNA de mudança de quadro SARS-CoV-2 (Figura 2A) com o fator trans-ação o isoforme curto da proteína antiviral de dedo de zinco (ZAP), que altera a tradução do RNA viral de um quadro de leitura alternativo43. Além disso, demonstra-se que ribossósicos rotulados por fluorescência podem ser empregados neste ensaio confocal OT, o que seria útil para monitorar a processividade e a velocidade da máquina de tradução. O método aqui apresentado pode ser usado para testar rapidamente o efeito de diferentes buffers, ligantes ou outros componentes celulares para estudar vários aspectos da tradução. Finalmente, são discutidas armadilhas experimentais comuns e como solucioná-las. Abaixo, alguns pontos cruciais no design experimental são delineados.
Projeto de construção
Em princípio, existem duas abordagens comuns para criar um RNA compatível com OT. A primeira abordagem emprega uma molécula de RNA longa que é hibridizada com alças de DNA complementares, produzindo assim um construto composto por duas regiões híbridas de RNA/DNA flanqueando uma sequência de RNA de uma única linha no meio (Figura 2B). Essa abordagem é empregada na maioria dos experimentos de RNA OT33,44,45.
A segunda abordagem aproveita as alças dsDNA com saliências curtas (em torno de 20 nt) 15,17. Essas saliências são então hibridizadas com a molécula de RNA. Embora mais complicado no design, o uso de alças dsDNA supera algumas das limitações do sistema DNA/RNA-híbrido. Em princípio, mesmo alças muito longas (>10kb) podem ser implementadas, o que é mais conveniente para medições confocal. Além disso, a molécula de RNA pode ser ligada às alças de DNA para aumentar a estabilidade das amarras.
Estratégia de rotulagem final
A construção deve ser amarrada a contas através de uma forte interação molecular. Embora existam abordagens disponíveis para a ligação covalente de alças a contas46, interações fortes, mas não covalentes, como streptavidin-biotina e digoxigenina-anticorpo são comumente usadas em experimentos OT15,33,35,45. No protocolo descrito, a construção é rotulada com biotina ou digoxigenina, e as contas são revestidas com streptavidina ou anticorpos contra digoxigenina, respectivamente (Figura 1A). Esta abordagem seria adequada para a aplicação de forças de até aproximadamente 60 pN (por tether)47. Além disso, o uso de diferentes estratégias de rotulagem de 5' e 3' permitem determinar a orientação da corda formada entre as contas17.
Rotulagem de proteínas para medidas de fluorescência
Para a imagem confocal, existem várias abordagens comumente utilizadas para rotulagem de fluorescência. Por exemplo, fluoroforos podem ser covalentemente ligados a resíduos de aminoácidos que são encontrados nativamente em proteínas ou introduzidos por mutagênese direcionada ao local através de um grupo orgânico reativo. Corantes de tiol ou amina-reativa podem ser usados para rotulagem de resíduos de cisteína e liseina, respectivamente. Existem vários métodos de proteção reversíveis para aumentar a especificidade da rotulagem48,49, porém as proteínas nativas normalmente seriam rotuladas em múltiplos resíduos. Embora o pequeno tamanho do fluorohore possa conferir uma vantagem, a rotulagem não específica pode interferir na atividade proteica e, portanto, a intensidade do sinal pode variar49. Além disso, dependendo da intensidade do sinal de eficiência de rotulagem pode diferir entre diferentes experimentos. Portanto, uma verificação de atividade deve ser realizada antes do experimento.
Caso a proteína de interesse contenha uma tag terminal N ou C, como uma tag His ou marca de estreptococos, a rotulagem específica dessas tags representa outra abordagem popular. Além disso, a rotulagem direcionada a tags reduz a chance do fluorohore interferir na atividade proteica e pode aumentar a solubilidade49. No entanto, a rotulagem específica de etiquetas geralmente produz proteínas rotuladas mono-fluorforo, o que pode ser desafiador de detectar. Outra forma de rotulagem específica pode ser realizada empregando anticorpos.
Configuração de microfluidos
A combinação de OT com um sistema de microfluidos permite uma transição rápida entre diferentes condições experimentais. Além disso, os sistemas atuais aproveitam a manutenção do fluxo laminar dentro da célula de fluxo, o que impede a mistura de líquidos de outros canais na direção perpendicular em relação à direção de fluxo. Portanto, o fluxo laminar é particularmente vantajoso para o design experimental. Atualmente, células de fluxo com até 5 canais são comumente empregadas (Figura 3).
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparação da amostra
- Clone a sequência de interesse no vetor contendo os fragmentos de DNA lambda, que servem como sequências de alça (Figura 2)43,50.
- Primeiro gerar um modelo de DNA para transcrição in vitro subsequente via PCR (Figura 2B; reação 1). Nesta etapa do PCR, o promotor T7 é adicionado no final de 5' da molécula de DNA de sentido32,33,43,50. Defina a reação do PCR de acordo com a Tabela 1. Execute o PCR em alíquotas de 50 μL com ciclos apropriados no termociclador.
- Prepare as alças por duas reações de PCR separadas (Tabela 1, Figura 2B; reação 2 e 3). Primeiro, gere a alça de 5' por PCR. Em seguida, gere a alça de 3' e rotule-a simultaneamente com digoxigenina usando uma cartilha de 5' com etiqueta de digoxigenina, 32,33,43,50.
- Depois do PCR, purificar o DNA usando colunas de giro de sílica.
- Realize a reação de transcrição in vitro usando a polimerase T7 RNA (Tabela 2)32,33,43,50. Incubar a reação a 37 °C por 2-4 h, dependendo do comprimento do RNA. Em seguida, adicione DNase I à reação e incubar a 37 °C por 30 min para digerir o modelo de DNA. Purifique o RNA usando colunas de giro de sílica.
- Durante a reação de rotulagem da alça de 5''(Tabela 3), adicione biotina-16-dUTP na extremidade de 3' da alça por polimerase de DNA T438,50. Realize a reação à temperatura ambiente durante 1-2 h. Depois, purificar o DNA usando colunas de giro de sílica.
NOTA: Uma vez que a alça de 5' deve ser rotulada no final de 3' (Figura 2B), a rotulagem não pode ser realizada durante o PCR. - Misture os componentes mencionados acima - alça de 5' (3' rotulada com biotina), alça de 3' (5' rotulada com digoxigenina) e RNA - em uma relação molar de 1:1:1 no tampão de corte (80% de formamida, 400 mM NaCl, 40 mM HEPES, pH 7.5, 0,5 mM EDTA, pH 8), para obter o híbrido RNA/DNA desejado (Tabela 4). Aqueça a mistura de ressarcimento até 85 °C por 10 min e depois esfrie lentamente até 4 °C.
- Misture a amostra encolhida com 1/10 de volume de acetato de sódio de 3 M (pH 5), 3 volumes de etanol gelado e incubar a -80 °C por pelo menos 1 h ou a -20 °C durante a noite.
- Centrifugar as amostras a 15.000 × g por 30 min a 4 °C. Descarte o sobrenante e seque a pelota (geralmente não visível) sob vácuo.
- Finalmente, resuspend, a pelota em 50 μL de água sem RNase e fazer alíquotas. Armazene as alíquotas a -80 °C até usar. Para armazenamento a curto prazo, as amostras também podem ser armazenadas a -20 °C.
2. Configuração do instrumento
NOTA: O protocolo a seguir é otimizado para o instrumento de pinça óptica comercial C-Trap da empresa LUMICKS. Portanto, ajustes nas etapas apresentadas podem ser necessários ao utilizar outros instrumentos ópticos de pinça. Se não for utilizado, o sistema de microfluidos da máquina é mantido em alvejante (solução de hipoclorito de sódio) e deve ser lavado antes do uso.
- Descarte o alvejante e encha as seringas com 1 mL de água sem RNase.
- Adicione 50 μL de 0,5 M de tiossulfito de sódio a pelo menos 1 mL da água sem RNase e lave completamente o sistema (1 barra, pelo menos 0,5 mL) para eliminar o restante do alvejante no sistema.
- Descarte a solução de tiossulfato de sódio das seringas. Substitua as seringas por as frescas e lave o sistema por pelo menos 0,5 mL de água sem RNase.
NOTA: Tenha cuidado, que o sistema de microfluidos nunca seque para evitar bolhas de ar no sistema. - Coloque 2 gotas de óleo de imersão (índice de refração de 1,33) ou aproximadamente 70 μL de água em cima do objetivo.
- Coloque a célula de fluxo dentro da estrutura de retenção em sua posição.
- Coloque 2 gotas de óleo de imersão (índice de refração de 1,51) em cima da célula de fluxo.
- Ligue o dispositivo laser na máquina de pinças. Uma vez que esteja em execução, ligue o laser de trapping na interface de software em 100%.
- Utilizando câmeras de diagnóstico (Z-finder), ajuste o eixo Z para o meio da câmara entre a segunda e a terceira reflexões (interfaces) onde os anéis de refração são os maiores, girando o micro parafuso.
NOTA: Cada vez que o objetivo é movido mais perto da câmara de medição e o plano focal do objetivo cruza a interface entre duas fases, um reflexo pode ser reconhecido no modo Z-finder. Há 4 interfaces possíveis: (i) óleo de água/imersão e vidro inferior (ii) vidro inferior e tampão dentro do tampão da câmara (iii) dentro da câmara e vidro superior (iv) vidro superior e óleo de imersão para condensador. - Ajuste a posição do condensador (ajuste o laser de trapping para aproximadamente 50%) para que o condensador toque o óleo de imersão em cima da câmara de medição.
- Ajuste o foco movendo-se lentamente para baixo/para cima com o condensador, de modo que aproximadamente 10 faixas de luz são mostradas no modo lua (câmeras de diagnóstico).
3. Medição da amostra
- Incubar contas revestidas de anti-digoxigenina (AD) com os construtos de amostra (3 μL de 0,1% (w/v) suspensão de contas de AD + 4 μL de amostra) e com 1 μL de inibidores de RNase e 8 μL do tampão de ensaio (300 mM KCl, 5 mM MgCl2, 20 mM HEPES, pH 7.6, 0,05% Tween 20, 5 mM DTT) na RT por 10-20 min. Após a incubação, diluir a amostra em 500 μL de tampão de ensaio.
NOTA: Recomenda-se adicionar catadores de oxigênio, particularmente durante as medições de fluorescência ao buffer, a fim de evitar danos oxidativos. Aqui foi utilizado sistema de catalase de oxigênio contendo glicose (8,3 mg/mL), glicose oxidase (40 U/mL) e catalase (185 U/mL). - Misture 0,8 μL de 1% (w/v) contas revestidas de streptavidin (SA) com 1 mL de tampão de ensaio.
- Descarte a água das seringas e encha as seringas com as respectivas suspensões/soluções. Lave por pelo menos 2 min em aproximadamente 1 bar, e depois comece a pegar contas.
NOTA: Dependendo da configuração experimental, podem ser utilizados diferentes arranjos de canais (Figura 3). Normalmente, um canal de fluxo é preenchido com contas anti-digoxigenina carregando a molécula de RNA. Um segundo canal está cheio com as contas revestidas de streptavidin. O canal buffer é usado para formar as amarras. Um quarto canal pode ser empregado para carregar a proteína de ligação RNA (Figura 3C), ou, alternativamente, o RBP pode ser adicionado diretamente no canal tampão (Figura 3B). - Para capturar as contas, mova as armadilhas ópticas umas das outras. Primeiro vá para o canal de AD e pegue uma conta em armadilha 1. Em seguida, mova o palco para o canal SA e pegue uma única conta SA por armadilha 2.
NOTA: Tente ficar na interface dos canais tampão e contas para evitar perder a conta já capturada, ou para evitar pegar várias contas pela mesma armadilha. - Uma vez que as contas do tamanho certo sejam capturadas, mova-se para o canal tampão e pare o fluxo laminar. Em seguida, execute a calibração da força para verificar a rigidez da armadilha. Os respectivos valores de rigidez não devem diferir no eixo x/y em mais de 10-15%.
NOTA: Ajuste a potência do laser ou a divisão do laser entre as armadilhas de acordo com o tamanho das contas. A calibração da força não precisa ser feita para cada par de contas, desde que os modelos de contas coinceram (pontuação de similaridade > 0,9). No entanto, deve ser realizado regularmente, ou pelo menos toda vez que as condições de ensaio são alteradas. - Comece a pescar por uma corda movendo as contas perto uma da outra, esperando por alguns segundos, e depois movendo-as de volta, repita até formar uma corda. Uma formação de corda resulta em um aumento de força medida ao puxar as duas contas para longe uma da outra.
NOTA: Para evitar a formação de múltiplas amarras, as contas não devem ser movidas muito perto. Ao pegar uma corda entre as duas contas, a qualidade da corda pode ser verificada encontrando o planalto de alongamento excessivo. O planalto deve ficar entre 50 a 60 pN para uma única corda. - Ao obter uma corda, inicie a medição. Dependendo do fenômeno estudado devem ser escolhidas diferentes configurações de medição (Figura 1B-D).
NOTA: Geralmente, no início do experimento, um experimento de rampa de força é realizado para verificar a qualidade da corda e sondar o comportamento. Depois, pode-se também iniciar os experimentos de força constante ou de posição constante para estudar ainda mais as transições do Estado. Uma vez que um número suficiente de medições tenha sido realizado em uma amostra de RNA para determinar seu comportamento, fatores rotulados podem ser adicionados ao sistema para realizar medições confocal. - Para realizar medições de fluorescência, ligue os lasers confocal e a unidade do contador de fótons no instrumento pinça óptica.
- Ligue o laser de excitação do comprimento de onda desejado na interface de software e defina a potência do laser para 5% ou mais, dependendo do fluoróforo.
NOTA: Embora não medindo a configuração de potência do laser de excitação para 0% para evitar fotodamagem excessiva para a amostra. - Comece a fotografar a amostra usando funções de imagem do software.
NOTA: Para obter imagens bem focadas, o plano focal do microscópio confocal e armadilhas ópticas devem ser alinhados. Para isso, pode-se empregar a autofluorescência das contas de poliestireno no canal laser azul. O plano focal das armadilhas ópticas é movido para cima ou para baixo no eixo z até que a imagem das contas atinja seu diâmetro mais alto. Nesta posição, o sinal de fluorescência da molécula amarrada entre as contas pode ser medido. - Para usar a função kymograph, especifique a posição x-y do eixo kymograph para permitir a detecção da corda entre as contas.
- Ao longo da medição, a composição do buffer pode ser facilmente alterada movendo as contas para diferentes canais ou alterando o buffer fornecido no sistema de microfluidos.
4. Análise de dados
- Pré-processamento de dados brutos
- Usando um script simples, desça os dados brutos o suficiente para (i) permitir o processamento de dados subsequente mais rápido, mas (ii) ainda contêm todas as informações críticas (Figura 4A). Normalmente, 100-5000 Hz é adequado para este fim.
NOTA: A frequência de coleta de dados em experimentos ópticos de pinças é muitas vezes maior do que é necessária para a análise - nos experimentos apresentados, a frequência de coleta de dados é definida como 78 125 Hz por padrão. Uma vez que o espaço de armazenamento é limitado, é conveniente e economia de tempo reduzir a taxa de amostragem dos dados. Aqui, os dados brutos foram derrubados por um fator de 30. - Em seguida, empregue um filtro de sinal para reduzir o ruído de medição de alta frequência do sinal (Figura 4A). Ajuste o grau do filtro e os parâmetros de frequência de corte de acordo para otimizar a saída de dados de diferentes experimentos (Figura 5).
NOTA: Entre os filtros de sinal, o filtro Butterworth51 é um dos mais utilizados. Um script python escrito sob medida permitindo o pré-processamento de dados brutos é fornecido nos dados complementares. Os parâmetros de redução e filtragem de sinal (frequência de corte, grau de filtro) precisam ser otimizados para diferentes experimentos.
- Usando um script simples, desça os dados brutos o suficiente para (i) permitir o processamento de dados subsequente mais rápido, mas (ii) ainda contêm todas as informações críticas (Figura 4A). Normalmente, 100-5000 Hz é adequado para este fim.
- Para análise de dados de rampa de força, use as seguintes etapas.
- Marque os passos manualmente, encontrando pontos correspondentes no enredo da trajetória da força ou usando scripts personalizados. Os passos desdobrados são caracterizados por uma queda brusca de força combinada com um aumento da distância na curva de distância de força (FD).
- Uma vez marcados os eventos desdobrado, encaixe diferentes regiões da curva FD usando modelos apropriados (Figura 4D).
NOTA: Para a região antes do primeiro passo de desdobramento, a corda pode ser considerada "duplamente encalhada" e é comumente adequada usando um modelo extensível de corrente semelhante a worm (WLC)47,52,53. As partes após o primeiro evento de desdobramento são consideradas uma combinação de nucleotídeos de dupla fita (alças) e nucleotídeos de fio único (molécula de RNA desdobrada). Portanto, o encaixe de dados é mais complexo - geralmente uma combinação de 2 modelos WLC ou WLC e modelos de cadeia livremente articulada (FJC) são empregados36,39,52. O modelo WLC extensível tem dois parâmetros principais de ajuste, o comprimento do contorno (LC) e o comprimento da persistência (LP). O comprimento do contorno corresponde ao comprimento da molécula totalmente esticada e o comprimento da persistência define as propriedades de dobra da molécula de interesse. O modelo pode ser descrito com a seguinte equação (1). O WLC pode ser usado para modelar o comportamento tanto de regiões dobradas quanto desdobradas, embora para cada um deles um modelo separado com parâmetros diferentes tenha que ser empregado.
(1)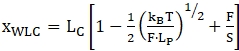
onde x é extensão, LC é comprimento de contorno, F é força, LP é comprimento de persistência, kB é boltzmann constante, T é temperatura termodinâmica, e S é módulo de estiramento.
O segundo modelo chamado cadeia livremente articulada (FJC) é comumente usado para descrever o comportamento de regiões únicas encalhadas desdobradas. Utiliza parâmetros semelhantes dos polímeros, mas trata cada unidade da "cadeia" como uma haste rígida, aqui correspondente aos nucleotídeos da região única encalhada desdobrada. A seguinte equação (2) descreve este modelo:
(2)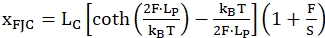
NOTA: Nosso laboratório desenvolveu recentemente um algoritmo que permite o processamento em lote dos dados de rampa de força bruta chamado "Prático Tweezers Analysis TOol (BATATA)54". O algoritmo diminui e filtra os dados, então identifica possíveis etapas de desdobramento e finalmente executa a montagem de dados. O BATATA é construído em uma interface de usuário gráfico amigável (GUI) (https://github.com/REMI-HIRI/POTATO).
- Processar dados de força constante da seguinte forma:
NOTA: As seguintes instruções podem ser aplicadas ana analogicamente em dados de posição constante.- Para os dados de força constante, plote a distância ao longo do tempo (Figura 5). Um histograma mostrando a frequência (contagem) de diferentes conformações sobre a mudança relativa de posição é uma maneira útil de caracterizar vários estados dominantes e menores (Figura 7).
- Ajuste o histograma usando (múltiplas) funções gaussianas para estimar a porcentagem global de conformadores individuais a uma determinada força (Figura 7C). O gaussiano se encaixa, posição média, e o desvio padrão descreve a relação relacionada à força entre diferentes populações.
NOTA: Um script python escrito sob medida que permite o pré-processamento e o encaixe bimodal básico de dados de força constante são fornecidos nos dados complementares. Os parâmetros (frequência de corte, grau de filtro, meios esperados, valores de desvio padrão e amplitudes) precisam ser otimizados para diferentes experimentos. - Em seguida, empregue o modelo Hidden Markov para analisar melhor os estados, o que pode descobrir intermediários dobráveis adicionais (conformadores)55. Para obter mais informações sobre o modelo De markov de força constante e hidden, pode-se referir-se a 55,56,57,58.
Access restricted. Please log in or start a trial to view this content.
Resultados
Nesta seção, o foco é dado principalmente em medidas de interações RNA-proteína/ligand pelas pinças ópticas fluorescência. Para uma descrição dos experimentos gerais de pinça óptica RNA e resultados representativos correspondentes, consulte32. Para uma discussão mais detalhada das interações RNA/DNA-proteína, consulte também 1,2,26,59,60.
Access restricted. Please log in or start a trial to view this content.
Discussão
Aqui, demonstramos o uso de pinças ópticas acopladas à fluorescência para estudar interações e comportamento dinâmico de moléculas de RNA com vários ligantes. Abaixo, são discutidas etapas críticas e limitações da presente técnica.
Passos críticos no protocolo
Como para muitos outros métodos, a qualidade da amostra é fundamental para obter dados confiáveis. Portanto, para obter as amostras de maior qualidade possível, vale a pena gastar tempo para otimiza...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos a Anuja Kibe e ao Prof. Redmond Smyth por revisarem criticamente o manuscrito. Agradecemos a Tatyana Koch pela assistência técnica especializada. Agradecemos a Kristyna Pekarkova pela ajuda na gravação de vídeos experimentais. O trabalho em nosso laboratório é apoiado pela Associação Helmholtz e financiamento do Conselho Europeu de Pesquisa (ERC) Grant Nr. 948636 (para a NC).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| E. coli HB101 | lab collection | N/A | cloning of the vectors |
| Chemicals and enzymes | |||
| Sodium chloride | Sigma-Aldrich | 31424 | Buffers |
| Biotin-16-dUTP | Roche | 11093070910 | Biotinylation |
| BSA | Sigma-Aldrich | A4737 | Buffers |
| Catalase | Lumicks | N/A | Oxygen scavanger system |
| Dithiothreitol (DTT) | Melford Labs | D11000 | Buffers |
| DNAse I from bovine pancreas | Sigma-Aldrich | D4527 | in vitro transcription |
| dNTPs | Th.Geyer | 11786181 | PCR |
| EDTA | Sigma-Aldrich | E9884 | Buffers |
| Formamide | Sigma-Aldrich | 11814320001 | Buffers |
| Glucose | Sigma-Aldrich | G8270-1KG | Oxygen scavanger system |
| Glucose-oxidase | Lumicks | N/A | Oxygen scavanger system |
| HEPES | Carl Roth | HN78.3 | Buffers |
| Magnesium chloride | Carl Roth | 2189.1 | Buffers |
| Phusion DNA polymerase | NEB | M0530L | Gibson assembly, cloning |
| Potassium chloride | Merck | 529552-1KG | Buffers |
| PrimeSTAR GXL DNA Polymerase | Takara Bio Clontech | R050A | PCR |
| Pyrophosphotase, thermostabile, inorganic | NEB | M0296L | in vitro transcription |
| RNase Inhibitor | Molox | 1000379515 | Buffers |
| rNTPS | life technologies | R0481 | in vitro transcription |
| Sodium thiosulophate | Sigma-Aldrich | S6672-500G | Bleach deactivation |
| Sytox Green | Lumicks | N/A | confocal measurements |
| T4 DNA Polymerase | NEB | M0203S | Biotinylation |
| T5 exonuclease | NEB | M0363S | Gibson assembly, cloning |
| T7 RNA polymerase | Produced in-house | N/A | in vitro transcription |
| Taq DNA polymerase | NEB | M0267S | PCR |
| Taq ligase | Biozym | L6060L | Gibson assembly, cloning |
| TWEEN 20 BioXtra | Sigma-Aldrich | P7949 | Buffers |
| Kits | |||
| Monolith Protein Labeling Kit RED-NHS 2nd Generation (Amine Reactive) | Nanotemper | MO-L011 | Used for ribosome labeling |
| Purefrex 2.0 | GeneFrontier | PF201-0.25-EX | Ribosomes used for the labeling |
| Oligonucleotides | |||
| 5' handle T7 forward | Microsynth | custom order | 5’ - CTTAATACGACTCACTATAGGTC CTTTCTGTGGACGCC - 3’, used to generate OT in vitro transcription template in PCR 1 |
| 3’ handle reverse | Microsynth | custom order | 5' - GTCAAAGTGCGCCCCGTTATCC - 3', used to generate OT in vitro transcription template in PCR 1 |
| 5' handle forward | Microsynth | custom order | 5' - TCCTTTCTGTGGACGCCGC - 3' , used to generate 5' handle in PCR 2 |
| 5’ handle reverse | Microsynth | custom order | 5’ - CATAAATACCTCTTTACTAATATA TATACCTTCGTAAGCTAGCGT - 3’, used to generate 5' handle in PCR 2 |
| 3’ handle forward | Microsynth | custom order | 5' - ATCCTGCAACCTGCTCTTCGCC AG - 3', used to generate 3' handle in PCR 3 |
| 3’ handle reverse 5’labeled with digoxigenin | Microsynth | custom order | 5' -[Dig]-GTCAAAGTGCGCCCCGTTATCC - 3', used to generate 3' handle in PCR 3 |
| DNA vectors | |||
| pMZ_OT | produced in-house | N/A | further description in "Structural studies of Cardiovirus 2A protein reveal the molecular basis for RNA recognition and translational control" Chris H. Hill, Sawsan Napthine, Lukas Pekarek, Anuja Kibe, Andrew E. Firth, Stephen C. Graham, Neva Caliskan, Ian Brierley bioRxiv 2020.08.11.245035; doi: https://doi.org/10.1101/2020.08.11.245035 |
| Software and Algorithms | |||
| Atom | https://atom.io/packages/ide-python | N/A | |
| Bluelake | Lumicks | N/A | |
| Graphpad | https://www.graphpad.com/ | N/A | |
| InkScape 0.92.3 | https://inkscape.org/ | N/A | |
| Matlab | https://www.mathworks.com/products/matlab.html | N/A | |
| POTATO | https://github.com/lpekarek/POTATO.git | N/A | |
| RNAstructure | https://rna.urmc.rochester.edu/RNAstructure.html | N/A | |
| Spyder | https://www.spyder-ide.org/ | N/A | |
| Other | |||
| Streptavidin Coated Polystyrene Particles, 1.5-1.9 µm, 5 ml, 1.0% w/v | Spherotech | SVP-15-5 | |
| Anti-digoxigenin Coated Polystyrene Particles, 2.0-2.4 µm, 2 ml, 0.1% w/v | Spherotech | DIGP-20-2 | |
| Syringes | VWR | TERUMO SS+03L1 | |
| Devices | |||
| C-trap | Lumicks | N/A | optical tweezers coupled with confocal microscopy |
Referências
- Balcerak, A., Trebinska-Stryjewska, A., Konopinski, R., Wakula, M., Grzybowska, E. A. RNA-protein interactions: disorder, moonlighting and junk contribute to eukaryotic complexity. Open Biology. 9 (6), 190096(2019).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118(2021).
- Firth, A. E., Brierley, I. Non-canonical translation in RNA viruses. Journal of General Virology. 93, Pt 7 1385-1409 (2012).
- Caliskan, N., Peske, F., Rodnina, M. V. Changed in translation: mRNA recoding by −1 programmed ribosomal frameshifting. Trends in Biochemical Sciences. 40 (5), 265-274 (2015).
- Jaafar, Z. A., Kieft, J. S. Viral RNA structure-based strategies to manipulate translation. Nature Reviews Microbiology. 17 (2), 110-123 (2019).
- Eswarappa, S. M., et al. Programmed translational readthrough generates antiangiogenic VEGF-Ax. Cell. 157 (7), 1605-1618 (2014).
- Rodnina, M. V., et al. Translational recoding: canonical translation mechanisms reinterpreted. Nucleic Acids Research. 48 (3), 1056-1067 (2020).
- Li, Y., et al. Transactivation of programmed ribosomal frameshifting by a viral protein. Proceedings of the National Academy of Sciences. 111 (21), 2172(2014).
- Napthine, S., et al. Protein-directed ribosomal frameshifting temporally regulates gene expression. Nature Communications. 8 (1), 15582(2017).
- Patel, A., et al. Molecular characterization of the RNA-protein complex directing -2/-1 programmed ribosomal frameshifting during arterivirus replicase expression. Journal of Biological Chemistry. 295 (52), 17904-17921 (2020).
- Napthine, S., Bell, S., Hill, C. H., Brierley, I., Firth, A. E. Characterization of the stimulators of protein-directed ribosomal frameshifting in Theiler's murine encephalomyelitis virus. Nucleic Acids Research. 47 (15), 8207-8223 (2019).
- Marshall, R. A., Aitken, C. E., Dorywalska, M., Puglisi, J. D. Translation at the Single-Molecule Level. Annual Review of Biochemistry. 77 (1), 177-203 (2008).
- Rodnina, M. V. The ribosome in action: Tuning of translational efficiency and protein folding. Protein science : A publication of the Protein Society. 25 (8), 1390-1406 (2016).
- Rodnina, M. V., Fischer, N., Maracci, C., Stark, H. Ribosome dynamics during decoding. Philosophical Transactions of Royal Society of London B Biological Sciences. 372 (1716), (2017).
- Yan, S., Wen, J. D., Bustamante, C., Tinoco, I. Ribosome excursions during mRNA translocation mediate broad branching of frameshift pathways. Cell. 160 (5), 870-881 (2015).
- Liu, T., et al. Direct measurement of the mechanical work during translocation by the ribosome. eLife. 3, 03406(2014).
- Desai, V. P., et al. Co-temporal force and fluorescence measurements reveal a ribosomal gear shift mechanism of translation regulation by structured mRNAs. Molecular Cell. 75 (5), 1007-1019 (2019).
- Choi, J., O'Loughlin, S., Atkins, J. F., Puglisi, J. D. The energy landscape of -1 ribosomal frameshifting. Science Advances. 6 (1), (2020).
- Prabhakar, A., Puglisi, E. V., Puglisi, J. D. Single-molecule fluorescence applied to translation. Cold Spring Harbor Perspectives in Biology. 11 (1), 032714(2019).
- Bao, C., et al. mRNA stem-loops can pause the ribosome by hindering A-site tRNA binding. Elife. 9, 55799(2020).
- Chen, J., Tsai, A., O'Leary, S. E., Petrov, A., Puglisi, J. D. Unraveling the dynamics of ribosome translocation. Current Opinion in Structural Biology. 22 (6), 804-814 (2012).
- Qu, X., et al. The ribosome uses two active mechanisms to unwind messenger RNA during translation. Nature. 475 (7354), 118-121 (2011).
- Zheng, Q., et al. Ultra-stable organic fluorophores for single-molecule research. Chemical Society Reviews. 43 (4), 1044-1056 (2014).
- Blanchard, S. C. Single-molecule observations of ribosome function. Current Opinion in Structural Biology. 19 (1), 103-109 (2009).
- Juette, M. F., et al. The bright future of single-molecule fluorescence imaging. Current Opinion in Chemical Biology. 20, 103-111 (2014).
- McCauley, M. J., Williams, M. C. Mechanisms of DNA binding determined in optical tweezers experiments. Biopolymers. 85 (2), 154-168 (2007).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters. 11 (5), 288-290 (1986).
- Bustamante, C., Smith, S. B., Liphardt, J., Smith, D. Single-molecule studies of DNA mechanics. Current Opinion in Structural Biology. 10 (3), 279-285 (2000).
- Choudhary, D., Mossa, A., Jadhav, M., Cecconi, C. Bio-molecular applications of recent developments in optical tweezers. Biomolecules. 9 (1), 23(2019).
- Moffitt, J. R., Chemla, Y. R., Smith, S. B., Bustamante, C. Recent advances in optical tweezers. Annual Reviews of Biochemistry. 77, 205-228 (2008).
- Li, P. T. X., Vieregg, J., Tinoco, I. How RNA Unfolds and Refolds. Annual Review of Biochemistry. 77 (1), 77-100 (2008).
- Stephenson, W., Wan, G., Tenenbaum, S. A., Li, P. T. Nanomanipulation of single RNA molecules by optical tweezers. Journal of Visualized Experiments. (90), e51542(2014).
- Halma, M. T. J., Ritchie, D. B., Cappellano, T. R., Neupane, K., Woodside, M. T. Complex dynamics under tension in a high-efficiency frameshift stimulatory structure. Proceedings of the National Academy of Sciences. 116 (39), 19500(2019).
- Hansen, T. M., Reihani, S. N. S., Oddershede, L. B., Sørensen, M. A. Correlation between mechanical strength of messenger RNA pseudoknots and ribosomal frameshifting. Proceedings of the National Academy of Sciences of the United States of America. 104 (14), 5830-5835 (2007).
- Zhong, Z., et al. Mechanical unfolding kinetics of the SRV-1 gag-pro mRNA pseudoknot: possible implications for -1 ribosomal frameshifting stimulation. Science Reports. 6, 39549(2016).
- McCauley, M. J., Rouzina, I., Li, J., Núñez, M. E., Williams, M. C. Significant differences in RNA structure destabilization by HIV-1 GagDp6 and NCp7 proteins. Viruses. 12 (5), 484(2020).
- de Messieres, M., et al. Single-molecule measurements of the CCR5 mRNA unfolding pathways. Biophysics Journal. 106 (1), 244-252 (2014).
- Yang, L., et al. Single-molecule mechanical folding and unfolding of RNA hairpins: Effects of single A-U to A·C pair substitutions and single proton binding and implications for mRNA structure-induced -1 ribosomal frameshifting. Journal of American Chemical Society. 140 (26), 8172-8184 (2018).
- McCauley, M. J., et al. Targeted binding of nucleocapsid protein transforms the folding landscape of HIV-1 TAR RNA. Proceedings of the National Academy of Sciences of the United States of America. 112 (44), 13555-13560 (2015).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. High-resolution "Fleezers": Dual-trap optical tweezers combined with single-molecule fluorescence detection. Methods in Molecular Biology. 1486, Clifton, N.J. 183-256 (2017).
- Yerramilli, V. S., Kim, K. H. Labeling RNAs in live cells using malachite green aptamer scaffolds as fluorescent probes. ACS Synthetic Biology. 7 (3), 758-766 (2018).
- Gross, P., Farge, G., Peterman, E. J., Wuite, G. J. Combining optical tweezers, single-molecule fluorescence microscopy, and microfluidics for studies of DNA-protein interactions. Methods in Enzymology. 475, 427-453 (2010).
- Zimmer, M. M., et al. The short isoform of the host antiviral protein ZAP acts as an inhibitor of SARS-CoV-2 programmed ribosomal frameshifting. Nature Communications. 12 (1), 7193(2021).
- Neupane, K., Yu, H., Foster, D. A. N., Wang, F., Woodside, M. T. Single-molecule force spectroscopy of the add adenine riboswitch relates folding to regulatory mechanism. Nucleic acids research. 39 (17), 7677-7687 (2011).
- Ritchie, D. B., Soong, J., Sikkema, W. K., Woodside, M. T. Anti-frameshifting ligand reduces the conformational plasticity of the SARS virus pseudoknot. Journal of the American Chemical Society. 136 (6), 2196-2199 (2014).
- Janissen, R., et al. Invincible DNA tethers: covalent DNA anchoring for enhanced temporal and force stability in magnetic tweezers experiments. Nucleic Acids Research. 42 (18), 137(2014).
- Smith, S. B., Cui, Y., Bustamante, C. Overstretching B-DNA: The elastic response of individual double-stranded and single-stranded DNA molecules. Science. 271 (5250), 795(1996).
- Puljung, M. C., Zagotta, W. N. Labeling of specific cysteines in proteins using reversible metal protection. Biophysical Journal. 100 (10), 2513-2521 (2011).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Hill, C. H., et al. Structural and molecular basis for Cardiovirus 2A protein as a viral gene expression switch. Nature Communications. 12 (1), 7166(2021).
- Butterworth, S. On the theory of filter amplifiers. Experimental Wireless and the Wireless Engineer. 7, 536-541 (1930).
- Wang, M. D., Yin, H., Landick, R., Gelles, J., Block, S. M. Stretching DNA with optical tweezers. Biophysics Journal. 72 (3), 1335-1346 (1997).
- Mukhortava, A., et al. Structural heterogeneity of attC integron recombination sites revealed by optical tweezers. Nucleic Acids Research. 47 (4), 1861-1870 (2019).
- Buck, S., Pekarek, L., Caliskan, N. POTATO: An automated pipeline for batch analysis of optical tweezers data. bioRxiv. , (2021).
- Zhang, Y., Jiao, J., Rebane, A. A. Hidden Markov modeling with detailed balance and its application to single protein folding. Biophysical Journal. 111 (10), 2110-2124 (2016).
- Sgouralis, I., Pressé, S. An introduction to infinite HMMs for single-molecule data analysis. Biophysics Journal. 112 (10), 2021-2029 (2017).
- Müllner, F. E., Syed, S., Selvin, P. R., Sigworth, F. J. Improved hidden Markov models for molecular motors, part 1: basic theory. Biophysical Journal. 99 (11), 3684-3695 (2010).
- Elms, P. J., Chodera, J. D., Bustamante, C. J., Marqusee, S. Limitations of constant-force-feedback experiments. Biophysical Journal. 103 (7), 1490-1499 (2012).
- Re, A., Joshi, T., Kulberkyte, E., Morris, Q., Workman, C. T. RNA-protein interactions: an overview. Methods Molecular Biology. 1097, 491-521 (2014).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature reviews. Molecular Cell Biology. 16 (9), 533-544 (2015).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Sunbul, M., Jäschke, A. SRB-2: a promiscuous rainbow aptamer for live-cell RNA imaging. Nucleic Acids Research. 46 (18), 110(2018).
- Sanchez de Groot, N., et al. RNA structure drives interaction with proteins. Nature Communications. 10 (1), 3246(2019).
- Zeffman, A., Hassard, S., Varani, G., Lever, A. The major HIV-1 packaging signal is an extended bulged stem loop whose structure is altered on interaction with the Gag polyprotein. Journal of Molecular Biology. 297 (4), 877-893 (2000).
- Mangeol, P., et al. Probing ribosomal protein-RNA interactions with an external force. Proceedings of the National Academy of Sciences. 108 (45), 18272(2011).
- Luo, X., et al. Molecular mechanism of RNA recognition by Zinc-Finger antiviral protein. Cell Reports. 30 (1), 46-52 (2020).
- Qu, X., Lancaster, L., Noller, H. F., Bustamante, C., Tinoco, I. Ribosomal protein S1 unwinds double-stranded RNA in multiple steps. Proceedings of the National Academy of Science U. S. A. 109 (36), 14458-14463 (2012).
- Chandra, V., Hannan, Z., Xu, H., Mandal, M. Single-molecule analysis reveals multi-state folding of a guanine riboswitch. Nature Chemical Biology. 13 (2), 194-201 (2017).
- Savinov, A., Perez, C. F., Block, S. M. Single-molecule studies of riboswitch folding. Biochimica et Biophysica Acta. 1839 (10), 1030-1045 (2014).
- Kelly, J. A., et al. Structural and functional conservation of the programmed ribosomal frameshift signal of SARS coronavirus 2 (SARS-CoV-2). Journal of Biological Chemistry. 295 (31), 10741-10748 (2020).
- Neupane, K., et al. Structural dynamics of single SARS-CoV-2 pseudoknot molecules reveal topologically distinct conformers. Nature Communications. 12 (1), 4749(2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zheng, Q., Jockusch, S., Zhou, Z., Blanchard, S. C. The contribution of reactive oxygen species to the photobleaching of organic fluorophores. Photochemistry and Photobiology. 90 (2), 448-454 (2014).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Rill, N., Mukhortava, A., Lorenz, S., Tessmer, I. Alkyltransferase-like protein clusters scan DNA rapidly over long distances and recruit NER to alkyl-DNA lesions. Proceedings of the National Academy of Science U. S. A. 117 (17), 9318-9328 (2020).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Wen, J. -D., et al. Force unfolding kinetics of RNA using optical tweezers. I. Effects of experimental variables on measured results. Biophysical journal. 92 (9), 2996-3009 (2007).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados