Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Pinces optiques pour étudier les interactions ARN-protéine dans la régulation de la traduction
Dans cet article
Résumé
Ce protocole présente un flux de travail expérimental complet pour l’étude des interactions ARN-protéine à l’aide de pinces optiques. Plusieurs configurations expérimentales possibles sont décrites, y compris la combinaison de pinces optiques avec la microscopie confocale.
Résumé
L’ARN adopte divers plis structurels, qui sont essentiels à ses fonctions et peuvent donc avoir un impact sur divers processus dans la cellule. En outre, la structure et la fonction d’un ARN peuvent être modulées par divers facteurs trans-agissants, tels que des protéines, des métabolites ou d’autres ARN. Les molécules d’ARN à décalage de trame, par exemple, sont des ARN régulateurs situés dans des régions codantes, qui dirigent la traduction des ribosomes en un cadre de lecture ouvert alternatif, et agissent ainsi comme des commutateurs de gènes. Ils peuvent également adopter différents plis après s’être liés à des protéines ou à d’autres facteurs trans. Pour disséquer le rôle des protéines de liaison à l’ARN dans la traduction et la façon dont elles modulent la structure et la stabilité de l’ARN, il est crucial d’étudier simultanément l’interaction et les caractéristiques mécaniques de ces complexes ARN-protéine. Ce travail illustre comment utiliser des pinces optiques couplées à une seule molécule et à fluorescence pour explorer le paysage conformationnel et thermodynamique des complexes ARN-protéine à haute résolution. À titre d’exemple, l’interaction de l’élément de décalage de trame ribosomique programmé par le SARS-CoV-2 avec l’isoforme courte du facteur trans-agissant de la protéine antivirale zinc-doigt est élaborée. De plus, les ribosomes marqués par fluorescence ont été surveillés à l’aide de l’unité confocale, ce qui permettrait finalement d’étudier l’allongement de la traduction. Le test OT couplé à la fluorescence peut être largement appliqué pour explorer divers complexes ARN-protéine ou facteurs trans-agissant régulant la traduction et pourrait faciliter les études de la régulation des gènes basée sur l’ARN.
Introduction
Le transfert de l’information génétique de l’ADN aux protéines à travers les ARNm est un processus biochimique complexe, qui est régulé avec précision à tous les niveaux par des interactions macromoléculaires à l’intérieur des cellules. Pour la régulation translationnelle, les interactions ARN-protéine confèrent un rôle essentiel pour réagir rapidement à divers stimuli et signaux1,2. Certaines interactions ARN-protéine affectent la stabilité de l’ARNm et modifient ainsi le temps pendant lequel un ARN est actif sur le plan translationnel. D’autres interactions ARN-protéine sont associées à des mécanismes de recodage tels que la lecture stop-codon, le contournement ou le décalage de trame ribosomique programmé (PRF)3,4,5,6,7. Récemment, il a été démontré qu’un certain nombre de protéines de liaison à l’ARN (RBP) interagissent avec les éléments de l’ARNm stimulants et la machinerie de traduction pour dicter quand et combien de recodage se produira dans la cellule7,8,9,10,11. Ainsi, pour disséquer le rôle des protéines de liaison à l’ARN dans la traduction et la façon dont elles modulent la structure et la stabilité de l’ARN, il est essentiel d’étudier en détail les principes d’interaction et les propriétés mécaniques de ces complexes ARN-protéine.
Des décennies de travail ont jeté les bases de l’étude du processus de traduction en plusieurs étapes et en plusieurs composants, qui repose sur une communication complexe entre les composants ARN et protéique de la machinerie de traduction pour atteindre la vitesse et la précision12,13,14. Une prochaine étape cruciale dans la compréhension des événements réglementaires complexes consiste à déterminer les forces, les échelles de temps et les déterminants structurels pendant la traduction avec une grande précision12,15,16,17. L’étude de la dynamique conformationnelle de l’ARN et en particulier de la façon dont les facteurs auxiliaires à action trans agissant sur la structure de l’ARN pendant la traduction ont été encore éclairées par l’émergence d’outils à molécule unique, y compris les pinces optiques ou les guides d’ondes en mode zéro16,17,18,19,20,21,22,23,24 ,25,26.
Les pinces optiques (OT) représentent une technique à molécule unique très précise, qui a été appliquée pour étudier de nombreux types de processus dynamiques dépendants de l’ARN, y compris la transcription et la traduction26,27,28,29,30,31,32. L’utilisation de pinces optiques a permis de sonder en détail les interactions moléculaires, les structures des acides nucléiques et les propriétés thermodynamiques, la cinétique et l’énergie de ces processus16,17,22,33,34,35,36,37,38,39 . Le test de pince à épiler optique est basé sur le piégeage d’objets microscopiques avec un faisceau laser focalisé. Dans une expérience d’OT typique, la molécule d’intérêt est attachée entre deux billes transparentes (généralement du polystyrène) (Figure 1A)27. Ces perles sont ensuite capturées par des pièges optiques, qui se comportent comme des ressorts. Ainsi, la force appliquée sur la molécule peut être calculée en fonction du déplacement de la perle du centre du faisceau laser focalisé (centre du piège). Récemment, des pinces optiques ont été combinées à la microscopie confocale (Figure 1B), permettant des mesures de fluorescence ou de transfert d’énergie par résonance de Förster (FRET)40,41,42. Cela ouvre un tout nouveau champ d’expériences possibles permettant une mesure simultanée et, par conséquent, une corrélation précise des données de spectroscopie de force et de fluorescence.
Ici, nous démontrons des expériences utilisant la pince à épiler optique combinée à la microscopie confocale pour étudier les interactions protéine-ARN régulant le décalage translationnel. Entre l’objectif et le condenseur, une cellule d’écoulement à cinq canaux permet une application continue de l’échantillon avec un flux laminaire. Grâce aux canaux microfluidiques, divers composants peuvent être injectés directement, ce qui réduit le temps de pratique et permet très peu de consommation d’échantillons tout au long de l’expérience.
Tout d’abord, une ligne directrice de base pour aider à la conception d’expériences OT est proposée et les avantages ainsi que les pièges de diverses configurations sont discutés. Ensuite, la préparation des échantillons et des flux de travail expérimentaux sont décrits, et un protocole pour l’analyse des données est fourni. Pour représenter un exemple, nous décrivons les résultats obtenus à partir d’expériences d’étirement de l’ARN pour étudier l’élément ARN de décalage de trame du SARS-CoV-2 (Figure 2A) avec le facteur de transaction l’isoforme courte de la protéine antivirale à doigts de zinc (ZAP), qui modifie la traduction de l’ARN viral à partir d’un cadre de lecture alternatif43. En outre, il est démontré que des ribosomes marqués par fluorescence peuvent être utilisés dans ce test confocal OT, ce qui serait utile pour surveiller la processivité et la vitesse de la machine de traduction. La méthode présentée ici peut être utilisée pour tester rapidement l’effet de différents tampons, ligands ou autres composants cellulaires afin d’étudier divers aspects de la traduction. Enfin, les pièges expérimentaux courants et la façon de les résoudre sont discutés. Ci-dessous, quelques points cruciaux de la conception expérimentale sont décrits.
Conception de construction
En principe, il existe deux approches communes pour créer une construction d’ARN compatible OT. La première approche utilise une longue molécule d’ARN qui est hybridée avec des poignées d’ADN complémentaires, donnant ainsi une construction composée de deux régions hybrides ARN/ADN flanquant une séquence d’ARN simple brin au milieu (Figure 2B). Cette approche est utilisée dans la plupart des expériences d’ARN OT33,44,45.
La deuxième approche tire parti des poignées dsDNA avec des porte-à-faux courts (environ 20 nt)15,17. Ces surplombs sont ensuite hybridés avec la molécule d’ARN. Bien que de conception plus compliquée, l’utilisation de poignées dsDNA surmonte certaines des limites du système hybride ADN/ARN. En principe, même des poignées très longues (>10kb) peuvent être mises en œuvre, ce qui est plus pratique pour les mesures confocales. De plus, la molécule d’ARN peut être ligaturée aux poignées d’ADN pour augmenter la stabilité de l’attache.
Stratégie d’étiquetage final
La construction doit être attachée aux perles via une forte interaction moléculaire. Bien qu’il existe des approches disponibles pour la liaison covalente des poignées aux billes46, des interactions fortes mais non covalentes telles que la streptavidine-biotine et la digoxigénine-anticorps sont couramment utilisées dans les expériences OT15,33,35,45. Dans le protocole décrit, la construction est marquée avec de la biotine ou de la digoxigénine, et les billes sont recouvertes de streptavidine ou d’anticorps contre la digoxigénine, respectivement (Figure 1A). Cette approche conviendrait à l’application de forces allant jusqu’à environ 60 pN (par attache)47. De plus, l’utilisation de différentes stratégies d’étiquetage de 5' et 3' permet de déterminer l’orientation de l’attache formée entre les perles17.
Marquage des protéines pour les mesures de fluorescence
Pour l’imagerie confocale, il existe plusieurs approches couramment utilisées pour le marquage par fluorescence. Par exemple, les fluorophores peuvent être attachés de manière covalente à des résidus d’acides aminés qui se trouvent nativement dans les protéines ou introduits par mutagénèse dirigée par le site à travers un groupe organique réactif. Les colorants thiol ou réactifs aux amines peuvent être utilisés pour le marquage des résidus de cystéine et de lysine, respectivement. Il existe plusieurs méthodes de protection réversibles pour augmenter la spécificité du marquage48,49, mais les protéines natives seraient généralement marquées à plusieurs résidus. Bien que la petite taille du fluorophore puisse conférer un avantage, un marquage non spécifique peut interférer avec l’activité protéique et, par conséquent, l’intensité du signal peut varier49. En outre, en fonction de l’efficacité de l’étiquetage, l’intensité du signal peut différer d’une expérience à l’autre. Par conséquent, une vérification de l’activité doit être effectuée avant l’expérience.
Dans le cas où la protéine d’intérêt contient une étiquette terminale N ou C, telle qu’une étiquette His ou une étiquette streptococcique, l’étiquetage spécifique de ces étiquettes représente une autre approche populaire. De plus, le marquage ciblé par étiquette réduit le risque que le fluorophore interfère avec l’activité des protéines et peut améliorer la solubilité49. Cependant, l’étiquetage spécifique à l’étiquette donne généralement des protéines marquées monofluorophore, ce qui peut être difficile à détecter. Une autre façon de marquer spécifiquement peut être réalisée en utilisant des anticorps.
Configuration de la microfluidique
La combinaison de l’OT avec un système microfluidique permet une transition rapide entre différentes conditions expérimentales. De plus, les systèmes actuels tirent parti du maintien du flux laminaire à l’intérieur de la cellule d’écoulement, ce qui empêche le mélange de liquides d’autres canaux dans la direction perpendiculaire par rapport à la direction d’écoulement. Par conséquent, l’écoulement laminaire est particulièrement avantageux pour la conception expérimentale. Actuellement, les cellules d’écoulement avec jusqu’à 5 canaux sont couramment utilisées (Figure 3).
Protocole
1. Préparation de l’échantillon
- Clonez la séquence d’intérêt dans le vecteur contenant les fragments d’ADN Lambda, qui servent de séquences de poignée (Figure 2)43,50.
- Générez d’abord un modèle d’ADN pour une transcription in vitro ultérieure par PCR (Figure 2B; réaction 1). À cette étape de PCR, le promoteur T7 est ajouté à l’extrémité 5' de la molécule d’ADN sensorielle32,33,43,50. Réglez la réaction pcR conformément au tableau 1. Exécutez la PCR dans des aliquotes de 50 μL avec les cycles appropriés dans le thermocycleur.
- Préparer les poignées par deux réactions PCR distinctes (tableau 1, figure 2B; réactions 2 et 3). Tout d’abord, générez la poignée 5' par PCR. Ensuite, générez la poignée 3' et étiquetez-la simultanément avec de la digoxigénine à l’aide d’un apprêt marqué à la digoxigénine 5'32,33,43,50.
- Après la PCR, purifiez l’ADN à l’aide de colonnes de spin de silice.
- Effectuer la réaction de transcription in vitro à l’aide de l’ARN polymérase T7 (Tableau 2)32,33,43,50. Incuber la réaction à 37 °C pendant 2-4 h en fonction de la longueur de l’ARN. Ensuite, ajoutez la DNase I à la réaction et incubez à 37 °C pendant 30 min pour digérer le gabarit d’ADN. Purifier l’ARN à l’aide de colonnes de spin de silice.
- Lors de la réaction de marquage de la poignée 5' (Tableau 3), ajouter la biotine-16-dUTP à l’extrémité 3' de la poignée par ADN polymérase T438,50. Effectuer la réaction à température ambiante pendant 1-2 h. Ensuite, purifiez l’ADN à l’aide de colonnes de spin de silice.
REMARQUE : Étant donné que la poignée de 5' doit être étiquetée à son extrémité de 3' (Figure 2B), l’étiquetage ne peut pas être effectué pendant la PCR. - Mélanger les composants mentionnés ci-dessus - poignée 5' (3' marquée avec biotine), poignée 3' (5' marquée avec digoxiénine), et ARN - dans un rapport molaire 1:1:1 dans un tampon de recuit (80% formamide, 400 mM NaCl, 40 mM HEPES, pH 7,5, 0,5 mM EDTA, pH 8), pour obtenir l’hybride ARN/ADN souhaité (Tableau 4). Chauffer le mélange de recuit jusqu’à 85 °C pendant 10 min, puis refroidir lentement jusqu’à 4 °C.
- Mélanger l’échantillon recuit avec 1/10 de volume d’acétate de sodium de 3 M (pH 5), 3 volumes d’éthanol glacé et incuber à -80 °C pendant au moins 1 h ou à -20 °C pendant la nuit.
- Centrifuger les échantillons à 15 000 × g pendant 30 min à 4 °C. Jetez le surnageant et séchez la pastille (généralement non visible) sous vide.
- Enfin, remettez en suspension la pastille dans 50 μL d’eau sans RNase et faites des aliquotes. Conserver les aliquotes à -80 °C jusqu’à ce qu’elles soient utilisées. Pour un stockage à court terme, les échantillons peuvent également être stockés à -20 °C.
2. Configuration de l’instrument
REMARQUE: Le protocole suivant est optimisé pour l’instrument de pince optique commerciale C-Trap de la société LUMICKS. Par conséquent, des ajustements aux étapes présentées peuvent être nécessaires lors de l’utilisation d’autres instruments de pince à épiler optiques. S’il n’est pas utilisé, le système microfluidique de la machine est conservé dans de l’eau de Javel (solution d’hypochlorite de sodium) et doit être lavé avant utilisation.
- Jetez l’eau de Javel et remplissez les seringues avec 1 mL d’eau sans RNase.
- Ajouter 50 μL de thiosulfate de sodium de 0,5 M à au moins 1 mL d’eau sans RNase et bien laver le système (1 bar, au moins 0,5 mL) pour éliminer l’eau de Javel restante dans le système.
- Jeter la solution de thiosulfate de sodium des seringues. Remplacez les seringues par des seringues fraîches et lavez le système avec au moins 0,5 mL d’eau sans RNase.
REMARQUE: Faites attention, que le système de microfluidique ne fonctionne jamais à sec pour éviter les bulles d’air dans le système. - Mettre 2 gouttes d’huile d’immersion (indice de réfraction de 1,33) ou environ 70 μL d’eau sur le dessus de l’objectif.
- Placez la cellule d’écoulement à l’intérieur du cadre de maintien dans sa position.
- Placez 2 gouttes d’huile d’immersion (indice de réfraction de 1,51) sur le dessus de la cellule d’écoulement.
- Allumez l’appareil laser dans la pince à épiler. Une fois qu’il est en cours d’exécution, allumez le laser de piégeage dans l’interface du logiciel à 100%.
- À l’aide de caméras de diagnostic (Z-finder), ajustez l’axe Z au milieu de la chambre entre la deuxième et la troisième réflexion (interfaces) où les anneaux de réfraction sont les plus grands, en tournant la microvis.
REMARQUE: Chaque fois que l’objectif est rapproché de la chambre de mesure et que le plan focal de l’objectif traverse l’interface entre deux phases, une réflexion peut être reconnue en mode Z-finder. Il y a 4 interfaces possibles: (i) huile d’eau / immersion et verre de fond (ii) verre inférieur et tampon à l’intérieur de la chambre (iii) tampon à l’intérieur de la chambre et verre supérieur (iv) verre supérieur et huile d’immersion pour condenseur. - Ajustez la position du condenseur (réglez le laser de piégeage à environ 50%) afin que le condenseur touche l’huile d’immersion sur le dessus de la chambre de mesure.
- Ajustez la mise au point en vous déplaçant lentement vers le bas / vers le haut avec le condenseur, de sorte qu’environ 10 bandes lumineuses sont affichées en mode lune (caméras de diagnostic).
3. Mesure de l’échantillon
- Incuber des billes enrobées d’anti-digoxigénine (AD) avec les constructions de l’échantillon (3 μL de suspension de billes AD à 0,1 % (p/v) + 4 μL d’échantillon) et avec 1 μL d’inhibiteurs de la RNase et 8 μL du tampon d’essai (300 mM KCl, 5 mM MgCl2, 20 mM HEPES, pH 7,6, 0,05 % Tween 20, 5 mM DTT) à RT pendant 10-20 min. Après l’incubation, diluer l’échantillon dans 500 μL de tampon d’essai.
REMARQUE: Il est recommandé d’ajouter des récupérateurs d’oxygène, en particulier lors des mesures de fluorescence au tampon afin de prévenir les dommages oxydatifs. Ici, un système de piégeage de l’oxygène contenant du glucose (8,3 mg / mL), de la glucose oxydase (40 U / mL) et de la catalase (185 U / mL) a été utilisé. - Mélanger 0,8 μL de billes enrobées de streptavidine (SA) à 1 % (p/v) avec 1 mL de tampon d’essai.
- Jetez l’eau des seringues et remplissez les seringues avec les suspensions/solutions respectives. Laver pendant au moins 2 minutes à environ 1 bar, puis commencer à attraper des perles.
REMARQUE: Selon la configuration expérimentale, différents arrangements de canaux peuvent être utilisés (Figure 3). Typiquement, un canal d’écoulement est rempli de billes d’anti-digoxigénine portant la molécule d’ARN. Un deuxième canal est rempli de perles enduites de streptavidine. Le canal tampon est utilisé pour former les attaches. Un quatrième canal peut être utilisé pour charger la protéine de liaison à l’ARN (Figure 3C), ou bien RBP peut être ajouté directement dans le canal tampon (Figure 3B). - Pour capturer les perles, éloignez les pièges optiques les uns des autres. Déplacez-vous d’abord vers le canal AD et attrapez une perle AD dans le piège 1. Ensuite, déplacez la scène vers le canal SA et attrapez une seule perle SA par le piège 2.
REMARQUE: Essayez de rester à l’interface des canaux tampon et perle pour éviter de perdre la perle déjà capturée ou pour éviter d’attraper plusieurs perles par le même piège. - Une fois que les billes de la bonne taille sont capturées, déplacez-vous vers le canal tampon et arrêtez le flux laminaire. Ensuite, effectuez un étalonnage de force pour vérifier la rigidité du piège. Les valeurs de rigidité respectives ne doivent pas différer de plus de 10 à 15 % dans l’axe x/y.
REMARQUE: Ajustez la puissance du laser ou la répartition laser entre les pièges en fonction de la taille du cordon. Il n’est pas nécessaire d’effectuer l’étalonnage de la force pour chaque paire de perles tant que les modèles de perles correspondent (score de similitude > 0,9). Cependant, il doit être effectué régulièrement, ou au moins chaque fois que les conditions de dosage sont modifiées. - Commencez à pêcher une attache en déplaçant les perles les unes près des autres, en attendant quelques secondes, puis en les séparant, répétez jusqu’à ce qu’une attache soit formée. Une formation d’attache entraîne une augmentation de la force mesurée en éloignant les deux perles l’une de l’autre.
REMARQUE: Pour éviter la formation de plusieurs attaches, les perles ne doivent pas être déplacées trop près. En attrapant une attache entre les deux perles, la qualité de l’attache peut être vérifiée en trouvant le plateau de surmenage. Le plateau doit être compris entre 50 et 60 pN pour une seule attache. - Après avoir obtenu une attache, commencez la mesure. En fonction du phénomène étudié, différentes configurations de mesure doivent être choisies (Figure 1B-D).
REMARQUE: Habituellement, au début de l’expérience, une expérience de rampe de force est menée pour vérifier la qualité de l’attache et sonder le comportement. Par la suite, on peut également commencer les expériences de force constante ou de position constante pour étudier plus avant les transitions d’état. Une fois qu’un nombre suffisant de mesures ont été effectuées sur un échantillon d’ARN pour déterminer son comportement, des facteurs marqués peuvent être ajoutés au système pour effectuer des mesures confocales. - Pour effectuer des mesures de fluorescence, allumez les lasers confocaux et l’unité de compteur de photons dans l’instrument de pince à épiler optique.
- Allumez le laser d’excitation de la longueur d’onde souhaitée dans l’interface logicielle et réglez la puissance du laser à 5% ou plus, en fonction du fluorophore.
REMARQUE: Tout en ne mesurant pas, abaissez le réglage de la puissance du laser d’excitation à 0% pour éviter des photodommages excessifs à l’échantillon. - Commencez à créer une image de l’échantillon à l’aide des fonctions d’image du logiciel.
REMARQUE: Afin d’obtenir des images bien focalisées, le plan focal du microscope confocal et les pièges optiques doivent être alignés. À cette fin, l’autofluorescence des billes de polystyrène dans le canal laser bleu peut être utilisée. Le plan focal des pièges optiques est déplacé vers le haut ou vers le bas dans l’axe z jusqu’à ce que l’image des perles atteigne son diamètre le plus élevé. À cette position, le signal de fluorescence de la molécule attachée entre les perles peut être mesuré. - Pour utiliser la fonction kymographe, spécifiez la position x-y de l’axe du kymographe afin qu’il permette la détection de l’attache entre les perles.
- Tout au long de la mesure, la composition du tampon peut être facilement modifiée en déplaçant les billes vers différents canaux ou en changeant le tampon fourni dans le système microfluidique.
4. Analyse des données
- Prétraitement des données brutes
- À l’aide d’un script simple, sous-échantillonnez suffisamment les données brutes pour (i) permettre un traitement ultérieur plus rapide des données, mais (ii) contenir toutes les informations critiques (Figure 4A). Habituellement, 100-5000 Hz convient à cet effet.
REMARQUE: La fréquence de collecte de données dans les expériences de pinces optiques est souvent plus élevée que nécessaire pour l’analyse - dans les expériences présentées, la fréquence de collecte de données est définie sur 78 125 Hz par défaut. Étant donné que l’espace de stockage est limité, il est pratique et rapide de réduire le taux d’échantillonnage des données. Ici, les données brutes ont été sous-échantillonnées d’un facteur 30. - Ensuite, utilisez un filtre de signal pour réduire le bruit de mesure à haute fréquence du signal (Figure 4A). Ajustez les paramètres de degré de filtre et de fréquence de coupure en conséquence pour optimiser la sortie des données de différentes expériences (Figure 5).
REMARQUE: Parmi les filtres de signal, le filtre Butterworth51 est l’un des plus utilisés. Un script python écrit sur mesure permettant le prétraitement des données brutes est fourni dans les données supplémentaires. Les paramètres de sous-échantillonnage et de filtrage du signal (fréquence de coupure, degré de filtre) doivent être optimisés pour différentes expériences.
- À l’aide d’un script simple, sous-échantillonnez suffisamment les données brutes pour (i) permettre un traitement ultérieur plus rapide des données, mais (ii) contenir toutes les informations critiques (Figure 4A). Habituellement, 100-5000 Hz convient à cet effet.
- Pour l’analyse des données de rampe de force, procédez comme suit.
- Marquez les étapes manuellement en trouvant les points correspondants sur le tracé de trajectoire de force ou en utilisant des scripts écrits sur mesure. Les étapes de dépliage sont caractérisées par une chute soudaine de la force combinée à une augmentation de la distance dans la courbe force-distance (FD).
- Une fois les événements de dépliage marqués, ajustez différentes régions de la courbe FD à l’aide de modèles appropriés (Figure 4D).
REMARQUE: Pour la région précédant la première étape de dépliage, l’attache peut être considérée comme « double brin » et est généralement ajustée à l’aide d’un modèle de chaîne extensible de type ver (WLC)47,52,53. Les parties après le premier événement de déploiement sont considérées comme une combinaison de nucléotides double brin (poignées) et de nucléotides simple brin (molécule d’ARN dépliée). Par conséquent, l’ajustement des données est plus complexe - généralement une combinaison de 2 modèles WLC ou WLC et de modèles à chaîne à jointure libre (FJC) est généralement utilisée36,39,52. Le modèle WLC extensible a deux paramètres d’ajustement principaux: la longueur du contour (LC) et la longueur de persistance (LP). La longueur du contour correspond à la longueur de la molécule entièrement étirée et la longueur de persistance définit les propriétés de flexion de la molécule d’intérêt. Le modèle peut être décrit avec l’équation suivante (1). WLC peut être utilisé pour modéliser le comportement des régions pliées et dépliées, bien que pour chacune d’entre elles, un modèle distinct avec des paramètres différents doive être utilisé.
(1)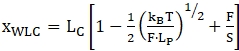
où x est l’extension, LC est la longueur du contour, F est la force, LP est la longueur de persistance, kB est la constante de Boltzmann, T est la température thermodynamique et S est le module d’étirement.
Le deuxième modèle appelé chaîne librement articulée (FJC) est couramment utilisé pour décrire le comportement des régions monocaténaires dépliées. Il utilise des paramètres similaires des polymères mais traite chaque unité de la « chaîne » comme une tige rigide, correspondant ici aux nucléotides de la région monocaténaire dépliée. L’équation suivante (2) décrit ce modèle :
(2)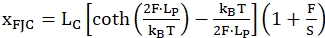
REMARQUE: Notre laboratoire a récemment développé un algorithme qui permet le traitement par lots des données brutes de rampe de force appelé « Analyse pratique de la pince optique TOol (POTATO)54 ». L’algorithme sous-échantillonne et filtre les données, puis il identifie les étapes de dépliage possibles et enfin effectue l’ajustement des données. Le POTATO est construit dans une interface utilisateur graphique (GUI) conviviale (https://github.com/REMI-HIRI/POTATO).
- Traitez les données à force constante comme suit :
REMARQUE : Les instructions suivantes peuvent être appliquées par analogie sur des données à position constante.- Pour les données de force constante, tracez la distance dans le temps (Figure 5). Un histogramme montrant la fréquence (nombre) de différentes conformations sur le changement relatif de position est un moyen utile de caractériser divers états dominants et mineurs (figure 7).
- Ajuster l’histogramme à l’aide de fonctions gaussiennes (multiples) pour estimer le pourcentage global de conformateurs individuels à une force donnée (Figure 7C). Les ajustements gaussiens, la position moyenne et l’écart-type décrivent la relation liée à la force entre différentes populations.
REMARQUE: Un script python écrit sur mesure permettant le prétraitement et l’ajustement gaussien bimodale de base des données à force constante est fourni dans les données supplémentaires. Les paramètres (fréquence de coupure, degré de filtre, moyennes attendues, valeurs d’écart-type et amplitudes) doivent être optimisés pour différentes expériences. - Ensuite, utilisez le modèle de Markov caché pour analyser plus en détail les états, ce qui peut révéler des intermédiaires de pliage supplémentaires (conformateurs)55. Pour plus d’informations sur la force constante et le modèle de Markov caché, on peut se référer à 55,56,57,58.
Résultats
Dans cette section, l’accent est principalement mis sur les mesures des interactions ARN-protéine/ligand par les pinces optiques à fluorescence. Pour une description des expériences générales de pinces optiques à ARN et des résultats représentatifs correspondants, voir 32. Pour une discussion plus détaillée des interactions ARN/ADN-protéine, voir également 1,2,26,59,60.
Discussion
Ici, nous démontrons l’utilisation de pinces optiques couplées à la fluorescence pour étudier les interactions et le comportement dynamique des molécules d’ARN avec divers ligands. Ci-dessous, les étapes critiques et les limites de la technique actuelle sont discutées.
Étapes critiques du protocole
Comme pour beaucoup d’autres méthodes, la qualité de l’échantillon est essentielle pour obtenir des données fiables. Par conséquent, pour obtenir des échant...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Anuja Kibe et Jun. Prof. Redmond Smyth d’avoir examiné le manuscrit de manière critique. Nous remercions Tatyana Koch pour son assistance technique experte. Nous remercions Kristyna Pekarkova pour son aide dans l’enregistrement de vidéos expérimentales. Les travaux de notre laboratoire sont soutenus par l’Association Helmholtz et le financement de la subvention Nr. 948636 (à NC) du Conseil européen de la recherche (ERC).
matériels
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| E. coli HB101 | lab collection | N/A | cloning of the vectors |
| Chemicals and enzymes | |||
| Sodium chloride | Sigma-Aldrich | 31424 | Buffers |
| Biotin-16-dUTP | Roche | 11093070910 | Biotinylation |
| BSA | Sigma-Aldrich | A4737 | Buffers |
| Catalase | Lumicks | N/A | Oxygen scavanger system |
| Dithiothreitol (DTT) | Melford Labs | D11000 | Buffers |
| DNAse I from bovine pancreas | Sigma-Aldrich | D4527 | in vitro transcription |
| dNTPs | Th.Geyer | 11786181 | PCR |
| EDTA | Sigma-Aldrich | E9884 | Buffers |
| Formamide | Sigma-Aldrich | 11814320001 | Buffers |
| Glucose | Sigma-Aldrich | G8270-1KG | Oxygen scavanger system |
| Glucose-oxidase | Lumicks | N/A | Oxygen scavanger system |
| HEPES | Carl Roth | HN78.3 | Buffers |
| Magnesium chloride | Carl Roth | 2189.1 | Buffers |
| Phusion DNA polymerase | NEB | M0530L | Gibson assembly, cloning |
| Potassium chloride | Merck | 529552-1KG | Buffers |
| PrimeSTAR GXL DNA Polymerase | Takara Bio Clontech | R050A | PCR |
| Pyrophosphotase, thermostabile, inorganic | NEB | M0296L | in vitro transcription |
| RNase Inhibitor | Molox | 1000379515 | Buffers |
| rNTPS | life technologies | R0481 | in vitro transcription |
| Sodium thiosulophate | Sigma-Aldrich | S6672-500G | Bleach deactivation |
| Sytox Green | Lumicks | N/A | confocal measurements |
| T4 DNA Polymerase | NEB | M0203S | Biotinylation |
| T5 exonuclease | NEB | M0363S | Gibson assembly, cloning |
| T7 RNA polymerase | Produced in-house | N/A | in vitro transcription |
| Taq DNA polymerase | NEB | M0267S | PCR |
| Taq ligase | Biozym | L6060L | Gibson assembly, cloning |
| TWEEN 20 BioXtra | Sigma-Aldrich | P7949 | Buffers |
| Kits | |||
| Monolith Protein Labeling Kit RED-NHS 2nd Generation (Amine Reactive) | Nanotemper | MO-L011 | Used for ribosome labeling |
| Purefrex 2.0 | GeneFrontier | PF201-0.25-EX | Ribosomes used for the labeling |
| Oligonucleotides | |||
| 5' handle T7 forward | Microsynth | custom order | 5’ - CTTAATACGACTCACTATAGGTC CTTTCTGTGGACGCC - 3’, used to generate OT in vitro transcription template in PCR 1 |
| 3’ handle reverse | Microsynth | custom order | 5' - GTCAAAGTGCGCCCCGTTATCC - 3', used to generate OT in vitro transcription template in PCR 1 |
| 5' handle forward | Microsynth | custom order | 5' - TCCTTTCTGTGGACGCCGC - 3' , used to generate 5' handle in PCR 2 |
| 5’ handle reverse | Microsynth | custom order | 5’ - CATAAATACCTCTTTACTAATATA TATACCTTCGTAAGCTAGCGT - 3’, used to generate 5' handle in PCR 2 |
| 3’ handle forward | Microsynth | custom order | 5' - ATCCTGCAACCTGCTCTTCGCC AG - 3', used to generate 3' handle in PCR 3 |
| 3’ handle reverse 5’labeled with digoxigenin | Microsynth | custom order | 5' -[Dig]-GTCAAAGTGCGCCCCGTTATCC - 3', used to generate 3' handle in PCR 3 |
| DNA vectors | |||
| pMZ_OT | produced in-house | N/A | further description in "Structural studies of Cardiovirus 2A protein reveal the molecular basis for RNA recognition and translational control" Chris H. Hill, Sawsan Napthine, Lukas Pekarek, Anuja Kibe, Andrew E. Firth, Stephen C. Graham, Neva Caliskan, Ian Brierley bioRxiv 2020.08.11.245035; doi: https://doi.org/10.1101/2020.08.11.245035 |
| Software and Algorithms | |||
| Atom | https://atom.io/packages/ide-python | N/A | |
| Bluelake | Lumicks | N/A | |
| Graphpad | https://www.graphpad.com/ | N/A | |
| InkScape 0.92.3 | https://inkscape.org/ | N/A | |
| Matlab | https://www.mathworks.com/products/matlab.html | N/A | |
| POTATO | https://github.com/lpekarek/POTATO.git | N/A | |
| RNAstructure | https://rna.urmc.rochester.edu/RNAstructure.html | N/A | |
| Spyder | https://www.spyder-ide.org/ | N/A | |
| Other | |||
| Streptavidin Coated Polystyrene Particles, 1.5-1.9 µm, 5 ml, 1.0% w/v | Spherotech | SVP-15-5 | |
| Anti-digoxigenin Coated Polystyrene Particles, 2.0-2.4 µm, 2 ml, 0.1% w/v | Spherotech | DIGP-20-2 | |
| Syringes | VWR | TERUMO SS+03L1 | |
| Devices | |||
| C-trap | Lumicks | N/A | optical tweezers coupled with confocal microscopy |
Références
- Balcerak, A., Trebinska-Stryjewska, A., Konopinski, R., Wakula, M., Grzybowska, E. A. RNA-protein interactions: disorder, moonlighting and junk contribute to eukaryotic complexity. Open Biology. 9 (6), 190096 (2019).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Firth, A. E., Brierley, I. Non-canonical translation in RNA viruses. Journal of General Virology. 93, 1385-1409 (2012).
- Caliskan, N., Peske, F., Rodnina, M. V. Changed in translation: mRNA recoding by −1 programmed ribosomal frameshifting. Trends in Biochemical Sciences. 40 (5), 265-274 (2015).
- Jaafar, Z. A., Kieft, J. S. Viral RNA structure-based strategies to manipulate translation. Nature Reviews Microbiology. 17 (2), 110-123 (2019).
- Eswarappa, S. M., et al. Programmed translational readthrough generates antiangiogenic VEGF-Ax. Cell. 157 (7), 1605-1618 (2014).
- Rodnina, M. V., et al. Translational recoding: canonical translation mechanisms reinterpreted. Nucleic Acids Research. 48 (3), 1056-1067 (2020).
- Li, Y., et al. Transactivation of programmed ribosomal frameshifting by a viral protein. Proceedings of the National Academy of Sciences. 111 (21), 2172 (2014).
- Napthine, S., et al. Protein-directed ribosomal frameshifting temporally regulates gene expression. Nature Communications. 8 (1), 15582 (2017).
- Patel, A., et al. Molecular characterization of the RNA-protein complex directing -2/-1 programmed ribosomal frameshifting during arterivirus replicase expression. Journal of Biological Chemistry. 295 (52), 17904-17921 (2020).
- Napthine, S., Bell, S., Hill, C. H., Brierley, I., Firth, A. E. Characterization of the stimulators of protein-directed ribosomal frameshifting in Theiler's murine encephalomyelitis virus. Nucleic Acids Research. 47 (15), 8207-8223 (2019).
- Marshall, R. A., Aitken, C. E., Dorywalska, M., Puglisi, J. D. Translation at the Single-Molecule Level. Annual Review of Biochemistry. 77 (1), 177-203 (2008).
- Rodnina, M. V. The ribosome in action: Tuning of translational efficiency and protein folding. Protein science : A publication of the Protein Society. 25 (8), 1390-1406 (2016).
- Rodnina, M. V., Fischer, N., Maracci, C., Stark, H. Ribosome dynamics during decoding. Philosophical Transactions of Royal Society of London B Biological Sciences. 372 (1716), (2017).
- Yan, S., Wen, J. D., Bustamante, C., Tinoco, I. Ribosome excursions during mRNA translocation mediate broad branching of frameshift pathways. Cell. 160 (5), 870-881 (2015).
- Liu, T., et al. Direct measurement of the mechanical work during translocation by the ribosome. eLife. 3, 03406 (2014).
- Desai, V. P., et al. Co-temporal force and fluorescence measurements reveal a ribosomal gear shift mechanism of translation regulation by structured mRNAs. Molecular Cell. 75 (5), 1007-1019 (2019).
- Choi, J., O'Loughlin, S., Atkins, J. F., Puglisi, J. D. The energy landscape of -1 ribosomal frameshifting. Science Advances. 6 (1), (2020).
- Prabhakar, A., Puglisi, E. V., Puglisi, J. D. Single-molecule fluorescence applied to translation. Cold Spring Harbor Perspectives in Biology. 11 (1), 032714 (2019).
- Bao, C., et al. mRNA stem-loops can pause the ribosome by hindering A-site tRNA binding. Elife. 9, 55799 (2020).
- Chen, J., Tsai, A., O'Leary, S. E., Petrov, A., Puglisi, J. D. Unraveling the dynamics of ribosome translocation. Current Opinion in Structural Biology. 22 (6), 804-814 (2012).
- Qu, X., et al. The ribosome uses two active mechanisms to unwind messenger RNA during translation. Nature. 475 (7354), 118-121 (2011).
- Zheng, Q., et al. Ultra-stable organic fluorophores for single-molecule research. Chemical Society Reviews. 43 (4), 1044-1056 (2014).
- Blanchard, S. C. Single-molecule observations of ribosome function. Current Opinion in Structural Biology. 19 (1), 103-109 (2009).
- Juette, M. F., et al. The bright future of single-molecule fluorescence imaging. Current Opinion in Chemical Biology. 20, 103-111 (2014).
- McCauley, M. J., Williams, M. C. Mechanisms of DNA binding determined in optical tweezers experiments. Biopolymers. 85 (2), 154-168 (2007).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters. 11 (5), 288-290 (1986).
- Bustamante, C., Smith, S. B., Liphardt, J., Smith, D. Single-molecule studies of DNA mechanics. Current Opinion in Structural Biology. 10 (3), 279-285 (2000).
- Choudhary, D., Mossa, A., Jadhav, M., Cecconi, C. Bio-molecular applications of recent developments in optical tweezers. Biomolecules. 9 (1), 23 (2019).
- Moffitt, J. R., Chemla, Y. R., Smith, S. B., Bustamante, C. Recent advances in optical tweezers. Annual Reviews of Biochemistry. 77, 205-228 (2008).
- Li, P. T. X., Vieregg, J., Tinoco, I. How RNA Unfolds and Refolds. Annual Review of Biochemistry. 77 (1), 77-100 (2008).
- Stephenson, W., Wan, G., Tenenbaum, S. A., Li, P. T. Nanomanipulation of single RNA molecules by optical tweezers. Journal of Visualized Experiments. (90), e51542 (2014).
- Halma, M. T. J., Ritchie, D. B., Cappellano, T. R., Neupane, K., Woodside, M. T. Complex dynamics under tension in a high-efficiency frameshift stimulatory structure. Proceedings of the National Academy of Sciences. 116 (39), 19500 (2019).
- Hansen, T. M., Reihani, S. N. S., Oddershede, L. B., Sørensen, M. A. Correlation between mechanical strength of messenger RNA pseudoknots and ribosomal frameshifting. Proceedings of the National Academy of Sciences of the United States of America. 104 (14), 5830-5835 (2007).
- Zhong, Z., et al. Mechanical unfolding kinetics of the SRV-1 gag-pro mRNA pseudoknot: possible implications for -1 ribosomal frameshifting stimulation. Science Reports. 6, 39549 (2016).
- McCauley, M. J., Rouzina, I., Li, J., Núñez, M. E., Williams, M. C. Significant differences in RNA structure destabilization by HIV-1 GagDp6 and NCp7 proteins. Viruses. 12 (5), 484 (2020).
- de Messieres, M., et al. Single-molecule measurements of the CCR5 mRNA unfolding pathways. Biophysics Journal. 106 (1), 244-252 (2014).
- Yang, L., et al. Single-molecule mechanical folding and unfolding of RNA hairpins: Effects of single A-U to A·C pair substitutions and single proton binding and implications for mRNA structure-induced -1 ribosomal frameshifting. Journal of American Chemical Society. 140 (26), 8172-8184 (2018).
- McCauley, M. J., et al. Targeted binding of nucleocapsid protein transforms the folding landscape of HIV-1 TAR RNA. Proceedings of the National Academy of Sciences of the United States of America. 112 (44), 13555-13560 (2015).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. High-resolution "Fleezers": Dual-trap optical tweezers combined with single-molecule fluorescence detection. Methods in Molecular Biology. 1486, 183-256 (2017).
- Yerramilli, V. S., Kim, K. H. Labeling RNAs in live cells using malachite green aptamer scaffolds as fluorescent probes. ACS Synthetic Biology. 7 (3), 758-766 (2018).
- Gross, P., Farge, G., Peterman, E. J., Wuite, G. J. Combining optical tweezers, single-molecule fluorescence microscopy, and microfluidics for studies of DNA-protein interactions. Methods in Enzymology. 475, 427-453 (2010).
- Zimmer, M. M., et al. The short isoform of the host antiviral protein ZAP acts as an inhibitor of SARS-CoV-2 programmed ribosomal frameshifting. Nature Communications. 12 (1), 7193 (2021).
- Neupane, K., Yu, H., Foster, D. A. N., Wang, F., Woodside, M. T. Single-molecule force spectroscopy of the add adenine riboswitch relates folding to regulatory mechanism. Nucleic acids research. 39 (17), 7677-7687 (2011).
- Ritchie, D. B., Soong, J., Sikkema, W. K., Woodside, M. T. Anti-frameshifting ligand reduces the conformational plasticity of the SARS virus pseudoknot. Journal of the American Chemical Society. 136 (6), 2196-2199 (2014).
- Janissen, R., et al. Invincible DNA tethers: covalent DNA anchoring for enhanced temporal and force stability in magnetic tweezers experiments. Nucleic Acids Research. 42 (18), 137 (2014).
- Smith, S. B., Cui, Y., Bustamante, C. Overstretching B-DNA: The elastic response of individual double-stranded and single-stranded DNA molecules. Science. 271 (5250), 795 (1996).
- Puljung, M. C., Zagotta, W. N. Labeling of specific cysteines in proteins using reversible metal protection. Biophysical Journal. 100 (10), 2513-2521 (2011).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Hill, C. H., et al. Structural and molecular basis for Cardiovirus 2A protein as a viral gene expression switch. Nature Communications. 12 (1), 7166 (2021).
- Butterworth, S. On the theory of filter amplifiers. Experimental Wireless and the Wireless Engineer. 7, 536-541 (1930).
- Wang, M. D., Yin, H., Landick, R., Gelles, J., Block, S. M. Stretching DNA with optical tweezers. Biophysics Journal. 72 (3), 1335-1346 (1997).
- Mukhortava, A., et al. Structural heterogeneity of attC integron recombination sites revealed by optical tweezers. Nucleic Acids Research. 47 (4), 1861-1870 (2019).
- Buck, S., Pekarek, L., Caliskan, N. POTATO: An automated pipeline for batch analysis of optical tweezers data. bioRxiv. , (2021).
- Zhang, Y., Jiao, J., Rebane, A. A. Hidden Markov modeling with detailed balance and its application to single protein folding. Biophysical Journal. 111 (10), 2110-2124 (2016).
- Sgouralis, I., Pressé, S. An introduction to infinite HMMs for single-molecule data analysis. Biophysics Journal. 112 (10), 2021-2029 (2017).
- Müllner, F. E., Syed, S., Selvin, P. R., Sigworth, F. J. Improved hidden Markov models for molecular motors, part 1: basic theory. Biophysical Journal. 99 (11), 3684-3695 (2010).
- Elms, P. J., Chodera, J. D., Bustamante, C. J., Marqusee, S. Limitations of constant-force-feedback experiments. Biophysical Journal. 103 (7), 1490-1499 (2012).
- Re, A., Joshi, T., Kulberkyte, E., Morris, Q., Workman, C. T. RNA-protein interactions: an overview. Methods Molecular Biology. 1097, 491-521 (2014).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature reviews. Molecular Cell Biology. 16 (9), 533-544 (2015).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Sunbul, M., Jäschke, A. SRB-2: a promiscuous rainbow aptamer for live-cell RNA imaging. Nucleic Acids Research. 46 (18), 110 (2018).
- Sanchez de Groot, N., et al. RNA structure drives interaction with proteins. Nature Communications. 10 (1), 3246 (2019).
- Zeffman, A., Hassard, S., Varani, G., Lever, A. The major HIV-1 packaging signal is an extended bulged stem loop whose structure is altered on interaction with the Gag polyprotein. Journal of Molecular Biology. 297 (4), 877-893 (2000).
- Mangeol, P., et al. Probing ribosomal protein-RNA interactions with an external force. Proceedings of the National Academy of Sciences. 108 (45), 18272 (2011).
- Luo, X., et al. Molecular mechanism of RNA recognition by Zinc-Finger antiviral protein. Cell Reports. 30 (1), 46-52 (2020).
- Qu, X., Lancaster, L., Noller, H. F., Bustamante, C., Tinoco, I. Ribosomal protein S1 unwinds double-stranded RNA in multiple steps. Proceedings of the National Academy of Science U. S. A. 109 (36), 14458-14463 (2012).
- Chandra, V., Hannan, Z., Xu, H., Mandal, M. Single-molecule analysis reveals multi-state folding of a guanine riboswitch. Nature Chemical Biology. 13 (2), 194-201 (2017).
- Savinov, A., Perez, C. F., Block, S. M. Single-molecule studies of riboswitch folding. Biochimica et Biophysica Acta. 1839 (10), 1030-1045 (2014).
- Kelly, J. A., et al. Structural and functional conservation of the programmed ribosomal frameshift signal of SARS coronavirus 2 (SARS-CoV-2). Journal of Biological Chemistry. 295 (31), 10741-10748 (2020).
- Neupane, K., et al. Structural dynamics of single SARS-CoV-2 pseudoknot molecules reveal topologically distinct conformers. Nature Communications. 12 (1), 4749 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zheng, Q., Jockusch, S., Zhou, Z., Blanchard, S. C. The contribution of reactive oxygen species to the photobleaching of organic fluorophores. Photochemistry and Photobiology. 90 (2), 448-454 (2014).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Rill, N., Mukhortava, A., Lorenz, S., Tessmer, I. Alkyltransferase-like protein clusters scan DNA rapidly over long distances and recruit NER to alkyl-DNA lesions. Proceedings of the National Academy of Science U. S. A. 117 (17), 9318-9328 (2020).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Wen, J. -. D., et al. Force unfolding kinetics of RNA using optical tweezers. I. Effects of experimental variables on measured results. Biophysical journal. 92 (9), 2996-3009 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon