JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
번역 조절에서 RNA 단백질 상호 작용을 연구하는 광학 핀셋
요약
이 프로토콜은 광학 핀셋을 사용하여 RNA 단백질 상호 작용을 연구하기위한 완벽한 실험 워크플로우를 제공합니다. 공초점 현미경 검사법과 광학 핀셋의 조합을 포함하여 몇 가지 가능한 실험 설정이 설명되어 있습니다.
초록
RNA는 기능에 필수적인 다양한 구조 접을 채택하여 세포의 다양한 프로세스에 영향을 미칠 수 있습니다. 또한, RNA의 구조 및 기능은 단백질, 대사산물 또는 기타 RNA와 같은 다양한 트랜스 작용 요인에 의해 변조될 수 있다. 프레임 변속 RNA 분자는, 예를 들면, 다른 개방독서 프레임으로 리보솜을 직접 번역하는 코딩 지구에 있는 규제 RNA이고, 그 에 의해 유전자 스위치로 작동합니다. 그(것)들은 또한 단백질 또는 그밖 트랜스 인자에 결합한 후에 다른 주름을 채택할 수 있습니다. 번역에서 RNA 결합 단백질의 역할을 해부하고 RNA 구조와 안정성을 조절하는 방법을 해부하기 위해 이러한 RNA 단백질 복합체의 상호 작용 및 기계적 특징을 동시에 연구하는 것이 중요합니다. 이 연구는 단일 분자 형광 결합 광학 핀셋을 사용하여 고해상도로 RNA 단백질 복합체의 형성 및 열역학 적 풍경을 탐구하는 방법을 보여줍니다. 예를 들어, ZS-CoV-2 프로그래밍된 리보소말 프레임 변속 요소와 아연 핑거 항바이러스 단백질의 짧은 이소폼을 결합한 상호작용이 정교화된다. 또한, 형광 라벨리보솜은 공초점 유닛을 사용하여 모니터링하여 궁극적으로 번역 신장의 연구를 가능하게 했습니다. 형광 결합 OT 분석은 다양한 RNA 단백질 복합체 또는 번역을 조절하는 트랜스 작용 인자를 탐구하기 위해 널리 적용 될 수 있으며 RNA 기반 유전자 조절의 연구를 용이하게 할 수 있습니다.
서문
MRNAs를 통해 DNA에서 단백질로 유전 정보의 전송은 세포 내의 거대 분자 상호 작용을 통해 모든 수준에서 정확하게 규제되는 복잡한 생화학 과정입니다. 번역 조절을 위해 RNA-단백질 상호 작용은 다양한 자극및 신호1,2에 신속하게 반응하는 중요한 역할을 부여합니다. 일부 RNA 단백질 상호 작용은 mRNA 안정성에 영향을 미치고 따라서 RNA가 번역 활성시간을 변경합니다. 그밖 RNA 단백질 상호 작용은 정지 코돈 판독과 같은 재코딩 기계장치와 연관됩니다, 우회, 또는 프로그램된 리보소말 프레임 변속 (PRF)3,4,5,6,7. 최근에는 다수의 RNA 결합 단백질(RBP)이 자극성 mRNA 원소 및 번역 기계와 상호 작용하여 셀7,8,9,10,11에서 언제 그리고 얼마나 많은 재코딩이 발생할지 지시하는 것으로 입증되었다. 따라서, 번역에서 RNA 결합 단백질의 역할을 해부하고 RNA 구조 및 안정성을 조절하는 방법, 이러한 RNA-단백질 복합체의 상호 작용 원리 및 기계적 특성을 자세히 연구하는 것은 중추적인 것이다.
수십 년의 작업은 속도와 정확성을 달성하기 위해 번역 기계의 RNA와 단백질 구성 요소 사이의 복잡한 통신에 의존하는 다단계 및 다중 구성 요소 번역 과정을 연구하는 토대를 마련했습니다12,13,14. 복잡한 규제 이벤트를 이해하는 중요한 다음 단계 는 고정밀12,15,16,17에서 번역 하는 동안 힘, 기간 및 구조 결정자를 결정 하는. RNA 형태 역학및 특히 번역 도중 RNA 구조 에 작용하는 트랜스 작용 보조 요인의 연구는 광학 핀셋 또는 제로 모드 파도관을 포함하여 단하나 분자 공구의 출현에 의해 더 조명되었습니다16,17,18,19,20,21,22,23,24 ,25,26.
광학 핀셋(OT)은 매우 정밀한 단일 분자 기법을 나타내며, 전사를 포함한 많은 종류의 RNA 의존적 동적 공정을 연구하고, 번역26,27,28,29,30,31,32를 연구한다. 광학 핀셋의 사용은 분자 상호 작용, 핵산 구조 및 열역학 적 특성, 운동학 및 이러한 공정의 정력적 16,17,17,22,34,35,35,37,38,38,39의 탐구를 허용했습니다. . 광학 핀셋 분석은 집중 된 레이저 빔과 미세한 물체의 함정을 기반으로합니다. 일반적인 OT 실험에서, 관심 있는 분자는 2개의 투명한 (일반적으로 폴리스티렌) 구슬 사이 테더드됩니다 (그림 1A)27. 이 구슬은 스프링처럼 행동 광학 트랩에 의해 잡힐 수 있습니다. 따라서, 분자상에 가해지는 힘은 집중된 레이저 빔(trap center)의 중심으로부터의 비드변위에 기초하여 계산될 수 있다. 최근에는 광학 핀셋이 공초점 현미경검사(도 1B)와 결합되어 형광 또는 Förster 공명 에너지 전달(FRET) 측정40,41,42를 가능하게 합니다. 이렇게 하면 동시 측정을 가능하게 하는 완전히 새로운 실험 분야가 열리므로 힘 분광법과 형광 데이터의 정확한 상관 관계가 있습니다.
여기서는, 우리는 번역 프레임 이동을 조절하는 단백질-RNA 상호 작용을 연구하기 위하여 공초점 현미경검사법과 결합된 광학 핀셋을 사용하여 실험을 보여줍니다. 목표와 응축기 사이에 5개의 채널이 있는 플로우 셀은 라미나르 흐름을 통해 연속 적인 샘플 응용을 가능하게 합니다. 미세 유체 채널을 통해 다양한 구성 요소를 직접 주입하여 실습 시간을 줄이면서 실험 전반에 걸쳐 샘플 소비가 거의 되지 않습니다.
첫째, OT 실험의 설계를 지원하는 기본 지침이 제안되고 다양한 설정의 함정뿐만 아니라 장점이 논의됩니다. 다음으로, 샘플 및 실험 워크플로우의 준비가 설명되고, 데이터 분석을 위한 프로토콜이 제공됩니다. 예를 나타내기 위해, 우리는 RNA 스트레칭 실험으로부터 얻은 결과를 설명하고, 다른 판독 프레임으로부터 바이러스 RNA의 번역을 변화시키는 아연 핑거 항바이러스 단백질(ZAP)의 짧은 이소폼을 트랜스-작용 인자로 SARS-CoV-2 프레임 이동 RNA 요소(도 2A)를 연구하는 결과를 설명한다. 또한, 형광 라벨리보솜이 OT 공초점 분석에서 사용될 수 있음을 입증하여 번역 기계의 공정성과 속도를 모니터링하는 데 유용할 것입니다. 여기에 제시된 방법은 번역의 다양한 측면을 연구하기 위해 다른 버퍼, 리간드 또는 기타 세포 구성 요소의 효과를 빠르게 테스트하는 데 사용할 수 있습니다. 마지막으로 일반적인 실험적 함정과 문제를 해결하는 방법에 대해 논의합니다. 아래, 실험 디자인의 몇 가지 중요한 포인트가 설명되어 있습니다.
구성 설계
원칙적으로 OT 호환 RNA 구조를 만드는 두 가지 일반적인 접근 방식이 있습니다. 첫 번째 접근법은 보완적인 DNA 손잡이로 혼성화되는 긴 RNA 분자를 채택하여 중간에 단일 좌초 RNA 서열을 측면시키는 2개의 RNA/DNA 하이브리드 영역으로 구성된 구조를 산출한다(도 2B). 이 접근법은 대부분의 OT RNA 실험에서 33,44,45에 사용됩니다.
두 번째 접근법은 짧은 (약 20 nt) overhangs15,17로 dsDNA 핸들을 활용합니다. 이 돌출은 RNA 분자로 혼성화됩니다. 디자인에서 더 복잡하더라도, dsDNA 처리의 사용은 DNA/RNA 하이브리드 시스템의 몇몇 한계를 극복합니다. 원칙적으로 매우 긴 핸들(>10kb)을 구현할 수 있어 공초점 측정에 더 편리합니다. 또한, RNA 분자는 테더 안정성을 높이기 위해 DNA 손잡이로 회개될 수 있다.
최종 라벨링 전략
구조는 강력한 분자 상호 작용을 통해 구슬에 묶여 있어야합니다. 구슬46에 손잡이의 공유 결합에 사용할 수있는 접근 방법이 있지만, 스트렙타비딘 - 비오틴 및 디곡시게닌 항체와 같은 강력하지만 비 공유 상호 작용은 OT 실험에서 일반적으로 사용된다15,33,35,45. 설명된 프로토콜에서, 구조는 비오틴 또는 디곡시게닌으로 표지되고, 구슬은 디곡시게닌에 대하여 스트렙타비딘 또는 항체로 각각 코팅된다(도 1A). 이 방법은 최대 약 60 pN(밧줄 당)47까지 힘을 적용하는 데 적합합니다. 더욱이, 다른 5' 및 3'라벨링 전략을 사용하면 구슬사이에 형성된 밧줄의 방향을 결정할 수 있다17.
형광 측정을 위한 단백질 라벨링
공초점 화상 진찰을 위해, 형광 표지를 위한 몇몇 일반적으로 이용된 접근이 있습니다. 예를 들어, 형광은 단백질에서 기본적으로 발견되거나 반응성 유기 그룹을 통해 현장 지향 돌연변이 발생에 의해 도입되는 아미노산 잔류물에 공유적으로 부착될 수 있다. 티올 또는 아민 반응성 염료는 각각 시스테인 및 리신 잔류물의 라벨링에 사용할 수 있습니다. 라벨링48,49의 특이성을 증가시키는 몇 가지 가역적인 보호 방법이 있지만, 토착 단백질은 일반적으로 여러 잔류물에서 표지될 것입니다. 형광의 작은 크기는 이점을 부여 할 수 있지만, 비 특이적 라벨은 단백질 활성을 방해할 수 있으며 따라서 신호 강도는 다를 수 있습니다49. 또한 라벨링 효율에 따라 신호 강도는 상이한 실험 간에 다를 수 있다. 따라서 실험 전에 활동 검사를 수행해야 합니다.
관심 있는 단백질에 His-tag 또는 스트렙 태그와 같은 N-또는 C 단말 태그가 포함된 경우 이러한 태그의 특정 라벨링은 또 다른 인기 있는 접근 방식을 나타냅니다. 더욱이, 태그 표적 라벨링은 플루오로포어가 단백질 활성을 방해할 가능성을 감소시키고 용해도를 향상시킬 수 있다49. 그러나 태그별 라벨링은 일반적으로 모노 플루오로포어 표지 단백질을 생성하므로 감지하기가 어려울 수 있습니다. 특정 라벨링의 또 다른 방법은 항체를 채택함으로써 달성될 수 있다.
미세 유체 설정
OT와 미세 유체 시스템의 조합은 다른 실험 조건 사이의 빠른 전환을 허용합니다. 더욱이, 현재 시스템은 유동 방향에 비해 수직 방향으로 다른 채널에서 액체의 혼합을 배제하는 유동 셀 내부의 라미나르 흐름을 유지하는 것을 활용한다. 따라서, 라미나르 유량은 실험 설계에 특히 유리하다. 현재 최대 5개의 채널을 사용하는 유동 셀이 일반적으로 사용됩니다(그림 3).
프로토콜
1. 샘플 준비
- 핸들 서열로 작용하는 람다 DNA 단편을 포함하는 벡터내관심서열을 복제한다(도 2)43,50.
- 먼저 PCR을 통해 후속 체외 전사를 위한 DNA 템플릿을 생성한다(도 2B; 반응 1). 이 PCR 단계에서, T7 프로모터는 감각 DNA 분자32,33,43,50의 끝에 추가됩니다. 표 1에 따라 PCR 반응을 설정합니다. 온도 사이클러에서 적절한 사이클로 50 μL 알리코에서 PCR을 실행합니다.
- 두 개의 별도의 PCR 반응(표 1, 그림 2B; 반응 2 및 3)으로 핸들을 준비합니다. 먼저 PCR별로 5' 핸들을 생성합니다. 이어서, 3' 핸들을 생성하고 동시에 5'디곡시게닌 라벨프라이머32,33,43,50을 사용하여 디곡시게닌으로 라벨을 붙입니다.
- PCR 후 실리카 스핀 컬럼을 사용하여 DNA를 정화합니다.
- T7 RNA 폴리머라제(표 2)를 이용한 체외 전사 반응을 수행한다(표 2)32,33,43,50. RNA의 길이에 따라 2-4 시간 동안 37°C에서 반응을 배양한다. 다음으로, 반응에 DNase I를 추가하고 DNA 템플릿을 소화하기 위해 37°C에서 30분 동안 배양한다. 실리카 스핀 컬럼을 사용하여 RNA를 정화합니다.
- 5' 핸들(표 3)의 라벨링 반응 동안, T4 DNA 폴리머라세38,50에 의해 핸들의 3'끝에 비오틴-16-dUTP를 추가한다. 1-2 시간 동안 실온에서 반응을 수행하십시오. 그 후 실리카 스핀 컬럼을 사용하여 DNA를 정화합니다.
참고: 5' 핸들은 3'끝(그림 2B)에 레이블을 지정해야 하므로 PCR 중에 레이블을 수행할 수 없습니다. - 상기 에 언급된 성분-5' 손잡이(3'biotin로 표시), 3'핸들(5' 디곡시게닌으로 표시), RNA-1:1:1 어닐링 버퍼(80% 포르마미드, 400mM NaCl, 40m HEPES, pH 7.5, 0.5m EDTA) 및 원하(RNA)에서 1:1:1 m 의 어퍼비로 혼합하여 원하는 RNA(8M EDTA)를 원하는 것으로 나타났다. 어닐링 혼합물을 최대 85°C까지 10분간 가열한 다음 천천히 식힙니다.
- 어닐드 샘플을 3M 아세테이트(pH 5)의 1/10 부피와 혼합하고, 3권의 얼음-차가운 에탄올을 혼합하고 -80°C에서 -80°C에서 하룻밤 사이에 -20°C 이상을 배양한다.
- 4°C에서 30분 동안 15,000 g 의 × 샘플을 원심분리합니다. 상체를 버리고 진공 상태에서 펠릿 (일반적으로 보이지 않음)을 건조시십시오.
- 마지막으로, RNase가없는 물의 50 μL의 펠릿을 다시 중단하고 알리쿼트합니다. 사용 될 때까지 -80 °C에 알리쿼트저장합니다. 단기 저장을 위해 시료도 -20°C로 저장할 수 있습니다.
2. 악기 설정
참고: 다음 프로토콜은 LUMICKS 회사의 상업용 광학 핀셋 계측기 C-Trap에 최적화되어 있습니다. 따라서 다른 광학 핀셋 계측기를 사용하는 동안 제시된 단계에 대한 조정이 필요할 수 있습니다. 사용하지 않을 경우 기계의 미세 유체 시스템은 표백제 (hypochlorite 용액) 보관되며 사용하기 전에 세척해야합니다.
- 표백제를 버리고 주사기를 RNase가없는 물 1 mL로 채웁니다.
- 0.5M 나트륨 티오술파테의 50μL을 RNase 가 없는 물의 최소 1mL에 추가하고 시스템의 나머지 표백제를 제거하기 위해 시스템(1bar, 적어도 0.5mL)을 철저히 세척합니다.
- 주사기에서 티오술파테 나트륨 용액을 버리십시오. 주사기를 신선한 것으로 바꾸고 RNase가 없는 물0.5mL이상으로 시스템을 세척하십시오.
참고: 미세 유체 시스템이 시스템의 기포를 피하기 위해 건조하게 실행되지 않도록 주의하십시오. - 2 방울의 침지 오일 (굴절률 1.33) 또는 목표 위에 약 70 μL의 물을 넣습니다.
- 흐름 셀을 유지 프레임 내부에 배치합니다.
- 침수 오일 2 방울(굴절률 1.51)을 유량 셀 위에 놓습니다.
- 핀셋 머신에서 레이저 장치를 켭니다. 실행되면 소프트웨어 인터페이스의 트래핑 레이저를 100%로 켭니다.
- 진단 카메라(Z-finder)를 사용하여, 마이크로 나사를 돌려 굴절 링이 가장 큰 두 번째 반사(인터페이스) 사이의 챔버 의 중간으로 Z축을 조정합니다.
참고: 목표가 측정 챔버에 더 가깝게 이동하고 목표의 초점 평면이 두 단계 사이의 인터페이스를 교차할 때마다 Z 파인더 모드에서 반사를 인식할 수 있습니다. 가능한 4 개의 인터페이스가 있습니다 : (i) 물 / 침수 오일 및 바닥 유리 (ii) 바닥 유리 및 챔버 내부버퍼 (iii) 챔버 내부 버퍼 및 상단 유리 (iv) 상단 유리 및 응축기용 침지 오일. - 응축기 위치(트랩핑 레이저를 약 50%로 설정)를 조정하여 응축기가 측정 챔버 위에 있는 침지 오일을 만지도록 합니다.
- 응축기로 천천히 아래/위로 이동하여 초점을 조정하므로 약 10 개의 라이트 밴드가 달 모드 (진단 카메라)에 표시됩니다.
3. 샘플 측정
- 시료 구조(0.1%(w/v) AD 비드 서스펜션 +4 μL+4 μL의 샘플과 분석 버퍼(300mM KCl)의 8μL을 가진 비착성 안티곡시게닌 코팅 구슬(AD)을 배양합니다. 5 mM MgCl2, 20 mM HEPES, pH 7.6, 0.05% Tween 20, 5 mM DTT) 10-20 분 RT. 인큐베이션 후, 분석 버퍼의 500 μL로 샘플을 희석.
참고: 산소 침전을 첨가하는 것이 좋습니다. 여기서 포도당(8.3 mg/mL), 포도당 산화제(40 U/mL) 및 카탈라아제(185 U/mL)를 포함하는 산소 제거제 시스템이 사용되었다. - 1%의 0.8 μL(w/v) 스트렙타비딘 코팅(SA) 구슬을 1mL의 분석 버퍼와 혼합합니다.
- 주사기에서 물을 버리고 주사기를 각각의 서스펜션/솔루션으로 채웁니다. 약 1 바에서 적어도 2 분 동안 씻은 다음 구슬을 잡기 시작합니다.
참고: 실험적인 설정에 따라 다양한 채널 배열이 사용될 수 있습니다(그림 3). 전형적으로, 하나의 유량 채널은 RNA 분자를 운반하는 항 디곡시겐 구슬로 채워져 있다. 두 번째 채널은 스트렙타비딘 코팅 구슬로 채워져 있습니다. 버퍼 채널은 테더를 형성하는 데 사용됩니다. 제4 채널은 RNA 결합 단백질(도 3C)을 로드하거나 대안적으로 RBP를 완충 채널에 직접 첨가할 수 있도록 사용될 수 있다(도 3B). - 구슬을 잡려면 광학 트랩을 서로 분리합니다. 먼저 AD 채널로 이동하여 트랩 1에서 AD 비드를 잡습니다. 다음으로, SA 채널로 무대를 이동하고 트랩 2에 의해 하나의 SA 비드를 잡을 수 있습니다.
참고: 버퍼 및 비드 채널의 인터페이스에 머무르거나 이미 잡힌 비드를 잃지 않도록 하거나 동일한 트랩으로 여러 구슬을 잡지 않도록 하십시오. - 올바른 크기의 구슬이 캡처되면 버퍼 채널로 이동하여 라미나르 흐름을 중지합니다. 다음으로, 트랩 강성을 확인하기 위해 힘 보정을 수행합니다. 각각의 강성 값은 x/y 축에서 10-15% 이상 차이가 없어야 합니다.
참고: 비드 크기에 따라 레이저 전원 또는 레이저 분할 트랩 사이를 조정합니다. 비드 템플릿이 일치하는 한 모든 비드 쌍에 대해 강제 보정을 수행할 필요가 없습니다(유사성 점수 > 0.9). 그러나, 그것은 정기적으로 수행 해야 합니다., 또는 적어도 때마다 분석 조건이 변경 됩니다. - 구슬을 서로 가까이 이동시키고 몇 초 동안 기다린 다음 다시 떨어져 이동하여 밧줄이 형성될 때까지 반복하여 밧줄낚시를 시작합니다. 밧줄 형성은 두 구슬을 서로 떼어낼 때 측정된 힘의 증가를 초래한다.
참고: 여러 테더의 형성을 피하려면 구슬을 너무 가깝게 이동해서는 안 됩니다. 두 구슬 사이의 밧줄을 잡으면서, 밧줄의 품질은 오버 스트레칭 고원을 찾아 확인할 수 있습니다. 고원은 단일 밧줄에 대해 50에서 60 pN 사이여야합니다. - 밧줄을 얻으면 측정을 시작합니다. 다른 측정 설정을 연구 현상에 따라 선택되어야한다 (그림 1B-D).
참고: 일반적으로 실험 의 시작 부분에서, 힘 램프 실험은 밧줄 의 품질을 확인하고 동작을 조사하기 위해 수행됩니다. 그 후, 하나는 또한 상태 전환을 더 연구하기 위해 상수 력 또는 상수 위치 실험을 시작할 수 있습니다. RNA 샘플에서 충분한 수의 측정이 수행되어 그 동작을 결정하면, 표지된 인자를 시스템에 추가하여 공초점 측정을 수행할 수 있습니다. - 형광 측정을 수행하려면 광학 핀셋 기기에서 공초점 레이저 및 광자 카운터 유닛을 켭니다.
- 소프트웨어 인터페이스에서 원하는 파장의 여기 레이저를 켜고 불소에 따라 레이저의 힘을 5% 이상으로 설정합니다.
참고: 저측정하지는 않지만 샘플에 대한 과도한 광손상을 방지하기 위해 여기 레이저의 전력 설정을 0%로 낮춥니다. - 소프트웨어의 이미지 기능을 사용하여 샘플 이미징을 시작합니다.
참고: 잘 초점을 맞춘 이미지를 얻으려면 공초점 현미경 및 광학 트랩의 초점 평면을 정렬해야 합니다. 이를 위해, 블루 레이저 채널에서 폴리스티렌 구슬의 자동 불발성을 사용할 수 있다. 광학 트랩의 초점 평면은 구슬의 이미지가 가장 높은 직경에 도달 할 때까지 z 축에서 위 또는 아래로 이동합니다. 이 위치에서, 구슬 사이에 테더드된 분자로부터의 형광 신호를 측정할 수 있다. - kymograph 함수를 사용하려면 구슬 사이의 밧줄을 감지할 수 있도록 kymograph 축의 x-y 위치를 지정합니다.
- 측정 전반에 걸쳐, 완충 조성물은 구슬을 다른 채널로 이동하거나 미세 유체 시스템에 제공된 버퍼를 변경하여 쉽게 변경할 수 있습니다.
4. 데이터 분석
- 원시 데이터 사전 처리
- 간단한 스크립트를 사용하여 원시 데이터를 다운샘플링하여 (i) 더 빠른 후속 데이터 처리를 허용하지만 (ii) 여전히 모든 중요한 정보(그림 4A)가 포함되어 있습니다. 일반적으로 100-5000 Hz는 이 목적에 적합합니다.
참고: 광학 핀셋 실험에서 데이터 수집 빈도는 분석에 필요한 것보다 더 높은 경우가 많습니다 - 제시된 실험에서 데이터 수집 빈도는 기본적으로 78 125Hz로 설정됩니다. 저장 공간이 제한되어 있으므로 데이터의 샘플링 속도를 줄이기 위해 편리하고 시간 절약이 가능합니다. 여기서 원시 데이터는 30배로 다운샘플링되었습니다. - 다음으로 신호 필터를 사용하여 신호에서 고주파 측정 노이즈를 줄입니다(그림 4A). 필터 정도 및 컷오프 주파수 매개변수를 적절히 조정하여 다양한 실험의 데이터 출력을 최적화합니다(그림 5).
참고: 신호 필터 중, 버터 워스 필터51 은 가장 널리 사용되는 중 하나입니다. 원시 데이터의 사전 처리를 허용하는 맞춤형 파이썬 스크립트가 추가 데이터에 제공됩니다. 다운샘플링 및 신호 필터링 매개 변수(컷오프 주파수, 필터 정도)는 다양한 실험에 최적화되어야 합니다.
- 간단한 스크립트를 사용하여 원시 데이터를 다운샘플링하여 (i) 더 빠른 후속 데이터 처리를 허용하지만 (ii) 여전히 모든 중요한 정보(그림 4A)가 포함되어 있습니다. 일반적으로 100-5000 Hz는 이 목적에 적합합니다.
- 강제 램프 데이터 분석을 보려면 다음 단계를 사용합니다.
- 힘 궤적 플롯에서 해당 점을 찾거나 사용자 지정 작성된 스크립트를 사용하여 수동으로 단계를 표시합니다. 전개 단계는 포스 거리(FD) 곡선의 거리 증가와 함께 급격한 힘의 하강이 특징입니다.
- 전개 이벤트가 표시되면 적절한 모델을 사용하여 FD 곡선의 다른 영역에 적합합니다(그림 4D).
참고: 첫 번째 전개 단계 이전의 영역의 경우 밧줄은 "이중 좌초"로 간주될 수 있으며 일반적으로 확장 가능한 웜 유사 체인 모델(WLC)47,52,53을 사용하여 적합합니다. 첫 번째 전개 사건 이후에 부품은 이중 가닥 뉴클레오티드(handles) 및 단일 가닥 뉴클레오티드(전개된 RNA 분자)의 조합으로 간주됩니다. 따라서 데이터 피팅은 더 복잡합니다 - 일반적으로 2개의 WLC 모델 또는 WLC 및 자유 조인트 체인(FJC) 모델의 조합이 36,39,52로 채용된다. 확장 가능한 WLC 모델에는 등고선 길이(LC)와 지속성 길이(LP)가 두 가지 주 맞춤 매개 변수가 있습니다. 윤곽 길이는 완전히 늘어난 분자의 길이와 지속성 길이에 해당하며 관심 있는 분자의 굽힘 특성을 정의합니다. 모델은 다음 방정식(1)으로 설명할 수 있다. WLC는 서로 다른 매개 변수를 가진 각각의 별도의 모델을 사용해야 하지만 접힌 영역과 전개된 영역의 동작을 모델링하는 데 사용할 수 있습니다.
(1)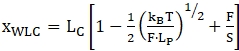
여기서 x는 확장, LC 는 윤곽 길이, F는 힘, LP 는 지속성 길이, kB 는 볼트만 상수, T는 열역학 온도, S는 스트레치 계수이다.
Freely-jointed 체인(FJC)이라고 하는 두 번째 모델은 일반적으로 전개된 단일 좌초 영역의 동작을 설명하는 데 사용됩니다. 그것은 중합체의 유사한 매개 변수를 사용하지만 "사슬"의 각 단위를 단단한 막대로 취급하며, 여기서 전개 된 단일 좌초 영역의 뉴클레오티드에 해당합니다. 다음 방정식(2)은 이 모델을 설명합니다.
(2)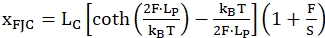
참고: 우리 연구소는 최근 "실용적인 광학 핀셋 분석 TOol (감자)54"라는 원시 힘 램프 데이터의 배치 처리를 허용하는 알고리즘을 개발했습니다. 알고리즘은 데이터를 다운샘플링하고 필터링한 다음 가능한 전개 단계를 식별하고 마지막으로 데이터 피팅을 수행합니다. 감자는 사용자 친화적인 그래픽 사용자 인터페이스(GUI) (https://github.com/REMI-HIRI/POTATO)로 구축되었습니다.
- 다음과 같이 일정한 힘 데이터를 처리합니다.
참고: 다음 지침은 상수 위치 데이터에 유사하게 적용할 수 있습니다.- 상수 력 데이터의 경우 시간별 거리를 플롯합니다(그림 5). 위치의 상대적 변화에 대한 상이한 적합성의 주파수(counts)를 나타내는 히스토그램은 다양한 지배적및 소소상태를 특성화하는 유용한 방법이다(도 7).
- (여러) 가우시안 함수를 사용하여 히스토그램에 맞게 지정된 힘에서 개별 순응의 전체 비율을 추정합니다(그림 7C). 가우시안은 적합, 평균 위치, 표준 편차는 다른 인구 사이의 힘 관련 관계를 설명합니다.
참고: 상수 데이터의 사전 처리 및 기본 바이모달 가우시안 피팅을 허용하는 맞춤형 파이썬 스크립트가 추가 데이터에 제공됩니다. 매개 변수(차단 주파수, 필터 정도, 예상 수단, 표준 편차 값 및 진폭)는 다양한 실험에 최적화되어야 합니다. - 다음으로, 숨겨진 마르코프 모델을 사용하여 상태를 추가로 분석하여 추가 접이식 중간(conformers)55를 발견할 수 있습니다. 상수력 및 히든 마르코프 모델에 대한 자세한 내용은 55,56,57,58을 참조할 수 있습니다.
결과
이 섹션에서는, 초점은 주로 형광 광학 핀셋에 의한 RNA 단백질/리간드 상호작용의 측정에 주어집니다. 일반적인 RNA 광학 핀셋 실험 및 해당 대표 결과에 대한 설명을 보려면 see32. RNA/DNA 단백질 상호 작용 에 대한 자세한 논의를 위해 1,26,59,60을 참조하십시오
토론
여기서는 다양한 리간드를 사용한 RNA 분자의 상호 작용과 동적 행동을 연구하기 위해 형광 결합 광학 핀셋의 사용을 입증합니다. 아래에서 현재 기술의 중요한 단계와 한계에 대해 설명합니다.
프로토콜의 중요한 단계
다른 많은 방법에 관해서는, 샘플의 품질은 신뢰할 수있는 데이터를 얻기 위해 중추적 인이다. 따라서 가능한 최고 품질의 샘플을 얻으려면 시...
공개
저자는 공개 할 것이 없습니다.
감사의 말
우리는 아누자 키베와 준 교수 레드몬드 Smyth 비판적으로 원고를 검토해 주셔서 감사합니다. 우리는 전문 기술 지원을 위한 Tatyana Koch에게 감사드립니다. 실험적인 비디오 녹화에 도움을 주신 크리스티나 페카르코바에게 감사드립니다. 우리의 실험실에서 작품은 헬름홀츠 협회에 의해 지원되고 유럽 연구 위원회 (ERC) 그랜트 nr. 948636 (NC에)에서 자금.
자료
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| E. coli HB101 | lab collection | N/A | cloning of the vectors |
| Chemicals and enzymes | |||
| Sodium chloride | Sigma-Aldrich | 31424 | Buffers |
| Biotin-16-dUTP | Roche | 11093070910 | Biotinylation |
| BSA | Sigma-Aldrich | A4737 | Buffers |
| Catalase | Lumicks | N/A | Oxygen scavanger system |
| Dithiothreitol (DTT) | Melford Labs | D11000 | Buffers |
| DNAse I from bovine pancreas | Sigma-Aldrich | D4527 | in vitro transcription |
| dNTPs | Th.Geyer | 11786181 | PCR |
| EDTA | Sigma-Aldrich | E9884 | Buffers |
| Formamide | Sigma-Aldrich | 11814320001 | Buffers |
| Glucose | Sigma-Aldrich | G8270-1KG | Oxygen scavanger system |
| Glucose-oxidase | Lumicks | N/A | Oxygen scavanger system |
| HEPES | Carl Roth | HN78.3 | Buffers |
| Magnesium chloride | Carl Roth | 2189.1 | Buffers |
| Phusion DNA polymerase | NEB | M0530L | Gibson assembly, cloning |
| Potassium chloride | Merck | 529552-1KG | Buffers |
| PrimeSTAR GXL DNA Polymerase | Takara Bio Clontech | R050A | PCR |
| Pyrophosphotase, thermostabile, inorganic | NEB | M0296L | in vitro transcription |
| RNase Inhibitor | Molox | 1000379515 | Buffers |
| rNTPS | life technologies | R0481 | in vitro transcription |
| Sodium thiosulophate | Sigma-Aldrich | S6672-500G | Bleach deactivation |
| Sytox Green | Lumicks | N/A | confocal measurements |
| T4 DNA Polymerase | NEB | M0203S | Biotinylation |
| T5 exonuclease | NEB | M0363S | Gibson assembly, cloning |
| T7 RNA polymerase | Produced in-house | N/A | in vitro transcription |
| Taq DNA polymerase | NEB | M0267S | PCR |
| Taq ligase | Biozym | L6060L | Gibson assembly, cloning |
| TWEEN 20 BioXtra | Sigma-Aldrich | P7949 | Buffers |
| Kits | |||
| Monolith Protein Labeling Kit RED-NHS 2nd Generation (Amine Reactive) | Nanotemper | MO-L011 | Used for ribosome labeling |
| Purefrex 2.0 | GeneFrontier | PF201-0.25-EX | Ribosomes used for the labeling |
| Oligonucleotides | |||
| 5' handle T7 forward | Microsynth | custom order | 5’ - CTTAATACGACTCACTATAGGTC CTTTCTGTGGACGCC - 3’, used to generate OT in vitro transcription template in PCR 1 |
| 3’ handle reverse | Microsynth | custom order | 5' - GTCAAAGTGCGCCCCGTTATCC - 3', used to generate OT in vitro transcription template in PCR 1 |
| 5' handle forward | Microsynth | custom order | 5' - TCCTTTCTGTGGACGCCGC - 3' , used to generate 5' handle in PCR 2 |
| 5’ handle reverse | Microsynth | custom order | 5’ - CATAAATACCTCTTTACTAATATA TATACCTTCGTAAGCTAGCGT - 3’, used to generate 5' handle in PCR 2 |
| 3’ handle forward | Microsynth | custom order | 5' - ATCCTGCAACCTGCTCTTCGCC AG - 3', used to generate 3' handle in PCR 3 |
| 3’ handle reverse 5’labeled with digoxigenin | Microsynth | custom order | 5' -[Dig]-GTCAAAGTGCGCCCCGTTATCC - 3', used to generate 3' handle in PCR 3 |
| DNA vectors | |||
| pMZ_OT | produced in-house | N/A | further description in "Structural studies of Cardiovirus 2A protein reveal the molecular basis for RNA recognition and translational control" Chris H. Hill, Sawsan Napthine, Lukas Pekarek, Anuja Kibe, Andrew E. Firth, Stephen C. Graham, Neva Caliskan, Ian Brierley bioRxiv 2020.08.11.245035; doi: https://doi.org/10.1101/2020.08.11.245035 |
| Software and Algorithms | |||
| Atom | https://atom.io/packages/ide-python | N/A | |
| Bluelake | Lumicks | N/A | |
| Graphpad | https://www.graphpad.com/ | N/A | |
| InkScape 0.92.3 | https://inkscape.org/ | N/A | |
| Matlab | https://www.mathworks.com/products/matlab.html | N/A | |
| POTATO | https://github.com/lpekarek/POTATO.git | N/A | |
| RNAstructure | https://rna.urmc.rochester.edu/RNAstructure.html | N/A | |
| Spyder | https://www.spyder-ide.org/ | N/A | |
| Other | |||
| Streptavidin Coated Polystyrene Particles, 1.5-1.9 µm, 5 ml, 1.0% w/v | Spherotech | SVP-15-5 | |
| Anti-digoxigenin Coated Polystyrene Particles, 2.0-2.4 µm, 2 ml, 0.1% w/v | Spherotech | DIGP-20-2 | |
| Syringes | VWR | TERUMO SS+03L1 | |
| Devices | |||
| C-trap | Lumicks | N/A | optical tweezers coupled with confocal microscopy |
참고문헌
- Balcerak, A., Trebinska-Stryjewska, A., Konopinski, R., Wakula, M., Grzybowska, E. A. RNA-protein interactions: disorder, moonlighting and junk contribute to eukaryotic complexity. Open Biology. 9 (6), 190096 (2019).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Firth, A. E., Brierley, I. Non-canonical translation in RNA viruses. Journal of General Virology. 93, 1385-1409 (2012).
- Caliskan, N., Peske, F., Rodnina, M. V. Changed in translation: mRNA recoding by −1 programmed ribosomal frameshifting. Trends in Biochemical Sciences. 40 (5), 265-274 (2015).
- Jaafar, Z. A., Kieft, J. S. Viral RNA structure-based strategies to manipulate translation. Nature Reviews Microbiology. 17 (2), 110-123 (2019).
- Eswarappa, S. M., et al. Programmed translational readthrough generates antiangiogenic VEGF-Ax. Cell. 157 (7), 1605-1618 (2014).
- Rodnina, M. V., et al. Translational recoding: canonical translation mechanisms reinterpreted. Nucleic Acids Research. 48 (3), 1056-1067 (2020).
- Li, Y., et al. Transactivation of programmed ribosomal frameshifting by a viral protein. Proceedings of the National Academy of Sciences. 111 (21), 2172 (2014).
- Napthine, S., et al. Protein-directed ribosomal frameshifting temporally regulates gene expression. Nature Communications. 8 (1), 15582 (2017).
- Patel, A., et al. Molecular characterization of the RNA-protein complex directing -2/-1 programmed ribosomal frameshifting during arterivirus replicase expression. Journal of Biological Chemistry. 295 (52), 17904-17921 (2020).
- Napthine, S., Bell, S., Hill, C. H., Brierley, I., Firth, A. E. Characterization of the stimulators of protein-directed ribosomal frameshifting in Theiler's murine encephalomyelitis virus. Nucleic Acids Research. 47 (15), 8207-8223 (2019).
- Marshall, R. A., Aitken, C. E., Dorywalska, M., Puglisi, J. D. Translation at the Single-Molecule Level. Annual Review of Biochemistry. 77 (1), 177-203 (2008).
- Rodnina, M. V. The ribosome in action: Tuning of translational efficiency and protein folding. Protein science : A publication of the Protein Society. 25 (8), 1390-1406 (2016).
- Rodnina, M. V., Fischer, N., Maracci, C., Stark, H. Ribosome dynamics during decoding. Philosophical Transactions of Royal Society of London B Biological Sciences. 372 (1716), (2017).
- Yan, S., Wen, J. D., Bustamante, C., Tinoco, I. Ribosome excursions during mRNA translocation mediate broad branching of frameshift pathways. Cell. 160 (5), 870-881 (2015).
- Liu, T., et al. Direct measurement of the mechanical work during translocation by the ribosome. eLife. 3, 03406 (2014).
- Desai, V. P., et al. Co-temporal force and fluorescence measurements reveal a ribosomal gear shift mechanism of translation regulation by structured mRNAs. Molecular Cell. 75 (5), 1007-1019 (2019).
- Choi, J., O'Loughlin, S., Atkins, J. F., Puglisi, J. D. The energy landscape of -1 ribosomal frameshifting. Science Advances. 6 (1), (2020).
- Prabhakar, A., Puglisi, E. V., Puglisi, J. D. Single-molecule fluorescence applied to translation. Cold Spring Harbor Perspectives in Biology. 11 (1), 032714 (2019).
- Bao, C., et al. mRNA stem-loops can pause the ribosome by hindering A-site tRNA binding. Elife. 9, 55799 (2020).
- Chen, J., Tsai, A., O'Leary, S. E., Petrov, A., Puglisi, J. D. Unraveling the dynamics of ribosome translocation. Current Opinion in Structural Biology. 22 (6), 804-814 (2012).
- Qu, X., et al. The ribosome uses two active mechanisms to unwind messenger RNA during translation. Nature. 475 (7354), 118-121 (2011).
- Zheng, Q., et al. Ultra-stable organic fluorophores for single-molecule research. Chemical Society Reviews. 43 (4), 1044-1056 (2014).
- Blanchard, S. C. Single-molecule observations of ribosome function. Current Opinion in Structural Biology. 19 (1), 103-109 (2009).
- Juette, M. F., et al. The bright future of single-molecule fluorescence imaging. Current Opinion in Chemical Biology. 20, 103-111 (2014).
- McCauley, M. J., Williams, M. C. Mechanisms of DNA binding determined in optical tweezers experiments. Biopolymers. 85 (2), 154-168 (2007).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters. 11 (5), 288-290 (1986).
- Bustamante, C., Smith, S. B., Liphardt, J., Smith, D. Single-molecule studies of DNA mechanics. Current Opinion in Structural Biology. 10 (3), 279-285 (2000).
- Choudhary, D., Mossa, A., Jadhav, M., Cecconi, C. Bio-molecular applications of recent developments in optical tweezers. Biomolecules. 9 (1), 23 (2019).
- Moffitt, J. R., Chemla, Y. R., Smith, S. B., Bustamante, C. Recent advances in optical tweezers. Annual Reviews of Biochemistry. 77, 205-228 (2008).
- Li, P. T. X., Vieregg, J., Tinoco, I. How RNA Unfolds and Refolds. Annual Review of Biochemistry. 77 (1), 77-100 (2008).
- Stephenson, W., Wan, G., Tenenbaum, S. A., Li, P. T. Nanomanipulation of single RNA molecules by optical tweezers. Journal of Visualized Experiments. (90), e51542 (2014).
- Halma, M. T. J., Ritchie, D. B., Cappellano, T. R., Neupane, K., Woodside, M. T. Complex dynamics under tension in a high-efficiency frameshift stimulatory structure. Proceedings of the National Academy of Sciences. 116 (39), 19500 (2019).
- Hansen, T. M., Reihani, S. N. S., Oddershede, L. B., Sørensen, M. A. Correlation between mechanical strength of messenger RNA pseudoknots and ribosomal frameshifting. Proceedings of the National Academy of Sciences of the United States of America. 104 (14), 5830-5835 (2007).
- Zhong, Z., et al. Mechanical unfolding kinetics of the SRV-1 gag-pro mRNA pseudoknot: possible implications for -1 ribosomal frameshifting stimulation. Science Reports. 6, 39549 (2016).
- McCauley, M. J., Rouzina, I., Li, J., Núñez, M. E., Williams, M. C. Significant differences in RNA structure destabilization by HIV-1 GagDp6 and NCp7 proteins. Viruses. 12 (5), 484 (2020).
- de Messieres, M., et al. Single-molecule measurements of the CCR5 mRNA unfolding pathways. Biophysics Journal. 106 (1), 244-252 (2014).
- Yang, L., et al. Single-molecule mechanical folding and unfolding of RNA hairpins: Effects of single A-U to A·C pair substitutions and single proton binding and implications for mRNA structure-induced -1 ribosomal frameshifting. Journal of American Chemical Society. 140 (26), 8172-8184 (2018).
- McCauley, M. J., et al. Targeted binding of nucleocapsid protein transforms the folding landscape of HIV-1 TAR RNA. Proceedings of the National Academy of Sciences of the United States of America. 112 (44), 13555-13560 (2015).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. High-resolution "Fleezers": Dual-trap optical tweezers combined with single-molecule fluorescence detection. Methods in Molecular Biology. 1486, 183-256 (2017).
- Yerramilli, V. S., Kim, K. H. Labeling RNAs in live cells using malachite green aptamer scaffolds as fluorescent probes. ACS Synthetic Biology. 7 (3), 758-766 (2018).
- Gross, P., Farge, G., Peterman, E. J., Wuite, G. J. Combining optical tweezers, single-molecule fluorescence microscopy, and microfluidics for studies of DNA-protein interactions. Methods in Enzymology. 475, 427-453 (2010).
- Zimmer, M. M., et al. The short isoform of the host antiviral protein ZAP acts as an inhibitor of SARS-CoV-2 programmed ribosomal frameshifting. Nature Communications. 12 (1), 7193 (2021).
- Neupane, K., Yu, H., Foster, D. A. N., Wang, F., Woodside, M. T. Single-molecule force spectroscopy of the add adenine riboswitch relates folding to regulatory mechanism. Nucleic acids research. 39 (17), 7677-7687 (2011).
- Ritchie, D. B., Soong, J., Sikkema, W. K., Woodside, M. T. Anti-frameshifting ligand reduces the conformational plasticity of the SARS virus pseudoknot. Journal of the American Chemical Society. 136 (6), 2196-2199 (2014).
- Janissen, R., et al. Invincible DNA tethers: covalent DNA anchoring for enhanced temporal and force stability in magnetic tweezers experiments. Nucleic Acids Research. 42 (18), 137 (2014).
- Smith, S. B., Cui, Y., Bustamante, C. Overstretching B-DNA: The elastic response of individual double-stranded and single-stranded DNA molecules. Science. 271 (5250), 795 (1996).
- Puljung, M. C., Zagotta, W. N. Labeling of specific cysteines in proteins using reversible metal protection. Biophysical Journal. 100 (10), 2513-2521 (2011).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Hill, C. H., et al. Structural and molecular basis for Cardiovirus 2A protein as a viral gene expression switch. Nature Communications. 12 (1), 7166 (2021).
- Butterworth, S. On the theory of filter amplifiers. Experimental Wireless and the Wireless Engineer. 7, 536-541 (1930).
- Wang, M. D., Yin, H., Landick, R., Gelles, J., Block, S. M. Stretching DNA with optical tweezers. Biophysics Journal. 72 (3), 1335-1346 (1997).
- Mukhortava, A., et al. Structural heterogeneity of attC integron recombination sites revealed by optical tweezers. Nucleic Acids Research. 47 (4), 1861-1870 (2019).
- Buck, S., Pekarek, L., Caliskan, N. POTATO: An automated pipeline for batch analysis of optical tweezers data. bioRxiv. , (2021).
- Zhang, Y., Jiao, J., Rebane, A. A. Hidden Markov modeling with detailed balance and its application to single protein folding. Biophysical Journal. 111 (10), 2110-2124 (2016).
- Sgouralis, I., Pressé, S. An introduction to infinite HMMs for single-molecule data analysis. Biophysics Journal. 112 (10), 2021-2029 (2017).
- Müllner, F. E., Syed, S., Selvin, P. R., Sigworth, F. J. Improved hidden Markov models for molecular motors, part 1: basic theory. Biophysical Journal. 99 (11), 3684-3695 (2010).
- Elms, P. J., Chodera, J. D., Bustamante, C. J., Marqusee, S. Limitations of constant-force-feedback experiments. Biophysical Journal. 103 (7), 1490-1499 (2012).
- Re, A., Joshi, T., Kulberkyte, E., Morris, Q., Workman, C. T. RNA-protein interactions: an overview. Methods Molecular Biology. 1097, 491-521 (2014).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature reviews. Molecular Cell Biology. 16 (9), 533-544 (2015).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Sunbul, M., Jäschke, A. SRB-2: a promiscuous rainbow aptamer for live-cell RNA imaging. Nucleic Acids Research. 46 (18), 110 (2018).
- Sanchez de Groot, N., et al. RNA structure drives interaction with proteins. Nature Communications. 10 (1), 3246 (2019).
- Zeffman, A., Hassard, S., Varani, G., Lever, A. The major HIV-1 packaging signal is an extended bulged stem loop whose structure is altered on interaction with the Gag polyprotein. Journal of Molecular Biology. 297 (4), 877-893 (2000).
- Mangeol, P., et al. Probing ribosomal protein-RNA interactions with an external force. Proceedings of the National Academy of Sciences. 108 (45), 18272 (2011).
- Luo, X., et al. Molecular mechanism of RNA recognition by Zinc-Finger antiviral protein. Cell Reports. 30 (1), 46-52 (2020).
- Qu, X., Lancaster, L., Noller, H. F., Bustamante, C., Tinoco, I. Ribosomal protein S1 unwinds double-stranded RNA in multiple steps. Proceedings of the National Academy of Science U. S. A. 109 (36), 14458-14463 (2012).
- Chandra, V., Hannan, Z., Xu, H., Mandal, M. Single-molecule analysis reveals multi-state folding of a guanine riboswitch. Nature Chemical Biology. 13 (2), 194-201 (2017).
- Savinov, A., Perez, C. F., Block, S. M. Single-molecule studies of riboswitch folding. Biochimica et Biophysica Acta. 1839 (10), 1030-1045 (2014).
- Kelly, J. A., et al. Structural and functional conservation of the programmed ribosomal frameshift signal of SARS coronavirus 2 (SARS-CoV-2). Journal of Biological Chemistry. 295 (31), 10741-10748 (2020).
- Neupane, K., et al. Structural dynamics of single SARS-CoV-2 pseudoknot molecules reveal topologically distinct conformers. Nature Communications. 12 (1), 4749 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zheng, Q., Jockusch, S., Zhou, Z., Blanchard, S. C. The contribution of reactive oxygen species to the photobleaching of organic fluorophores. Photochemistry and Photobiology. 90 (2), 448-454 (2014).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Rill, N., Mukhortava, A., Lorenz, S., Tessmer, I. Alkyltransferase-like protein clusters scan DNA rapidly over long distances and recruit NER to alkyl-DNA lesions. Proceedings of the National Academy of Science U. S. A. 117 (17), 9318-9328 (2020).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Wen, J. -. D., et al. Force unfolding kinetics of RNA using optical tweezers. I. Effects of experimental variables on measured results. Biophysical journal. 92 (9), 2996-3009 (2007).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유