È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Pinzette ottiche per studiare le interazioni RNA-proteina nella regolazione della traduzione
In questo articolo
Riepilogo
Questo protocollo presenta un flusso di lavoro sperimentale completo per lo studio delle interazioni RNA-proteina utilizzando pinzette ottiche. Sono delineate diverse possibili configurazioni sperimentali, tra cui la combinazione di pinzette ottiche con microscopia confocale.
Abstract
L'RNA adotta diverse pieghe strutturali, che sono essenziali per le sue funzioni e quindi possono avere un impatto su diversi processi nella cellula. Inoltre, la struttura e la funzione di un RNA possono essere modulate da vari fattori trans-agenti, come proteine, metaboliti o altri RNA. Le molecole di RNA frameshifting, ad esempio, sono RNA regolatori situati in regioni codificanti, che dirigono la traduzione dei ribosomi in un frame di lettura aperto alternativo, e quindi agiscono come interruttori genici. Possono anche adottare pieghe diverse dopo il legame a proteine o altri fattori trans. Per analizzare il ruolo delle proteine leganti l'RNA nella traduzione e il modo in cui modulano la struttura e la stabilità dell'RNA, è fondamentale studiare contemporaneamente l'interazione e le caratteristiche meccaniche di questi complessi RNA-proteina. Questo lavoro illustra come impiegare pinzette ottiche accoppiate a singola molecola-fluorescenza per esplorare il panorama conformazionale e termodinamico dei complessi RNA-proteina ad alta risoluzione. Ad esempio, viene elaborata l'interazione dell'elemento di frameshifting ribosomiale programmato SARS-CoV-2 con l'isoforma breve del fattore trans-agente della proteina antivirale zinco-dito. Inoltre, i ribosomi marcati con fluorescenza sono stati monitorati utilizzando l'unità confocale, che alla fine avrebbe permesso lo studio dell'allungamento della traduzione. Il test OT accoppiato a fluorescenza può essere ampiamente applicato per esplorare diversi complessi RNA-proteina o fattori trans-agenti che regolano la traduzione e potrebbe facilitare gli studi sulla regolazione genica basata sull'RNA.
Introduzione
Il trasferimento di informazioni genetiche dal DNA alle proteine attraverso gli mRNA è un processo biochimico complesso, che è regolato con precisione a tutti i livelli attraverso interazioni macromolecolari all'interno delle cellule. Per la regolazione traslazionale, le interazioni RNA-proteina conferiscono un ruolo critico per reagire rapidamente a vari stimoli e segnali1,2. Alcune interazioni RNA-proteina influenzano la stabilità dell'mRNA e quindi alterano il tempo in cui un RNA è attivo per via traslazionale. Altre interazioni RNA-proteina sono associate a meccanismi di ricodifica come il readthrough stop-codone, il bypassing o il frameshifting ribosomiale programmato (PRF)3,4,5,6,7. Recentemente, un certo numero di proteine leganti l'RNA (RBP) hanno dimostrato di interagire con gli elementi dell'mRNA stimolatorio e il meccanismo di traduzione per dettare quando e quanta ricodifica si verificherà nella cellula7,8,9,10,11. Pertanto, per analizzare il ruolo delle proteine leganti l'RNA nella traduzione e il modo in cui modulano la struttura e la stabilità dell'RNA, è fondamentale studiare in dettaglio i principi di interazione e le proprietà meccaniche di questi complessi RNA-proteina.
Decenni di lavoro hanno gettato le basi per studiare il processo di traduzione multi-fase e multi-componente, che si basa su una comunicazione intricata tra l'RNA e i componenti proteici del meccanismo di traduzione per raggiungere velocità e precisione12,13,14. Un passo successivo cruciale nella comprensione di eventi normativi complessi è determinare le forze, i tempi e i determinanti strutturali durante la traduzione ad alta precisione12,15,16,17. Lo studio della dinamica conformazionale dell'RNA e in particolare di come i fattori ausiliari trans-agenti agiscono sulla struttura dell'RNA durante la traslazione sono stati ulteriormente illuminati dall'emergere di strumenti a singola molecola, tra cui pinzette ottiche o guide d'onda in modalità zero16,17,18,19,20,21,22,23,24 ,25,26.
Le pinzette ottiche (OT) rappresentano una tecnica a singola molecola altamente precisa, che è stata applicata per studiare molti tipi di processi dinamici dipendenti dall'RNA, tra cui la trascrizione e la traduzione26,27,28,29,30,31,32. L'uso di pinzette ottiche ha permesso di sondare in dettaglio le interazioni molecolari, le strutture degli acidi nucleici e le proprietà termodinamiche, la cinetica e l'energetica di questi processi16,17,22,33,34,35,36,37,38,39 . Il test delle pinzette ottiche si basa sull'intrappolamento di oggetti microscopici con un raggio laser focalizzato. In un tipico esperimento OT, la molecola di interesse è legata tra due perle trasparenti (di solito polistirene) (Figura 1A)27. Queste perle vengono poi catturate da trappole ottiche, che si comportano come molle. Pertanto, la forza applicata sulla molecola può essere calcolata in base allo spostamento del tallone dal centro del raggio laser focalizzato (centro trappola). Recentemente, le pinzette ottiche sono state combinate con la microscopia confocale (Figura 1B), consentendo misurazioni di fluorescenza o trasferimento di energia di risonanza di Förster (FRET) 40,41,42. Questo apre un campo completamente nuovo di possibili esperimenti che consentono la misurazione simultanea e, quindi, una correlazione precisa della spettroscopia di forza e dei dati di fluorescenza.
Qui, dimostriamo esperimenti che utilizzano le pinzette ottiche combinate con la microscopia confocale per studiare le interazioni proteina-RNA che regolano il frameshifting traslazionale. Tra l'obiettivo e il condensatore, una cella di flusso con cinque canali consente l'applicazione continua del campione con flusso laminare. Attraverso i canali microfluidici, vari componenti possono essere iniettati direttamente, il che riduce il tempo di hands-on e consente un consumo di campioni molto ridotto durante l'esperimento.
In primo luogo, viene proposta una linea guida di base per assistere la progettazione di esperimenti OT e vengono discussi i vantaggi e le insidie di varie configurazioni. Successivamente, viene descritta la preparazione di campioni e flussi di lavoro sperimentali e viene fornito un protocollo per l'analisi dei dati. Per rappresentare un esempio, delineiamo i risultati ottenuti da esperimenti di stretching dell'RNA per studiare l'elemento RNA frameshifting SARS-CoV-2 (Figura 2A) con il fattore trans-agente l'isoforma corta della proteina antivirale zinco-dito (ZAP), che altera la traduzione dell'RNA virale da un frame di lettura alternativo43. Inoltre, è dimostrato che i ribosomi marcati con fluorescenza possono essere impiegati in questo saggio confocale OT, che sarebbe utile per monitorare la processività e la velocità del macchinario di traduzione. Il metodo qui presentato può essere utilizzato per testare rapidamente l'effetto di diversi tamponi, ligandi o altri componenti cellulari per studiare vari aspetti della traduzione. Infine, vengono discusse le insidie sperimentali comuni e come risolverle. Di seguito, vengono delineati alcuni punti cruciali nella progettazione sperimentale.
Progettazione di costruzioni
In linea di principio, ci sono due approcci comuni per creare un costrutto di RNA compatibile ot. Il primo approccio impiega una lunga molecola di RNA che viene ibridata con maniglie di DNA complementari, producendo così un costrutto costituito da due regioni ibride RNA / DNA che fiancheggiano una sequenza di RNA a singolo filamento nel mezzo (Figura 2B). Questo approccio è impiegato nella maggior parte degli esperimenti ot RNA33,44,45.
Il secondo approccio sfrutta le maniglie dsDNA con sporgenze corte (circa 20 nt)15,17. Queste sporgenze vengono poi ibridate con la molecola di RNA. Sebbene più complicato nella progettazione, l'uso di maniglie dsDNA supera alcune delle limitazioni del sistema ibrido DNA / RNA. In linea di principio, è possibile implementare anche maniglie molto lunghe (>10kb), il che è più conveniente per le misurazioni confocali. Inoltre, la molecola di RNA può essere legata alle maniglie del DNA per aumentare la stabilità del legame.
Strategia di etichettatura finale
Il costrutto deve essere legato a perline tramite una forte interazione molecolare. Mentre ci sono approcci disponibili per il legame covalente delle maniglie alle perline46, interazioni forti ma non covalenti come streptavidina-biotina e digoxigenina-anticorpo sono comunemente usate negli esperimenti OT15,33,35,45. Nel protocollo descritto, il costrutto è etichettato con biotina o digossigenina e le perline sono rivestite rispettivamente con streptavidina o anticorpi contro la digossigenina (Figura 1A). Questo approccio sarebbe adatto per applicare forze fino a circa 60 pN (per tether)47. Inoltre, l'uso di diverse strategie di etichettatura 5' e 3' consente di determinare l'orientamento del cavo formato tra le perline17.
Etichettatura delle proteine per misure di fluorescenza
Per l'imaging confocale, ci sono diversi approcci comunemente usati per l'etichettatura a fluorescenza. Ad esempio, i fluorofori possono essere attaccati covalentemente a residui di amminoacidi che si trovano nativamente nelle proteine o introdotti dalla mutagenesi diretta dal sito attraverso un gruppo organico reattivo. I coloranti tiolici o ammino-reattivi possono essere utilizzati per l'etichettatura dei residui di cisteina e lisina, rispettivamente. Esistono diversi metodi di protezione reversibili per aumentare la specificità dell'etichettatura48,49, tuttavia le proteine native sarebbero tipicamente etichettate a più residui. Sebbene le piccole dimensioni del fluoroforo possano conferire un vantaggio, l'etichettatura non specifica potrebbe interferire con l'attività proteica e quindi l'intensità del segnale potrebbe variare49. Inoltre, a seconda dell'efficienza di etichettatura, l'intensità del segnale può differire tra i diversi esperimenti. Pertanto, un controllo dell'attività deve essere eseguito prima dell'esperimento.
Nel caso in cui la proteina di interesse contenga un tag N- o C-terminale, come un His-tag o strep-tag, l'etichettatura specifica di questi tag rappresenta un altro approccio popolare. Inoltre, l'etichettatura mirata ai tag riduce la possibilità che il fluoroforo interferisca con l'attività proteica e può migliorare la solubilità49. Tuttavia, l'etichettatura specifica del tag di solito produce proteine marcate con monofluoroforo, che potrebbero essere difficili da rilevare. Un altro modo di etichettatura specifica può essere realizzato impiegando anticorpi.
Configurazione della microfluidica
La combinazione di OT con un sistema di microfluidica consente una rapida transizione tra diverse condizioni sperimentali. Inoltre, i sistemi di corrente sfruttano il mantenimento del flusso laminare all'interno della cella di flusso, che preclude la miscelazione di liquidi da altri canali nella direzione perpendicolare rispetto alla direzione del flusso. Pertanto, il flusso laminare è particolarmente vantaggioso per la progettazione sperimentale. Attualmente, le celle di flusso con un massimo di 5 canali sono comunemente utilizzate (Figura 3).
Protocollo
1. Preparazione del campione
- Clonare la sequenza di interesse nel vettore contenente i frammenti di DNA Lambda, che fungono da sequenze di maniglia (Figura 2)43,50.
- Per prima cosa generare un modello di DNA per la successiva trascrizione in vitro tramite PCR (Figura 2B; reazione 1). In questa fase della PCR, il promotore T7 viene aggiunto all'estremità 5' della molecola di DNA del senso32,33,43,50. Impostare la reazione PCR in base alla Tabella 1. Eseguire la PCR in aliquote da 50 μL con cicli appropriati nel termociclatore.
- Preparare le maniglie mediante due reazioni PCR separate (Tabella 1, Figura 2B; reazione 2 e 3). Innanzitutto, genera l'handle da 5' tramite PCR. Quindi, generare la maniglia da 3' e contemporaneamente etichettarla con digoxigenina utilizzando un primer marcato con digossigenina da 5'32,33,43,50.
- Dopo la PCR, purificare il DNA usando colonne di spin di silice.
- Effettuare la reazione di trascrizione in vitro utilizzando T7 RNA polimerasi (Tabella 2)32,33,43,50. Incubare la reazione a 37 °C per 2-4 ore a seconda della lunghezza dell'RNA. Quindi, aggiungere DNasi I alla reazione e incubare a 37 °C per 30 minuti per digerire il modello di DNA. Purificare l'RNA usando colonne di spin di silice.
- Durante la reazione di marcatura del manico da 5' (Tabella 3), aggiungere biotina-16-dUTP all'estremità 3' del manico con T4 DNA polimerasi38,50. Eseguire la reazione a temperatura ambiente per 1-2 ore. Successivamente, purificare il DNA usando colonne di spin di silice.
NOTA: poiché l'impugnatura da 5' deve essere etichettata all'estremità 3' (Figura 2B), l'etichettatura non può essere eseguita durante la PCR. - Mescolare i componenti sopra menzionati - 5' maniglia (3' etichettata con biotina), 3' maniglia (5' etichettata con digoxigenina) e RNA - in un rapporto molare 1:1:1 in tampone ricottura (80% formammide, 400 mM NaCl, 40 mM HEPES, pH 7,5, 0,5 mM EDTA, pH 8), per ottenere l'ibrido RNA/DNA desiderato (Tabella 4). Riscaldare la miscela di ricottura fino a 85 °C per 10 minuti e poi raffreddare lentamente fino a 4 °C.
- Mescolare il campione ricotto con 1/10 di volume di 3 M di acetato di sodio (pH 5), 3 volumi di etanolo ghiacciato e incubare a -80 °C per almeno 1 ora o a -20 °C durante la notte.
- Centrifugare i campioni a 15.000 × g per 30 minuti a 4 °C. Scartare il surnatante e asciugare il pellet (di solito non visibile) sotto vuoto.
- Infine, risospendere, il pellet in 50 μL di acqua priva di RNasi e fare aliquote. Conservare le aliquote a -80 °C fino all'uso. Per la conservazione a breve termine, i campioni possono anche essere conservati a -20 °C.
2. Configurazione dello strumento
NOTA: Il seguente protocollo è ottimizzato per lo strumento commerciale di pinzette ottiche C-Trap della società LUMICKS. Pertanto, potrebbero essere necessarie regolazioni dei passaggi presentati durante l'utilizzo di altri strumenti ottici a pinzetta. Se non utilizzato, il sistema di microfluidica della macchina viene mantenuto in candeggina (soluzione di ipoclorito di sodio) e deve essere lavato prima dell'uso.
- Scartare la candeggina e riempire le siringhe con 1 mL di acqua priva di RNasi.
- Aggiungere 50 μL di tiosolfato di sodio 0,5 M ad almeno 1 mL di acqua priva di RNasi e lavare accuratamente il sistema (1 bar, almeno 0,5 mL) per eliminare la candeggina rimanente nel sistema.
- Eliminare la soluzione di tiosolfato di sodio dalle siringhe. Sostituire le siringhe con quelle fresche e lavare il sistema con almeno 0,5 ml di acqua priva di RNasi.
NOTA: Fare attenzione che il sistema di microfluidica non si esaurisca mai per evitare bolle d'aria nel sistema. - Mettere 2 gocce di olio per immersione (indice di rifrazione di 1,33) o circa 70 μL di acqua sopra l'obiettivo.
- Posizionate la cella di flusso all'interno del telaio di tenuta nella sua posizione.
- Mettere 2 gocce di olio ad immersione (indice di rifrazione di 1,51) sopra la cella di flusso.
- Accendere il dispositivo laser nella pinzetta. Una volta in esecuzione, attivare il laser di trapping nell'interfaccia software al 100%.
- Utilizzando telecamere diagnostiche (Z-finder), regolare l'asse Z al centro della camera tra la seconda e la terza riflessione (interfacce) dove gli anelli di rifrazione sono i più grandi, ruotando la micro vite.
NOTA: Ogni volta che l'obiettivo viene avvicinato alla camera di misura e il piano focale dell'obiettivo attraversa l'interfaccia tra due fasi, è possibile riconoscere un riflesso nella modalità Z-finder. Sono possibili 4 interfacce: (i) olio acqua/immersione e vetro inferiore (ii) vetro inferiore e tampone all'interno della camera (iii) tampone all'interno della camera e vetro superiore (iv) vetro superiore e olio ad immersione per condensatore. - Regolare la posizione del condensatore (impostare il laser di intrappolamento a circa il 50%) in modo che il condensatore tocchi l'olio di immersione sulla parte superiore della camera di misura.
- Regolare la messa a fuoco muovendosi lentamente verso il basso / verso l'alto con il condensatore, in modo che circa 10 bande di luce vengano visualizzate in modalità luna (telecamere diagnostiche).
3. Misurazione del campione
- Incubare perline rivestite di anti-digoxigenina (AD) con i costrutti del campione (3 μL di sospensione di perline AD allo 0,1% (p/v) + 4 μL di campione) e con 1 μL di inibitori della RNasi e 8 μL del tampone di dosaggio (300 mM KCl, 5 mM MgCl2, 20 mM HEPES, pH 7,6, 0,05% Tween 20, 5 mM DTT) a RT per 10-20 min. Dopo l'incubazione, diluire il campione in 500 μL di tampone di dosaggio.
NOTA: Si consiglia di aggiungere spazzini di ossigeno, in particolare durante le misurazioni di fluorescenza al tampone al fine di prevenire danni ossidativi. Qui è stato utilizzato il sistema di scavenger di ossigeno contenente glucosio (8,3 mg / mL), glucosio ossidasi (40 U / mL) e catalasi (185 U / mL). - Miscelare 0,8 μL di perle rivestite di streptavidina (SA) all'1% (p/v) con 1 mL di tampone di dosaggio.
- Scartare l'acqua dalle siringhe e riempire le siringhe con le rispettive sospensioni/soluzioni. Lavare per almeno 2 minuti a circa 1 bar, quindi iniziare a catturare perline.
NOTA: a seconda dell'impostazione sperimentale, possono essere utilizzate diverse disposizioni di canale (Figura 3). Tipicamente, un canale di flusso è riempito con perline anti-digoxigenina che trasportano la molecola di RNA. Un secondo canale è riempito con le perle rivestite di streptavidin. Il canale buffer viene utilizzato per formare i cavi. Un quarto canale può essere impiegato per caricare la proteina legante l'RNA (Figura 3C), o in alternativa RBP può essere aggiunto direttamente nel canale tampone (Figura 3B). - Per catturare le perline, sposta le trappole ottiche l'una dall'altra. Per prima cosa spostati sul canale AD e cattura un tallone AD nella trappola 1. Quindi, sposta il palco sul canale SA e cattura una singola perla SA con la trappola 2.
NOTA: cercare di rimanere all'interfaccia dei canali buffer e bead per evitare di perdere il tallone già catturato o per evitare di catturare più perline dalla stessa trappola. - Una volta catturate le perline della giusta dimensione, spostarsi sul canale buffer e arrestare il flusso laminare. Quindi, eseguire la calibrazione della forza per verificare la rigidità della trappola. I rispettivi valori di rigidità non devono differire nell'asse x/y di oltre il 10-15%.
NOTA: Regolare la potenza del laser o la divisione laser tra le trappole in base alle dimensioni del tallone. La calibrazione della forza non deve essere eseguita per ogni coppia di perline purché i modelli di perline corrispondano (punteggio di somiglianza > 0,9). Tuttavia, dovrebbe essere eseguito regolarmente, o almeno ogni volta che le condizioni del test vengono modificate. - Inizia a pescare un laccio spostando le perline l'una vicino all'altra, aspettando alcuni secondi, quindi allontanandole di nuovo, ripeti fino a quando non si forma un cavo. Una formazione di cavi si traduce in un aumento della forza misurata dopo aver tirato le due perle lontano l'una dall'altra.
NOTA: per evitare la formazione di più cavi, le perline non devono essere spostate troppo vicine. Dopo aver catturato un cavo tra le due perline, la qualità del cavo può essere controllata trovando l'altopiano sovraccarico. Il plateau dovrebbe essere compreso tra 50 e 60 pN per un singolo cavo. - Dopo aver ottenuto un cavo, avviare la misurazione. A seconda del fenomeno studiato, è necessario scegliere diverse configurazioni di misurazione (Figura 1B-D).
NOTA: Di solito all'inizio dell'esperimento, viene condotto un esperimento di rampa di forza per verificare la qualità del cavo e sondare il comportamento. Successivamente, si possono anche iniziare gli esperimenti di forza costante o posizione costante per studiare ulteriormente le transizioni di stato. Una volta che è stato eseguito un numero sufficiente di misurazioni su un campione di RNA per determinarne il comportamento, è possibile aggiungere fattori etichettati al sistema per eseguire misurazioni confocali. - Per eseguire misurazioni di fluorescenza, accendere i laser confocali e l'unità contatore di fotoni nello strumento delle pinzette ottiche.
- Accendere il laser di eccitazione della lunghezza d'onda desiderata nell'interfaccia software e impostare la potenza del laser al 5% o superiore, a seconda del fluoroforo.
NOTA: mentre non si misura, abbassare l'impostazione di potenza del laser di eccitazione allo 0% per evitare un eccessivo fotodanneggiamento al campione. - Avviare l'imaging del campione utilizzando le funzioni immagine del software.
NOTA: Per ottenere immagini ben focalizzate, il piano focale del microscopio confocale e le trappole ottiche devono essere allineati. A tale scopo, è possibile utilizzare l'autofluorescenza delle perle di polistirene nel canale laser blu. Il piano focale delle trappole ottiche viene spostato verso l'alto o verso il basso nell'asse z fino a quando l'immagine delle perline raggiunge il suo diametro più alto. In questa posizione, è possibile misurare il segnale di fluorescenza dalla molecola legata tra le perline. - Per utilizzare la funzione kymograph, specificare la posizione x-y dell'asse del kymograph in modo che consenta il rilevamento del cavo tra le perline.
- Durante la misurazione, la composizione del buffer può essere facilmente modificata spostando le perline su canali diversi o modificando il buffer fornito nel sistema di microfluidica.
4. Analisi dei dati
- Pre-elaborazione dei dati grezzi
- Utilizzando un semplice script, eseguire il downsampling dei dati grezzi in modo sufficiente a (i) consentire un'elaborazione dei dati successiva più rapida, ma (ii) contenere ancora tutte le informazioni critiche (Figura 4A). Di solito, 100-5000 Hz è adatto a questo scopo.
NOTA: La frequenza di raccolta dei dati negli esperimenti di pinzette ottiche è spesso superiore a quella necessaria per l'analisi - negli esperimenti presentati, la frequenza di raccolta dei dati è impostata su 78 125 Hz per impostazione predefinita. Poiché lo spazio di archiviazione è limitato, è conveniente e richiede un risparmio di tempo ridurre la frequenza di campionamento dei dati. Qui, i dati grezzi sono stati decampionati di un fattore 30. - Quindi, utilizzare un filtro del segnale per ridurre il rumore di misurazione ad alta frequenza dal segnale (Figura 4A). Regolare di conseguenza il grado del filtro e i parametri di frequenza di taglio per ottimizzare l'output dei dati di diversi esperimenti (Figura 5).
NOTA: Tra i filtri di segnale, il filtro Butterworth51 è uno dei più utilizzati. Uno script python scritto su misura che consente la pre-elaborazione dei dati grezzi è fornito nei dati supplementari. I parametri di downsampling e filtraggio del segnale (frequenza di cut-off, grado di filtro) devono essere ottimizzati per diversi esperimenti.
- Utilizzando un semplice script, eseguire il downsampling dei dati grezzi in modo sufficiente a (i) consentire un'elaborazione dei dati successiva più rapida, ma (ii) contenere ancora tutte le informazioni critiche (Figura 4A). Di solito, 100-5000 Hz è adatto a questo scopo.
- Per l'analisi dei dati della rampa di forza, attenersi alla seguente procedura.
- Contrassegnare i passaggi manualmente trovando i punti corrispondenti sul plottaggio della traiettoria di forza o utilizzando script scritti in modo personalizzato. I passi di apertura sono caratterizzati da un improvviso calo di forza combinato con un aumento della distanza nella curva forza-distanza (FD).
- Una volta che gli eventi di dispiegamento sono stati contrassegnati, adattare diverse regioni della curva FD utilizzando modelli appropriati (Figura 4D).
NOTA: per la regione prima della prima fase di dispiegamento, il cavo può essere considerato "a doppio filamento" ed è comunemente adatto utilizzando un modello a catena simile a worm (WLC) estensibile 47,52,53. Le parti dopo il primo evento di dispiegamento sono considerate una combinazione di nucleotidi a doppio filamento (maniglie) e nucleotidi a singolo filamento (molecola di RNA dispiegata). Pertanto, l'adattamento dei dati è più complesso - di solito viene utilizzata una combinazione di 2 modelli WLC o WLC e modelli A catena libera (FJC)36,39,52. Il modello WLC estensibile ha due parametri di adattamento principali: la lunghezza del contorno (LC) e la lunghezza di persistenza (LP). La lunghezza del contorno corrisponde alla lunghezza della molecola completamente allungata e la lunghezza di persistenza definisce le proprietà di flessione della molecola di interesse. Il modello può essere descritto con la seguente equazione (1). WLC può essere utilizzato per modellare il comportamento sia delle regioni piegate che di quelle spiegate, sebbene per ciascuna di queste debba essere utilizzato un modello separato con parametri diversi.
(1)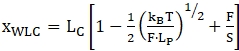
dove x è estensione, LC è lunghezza del contorno, F è forza, LP è lunghezza di persistenza, kB è costante di Boltzmann, T è temperatura termodinamica e S è modulo di allungamento.
Il secondo modello chiamato Catena a giunti liberi (FJC) è comunemente usato per descrivere il comportamento delle regioni a singolo filamento dispiegate. Utilizza parametri simili dei polimeri ma tratta ogni unità della "catena" come un'asta rigida, qui corrispondente ai nucleotidi della regione a singolo filamento dispiegata. La seguente equazione (2) descrive questo modello:
(2)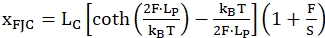
NOTA: Il nostro laboratorio ha recentemente sviluppato un algoritmo che consente l'elaborazione in batch dei dati grezzi della rampa di forza chiamato "Practical Optical Tweezers Analysis TOol (POTATO)54". L'algoritmo esegue il downsampling e filtra i dati, quindi identifica i possibili passaggi di dispiegamento e infine esegue il fitting dei dati. Il POTATO è costruito in un'interfaccia utente grafica (GUI) user-friendly (https://github.com/REMI-HIRI/POTATO).
- Elaborare i dati a forza costante come segue:
NOTA: le seguenti istruzioni possono essere applicate in modo analogico sui dati di posizione costante.- Per i dati a forza costante, tracciare la distanza nel tempo (Figura 5). Un istogramma che mostra la frequenza (conta) di diverse conformazioni sul cambiamento relativo di posizione è un modo utile per caratterizzare vari stati dominanti e minori (Figura 7).
- Adattare l'istogramma utilizzando funzioni gaussiane (multiple) per stimare la percentuale complessiva di singoli conformisti a una data forza (Figura 7C). L'adattamento gaussiano, la posizione media e la deviazione standard delineano la relazione correlata alla forza tra le diverse popolazioni.
NOTA: nei dati supplementari viene fornito uno script python scritto su misura che consente la pre-elaborazione e il montaggio gaussiano bimodale di base dei dati a forza costante. I parametri (frequenza di cut-off, grado di filtro, mezzi attesi, valori di deviazione standard e ampiezze) devono essere ottimizzati per diversi esperimenti. - Successivamente, utilizzare il modello di Markov nascosto per analizzare ulteriormente gli stati, che possono scoprire ulteriori intermedi di piegatura (conformisti)55. Per ulteriori informazioni sulla forza costante e sul modello di Markov nascosto, si può fare riferimento a55,56,57,58.
Risultati
In questa sezione, l'attenzione si concentra principalmente sulle misurazioni delle interazioni RNA-proteina/ligando da parte delle pinzette ottiche a fluorescenza. Per una descrizione degli esperimenti generali sulle pinzette ottiche a RNA e dei corrispondenti risultati rappresentativi, vedere32. Per una discussione più dettagliata delle interazioni RNA/DNA-proteina, vedere anche1,2,26,59,60....
Discussione
Qui, dimostriamo l'uso di pinzette ottiche accoppiate a fluorescenza per studiare le interazioni e il comportamento dinamico delle molecole di RNA con vari ligandi. Di seguito, vengono discussi i passaggi critici e le limitazioni della presente tecnica.
Passaggi critici nel protocollo
Come per molti altri metodi, la qualità del campione è fondamentale per ottenere dati affidabili. Pertanto, per ottenere campioni della massima qualità possibile, vale la pena dedicare del ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Anuja Kibe e Jun. Prof. Redmond Smyth per aver esaminato criticamente il manoscritto. Ringraziamo Tatyana Koch per l'assistenza tecnica esperta. Ringraziamo Kristyna Pekarkova per l'aiuto nella registrazione di video sperimentali. Il lavoro nel nostro laboratorio è supportato dall'Associazione Helmholtz e dal finanziamento della sovvenzione del Consiglio europeo della ricerca (CER) Nr. 948636 (a NC).
Materiali
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| E. coli HB101 | lab collection | N/A | cloning of the vectors |
| Chemicals and enzymes | |||
| Sodium chloride | Sigma-Aldrich | 31424 | Buffers |
| Biotin-16-dUTP | Roche | 11093070910 | Biotinylation |
| BSA | Sigma-Aldrich | A4737 | Buffers |
| Catalase | Lumicks | N/A | Oxygen scavanger system |
| Dithiothreitol (DTT) | Melford Labs | D11000 | Buffers |
| DNAse I from bovine pancreas | Sigma-Aldrich | D4527 | in vitro transcription |
| dNTPs | Th.Geyer | 11786181 | PCR |
| EDTA | Sigma-Aldrich | E9884 | Buffers |
| Formamide | Sigma-Aldrich | 11814320001 | Buffers |
| Glucose | Sigma-Aldrich | G8270-1KG | Oxygen scavanger system |
| Glucose-oxidase | Lumicks | N/A | Oxygen scavanger system |
| HEPES | Carl Roth | HN78.3 | Buffers |
| Magnesium chloride | Carl Roth | 2189.1 | Buffers |
| Phusion DNA polymerase | NEB | M0530L | Gibson assembly, cloning |
| Potassium chloride | Merck | 529552-1KG | Buffers |
| PrimeSTAR GXL DNA Polymerase | Takara Bio Clontech | R050A | PCR |
| Pyrophosphotase, thermostabile, inorganic | NEB | M0296L | in vitro transcription |
| RNase Inhibitor | Molox | 1000379515 | Buffers |
| rNTPS | life technologies | R0481 | in vitro transcription |
| Sodium thiosulophate | Sigma-Aldrich | S6672-500G | Bleach deactivation |
| Sytox Green | Lumicks | N/A | confocal measurements |
| T4 DNA Polymerase | NEB | M0203S | Biotinylation |
| T5 exonuclease | NEB | M0363S | Gibson assembly, cloning |
| T7 RNA polymerase | Produced in-house | N/A | in vitro transcription |
| Taq DNA polymerase | NEB | M0267S | PCR |
| Taq ligase | Biozym | L6060L | Gibson assembly, cloning |
| TWEEN 20 BioXtra | Sigma-Aldrich | P7949 | Buffers |
| Kits | |||
| Monolith Protein Labeling Kit RED-NHS 2nd Generation (Amine Reactive) | Nanotemper | MO-L011 | Used for ribosome labeling |
| Purefrex 2.0 | GeneFrontier | PF201-0.25-EX | Ribosomes used for the labeling |
| Oligonucleotides | |||
| 5' handle T7 forward | Microsynth | custom order | 5’ - CTTAATACGACTCACTATAGGTC CTTTCTGTGGACGCC - 3’, used to generate OT in vitro transcription template in PCR 1 |
| 3’ handle reverse | Microsynth | custom order | 5' - GTCAAAGTGCGCCCCGTTATCC - 3', used to generate OT in vitro transcription template in PCR 1 |
| 5' handle forward | Microsynth | custom order | 5' - TCCTTTCTGTGGACGCCGC - 3' , used to generate 5' handle in PCR 2 |
| 5’ handle reverse | Microsynth | custom order | 5’ - CATAAATACCTCTTTACTAATATA TATACCTTCGTAAGCTAGCGT - 3’, used to generate 5' handle in PCR 2 |
| 3’ handle forward | Microsynth | custom order | 5' - ATCCTGCAACCTGCTCTTCGCC AG - 3', used to generate 3' handle in PCR 3 |
| 3’ handle reverse 5’labeled with digoxigenin | Microsynth | custom order | 5' -[Dig]-GTCAAAGTGCGCCCCGTTATCC - 3', used to generate 3' handle in PCR 3 |
| DNA vectors | |||
| pMZ_OT | produced in-house | N/A | further description in "Structural studies of Cardiovirus 2A protein reveal the molecular basis for RNA recognition and translational control" Chris H. Hill, Sawsan Napthine, Lukas Pekarek, Anuja Kibe, Andrew E. Firth, Stephen C. Graham, Neva Caliskan, Ian Brierley bioRxiv 2020.08.11.245035; doi: https://doi.org/10.1101/2020.08.11.245035 |
| Software and Algorithms | |||
| Atom | https://atom.io/packages/ide-python | N/A | |
| Bluelake | Lumicks | N/A | |
| Graphpad | https://www.graphpad.com/ | N/A | |
| InkScape 0.92.3 | https://inkscape.org/ | N/A | |
| Matlab | https://www.mathworks.com/products/matlab.html | N/A | |
| POTATO | https://github.com/lpekarek/POTATO.git | N/A | |
| RNAstructure | https://rna.urmc.rochester.edu/RNAstructure.html | N/A | |
| Spyder | https://www.spyder-ide.org/ | N/A | |
| Other | |||
| Streptavidin Coated Polystyrene Particles, 1.5-1.9 µm, 5 ml, 1.0% w/v | Spherotech | SVP-15-5 | |
| Anti-digoxigenin Coated Polystyrene Particles, 2.0-2.4 µm, 2 ml, 0.1% w/v | Spherotech | DIGP-20-2 | |
| Syringes | VWR | TERUMO SS+03L1 | |
| Devices | |||
| C-trap | Lumicks | N/A | optical tweezers coupled with confocal microscopy |
Riferimenti
- Balcerak, A., Trebinska-Stryjewska, A., Konopinski, R., Wakula, M., Grzybowska, E. A. RNA-protein interactions: disorder, moonlighting and junk contribute to eukaryotic complexity. Open Biology. 9 (6), 190096 (2019).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Firth, A. E., Brierley, I. Non-canonical translation in RNA viruses. Journal of General Virology. 93, 1385-1409 (2012).
- Caliskan, N., Peske, F., Rodnina, M. V. Changed in translation: mRNA recoding by −1 programmed ribosomal frameshifting. Trends in Biochemical Sciences. 40 (5), 265-274 (2015).
- Jaafar, Z. A., Kieft, J. S. Viral RNA structure-based strategies to manipulate translation. Nature Reviews Microbiology. 17 (2), 110-123 (2019).
- Eswarappa, S. M., et al. Programmed translational readthrough generates antiangiogenic VEGF-Ax. Cell. 157 (7), 1605-1618 (2014).
- Rodnina, M. V., et al. Translational recoding: canonical translation mechanisms reinterpreted. Nucleic Acids Research. 48 (3), 1056-1067 (2020).
- Li, Y., et al. Transactivation of programmed ribosomal frameshifting by a viral protein. Proceedings of the National Academy of Sciences. 111 (21), 2172 (2014).
- Napthine, S., et al. Protein-directed ribosomal frameshifting temporally regulates gene expression. Nature Communications. 8 (1), 15582 (2017).
- Patel, A., et al. Molecular characterization of the RNA-protein complex directing -2/-1 programmed ribosomal frameshifting during arterivirus replicase expression. Journal of Biological Chemistry. 295 (52), 17904-17921 (2020).
- Napthine, S., Bell, S., Hill, C. H., Brierley, I., Firth, A. E. Characterization of the stimulators of protein-directed ribosomal frameshifting in Theiler's murine encephalomyelitis virus. Nucleic Acids Research. 47 (15), 8207-8223 (2019).
- Marshall, R. A., Aitken, C. E., Dorywalska, M., Puglisi, J. D. Translation at the Single-Molecule Level. Annual Review of Biochemistry. 77 (1), 177-203 (2008).
- Rodnina, M. V. The ribosome in action: Tuning of translational efficiency and protein folding. Protein science : A publication of the Protein Society. 25 (8), 1390-1406 (2016).
- Rodnina, M. V., Fischer, N., Maracci, C., Stark, H. Ribosome dynamics during decoding. Philosophical Transactions of Royal Society of London B Biological Sciences. 372 (1716), (2017).
- Yan, S., Wen, J. D., Bustamante, C., Tinoco, I. Ribosome excursions during mRNA translocation mediate broad branching of frameshift pathways. Cell. 160 (5), 870-881 (2015).
- Liu, T., et al. Direct measurement of the mechanical work during translocation by the ribosome. eLife. 3, 03406 (2014).
- Desai, V. P., et al. Co-temporal force and fluorescence measurements reveal a ribosomal gear shift mechanism of translation regulation by structured mRNAs. Molecular Cell. 75 (5), 1007-1019 (2019).
- Choi, J., O'Loughlin, S., Atkins, J. F., Puglisi, J. D. The energy landscape of -1 ribosomal frameshifting. Science Advances. 6 (1), (2020).
- Prabhakar, A., Puglisi, E. V., Puglisi, J. D. Single-molecule fluorescence applied to translation. Cold Spring Harbor Perspectives in Biology. 11 (1), 032714 (2019).
- Bao, C., et al. mRNA stem-loops can pause the ribosome by hindering A-site tRNA binding. Elife. 9, 55799 (2020).
- Chen, J., Tsai, A., O'Leary, S. E., Petrov, A., Puglisi, J. D. Unraveling the dynamics of ribosome translocation. Current Opinion in Structural Biology. 22 (6), 804-814 (2012).
- Qu, X., et al. The ribosome uses two active mechanisms to unwind messenger RNA during translation. Nature. 475 (7354), 118-121 (2011).
- Zheng, Q., et al. Ultra-stable organic fluorophores for single-molecule research. Chemical Society Reviews. 43 (4), 1044-1056 (2014).
- Blanchard, S. C. Single-molecule observations of ribosome function. Current Opinion in Structural Biology. 19 (1), 103-109 (2009).
- Juette, M. F., et al. The bright future of single-molecule fluorescence imaging. Current Opinion in Chemical Biology. 20, 103-111 (2014).
- McCauley, M. J., Williams, M. C. Mechanisms of DNA binding determined in optical tweezers experiments. Biopolymers. 85 (2), 154-168 (2007).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters. 11 (5), 288-290 (1986).
- Bustamante, C., Smith, S. B., Liphardt, J., Smith, D. Single-molecule studies of DNA mechanics. Current Opinion in Structural Biology. 10 (3), 279-285 (2000).
- Choudhary, D., Mossa, A., Jadhav, M., Cecconi, C. Bio-molecular applications of recent developments in optical tweezers. Biomolecules. 9 (1), 23 (2019).
- Moffitt, J. R., Chemla, Y. R., Smith, S. B., Bustamante, C. Recent advances in optical tweezers. Annual Reviews of Biochemistry. 77, 205-228 (2008).
- Li, P. T. X., Vieregg, J., Tinoco, I. How RNA Unfolds and Refolds. Annual Review of Biochemistry. 77 (1), 77-100 (2008).
- Stephenson, W., Wan, G., Tenenbaum, S. A., Li, P. T. Nanomanipulation of single RNA molecules by optical tweezers. Journal of Visualized Experiments. (90), e51542 (2014).
- Halma, M. T. J., Ritchie, D. B., Cappellano, T. R., Neupane, K., Woodside, M. T. Complex dynamics under tension in a high-efficiency frameshift stimulatory structure. Proceedings of the National Academy of Sciences. 116 (39), 19500 (2019).
- Hansen, T. M., Reihani, S. N. S., Oddershede, L. B., Sørensen, M. A. Correlation between mechanical strength of messenger RNA pseudoknots and ribosomal frameshifting. Proceedings of the National Academy of Sciences of the United States of America. 104 (14), 5830-5835 (2007).
- Zhong, Z., et al. Mechanical unfolding kinetics of the SRV-1 gag-pro mRNA pseudoknot: possible implications for -1 ribosomal frameshifting stimulation. Science Reports. 6, 39549 (2016).
- McCauley, M. J., Rouzina, I., Li, J., Núñez, M. E., Williams, M. C. Significant differences in RNA structure destabilization by HIV-1 GagDp6 and NCp7 proteins. Viruses. 12 (5), 484 (2020).
- de Messieres, M., et al. Single-molecule measurements of the CCR5 mRNA unfolding pathways. Biophysics Journal. 106 (1), 244-252 (2014).
- Yang, L., et al. Single-molecule mechanical folding and unfolding of RNA hairpins: Effects of single A-U to A·C pair substitutions and single proton binding and implications for mRNA structure-induced -1 ribosomal frameshifting. Journal of American Chemical Society. 140 (26), 8172-8184 (2018).
- McCauley, M. J., et al. Targeted binding of nucleocapsid protein transforms the folding landscape of HIV-1 TAR RNA. Proceedings of the National Academy of Sciences of the United States of America. 112 (44), 13555-13560 (2015).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. High-resolution "Fleezers": Dual-trap optical tweezers combined with single-molecule fluorescence detection. Methods in Molecular Biology. 1486, 183-256 (2017).
- Yerramilli, V. S., Kim, K. H. Labeling RNAs in live cells using malachite green aptamer scaffolds as fluorescent probes. ACS Synthetic Biology. 7 (3), 758-766 (2018).
- Gross, P., Farge, G., Peterman, E. J., Wuite, G. J. Combining optical tweezers, single-molecule fluorescence microscopy, and microfluidics for studies of DNA-protein interactions. Methods in Enzymology. 475, 427-453 (2010).
- Zimmer, M. M., et al. The short isoform of the host antiviral protein ZAP acts as an inhibitor of SARS-CoV-2 programmed ribosomal frameshifting. Nature Communications. 12 (1), 7193 (2021).
- Neupane, K., Yu, H., Foster, D. A. N., Wang, F., Woodside, M. T. Single-molecule force spectroscopy of the add adenine riboswitch relates folding to regulatory mechanism. Nucleic acids research. 39 (17), 7677-7687 (2011).
- Ritchie, D. B., Soong, J., Sikkema, W. K., Woodside, M. T. Anti-frameshifting ligand reduces the conformational plasticity of the SARS virus pseudoknot. Journal of the American Chemical Society. 136 (6), 2196-2199 (2014).
- Janissen, R., et al. Invincible DNA tethers: covalent DNA anchoring for enhanced temporal and force stability in magnetic tweezers experiments. Nucleic Acids Research. 42 (18), 137 (2014).
- Smith, S. B., Cui, Y., Bustamante, C. Overstretching B-DNA: The elastic response of individual double-stranded and single-stranded DNA molecules. Science. 271 (5250), 795 (1996).
- Puljung, M. C., Zagotta, W. N. Labeling of specific cysteines in proteins using reversible metal protection. Biophysical Journal. 100 (10), 2513-2521 (2011).
- Toseland, C. P. Fluorescent labeling and modification of proteins. Journal of Chemical Biology. 6 (3), 85-95 (2013).
- Hill, C. H., et al. Structural and molecular basis for Cardiovirus 2A protein as a viral gene expression switch. Nature Communications. 12 (1), 7166 (2021).
- Butterworth, S. On the theory of filter amplifiers. Experimental Wireless and the Wireless Engineer. 7, 536-541 (1930).
- Wang, M. D., Yin, H., Landick, R., Gelles, J., Block, S. M. Stretching DNA with optical tweezers. Biophysics Journal. 72 (3), 1335-1346 (1997).
- Mukhortava, A., et al. Structural heterogeneity of attC integron recombination sites revealed by optical tweezers. Nucleic Acids Research. 47 (4), 1861-1870 (2019).
- Buck, S., Pekarek, L., Caliskan, N. POTATO: An automated pipeline for batch analysis of optical tweezers data. bioRxiv. , (2021).
- Zhang, Y., Jiao, J., Rebane, A. A. Hidden Markov modeling with detailed balance and its application to single protein folding. Biophysical Journal. 111 (10), 2110-2124 (2016).
- Sgouralis, I., Pressé, S. An introduction to infinite HMMs for single-molecule data analysis. Biophysics Journal. 112 (10), 2021-2029 (2017).
- Müllner, F. E., Syed, S., Selvin, P. R., Sigworth, F. J. Improved hidden Markov models for molecular motors, part 1: basic theory. Biophysical Journal. 99 (11), 3684-3695 (2010).
- Elms, P. J., Chodera, J. D., Bustamante, C. J., Marqusee, S. Limitations of constant-force-feedback experiments. Biophysical Journal. 103 (7), 1490-1499 (2012).
- Re, A., Joshi, T., Kulberkyte, E., Morris, Q., Workman, C. T. RNA-protein interactions: an overview. Methods Molecular Biology. 1097, 491-521 (2014).
- Jankowsky, E., Harris, M. E. Specificity and nonspecificity in RNA-protein interactions. Nature reviews. Molecular Cell Biology. 16 (9), 533-544 (2015).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Sunbul, M., Jäschke, A. SRB-2: a promiscuous rainbow aptamer for live-cell RNA imaging. Nucleic Acids Research. 46 (18), 110 (2018).
- Sanchez de Groot, N., et al. RNA structure drives interaction with proteins. Nature Communications. 10 (1), 3246 (2019).
- Zeffman, A., Hassard, S., Varani, G., Lever, A. The major HIV-1 packaging signal is an extended bulged stem loop whose structure is altered on interaction with the Gag polyprotein. Journal of Molecular Biology. 297 (4), 877-893 (2000).
- Mangeol, P., et al. Probing ribosomal protein-RNA interactions with an external force. Proceedings of the National Academy of Sciences. 108 (45), 18272 (2011).
- Luo, X., et al. Molecular mechanism of RNA recognition by Zinc-Finger antiviral protein. Cell Reports. 30 (1), 46-52 (2020).
- Qu, X., Lancaster, L., Noller, H. F., Bustamante, C., Tinoco, I. Ribosomal protein S1 unwinds double-stranded RNA in multiple steps. Proceedings of the National Academy of Science U. S. A. 109 (36), 14458-14463 (2012).
- Chandra, V., Hannan, Z., Xu, H., Mandal, M. Single-molecule analysis reveals multi-state folding of a guanine riboswitch. Nature Chemical Biology. 13 (2), 194-201 (2017).
- Savinov, A., Perez, C. F., Block, S. M. Single-molecule studies of riboswitch folding. Biochimica et Biophysica Acta. 1839 (10), 1030-1045 (2014).
- Kelly, J. A., et al. Structural and functional conservation of the programmed ribosomal frameshift signal of SARS coronavirus 2 (SARS-CoV-2). Journal of Biological Chemistry. 295 (31), 10741-10748 (2020).
- Neupane, K., et al. Structural dynamics of single SARS-CoV-2 pseudoknot molecules reveal topologically distinct conformers. Nature Communications. 12 (1), 4749 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Zheng, Q., Jockusch, S., Zhou, Z., Blanchard, S. C. The contribution of reactive oxygen species to the photobleaching of organic fluorophores. Photochemistry and Photobiology. 90 (2), 448-454 (2014).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Rill, N., Mukhortava, A., Lorenz, S., Tessmer, I. Alkyltransferase-like protein clusters scan DNA rapidly over long distances and recruit NER to alkyl-DNA lesions. Proceedings of the National Academy of Science U. S. A. 117 (17), 9318-9328 (2020).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Wen, J. -. D., et al. Force unfolding kinetics of RNA using optical tweezers. I. Effects of experimental variables on measured results. Biophysical journal. 92 (9), 2996-3009 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon