Method Article
G-Dörtlülerinde DNA Polimeraz Dinamiğinin Tek Moleküllü Floresan Görselleştirmesi
Bu Makalede
Özet
Bu protokol, DNA polimerazlar ve G-dörtlü yapılar gibi engeller arasındaki etkileşimlerin gerçek zamanlı görselleştirilmesini sağlayan floresan mikroskobu tabanlı tek moleküllü DNA replikasyon testini ana hatlarıyla belirtir.
Özet
Ökaryotik DNA replikasyonunda yer alan proteinlerin engellerin üstesinden gelme yeteneği - protein ve DNA 'barikatları' gibi - sadık genom duplikasyonunu sağlamak için kritik öneme sahiptir. G-dörtlüleri, DNA'nın guanin açısından zengin bölgelerinde oluşan ve genomik idame yollarına müdahale ederek engel teşkil ettiği gösterilen daha yüksek dereceli nükleik asit yapılarıdır. Bu çalışma, G-dörtlü yapılarla DNA polimeraz etkileşimlerini gözlemlemek için gerçek zamanlı, floresan mikroskobu tabanlı bir yöntem sunmaktadır. Bir G-dörtlüsü içeren kısa, astarlanmış DNA oligonükleotidleri, bir mikroakışkan akış hücresi içinde işlevselleştirilmiş cam lameller üzerinde hareketsiz hale getirildi. Floresan etiketli DNA polimerazlar tanıtıldı, bu da davranışlarının ve stokiyometrilerinin zaman içinde izlenmesine izin verdi. Bu yaklaşım, bir G-dörtlüsü tarafından durdurulduğu için polimeraz davranışının gözlemlenmesini sağladı. Spesifik olarak, floresan etiketli maya polimeraz δ kullanılarak, bir G-dörtlüsü ile karşılaşıldığında, polimerazın sürekli bir bağlanma ve ayrılma döngüsüne girdiği bulundu. Bu tek moleküllü tahlil, çeşitli DNA bakım proteinleri ve DNA substratı üzerindeki engeller arasındaki etkileşimleri incelemek için uyarlanabilir.
Giriş
DNA polimerazlar, DNA 1,2,3,4,5'i çoğaltmak için nükleosit trifosfatların dahil edilmesini katalize eden enzimlerdir. Bu nedenle, DNA replikasyonu 6,7,8 ve onarım 9,10,11,12 dahil olmak üzere temel DNA bakım süreçlerinde kilit bir rol oynarlar. DNA polimerazlar, genomik bütünlüğü sağlamak için genomu doğru ve verimli bir şekilde çoğaltmalı ve genomda mutasyon birikimini önlemelidir. Sentez sırasında, polimerazlar genellikle DNA'ya bağlı proteinler veya ikincil DNA yapıları gibi "barikatlar" ile karşılaşırlar13. Bu barikatlar polimeraz ilerlemesini yavaşlatabilir ve hatta engelleyebilir14. Bu engellerin üstesinden gelmek, sadık genom duplikasyonunu sağlamak için önemlidir, çünkü bunun yapılmaması genomik kararsızlığa yol açabilir 15,16.
Başlıca engel sınıflarından biri, insan genomu 17 içindeki guanin açısından zengin dizilerde oluştuğu gösterilen kanonik olmayan ikincil DNA yapıları olan G-dörtlüleridir(G4). İnsan genomunda, telomerler ve onkogen promotörleri18 içindeki bölgeler de dahil olmak üzere bir G4 oluşturabilen 700.000'den fazla farklı dizi vardır. Bu DNA yapıları, nükleotid dizisine, uzunluğuna ve bağlı metal katyonunabağlı olarak çeşitli konformasyonları benimser 19,20,21. Bu çeşitlilik, polimerazların, potansiyel olarak değişen derecelerde verimlilikle bir dizi farklı G4 topolojisinin üstesinden gelmesi gerektiği anlamına gelir. Polimerazların bir G4 yapısının üstesinden gelememesinin veya atlayamamasının, in vivo replikasyon çatalı ilerlemesini engellediği ve genomik instabiliteye yol açtığı gösterilmiştir22. İn vitro çalışmalar, G4 yapılarının maya polimerazlarını durdurabildiğini veya tamamen bloke edebildiğinigöstermiştir 23,24,25,26,27. G4 yapılarının DNA polimerazları durdurma veya bloke etme yeteneği tamamen kinetik ve termodinamik stabilitelerine bağlıdır ve bazı polimerazlar belirli G4'leri28 açabilmektedir. Bu çalışmalar, polimerazların G-dörtlü barikatların üstesinden gelme yeteneği hakkında fikir verirken, bir G4 ile karşılaştıklarında polimerazın davranışını doğrudan görselleştirme yeteneğinden yoksundurlar. Polimerazların kaderi - bağlı kalmaları, düşmeleri veya dinamik olarak değişip değişmemeleri - G4'ü çözmek için hangi aşağı akış süreçlerinin erişilebilir olduğunu belirler.
Bu çalışmada, DNA polimerazın G4 yapıları ile gerçek zamanlı olarak bağlanmasını doğrudan görselleştirmek ve izlemek için floresan tabanlı tek moleküllü bir mikroskopi testi geliştirilmiştir. Bu tahlil, G4 oluşturan DNA şablonlarının, DNA sentezini başlatmak için floresan etiketli DNA polimerazların eklenebileceği bir mikroakışkan akış hücresindeki biyotinile edilmiş bir lamele bağlanmasını içerir. Polimerazların zaman içindeki floresansını ölçerek, bir G4 yapısıyla karşılaştıklarında davranışları doğrudan gözlemlenebilir. C-MYC kanser onkogeninde bulunan G4 yapısı, yüksek stabilite seviyesi nedeniyle bu test için seçilmiştir. Bu protokol artık farklı G4 topolojileri ve stabiliteleri ile ilişkili yaşamın tüm alanlarında çeşitli polimerazların davranışını kataloglamak için uyarlanabilir. Bu tahlil, DNA polimerazların DNA engellerini aştığı mekanizmaları aydınlatmak için yenilikçi ve yüksek verimli bir yaklaşım sunar ve polimeraz dinamiklerinin anlaşılmasını ilerletmek için güçlü bir araç sağlar.
Protokol
Reaktiflerin ve kullanılan ekipmanın ayrıntıları Malzeme Tablosunda listelenmiştir.
1. Dairesel dikroizm spektroskopisi
NOT: Testi geliştirmeden önce, doğru katlamayı sağlamak için seçilen G4 dizisi üzerinde dairesel dikroizm (CD) spektroskopisi yapmak gerekliydi. 22 nt dizisi (5′-TGAGGGTGGGGGGGGGGAA-3′), c-MYC kanser onkogeninden türetilen G4 yapısını oluşturur. CD spektroskopisi ayrıca, G4 oluşumunu önleyen anahtar nükleotidlerin değiştirildiği bir kontrol dizisi (5'- TGAGTGTGAAGACGATGTAGAA -3) üzerinde de gerçekleştirildi.
- DNA şablonlarını CD spektroskopisi için hazırlamak için, 200 μL potasyum klorür (KCl) ile desteklenmiş 360 μL Tris-HCl (pH 8.0), etilendiamintetraasetik asit (EDTA) (TE) tamponuna 40 μL 100 μM 22 nt şablonu ekleyin. CD spektroskopisi deneyleri için 10 μM'lik bir nihai DNA konsantrasyonu arzu edilir.
NOT: Şablondaki G4 yapısını stabilize etmek için K+ iyonlarının varlığı çok önemlidir. - DNA solüsyonlarını dijital kuru banyoda 15 dakika boyunca 95 ° C'ye ısıtın. Tamamlandığında, ısıtma bloğunu polistiren içine yerleştirerek çözeltileri gece boyunca yavaşça soğutun. Bu yavaş soğutma işlemi, G4'ün katlanmasından sorumludur.
- 400 μL'lik G4 oluşturucu ve kontrol dizisi solüsyonlarını gaz geçirmez bir şırınga kullanarak 0,1 cm yol uzunluğunda bir kuvars küvete aktarın. Hava kabarcıklarının oluşmasını önlemek için küveti yavaşça doldurun.
- 25 ° C'de bir spektropolarimetre kullanarak 200 nm ile 400 nm arasındaki CD spektrumunu ölçün. Bu ölçüm için parametreler ve koşullar daha önce açıklanmıştır29. Sonuçlar Ek Şekil 1'de bulunabilir.
NOT: Boş olarak işlev görmesi için yalnızca arabellek için CD spektrumunu kaydettiğinizden emin olun.
2. DNA şablonlarının hazırlanması
NOT: Çoğaltılabilir özellikteki şablonlar, standart moleküler biyoloji teknikleri kullanılarak hazırlanmış kısa, 100 nt astarlı lineer şablonlardır. G4 oluşturan şablon (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGTGGGGGTGTGTGTGT
GGGACCTTAGCTTCGAGTTCCGAT-3'), merkezinde MYC'den türetilmiş bir G-dörtlüsü (1. adımdan itibaren) oluşturabilen diziyi içerir. Kontrol şablonu (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3") merkezinde de aynı kontrol bölgesine (1. adımdan itibaren) sahiptir.
- Primeri G4 oluşturan ve kontrol DNA şablonlarına tavlamak için, 20 μM primer (5 μ - Biotin-AF647-TCTTAATGTAAATTTAAAATGTC-3 "), 200 mM KCl ile desteklenmiş TE tamponunda 100 nt şablonlarının 20 μM'sini karıştırın.
- DNA şablon solüsyonlarını dijital kuru banyoda 15 dakika boyunca 95 °C'ye ısıtın. Tamamlandığında, G4'ün katlanmasına ve iki telin tavlanmasına izin vermek için ısıtma bloğunu polistirene yerleştirerek çözeltileri gece boyunca yavaşça soğutun.
3. DNA polimerazın AF647 ile etiketlenmesi
- Polimerazın boya ile etiketlenmesi
NOT: Pol δ mayası, daha önce tarif edildiği gibi ifade edilmiş ve saflaştırılmıştır30. Polimerazın daha sonra tek molekül seviyesinde görselleştirmeye izin vermek için Alexa Fluor NHS ester mono-reaktif boya (AF647) ile etiketlenmesi gerekiyordu. Bu, daha önce açıklandığı gibi tamamlandı5.- N-terminalini etiketlemek için, 30 mM Tris-HCl (pH 7.6), 2 mM ditiyotreitol (DTT), 300 mM sodyum klorür (NaCl), 50 mM potasyum glutamat ve% 10 (h/h) gliserol içeren bir tamponda 320 μL 58.5 μM Pol δ ile 3 kat fazla AF647 boyasını inkübe edin 4 ° C'de 10 dakika boyunca. Kapsamlı bir karıştırma sağlamak ve etiketleme verimliliğini artırmak için çözeltiyi yavaşça döndürün.
NOT: Protein dizisi, lizin amino asitlerinde ek birincil aminler içerebilir. PH gibi reaksiyon koşullarını ayarlayarak, etiket, lizin kalıntılarının aksine tercihen N-terminaline yapıştırılabilir. Bu, etiketleme verimliliğini artırmak için daha fazla optimizasyon gerektirebilir. - 0,5 mL spin tuzdan arındırma kolonlarını (7 K moleküler ağırlık kesilmiş) hazırlamak için, depolama solüsyonunu çıkarmak için önce kolonları 1500 x g'da 1 dakika santrifüjleyin. Santrifüjlemeden sonra toplama tüpünde biriken çözeltileri atın.
NOT: Santrifüjleme adımları sırasında döndürme kolonlarının kapağını gevşettiğinizden emin olun. Bu, sütunların düzgün bir şekilde ayrıştırılmasını sağlayacaktır. - Kolonu 30 μL tampon ekleyerek 30 mM Tris-HCl (pH 7.6), 300 mM NaCl, 3 mM DTT, 50 mM potasyum glutamat ve %5 (h/h) gliserol içeren bir tamponda dengeleyin. Kolonu 1 dakika boyunca 1500 x g'da santrifüjleyin ve akışı atın. Bu işlemi iki kez daha tekrarlayın.
- Kolona her bir Pol δ numunesinden 200 μL uygulayın. Sütunları 1500 x g'da 2 dakika santrifüjleyin ve saflaştırılmış Pol δ 1.5 mL'lik bir mikrosantrifüj tüpünde toplayın.
- Saflaştırılmış fraksiyonları sıvıN2 içinde dondurun ve zamanla protein bozulmasını önlemek için alikotları -80 °C'de saklayın.
- N-terminalini etiketlemek için, 30 mM Tris-HCl (pH 7.6), 2 mM ditiyotreitol (DTT), 300 mM sodyum klorür (NaCl), 50 mM potasyum glutamat ve% 10 (h/h) gliserol içeren bir tamponda 320 μL 58.5 μM Pol δ ile 3 kat fazla AF647 boyasını inkübe edin 4 ° C'de 10 dakika boyunca. Kapsamlı bir karıştırma sağlamak ve etiketleme verimliliğini artırmak için çözeltiyi yavaşça döndürün.
- Etiketleme verimliliğinin belirlenmesi
- Şimdi AF2 etiketli Pol δ numunesinden 647 μL'yi bir UV-vis spektrofotometresine31 yükleyin.
NOT: Önce Pol δ tamponunun boşluğunun ölçüldüğünden emin olun. - Spektrofotometreyi kullanarak AF647 boyası için 650 nm ve Pol δ için 280 nm'lik uyarma dalga boylarını ölçün.
- Beer-Lambert yasasını ve AF647 boyası için 270.000 cm-1 M-1 ve polimeraz için 195.960 cm-1 M-1 molar sönme katsayılarını kullanarak etiketleme verimliliğini hesaplayın.
NOT: Bu testte kullanılan AF647 etiketli maya Pol δ için belirlenen etiketleme verimliliği yaklaşık %67 olarak hesaplanmıştır.
- Şimdi AF2 etiketli Pol δ numunesinden 647 μL'yi bir UV-vis spektrofotometresine31 yükleyin.
4. Topluluk primer uzatma testi
NOT: Tek moleküllü replikasyon deneylerini gerçekleştirmeden önce, DNA polimerazın toplu replikasyon deneyleri yoluyla G-dörtlüsü tarafından bloke edildiğini doğrulamak gerekir.
- Dijital kuru banyodaki bir ısıtma bloğunun sıcaklığını 30 °C'ye ayarlayın. Bu, maya Pol δ'in maksimum aktivitesini sağlayacaktır.
- Replikasyon tamponunda (25 mM Tris-HCl (pH 7.6), 10 mM magnezyum asetat, 50 mM potasyum glutamat, 40 μg/mL sığır serum albümini (BSA), 0.1 mM EDTA, 5 mM DTT ve %0.0025 (h/h) Tween-20). Her karışımı 1 mM DTT, her biri 250 μM dTTP, dCTP, dATP, dGTP (dNTP'ler) ve 10 nM ilgili DNA şablonu (G4 oluşturucu veya kontrol) ile destekleyin. 30 °C'ye ulaştıklarından emin olmak için bunları bir ısıtma bloğuna yerleştirin.
- T = 0 dk kontrol olarak işlev görmek için 12 μL formamid yükleme tamponu (%80 (a/h) formamid, 10 mM EDTA) ile her ana karışımın 12 μL'sini söndürün.
- DNA sentezini başlatmak için, her bir ana karışıma (20 nM nihai konsantrasyon) AF647 etiketli maya Pol δ ekleyin. DNA polimerazların şablonları maksimum verimlilikle çoğaltmasını sağlamak için iyice karıştırın.
NOT: Enzim aktivitesini korumak için kullanım sırasında mayayı Pol δ buz üzerinde tutun. - 30 sn, 60 sn ve 180 s'den sonra, her bir ana karışımdan 12 μL'yi çıkarın ve 12 μL formamid yükleme tamponu ile söndürün. Ana karışımın formamid yükleme tamponuna 1:1 oranı, replikasyon reaksiyonunun bir kez söndürüldükten sonra devam etmemesini sağlar.
- Şimdi söndürülmüş çözeltileri 30 °C ısıtma bloğundan çıkarın ve dsDNA'yı ssDNA'ya denatüre etmek yerine 98 °C'lik bir ısıtma bloğuna yerleştirin. 10 dakika geçtikten sonra, çözeltilerin denatüre edici% 15 tris-borat-EDTA (TBE) -üre poliakrilamid (PAGE) jel32'ye yüklenmesi için tüm ısıtma bloğunu jel tankına getirin.
NOT: PAGE jelinin, numunelerin yüklenmesinden önce, jel elektroforezini 180 V'ta 30 x TBE tamponunda 1 dakika çalıştırarak ısıtması gerekir. Tamamlandığında, üreyi çıkarmak için her bir kuyuyu durulayın. - Formamid yükleme tamponunda 0.02 μM 20 nt, 60 nt ve 100 nt etiketli oligonükleotid içeren bir merdivenin yanında her numuneden 15 μL jel üzerine yükleyin.
- Tam ayrılmayı sağlamak için jeli 1x TBE tamponunda 180 V'ta 60 dakika çalıştırın.
- Jeli bir biyomoleküler görüntüleyicinin Cy5 kanalında görüntüleyin. Bu, AF-647 etiketini içeren kopyalanmış DNA zincirinin ölçülmesine izin verecektir.
5. Tek moleküllü floresan mikroskobu
- Lamel işlevselleştirme
NOT: DNA şablonlarını cam lamele takmak için önce aminosilan ile işlevselleştirilmeli, ardından biyotinile PEG moleküllerinin bağlanması gerekir. Bu işlem, DNA ve/veya proteinler ile yüzey arasındaki spesifik olmayan etkileşimleri en aza indirir.- Lamelleri (24 x 24 mm) boyama kavanozlarına koyarak ve etanol dökerek temizleyin. Lamelleri saf suyla durulamadan önce kavanozları 30 dakika boyunca sonikleştirin. Tekrar durulamadan önce bu işlemi 1 M potasyum hidroksit (KOH) ile tekrarlayın. Bu temizleme adımlarını bir kez daha tekrarlayın.
- Durulayın ve yeni bir kavanozu asetonla doldurun ve lamelleri içine yerleştirin. % 2 v / v çözeltisi oluşturmak için 3-aminopropiltrietoksisilanı kavanozun içine yayın. Reaksiyonu çok fazla su ile söndürmeden önce kavanozu 3 dakika çalkalayın.
- Lamelleri N2 ile kurulayın ve ayrı ayrı su dolu nemli bir kutuya yerleştirin. Bu, lamellerin kurumasını önleyecektir.
- 100 mM NaHCO3 (pH 8.2) içinde 1:25 Biotin-PEG-SVA:mPEG-SVA esteri hazırlayın. İyice karıştırmayı sağlamak için bu karışımı 20 saniye boyunca vorteksleyin.
- 40 μL PEG solüsyonunu kuru bir lamel üzerine pipetleyin. Çözeltiyi iki lamel arasına "sandviç" yapmak için üstüne başka bir lamel yerleştirin. Bu, lamel yüzlerini iç kısımda işlevsel hale getirecektir. Tüm lameller için tekrarlayın.
- Lamelleri karanlıkta 3 saat inkübe edin. İnkübasyondan sonra, lamel çiftlerini ayırın, fazla su ile durulayın ve sıkıştırılmış N2 gazı ile kurulayın.
- Lamellere ikinci bir PEG katmanı uygulamak için 4.1.4-4.1.5 adımlarını tekrarlayın. Önceden işlevselleştirilmiş kenarları birbirine sandviçlediğinizden emin olun, bu nedenle yıkama adımları sırasında yanlışlıkla çevirmemeye dikkat edin. Solüsyonları durulamadan ve kurutmadan önce gece boyunca karanlıkta inkübe edin.
- İşlevselliklerini korumak için lamelleri bir vakumda saklayın. İşlevselleştirilmiş lameller, doğru şekilde saklandığında bir ay boyunca stabildir.
- Mikroakışkan akış hücrelerinin hazırlanması
NOT: Tek moleküllü deneyler için bir mikroakışkan akış hücresi33 inşa edilmiştir (bkz. Ek Şekil 2). Bu, tamponların, DNA şablonlarının ve proteinlerin işlevselleştirilmiş bir lamel ile temas etmesine izin verecektir (bkz. adım 5.1).- İşlevselleştirilmiş bir lameli elektrikli süpürgeden çıkarın ve kısmen suyla dolu bir mikrotüp rafına (nemli kutu) yerleştirin. 100 μL'lik bir bloke edici tamponu (50 mM Tris-HCl (pH 7.6), 50 mM KCl, %2 (h/h) Tween-20) ile 25 μL 1 mg/mL NeutrAvidin çözeltisi (%10 PBS) karıştırın ve bunu lamel yüzeyine yayın. Bunu nemli kutuda oda sıcaklığında 15 dakika inkübe edin.
- Lameli suda yıkayın ve N2'de kurutun. Lamelin yalnızca bir tarafının işlevsel olduğunu unutmayın, bu nedenle hangi taraf olduğunu unutmayın.
- Özel yapım polidimetilsiloksan (PDMS) bloğunu34 akış hücresi tutucusu içindeki lamel üzerine yerleştirin. Bu, 100 μm yüksekliğinde ve 1 mm genişliğinde bir akış kanalı oluşturacaktır. Polietilen borular (PE-60: 0,76 mm giriş çapı ve 1,22 mm dış çap) daha sonra tamponlara ve alt tabakalara erişim sağlamak için akış hücresindeki deliklere yerleştirilebilir.
- Tek moleküllü floresan görüntüleme
NOT: Bu deneyler hem G4 şekillendirme hem de kontrol şablonlarıyla gerçekleştirilmelidir. Bu, her iki koşulun da (Pol δ'nin bir G4 yapısı tarafından bloke edilmesi ve blokaj olmaması) toplam iç yansıma (TIRF) mikroskobu kullanılarak tek molekül seviyesinde görüntülenmesine izin verecektir.- 1 mL alikotları ısıtın Tween bloke edici tampon (50 mM Tris-HCl (pH 7.6), 50 mM KCl, %2 (h/h) Tween-20) ve 500 μL alikot yıkama tamponu (25 mM Tris-HCl (pH 7.6), 10 mM magnezyum asetat, 250 mM potasyum glutamat, 40 μg/mL BSA, 0.1 mM EDTA, 5 mM DTT, %0,0025 (h/h) Tween-20) ve 15 dakika boyunca 40 ° C'ye kadar çoğaltma tamponu. Bu, gazları çözeltilerden kurtaracaktır. Çözeltileri bir vakum odasında atmosferik basıncın 800 mbar altında 15 dakika daha gazdan arındırın.
- Oluşturulan akış hücresini alın (bkz. adım 4.2) ve mikroskobun tablasına yerleştirin. Hedefin üzerine bir damla yağ koyduktan sonra, lamel ile buluşmak için hedefi kaldırın.
NOT: Objektif ve akış hücresinin 31 °C'ye ulaşması için zamana sahip olduğundan emin olun. Bu, maya Pol δ'nin aktivitesini artıracak ve tekrarlanabilir verilerin sağlanmasına yardımcı olacaktır. - Giriş borusunu gazı alınmış Tween engelleme tamponuna yerleştirin ve çıkışı şırınga pompasına bağlayın. Tween engelleme tamponunu borudan kanala çekmek için şırıngayı geri çekin. Spesifik olmayan etkileşimleri en aza indirmek için bu tamponun en az 30 dakika inkübe etmesine izin verin.
- 200 μL'lik gazdan arındırılmış yıkama tamponunu 100 μL/dk hızında kanala akıtın. Bu, Ara engelleme arabelleğini temizleyecektir.
- DNA şablon çözeltilerini 500 μL replikasyon tamponunda 0.5 pM'ye seyreltin. Kanala 10 μL/dk hızında 150 μL akıtın. Tek tek DNA şablonlarını görselleştirmek için numune düzleminde yaklaşık 900 mWcm-2'de 647 nm'lik bir lazer kullanarak numuneyi aydınlatın.
NOT: Numune ile görüş alanı (FOV) tamamen odakta değilse, kapak kayması, inşa edilen akış hücresinin altına aynı hizada oturmuyor olabilir. Böyle bir durumda yeni bir lamel kullanarak yeni bir akış hücresi oluşturun. - Yeterli bir nokta yoğunluğu (10 μm2 başına yaklaşık 1 nokta) göründüğünde, fazla DNA'yı yıkamak için kanala taze bir replikasyon tamponu çözeltisi (1 mM DTT ile desteklenmiş) içinde akıtın. 250 μL'lik bir hacim, bağlanmamış tüm DNA'yı tamamen çıkarmak için yeterlidir.
- Etiketli polimeraz ile DNA substratı arasındaki kolokalizasyon derecesini belirlemek için yeni bir FOV'a geçin ve DNA'nın bir görüntüsünü yakalayın. Tamamlandığında, kalan lekeleri foto-ağartmak için lazer gücünü artırın.
- 200 μL replikasyon tamponunda 1 mM DTT, 250 μM dNTP ve 20 nM AF647 etiketli Pol δ içeren bir polimeraz çözeltisi hazırlayın. 100 μL polimeraz çözeltisini 5 μL/dk hızında kanala akıtın.
- Numune odaklandıktan ve TIRF açısı ayarlandıktan sonra, 647 nm'lik bir lazerin lazer gücünü numune düzleminde yaklaşık 900 mWcm-2'ye ayarlayın. Ardından, FOV'u istenen süre boyunca görüntülemeye başlayın. Tüm çoğaltma olaylarını yakalamak için 10-20 dakika boyunca 1-5 kare/sn arasında çekim yapın.
- Veri analizi
NOT: Tüm analizler Python (v. 3.11.7) kullanılarak gerçekleştirilmiştir. Veri analizi için kullanılan özel kod burada mevcuttur: https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis.- İlk olarak, DNA şablon noktalarını, etiketli maya Pol δ'nin floresan noktalarıyla birlikte lokalize edin. Sürüklenme düzeltmesinden sonra mesafeleri 3 pikselden ≤ ise iki tepe noktasının birlikte lokalize olduğu belirlendi. Yalnızca DNA şablonlarını birlikte lokalize eden Pol δ noktaları, bağlanma kinetikleri ve genel davranış eğilimleri açısından analiz edilir.
- Bağlayıcı olay verilerini belirlemek için, film boyunca "açık/kapalı" davranışı sergileyen her bir noktanın zaman içindeki yoğunluğunu ölçün. Bu, tek bir DNA şablonunda Pol δ bağlanmasının kaç kez gerçekleştiğinin belirlenmesini sağlar.
NOT: Bu analiz, bağlama dinamiklerini karşılaştırmak için hem G4 oluşturan hem de kontrol şablonu deneyleri için yapılmıştır.
NOT: G-dörtlüsüne veya şablonun kendisine (23,2 sn ± 4,9 s) spesifik olmayan bağlanma olaylarının bağlanma olaylarının analizine dahil edilmemesini sağlayan kontrol deneyleri yapılmıştır.
Sonuçlar
Bu test için, floresan olarak işaretlenmiş 20-nt primerli iki DNA substratı tasarlandı: biri G4 oluşturan bir dizi içeren (Şekil 1A, solda) ve diğeri bu dizilimden yoksun (Şekil 1A, sağda). G-dörtlüsünün polimeraz aktivitesi için etkili bir engel olduğunu doğrulamak için, Pol δ ile DNA sentezi, denatüre edici bir PAGE jeli üzerinde izlendi. Saflaştırılmış etiketli Pol δ'nin DNA substratları üzerindeki aktivitesi jel elektroforezi ile incelendi. Şekil 1B (solda), floresan etiketli Pol δ'nin G-dörtlüsünü geçemediğini göstermektedir. Sentezin başlamasından önce (t = 0 dk), şablon iplikten denatüre edilmiş etiketli primeri temsil eden 20 nt'ye karşılık gelen bir bant mevcuttur. 3 dakika sonra, bu 20-nt bandı 60-nt bandına dönüştürüldü, bu da tüm DNA'da sentezin meydana geldiğini ve polimerazın G-dörtlü yapı tarafından tamamen bloke edildiğini doğruladı. Bu blokaj, polimerazın yapıyı ne açabileceğini ne de atlayabileceğini ima eder. Buna karşılık, G-dörtlü oluşturan diziyi içermeyen bir kontrol şablonunun sentezi (Şekil 1A, sağ), 3 dakika sonra 100 nt'lik bir bant üretti (Şekil 1B, sağ).
Şablonlar üzerinde sentezi doğruladıktan ve G-dörtlüsü tarafından verimli bir şekilde bloke edildikten sonra, bu ölçümler polimeraz davranışını izlemek için tek moleküllü bir floresan mikroskobu üzerinde tekrarlandı. DNA şablonları, bir mikroakışkan akış hücresinde işlevselleştirilmiş lamellere (Şekil 2A) bağlandı ve her bir substratın konumu, floresan etiketli primerin görselleştirilmesiyle belirlendi. Daha sonra, sentezi başlatmak için dNTP'lerin varlığında etiketli Pol δ yüklendi. Tipik bir FOV içinde, Pol δ'un DNA'ya ne sıklıkla bağlandığını ve DNA'dan ayrıldığını ölçmek için tek tek noktalar izlenir (Şekil 2B, C). Her bir DNA substratındaki yoğunluğu zamanın bir fonksiyonu olarak ölçerek, tek moleküllü yörüngeler oluşturulabilir (Şekil 2C, Ek Şekil 3). Karakteristik "bekleme süresi" veya Pol δ'ın şablona bağlı kaldığı ortalama süre ölçülebilir. G4 substratı için bekleme süresi 6 s ± 2 s olarak belirlenirken, kontrol substratı için 10 s ± 3 s idi (Şekil 2D, Ek Şekil 4). Ek olarak, her yörünge için Pol δ'ın şablona kaç kez bağlandığı ölçülebilir. G4 substratına bağlanma olaylarının sayısı, kontrol şablonuna kıyasla çok daha yüksektir (Şekil 2E). Skolastik bağlanma ve çözmeden dolayı kontrol şablonunda birden fazla bağlama olayının örnekleri olsa da, G4 oluşturan şablon için ortalama olarak bağlama olaylarının sayısında net bir artış vardır. Bu, sentez G-dörtlüsü tarafından durdurulduktan sonra, çözeltiden yeni polimerazlar sürekli bir bağlanma ve bağlanma döngüsüne girmeden önce bağlı polimerazın DNA'dan ayrıldığını göstermektedir. Bu nedenle, bu tek moleküllü tahlil, DNA polimerazların DNA barikatlarına nasıl tepki verdiğine dair benzersiz bir içgörü sağlar.

Şekil 1: Topluluk primer uzatma testi. (A) DNA substratlarının şematik gösterimi. G4 substratı (solda) bir G-dörtlüsü oluşturan dizi içerirken, kontrol substratı (sağda) içermez. (B) Floresan etiketli maya Pol δ'nin G-dörtlüsü tarafından tamamen bloke edildiğini gösteren astarlanmış G4 ve kontrol DNA substratlarının topluluk DNA primaz uzatma testi. PAGE jeli, DNA şablonlarının zaman içindeki replikasyonunu gösterir, bu da etiketli 20 nt primerin G4 substratı için 60 nt ürüne (solda) ve kontrol şablonu için 100 nt ürüne (sağda) kaymasına neden olur. M, etiketli 20 nt, 60 nt ve 100 nt oligoları içeren bir merdiveni temsil eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
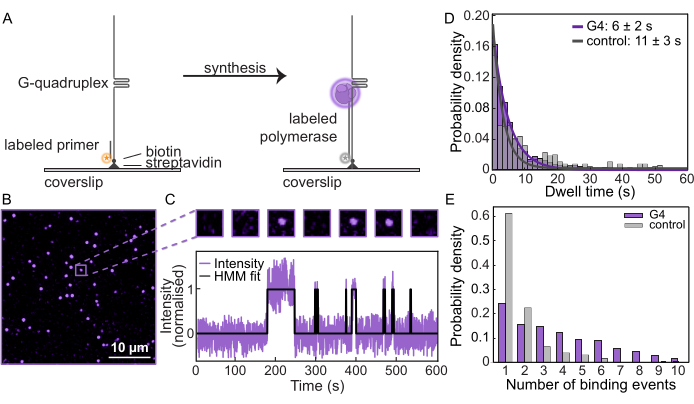
Şekil 2: Tek moleküllü floresan mikroskobu. (A) AF647 etiketli maya Pol δ kullanılarak tek moleküllü primer uzatma testinin şematik gösterimi. (B) Tek moleküllü bir testten elde edilen tipik FOV. Her nokta, bir DNA şablonuna bağlanan floresan etiketli bir Pol δ temsil eder. Ölçek çubuğu: 10 μm. (C) (Üst) Pol δ değişimleri olarak açık/kapalı bağlanmayı gösteren tek moleküllü tahlilden örnek molekül. (Altta) Aynı molekülün deneyin zaman boyunca yoğunluğunu gösteren tek moleküllü yörüngesi. Siyah çizgi, verilere uygun Gizli Markov Modeli'ni (HMM) temsil eder. (D) Pol δ'nin G4 alt tabakası (mor) ve kontrol alt tabakası (gri) üzerinde bekleme süresi. Çizgiler üstel uyumları temsil eder ve G4 alt tabakasında 6 sn ± 2 sn ve kontrol alt tabakası üzerinde 10 sn ± 3 sn kullanım ömrü sağlar. (E) Tek DNA substratlarına Pol δ bağlanma olaylarının sayısı. G4 substratı (mor) üzerindeki ortalama bağlanma olayı sayısı, kontrol substratı 1 (gri) ile karşılaştırıldığında 3.5'tir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Şekil 1: CD spektroskopisi. 25 ° C'de 200 mM KCl içeren TE tamponunda G4 oluşturan (mor) ve kontrol dizisinin (gri) CD spektrumları. 260 nm'deki karakteristik tepe noktası, ardından 240 nm'deki negatif tepe noktası, paralel, molekül içi bir GQ'nun karakteristiğidir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 2: Akış hücresi yapısı. (A) Akış hücresinin tek tek parçalarının şeması. Kare PDMS bloğu, mikroskop lamel üzerine oturur. Bu parçalar daha sonra kapağın içine ve akış hücresi tutucusunun altına yerleştirilir. (B) Tam akış hücresi. Tamponların mikroakışkanlar aracılığıyla cihazdan geçirilebileceği kanalları oluşturmak için PDMS bloğunun her iki tarafına tüpler yerleştirilebilir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 3: Tek moleküllü yörüngeler. (A) Deneyin 10 dakikalık zaman dilimi boyunca tek bir bağlanma olayına uğrayan bir molekülün örnek yörüngesi. (B) Deneyin 10 dakikalık zaman dilimi boyunca sıfır bağlanma olayına uğrayan bir molekülün örnek yörüngesi. (A) ve (B)'deki siyah çizgi, verilere uyan Gizli Markov Modelini (HMM) temsil eder. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 4: Bekleme süreleri. (A) Pol δ'in G4 substratı üzerinde bekleme süresi. Çizgi, üstel uyumu temsil eder ve 6 sn ± 2 sn arasında bir kullanım ömrü sağlar. (B) Pol δ'nin kontrol substratı üzerinde bekleme süresi. Üstel uyum, 10 sn ± 3 sn kullanım ömrü sağlar. Bu Dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 5: Foto ağartma kontrolü. Grafik, tek bir AF647 etiketli maya Pol δ enziminin 647 nm lazer tarafından fotoağartılması için ortalama süreyi göstermektedir. Çizgi, üstel uyumu temsil eder ve 39 ± 6 sn kullanım ömrü sağlar. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Burada, bir DNA polimerazın bir G-dörtlüsü ile karşılaştığında davranışı hakkında fikir veren tek moleküllü floresan bazlı bir test tanımlanmıştır. DNA şablonu oluşturma, Pol δ etiketleme ve toplu DNA replikasyon tahlillerinin protokollerinin tümü basit olsa da, tek moleküllü mikroskopi tahlillerinin yürütülmesi teknik olarak daha zordur. Tek moleküllü tekniklerin doğası gereği, toz, kirlilik veya hava kabarcıklarının girmesini önlemek için büyük özen gösterilmelidir, çünkü bunlar FOV'u gizleyecek ve veri toplamayı engelleyecektir.
Tek moleküllü TIRF mikroskobu deneylerinin bir sınırlaması, ilgilenilen biyomoleküllere kovalent olarak bağlanan floroforların foto-ağartılmasıdır. Foto ağartma, kalıcı floresan kaybına yol açan geri dönüşü olmayan bir işlemdir35. Deneyler sırasında bunu azaltmak için lazere maruz kalma süresini sınırlamak, lazer yoğunluğunu ayarlamak ve görüntüleme zamanlamasını optimize etmek çok önemlidir. Bu stratejiler, floresan sinyallerinin korunmasına yardımcı olarak daha güvenilir ve uzun gözlem süreleri sağlar. Bu parametrelere ince ayar yapıldığında, Pol δ sinyali ölçüm süresince kalır. Tek moleküllü floresan mikroskobu sentez tahlilleri için lazer gücünü optimize etmek için, etiketli polimerazı temiz bir cam lamel üzerinde görüntüleyerek fotoağartma hızının ölçülmesi tavsiye edilir. Lazer gücünü sistematik olarak değiştirerek ve görüş alanı boyunca foto ağartma oranını değerlendirerek, florofor sinyal gücünü foto ağartmaya dirençle dengeleyen optimal lazer yoğunluğu belirlenebilir (bkz. Ek Şekil 5).
Bu tek moleküllü yaklaşımın geleneksel topluluk tabanlı yöntemlere göre en önemli avantajı, tek bir DNA polimerazın bir G4 yapısıyla karşılaştığında ve etkileşime girdiğinde doğrudan görselleştirme yeteneğidir. Geleneksel topluluk tabanlı yöntemler (jel elektroforezi gibi), G4 yapılarının DNA polimerazları bloke etme kapasitesini göstermiştir 23,36,37. Bununla birlikte, bu teknikler, farklı moleküler kinetik adımları ve sonuçları çözmek için gerekli olan bu etkileşimin gerçek zamanlı kinetik ve mekanik bilgilerini sağlamada başarısız olur. Tek moleküllü teknikler, genellikle ortalama38 kişilik topluluk tarafından gizlenen biyomoleküllerin kinetiği, mekanizmaları ve davranışları hakkında benzersiz bir içgörü sunar. Artık DNA polimerazların gerçek zamanlı olarak nasıl davrandığını görmek mümkün - değiş tokuş edip etmediklerini, durdurup durmadıklarını, ayrışıp ayrışmadıklarını veya DNA barikatlarını atlayıp atlamadıklarınıgörmek mümkün 39. Bu protokol oluşturulduktan sonra, G4'ün kimliği, seçilen paralel c-MYC yapısından herhangi bir paralel, anti-paralel veya hibrit topolojiye kolayca değiştirilebilir. Bu tek moleküllü testin uygulanması, aynı DNA polimerazların alternatif G4 topolojileriyle karşılaştığında farklı davranıp davranmadığını ortaya çıkaracaktır. Bu nedenle, tek moleküllü yöntemler, vücudun proteinlerinin ve DNA'nın nasıl etkileşime girdiğine dair belirli soruları yanıtlamak için hayati önem taşır.
DNA polimeraz etkileşimlerinin G-dörtlüleri ile doğrudan görselleştirilmesi yoluyla, maya Pol δ için daha önce karakterize edilmemiş bir değişim yolu tanımlanmıştır. Bu keşif, polimerazın bir G-dörtlüsü ile karşılaştığında ayrıldığını ve DNA sentezini yeniden başlatmadan önce yapıyı çözmek için başka bir proteinin müdahalesini beklediğini göstermektedir. Bu protokol, çeşitli genom idame proteinleri ve DNA engelleri arasındaki etkileşimleri araştırmak için uyarlanabilir ve hücresel enzimlerin genomik engelleri nasıl aştığına dair benzersiz bilgiler sunar. Örneğin, bu testin barikatı, bir G4 yapısından, bir proteinin DNA'ya geri dönüşümsüz olarak kovalent olarak bağlandığı ve DNA replikasyonu için bir engel görevi gören bir tür DNA lezyonu olan bir protein-DNA çapraz bağına değiştirilebilir40. Bu tür incelemeler, DNA replikasyonu, onarımı ve rekombinasyonunun temel süreçlerini anlamak için çok önemlidir. DNA-protein dinamiklerinin moleküler düzeyde incelenmesini mümkün kılan bu tahlil, genomik bütünlüğün altında yatan mekanizmaları aydınlatmak için güçlü bir araç sağlar.
Açıklamalar
Yazarlar, rekabet eden hiçbir mali çıkarları olmadığını beyan ederler.
Teşekkürler
N.K.-A. Avustralya Hükümeti Araştırma Eğitim Programı Bursu tarafından verilen finansmanı kabul eder. L.M.S., Ulusal Sağlık ve Tıbbi Araştırma Konseyi'nden (Investigator Grant 2007778) aldığı fon için minnettardır. J.S.L, Avustralya Hükümeti tarafından finanse edilen Keşif Erken Kariyer Ödülü (DE240100780) ve NHMRC Araştırmacısı EL1'in (2025412) sahibi olduğu için minnettardır. SHM, Bruce Warren Molecular Horizons Ealy kariyer bursunun alıcısı olduğu için minnettardır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
Referanslar
- Sutton, M. D., Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci. 98 (15), 8342-8349 (2001).
- Burgers Garg, P., J, P. M. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M. -D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O., et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Sugino Kawasaki, Y., A, Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Shivji Wood, R. D., K, M. Which DNA polymerases are used for DNA repair in eukaryotes. Carcinogenesis. 18 (4), 605-610 (1997).
- Campbell Budd, M. E., L, J. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Gao Yang, W., Y, Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9. 968424, (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Monnat Loeb, L. A., J, R. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Bryan Oganesian, L., M, T. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I., et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Clayton Kaguni, L. S., A, D. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), (2021).
- Castillo Bosch,, P,, et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, J. ohnA., Zakian, V. irginiaA. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L., et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh. 11 (9), (2024).
- Pham, S. Q. T., et al. A new class of quadruplex DNA-binding nickel Schiff base complexes). Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E., et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Hansen, J. B., Au, Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J. , Springer. New York, New York, NY. 219-238 (2015).
- Lewis, J. S., et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, 23932(2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. , 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664(2014).
- Mueller, S. H., et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5(2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır