Method Article
Visualizzazione a fluorescenza a singola molecola della dinamica della DNA polimerasi ai G-quadruplex
In questo articolo
Riepilogo
Questo protocollo delinea un saggio di replicazione del DNA a singola molecola basato su microscopia a fluorescenza, che consente la visualizzazione in tempo reale delle interazioni tra le DNA polimerasi e gli ostacoli come le strutture G-quadruplex.
Abstract
La capacità delle proteine coinvolte nella replicazione del DNA eucariotico di superare gli ostacoli, come i "blocchi" proteici e del DNA, è fondamentale per garantire una duplicazione fedele del genoma. I G-quadruplex sono strutture di acidi nucleici di ordine superiore che si formano nelle regioni ricche di guanina del DNA e hanno dimostrato di agire come ostacoli, interferendo con le vie di mantenimento genomico. Questo studio introduce un metodo basato sulla microscopia a fluorescenza in tempo reale per osservare le interazioni della DNA polimerasi con le strutture G-quadruplex. Oligonucleotidi di DNA corti e innescati contenenti un G-quadruplex sono stati immobilizzati su vetrini di vetro funzionalizzati all'interno di una cella a flusso microfluidico. Sono state introdotte DNA polimerasi marcate in fluorescenza, che hanno permesso di monitorarne il comportamento e la stechiometria nel tempo. Questo approccio ha permesso l'osservazione del comportamento della polimerasi in quanto è stato bloccato da un G-quadruplex. In particolare, utilizzando δ di lievito polimerasi marcata in fluorescenza, si è scoperto che dopo aver incontrato un G-quadruplex, la polimerasi subisce un ciclo continuo di legame e slegatura. Questo saggio a singola molecola può essere adattato per studiare le interazioni tra varie proteine di mantenimento del DNA e gli ostacoli sul substrato del DNA.
Introduzione
Le DNA polimerasi sono enzimi che catalizzano l'incorporazione di nucleosidi trifosfati per duplicare il DNA 1,2,3,4,5. In quanto tali, svolgono un ruolo chiave nei processi essenziali di mantenimento del DNA, tra cui la replicazione del DNA 6,7,8 e la riparazione 9,10,11,12. Le DNA polimerasi devono replicare il genoma in modo accurato ed efficiente per garantire l'integrità genomica, prevenendo l'accumulo di mutazioni nel genoma. Durante la sintesi, le polimerasi incontrano spesso "ostacoli" come le proteine legate al DNA o le strutture secondarie del DNA13. Questi ostacoli possono rallentare o addirittura bloccare la progressione della polimerasi14. Superare questi ostacoli è importante per garantire una duplicazione fedele del genoma, poiché in caso contrario può portare a instabilità genomica15,16.
Una delle principali classi di ostacoli è rappresentata dai G-quadruplex (G4), strutture secondarie non canoniche del DNA che hanno dimostrato di formarsi in sequenze ricche di guanina all'interno del genoma umano17. Ci sono oltre 700.000 diverse sequenze nel genoma umano in grado di formare un G4, comprese le regioni all'interno dei telomeri e i promotori dell'oncogene18. Queste strutture di DNA adottano varie conformazioni a seconda della sequenza nucleotidica, della lunghezza e del catione metallico legato 19,20,21. Questa diversità significa che le polimerasi devono superare una gamma di diverse topologie G4, potenzialmente con vari gradi di efficienza. È stato dimostrato che l'incapacità delle polimerasi di superare o bypassare una struttura G4 impedisce la progressione della forcella di replicazione in vivo, portando all'instabilità genomica22. Studi in vitro hanno dimostrato che le strutture G4 possono bloccare o bloccare completamente le polimerasi di lievito 23,24,25,26,27. La capacità delle strutture G4 di bloccare o bloccare le DNA polimerasi dipende interamente dalla loro stabilità cinetica e termodinamica, con alcune polimerasi in grado di dispiegare alcuniG4 28. Sebbene questi studi forniscano informazioni sulla capacità delle polimerasi di superare gli ostacoli del G-quadruplex, non hanno la capacità di visualizzare direttamente il comportamento della polimerasi all'incontro con un G4. Il destino delle polimerasi - se rimangono legate, cadono o si scambiano dinamicamente - determina quali processi a valle sono accessibili per risolvere il G4.
In questo studio, è stato sviluppato un saggio di microscopia a singola molecola basato sulla fluorescenza per visualizzare e monitorare direttamente il legame della DNA polimerasi con le strutture G4 in tempo reale. Questo test prevede il legare i modelli di DNA che formano G4 a un vetrino coprioggetti biotinilato in una cella a flusso microfluidico, dove possono essere introdotte DNA polimerasi marcate in fluorescenza per avviare la sintesi del DNA. Misurando la fluorescenza delle polimerasi nel tempo, il loro comportamento all'incontro con una struttura G4 può essere osservato direttamente. La struttura G4 trovata nell'oncogene tumorale c-MYC è stata scelta per questo test per il suo alto livello di stabilità. Questo protocollo può ora essere adattato per catalogare il comportamento di una varietà di polimerasi in tutti i domini della vita associati a diverse topologie e stabilità G4. Questo test offre un approccio innovativo e ad alto rendimento per chiarire i meccanismi con cui le DNA polimerasi superano gli ostacoli del DNA, fornendo un potente strumento per far progredire la comprensione della dinamica della polimerasi.
Protocollo
I dettagli dei reagenti e delle attrezzature utilizzate sono elencati nella Tabella dei Materiali.
1. Spettroscopia di dicroismo circolare
NOTA: Prima di sviluppare il test, è stato necessario condurre una spettroscopia di dicroismo circolare (CD) sulla sequenza G4 selezionata per garantire il corretto ripiegamento. La sequenza di 22 nt (5′-TGAGGGTGGGGAGGGGGGGAA-3′) forma la struttura G4 derivata dall'oncogene del cancro c-MYC . La spettroscopia CD è stata eseguita anche su una sequenza di controllo (5′- TGAGTGTGAAGACGATGTAGAA -3) che ha nucleotidi chiave alterati che impediscono la formazione di G4.
- Per preparare i modelli di DNA per la spettroscopia CD, aggiungere 40 μL di 100 μM dei 22 modelli nt a 360 μL di Tris-HCl (pH 8,0), tampone acido etilendiamminotetraacetico (EDTA) (TE) integrato con 200 mM di cloruro di potassio (KCl). Per gli esperimenti di spettroscopia CD è necessario un 10 μM di concentrazione finale di DNA.
NOTA: La presenza di ioni K+ è essenziale per stabilizzare la struttura G4 nel modello. - Riscaldare le soluzioni di DNA a 95 °C in un bagno secco digitale per 15 minuti. Una volta completate, raffreddare lentamente le soluzioni durante la notte posizionando il blocco riscaldante nel polistirolo. Questo lento processo di raffreddamento è responsabile della piegatura del G4.
- Trasferire 400 μl delle soluzioni di formazione G4 e della sequenza di controllo in una cuvetta di quarzo lunga 0,1 cm utilizzando una siringa a tenuta di gas. Riempire lentamente la cuvetta per evitare la formazione di bolle d'aria.
- Misurare lo spettro CD tra 200 nm e 400 nm utilizzando uno spettropolarimetro a 25 °C. I parametri e le condizioni per questa misurazione sono stati descritti in precedenza29. I risultati sono riportati nella Figura 1 supplementare.
NOTA: Assicurarsi di registrare lo spettro del CD in modo che il buffer da solo funga da vuoto.
2. Preparazione di modelli di DNA
NOTA: I modelli che possono essere replicati sono modelli lineari corti da 100 nt preparati utilizzando tecniche standard di biologia molecolare. Il modello di formazione G4 (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGTGGGAGGGGT
GGGACCTTAGCTTCGAGTTCCGAT-3′) contiene al suo centro la sequenza in grado di formare un G-quadruplex derivato da MYC (dal passaggio 1). Il modello di controllo (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3′) ha la stessa regione di controllo (dal passaggio 1) anche nel suo centro.
- Per ricuocere il primer nei modelli di DNA di formazione G4 e di controllo, miscelare 20 μM del primer (5′-Biotin-AF647-TCTTAATGTAAATTTAAAATGTC-3′) a 20 μM dei modelli da 100 nt in tampone TE integrato con 200 mM KCl.
- Riscaldare le soluzioni del DNA a 95 °C in un bagno secco digitale per 15 minuti. Una volta completate, raffreddare lentamente le soluzioni durante la notte posizionando il blocco riscaldante nel polistirolo per consentire la piegatura del G4 e la ricottura dei due fili.
3. Marcatura della DNA polimerasi con AF647
- Marcatura della polimerasi con il colorante
NOTA: Il lievito Pol δ è stato estratto e purificato come precedentemente descritto30. La polimerasi doveva quindi essere marcata con il colorante monoreattivo estere Alexa Fluor NHS (AF647) per consentire la visualizzazione a livello di singola molecola. Questo è stato completato come descritto in precedenza5.- Per marcare l'N-terminale, incubare un eccesso di 3 volte del colorante AF647 con 320 μL di 58,5 μM di Pol δ in un tampone contenente 30 mM di Tris-HCl (pH 7,6), 2 mM di ditiotreitolo (DTT), 300 mM di cloruro di sodio (NaCl), 50 mM di glutammato di potassio e glicerolo al 10% (v/v) per 10 minuti a 4 °C. Ruotare delicatamente la soluzione per garantire una miscelazione accurata e migliorare l'efficienza dell'etichettatura.
NOTA: La sequenza proteica può contenere ulteriori ammine primarie negli amminoacidi della lisina. Regolando le condizioni di reazione, come il pH, l'etichetta può essere attaccata preferenzialmente all'N-terminale rispetto ai residui di lisina. Ciò potrebbe richiedere un'ulteriore ottimizzazione per aumentare l'efficienza dell'etichettatura. - Per preparare le colonne di desalinizzazione centrifughe da 0,5 mL (taglio del peso molecolare di 7 K), centrifugare prima le colonne a 1500 x g per 1 minuto per rimuovere la soluzione di conservazione. Eliminare le soluzioni depositate nella provetta di raccolta dopo la centrifugazione.
NOTA: Assicurarsi di allentare il tappo delle colonne di centrifugazione durante le fasi di centrifugazione. Ciò garantirà che le colonne siano eluite correttamente. - Equilibrare le colonne in un tampone contenente 30 mM di Tris-HCl (pH 7,6), 300 mM di NaCl, 3 mM di DTT, 50 mM di glutammato di potassio e glicerolo al 5% (v/v) aggiungendo 300 μL di tampone alla sommità della colonna. Centrifugare la colonna a 1500 x g per 1 minuto ed eliminare il flusso. Ripeti questo processo altre due volte.
- Applicare 200 μl di ciascun campione di Pol δ sulla colonna. Centrifugare le colonne a 1500 x g per 2 minuti e raccogliere il Pol δ purificato in una provetta da microcentrifuga da 1,5 mL.
- Congelare le frazioni purificate nel liquido N2 e conservare le aliquote a -80 °C per evitare la degradazione delle proteine nel tempo.
- Per marcare l'N-terminale, incubare un eccesso di 3 volte del colorante AF647 con 320 μL di 58,5 μM di Pol δ in un tampone contenente 30 mM di Tris-HCl (pH 7,6), 2 mM di ditiotreitolo (DTT), 300 mM di cloruro di sodio (NaCl), 50 mM di glutammato di potassio e glicerolo al 10% (v/v) per 10 minuti a 4 °C. Ruotare delicatamente la soluzione per garantire una miscelazione accurata e migliorare l'efficienza dell'etichettatura.
- Determinazione dell'efficienza dell'etichettatura
- Caricare 2 μl del campione di Pol δ, ora marcato con AF647, su uno spettrofotometro UV-vis31.
NOTA: Assicurarsi che venga misurato prima uno spazio vuoto del tampone Pol δ. - Misurare le lunghezze d'onda di eccitazione di 650 nm per il colorante AF647 e di 280 nm per il Pol δ utilizzando lo spettrofotometro.
- Utilizzando la legge di Beer-Lambert e i coefficienti di estinzione molare di 270.000 cm-1 M-1 per il colorante AF647 e 195.960 cm-1 M-1 per la polimerasi, calcolare l'efficienza di marcatura.
NOTA: L'efficienza di marcatura determinata per il lievito marcato AF647 Pol δ utilizzato in questo test è stata calcolata in circa il 67%.
- Caricare 2 μl del campione di Pol δ, ora marcato con AF647, su uno spettrofotometro UV-vis31.
4. Saggio di estensione del primer d'insieme
NOTA: Prima di eseguire esperimenti di replicazione di una singola molecola, è necessario confermare che la DNA polimerasi è bloccata dal G-quadruplex tramite saggi di replicazione di massa.
- Impostare la temperatura di un blocco riscaldante all'interno di un bagno secco digitale a 30 °C. Ciò garantirà la massima attività del lievito Pol δ.
- Preparare "miscele master" per i modelli di formazione G4 e di controllo nel tampone di replicazione (25 mM di Tris-HCl (pH 7,6), 10 mM di acetato di magnesio, 50 mM di glutammato di potassio, 40 μg/mL di albumina sierica bovina (BSA), 0,1 mM di EDTA, 5 mM di DTT e 0,0025% (v/v) Tween-20). Integrare ogni miscela con 1 mM di DTT, 250 μM ciascuno di dTTP, dCTP, dATP, dGTP (dNTP) e 10 nM del rispettivo stampo di DNA (G4-forming o controllo). Posizionarli in un blocco riscaldante per assicurarsi che raggiungano i 30 °C.
- Temprare 12 μL di ciascuna miscela master con 12 μL di tampone di caricamento della formammide (80% (p/v) di formammide, 10 mM di EDTA) per fungere da controllo T = 0 min.
- Per avviare la sintesi del DNA, aggiungere il lievito Pol δ marcato con AF647 a ciascuna miscela master (concentrazione finale di 20 nM). Mescolare accuratamente per garantire che le DNA polimerasi replichino i modelli con la massima efficienza.
NOTA: Conservare il lievito Pol δ sul ghiaccio durante l'uso per preservare l'attività enzimatica. - Dopo 30 s, 60 s e 180 s, rimuovere 12 μl di ciascuna miscela master e spegnere con 12 μl di tampone di caricamento della formammide. Il rapporto 1:1 tra la miscela master e il tampone di caricamento della formammide assicura che la reazione di replicazione non possa continuare una volta temprata.
- Rimuovete le soluzioni ora temprate dal blocco riscaldante a 30 °C e posizionatele in un blocco riscaldante a 98 °C per denaturare il dsDNA in ssDNA. Trascorsi 10 minuti, portare l'intero blocco riscaldante nel serbatoio del gel per il caricamento delle soluzioni in un gel denaturante al 15% di tris-borato-EDTA (TBE)-urea poliacrilammide (PAGE)32.
NOTA: Il gel PAGE deve essere riscaldato prima del caricamento dei campioni eseguendo l'elettroforesi del gel a 180 V per 30 minuti in 1x tampone TBE. Una volta completato, sciacquare bene ogni pozzetto per rimuovere l'urea. - Caricare 15 μL di ciascun campione sul gel insieme a una scala contenente 0,02 μM di oligonucleotidi marcati con 20 nt, 60 nt e 100 nt in tampone di caricamento della formammide.
- Far funzionare il gel per 60 minuti a 180 V in 1x tampone TBE per garantire una separazione completa.
- Immagine del gel nel canale Cy5 di un imager biomolecolare. Ciò consentirà di misurare il filamento di DNA replicato contenente l'etichetta AF-647.
5. Microscopia a fluorescenza a singola molecola
- Funzionalizzazione vetrino coprioggetti
NOTA: Per attaccare i modelli di DNA al vetrino coprioggetti, è necessario prima funzionalizzarlo con aminosilano, seguito dal legare le molecole di PEG biotinilate. Questo processo riduce al minimo le interazioni aspecifiche tra il DNA e/o le proteine e la superficie.- Pulire i vetrini coprioggetti (24 x 24 mm) inserendoli nei barattoli di colorazione e versando l'etanolo. Sonicare i barattoli per 30 minuti prima di sciacquare i vetrini coprioggetti con acqua pura. Ripetere questo processo con 1 M di idrossido di potassio (KOH) prima di risciacquare nuovamente. Ripetere ancora una volta questi passaggi di pulizia.
- Sciacquare e riempire un nuovo barattolo con acetone e posizionare i vetrini coprioggetti all'interno. Disseminare il 3-amminopropiltrietossisilano nel barattolo per formare una soluzione al 2% v/v. Agitare il barattolo per 3 minuti prima di spegnere la reazione con un grande eccesso di acqua.
- Asciugare i vetrini coprioggetti con N2 e posizionarli singolarmente su una scatola umida piena d'acqua. In questo modo si eviterà che i vetrini coprioggetti si secchino.
- Preparare un estere 1:25 di Biotina-PEG-SVA:mPEG-SVA in 100 mM di NaHCO3 (pH 8,2). Agitare questa miscela per 20 s per garantire una miscelazione accurata.
- Pipettare 40 μl della soluzione PEG su un vetrino coprioggetti asciutto. Posizionare un altro vetrino coprioggetti sopra per "inserire" la soluzione tra i due vetrini. Questo funzionalizzerà le facce del vetrino coprioggetti all'interno. Ripetere l'operazione per tutti i vetrini.
- Incubare i vetrini al buio per 3 ore. Dopo l'incubazione, separare le coppie di vetrini, sciacquarle con acqua in eccesso e asciugarle con gas N2 compresso.
- Ripetere i passaggi 4.1.4-4.1.5 per applicare un secondo strato di PEG sui vetrini. Assicurati di unire i lati precedentemente funzionalizzati, quindi fai attenzione a non capovolgerli accidentalmente durante le fasi di lavaggio. Incubare le soluzioni al buio per una notte prima di risciacquarle e asciugarle.
- Conservare i vetrini coprioggetti sottovuoto per preservarne la funzionalità. I vetrini coprioggetti funzionalizzati sono stabili per un mese se conservati correttamente.
- Preparazione di celle a flusso microfluidiche
NOTA: Una cella a flusso microfluidica33 è costruita per esperimenti su singole molecole (vedi Figura 2 supplementare). Ciò consentirà ai tamponi, ai modelli di DNA e alle proteine di entrare in contatto con un vetrino coprioggetti funzionalizzato (vedere il passaggio 5.1).- Rimuovere un vetrino coprioggetti funzionalizzato da un aspirapolvere e posizionarlo su un rack per microprovette parzialmente riempito d'acqua (scatola umida). Miscelare 100 μL di tampone bloccante (50 mM di Tris-HCl (pH 7,6), 50 mM di KCl, 2% (v/v) Tween-20) con 25 μL di soluzione di NeutrAvidin 1 mg/mL (10% PBS) e stenderlo sulla superficie del vetrino coprioggetti. Lasciate incubare per 15 minuti a temperatura ambiente nella scatola umida.
- Lavare il vetrino coprioggetti in acqua e asciugarlo in N2. Ricorda che solo un lato del vetrino è funzionalizzato, quindi ricorda di quale lato si tratta.
- Posizionare il blocco34 di polidimetilsilossano (PDMS) su misura sul vetrino coprioggetti all'interno del supporto della cella di flusso. In questo modo si otterrà un canale di flusso alto 100 μm e largo 1 mm. I tubi in polietilene (PE-60: diametro di ingresso di 0,76 mm e diametro esterno di 1,22 mm) possono quindi essere inseriti nei fori della cella di flusso per fornire l'accesso a tamponi e substrati.
- Imaging a fluorescenza a singola molecola
NOTA: Questi esperimenti devono essere eseguiti sia con i modelli di formazione G4 che con quelli di controllo. Ciò consentirà di visualizzare entrambe le condizioni (blocco della δ Pol da parte di una struttura G4 e nessun blocco) a livello di singola molecola utilizzando la microscopia a riflessione interna totale (TIRF).- Riscaldare 1 mL di aliquote di un tampone bloccante Tween (50 mM di Tris-HCl (pH 7,6), 50 mM di KCl, 2% (v/v) Tween-20) e 500 μL di aliquote di un tampone di lavaggio (25 mM di Tris-HCl (pH 7,6), 10 mM di acetato di magnesio, 250 mM di glutammato di potassio, 40 μg/mL di BSA, 0,1 mM di EDTA, 5 mM di DTT, 0,0025% (v/v) Tween-20) e tampone di replicazione a 40 °C per 15 min. In questo modo i gas saranno liberati dalle soluzioni. Degassare le soluzioni in una camera a vuoto per altri 15 minuti a 800 mbar al di sotto della pressione atmosferica.
- Prendere la cella di flusso costruita (vedere il passaggio 4.2) e posizionarla sul tavolino del microscopio. Dopo aver posizionato una goccia d'olio sull'obiettivo, sollevare l'obiettivo per incontrare il vetrino.
NOTA: Assicurarsi che l'obiettivo e la cella di flusso abbiano il tempo di raggiungere i 31 °C. Ciò aumenterà l'attività del lievito Pol δ e contribuirà a garantire dati riproducibili. - Inserire il tubo di ingresso nel tampone di blocco Tween degassato e collegare l'uscita alla pompa a siringa. Tirare indietro la siringa per aspirare il tampone di blocco Tween attraverso il tubo nel canale. Lasciare incubare questo tampone per almeno 30 minuti per ridurre al minimo le interazioni non specifiche.
- Fluire in 200 μl di tampone di lavaggio degassato nel canale a una velocità di 100 μl/min. In questo modo il buffer di blocco dell'interpolazione verrà eliminato.
- Diluire le soluzioni del DNA stampo a 0,5 pM in 500 μL di tampone di replicazione. Flusso di 150 μl nel canale a una velocità di 10 μl/min. Illuminare il campione utilizzando un laser da 647 nm a circa 900 mWcm-2 sul piano del campione per visualizzare i singoli modelli di DNA.
NOTA: Se il campo visivo (FOV) con il campione non è completamente a fuoco, il vetrino coprioggetto potrebbe non essere posizionato a filo contro il fondo della cella di flusso costruita. Costruire una nuova cella a flusso utilizzando un nuovo vetrino coprioggetti, se ciò si verifica. - Una volta che è visibile una densità sufficiente di macchie (circa 1 punto per 10 μm2), far fluire una nuova soluzione di tampone di replicazione (integrata con 1 mM di DTT) nel canale per eliminare il DNA in eccesso. Un volume di 250 μl è sufficiente per rimuovere completamente tutto il DNA non legato.
- Passa a un nuovo FOV e acquisisci un'immagine del DNA per determinare il grado di colocalizzazione tra la polimerasi marcata e il substrato del DNA. Una volta completato, aumentare la potenza del laser per sbiancare le macchie rimanenti.
- Preparare una soluzione di polimerasi contenente 1 mM di DTT, 250 μM di dNTP e 20 nM di Pol δ marcato con AF647 in 200 μL di tampone di replicazione. Flusso in 100 μl della soluzione di polimerasi ad una velocità di 5 μl/min nel canale.
- Una volta che il campione è a fuoco e l'angolo TIRF è stato regolato, impostare la potenza del laser a 647 nm su circa 900 mWcm-2 sul piano del campione. Quindi, inizia a visualizzare il FOV per il periodo di tempo desiderato. Acquisisci tra 1 e 5 fotogrammi/s per 10-20 minuti per acquisire tutti gli eventi di replica.
- Analisi dei dati
NOTA: Tutte le analisi sono state effettuate utilizzando Python (v. 3.11.7). Il codice personalizzato utilizzato per l'analisi dei dati è disponibile qui: https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis.- Innanzitutto, colocalizzare le macchie del DNA stampo con le macchie fluorescenti del lievito marcato Pol δ. Due picchi sono stati determinati come colocalizzati se la loro distanza dopo la correzione della deriva era ≤ 3 pixel. Solo i punti di Pol δ che colocalizzano i modelli di DNA vengono analizzati per la loro cinetica di legame e le tendenze comportamentali generali.
- Per determinare i dati dell'evento di binding, misurare l'intensità nel tempo di ogni punto in quanto mostrano un comportamento "on/off" durante il filmato. Ciò consente di determinare quante volte il legame del δ Pol si verifica su un singolo modello di DNA.
NOTA: Questa analisi viene eseguita sia per gli esperimenti di formazione G4 che per quelli con il modello di controllo per confrontare le dinamiche di legame.
NOTA: Sono stati condotti esperimenti di controllo, che assicurano che gli eventi di legame non specifici al G-quadruplex o al modello stesso (23,2 s ± 4,9 s) non siano inclusi nell'analisi degli eventi di legame.
Risultati
Per questo saggio, sono stati progettati due substrati di DNA con un primer da 20 nt marcato in fluorescenza: uno contenente una sequenza di formazione G4 (Figura 1A, a sinistra) e uno privo di questa sequenza (Figura 1A, a destra). Per confermare che il G-quadruplex è un efficace ostacolo all'attività della polimerasi, la sintesi del DNA da parte di Pol δ è stata monitorata su un gel denaturante PAGE. L'attività del δ di Pol marcato purificato sui substrati di DNA è stata esaminata mediante elettroforesi su gel. La Figura 1B (a sinistra) mostra che il Pol marcato in fluorescenza δ non è in grado di sintetizzare oltre il G-quadruplex. Prima dell'inizio della sintesi (t = 0 min), è presente una banda corrispondente a 20 nt, che rappresenta il primer marcato che è stato denaturato dal filamento stampo. Dopo 3 minuti, questa banda di 20 nt è stata convertita in una banda di 60 nt, indicando che la sintesi è avvenuta su tutto il DNA e confermando che la polimerasi era completamente bloccata dalla struttura G-quadruplex. Questo blocco implica che la polimerasi non potrebbe né dispiegarsi né bypassare la struttura. Al contrario, la sintesi di un modello di controllo che non contiene la sequenza di formazione G-quadruplex (Figura 1A, a destra) ha prodotto una banda di 100 nt dopo 3 minuti (Figura 1B, a destra).
Dopo aver confermato la sintesi sui modelli e il blocco efficiente da parte del G-quadruplex, queste misurazioni sono state ripetute su un microscopio a fluorescenza a singola molecola per monitorare il comportamento della polimerasi. I modelli di DNA sono stati legati a vetrini coprioggetti funzionalizzati (Figura 2A) in una cella a flusso microfluidico e la posizione di ciascun substrato è stata determinata visualizzando il primer marcato in fluorescenza. Quindi, il Pol δ marcato è stato caricato in presenza di dNTP per avviare la sintesi. All'interno di un FOV tipico, i singoli punti vengono tracciati per quantificare la frequenza con cui Pol δ si lega e si dissocia dal DNA (Figura 2B, C). Misurando l'intensità in funzione del tempo su ciascun substrato di DNA, è possibile generare traiettorie di singole molecole (Figura 2C, Figura 3 supplementare). È possibile misurare il caratteristico "tempo di permanenza", ovvero la durata media in cui Pol δ rimane legato al modello. Per il substrato G4, il tempo di permanenza è stato determinato in 6 s ± 2 s, mentre per il substrato di controllo è stato di 10 s ± 3 s (Figura 2D, Figura 4 supplementare). Inoltre, per ogni traiettoria, è possibile quantificare il numero di volte in cui Pol δ si lega al modello. Il numero di eventi di legame al substrato G4 è molto più elevato rispetto al modello di controllo (Figura 2E). Mentre ci sono casi di più di un evento di legame nel modello di controllo a causa del legame e dello svincolo scolastico, c'è un chiaro aumento del numero di eventi di legame in media per il modello di formazione G4. Ciò suggerisce che, dopo che la sintesi è stata interrotta dal G-quadruplex, la polimerasi legata si dissocia dal DNA prima che le nuove polimerasi dalla soluzione inizino un ciclo continuo di legame e dislegamento. Pertanto, questo saggio a singola molecola fornisce una visione senza precedenti di come le DNA polimerasi rispondono agli ostacoli del DNA.

Figura 1: Saggio di estensione del primer d'insieme. (A) Rappresentazione schematica dei substrati del DNA. Il substrato G4 (a sinistra) contiene una sequenza G-quadruplex-forming, mentre il substrato di controllo (a destra) no. (B) Saggio di estensione della primasi del DNA d'insieme del G4 innescato e dei substrati del DNA di controllo che mostrano che il lievito Pol δ marcato in fluorescenza è completamente bloccato dal G-quadruplex. Il gel PAGE mostra la replicazione dei modelli di DNA nel tempo, determinando uno spostamento del primer da 20 nt marcato nel prodotto da 60 nt per il substrato G4 (a sinistra) e dal prodotto da 100 nt per il modello di controllo (a destra). M rappresenta una scala contenente oligo marcati 20 nt, 60 nt e 100 nt. Clicca qui per visualizzare una versione più grande di questa figura.
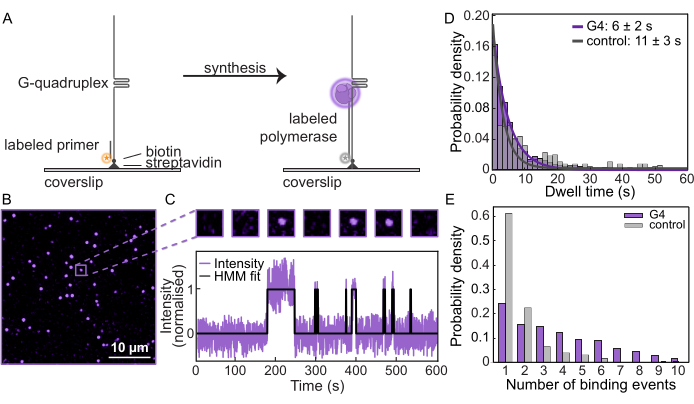
Figura 2: Microscopia a fluorescenza a singola molecola. (A) Rappresentazione schematica del saggio di estensione del primer a singola molecola utilizzando il Pol δ di lievito marcato con AF647. (B) FOV tipico ottenuto da un saggio a singola molecola. Ogni punto rappresenta un δ Pol marcato in fluorescenza che si lega a un modello di DNA. Barra della scala: 10 μm. (C) (In alto) Esempio di molecola del saggio a singola molecola che mostra il legame on/off durante gli scambi di Pol δ. (In basso) Traiettoria di una singola molecola della stessa molecola che mostra l'intensità nel corso del tempo dell'esperimento. La linea nera rappresenta l'adattamento del modello di Markov nascosto (HMM) ai dati. (D) Tempo di permanenza di Pol δ sul substrato G4 (viola) e sul substrato di controllo (grigio). Le linee rappresentano adattamenti esponenziali, con una durata di 6 s ± 2 s sul substrato G4 e di 10 s ± 3 s sul substrato di controllo. (E) Numero di eventi di legame di Pol δ a singoli substrati di DNA. Il numero mediano di eventi di legame sul substrato G4 (viola) è 3,5, rispetto al substrato di controllo 1 (grigio). Clicca qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1: Spettroscopia CD. Spettri CD della sequenza di formazione G4 (viola) e di controllo (grigio) in tampone TE contenente 200 mM di KCl a 25 °C. Il picco caratteristico a 260 nm, seguito dal picco negativo a 240 nm, è caratteristico di un GQ intramolecolare parallelo. Clicca qui per scaricare questo file.
Figura 2 supplementare: Costruzione di celle a flusso. (A) Schema dei singoli pezzi della cella a flusso. Il blocco PDMS quadrato si trova sopra il vetrino coprioggetti del microscopio. Questi pezzi vengono quindi inseriti insieme all'interno del coperchio e del fondo del supporto della cella di flusso. (B) La cella a flusso completa. I tubi possono essere inseriti in ciascun lato del blocco PDMS per formare i canali attraverso i quali i tamponi possono essere fatti fluire attraverso il dispositivo tramite microfluidica. Clicca qui per scaricare questo file.
Figura 3 supplementare: Traiettorie di singole molecole. (A) Esempio di traiettoria di una molecola sottoposta a un singolo evento di legame durante l'intervallo di tempo di 10 minuti dell'esperimento. (B) Esempio di traiettoria di una molecola che subisce zero eventi di legame durante l'intervallo di tempo di 10 minuti dell'esperimento. La linea nera in (A) e (B) rappresenta l'adattamento del modello di Markov nascosto (HMM) ai dati. Clicca qui per scaricare questo file.
Figura supplementare 4: Tempi di sosta. (A) Tempo di permanenza di Pol δ sul substrato G4. La linea rappresenta l'adattamento esponenziale, fornendo una durata di 6 s ± 2 s. (B) Tempo di permanenza di Pol δ sul substrato di controllo. L'adattamento esponenziale garantisce una durata di 10 s ± 3 s. Clicca qui per scaricare questo file.
Figura supplementare 5: Controllo del fotosbiancamento. Il grafico mostra il tempo medio per la fotosbiancamento di un singolo enzima Pol δ di lievito marcato con AF647 dal laser a 647 nm. La linea rappresenta l'adattamento esponenziale, dando una durata di 39 ± 6 s. Clicca qui per scaricare questo file.
Discussione
Qui, è stato descritto un saggio basato sulla fluorescenza a singola molecola che fornisce informazioni sul comportamento di una DNA polimerasi quando incontra un G-quadruplex. Mentre i protocolli di generazione del modello di DNA, la marcatura del δ Pol e i saggi di replicazione del DNA di massa sono tutti semplici, l'esecuzione di saggi di microscopia a singola molecola è tecnicamente più impegnativa. A causa della natura delle tecniche a singola molecola, è necessario prestare molta attenzione per evitare l'introduzione di polvere, contaminazione o bolle d'aria, poiché oscureranno il FOV e ostacoleranno la raccolta dei dati.
Un limite degli esperimenti di microscopia TIRF a singola molecola è il fotosbiancamento dei fluorofori che sono accoppiati covalentemente alle biomolecole di interesse. Il fotosbiancamento è un processo irreversibile che porta alla perdita permanente della fluorescenza35. Per mitigare questo problema durante gli esperimenti, è essenziale limitare la durata dell'esposizione al laser, regolare l'intensità del laser e ottimizzare i tempi di imaging. Queste strategie aiutano a preservare i segnali di fluorescenza, garantendo periodi di osservazione più affidabili e prolungati. Regolando con precisione questi parametri, il segnale Pol δ rimane per tutta la durata della misurazione. Per ottimizzare la potenza del laser per i saggi di sintesi al microscopio a fluorescenza a singola molecola, si consiglia di misurare il tasso di fotosbiancamento visualizzando la polimerasi marcata su un vetrino di vetro pulito. Variando sistematicamente la potenza del laser e valutando il tasso di fotosbiancamento in tutto il campo visivo, è possibile identificare l'intensità laser ottimale che bilancia l'intensità del segnale del fluoroforo con la resistenza al fotosbiancamento (vedere la Figura 5 supplementare).
Il vantaggio principale di questo approccio a singola molecola rispetto ai tradizionali metodi basati sull'ensemble è la sua capacità di visualizzare direttamente quando una singola DNA polimerasi incontra e interagisce con una struttura G4. I metodi tradizionali basati sull'ensemble (come l'elettroforesi su gel) hanno dimostrato la capacità delle strutture G4 di bloccare le DNA polimerasi 23,36,37. Queste tecniche, tuttavia, non riescono a fornire le informazioni cinetiche e meccanicistiche in tempo reale di questa interazione, che sono necessarie per districare i diversi passaggi e risultati cinetici molecolari. Le tecniche a singola molecola offrono una visione senza precedenti della cinetica, dei meccanismi e dei comportamenti delle biomolecole spesso nascoste da un insieme con una media di38. Ora è possibile vedere come le DNA polimerasi agiscono in tempo reale, sia che scambino, blocchino, dissocino o aggirino i blocchi stradalidel DNA 39. Con questo protocollo stabilito, l'identità del G4 può essere facilmente modificata dalla struttura c-MYC parallela scelta a qualsiasi topologia parallela, anti-parallela o ibrida. L'applicazione di questo saggio a singola molecola rivelerà se le stesse DNA polimerasi si comportano in modo diverso quando incontrano topologie G4 alternative. Pertanto, i metodi a singola molecola sono fondamentali per rispondere a domande specifiche su come interagiscono le proteine e il DNA del corpo.
Attraverso la visualizzazione diretta delle interazioni della DNA polimerasi con i G-quadruplex, è stata identificata una via di scambio precedentemente non caratterizzata per il δ Pol del lievito. Questa scoperta suggerisce che la polimerasi si disinnesta all'incontro con un G-quadruplex, in attesa dell'intervento di un'altra proteina per risolvere la struttura prima di ricominciare la sintesi del DNA. Questo protocollo può essere adattato per studiare le interazioni tra varie proteine di mantenimento del genoma e ostacoli al DNA, offrendo intuizioni senza precedenti su come gli enzimi cellulari navigano negli impedimenti genomici. Ad esempio, l'ostacolo di questo test può essere modificato da una struttura G4 a un legame incrociato proteina-DNA, un tipo di lesione del DNA in cui una proteina è irreversibilmente legata in modo covalente al DNA, fungendo da ostacolo per la replicazione del DNA40. Tali esami sono fondamentali per comprendere i processi fondamentali della replicazione, riparazione e ricombinazione del DNA. Consentendo lo studio della dinamica DNA-proteina a livello molecolare, questo saggio fornisce un potente strumento per chiarire i meccanismi alla base dell'integrità genomica.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
N.K.-A. riconosce il finanziamento fornito dalla borsa di studio del programma di formazione alla ricerca del governo australiano. L.M.S. è grata per il finanziamento che ha ricevuto dal National Health and Medical Research Council (Investigator Grant 2007778). J.S.L è grata di aver ricevuto il Discovery Early Career Award (DE240100780) e l'NHMRC Investigator EL1 (2025412) finanziati dal governo australiano. S.H.M è grata di essere la destinataria della borsa di studio Bruce Warren Molecular Horizons Ealy.
Materiali
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
Riferimenti
- Sutton, M. D., Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci. 98 (15), 8342-8349 (2001).
- Burgers Garg, P., J, P. M. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M. -D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O., et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Sugino Kawasaki, Y., A, Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Shivji Wood, R. D., K, M. Which DNA polymerases are used for DNA repair in eukaryotes. Carcinogenesis. 18 (4), 605-610 (1997).
- Campbell Budd, M. E., L, J. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Gao Yang, W., Y, Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9. 968424, (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Monnat Loeb, L. A., J, R. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Bryan Oganesian, L., M, T. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I., et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Clayton Kaguni, L. S., A, D. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), (2021).
- Castillo Bosch,, P,, et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, J. ohnA., Zakian, V. irginiaA. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L., et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh. 11 (9), (2024).
- Pham, S. Q. T., et al. A new class of quadruplex DNA-binding nickel Schiff base complexes). Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E., et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Hansen, J. B., Au, Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J. , Springer. New York, New York, NY. 219-238 (2015).
- Lewis, J. S., et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, 23932(2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. , 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664(2014).
- Mueller, S. H., et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5(2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon