Method Article
G-Quadruplexes에서 DNA 중합효소 역학의 단일 분자 형광 시각화
요약
이 프로토콜은 형광 현미경 기반 단일 분자 DNA 복제 분석의 개요를 설명하여 DNA 중합효소와 G-4중 구조와 같은 장애물 간의 상호 작용을 실시간으로 시각화할 수 있습니다.
초록
진핵생물 DNA 복제에 관여하는 단백질이 단백질 및 DNA '장애물'과 같은 장애물을 극복하는 능력은 충실한 게놈 복제를 보장하는 데 매우 중요합니다. G-quadruplexes는 구아닌이 풍부한 DNA 영역에서 형성되는 고차 핵산 구조이며 게놈 유지 경로를 방해하는 장애물로 작용하는 것으로 나타났습니다. 본 연구에서는 DNA 중합효소와 G-사중극자 구조의 상호작용을 관찰하기 위한 실시간 형광 현미경 기반 방법을 소개합니다. G-quadruplex를 포함하는 짧고 프라이밍된 DNA 올리고뉴클레오티드는 미세유체 플로우 셀 내의 기능화된 유리 커버슬립에 고정되었습니다. 형광 표지된 DNA 중합효소가 도입되어 시간이 지남에 따라 이들의 거동과 화학량론을 모니터링할 수 있게 되었습니다. 이 접근법은 G-quadruplex에 의해 지연된 중합효소 거동을 관찰할 수 있게 했습니다. 구체적으로, 형광 표지된 효모 중합효소 δ를 사용하여 G-quadruplex를 만나면 중합효소가 결합과 결합이 해제되는 연속적인 주기를 겪는 것으로 나타났습니다. 이 단일 분자 분석은 다양한 DNA 유지 단백질과 DNA 기질의 장애물 간의 상호 작용을 연구하는 데 적용할 수 있습니다.
서문
DNA 중합효소는 DNA 1,2,3,4,5를 복제하기 위해 뉴클레오시드 삼인산의 통합을 촉매하는 효소입니다. 따라서 그들은 DNA 복제 6,7,8 및 복구 9,10,11,12를 포함한 필수 DNA 유지 과정에서 중요한 역할을 합니다. DNA 중합효소는 게놈 무결성을 보장하기 위해 게놈을 정확하고 효율적으로 복제하여 게놈에 돌연변이가 축적되는 것을 방지해야 합니다. 합성하는 동안 중합효소는 종종 DNA 결합 단백질 또는 2차 DNA 구조와 같은 "장애물"에 부딪히게 됩니다13. 이러한 장애물은 중합효소 진행을 늦추거나 심지어 차단할 수 있다14. 이러한 장애물을 극복하는 것은 충실한 게놈 복제를 보장하는 데 중요한데, 그렇게 하지 않으면 게놈 불안정으로 이어질 수 있기 때문이다 15,16.
장애물의 주요 부류 중 하나는 G-quadruplexes (G4)로, 인간 게놈 내에서 guanine이 풍부한 서열로 형성되는 것으로 나타난 non-canonical secondary DNA 구조입니다17. 인간 게놈에는 텔로미어(telomeres)와 종양유전자 프로모터(oncogene promoter) 내의 영역을 포함하여 G4를 형성할 수 있는 700,000개 이상의 서로 다른 염기서열이 있습니다18. 이러한 DNA 구조는 뉴클레오티드 서열, 길이 및 결합 금속 양이온 19,20,21에 따라 다양한 형태를 채택합니다. 이러한 다양성은 중합효소가 잠재적으로 다양한 수준의 효율성으로 다양한 G4 토폴로지를 극복해야 함을 의미합니다. 중합효소가 G4 구조를 극복하거나 우회하지 못하면 생체 내에서 복제 분기점 진행을 방해하여 유전체 불안정성을 유발하는 것으로 나타났습니다22. 체외 연구는 G4 구조가 효모 중합효소를 지연시키거나 완전히 차단할 수 있음을 보여주었습니다 23,24,25,26,27. DNA 중합효소를 지연시키거나 차단하는 G4 구조의 능력은 전적으로 역학 및 열역학적 안정성에 의존하며, 일부 중합효소는 특정 G4를 펼칠 수 있습니다28. 이러한 연구는 G-quadruplex 장애물을 극복하는 중합효소의 능력에 대한 통찰력을 제공하지만, G4를 만났을 때 중합효소의 거동을 직접 시각화할 수 있는 능력은 부족합니다. 중합효소의 운명은 결합된 상태로 유지되는지, 떨어지거나 동적으로 교환되는지에 따라 G4를 해결하기 위해 어떤 다운스트림 프로세스에 접근할 수 있는지가 결정됩니다.
본 연구에서는 DNA polymerraz와 G4 structure의 결합을 실시간으로 직접 시각화하고 모니터링하기 위해 형광 기반 단일분자 현미경 분석을 개발하였습니다. 이 분석은 G4 형성 DNA 템플릿을 미세유체 플로우 셀의 비오틴화 커버슬립에 테더링하는 것을 포함하며, 여기서 형광 표지된 DNA 중합효소를 도입하여 DNA 합성을 시작할 수 있습니다. 시간 경과에 따른 중합효소의 형광을 측정함으로써 G4 구조를 만났을 때의 거동을 직접 관찰할 수 있습니다. c-MYC 암 종양 유전자에서 발견된 G4 구조는 높은 수준의 안정성으로 인해 이 분석을 위해 선택되었습니다. 이 프로토콜은 이제 서로 다른 G4 토폴로지 및 안정성과 관련된 모든 생명 영역에서 다양한 중합효소의 거동을 목록화하도록 조정할 수 있습니다. 이 분석은 DNA 중합효소가 DNA 장애물을 탐색하는 메커니즘을 규명하기 위한 혁신적이고 처리량이 많은 접근 방식을 제공하여 중합효소 역학에 대한 이해를 증진하기 위한 강력한 도구를 제공합니다.
프로토콜
시약 및 사용된 장비에 대한 자세한 내용은 재료 표에 나열되어 있습니다.
1. 원형 이색성 분광법
참고: 분석을 개발하기 전에 올바른 폴딩을 보장하기 위해 선택한 G4 염기서열에 대해 원이색성(CD) 분광법을 수행해야 했습니다. 22 nt 서열(5'-TGAGGGTGGGGGGGGGGGGAA-3')은 c-MYC 암 종양 유전자에서 유래한 G4 구조를 형성합니다. CD 분광법은 또한 G4 형성을 방지하는 주요 뉴클레오티드가 변경된 대조 서열(5'- TGAGTGTGAAGACGATGTAGAA -3)에 대해 수행되었습니다.
- CD 분광법을 위한 DNA 템플렛을 준비하기 위하여는, 22개의 nt 템플렛 100 μM 40 μL를 360 μL의 Tris-HCl (PH 8.0), 에틸렌디아민테트라아세트산(EDTA) (200 mM의 염화칼륨 (KCl)으로 보충된 에틸렌디아민테트라아세트산(EDTA) (TE) 완충액에 첨가합니다. CD 분광법 실험에는 10μM의 최종 DNA 농도가 필요합니다.
참고: K+ 이온의 존재는 템플릿에서 G4 구조를 안정화하는 데 필수적입니다. - DNA 용액을 디지털 건조 수조에서 95분 동안 15°C로 가열합니다. 완료되면 가열 블록을 폴리스티렌에 넣어 용액을 밤새 천천히 냉각시킵니다. 이 느린 냉각 과정은 G4의 접힘을 담당합니다.
- 기밀 주사기를 사용하여 400μL의 G4 형성 및 제어 시퀀스 용액을 0.1cm 경로 길이의 석영 큐벳으로 옮깁니다. 기포가 형성되지 않도록 큐벳을 천천히 채우십시오.
- 25°C에서 분광 편광계를 사용하여 200nm와 400nm 사이의 CD 스펙트럼을 측정합니다. 이 측정을 위한 파라미터 및 조건은 앞서설명되었다(29). 결과는 보충 그림 1에서 확인할 수 있습니다.
참고: 버퍼만 블랭크 역할을 할 수 있도록 CD 스펙트럼을 기록해야 합니다.
2. DNA 템플릿 준비
참고: 복제할 수 있는 템플릿은 표준 분자 생물학 기술을 사용하여 준비된 짧은 100nt 프라이밍 선형 템플릿입니다. G4 형성 템플릿(5'-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGTGGGGG
GGGACCTTAGCTTCGAGTTCCGAT-3')는 중심에 MYC 유래 G-4중(1단계에서)을 형성할 수 있는 서열을 포함합니다. 컨트롤 템플릿(5'-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3')의 중심에도 동일한 제어 영역(1단계)이 있습니다.
- 프라이머를 G4 형성 및 제어 DNA 템플릿에 어닐링하려면 20μM의 프라이머(5'-Biotin-AF647-TCTTAATGTAAATTTAAAATGTC-3')를 200mM KCl이 보충된 TE 완충액의 100nt 템플릿 20μM에 혼합합니다.
- DNA 템플릿 용액을 디지털 건조 수조에서 95°C로 15분 동안 가열합니다. 완료되면 가열 블록을 폴리스티렌에 넣어 G4를 접고 두 가닥을 어닐링할 수 있도록 하여 용액을 밤새 천천히 냉각합니다.
3. AF647을 사용한 DNA 중합효소 라벨링
- 염료로 중합효소 라벨링
참고: 효모 Pol δ는 앞서 기술한 바와 같이 발현되고 정제되었다30. 그런 다음 단일 분자 수준에서 시각화할 수 있도록 Alexa Fluor NHS 에스테르 단일 반응성 염료(AF647)로 중합효소를 라벨링해야 했습니다. 이것은 앞서 설명한대로 완료되었습니다 5.- N-terminus를 라벨링하려면 30mM의 Tris-HCl(pH 7.6), 2mM의 디티오트레이톨(DTT), 300mM의 염화나트륨(NaCl), 50mM의 글루타민산칼륨 및 10%(v/v) 글리세롤을 포함하는 완충액에 320μL의 58.5μM Pol δ와 함께 AF647 염료의 3배 초과량을 4°C에서 10분 동안 배양합니다. 용액을 부드럽게 회전시켜 철저한 혼합을 보장하고 라벨링 효율성을 개선합니다.
참고: 단백질 서열은 라이신 아미노산에 추가 1차 아민을 포함할 수 있습니다. pH와 같은 반응 조건을 조정함으로써, 라벨은 라이신 잔기와 반대로 N-말단에 우선적으로 부착될 수 있습니다. 이를 위해서는 라벨링 효율성을 높이기 위해 추가 최적화가 필요할 수 있습니다. - 0.5mL 스핀 탈염 컬럼(7K 분자량 절단)을 준비하려면 먼저 1500 x g 에서 컬럼을 1분 동안 원심분리하여 저장 용액을 제거합니다. 원심분리 후 수집 튜브에 침전된 용액을 폐기하십시오.
알림: 원심분리 단계에서 스핀 컬럼의 캡을 풀어야 합니다. 이렇게 하면 컬럼이 적절하게 용리됩니다. - 300μL의 완충액을 컬럼 상단에 추가하여 30mM의 Tris-HCl(pH 7.6), 300mM의 NaCl, 3mM의 DTT, 50mM의 글루타메이트칼륨 및 5%(v/v) 글리세롤이 포함된 완충액에서 컬럼을 평형화합니다. 1500 x g 에서 컬럼을 1분 동안 원심분리하고 플로우스루(flow-through)를 버립니다. 이 과정을 두 번 더 반복합니다.
- 각 Pol δ 샘플 200μL를 컬럼에 적용합니다. 1500 x g 에서 2분 동안 컬럼을 원심분리하고 정제된 Pol δ를 1.5mL 마이크로 원심분리 튜브에 수집합니다.
- 정제된 분획을 액체 N2 에 동결하고 부분 표본을 -80 °C에 보관하여 시간이 지남에 따라 단백질 분해를 방지합니다.
- N-terminus를 라벨링하려면 30mM의 Tris-HCl(pH 7.6), 2mM의 디티오트레이톨(DTT), 300mM의 염화나트륨(NaCl), 50mM의 글루타민산칼륨 및 10%(v/v) 글리세롤을 포함하는 완충액에 320μL의 58.5μM Pol δ와 함께 AF647 염료의 3배 초과량을 4°C에서 10분 동안 배양합니다. 용액을 부드럽게 회전시켜 철저한 혼합을 보장하고 라벨링 효율성을 개선합니다.
- 라벨링 효율성 결정
- 현재 AF647 표지된 Pol δ 샘플 2μL를 UV-vis 분광 광도계31에 로드합니다.
알림: Pol δ 버퍼의 블랭크가 먼저 측정되었는지 확인하십시오. - 분광 광도계를 사용하여 AF647 염료의 경우 650nm, Pol δ의 경우 280nm의 여기 파장을 측정합니다.
- Beer-Lambert 법칙과 AF647 염료의 경우 270,000cm-1 M-1, 중합효소의 경우 195,960cm-1 M-1의 몰 흡광 계수를 사용하여 라벨링 효율을 계산합니다.
참고: 이 분석에 사용된 AF647 표지 효모 Pol δ에 대해 측정된 라벨링 효율은 약 67%로 계산되었습니다.
- 현재 AF647 표지된 Pol δ 샘플 2μL를 UV-vis 분광 광도계31에 로드합니다.
4. 앙상블 프라이머 확장 분석
참고: 단일 분자 복제 실험을 수행하기 전에 DNA 중합효소가 벌크 복제 분석을 통해 G-quadruplex에 의해 차단되었는지 확인해야 합니다.
- 디지털 건식조 내 가열 블록의 온도를 30°C로 설정합니다. 이것은 효모 Pol δ의 최대 활성을 보장합니다.
- 복제 완충액(Tris-HCl(pH 7.6) 25mM, 마그네슘 아세테이트 10mM, 글루타민산칼륨 50mM, 소 혈청 알부민(BSA) 40μg/mL, EDTA 0.1mM, DTT 5mM 및 0.0025%(v/v) Tween-20)에서 G4 형성 및 제어 템플릿을 위한 "마스터 믹스"를 준비합니다. 각 혼합물에 1mM의 DTT, 각각 250μM의 dTTP, dCTP, dATP, dGTP(dNTP) 및 10nM의 각 DNA 템플릿(G4 형성 또는 대조군)을 보충합니다. 30°C에 도달하도록 가열 블록에 놓습니다.
- 각 마스터 믹스 12μL를 12μL의 포름아미드 로딩 버퍼(80%(w/v) 포름아미드, 10mM의 EDTA)와 함께 T = 0분 대조군으로 소멸합니다.
- DNA 합성을 시작하려면 AF647 표지된 효모 Pol δ를 각 마스터 믹스(20nM 최종 농도)에 첨가합니다. DNA 중합효소가 최대 효율로 주형을 복제할 수 있도록 철저히 혼합합니다.
알림: 효소 활성을 보존하기 위해 사용하는 동안 효모 Pol δ를 얼음에 보관하십시오. - 30초, 60초 및 180초 후 각 마스터 믹스 12μL를 제거하고 12μL의 포름아미드 로딩 버퍼로 퀀칭합니다. 포름아미드 로딩 버퍼에 대한 마스터 믹스의 1:1 비율은 담금질된 후 복제 반응이 계속될 수 없도록 합니다.
- 30°C 가열 블록에서 이제 담금질된 용액을 제거하고 dsDNA를 ssDNA로 변성시키는 대신 98°C 가열 블록에 넣습니다. 10분이 경과하면 전체 가열 블록을 젤 탱크로 가져와 용액을 변성 15% 트리스-붕산염-EDTA(TBE)-우레아 폴리아크릴아미드(PAGE) 젤32에 로드합니다.
참고: PAGE 젤은 1x TBE 버퍼에서 180V에서 30분 동안 겔 전기영동을 실행하여 샘플을 로드하기 전에 예열해야 합니다. 완료되면 각 웰을 헹구어 요소를 제거합니다. - 포름아미드 로딩 버퍼에서 0.02μM의 20nt, 60nt 및 100nt 표지된 올리고뉴클레오티드가 포함된 래더를 따라 각 샘플의 15μL를 겔에 로드합니다.
- 완전한 분리를 위해 1x TBE 버퍼에서 180V로 60분 동안 젤을 실행합니다.
- 생체 분자 이미저의 Cy5 채널에 있는 겔을 이미지화합니다. 이를 통해 AF-647 라벨을 포함하는 복제된 DNA 가닥을 측정할 수 있습니다.
5. 단 하나 분자 형광 현미경 검사법 검사법
- 커버슬립 기능화
참고: DNA 템플릿을 유리 커버슬립에 부착하려면 먼저 아미노실란으로 기능화한 다음 비오틴화된 PEG 분자를 테더링해야 합니다. 이 과정은 DNA 및/또는 단백질과 표면 사이의 비특이적 상호 작용을 최소화합니다.- 커버 슬립 (24 x 24mm)을 염색 항아리에 넣고 에탄올을 부어 청소합니다. 커버슬립을 순수한 물로 헹구기 전에 항아리를 30분 동안 초음파 처리합니다. 다시 헹구기 전에 1M 수산화칼륨(KOH)으로 이 과정을 반복합니다. 이 청소 단계를 한 번 더 반복합니다.
- 새 병을 헹구고 아세톤을 채우고 커버 슬립을 안에 넣습니다. 3-아미노프로필트리에톡시실란을 항아리에 퍼뜨려 2% v/v 용액을 형성합니다. 많은 양의 물로 반응을 담금질하기 전에 항아리를 3분 동안 교반합니다.
- N2 로 커버슬립을 말리고 물이 채워진 습한 상자에 개별적으로 놓습니다. 이렇게 하면 커버슬립이 마르는 것을 방지할 수 있습니다.
- 100mM의 NaHCO3 (pH 8.2)에 1:25 비오틴-PEG-SVA:mPEG-SVA 에스테르를 준비합니다. 철저한 혼합을 보장하기 위해 이 혼합물을 20초 동안 소용돌이치십시오.
- PEG 용액 40μL를 드라이 커버슬립에 피펫팅합니다. 두 커버슬립 사이에 용액을 "샌드위치"하기 위해 다른 커버슬립을 위에 놓습니다. 이렇게 하면 내부에 있는 커버슬립의 면이 기능화됩니다. 모든 커버슬립에 대해 반복합니다.
- 커버슬립을 어둠 속에서 3시간 동안 배양합니다. 배양 후 커버 슬립 쌍을 분리하고 여분의 물로 헹구고 압축 된 N2 가스로 건조시킵니다.
- 4.1.4-4.1.5단계를 반복하여 커버슬립에 PEG의 두 번째 층을 적용합니다. 이전에 기능화된 면을 함께 끼워 넣어야 하므로 세탁 단계에서 실수로 뒤집히지 않도록 주의하십시오. 용액을 헹구고 건조하기 전에 밤새 어둠 속에서 배양하십시오.
- 커버슬립은 기능을 보존하기 위해 진공 청소기에 보관하십시오. 기능화된 커버슬립은 올바르게 보관하면 한 달 동안 안정적입니다.
- 미세유체 플로우 셀(microfluidic flow cells)의 준비
참고: 미세유체 플로우 셀(33 )은 단일-분자 실험을 위해 구성된다( 보충 도 2 참조). 이를 통해 완충액, DNA 템플릿 및 단백질이 기능화된 커버슬립과 접촉할 수 있습니다(5.1단계 참조).- 진공 청소기에서 기능적인 커버슬립을 제거하고 부분적으로 물로 채워진 마이크로튜브 랙(습한 상자)에 놓습니다. 100μL의 블로킹 버퍼(50mM의 Tris-HCl(pH 7.6), 50mM의 KCl, 2%(v/v) Tween-20)를 25μL의 1mg/mL NeutrAvidin 용액(10% PBS)과 혼합하여 커버슬립 표면에 펴 바릅니다. 습기가 많은 상자에서 실온에서 15분 동안 배양합니다.
- 커버슬립을 물로 씻고 N2에서 건조시킵니다. 커버슬립의 한쪽 면만 기능화되어 있으므로 어느 면인지 기억하십시오.
- 맞춤형 폴리디메틸실록산(PDMS) 블록34 를 플로우 셀 홀더 내의 커버슬립에 놓습니다. 이렇게 하면 높이 100μm, 너비 1mm의 흐름 채널이 생성됩니다. 그런 다음 폴리에틸렌 튜브(PE-60: 입구 직경 0.76mm 및 외경 1.22mm)를 플로우 셀의 구멍에 삽입하여 버퍼와 기판에 접근할 수 있습니다.
- 단일분자 형광 이미징
참고: 이러한 실험은 G4 형성 및 대조군 템플릿 모두에서 수행해야 합니다. 이를 통해 두 조건(G4 구조에 의한 Pol δ 차단과 차단 없음)을 TIRF(Total Internal Reflection) 현미경을 사용하여 단일 분자 수준에서 시각화할 수 있습니다.- Tween 차단 완충액의 1mL 분취액(Tris-HCl(pH 7.6) 50mM, KCl 50mM, 2%(v/v) Tween-20) 및 세척 완충액 500μL 분취액(Tris-HCl(pH 7.6) 25mM, 마그네슘 아세테이트 10mM, 글루타민산칼륨 250mM, 40μg/mL BSA, EDTA 0.1mM, DTT 5mM, 0.0025% (v/v) Tween-20) 및 복제 버퍼를 40°C에서 15분 동안 가열합니다. 이것은 용액에서 가스를 해방시킬 것입니다. 대기압보다 800mbar 낮은 곳에서 15분 동안 진공 챔버에서 용액의 가스를 제거합니다.
- 구성된 플로우 셀(flow cell)을 가져다가 현미경의 스테이지에 놓습니다. 대물렌즈에 기름 한 방울을 떨어뜨린 후 대물렌즈를 올려 커버슬립에 맞도록 합니다.
알림: 대물렌즈와 플로우 셀이 31°C에 도달할 시간이 있는지 확인합니다. 이렇게 하면 효모 Pol δ의 활성이 증가하고 재현 가능한 데이터를 보장하는 데 도움이 됩니다. - Inlet Tube를 탈기된 Tween 차단 버퍼에 삽입하고 Outlet을 실린지 펌프에 연결합니다. 주사기를 뒤로 당겨 튜브를 통해 Tween 차단 버퍼를 채널로 끌어들입니다. 이 버퍼가 비특이적 상호 작용을 최소화하기 위해 최소 30분 동안 배양하도록 합니다.
- 200μL의 탈기 세척 버퍼를 100μL/분의 속도로 채널로 유입합니다. 이렇게 하면 Tween 블로킹 버퍼가 플러시됩니다.
- DNA 템플릿 용액을 500μL 복제 완충액에서 0.5pM으로 희석합니다. 10 μL/min의 속도로 150 μL를 채널로 흘려보냅니다. 샘플 평면에서 약 900mWcm-2에서 647nm 레이저를 사용하여 샘플을 조명하여 개별 DNA 템플릿을 시각화합니다.
참고: 샘플의 시야(FOV)에 초점이 완전히 맞춰지지 않은 경우 커버 슬립이 구성된 플로우 셀의 바닥과 같은 높이에 있지 않을 수 있습니다. 이 경우 새로운 커버슬립(coverslip)을 사용하여 새 플로우 셀을 구성합니다. - 충분한 반점 밀도(10μm2당 ca 1 spot)가 보이면 복제 완충액(1mM의 DTT가 보충됨)의 새로운 용액을 채널로 유입시켜 과도한 DNA를 씻어냅니다. 250 μL의 부피는 모든 결합되지 않은 DNA를 완전히 제거하기에 충분합니다.
- 새로운 FOV로 이동하고 DNA 이미지를 캡처하여 표지된 중합효소와 DNA 기질 사이의 공동 국소화 정도를 결정합니다. 완료되면 레이저 출력을 높여 나머지 반점을 광표백합니다.
- 200μL의 복제 완충액에 1mM의 DTT, 250μM의 dNTP 및 20nM의 AF647 표지 Pol δ을 함유하는 중합효소 용액을 준비합니다. 100μL의 중합효소 용액을 5μL/분의 속도로 채널로 유입합니다.
- 샘플에 초점이 맞춰지고 TIRF 각도가 조정되면 샘플 평면에서 647nm 레이저의 레이저 출력을 약 900mWcm-2로 설정합니다. 그런 다음 원하는 시간 동안 FOV 이미징을 시작합니다. 10-20분 동안 1-5 프레임/초를 획득하여 모든 복제 이벤트를 캡처합니다.
- 데이터 분석
참고: 모든 분석은 Python(v. 3.11.7)을 사용하여 수행되었습니다. 데이터 분석에 사용되는 사용자 지정 코드는 https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis 에서 사용할 수 있습니다.- 먼저, DNA 템플릿 스팟을 라벨링된 효모 Pol δ의 형광 스폿과 공동 국소화합니다. 두 개의 피크는 드리프트 보정 후 거리가 3픽셀인 경우 공동 국소화≤되는 것으로 확인되었습니다. DNA 주형을 공동 국소화하는 Pol δ 스폿만 결합 역학 및 전반적인 행동 경향에 대해 분석됩니다.
- 바인딩 이벤트 데이터를 확인하려면 영화 전체에서 "켜기/끄기" 동작을 보이는 각 지점의 시간 경과에 따른 강도를 측정합니다. 이를 통해 단일 DNA 템플릿에서 Pol δ 결합이 발생하는 횟수를 측정할 수 있습니다.
참고: 이 분석은 결합 역학을 비교하기 위해 G4 형성 및 대조군 템플릿 실험 모두에 대해 수행됩니다.
참고: G-quadruplex 또는 템플릿 자체(23.2초 ± 4.9초)에 대한 비특이적 결합 이벤트가 결합 이벤트 분석에 포함되지 않도록 하는 대조군 실험이 수행되었습니다.
결과
이 분석을 위해 형광 표지된 20-nt 프라이머가 있는 두 개의 DNA 기질이 설계되었는데, 하나는 G4 형성 서열을 포함하고(그림 1A, 왼쪽) 이 서열이 없는 기질(그림 1A, 오른쪽)을 포함합니다. G-quadruplex가 중합효소 활성에 효과적인 장애물임을 확인하기 위해 Pol δ에 의한 DNA 합성을 변성 PAGE 겔에서 모니터링했습니다. DNA 기질에 대한 정제된 표지된 Pol δ의 활성을 겔 전기영동에 의해 검사했습니다. 그림 1B (왼쪽)는 형광 표지된 Pol δ이 G-quadruplex를 지나 합성할 수 없음을 보여줍니다. 합성이 시작되기 전에(t = 0 min), 20 nt에 해당하는 밴드가 존재하며, 이는 템플릿 가닥에서 변성된 라벨링된 프라이머를 나타냅니다. 3분 후 이 20-nt 밴드는 60-nt 밴드로 변환되어 모든 DNA에서 합성이 발생했음을 나타내고 중합효소가 G-사중플렉스 구조에 의해 완전히 차단되었음을 확인했습니다. 이 차단은 중합효소가 구조를 펼치거나 우회할 수 없음을 의미합니다. 대조적으로, G-quadruplex-forming sequence(그림 1A, 오른쪽)를 포함하지 않는 control template의 합성은 3분 후에 100-nt band를 생성했습니다(그림 1B, 오른쪽).
템플릿에서 합성을 확인하고 G-quadruplex에 의한 효율적인 차단을 확인한 후, 이러한 측정을 단일 분자 형광 현미경에서 반복하여 중합효소 거동을 모니터링했습니다. DNA 템플릿은 미세유체 플로우 셀(microfluidic flow cell)에서 기능화된 커버슬립(coverslip)에 테더링되었으며, 각 기질의 위치는 형광 표지된 프라이머를 시각화하여 결정되었습니다. 그런 다음 Pol δ로 표시된 것을 dNTP가 있는 곳에 로드하여 합성을 시작했습니다. 일반적인 FOV 내에서 개별 스폿을 추적하여 Pol δ이 DNA에서 얼마나 자주 결합하고 해리되는지 정량화합니다(그림 2B, C). 각 DNA 기질에서 시간 함수로 강도를 측정함으로써 단일 분자 궤적을 생성할 수 있습니다(그림 2C, 보충 그림 3). 특징적인 "체류 시간" 또는 Pol δ이 템플릿에 바인딩된 상태로 유지되는 평균 지속 시간을 측정할 수 있습니다. G4 기판의 경우 체류 시간이 6초 ± 2초로 결정된 반면, 대조 기판의 경우 10초 ± 3초로 결정되었습니다(그림 2D, 보충 그림 4). 또한 각 궤적에 대해 Pol δ이 템플릿에 바인딩하는 횟수를 정량화할 수 있습니다. G4 기질에 대한 결합 이벤트의 수는 대조군 템플릿에 비해 훨씬 높습니다(그림 2E). 스콜라스틱 바인딩 및 바인딩 해제로 인해 컨트롤 템플릿에 둘 이상의 바인딩 이벤트 인스턴스가 있지만, G4 형성 템플릿에 대한 바인딩 이벤트의 수가 평균적으로 확실히 증가하고 있습니다. 이것은 합성이 G-quadruplex에 의해 중단된 후 결합된 중합효소가 DNA에서 해리되기 전에 용액의 새로운 중합효소가 결합과 해제의 연속적인 주기를 결합한다는 것을 시사합니다. 따라서 이 단일 분자 분석은 DNA 중합효소가 DNA 장애물에 어떻게 반응하는지에 대한 탁월한 통찰력을 제공합니다.

그림 1: 앙상블 프라이머 확장 분석. (A) DNA 기질의 개략적 표현. G4 기질(왼쪽)에는 G-4중플렉스 형성 서열이 포함되어 있지만 대조 기질(오른쪽)에는 포함되어 있지 않습니다. (B) 프라이밍된 G4 및 대조군 DNA 기질의 앙상블 DNA 프리마즈 확장 분석은 형광 표지된 효모 Pol δ이 G-사중플렉스에 의해 완전히 차단되었음을 보여줍니다. PAGE 겔은 시간 경과에 따른 DNA 템플릿의 복제를 보여주며, 그 결과 라벨링된 20 nt 프라이머가 G4 기질의 경우 60-nt 산물(왼쪽)로, 대조군 주형의 경우 100-nt 산물(오른쪽)로 이동했습니다. M은 라벨링된 20nt, 60nt 및 100nt 올리고를 포함하는 래더를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
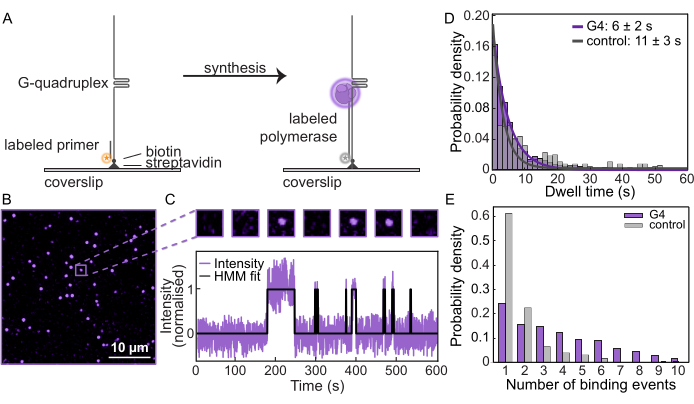
그림 2: 단일분자 형광 현미경 검사. (A) AF647 표지 효모 Pol δ을 사용한 단일분자 프라이머 확장 분석의 개략도. (B) 단일분자 분석에서 얻은 일반적인 FOV. 각 반점은 DNA 주형에 결합하는 형광 표지된 Pol δ를 나타냅니다. 스케일 바: 10μm. (C) (상단) Pol δ 교환에 따른 on/off 결합을 보여주는 단일 분자 분석의 예시 분자. (아래) 실험의 시간 경과에 따른 강도를 보여주는 동일한 분자의 단일 분자 궤적. 검은색 선은 데이터에 맞는 HMM(Hidden Markov Model)을 나타냅니다. (D) G4 기판(자주색)과 대조 기판(회색)에 대한 Pol δ의 체류 시간. 선은 지수 적합을 나타내며 G4 기판에서 6초 ± 2초, 대조 기판에서 10초 ± 3초의 수명을 제공합니다. (E) 단일 DNA 기질에 대한 Pol δ 결합 이벤트의 수. G4 기질(보라색)의 결합 이벤트 중앙값은 대조군 기질 1(회색)과 비교하여 3.5입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 그림 1 : CD 분광법. 25°C에서 200mM의 KCl을 포함하는 TE 완충액의 G4 형성(보라색) 및 제어 순서(회색)의 CD 스펙트럼. 260nm에서 특징적인 피크와 240nm에서 음의 피크가 이어지는 것은 병렬 분자 내 GQ의 특징입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 2: 플로우 셀 구조. (A) 플로우 셀(flow cell)의 개별 조각에 대한 개략도. 사각형 PDMS 블록은 현미경 커버슬립 위에 있습니다. 그런 다음 이 조각들을 플로우 셀 홀더의 뚜껑과 바닥 내부에 함께 끼웁니다. (B) 완전한 플로우 셀. 튜브를 PDMS 블록의 각 측면에 삽입하여 미세유체역학을 통해 장치를 통해 버퍼가 흐를 수 있는 채널을 형성할 수 있습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 3: 단일 분자 궤적. (A) 실험의 10분 기간 동안 단일 결합 이벤트를 겪는 분자의 궤적 예. (B) 실험의 10분 기간 동안 0 결합 이벤트를 겪는 분자의 궤적 예. (A)와 (B)의 검은색 선은 데이터에 대한 HMM(Hidden Markov Model)을 나타냅니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 4: 체류 시간. (A) G4 기판에 δ Pol의 체류 시간. 선은 지수 적합도를 나타내며 6초 ± 2초의 수명을 제공합니다.(B) 제어 기판에 대한 Pol δ의 체류 시간. 지수 맞춤은 10초 ± 3초의 수명을 제공합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 5: 광표백 대조군. 이 플롯은 개별 AF647 표지 효모 Pol δ 효소가 647nm 레이저에 의해 광표백되는 평균 시간을 보여줍니다. 선은 지수 적합도를 나타내며 39 ± 6초의 수명을 제공합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
여기에서는 DNA 중합효소가 G-4중플렉스를 만날 때 DNA 중합효소의 거동에 대한 통찰력을 제공하는 단일 분자 형광 기반 분석법이 설명되었습니다. DNA 템플릿 생성, Pol δ 라벨링 및 벌크 DNA 복제 분석의 프로토콜은 모두 간단하지만 단일 분자 현미경 분석을 실행하는 것은 기술적으로 더 까다롭습니다. 단일 분자 기술의 특성으로 인해 먼지, 오염 또는 기포가 유입되지 않도록 세심한 주의를 기울여야 하며, 이는 FOV를 모호하게 하고 데이터 수집을 방해할 수 있습니다.
단일분자 TIRF 현미경 실험의 한계는 관심 생체 분자에 공유 결합되어 있는 형광단의 광표백입니다. 광표백은 영구적인 형광 손실로 이어지는 비가역적 과정입니다35. 실험 중 이 문제를 완화하려면 레이저 노출 시간을 제한하고, 레이저 강도를 조정하고, 이미징 타이밍을 최적화하는 것이 필수적입니다. 이러한 전략은 형광 신호를 보존하는 데 도움이 되어 보다 안정적이고 확장된 관찰 기간을 보장합니다. 이러한 파라미터를 미세 조정하면 Pol δ 신호가 측정 기간 동안 유지됩니다. 단일분자 형광 현미경 검사 합성 분석을 위한 레이저 출력을 최적화하려면 깨끗한 유리 커버슬립에서 표지된 중합효소를 이미징하여 광표백 속도를 측정하는 것이 좋습니다. 레이저 출력을 체계적으로 변화시키고 시야 전반에 걸쳐 광표백 속도를 평가함으로써 형광단 신호 강도와 광표백에 대한 저항성의 균형을 맞추는 최적의 레이저 강도를 식별할 수 있습니다( 보충 그림 5 참조).
기존 앙상블 기반 방법에 비해 이 단일 분자 접근 방식의 주요 이점은 개별 DNA 중합효소가 G4 구조를 만나 상호 작용할 때 직접 시각화할 수 있다는 것입니다. 전통적인 앙상블 기반 방법(예: 겔 전기영동)은 G4 구조가 DNA 중합효소 23,36,37을 차단하는 능력을 입증했습니다. 그러나 이러한 기술은 서로 다른 분자 운동 단계와 결과를 분리하는 데 필요한 이 상호 작용의 실시간 운동 및 기계론적 정보를 제공하지 못합니다. 단일 분자 기술은 평균 앙상블38에 의해 종종 숨겨져 있는 생체 분자의 동역학, 메커니즘 및 행동에 대한 비할 데 없는 통찰력을 제공합니다. 이제 DNA 중합효소가 어떻게 작용하는지 실시간으로 확인할 수 있습니다 - DNA 장애물을 교환하거나, 지연시키거나, 해리하거나, 우회하는지 여부39. 이 프로토콜이 설정되면 G4의 ID를 선택한 병렬 c-MYC 구조에서 병렬, 반병렬 또는 하이브리드 토폴로지로 쉽게 변경할 수 있습니다. 이 단일 분자 분석을 적용하면 동일한 DNA 중합효소가 대체 G4 토폴로지를 만날 때 다르게 동작하는지 여부를 확인할 수 있습니다. 이와 같이 단일 분자 방법은 신체의 단백질과 DNA가 어떻게 상호 작용하는지에 대한 구체적인 질문에 답하는 데 필수적입니다.
DNA 중합효소와 G-사중플렉스의 직접적인 시각화를 통해 이전에는 특성화되지 않았던 효모 Pol δ에 대한 교환 경로가 확인되었습니다. 이 발견은 중합효소가 G-quadruplex를 만나면 분리되어 DNA 합성을 다시 시작하기 전에 구조를 해결하기 위해 다른 단백질의 개입을 기다린다는 것을 시사합니다. 이 프로토콜은 다양한 게놈 유지 단백질과 DNA 장애물 간의 상호 작용을 조사하는 데 적용할 수 있으며, 세포 효소가 게놈 장애를 탐색하는 방법에 대한 탁월한 통찰력을 제공합니다. 예를 들어, 이 분석법의 장애물은 G4 구조에서 단백질-DNA 가교결합(protein-DNA crosslink)으로 변경될 수 있는데, 이는 단백질이 DNA에 비가역적으로 공유 결합되어 DNA 복제에 대한 장애물로 작용하는 DNA 병변의 일종입니다40. 이러한 검사는 DNA 복제, 복구 및 재조합의 기본 과정을 이해하는 데 중요합니다. 분자 수준에서 DNA-단백질 역학 연구를 가능하게 함으로써 이 분석은 게놈 무결성의 기저에 있는 메커니즘을 설명하기 위한 강력한 도구를 제공합니다.
공개
저자들은 자신들이 경쟁하는 재정적 이해관계가 없다고 선언한다.
감사의 말
N.K.-A. 호주 정부 연구 훈련 프로그램 장학금에서 제공하는 자금을 인정합니다. L.M.S.는 National Health and Medical Research Council(Investigator Grant 2007778)으로부터 받은 자금에 감사하고 있습니다. J.S.L은 호주 정부가 후원하는 Discovery Early Career Award(DE240100780)와 NHMRC Investigator EL1(2025412)을 수상하게 된 것을 감사하게 생각합니다. S.H.M은 Bruce Warren Molecular Horizons Ealy 커리어 펠로우십의 수혜자가 된 것을 감사하게 생각합니다.
자료
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
참고문헌
- Sutton, M. D., Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci. 98 (15), 8342-8349 (2001).
- Burgers Garg, P., J, P. M. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M. -D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O., et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Sugino Kawasaki, Y., A, Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Shivji Wood, R. D., K, M. Which DNA polymerases are used for DNA repair in eukaryotes. Carcinogenesis. 18 (4), 605-610 (1997).
- Campbell Budd, M. E., L, J. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Gao Yang, W., Y, Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9. 968424, (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Monnat Loeb, L. A., J, R. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Bryan Oganesian, L., M, T. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I., et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Clayton Kaguni, L. S., A, D. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), (2021).
- Castillo Bosch,, P,, et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, J. ohnA., Zakian, V. irginiaA. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L., et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh. 11 (9), (2024).
- Pham, S. Q. T., et al. A new class of quadruplex DNA-binding nickel Schiff base complexes). Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E., et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Hansen, J. B., Au, Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J. , Springer. New York, New York, NY. 219-238 (2015).
- Lewis, J. S., et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, 23932(2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. , 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664(2014).
- Mueller, S. H., et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5(2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유