Method Article
Einzelmolekül-Fluoreszenz-Visualisierung der DNA-Polymerase-Dynamik an G-Quadruplexen
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt einen auf Fluoreszenzmikroskopie basierenden Einzelmolekül-DNA-Replikationsassay dar, der die Echtzeit-Visualisierung von Wechselwirkungen zwischen DNA-Polymerasen und Hindernissen wie G-Quadruplex-Strukturen ermöglicht.
Zusammenfassung
Die Fähigkeit von Proteinen, die an der eukaryotischen DNA-Replikation beteiligt sind, Hindernisse – wie Protein- und DNA-"Hindernisse" – zu überwinden, ist entscheidend für die Gewährleistung einer getreuen Genomduplizierung. G-Quadruplexe sind Nukleinsäurestrukturen höherer Ordnung, die sich in guaninreichen Regionen der DNA bilden und nachweislich als Hindernisse wirken und die genomischen Erhaltungswege beeinträchtigen. In dieser Studie wird eine auf Fluoreszenzmikroskopie basierende Echtzeitmethode zur Beobachtung von DNA-Polymerase-Wechselwirkungen mit G-Quadruplex-Strukturen vorgestellt. Kurze, geprimte DNA-Oligonukleotide, die ein G-Quadruplex enthalten, wurden auf funktionalisierten Glasdeckgläsern in einer mikrofluidischen Durchflusszelle immobilisiert. Fluoreszenzmarkierte DNA-Polymerasen wurden eingeführt, mit denen ihr Verhalten und ihre Stöchiometrie über die Zeit überwacht werden konnten. Dieser Ansatz ermöglichte die Beobachtung des Polymerase-Verhaltens, da es durch einen G-Quadruplex blockiert wurde. Konkret wurde unter Verwendung von fluoreszenzmarkierter Hefepolymerase δ festgestellt, dass die Polymerase beim Auftreffen auf ein G-Quadruplex einen kontinuierlichen Zyklus der Bindung und Entbindung durchläuft. Dieser Einzelmolekül-Assay kann angepasst werden, um Wechselwirkungen zwischen verschiedenen DNA-Erhaltungsproteinen und Hindernissen auf dem DNA-Substrat zu untersuchen.
Einleitung
DNA-Polymerasen sind Enzyme, die den Einbau von Nukleosidtriphosphaten katalysieren, um die DNA 1,2,3,4,5 zu duplizieren. Als solche spielen sie eine Schlüsselrolle bei essentiellen DNA-Erhaltungsprozessen, einschließlich der DNA-Replikation 6,7,8 und der Reparatur 9,10,11,12. DNA-Polymerasen müssen das Genom genau und effizient replizieren, um die genomische Integrität zu gewährleisten und die Anhäufung von Mutationen im Genom zu verhindern. Während der Synthese stoßen Polymerasen häufig auf "Hindernisse" wie DNA-gebundene Proteine oder sekundäre DNA-Strukturen13. Diese Hindernisse können das Fortschreiten der Polymerase verlangsamen oder sogar blockieren14. Die Überwindung dieser Hindernisse ist wichtig, um eine getreue Genomduplizierung zu gewährleisten, da dies nicht zu genomischer Instabilität führen kann15,16.
Eine wichtige Klasse von Hindernissen sind G-Quadruplexe (G4), nicht-kanonische sekundäre DNA-Strukturen, die sich nachweislich in guaninreichen Sequenzen innerhalb des menschlichen Genoms bilden17. Es gibt über 700.000 verschiedene Sequenzen im menschlichen Genom, die in der Lage sind, ein G4 zu bilden, einschließlich Regionen innerhalb der Telomere und Onkogen-Promotoren18. Diese DNA-Strukturen nehmen in Abhängigkeit von der Nukleotidsequenz, der Länge und dem gebundenen Metallkation 19,20,21 verschiedene Konformationen an. Diese Vielfalt bedeutet, dass Polymerasen eine Reihe verschiedener G4-Topologien überwinden müssen, möglicherweise mit unterschiedlichem Wirkungsgrad. Es wurde gezeigt, dass das Versagen von Polymerasen, eine G4-Struktur zu überwinden oder zu umgehen, die Progression der Replikationsgabel in vivo behindert, was zu genomischer Instabilität führt22. In-vitro-Studien haben gezeigt, dass G4-Strukturen Hefepolymerasen blockieren oder vollständig blockieren können 23,24,25,26,27. Die Fähigkeit von G4-Strukturen, DNA-Polymerasen zu blockieren, hängt vollständig von ihrer kinetischen und thermodynamischen Stabilität ab, wobei einige Polymerasen in der Lage sind, bestimmte G4-Polymerasen zu entfalten28. Während diese Studien einen Einblick in die Fähigkeit der Polymerasen geben, G-Quadruplex-Hindernisse zu überwinden, fehlt ihnen die Fähigkeit, das Verhalten der Polymerase beim Auftreffen auf eine G4 direkt zu visualisieren. Das Schicksal der Polymerasen - ob sie gebunden bleiben, abfallen oder dynamisch austauschen - bestimmt, welche nachgelagerten Prozesse zur Auflösung des G4 zur Verfügung stehen.
In dieser Studie wurde ein fluoreszenzbasierter Einzelmolekül-Mikroskopie-Assay entwickelt, um die Bindung der DNA-Polymerase an G4-Strukturen in Echtzeit direkt sichtbar zu machen und zu überwachen. Bei diesem Assay werden G4-bildende DNA-Templates an ein biotinyliertes Deckglas in einer mikrofluidischen Durchflusszelle gebunden, in die fluoreszenzmarkierte DNA-Polymerasen eingeführt werden können, um die DNA-Synthese zu initiieren. Durch die Messung der Fluoreszenz der Polymerasen über die Zeit kann ihr Verhalten beim Auftreffen auf eine G4-Struktur direkt beobachtet werden. Die G4-Struktur, die im c-MYC-Krebsonkogen gefunden wurde, wurde aufgrund ihrer hohen Stabilität für diesen Assay ausgewählt. Dieses Protokoll kann nun angepasst werden, um das Verhalten einer Vielzahl von Polymerasen in allen Lebensbereichen zu katalogisieren, die mit unterschiedlichen G4-Topologien und -Stabilitäten verbunden sind. Dieser Assay bietet einen innovativen und hochdurchsatzfähigen Ansatz zur Aufklärung der Mechanismen, mit denen DNA-Polymerasen DNA-Hindernisse überwinden, und bietet ein leistungsstarkes Werkzeug, um das Verständnis der Polymerasedynamik zu verbessern.
Protokoll
Einzelheiten zu den verwendeten Reagenzien und den verwendeten Geräten sind in der Materialtabelle aufgeführt.
1. Zirkulardichroismus-Spektroskopie
HINWEIS: Vor der Entwicklung des Assays war es notwendig, eine Zirkulardichroismus-Spektroskopie (CD) an der ausgewählten G4-Sequenz durchzuführen, um eine korrekte Faltung zu gewährleisten. Die 22 nt-Sequenz (5′-TGAGGGTGGGGAGGGTGGGGAA-3′) bildet die G4-Struktur, die vom c-MYC-Krebsonkogen abgeleitet ist. Die Zöliakiespektroskopie wurde auch an einer Kontrollsequenz (5′- TGAGTGTGAAGACGATGTAGAA -3) durchgeführt, bei der wichtige Nukleotide verändert sind, die die G4-Bildung verhindern.
- Um die DNA-Templates für die CD-Spektroskopie vorzubereiten, fügen Sie 40 μl von 100 μM der 22 nt-Templates zu 360 μl Tris-HCl (pH 8,0), Ethylendiamintetraessigsäure (EDTA) (TE)-Puffer, ergänzt mit 200 mM Kaliumchlorid (KCl), hinzu. Für CD-Spektroskopie-Experimente ist eine DNA-Endkonzentration von 10 μM erwünscht.
HINWEIS: Das Vorhandensein von K+ -Ionen ist für die Stabilisierung der G4-Struktur in der Vorlage unerlässlich. - Erhitzen Sie die DNA-Lösungen in einem digitalen Trockenbad 15 Minuten lang auf 95 °C. Wenn Sie fertig sind, kühlen Sie die Lösungen über Nacht langsam ab, indem Sie den Heizblock in Styropor legen. Dieser langsame Abkühlprozess ist für das Falten des G4 verantwortlich.
- 400 μl der G4-bildenden und Kontrollsequenzlösungen werden mit einer gasdichten Spritze in eine Quarzküvette mit einer Schichtdicke von 0,1 cm überführt. Füllen Sie die Küvette langsam, um die Bildung von Luftblasen zu vermeiden.
- Messen Sie das CD-Spektrum zwischen 200 nm und 400 nm mit einem Spektropolarimeter bei 25 °C. Die Parameter und Bedingungen für diese Messung wurden bereits beschrieben29. Die Ergebnisse sind in der ergänzenden Abbildung 1 zu finden.
HINWEIS: Stellen Sie sicher, dass das CD-Spektrum nur für den Puffer aufgezeichnet wird, damit es als Leerzeichen fungiert.
2. Erstellung von DNA-Templates
HINWEIS: Bei den replizierbaren Vorlagen handelt es sich um kurze, mit 100 nt geprimte lineare Vorlagen, die mit molekularbiologischen Standardtechniken hergestellt wurden. Die G4-bildende Schablone (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGGGGGGGGGGGGT
GGGACCTTAGCTTCGAGTTCCGAT-3′) enthält in seinem Zentrum die Sequenz, die in der Lage ist, ein MYC-abgeleitetes G-Quadruplex (aus Schritt 1) zu bilden. Die Kontrollschablone (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3′) hat die gleiche Kontrollregion (aus Schritt 1) ebenfalls in seinem Zentrum.
- Um den Primer an die G4-bildenden und Kontroll-DNA-Templates zu binden, mischen Sie 20 μM des Primers (5′-Biotin-AF647-TCTTAATGTAAATTTAAAATGTC-3′) mit 20 μM der 100 nt-Templates in TE-Puffer, ergänzt mit 200 mM KCl.
- Erhitzen Sie die DNA-Template-Lösungen in einem digitalen Trockenbad für 15 min auf 95 °C. Sobald die Lösung fertig ist, kühlen Sie sie über Nacht langsam ab, indem Sie den Heizblock in Styropor legen, um das Falten des G4 und das Glühen der beiden Stränge zu ermöglichen.
3. Markierung der DNA-Polymerase mit AF647
- Markierung der Polymerase mit dem Farbstoff
HINWEIS: Die Hefe Pol δ wurde wie zuvor beschrieben exprimiert und gereinigt30. Die Polymerase musste dann mit dem Monoreaktivfarbstoff Alexa Fluor NHS Ester (AF647) markiert werden, um eine Visualisierung auf Einzelmolekülebene zu ermöglichen. Dies wurde wie zuvor beschrieben abgeschlossen5.- Um den N-Terminus zu markieren, inkubieren Sie einen 3-fachen Überschuss des Farbstoffs AF647 mit 320 μl 58,5 μM Pol δ in einem Puffer, der 30 mM Tris-HCl (pH 7,6), 2 mM Dithiothreitol (DTT), 300 mM Natriumchlorid (NaCl), 50 mM Kaliumglutamat und 10 % (v/v) Glycerin für 10 min bei 4 °C enthält. Drehen Sie die Lösung vorsichtig, um eine gründliche Durchmischung zu gewährleisten und die Etikettiereffizienz zu verbessern.
HINWEIS: Die Proteinsequenz kann zusätzliche primäre Amine in Lysin-Aminosäuren enthalten. Durch Einstellen der Reaktionsbedingungen, wie z. B. des pH-Werts, kann die Markierung bevorzugt an den N-Terminus gegenüber den Lysinresten angehängt werden. Dies kann weitere Optimierungen erfordern, um die Etikettiereffizienz zu erhöhen. - Zur Vorbereitung der 0,5 mL Spin-Entsalzungssäulen (7 K Molekulargewicht abgeschnitten) zentrifugieren Sie die Säulen zunächst 1 min lang bei 1500 x g , um die Lagerlösung zu entfernen. Die im Auffangröhrchen abgelagerten Lösungen sind nach dem Zentrifugieren zu verwerfen.
HINWEIS: Stellen Sie sicher, dass die Kappe der Spin-Säulen während der Zentrifugationsschritte gelöst wird. Dadurch wird sichergestellt, dass die Säulen ordnungsgemäß eluiert werden. - Die Säulen werden in einem Puffer äquilibriert, der 30 mM Tris-HCl (pH 7,6), 300 mM NaCl, 3 mM DTT, 50 mM Kaliumglutamat und 5 % (v/v) Glycerin enthält, indem 300 μl Puffer am oberen Rand der Säule hinzugefügt werden. Zentrifugieren Sie die Säule bei 1500 x g für 1 min und verwerfen Sie den Durchfluss. Wiederholen Sie diesen Vorgang zwei weitere Male.
- Geben Sie 200 μl jeder Pol δ Probe auf die Säule. Zentrifugieren Sie die Säulen bei 1500 x g für 2 min und sammeln Sie das gereinigte Pol δ in einem 1,5 mL Mikrozentrifugenröhrchen.
- Frieren Sie die gereinigten Fraktionen in flüssigem N2 ein und lagern Sie die Aliquote bei -80 °C, um einen Proteinabbau im Laufe der Zeit zu verhindern.
- Um den N-Terminus zu markieren, inkubieren Sie einen 3-fachen Überschuss des Farbstoffs AF647 mit 320 μl 58,5 μM Pol δ in einem Puffer, der 30 mM Tris-HCl (pH 7,6), 2 mM Dithiothreitol (DTT), 300 mM Natriumchlorid (NaCl), 50 mM Kaliumglutamat und 10 % (v/v) Glycerin für 10 min bei 4 °C enthält. Drehen Sie die Lösung vorsichtig, um eine gründliche Durchmischung zu gewährleisten und die Etikettiereffizienz zu verbessern.
- Bestimmung der Etikettiereffizienz
- Laden Sie 2 μL der nun AF647-markierten Pol δ-Probe auf ein UV-Vis-Spektralphotometer31.
HINWEIS: Stellen Sie sicher, dass zuerst ein Leerzeichen des Pol δ-Puffers gemessen wird. - Messen Sie mit dem Spektralphotometer die Anregungswellenlängen von 650 nm für den Farbstoff AF647 und 280 nm für den Pol-δ.
- Berechnen Sie unter Verwendung des Beer-Lambert-Gesetzes und der molaren Extinktionskoeffizienten von 270.000 cm-1 M-1 für den Farbstoff AF647 und 195.960 cm-1 M-1 für die Polymerase die Markierungseffizienz.
HINWEIS: Die Markierungseffizienz, die für die in diesem Assay verwendete AF647-markierte Hefe Pol δ bestimmt wurde, wurde auf etwa 67 % berechnet.
- Laden Sie 2 μL der nun AF647-markierten Pol δ-Probe auf ein UV-Vis-Spektralphotometer31.
4. Ensemble-Primer-Verlängerungsassay
HINWEIS: Vor der Durchführung von Einzelmolekül-Replikationsexperimenten muss bestätigt werden, dass die DNA-Polymerase durch den G-Quadruplex über Massenreplikationsassays blockiert wird.
- Stellen Sie die Temperatur eines Heizblocks in einem digitalen Trockenbad auf 30 °C ein. Dadurch wird die maximale Aktivität der Hefe Pol δ gewährleistet.
- Bereiten Sie "Mastermixe" für die G4-bildenden und Kontroll-Templates in Replikationspuffer vor (25 mM Tris-HCl (pH 7,6), 10 mM Magnesiumacetat, 50 mM Kaliumglutamat, 40 μg/ml Rinderserumalbumin (BSA), 0,1 mM EDTA, 5 mM DTT und 0,0025 % (v/v) Tween-20). Ergänzen Sie jeden Mix mit je 1 mM DTT, je 250 μM dTTP, dCTP, dATP, dGTP (dNTPs) und 10 nM des jeweiligen DNA-Templates (G4-bildend oder Kontrolle). Legen Sie diese in einen Heizblock, um sicherzustellen, dass sie 30 °C erreichen.
- Quenden Sie 12 μl jedes Mastermixes mit 12 μl Formamid-Ladepuffer (80 % (w/v) Formamid, 10 mM EDTA) ab, um als T = 0 min-Kontrolle zu fungieren.
- Um die DNA-Synthese zu starten, fügen Sie jedem Mastermix AF647-markierte Hefe Pol δ hinzu (Endkonzentration von 20 nM). Gründlich mischen, um sicherzustellen, dass die DNA-Polymerasen die Templates mit maximaler Effizienz replizieren.
HINWEIS: Bewahren Sie die Hefe Pol während des Gebrauchs δ auf Eis auf, um die Enzymaktivität zu erhalten. - Entfernen Sie nach 30 s, 60 s und 180 s 12 μl jedes Mastermixes und schrecken Sie mit 12 μl Formamid-Ladepuffer ab. Das Verhältnis von 1:1 zwischen dem Mastermix und dem Formamid-Ladepuffer stellt sicher, dass die Replikationsreaktion nach dem Quenchieren nicht fortgesetzt werden kann.
- Nehmen Sie die nun abgeschreckten Lösungen aus dem 30 °C-Heizblock und geben Sie sie stattdessen in einen 98 °C-Heizblock, um die dsDNA zu ssDNA zu denaturieren. Nach Ablauf von 10 Minuten wird der gesamte Heizblock in den Geltank gebracht, um die Lösungen in ein denaturierendes 15%iges Tris-Borat-EDTA (FSME)-Harnstoff-Polyacrylamid (PAGE)-Gel32 zu laden.
HINWEIS: Das PAGE-Gel muss vor dem Laden der Proben erwärmt werden, indem die Gelelektrophorese 30 Minuten lang bei 180 V in 1x TBE-Puffer durchgeführt wird. Wenn Sie fertig sind, spülen Sie jede Vertiefung aus, um Harnstoff zu entfernen. - Laden Sie 15 μl jeder Probe auf das Gel neben einer Leiter mit 0,02 μM 20 nt-, 60 nt- und 100 nt-markierten Oligonukleotiden in Formamid-Ladepuffer.
- Lassen Sie das Gel 60 min lang bei 180 V in 1x TBE-Puffer laufen, um eine vollständige Trennung zu gewährleisten.
- Bilden Sie das Gel im Cy5-Kanal eines biomolekularen Imagers ab. Auf diese Weise kann der replizierte DNA-Strang, der die AF-647-Markierung enthält, gemessen werden.
5. Einzelmolekül-Fluoreszenzmikroskopie
- Funktionalisierung von Deckgläsern
HINWEIS: Um die DNA-Templates auf dem Glasdeckglas zu befestigen, muss es zunächst mit Aminosilan funktionalisiert werden, gefolgt von der Tethering von biotinylierten PEG-Molekülen. Dieser Prozess minimiert die unspezifischen Wechselwirkungen zwischen der DNA und/oder den Proteinen und der Oberfläche.- Reinigen Sie die Deckgläser (24 x 24 mm), indem Sie sie in Färbegläser legen und mit Ethanol aufgießen. Beschallen Sie die Gläser 30 Minuten lang, bevor Sie die Deckgläser mit reinem Wasser ausspülen. Wiederholen Sie diesen Vorgang mit 1 M Kaliumhydroxid (KOH), bevor Sie erneut spülen. Wiederholen Sie diese Reinigungsschritte noch einmal.
- Spülen Sie ein neues Glas aus, füllen Sie es mit Aceton und legen Sie die Deckgläser hinein. Disseminieren Sie 3-Aminopropyltriethoxysilan in das Glas, um eine 2%ige v/v-Lösung zu bilden. Rühren Sie das Glas 3 Minuten lang, bevor Sie die Reaktion mit einem großen Überschuss an Wasser abschrecken.
- Trocknen Sie die Deckgläser mit N2 und legen Sie sie einzeln auf eine mit Wasser gefüllte Feuchtbox. So wird ein Austrocknen der Deckgläser verhindert.
- Bereiten Sie einen 1:25 Biotin-PEG-SVA:mPEG-SVA-Ester in 100 mM NaHCO3 (pH 8,2) vor. Wirbeln Sie diese Mischung 20 s lang, um eine gründliche Durchmischung zu gewährleisten.
- Pipettieren Sie 40 μl der PEG-Lösung auf ein trockenes Deckglas. Legen Sie ein weiteres Deckglas darauf, um die Lösung zwischen den beiden Deckgläsern zu "klemmen". Dadurch werden die Flächen des Deckglases auf der Innenseite funktionalisiert. Wiederholen Sie den Vorgang für alle Deckgläser.
- Inkubieren Sie die Deckgläser 3 h lang im Dunkeln. Trennen Sie nach der Inkubation die Deckglaspaare, spülen Sie sie mit überschüssigem Wasser ab und trocknen Sie sie mit komprimiertem N2 -Gas.
- Wiederholen Sie die Schritte 4.1.4-4.1.5, um eine zweite Schicht PEG auf die Deckgläser aufzutragen. Achten Sie darauf, die zuvor funktionalisierten Seiten miteinander zu verbinden, also achten Sie darauf, dass sie während der Waschschritte nicht versehentlich umgedreht werden. Inkubieren Sie die Lösungen über Nacht im Dunkeln, bevor Sie sie spülen und trocknen.
- Bewahren Sie die Deckgläser im Vakuum auf, um ihre Funktionalität zu erhalten. Funktionalisierte Deckgläser sind bei richtiger Lagerung einen Monat lang stabil.
- Vorbereitung von mikrofluidischen Durchflusszellen
HINWEIS: Eine mikrofluidische Durchflusszelle33 ist für Einzelmolekülexperimente konstruiert (siehe Ergänzende Figur 2). Dadurch können Puffer, DNA-Templates und Proteine mit einem funktionalisierten Deckglas in Kontakt kommen (siehe Schritt 5.1).- Nehmen Sie ein funktionalisiertes Deckglas aus dem Vakuum und legen Sie es auf ein teilweise mit Wasser gefülltes Mikroröhrchengestell (Feuchtbox). Mischen Sie 100 μl eines Blockierungspuffers (50 mM Tris-HCl (pH 7,6), 50 mM KCl, 2% (v/v) Tween-20) mit 25 μl 1 mg/ml NeutrAvidin-Lösung (10% PBS) und verteilen Sie diese auf der Oberfläche des Deckglases. Lassen Sie diese 15 min bei Raumtemperatur in der Feuchtbox inkubieren.
- Waschen Sie den Deckbezug mit Wasser und trocknen Sie ihn in N2. Denken Sie daran, dass nur eine Seite des Deckglases funktionalisiert ist, also denken Sie daran, um welche Seite es sich handelt.
- Platzieren Sie den maßgefertigten Polydimethylsiloxan (PDMS) Block34 auf dem Deckglas innerhalb des Durchflusszellenhalters. Dadurch entsteht ein 100 μm hoher und 1 mm breiter Strömungskanal. Polyethylenrohre (PE-60: 0,76 mm Einlaufdurchmesser und 1,22 mm Außendurchmesser) können dann in die Löcher in der Durchflusszelle eingeführt werden, um den Zugang zu Puffern und Substraten zu ermöglichen.
- Einzelmolekül-Fluoreszenzbildgebung
HINWEIS: Diese Versuche sind sowohl mit den G4-Formungs- als auch mit den Kontrollschablonen durchzuführen. Auf diese Weise können beide Bedingungen (Blockierung des Pols δ durch eine G4-Struktur und keine Blockierung) mit Hilfe der Totalreflexionsmikroskopie (TIRF) auf Einzelmolekülebene sichtbar gemacht werden.- Erhitzen Sie 1 mL Aliquots eines Tween-Blockierungspuffers (50 mM Tris-HCl (pH 7,6), 50 mM KCl, 2 % (v/v) Tween-20) und 500 μL Aliquots eines Waschpuffers (25 mM Tris-HCl (pH 7,6), 10 mM Magnesiumacetat, 250 mM Kaliumglutamat, 40 μg/mL BSA, 0,1 mM EDTA, 5 mM DTT, 0,0025 % (v/v) Tween-20) und Replikationspuffer auf 40 °C für 15 min. Dadurch werden die Gase aus den Lösungen freigesetzt. Entgasen Sie die Lösungen in einer Vakuumkammer für weitere 15 min bei 800 mbar unter Atmosphärendruck.
- Nehmen Sie die konstruierte Durchflusszelle (siehe Schritt 4.2) und stellen Sie sie auf den Tisch des Mikroskops. Nachdem Sie einen Tropfen Öl auf das Objektiv gegeben haben, heben Sie das Objektiv an, sodass es auf das Deckglas trifft.
HINWEIS: Stellen Sie sicher, dass das Objektiv und die Durchflusszelle Zeit haben, 31 °C zu erreichen. Dies erhöht die Aktivität der Hefe Pol δ und trägt dazu bei, reproduzierbare Daten zu gewährleisten. - Führen Sie den Einlassschlauch in den entgasten Tween-Blockierpuffer ein und verbinden Sie den Auslass mit der Spritzenpumpe. Ziehen Sie die Spritze zurück, um den Tween-Blockierungspuffer durch den Schlauch in den Kanal zu ziehen. Lassen Sie diesen Puffer mindestens 30 Minuten lang inkubieren, um unspezifische Wechselwirkungen zu minimieren.
- Fließen Sie 200 μl entgasten Waschpuffer mit einer Geschwindigkeit von 100 μl/min in den Kanal ein. Dadurch wird der Tween-Blockierungspuffer gelöscht.
- Verdünnen Sie die DNA-Matrizenlösungen auf 0,5 pM in 500 μl Replikationspuffer. Fließen Sie 150 μl mit einer Geschwindigkeit von 10 μl/min in den Kanal. Beleuchten Sie die Probe mit einem 647-nm-Laser bei ca. 900 mWcm-2 in der Probenebene, um einzelne DNA-Templates zu visualisieren.
HINWEIS: Wenn das Sichtfeld (FOV) mit der Probe nicht vollständig scharf ist, sitzt das Deckglas möglicherweise nicht bündig mit dem Boden der konstruierten Durchflusszelle. Konstruieren Sie in diesem Fall eine neue Durchflusszelle mit einem frischen Deckglas. - Sobald eine ausreichende Dichte von Spots (ca. 1 Spot pro 10μm 2) sichtbar ist, wird eine frische Lösung aus Replikationspuffer (ergänzt mit 1 mM DTT) in den Kanal eingegossen, um überschüssige DNA auszuwaschen. Ein Volumen von 250 μL reicht aus, um die gesamte ungebundene DNA vollständig zu entfernen.
- Wechseln Sie zu einem neuen FOV und nehmen Sie ein Bild der DNA auf, um den Grad der Kolokalisation zwischen der markierten Polymerase und dem DNA-Substrat zu bestimmen. Sobald der Vorgang abgeschlossen ist, erhöhen Sie die Laserleistung, um die verbleibenden Flecken zu photobleichen.
- Bereiten Sie eine Polymeraselösung vor, die 1 mM DTT, 250 μM dNTPs und 20 nM AF647-markiertes Pol δ in 200 μl Replikationspuffer enthält. 100 μl der Polymeraselösung mit einer Geschwindigkeit von 5 μl/min in den Kanal einfließen lassen.
- Sobald die Probe scharf gestellt ist und der TIRF-Winkel eingestellt wurde, stellen Sie die Laserleistung eines 647-nm-Lasers in der Probenebene auf ca. 900 mWcm-2 ein. Beginnen Sie dann mit der Abbildung des FOV für die gewünschte Zeitspanne. Erfassen Sie zwischen 1 und 5 Frames/s für 10 bis 20 Minuten, um alle Replikationsereignisse zu erfassen.
- Datenanalyse
HINWEIS: Alle Analysen wurden mit Python (v. 3.11.7) durchgeführt. Der benutzerdefinierte Code, der für die Datenanalyse verwendet wird, ist hier verfügbar: https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis.- Kolokalisieren Sie zunächst die DNA-Template-Spots mit den fluoreszierenden Spots der markierten Hefe Pol δ. Zwei Peaks wurden als kolokalisiert bestimmt, wenn ihr Abstand nach der Driftkorrektur ≤ 3 Pixel betrug. Nur die Pol-δ-Spots, die DNA-Templates kolokalisieren, werden auf ihre Bindungskinetik und allgemeine Verhaltenstrends analysiert.
- Um Bindungsereignisdaten zu bestimmen, messen Sie die Intensität der einzelnen Spots im Zeitverlauf, da sie während des gesamten Films ein Ein-/Ausschaltverhalten aufweisen. Auf diese Weise kann bestimmt werden, wie oft die Pol δ-Bindung an einem einzelnen DNA-Template stattfindet.
HINWEIS: Diese Analyse wird sowohl für die G4-bildenden als auch für die Kontrollschablonenexperimente durchgeführt, um die Bindungsdynamik zu vergleichen.
HINWEIS: Es wurden Kontrollexperimente durchgeführt, die sicherstellen, dass unspezifische Bindungsereignisse für den G-Quadruplex oder das Template selbst (23,2 s ± 4,9 s) nicht in die Analyse der Bindungsereignisse einbezogen werden.
Ergebnisse
Für diesen Assay wurden zwei DNA-Substrate mit einem fluoreszenzmarkierten 20-nt-Primer entworfen: eines mit einer G4-bildenden Sequenz (Abbildung 1A, links) und eines ohne diese Sequenz (Abbildung 1A, rechts). Um zu bestätigen, dass das G-Quadruplex ein wirksames Hindernis für die Polymeraseaktivität darstellt, wurde die DNA-Synthese durch Pol δ an einem denaturierenden PAGE-Gel überwacht. Die Aktivität des gereinigten markierten Pol-δ auf den DNA-Substraten wurde mittels Gelelektrophorese untersucht. Abbildung 1B (links) zeigt, dass das fluoreszenzmarkierte Pol δ nicht in der Lage ist, am G-Quadruplex vorbei zu synthetisieren. Vor Beginn der Synthese (t = 0 min) liegt eine Bande vor, die 20 nt entspricht und den markierten Primer darstellt, der aus dem Template-Strang denaturiert wurde. Nach 3 Minuten wurde diese 20-nt-Bande in eine 60-nt-Bande umgewandelt, was darauf hindeutet, dass die Synthese auf der gesamten DNA stattgefunden hat und bestätigt, dass die Polymerase vollständig durch die G-Quadruplex-Struktur blockiert wurde. Diese Blockierung impliziert, dass sich die Polymerase weder entfalten noch die Struktur umgehen konnte. Im Gegensatz dazu ergab die Synthese eines Kontroll-Templates, das die G-Quadruplex-bildende Sequenz nicht enthält (Abbildung 1A, rechts), nach 3 min eine 100-nt-Bande (Abbildung 1B, rechts).
Nach Bestätigung der Synthese an den Templates und der effizienten Blockierung durch das G-Quadruplex wurden diese Messungen an einem Einzelmolekül-Fluoreszenzmikroskop wiederholt, um das Verhalten der Polymerase zu überwachen. Die DNA-Templates wurden in einer mikrofluidischen Durchflusszelle an funktionalisierte Deckgläser gebunden (Abbildung 2A), und die Position jedes Substrats wurde durch Visualisierung des fluoreszenzmarkierten Primers bestimmt. Dann wurde markiertes Pol δ in Gegenwart von dNTPs geladen, um die Synthese zu initiieren. Innerhalb eines typischen FOV werden einzelne Spots verfolgt, um zu quantifizieren, wie oft Pol δ an die DNA bindet und von ihr dissoziiert (Abbildung 2B, C). Durch die Messung der Intensität als Funktion der Zeit an jedem DNA-Substrat können Einzelmolekül-Trajektorien erzeugt werden (Abbildung 2C, Ergänzende Abbildung 3). Gemessen werden kann die charakteristische "Verweildauer", also die durchschnittliche Dauer, in der Pol δ an die Vorlage gebunden bleibt. Für das G4-Substrat wurde die Verweilzeit mit 6 s ± 2 s bestimmt, während sie für das Kontrollsubstrat 10 s ± 3 s betrug (Abbildung 2D, Ergänzende Abbildung 4). Darüber hinaus kann für jede Trajektorie quantifiziert werden, wie oft Pol δ an das Template bindet. Die Anzahl der Bindungsereignisse an das G4-Substrat ist im Vergleich zum Kontroll-Template viel höher (Abbildung 2E). Während es Instanzen von mehr als einem Bindungsereignis in der Kontrollvorlage gibt, die auf schulische Bindung und Aufhebung der Bindung zurückzuführen sind, ist die Anzahl der Bindungsereignisse im Durchschnitt für die G4-bildende Vorlage deutlich gestiegen. Dies deutet darauf hin, dass die gebundene Polymerase nach dem Stoppen der Synthese durch das G-Quadruplex von der DNA dissoziiert, bevor neue Polymerasen aus der Lösung einen kontinuierlichen Zyklus der Bindung und Entbindung in Gang setzen. Daher bietet dieser Einzelmolekül-Assay einen beispiellosen Einblick in die Reaktion von DNA-Polymerasen auf DNA-Blockaden.

Abbildung 1: Ensemble-Primer-Verlängerungsassay. (A) Schematische Darstellung der DNA-Substrate. Das G4-Substrat (links) enthält eine G-Quadruplex-bildende Sequenz, während das Kontrollsubstrat (rechts) dies nicht tut. (B) Ensemble-DNA-Primase-Extensions-Assay der geprimten G4- und Kontroll-DNA-Substrate, der zeigt, dass die fluoreszenzmarkierte Hefe Pol δ vollständig durch den G-Quadruplex blockiert wird. Das PAGE-Gel zeigt die Replikation der DNA-Templates im Laufe der Zeit, was zu einer Verschiebung des markierten 20-nt-Primers in das 60-nt-Produkt für das G4-Substrat (links) und das 100-nt-Produkt für das Kontroll-Template (rechts) führt. M stellt eine Leiter dar, die markierte 20 nt-, 60 nt- und 100 nt-Oligos enthält. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
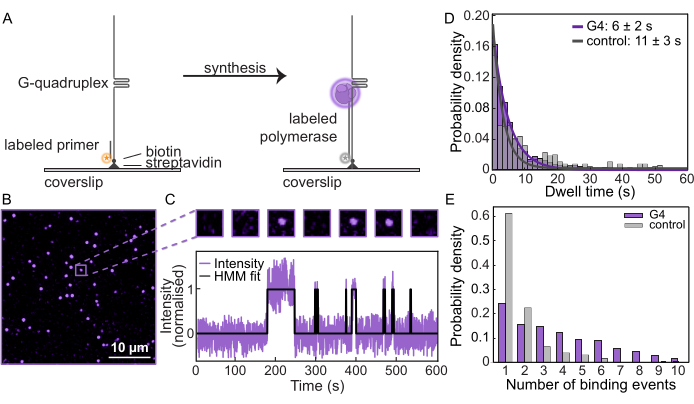
Abbildung 2: Einzelmolekül-Fluoreszenzmikroskopie. (A) Schematische Darstellung des Einzelmolekül-Primer-Verlängerungsassays unter Verwendung von AF647-markierter Hefe Pol δ. (B) Typisches FOV, das aus einem Einzelmolekül-Assay gewonnen wurde. Jeder Spot stellt eine fluoreszenzmarkierte Pol-δ dar, die an eine DNA-Matrize bindet. Maßstabsbalken: 10 μm. (C) (Oben) Beispielmolekül aus dem Einzelmolekül-Assay, das eine Ein-/Aus-Bindung zeigt, wenn der Pol δ austauscht. (Unten) Einzelmolekül-Trajektorie desselben Moleküls, die die Intensität über den Zeitverlauf des Experiments zeigt. Die schwarze Linie stellt das Hidden-Markov-Modell (HMM) dar, das an die Daten angepasst wurde. (D) Verweilzeit von Pol δ auf dem G4-Substrat (violett) und dem Kontrollsubstrat (grau). Die Linien stellen exponentielle Passungen dar, was eine Lebensdauer von 6 s ± 2 s auf dem G4-Substrat und 10 s ± 3 s auf dem Kontrollsubstrat ergibt. (E) Anzahl der Pol δ Bindungsereignisse an einzelne DNA-Substrate. Die mediane Anzahl der Bindungsereignisse auf dem G4-Substrat (violett) beträgt 3,5 im Vergleich zum Kontrollsubstrat 1 (grau). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: CD-Spektroskopie. CD-Spektren der G4-bildenden (violett) und Kontrollsequenz (grau) in TE-Puffer mit 200 mM KCl bei 25 °C. Der charakteristische Peak bei 260 nm, gefolgt vom negativen Peak bei 240 nm, ist charakteristisch für einen parallelen, intramolekularen GQ. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2: Konstruktion der Durchflusszelle. (A) Schematische Darstellung der einzelnen Teile der Durchflusszelle. Der quadratische PDMS-Block befindet sich auf dem Deckglas des Mikroskops. Diese Teile werden dann im Deckel und Boden des Durchflusszellenhalters zusammengefügt. (B) Die komplette Durchflusszelle. In jede Seite des PDMS-Blocks können Röhrchen eingeführt werden, um die Kanäle zu bilden, durch die Puffer mittels Mikrofluidik durch das Gerät geleitet werden können. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 3: Trajektorien von Einzelmolekülen. (A) Beispiel für die Trajektorie eines Moleküls, das während des 10-minütigen Zeitrahmens des Experiments ein einzelnes Bindungsereignis durchläuft. (B) Beispiel für die Trajektorie eines Moleküls, das während des 10-minütigen Zeitrahmens des Experiments Null-Bindungsereignisse durchläuft. Die schwarze Linie in (A) und (B) stellt das an die Daten angepasste Hidden-Markov-Modell (HMM) dar. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 4: Verweilzeiten. (A) Verweilzeit von Pol δ auf dem G4-Substrat. Die Linie stellt die exponentielle Anpassung dar, die eine Lebensdauer von 6 s ± 2 s ergibt. (B) Verweilzeit von Pol δ auf dem Kontrollsubstrat. Die exponentielle Anpassung ergibt eine Lebensdauer von 10 s ± 3 s. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 5: Kontrolle des Photobleachings. Das Diagramm zeigt die durchschnittliche Zeit, die ein einzelnes AF647-markiertes Hefe-Pol-δ-Enzym benötigt, um mit dem 647-nm-Laser photogebleicht zu werden. Die Linie stellt die exponentielle Anpassung dar, was eine Lebensdauer von 39 ± 6 s ergibt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In dieser Arbeit wurde ein Einzelmolekül-Fluoreszenz-basierter Assay beschrieben, der einen Einblick in das Verhalten einer DNA-Polymerase gibt, wenn sie auf einen G-Quadruplex trifft. Während die Protokolle für die Generierung von DNA-Templates, die Pol-δ-Markierung und die Bulk-DNA-Replikationsassays alle unkompliziert sind, ist die Durchführung von Einzelmolekül-Mikroskopie-Assays eine größere technische Herausforderung. Aufgrund der Natur von Einzelmolekültechniken muss große Sorgfalt darauf verwendet werden, das Eindringen von Staub, Verunreinigungen oder Luftblasen zu vermeiden, da diese das Sichtfeld verdecken und die Datenerfassung behindern.
Eine Einschränkung von Einzelmolekül-TIRF-Mikroskopie-Experimenten ist das Photobleaching der Fluorophore, die kovalent an die interessierenden Biomoleküle gekoppelt sind. Photobleaching ist ein irreversibler Prozess, der zu einem dauerhaften Fluoreszenzverlust führt35. Um dies während der Experimente zu mildern, ist es wichtig, die Belichtungsdauer des Lasers zu begrenzen, die Laserintensität anzupassen und das Bildgebungstiming zu optimieren. Diese Strategien tragen dazu bei, Fluoreszenzsignale zu erhalten und sorgen für zuverlässigere und längere Beobachtungszeiträume. Durch die Feinabstimmung dieser Parameter bleibt das Pol δ Signal für die Dauer der Messung erhalten. Um die Laserleistung für die Einzelmolekül-Fluoreszenzmikroskopie-Synthese-Assays zu optimieren, empfiehlt es sich, die Photobleaching-Rate zu messen, indem die markierte Polymerase auf einem sauberen Glasdeckglas abgebildet wird. Durch systematische Variation der Laserleistung und Bewertung der Photobleichrate über das gesamte Sichtfeld kann die optimale Laserintensität identifiziert werden, die die Signalstärke des Fluorophors mit der Beständigkeit gegen Photobleichung in Einklang bringt (siehe Ergänzende Abbildung 5).
Der Hauptvorteil dieses Einzelmolekül-Ansatzes gegenüber herkömmlichen Ensemble-basierten Methoden ist seine Fähigkeit, direkt zu visualisieren, wann eine einzelne DNA-Polymerase auf eine G4-Struktur trifft und mit ihr interagiert. Traditionelle Ensemble-basierte Methoden (wie z. B. die Gelelektrophorese) haben die Fähigkeit von G4-Strukturen gezeigt, DNA-Polymerasen zu blockieren 23,36,37. Diese Techniken liefern jedoch nicht die kinetischen und mechanistischen Echtzeitinformationen dieser Wechselwirkung, die notwendig sind, um verschiedene molekulare kinetische Schritte und Ergebnisse zu entwirren. Einzelmolekültechniken bieten einen beispiellosen Einblick in die Kinetik, Mechanismen und Verhaltensweisen von Biomolekülen, die oft durch die Ensemble-Mittelungverborgen werden 38. Es ist nun möglich zu sehen, wie DNA-Polymerasen in Echtzeit agieren - ob sie DNA-Hindernisse austauschen, blockieren, dissoziieren oder umgehen39. Wenn dieses Protokoll etabliert ist, kann die Identität des G4 leicht von der gewählten parallelen c-MYC-Struktur in eine parallele, antiparallele oder hybride Topologie geändert werden. Die Anwendung dieses Einzelmolekül-Assays wird zeigen, ob sich dieselben DNA-Polymerasen unterschiedlich verhalten, wenn sie auf alternative G4-Topologien treffen. Daher sind Einzelmolekülmethoden von entscheidender Bedeutung, um spezifische Fragen zum Zusammenspiel von Proteinen und DNA des Körpers zu beantworten.
Durch die direkte Visualisierung von DNA-Polymerase-Wechselwirkungen mit G-Quadruplexen wurde ein bisher nicht charakterisierter Austauschweg für Hefe Pol δ identifiziert. Diese Entdeckung deutet darauf hin, dass sich die Polymerase löst, wenn sie auf einen G-Quadruplex trifft, und auf den Eingriff eines anderen Proteins wartet, um die Struktur aufzulösen, bevor sie die DNA-Synthese wieder aufnimmt. Dieses Protokoll kann angepasst werden, um Wechselwirkungen zwischen verschiedenen Genomerhaltungsproteinen und DNA-Hindernissen zu untersuchen, und bietet beispiellose Einblicke in die Art und Weise, wie zelluläre Enzyme genomische Hindernisse überwinden. Zum Beispiel kann die Blockade dieses Assays von einer G4-Struktur zu einer Protein-DNA-Vernetzung verändert werden, einer Art von DNA-Läsion, bei der ein Protein irreversibel kovalent an die DNA gebunden ist, was als Hindernis für die DNA-Replikation wirkt40. Solche Untersuchungen sind entscheidend für das Verständnis der grundlegenden Prozesse der DNA-Replikation, -Reparatur und -Rekombination. Durch die Untersuchung der DNA-Protein-Dynamik auf molekularer Ebene bietet dieser Assay ein leistungsfähiges Werkzeug zur Aufklärung der Mechanismen, die der genomischen Integrität zugrunde liegen.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
N.K.-A. würdigt die Finanzierung durch das Stipendium des Australian Government Research Training Program. L.M.S. ist dankbar für die Finanzierung, die sie vom National Health and Medical Research Council (Investigator Grant 2007778) erhalten hat. J.S.L ist dankbar, dass sie mit dem Discovery Early Career Award (DE240100780) und dem NHMRC Investigator EL1 (2025412) ausgezeichnet wurde, der von der australischen Regierung finanziert wird. S.H.M ist dankbar, Empfänger des Bruce Warren Molecular Horizons Ealy Career Fellowship zu sein.
Materialien
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
Referenzen
- Sutton, M. D. Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci U. S. A. 98 (15), 8342-8349 (2001).
- Garg, P. Burgers, P. M. J. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M.-D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S. et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O. et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Kawasaki, Y. Sugino, A. Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Wood, R. D. Shivji, M. K. Which DNA polymerases are used for DNA repair in eukaryotes? Carcinogenesis. 18 (4), 605-610 (1997).
- Budd, M. E. Campbell, J. L. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Yang, W. Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9, 968424 (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Loeb, L. A. Monnat, R. J. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Oganesian, L. Bryan, T. M. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S. et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), 1579 (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I. et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Kaguni, L. S. Clayton, D. A. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), eabf8653 (2021).
- Castillo Bosch, P. et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, John A., Zakian, Virginia A. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L. et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh.). 11 (9), e2307696 (2024).
- Pham, S. Q. T. et al. A new class of quadruplex DNA-binding nickel Schiff base complexes. Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E. et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Au - Desjardins, P., Au - Hansen, J. B., Au - Allen, M. Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, e1610 (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, e1485 (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J.) 219-238, Springer New York, New York, NY (2015).
- Lewis, J. S. et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, e23932 (2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. 204, 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664 (2014).
- Mueller, S. H. et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5 (2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten